1.本发明公开了一种马氏珠母贝珍珠珍珠层厚度性状基因组选择育种方法,属于分子标记辅助育种技术领域。
背景技术:
2.马氏珠母贝pinctada fucata martensii是我国培育海水有核珍珠的主要贝类。在我国海水珍珠产量持续下行的阶段,优良品种培育是应对产业难题的有效途径之一。传统育种方法与基因分型技术相结合能提高性状选择的准确性与精确性,更重要的是大大缩短了育种时间,已成为当前育种的主流方向。培育适合当地海区养殖新品种,改良养殖群体的生长与育珠性状,这是解决产业困境的首要任务。
3.目前,已有研究者通过连锁分析和关联分析对软体动物的珍珠性状进行了定位。利用构建的马氏珠母贝遗传连锁图谱,结合连锁与关联分析定位到1个qtl与壳珍珠层厚度相关。利用est
‑
ssr标记对马氏珠母贝珍珠层表型进行关联分析,发现微卫星标记pmarte64与珍珠层厚度性状相关。马氏珠母贝p.f.martensii珍珠珍珠层厚度性状和珍珠质层厚度与prismalin
‑
14、n19和msi60矿化基因snps多态性相关,其中在3个家系中prismalin
‑
14基因的snp位点与贝壳珍珠质层厚度呈显著相关。
4.尽管马氏珠母贝传统育种取得了较大进展,但大都基于对表型性状的选择。表型性状由基因与环境共同决定,环境干扰常常影响选择效果,降低选择的准确性。对于珍珠性状的选育,目前多见于利用对珍珠珍珠层厚度性状的选择从而实现对珍珠性状的间接选择。马氏珠母贝珍珠性状属于数量性状,由多基因控制,本发明直接对珍珠珍珠层厚度性状进行测量及分析,则不存在该问题。另外利用基因组范围内的标记进行表型选择可加大对珍珠性状的准确性,缩短选择时间,是进行良种培育的一种新的育种技术。
技术实现要素:
5.本发明的目的在于克服上述现有技术的不足,提供了一种基于全基因组选择的马氏珠母贝珍珠珍珠层厚度优良性状培育方法,以改善传统选育手段中存在的准确性低、进展缓慢的问题,为培育珍珠珍珠层性状优异的马氏珠母贝提供分子育种方法,推动马氏珠母贝珍珠性状选育的快速发展,保障马氏珠母贝海水珍珠养殖产业平稳、健康发展。
6.为了实现上述目的,本发明采用的技术方案是:
7.一种基于全基因组选择的马氏珠母贝珍珠珍珠层厚度优良性状培育方法,所述培育方法包括以下步骤:
8.步骤一,马氏珠母贝珍珠珍珠层厚度性状测量及参考群体构建;
9.步骤二,对马氏珠母贝珍珠珍珠层厚度性状参考群体进行基因组重测序和全基因组snp标记鉴定;
10.步骤三,计算每个snp标记的效应值,对验证群进行育种值估计;结合验证群的表
型值,计算其相关性及回归系数,选出最佳的预测计算模型;
11.步骤四,利用步骤三选择的最佳预测模型用于育种群育种值估计,并用于最终的选种。
12.进一步地,所述步骤一中,利用oct系统对马氏珠母贝珍珠进行珍珠层厚度性状测量,依据珍珠的珍珠层厚度性状构建参考群体,用于后续的分析、估算。
13.进一步地,所述步骤二中,利用基因组重测序方法,测序深度大于10
×
,进行snp位点标记鉴定,获得全基因组范围内超高密度snp位点。首先对参考群体的基因组dna并建库、测序,对下机的原始数据进行过滤,再将过滤后的reads比对至马氏珠母贝参考基因组上,之后利用gatk的unified genotyper检测snp信息,并进行过滤获得高可信度的snp数据集用于基因组选择的计算。
14.进一步地,所述步骤三中,利用参考群的表型值和基因型信息,加入到基于rrblup、bayesa和bayesb分析方法的基因组预测模型,计算每个snp标记的育种效应值,将rrblup、bayesa和bayesb作为基因组选择方法的代表,对三种基因组选择方法的预测准确性进行评估,利用验证群全基因组snp分型结果和snp的效应值,计算验证群每个个体的育种值;根据验证群的育种值及表型值,计算两者的相关性及回归系数,筛选其中最佳的基因组预测模型。
15.进一步地,所述步骤四具体为选用步骤三中确定的预测准确性最高的基因组选择方法,预测育种群体中每个个体的育种值,并根据基因组育种值从高到低进行个体选择。
16.本发明的有益效果是:本发明的方法预测准确率高,可在个体幼年时期完成选择过程,降低生产成本,缩短选育进程。本发明改善了传统选育手段中存在的准确性低、进展缓慢的问题,为培育珍珠珍珠层性状优异的马氏珠母贝提供分子育种方法,推动马氏珠母贝珍珠性状选育的快速发展,保障马氏珠母贝珍珠养殖业平稳、健康发展。
附图说明
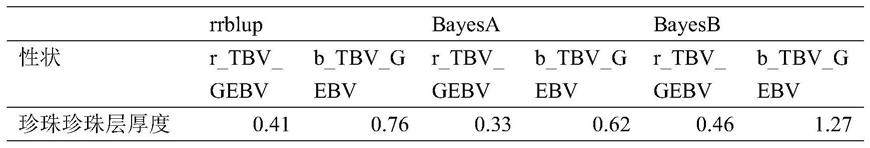
17.图1是rrblub、bayesa和bayesb预测准确性表;
18.其中:r_tbv_gebv:gebv与真实育种值(tbv)间的相关系数,代表其准确性,其平方称为可靠性;b_tbv_gebv:tbv对gebv的回归系数,代表其无偏性,若b_tbv_gebv=1表明无偏,否则有偏。
具体实施方式
19.下面结合实施例对本发明作进一步的详细说明,一种基于全基因组选择的马氏珠母贝珍珠珍珠层厚度优良性状培育方法,该方法包括以下步骤。
20.一、马氏珠母贝珍珠珍珠层厚度性状测量及参考群体构建。
21.选取马氏珠母贝“海选1号”新品种作为研究材料,利用oct系统进行珍珠的珍珠层厚度性状测量。
22.二、参考群体全基因组重测序、基因型采集及分析处理。对马氏珠母贝珍珠珍珠层厚度性状参考群体进行基因组重测序和全基因组snp标记鉴定。对参考群体进行基因组dna提取并建库测序。包括以下具体操作方法:
23.1)基因组dna提取
24.使用tiangen的海洋动物组织基因组dna提取试剂盒提取基因组dna,具体步骤包括:
25.(1)取0.05g闭壳肌,放入装有200μl ga缓冲液的高压灭菌过的1.5ml离心管;
26.(2)剪碎肌肉组织,加入4μl核糖核酸酶a(100mg/ml)溶液,振荡15sec,室温放置5min;
27.(3)加入20μl蛋白酶k(20mg/ml)溶液,涡旋混匀,简短离心,56℃放置3h,每小时振荡混合样品2
‑
3次,每次振荡混匀15sec;
28.(4)加入200μl缓冲液,充分颠倒混匀,70℃放置10min,简短离心;
29.(5)加人200μl无水乙醇,充分颠倒混匀,简短离心;
30.(6)将上一步所得溶液和絮状沉淀都加入一个吸附柱中(吸附柱放入收集管中),12,000rpm/min离心30sec,倒掉废液,将吸附柱放回收集管中;
31.(7)向吸附柱中加入500μl缓冲液,12,000rpm/min离心30sec,倒掉废液,将吸附柱放入收集管中;
32.(8)向吸附柱中加入600μl漂洗液,12,000rpm/min离心30sec,倒掉废液,将吸附柱放入收集管中;
33.(9)重复操作步骤7;
34.(10)将吸附柱放回收集管中,12,000rpm/min离心2min,倒掉废液。将吸附柱置于室温放置10分钟;
35.(11)将吸附柱转入一个干净的离心管中,向吸附膜的中间部位悬空滴加100μl洗脱缓冲液,室温放置5min,12,000rpm/min离心2min,将溶液收集到离心管中。
36.2)snp分子标记的检测
37.利用超声波高性能样品处理系统covaris对检测合格的dna样品进行随机打断,纯化后连接测序接头,桥式pcr制备cluster,利用illumina hiseqtm 2000平台进行测序,获得测序原始数据。为保证测序数据质量,在信息分析前通过分析碱基组成和质量分布对原始数据进行质控,之后对利用soapnukel软件对下机原始数据(raw data)进行过滤,获得有效数据(clean data)。测序原始数据经过质控和数据过滤后,应用bwa比对将clean data比对到参考基因组上。统计分析参考基因组碱基的测序深度分布和各染色体区域的覆盖度。应用samtools,reseqtools和picard
‑
tools对比对结果进行统计,预处理(排序,去重复、加id等)。之后应用gatk的unified genotyper检测snp信息。之后基于比对后获得的样本的所有snp信息,将所测基因型与参考序列之间有多态性的位点进行过滤,得到高可信度的snp数据集。
38.三、计算每个snp标记的效应值,对验证群进行育种值估计;结合验证群的表型值,计算其相关性及回归系数,选出最佳的预测计算模型。
39.根据参考群snp数据集及表型值,利用rrblup、bayesa和bayesb三种方法估算snp的效应值。利用步骤二中方法获得验证群的snp数据集,并利用snp效应值及三种计算模型计算验证群的育种值,并根据育种值与表型值的相关性及回归系数分析三种方法的准确性。经比较,bayesb方法获得的育种值与表型值直接的相关系数均大于bayesa和rrblub方法,如图1所示,因此最终选定bayesb方法计算基因组估计育种值gebv。
40.四、利用步骤三选择的最佳预测模型用于育种群育种值估计,并用于最终的选种。
41.选用步骤三中确定的预测准确性最高的方法,估算育种群体中每个个体的育种值,并根据基因组育种值从高到低进行个体选择,可根据个体育种值的高低为留种及育种方案定制提供参考。
42.以上内容仅用以说明本发明的技术方案,本领域的普通技术人员对本发明的技术方案进行的简单修改或者等同替换,均不脱离本发明技术方案的实质和范围。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。