1.本发明属于生物技术领域,具体涉及一种海水样本宏基因组建库检测技术。
背景技术:
2.海水样本宏基因组建库主要是针对海水中的微生物,海水微生物是生物活性物质的重要源泉。通常在海水微生物采样时最常用的方式是过滤,需要用不同孔径的滤膜对海水进行多次过滤,通过过滤大量海水得到更完整的微生物群落组成,进而提取海水微生物dna。但海水微生物含有大量的无机盐离子、无机化合物、有机化合物、重金属离子等干扰提取反应进行的杂质。且不同水域的海水微生物种类不同,这都为从海水微生物群中提取dna带来了困难。
3.对于海水样本宏基因组建库目前主要存在以下问题:(1)过滤时滤膜的孔径较小,不能满足高流量和高通量的要求;(2)大量海水的过滤加上孔径较小使滤膜容易堵塞,降低了工作效率;(3)海水至少过滤两次,增加工作量的同时费时费力;(4)构建文库步骤繁琐,时间长。
4.中国专利201610706216.9公开了一种海水微生物宏基因组的提取方法。针对海水这一特殊的样本给予去盐离子处理、双重滤膜过滤再回收处理相结合,旨在提供一种高效、快速的提取海水微生物宏基因组的方法,具体包括以下几个步骤:海水中杂质的去除、海水微生物的裂解、dna的游离与吸附、dna洗脱。能够有效的去除海水中较多的盐离子的干扰;充分裂解海水中的微生物细胞,使dna充分释放。该方法提取的dna,其a260/280比值维持在1.8
‑
2.0之间,所得dna质量好、纯度高;能够满足基因文库构建和qpcr以及高通量测序的需求,为研究微生物基因组遗传变异多样性提供了良好的基础。但双重滤膜过滤效率较低。
5.中国专利201410799477.0中公开了一种基于宏基因技术的石油污染海洋生态环境评价方法,该方法包括步骤:采集海水样品;对样品进行宏基因组dna的提取并测序;对测序数据进行有效序列数据统计分析和优化序列数据统计分析;使用mothur及chopseq软件进行otu
‑
based分析;进行alpha
‑
diversity分析,该分析过程包括对菌群丰度、菌群多样性、测序覆盖率进行分析;进行稀释性曲线分析;进行分类学分析和群落结构分析;进行heatmap聚类分析,获得样品微生物区系结构和种群数量参数值,用于评价不同样品中污染相似度及危害性。该方法可客观评价溢油污染物对海洋生态系统的损害程度以及生态系统对溢油污染物的响应变化。但其仅采用离心富集海水中微生物,精度较低。
技术实现要素:
6.为了解决上述问题,本发明提供了一种海水样本宏基因组建库检测技术,减少海水样本采样量,提高工作效率,改进的过滤方法也能得到更完整的微生物群落,同时在构建文库的方法上进行了改进,从而更符合宏基因组测序标准。
7.一方面,本发明提供了一种海水样本宏基因组建库检测技术。
8.具体地,所述的海水样本宏基因组建库检测技术中包括:滤膜的前处理、过滤步
骤、tn5转座子构建文库。
9.具体地,所述的滤膜的前处理为使滤膜带有正电荷。
10.进一步具体地,所述的滤膜的前处理为使用含有壳聚糖的处理液处理滤膜。
11.所述的滤膜的前处理包括但不限于:使滤膜浸泡在含有壳聚糖的处理液中;或者将含有壳聚糖的处理液喷涂到滤膜表面。
12.所述的含有壳聚糖的处理液中壳聚糖的浓度为:1.5mg/ml
‑
2.5mg/ml,优选为2mg/ml。
13.具体地,所述的过滤步骤为:使样本通过过滤装置。
14.进一步具体地,所述的过滤装置带有紧贴于滤膜的搅拌装置,从而减少堵塞,加速过滤。
15.优选地,所述的tn5转座子构建文库的方法为:利用fireseq
‑
转座法dna建库试剂盒进行文库的构建。
16.在一些实施例中,所述的海水样本宏基因组建库检测技术包括以下步骤:
17.(1)用含有壳聚糖的处理液处理滤膜,清除多余处理液后烘干;
18.(2)水样抽滤,抽滤完成后用裂解液覆盖滤器,冻存;
19.(3)步骤(2)样品解冻后,添加溶菌酶;
20.(4)添加蛋白酶k、十二烷基硫酸钠后封闭孵育;
21.(5)萃取;
22.(6)超滤收集水相,提取dna;
23.(7)tn5转座子构建文库;
24.(8)上机测序。
25.所述的步骤(2)中的裂解液中的成分及用量为:50mm tris
‑
hcl,40mm edta,0.75m蔗糖。
26.所述的步骤(5)中的萃取包括两种萃取液,第一种萃取液成分及比例为苯酚:氯仿:异戊醇(25:24:1);第二种萃取液的成分及比例为氯仿:异戊醇(24:1)。
27.所述的步骤(8)中包括:dna与流动槽的附着,桥式pcr,解链切除,测序。
28.另一方面,本发明提供了前述的海水样本宏基因组建库检测技术在制备基于宏基因组的检测试剂和/或试剂盒中的应用。
29.又一方面,本发明提供了一种基于宏基因组学的检测试剂盒。
30.所述的基于宏基因组学的检测试剂盒包括但不限于:gennature library preparation kit、collibri ps dna library prep kit、fireseqtm tn5 dna library prep kit for illumina。
31.具体地,所述的试剂盒中包括前述的海水样本宏基因组建库检测技术中所使用的检测试剂。
32.优选地,所述的试剂盒中包括文库接头。
33.所述的文库接头选自p5’(seq id no.1)、p7’(seq id no.2)、p5(seq id no.3)、p7’(seq id no.4)。
34.所述的试剂盒中还包括但不限于:dna聚合酶、甲酰胺基嘧啶糖苷酶、蛋白酶k、溶菌酶、裂解液、苯酚、氯仿、异戊醇、dntp、ddntp。
35.本发明的有益效果:
36.1、本发明用壳聚糖作为处理液处理滤膜,从而使滤膜带有正电荷,通过正负电荷之间的相互吸附,在不影响其他微生物吸附效果的同时富集更多带有负电荷的微生物。
37.2、本发明在过滤装置中使用带有紧贴于滤膜的搅拌装置,从而减少堵塞,加速过滤。
38.3、本发明可以吸附比滤膜孔径小的带负电荷的微生物,从而降低对孔径的要求,适当提高流速和流量。
39.4、本发明通过tn5转座子构建文库,省去了片段化、末端修复、加da尾、接头连接及多步纯化过程,极大缩短建库时间,提高工作效率。
40.5、本发明的一种基于宏基因组学的检测试剂盒,根据上述建库方法配置试剂,可适用于不同类型样本,简化了使用操作步骤,降低了对使用人员的要求,具有检测准确度高和便捷的优势。
具体实施方式
41.下面结合具体实施例,对本发明作进一步详细的阐述,下述实施例不用于限制本发明,仅用于说明本发明。以下实施例中所使用的实验方法如无特殊说明,实施例中未注明具体条件的实验方法,通常按照常规条件,下述实施例中所使用的材料、试剂等,如无特殊说明,均可从商业途径得到。
42.实施例1一种海水样本宏基因组建库检测技术
43.按以下步骤操作:
44.1、用孔径0.45μm混合纤维素酯滤膜为基膜,浸泡在含有浓度2.0mg/ml壳聚糖的处理液中(此步亦可替换为用含有壳聚糖的处理液喷涂到膜表面),从而使滤膜吸附饱和的处理液。
45.2、取出滤膜通过按压、刮除的方法清理膜表面多余处理液。
46.3、将滤膜在温度为70℃的烘箱中烘干,得产品待用。
47.4、水样抽滤,抽滤完成后用裂解液(50mm tris
‑
hcl,40mm edta,0.75m蔗糖)覆盖steripak
‑
gp20滤器,并置于
‑
80℃冻存待用。
48.5、解冻后,将5mg/ml溶菌酶(购自晓柚生物,货号为x11378)溶于3ml裂解液中,并添加到steripak
‑
gp20滤器中,并在37℃下处理30分钟。
49.6、在无菌水中添加蛋白酶k(终浓度为0.5mg/ml,蛋白酶k购自sigma,货号为p6556)注入steripak
‑
gp20滤器,然后添加sds(十二烷基硫酸钠,sigma)至最终浓度为1%。
50.7、封闭滤筒,55℃孵育20min,70℃孵育5min,进一步促进细胞裂解。
51.8、将裂解液从滤盒中取出,用苯酚:氯仿:异戊醇(25:24:1)萃取2次,氯仿:异戊醇(24:1)萃取1次。
52.9、使用centricon 100过滤器(millipore),超滤收集水相。提取dna(2mg)用于构建文库。
53.10、利用fireseq
‑
转座法dna建库试剂盒(购自fireseq,货号为fg0812
‑
m)构建文库,步骤如下:片段化(55℃,10min),纯化(30min),文库扩增(5
‑
15个循环,约20min),分选(30min)。
54.11、上机测序:
55.接头序列包括p5’:seq id no.1;p7’:seq id no.2;p5:seq id no.3;p7’:seq id no.4。
56.dna与流动槽(flow cell)的附着:流动槽是一种含有8条泳道(lane)的芯片,每条lane的表面固定有很多(p5’/p7)接头(引物)。测序时利用微注射系统将已经加过(p5/p7)接头的ssdna片段随机添加到流动槽内。ssdna片段的p5接头序列与芯片表面的p5’引物互补,一端被“固定”在芯片上。
57.桥式pcr(bridge pcr):向流动槽中添加未标记的dntp和dna聚合酶(购自西典实验,货号为kk1006),以ssdna片段为模板合成出一条全新的dna链(p5
’‑
p7’互补链),然后再加入naoh溶液,使dna双链解链,由于模板链没有结合在lane上,模板链会被溶液流洗脱,但互补链仍固定在lane上。加入中性缓冲溶液,环境变成中性后,互补链的p7’与lane上p7接头互补杂交,形成桥型ssdna,接下来加入dntp和dna聚合酶,聚合酶就沿着p7接头合成出一条新的链(桥型ssdna扩增为桥型dsdna)。再加入naoh碱溶液,使dna双链解链,然后再加中和液,继续扩增,经过不断的扩增和变性循环(大约35个循环)后,最终每种ssdna都将在各自的位置上集中成束(cluster),每一束都含有单个dna模板的500
‑
1000个拷贝,从而达到测序所需信号强度的模板量。
58.桥式pcr完成之后,再次强碱解链,采用甲酰胺基嘧啶糖苷酶(糖基化酶,购自盖德化工,货号为btn130644)选择性的切掉lane上p5’连接的链,只留下了与lane p7连接的链
–
forward strand。同时,添加ddntp阻断延伸。形成cluster测序单元,可直接用于边合成边测序反应。
59.测序:向反应体系中添加dna聚合酶、接头引物(p5’/p7’)和带有荧光标记的4种dntp(可逆终止子:dntp的3’羟基被叠氮基团替代,只容许每次添加一个dntp)。dntp被添加到合成链上之后,将剩余游离dntp和dna聚合酶洗脱。然后加入激发荧光所需的缓冲液,用激光激发荧光信号,光学设备记录荧光信号,计算机分析并将其转化为测序结果。再加入化学试剂猝灭荧光信号,并使dntp的3’叠氮基团变成羟基,继续进行下一轮测序反应。
60.本实施例中用于检测的水样采自山东省烟台市养马岛,东经121.6度;北纬37.5度。
61.检测结果如下:
[0062][0063]
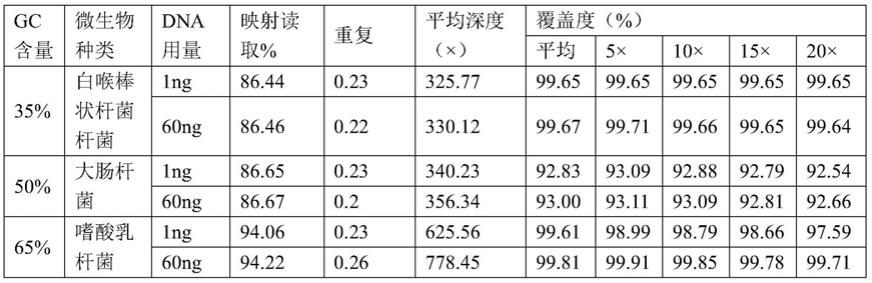
对不同gc含量的微生物gdna样本建库,并使用hiseq 4000测序,结果显示:无论样本起始量及gc含量,所建文库均获得碱基质量好且覆盖度均一的测序数据。
[0064]
对比例
[0065]
参照实施例1设置对比例,具体如下:
[0066] 与实施例1的区别之处对比例1处理液中壳聚糖浓度为1.3mg/ml。对比例2处理液中壳聚糖浓度为2.7mg/ml。
[0067]
对比例1的效果与实施例1相比较:微生物的丰度明显低于实施例1;
[0068]
对比例2的效果与实施例1相比较:微生物的丰度明显低于实施例1。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。