1.本发明涉及生物医药领域,具体涉及一种生物标志物及应用了该生物标志物进行肝内胆管细胞癌预后预测的模型、产品及系统。
背景技术:
2.肝内胆管癌(intrahepatic cholangiocarcinoma,icc)是肝脏自身产生的第二常见的恶性肿瘤,占原发性肝癌的4.8~12.0%,且预后较差。在过去的几十年中,肝内胆管癌的发病率和死亡率持续上升。完全的手术切除是患有可切除的肝内胆管癌患者长期生存的唯一选择,且部分患者在术后5年的总生存率(overall survival,os)约为25.0~39.8%。
3.肝内胆管癌的高度侵略性的生物学行为以及缺乏特定的症状和体征的特点,使得大多数患者在初次诊断时即表现出相对晚期的疾病,因而只有少数的患者有机会进行手术切除。然而,治愈后的高复发率将导致预后不良,即使进行了根治性切除,仍然会有57.9~73.4%的患者复发,而41.3~42.5%的复发患者会死于复发。因此,确定根治性切除术后死亡或复发风险高的患者,并针对性地探索适当的辅助治疗策略至关重要。
[0004]5’
甲基胞嘧啶(5
‑
mc)中异常的dna甲基化与肝内胆管细胞癌的发生和发展过程相关。但是,现有的关于甲基化与肝内胆管癌的研究主要集中于单个或多个常见的肿瘤抑制基因的甲基化水平上,例如cdh1、socs3、p15、hmlh1、apc、arideaopcml等,对其余的基因,以及基因上特定的基因区域的甲基化水平在预测肝内胆管细胞癌的长期预后方面的作用知之甚少。
技术实现要素:
[0005]
本发明一个目的在于提供一种生物标志物,所述生物标志物涉及1至17种基因区域,所述基因区域对应于1至18种基因,其中,基因区域可以是增强子、启动子、外显子、内含子、5’非翻译区和3’非翻译区中的一种。通过检测上述生物标志物的甲基化水平,能够基于各基因区域的甲基化水平计算得到基因组甲基化评分(genomic methylation score,gms),并通过基因组甲基化评分将进行肝内胆管癌预后。
[0006]
上述目的通过下述技术方案实现:
[0007]
一种生物标志物,所述生物标志物选自lix1l、linc01822、tuba4a、ac023481.1、mfsd1、ac097173.1、fam153b、steap2/steap2
‑
as1、cdkn2b、fam205bp、gad2、letmd1、rassf3、ac073592.4、golga6b、orc6、tmem106a中的至少一种基因的基因区域,所述基因区域为基因的增强子、启动子、外显子、内含子、5’非翻译区或3’非翻译区。
[0008]
传统的关于肝内胆管细胞癌dna甲基化的研究主要集中于数个基因或者450k芯片结果,样本量小,无法完整地反映出基因上特定的基因区域的甲基化水平对肝内胆管癌的作用和影响。
[0009]
本技术中,对选自四川大学华西医院、复旦大学中山医院和天津医科大学肿瘤医院的患者采用全基因组dna甲基化测序(whole genome bisulfite sequencing,wgbs),在
整个基因组范围内描述了肝内胆管细胞癌整体的表观遗传学改变,并系统地评估了各基因区域的甲基化水平的预后价值,基于甲基化水平的预后价值不同,构建了基因组甲基化评分以预测患者的预后。
[0010]
具体地,本技术方案中,通过wgbs检测基因区域甲基化水平,确定了1606362个基因区域,对应于61076个蛋白质编码基因和非编码基因。接下来,去除甲基化水平为0的和完全重叠的基因区域,以及单变量cox分析、c指数计算和cv计算,确定了350个符合要求的基因区域。最后采用lasso cox算法,选择了对应于18种基因的17个基因区域来构建基因组甲基化评分。上述17个基因区域分别对应于基因lix1l、linc01822、tuba4a、ac023481.1、mfsd1、ac097173.1、fam153b、steap2/steap2
‑
as1、cdkn2b、fam205bp、gad2、letmd1、rassf3、ac073592.4、golga6b、orc6、tmem106a中的增强子、启动子、外显子、内含子、5’非翻译区或3’非翻译区。
[0011]
在部分实施例中,基因组甲基化评分基于上述全部17个基因区域及对应的权重系数进行计算。在一个或多个实施例中,所述基因组甲基化评分也可以基于部分权重系数较高的基因区域进行计算,例如,基于基因lix1l的第1个外显子的甲基化分数,又例如,基因orc6的第1个内含子的甲基化分数。
[0012]
前述选定的基因区域所对应的18种基因中,包括12个蛋白质编码基因、3个lncrna基因、2个假基因和1个待实验确认基因(to be experimentally confirmed,tec)。
[0013]
其中,cdkn2b作为编码p15蛋白的基因,是一种常见的抑癌基因(tumour suppressor gene,tsg),cdkn2b的启动子的甲基化及其预后价值已在其他的多种癌症中得到了验证。letmd1是人类宫颈癌癌基因,不仅能够通过稳定p53促进细胞的增殖和存活,而且还通过负调控巨噬细胞功能参与到肿瘤微环境的调节。基因rassf3也是一种tsg,其可通过稳定p53诱导凋亡和g1
‑
s阻滞,基因rassf3的下调能够促进肺癌的恶性表型。基因orc6在功能性人类orc的靶向、定位和组装过程中发挥着重要的作用,因而参与了dna复制和细胞周期。lix1l是一种新型的间充质基因,与多种癌症的emt指数相关,此外,lix1l在多种肿瘤样品中高表达,且体外实验中lix1l的敲低能够阻碍癌细胞的增殖、侵袭和迁移能力。基因steap2的上调能够抑制恶性表型乳腺癌细胞,steap2或steap2
‑
as1的预后价值在多种癌症中已得到验证。基因tmem106a除了在癌症过程中抑制emt和pi3k/akt/nf
‑
κb通路,还通过mapk和nf
‑
κb信号通路维持巨噬细胞稳态以调节肿瘤微环境,类似的结果也在基因gad2和tuba4a中存在。
[0014]
除了上述基因外,传统被认定为无功能的背景噪音或垃圾基因的长非编码rna(lncrna)和假基因也逐渐受到人们关注。本技术方案中,基因组甲基化评分的构建采用了3种lncrna和2种假基因,这5种基因中,除了steap2
‑
as1外,均未见报道与肿瘤的发生或进展有关。另外,其余的编码基因与tec基因也未证实与肿瘤的发生或进展相关。
[0015]
由此可见,本技术筛选获得的基因区域中,一部分基因区域对应的基因已被验证与肿瘤生物学相关,并具有能够作为癌症的预后生物标志物,虽然其未公开本技术中基因上与肝内胆管癌预后密切相关的基因区域,但一定程度上证明了本技术中的基因组甲基化评分的可靠性;而另一部分基因区域及其对应的基因通过本技术证明其在肝内胆管细胞癌,甚至其他类型的癌症中具有重要的生物学功能。
[0016]
进一步地,所述ac097173.1、steap2/steap2
‑
as1、rassf3、ac073592.4、golga6b的
基因区域为增强子,所述mfsd1、fam153b、gad2、orc6的基因区域为内含子,所述lix1l、tuba4a的基因区域为外显子,所述linc01822的基因区域为启动子,所述tmem106a的基因区域为5’非翻译区,所述ac023481.1、cdkn2b、fam205bp的基因区域为5’非翻译区或外显子,所述letmd1的基因区域为3’非翻译区或外显子。现有技术中,虽然部分编码基因已被证实为抑癌基因,例如cdkn2b,但关于其基因区域的甲基化及预后价值的研究主要集中于该基因的启动子。本技术方案中,cdkn2b的基因区域优选为5’非翻译区或外显子。同时,发明人在实验中发现,在350个满足符合要求的基因区域中,增强子的占比明显高于其他几类基因区域,且5’非翻译区和3’非翻译区的占比也高于启动子。最终,通过lasso cox算法选定的17个基因区域中,大部分基因区域为增强子、外显子、内含子、5’非翻译区和3’非翻译区,启动子的数量只有1个。
[0017]
进一步地,所述lix1l的基因区域为chr1:145957635
‑
145958017,linc01822的基因区域为chr2:21710623
‑
21712623,tuba4a的基因区域为chr2:219251586
‑
219251713,ac023481.1的基因区域为chr3:8365593
‑
8365799,mfsd1的基因区域为chr3:158804371
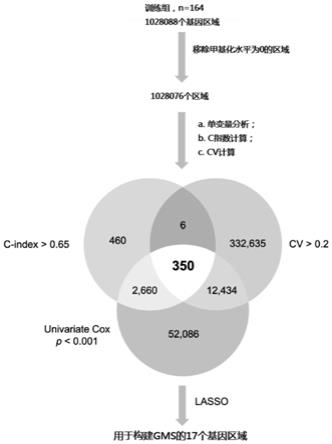
‑
158805340,ac097173.1的基因区域为chr4:119872612
‑
119873113,fam153b的基因区域为chr5:176114061
‑
176115592,steap2/steap2
‑
as1的基因区域为chr7:90178863
‑
90179364,cdkn2b的基因区域为chr9:22005202
‑
22006271,fam205bp的基因区域为chr9:34838349
‑
34838586,gad2的基因区域为chr10:26223977
‑
26224538,letmd1的基因区域为chr12:51056370
‑
51056502,rassf3的基因区域为chr12:64671189
‑
64671690,ac073592.4的基因区域为chr12:124626183
‑
124626684,golga6b的基因区域为chr15:72237361
‑
72237862,orc6的基因区域为chr16:46689829
‑
46690990,tmem106a的基因区域为chr17:43213020
‑
43213041。
[0018]
本技术方案中,lix1l的基因区域为其第1个外显子,对应的区域为1号染色体上第145957635个至第145958017个碱基;linc01822的基因区域为启动子,对应的区域为2号染色体上第21710623个至第21712623个碱基;tuba4a的基因区域为其第3个外显子,对应的区域为2号染色体上第219251586个至第219251713个碱基;ac023481.1的基因区域为其第2个外显子或5’非翻译区,对应的区域为3号染色体上第8365593个至第8365799个碱基;mfsd1的基因区域为第2个内含子,对应的区域为3号染色体上第158804371个至第158805340个碱基;ac097173.1的基因区域为增强子,对应的区域为4号染色体上第119872612个至第119873113个碱基;fam153b的基因区域为第22个内含子,对应的区域为5号染色体上第176114061个至第176115592个碱基;steap2/steap2
‑
as1的基因区域为增强子,对应的区域为7号染色体上第90178863个至第90179364个碱基;cdkn2b的基因区域为第1个外显子或5’非翻译区,对应的区域为9号染色体上第22005202个至第22006271个碱基;fam205bp的基因区域为第1个外显子或5’非翻译区,对应的区域为9号染色体上第34838349个至第34838586个碱基;gad2的基因区域为第5个内含子,对应的区域为10号染色体上第26223977个至第26224538个碱基;letmd1的基因区域为第1个外显子或3’非翻译区,对应的区域为12号染色体上第51056370个至第51056502个碱基;rassf3的基因区域为增强子,对应的区域为12号染色体上第64671189个至第64671690个碱基;ac073592.4的基因区域为增强子,对应的区域为12号染色体上第124626183个至第124626684个碱基;golga6b的基因区域为增强子,对应的区域为15号染色体上第72237361个至第72237862个碱基;orc6的基因区域为第1个内
含子,对应的区域为16号染色体上第46689829个至第46690990个碱基;tmem106a的基因区域为5’非翻译区,对应的区域为17号染色体上第43213020个至第43213041个碱基。上述基因区域的位置明确且一定。基于上述基因区域的甲基化水平检测结果,能够计算基因组甲基化评分,用于预测患者的肝内胆管细胞癌的预后。
[0019]
进一步地,所述生物标志物包括lix1l、ac097173.1、steap2/steap2
‑
as1、cdkn2b、orc6和tmem106a的基因区域。构建的基因组甲基化评分为基因区域的甲基化程度与权重系数的乘积的求和。本技术方案中,lix1l、ac097173.1、steap2/steap2
‑
as1、cdkn2b、orc6和tmem106a的权重系数相对更高,仅采用这几种基因的基因区域计算gms也能够对预后进行预测评估。优选地,在部分实施例中,生物标志物除了包括lix1l、ac097173.1、steap2/steap2
‑
as1、cdkn2b、orc6和tmem106a的基因区域外,还包括linc01822、tuba4a、fam153b、fam205bp、gad2和golga6b的基因区域。在一个或多个实施例中,生物标志物包括前述17种基因区域。
[0020]
本发明提供了前述任一种生物标志物在肝内胆管细胞癌预后预测中的应用,在应用中,检测所述生物标志物的甲基化水平,基于所述甲基化水平计算基因组甲基化评分,根据所述基因组甲基化评分进行肝内胆管细胞癌预后。
[0021]
本技术方案中,首先检测患者的生物标志物的甲基化水平。甲基化水平的检测方法可以采用现有的任一种甲基化检测方法进行。在部分实施例中,甲基化检测可以采用全基因组dna甲基化测序或者焦磷酸测序法等其他甲基化测序法检测,可以采用基因芯片法进行甲基化检测,也可以采用特异性的甲基化qpcr法进行甲基化检测。优选地,采用wgbs检测患者基因区域的甲基化水平。
[0022]
进一步地,基因区域的甲基化水平为生物样本中甲基化胞嘧啶的百分比。本技术方案中,基因区域的甲基化水平的取值为0~1,其具体指生物样本,例如组织中细胞的基因区域上甲基化的胞嘧啶数量除以甲基化的胞嘧啶数量与未甲基化的胞嘧啶数量之和。相较于设置截断值,并根据截断值将甲基化水平直接取值为0或1的方式,本技术方案中的甲基化水平的检测连续,不同患者的基因组甲基化评分的差异更明显,进而更有效地体现出在肝内胆管细胞癌预后预测中的差异。在部分实施例中,甲基化水平也可以采用其他方式来表示,例如当采用qpcr进行甲基化检测时,甲基化水平表示为甲基化靶向基因数量除以内参基因数量的比值。
[0023]
得到基因区域的甲基化水平后,利用基因组甲基化评分的计算公式计算患者的基因组甲基化评分gms。具体地,所述计算公式为:其中,n为用于计算基因组甲基化评分的基因区域数量,gr
i
为第i个基因区域的甲基化水平,w
i
为第i个基因区域的系数,i=1,2,
…
,n。
[0024]
优选地,上述gms公式中,基因lix1l的基因区域的系数为
‑
2.21,基因linc01822的基因区域的系数为
‑
0.35,基因tuba4a的基因区域的系数为
‑
0.47,基因ac023481.1的基因区域的系数为
‑
0.25,基因mfsd1的基因区域的系数为
‑
0.23,基因ac097173.1的基因区域的系数为0.64,基因fam153b的基因区域的系数为
‑
0.57,基因steap2或steap2
‑
as1的基因区域的系数为
‑
0.62,基因cdkn2b的基因区域的系数为
‑
0.88,基因fam205bp的基因区域的系数为
‑
0.30,基因gad2的基因区域的系数为
‑
0.48,基因letmd1的基因区域的系数为
‑
0.21,基因rassf3的基因区域的系数为
‑
0.28,基因ac073592.4的基因区域的系数为
‑
0.11,基因
golga6b的基因区域的系数为
‑
0.54,基因orc6的基因区域的系数为
‑
15.04,基因tmem106a的基因区域的系数为
‑
0.73。经survminer包中的surv_cutpoint函数确定截断值,gms低于截断值的低值组中患者的预后优于gms高于截断值的高值组中患者的预后。优选地,当gms的计算公式采用上述系数时,gms的最佳截断值为
‑
3.10。
[0025]
本发明中,将三个独立肝胆中心的患者分为训练组和验证组,通过训练组构建gms模型,确定gms模型中的基因区域及基因区域对应的权重系数,再将gms模型在验证组中进行验证,证实了gms在肝内胆管细胞癌预后预测中的作用。此外,将构建的gms模型与列线图、以及美国癌症联合委员会(ajcc)tnm分期系统(第八版)进行比较,通过c指数、auc和kaplan
‑
meier生存曲线证明了所述gms模型出色的预后性能。
[0026]
本发明提供了一种肝内胆管细胞癌预后产品,所述预后产品包括检测试剂,所述检测试剂用于检测前述的生物标志物的甲基化水平。在部分实施例中,所述产品可以是试剂盒,该试剂盒包括用于检测前述生物标志物的甲基化水平的引物或芯片。在部分实施例中,所述产品也可以是基因芯片,通过基因芯片检测生物标志物的甲基化水平。
[0027]
本发明还提供一种肝内胆管细胞癌预后系统,该系统包括检测单元、计算单元和分析单元,其中:
[0028]
所述检测单元,用于检测生物样本中的生物标志物的甲基化水平;
[0029]
所述计算单元,用于基于所述甲基化水平计算基因组甲基化评分;
[0030]
所述分析单元,用于比较所述基因组甲基化评分和截断值的大小,根据比较结果将患者分入低值组和高值组,所述低值组的患者的预后优于所述高值组的患者的预后。
[0031]
进一步地,所述计算单元计算基因组甲基化评分的计算公式为:其中,n为用于计算基因组甲基化评分的基因区域数量,gr
i
为第i个基因区域的甲基化水平,w
i
为第i个基因区域的系数,i=1,2,
…
,n。
[0032]
在一个或多个实施例中,带入基因区域的权重系数后,所述gms的计算公式为:(
‑
2.21)*chr1:145957635
‑
145958017 (
‑
0.35)*chr2:21710623
‑
21712623 (
‑
0.47)*chr2:219251586
‑
219251713 (
‑
0.25)*chr3:8365593
‑
8365799 (
‑
0.23)*chr3:158804371
‑
158805340 0.64*chr4:119872612
‑
119873113 (
‑
0.57)*chr5:176114061
‑
176115592 (
‑
0.62)*chr7:90178863
‑
90179364 (
‑
0.88)*chr9:22005202
‑
22006271 (
‑
0.30)*chr9:34838349
‑
34838586 (
‑
0.48)*chr10:26223977
‑
26224538 (
‑
0.21)*chr12:51056370
‑
51056502 (
‑
0.28)*chr12:64671189
‑
64671690 (
‑
0.11)*chr12:124626183
‑
124626684 (
‑
0.54)*chr15:72237361
‑
72237862 (
‑
15.04)*chr16:46689829
‑
46690990 (
‑
0.73)*chr17:43213020
‑
43213041。
[0033]
进一步地,所述基因区域的甲基化水平为生物样本中甲基化胞嘧啶的百分比。
[0034]
本发明与现有技术相比,具有如下的优点和有益效果:
[0035]
1、本发明采用全基因组dna甲基化测序,在整个基因组范围内描述了肝内胆管细胞癌整体的表观遗传学改变,并系统地评估出17个基因区域的甲基化水平在肝内胆管细胞癌预后中的价值;
[0036]
2、本发明中选定的基因区域包括增强子、启动子、外显子、内含子、5’非翻译区和3’非翻译区,证明了多种基因区域的甲基化能够作为生存监测的生物标志物,同时,相较于现有技术中专注于单个cpg的位点,本发明对基因组特征的选择更加全面,使得通过这些基
因区域构建的基因组甲基化评分能够更加准确、可靠地对肝内胆管细胞癌预后进行预测;
[0037]
3、本发明中,基于选定的基因区域构建的基因组甲基化评分,也即gms模型,能够有效地预测手术后的长期存活率和患者对辅助治疗的反应,该效果在验证组中得到了验证,此外,通过将构建的gms模型与列线图、以及美国癌症联合委员会(ajcc)tnm分期系统进行比较,通过c指数、auc和kaplan
‑
meier生存曲线证明了该gms模型出色的预后性能;
[0038]
4、本发明依据gms从高到低的排名,将患者分为两组,第一组的排名位列前30%,第二组的排名位列后70%,通过分析发现gms排名前30%的患者能够受益于辅助的治疗,而gms排名在后70%的患者则不会受益于辅助治疗,通过对患者的区分,能够更加准确地对能够对辅助治疗产生反应的患者开展针对性的辅助治疗;
[0039]
5、本发明的甲基化水平采用生物样本中甲基化胞嘧啶的百分比的方式,相较于现有技术中设置截断值的方式,甲基化水平的连续变化使得不同患者的基因组甲基化评分的差异更明显,进而更有效地体现出在肝内胆管细胞癌预后预测中的差异。
附图说明
[0040]
此处所说明的附图用来提供对本发明实施例的进一步理解,构成本技术的一部分,并不构成对本发明实施例的限定。在附图中:
[0041]
图1为经预处理筛选后进一步通过单变量分析、c指数计算、cv计算以及lasso筛选出用于构建gms模型的17个基因区域的流程示意图;
[0042]
图2为采用lasso回归分析进一步从350个基因区域中筛选出17个基因区域,包括基因lix1l、linc01822、tuba4a、ac023481.1、mfsd1、ac097173.1、fam153b、steap2/steap2
‑
as1、cdkn2b、fam205bp、gad2、letmd1、rassf3、ac073592.4、golga6b、orc6和tmem106a的基因区域;
[0043]
图3示出了训练组(wchsu cohort)、验证组(zshfu&tmucih cohort)以及外部验证组(seae 450k cohort)的roc曲线的1年、2年和3年的曲线下面积(auc),并且示出了三个组中,gms高值组/sgms高值组和gms低值组/sgms低值组的生存曲线;
[0044]
图4示出了采用gms模型、wchsu列线图、jhusm列线图、ehbsh列线图和tnm分期系统对训练组和验证组进行评分的1年、2年和3年的roc曲线和曲线下面积;
[0045]
图5示出了依据gms模型、wchsu列线图、jhusm列线图、ehbsh列线图和tnm分期系统的评分结果对患者进行四等分分组后,各分组的生存曲线;
[0046]
图6示出了依据gms模型、wchsu列线图、jhusm列线图、ehbsh列线图和tnm分期系统的评分结果获得的评分靠前30%的患者在接受辅助治疗或不接受辅助治疗的生存曲线;
[0047]
图7示出了本发明一个或多个实施例中预后系统的框图。
具体实施方式
[0048]
为使本发明的目的、技术方案和优点更加清楚明白,下面结合实施例和附图,对本发明作进一步的详细说明,本发明的示意性实施方式及其说明仅用于解释本发明,并不作为对本发明的限定。
[0049]
在本发明的描述中,生物样本是指细胞、组织、脏器、体液、消化液、咳痰、肺胞支气管清洗液、尿液、粪便中的一种或多种。在一个或多个实施例中,生物样本为患者的组织。
[0050]
在本发明中,预后是指关于医学发展的预期,例如长期存活的可能性、无疾病存活率等,预后包括消极预后和积极预后,其中,所述消极预后包括疾病进展如复发、肿瘤生长、转移和耐药死亡率,而所述积极预后包括疾病缓解和疾病改善,例如无疾病状态、肿瘤消退或稳定。
[0051]
下述实施例中,数据的统计采用rstudio 1.1.463,spss 25.0和graphpad prism 8执行。r包“survival”用于单变量cox分析,r包“rms”用于一致性指数(c指数)计算。对于所有的实验,双尾p值小于0.05被认为具有统计学意义。
[0052]
本发明中,为探讨dna甲基化在但内胆管细胞癌中的预后价值,对来自四川大学华西医院、复旦大学中山医院和天津医科大学肿瘤医院的共计334位患者采用全基因组dna甲基化测序(wgbs)。其中,基因组dna的分离,以及wgbs的建库和测序由北京诺禾致源科技股份有限公司(novogene)完成;采用fastqc生成原始wgbs测序读取的质量报告,并运用fastp进行质量控制和低质量读取的过滤。采用软件moabs进行dna甲基化分析。其余未注明具体条件的实验方法,采用现有技术中的常规实验条件、方法或产品,或者按照制造厂商建议的方式完成。
[0053]
【实施例1】
[0054]
本实施例包括多中心和回顾性队列的患者。研究了从2010年5月至2019年7月,在四川大学华西医院、复旦大学中山医院和天津医科大学肿瘤医院接受了治愈性肝切除术后的患者。考虑到临床病理和分子异质性,仅纳入了肝内胆管细胞癌患者。所有患者均在组织学上首次被诊断为肝内胆管细胞癌,并且未包括复发的肝内胆管细胞癌患者。该研究方案已获得三个肝胆中心的伦理委员会的批准,并在手术前获得了每位患者的书面知情同意书。
[0055]
将选自三个肝胆中心的共计334位患者分为训练组和验证组。其中,四川大学华西医院(wchsu cohort)的164名患者为训练组,复旦大学中山医院(zshfu cohort)的117名患者和天津医科大学肿瘤医院(tmucih cohort)的53名患者,共计170名患者构成验证组。训练组和验证组中患者的病理参数如表1所示。训练组的中位随访时间为28.5个月,有106名患者在随访期内死亡;验证组的中位随访时间为19.0个月,有62名患者在随访期内死亡。
[0056]
表1
[0057]
[0058][0059]
【实施例2】
[0060]
对训练组的患者采用全基因组dna甲基化测序,获得基因组的甲基化水平,移除cpg位点完全相同的区域、na数量不小于16的区域、甲基化水平为0的区域等基因区域后,从1606362个基因区域中初步筛选出1028088个基因区域。
[0061]
随后,从初步筛选出的基因区域中进一步筛选。如图1所示,在进一步移除12个甲基化水平为0的基因区域后,利用单变量cox分析、一致性指数计算和变异系数计算筛选得到350个基因区域。该350个基因区域符合下述条件:(1)在单变量cox分析中,p值小于0.001;(2)c指数大于0.65;且(3)cv值大于0.2。
[0062]
最后,利用lasso cox算法压缩高维数据,基于患者病理参数和甲基化水平选定候选基因区域,如图2(a)和(b)所示,最终得到17个基因区域以及各基因区域对应的权重系数,17个基因区域如表2所示:
[0063]
表2
[0064]
[0065][0066]
基于表2中的基因区域以及各基因区域的系数,得到下述gms模型:
[0067]
gms=(
‑
2.21)*chr1:145957635
‑
145958017 (
‑
0.35)*chr2:21710623
‑
21712623 (
‑
0.47)*chr2:219251586
‑
219251713 (
‑
0.25)*chr3:8365593
‑
8365799 (
‑
0.23)*chr3:158804371
‑
158805340 0.64*chr4:119872612
‑
119873113 (
‑
0.57)*chr5:176114061
‑
176115592 (
‑
0.62)*chr7:90178863
‑
90179364 (
‑
0.88)*chr9:22005202
‑
22006271 (
‑
0.30)*chr9:34838349
‑
34838586 (
‑
0.48)*chr10:26223977
‑
26224538 (
‑
0.21)*chr12:51056370
‑
51056502 (
‑
0.28)*chr12:64671189
‑
64671690 (
‑
0.11)*chr12:124626183
‑
124626684 (
‑
0.54)*chr15:72237361
‑
72237862 (
‑
15.04)*chr16:46689829
‑
46690990 (
‑
0.73)*chr17:43213020
‑
43213041
[0068]
【实施例3】
[0069]
为了验证gms模型的稳定性,首先在训练组(wchsu cohort)中进行gms调查,总生存期(overall survival,os)的c指数为0.779(95%ci:0.738~0.820)。
[0070]
如图3(a)所示,训练组中,总生存期的1年、2年、3年的曲线下面积(auc)分别为0.859、0.842和0.880。应用gms模型时,根据“survminer”包中的surv_cutpoint函数确定该gms模型的最佳截断值为
‑
3.10。通过比较患者的gms评分和截断值的大小,将患者分为gms低值组(gms
‑
low)和gms高值组(gms
‑
high)。其中,gms低值组中有98名患者,gms高值组中有66名患者。如图3(b)所示,gms低值组中98名患者的总生存期中位数为55.5
±
3.5个月,1年、3年和5年的生存率分别为93.9%、74.1%、46.3%;而gms高值组中66名患者的总生存期中位数为10.5
±
1.5个月,1年、3年和5年的生存率分别为45.0%、7.5%和0%。由此可见,gms低值组中患者的总生存期明显优于gms高值组中患者的总生存期。
[0071]
随后,在验证组(zshfu&tmucih cohort)中检测与训练组检测的相同的基因区域,并带入至相同的gms模型中进行计算,得到总生存期的c指数为0.739(95%ci:0.675~0.803)。
[0072]
如图3(c)所示,总生存期的1年、2年、3年的曲线下面积(auc)分别为0.787、0.770和0.786。类似地,基于gms分数以及与训练组相同的截断值成功将验证组中170名患者分为gms低值组和gms高值组。其中,gms低值组中有111名患者,gms高值组中有59名患者,另有10名患者的报告暂时无法获取(表1中验证组部分参数缺失原因相同)。如图3(d)所示,gms低值组的总生存期中位数为53.0
±
11.5个月,1年、3年和5年的生存率分别为92.2%、70.8%和41.8%;而gms高值组的总生存期中位数为16.9
±
2.4个月,1年、3年和5年的生存率分别为65.6%、20.7%和20.7%。gms低值组中患者的总生存期明显优于gms高值组中患者的总生存期。
[0073]
之后,基于另一个外部的验证组(seae 450cohort)对gms模型的稳定性进行验证,该验证组是apinyajusakul等人报道的迄今为止最大的具有高通量甲基化数据的icc队列(总生存期大于1个月),主要由东南亚和欧洲的icc患者构成。该验证组的甲基化数据由infinium公司的human methylation 450beadchip(450k序列)生成,如表1所示,该验证组仅覆盖6个具有代表性的cpg位点的基因区域,即基因lix1l、fam153b、cdkn2b、fam205bp、ac073592.4和orc6的基因区域。因此,采用与训练组的gms模型中这6个基因区域相同的系数,得到简化的sgms模型以计算基因组甲基化评分。所述sgms的总生存期的c指数为0.662(95%ci:0.575~0.750),且1年、2年、3年的曲线下面积(auc)分别为0.670、0.726和0.773,如图3(e)所示。
[0074]
进一步地,将sgms的中位数作为截止点,将患者分为sgms低值组(sgms
‑
low)和sgms高值组(sgms
‑
high),其中,sgms低值组的患者数量为45名,sgms高值组的患者数量为46名。如图3(f)所示,sgms低值组的平均os为70.5
±
7.9个月,1年、3年和5年生存率分别为86.5%、72.4%和55.3%,而sgms高值组的平均os为18.0
±
1.5个月,1年、3年和5年生存率分别为65.8%、34.5%和27.6%。sgms低值组中患者的总生存期明显优于sgms高值组中患者的总生存期。
[0075]
综上,gms模型可以准确地预测患者的生存率,且其效果在来自不同地区和国家的
两个独立的验证组中得到证实,稳定性高;同时,采用gms模型将患者分为低值组和高值组,能够对患者的总生存期和生存率进行预测和区分;不仅如此,简化的sgms模型虽然没有应用全部的17个基因区域,但在仅采用了6个基因区域的前提下也能够实现较准确的预后,并将患者划分为低值组和高值组,进一步表明了gms模型具有更高的稳定性和准确率。
[0076]
【实施例4】
[0077]
ajcc tnm分期系统和列线图(nomogram)通常被用于预测患者的生存率。目前,hyper等人在《a nomogram to predict long
‑
term survival after resection for intrahepatic cholangiocarcinoma》中公开的jhusm列线图和wang等人在《prognostic nomogram for intrahepatic cholangiocarcinoma after partial hepatectomy》公开的ehbsh列线图是两个被高度引用的列线图。为了验证gms模型的性能,利用jhusm列线图、ehbsh列线图、ajcc tnm分期系统以及四川大学华西医院的wchsu列线图分析训练组和验证组的临床参数。
[0078]
表3示出了训练组和验证组的单变量和多变量分析数据,从表3可以看出,在包含了临床参数的多变量分析中,gms模型是总生存期的独立预测指标(p<0.001,hr:3.201,95%ci:2.451~4.180)。
[0079]
表3:
[0080]
[0081]
[0082][0083]
进一步地,通过两两比对证明,gms的c指数(0.769,95%ci:0.733~0.804)高于wchsu列线图的c指数(0.715,95%ci:0.675~0.755,p=0.013)、高于jhusm列线图的c指数(0.686,95%ci:0.642~0.729,p<0.001)、高于ehbsh列线图的c指数(0.674,95%ci:0.632~0.716,p<0.001)、以及高于ajcc tnm分期系统的c指数(0.605,95%ci:0.559~0.652,p<0.001)。
[0084]
图4的(a)~(c)分别示出了1年、2年和3年的gms模型、各列线图和分期系统的roc曲线及对应的auc。由图可知,gms模型的1年、2年和3年的auc分别为0.833、0.822和0.853;wchsu列线图的1年、2年和3年的auc分别为0.747、0.747和0.744;jhusm列线图的1年、2年和3年的auc分别为0.722、0.724和0.702;ehbsh列线图的1年、2年和3年的auc分别为0.707、0.709和0.719;ajcc tnm分期系统的1年、2年和3年的auc分别为0.606、0.622和0.608。可以看出,gms模型的性能优于wchsu列线图、jhusm列线图、ehbsh列线图和ajcc tnm分期系统。
[0085]
此外,gms模型、wchsu列线图、jhusm列线图、ehbsh列线图和ajcc tnm分期系统评分后,依据评分从低到高,将患者分为quartile1、quartile2、quartile3和quartile4共4部分,各部分患者数量为总数量的25%。如图5(a)所示,gms模型的quartile1至quartile4的4条生存曲线能够完全分离,且生存率依次降低(整体p<0.001)。并且,如图所示,quartile 2比quartile 1的p值小于0.001,quartile 3比quartile 2的p值等于0.015,quartile 4比quartile 3的p值小于0.001,也表明两两分组之间存在明显差异。相反,如图5(b)~(e)所示,尽管wchsu列线图、jhusm列线图、ehbsh列线图和ajcc tnm分期系统的整体的p值均达到了统计学上的显著性,但是生存曲线均没有如gsm模型一样呈现出完全分离,且生存率依次降低,例如wchsu列线图中,quartile 3与quartile 2的生存曲线存在交叉,又例如jhusm列线图中,quartile 2与quartile 1的生存曲线存在交叉。此外,从相邻两个分组的比值的p值也可以看出,相邻两组之间不具备明显差异。
[0086]
【实施例5】
[0087]
gms模型还可以用于识别能够在术后从辅助治疗(adjuvant therapy,at)中获益的icc患者。通过尝试不同的临界值(10%~90%),发明人发现,gms评分从高到低排名靠前的30%的患者能够受益于辅助治疗,而排名靠后的70%的患者没有从辅助治疗中受益。如图6a和图6b所示,gms评分更高的前30%的患者的接受辅助治疗和未接受辅助治疗的生存曲线完全分离,hr=0.533,95%ci:0.329~0.866,log
‑
rank p=0.032;gms评分更低的后70%的患者的接受辅助治疗和未接受辅助治疗的生存曲线出现交叉,hr=1.264,95%ci:0.720~2.220,log
‑
rank p=0.373。
[0088]
如图6c至6f所示,在采用相同的临界值30%时,wchsu列线图(hr=0.639,95%ci:0.379~1.075,log
‑
rank p=0.131)、jhusm列线图(hr=0.732,95%ci:0.426~1.257,log
‑
rank p=0.294)、ehbsh列线图(hr=0.784,95%ci:0.464
–
1324,log
‑
rank p=0.388)和tnm分期系统(hr=0.948,95%ci:0.533~1.685,log
‑
rank p=0.855)的评分前30%的患者在接受辅助治疗和未接受辅助治疗的生存曲线均出现较差,故无法区分能够或不能够对辅助治疗产生反应的患者。因此,通过gms模型对患者进行区分,能够更加准确地对能够对辅助治疗产生反应的患者开展针对性的辅助治疗。
[0089]
【实施例6】
[0090]
如图7所示的一种肝内胆管细胞癌预后系统,该系统包括检测单元、计算单元和分析单元,其中:
[0091]
所述检测单元,用于检测生物样本中的前述实施例中任一种或多种生物标志物的甲基化水平;
[0092]
所述计算单元,用于基于所述甲基化水平计算基因组甲基化评分;
[0093]
所述分析单元,用于比较所述基因组甲基化评分和截断值的大小,根据比较结果将患者分入低值组和高值组,所述低值组的患者的预后优于所述高值组的患者的预后。
[0094]
在一个或多个实施例中,甲基化检测可以采用全基因组dna甲基化测序或者焦磷酸测序法等其他甲基化测序法检测,可以采用基因芯片法进行甲基化检测,也可以采用特异性的甲基化qpcr法进行甲基化检测。优选地,采用wgbs检测患者基因区域的甲基化水平。
[0095]
在部分实施例中,所述计算单元计算基因组甲基化评分的计算公式为:在部分实施例中,所述计算单元计算基因组甲基化评分的计算公式为:其中,n为用于计算基因组甲基化评分的基因区域数量,gri为第i个基因区域的甲基化水平,w
i
为第i个基因区域的系数,i=1,2,
…
,n。
[0096]
在部分实施例中,所述基因区域的甲基化水平为生物样本中甲基化胞嘧啶的百分比。在一个实施例中,甲基化水平也可以采用其他方式来表示,例如当采用qpcr进行甲基化检测时,甲基化水平表示为甲基化靶向基因数量除以内参基因数量的比值。
[0097]
在部分实施例中,计算单元除了计算gms评分外,还根据临床参数计算wchsu列线图,并结合gms评分和wchsu列线图共同进行预后预测,以进一步提高预后的准确性。
[0098]
以上所述的具体实施方式,对本发明的目的、技术方案和有益效果进行了进一步详细说明,所应理解的是,以上所述仅为本发明的具体实施方式而已,并不用于限定本发明的保护范围,凡在本发明的精神和原则之内,所做的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。