用于预测肝癌复发的dna甲基化标志物及其用途
技术领域
1.本发明涉及一种利用基因组dna甲基化水平差异根据肝癌预后预测肝癌复发的dna甲基化标志物及其用途,这具体涉及一种用于预测肝癌患者治疗后的正常肝组织的肝癌复发的dna甲基化标志物。
背景技术:
2.肝癌大致可分为由肝细胞自身产生的原发性肝癌(肝细胞癌、肝癌)和其他组织的癌症已转移至肝脏的转移性肝癌。大约90%以上的肝癌是原发性肝癌,并且通常理解的术语肝癌是指原发性肝癌。根据韩国统计局2018年9月发布的“2017年死亡原因统计数据”,以10万人口计,2017年癌症死亡人数为153.9人,比上年增加1.0(0.6%)。癌症死亡率按照从高到低排序为肺癌(35.1)、肝癌(20.9)、结直肠癌(17.1)、胃癌(15.7)和胰腺癌(11.3)。
3.与其他癌症一样,肝癌的治疗主要分为根治治疗和保守治疗。这里,根治是指完全消除癌症本身的治疗,包括肝切除手术、肝移植、射频消融等。此外,如果无法进行这些治疗,则进行颈动脉化疗栓塞、放射治疗和抗癌治疗。然而,这种治疗方法几乎难以彻底治疗,因此医学检查的目的是通过早期发现提供根治性治疗。在肝切除手术的情况下,大约50%的患者会出现癌症复发,因为切除的肝脏也暴露于肝炎,而不是正常肝脏。
4.肝癌的预后是指在肝癌确诊后,根据肝癌预测患者的各种情况,如肝癌完全治疗的可能性、治疗后复发的可能性和患者的生存可能性,这取决于各种情况,例如疾病的严重程度、诊断时间和治疗过程。只有根据预后适当应用各种治疗方法,才能有效治疗肝癌。例如,对于预期预后良好的患者,要避免可能对患者造成严重副作用的风险性治疗方法,如积极的化疗或手术和放疗,而应选择相对温和、保守、安全的治疗方法。反之,对于预计预后不良的患者,应通过积极动员如化疗或手术以及放疗的治疗方法以努力增加生存期或生存率。
5.然而,根据现有技术很难准确预测肝癌患者的预后。为了准确预测预后,需要一种分析方法,按风险组对患者进行分类,但到目前为止,还存在仅由诊断和初步手术治疗时肝癌的临床病理分期决定预后的情况,而没有手段准确预测肝癌的预后。
6.作为解决该问题的努力的结果,本发明人准备了一种能够基于肝癌组织的dna甲基化数据预测肝癌患者预后的方法。
技术实现要素:
7.技术问题
8.本发明的目的是提供一种包含dna甲基化标志物的用于预测肝癌复发的组合物和一种使用该组合物预测肝癌复发可能性的方法。
9.技术方案
10.为了实现上述目的,本发明的一个方面提供了一种用于预测肝癌复发的组合物,其包含一种或多种选自由seq id no:1至4所表示的dna甲基化标志物组成的组的dna甲基
化标志物。
11.在本发明中,上述seq id no:1至4所表示的dna甲基化标志物分别是指cg21325760、cg10544510、cg06702718和cg15997204,其是由cg基因座符号表示的dna甲基化标志物(infinium humanmethylation 850k微珠阵列cpg id)。
12.如本文所用,术语“肝癌”是发生在肝组织中的癌症的通用术语,肝癌可被分为由肝细胞自身产生的原发性肝癌(肝细胞癌、肝癌)和其他组织的癌症已转移至肝脏的转移性肝癌。多种治疗方法如肝切除、肝移植、射频消融以及颈动脉化疗栓塞可被用于肝癌的治疗,但5年生存率仅为33.6%(2011
‑
2015年的平均值;来自卫生和福利部的统计数据),并且复发率也达到70%。与其他癌相比,肝癌的5年生存率较低,复发率较高,因此需要一种能够预测复发的方法。
13.如本文所用,术语“dna甲基化”是指甲基与基因组dna中胞嘧啶碱基的c5位共价结合。甲基化水平是指,例如,所有基因组区域和一些非基因组区域的dna碱基序列中存在的甲基化量,而在本发明中,是指seq id no:1至4所示的dna甲基化标志物的甲基化程度。在seq id no:1至4的dna甲基化标志物中,甲基化可能发生在整个序列中。
14.在本发明中,用于预测肝癌复发的组合物可以是包含一种或多种、两种或多种、三种或多种或四种dna甲基化标志物的组合物。具体地,用于预测肝癌复发的组合物可分别包含以下dna甲基化标志物之一:cg21325760、cg10544510、cg06702718和cg15997204,并且可以包括cg21325760和cg10544510;cg21325760和cg06702718;cg21325760和cg15997204;cg10544510和cg06702718;cg10544510和cg15997204;或cg06702718和cg15997204的组合,其是两种标志物的组合。此外,用于预测肝癌复发的组合物可包含所述dna甲基化标志物的三种,例如cg21325760、cg10544510和cg06702718;cg21325760、cg10544510和cg15997204;cg21325760cg06702718和cg15997204;或cg10544510、cg06702718和cg15997204的组合此外,用于预测肝癌复发的组合物可以包含所有的dna甲基化标志物cg21325760、cg10544510、cg06702718和cg15997204。
15.本发明中,dna甲基化标志物cg15997204是存在于myt1l基因中的标志物,而myt1l(髓鞘转录因子1样(myelin transcription factor 1like))基因编码属于c
‑
c
‑
h
‑
c锌指蛋白的转录因子。根据tcga(the cancer genome atlas)发表的数据,myt1l在多种癌症中均有表达,但该基因的甲基化水平与肝癌复发之间的相关性尚不清楚。
16.在本发明中,dna甲基化标志物cg21325760存在于magel2(mage(黑色素瘤相关抗原)家族成员l2)基因中,并且根据tcga数据在睾丸癌中高度表达,但该基因的甲基化水平与肝癌复发之间的相关性尚不清楚。
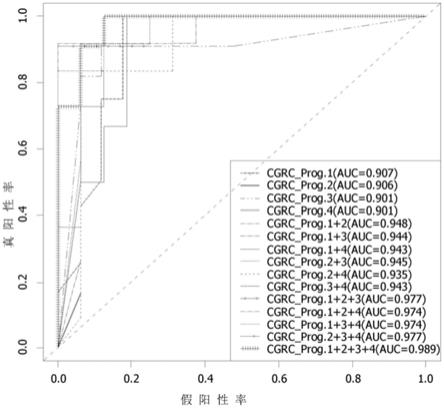
17.在本发明的一个实施方案中,本发明人在从肝癌组织中分离基因组dna后用infinium humanmethylation 850k微珠阵列确认了甲基化水平。结果证实,根据甲基化水平将肝癌组织分为两组,并且可知所述两组在肝癌复发率上存在显著差异。此后,通过随机森林等机器学习技术在两组中鉴定出甲基化水平显著不同的dna甲基化标志物,并发现了dna甲基化标志物(见表3)。此外,本发明通过确认当四种dna甲基化标志物一起使用而不是使用一种dna甲基化标志物时,预测肝癌复发的准确度收敛到1,从而完成了本发明(图5和表2)。
18.在本发明中,肝癌可以是由肝细胞自身产生的肝细胞癌,也可以是其他组织的癌
转移到肝脏的转移性肝癌,但优选为肝细胞癌。肝细胞癌约占肝脏恶性肿瘤的90%,在韩国、日本、东南亚、中国等多发。大多数发生在肝硬化状态,但也有一些是由慢性乙型或丙型肝炎引起的。
19.如本文所用,术语“复发”是指肝癌患者在治疗肝癌后,细胞增殖异常,即肝脏再次发生肝癌。作为肝癌患者的治疗方法,可以采用肝切除术、射频消融术、颈动脉化疗栓塞术、经皮乙醇注射等,但不限于此。
20.在本发明中,用于预测肝癌预后的组合物可以以用于预测肝癌复发的试剂盒的形式实施,该试剂盒还包括鉴定dna甲基化标志物的甲基化水平所必需的试剂。
21.本发明的一个方面提供了一种用于预测肝癌复发的方法,其包括以下步骤:
22.(a)从待分析的肝脏组织中分离基因组dna的步骤;
23.(b)检测一种或多种dna甲基化标志物的甲基化水平的步骤,所述dna甲基化标志物选自由seq id no:1至4表示的dna甲基化标志物组成的组;和
24.(c)比较检测到的dna甲基化标志物的甲基化水平与正常肝组织中检测到的dna甲基化标志物的甲基化水平的步骤。
25.在本发明中,由seq id no:1至4所表示的dna甲基化标志物分别是指cg21325760、cg10544510、cg06702718和cg15997204,其是由cg基因座符号表示的dna 甲基化标志物(infinium humanmethylation 850k微珠阵列cpg id)。
26.在本发明的一个实施方案中,待分析的肝组织可以是肝癌组织、肝细胞癌组织或转移性肝癌或肝癌已转移的部位的转移性癌组织,优选为肝细胞癌组织。作为从待分析受试者分离待分析肝组织的方法,可使用本发明所属技术领域中已知的经皮活检、颈静脉活检等方法。
27.在本发明的一个实施方案中,用于预测肝癌预后的方法可以是步骤(b)中鉴定至少两种dna甲基化标志物的甲基化的方法。发明人发现了四种能够预测肝癌复发的dna甲基化标志物(探针)(见表2),并根据标志物的数量评价了预测肝癌复发的性能。作为评价的结果,确认了存在两种或更多种标志物时,精度、灵敏度等性能收敛于0.9,而当标志物数量为3种或更多种时,肝癌复发预测性能收敛于1(图5)。
28.由该结果可知,本发明的dna甲基化标志物即使单独使用也具有优异的肝癌预后预测性能,但在多个使用时其性能是显著优异的。因此,本发明的dna甲基化标志物可单独使用或将两种或多种标志物组合使用以用于预测肝癌复发。
29.具体地,对于本发明的dna甲基化标志物,cg21325760、cg10544510、cg06702718和cg15997204可以分别以单独标志物使用,也可以两种标志物的组合(cg21325760和cg10544510;cg21325760和cg06702718;cg21325760和cg15997204;cg067027510和cg06702718;cg067027510和cg15997204;或cg06702718和cg15997204)或以三种标志物的组合(cg21325760、cg10544510和cg06702718;cg21325760、cg10544510和cg15997204;cg21325760、cg06702718和cg15997204;或cg10544510、cg06702718和cg15997204)使用。此外,可以组合并使用所有四种标志物。
30.在本发明的一个实施方案中,预测肝癌预后的方法还可以包括,在步骤(c)之后,确定当待分析样本中鉴定的dna甲基化标志物的甲基化水平低于正常肝组织的甲基化水平时,肝癌复发的可能性较高的步骤。
31.在本发明中,鉴定dna甲基化标志物的甲基化水平的方法可以通过本领域公知的pcr、甲基化特异性pcr、实时甲基化特异性pcr、和使用甲基化dna特异性结合蛋白的pcr、定量pcr、dna芯片、焦磷酸测序、市售芯片等进行,但不限于此。
32.本发明人已经证实,在复发性肝癌(mg2)患者中,dna甲基化标志物的甲基化水平明显低于正常肝组织(n)(图3、7和8),因此,dna甲基化标志物可有效地用作预测肝癌复发的用途。
33.有益效果
34.当使用本发明的用于肝癌复发预测的dna甲基化标志物时,可以容易地预测肝癌的复发,并且可以为肝癌复发高风险患者提供更加个性化的治疗,并且可以提供临床信息而避免不必要的过度治疗。
附图说明
35.图1示出了鉴定从肝细胞癌样本(a;n=140))和正常肝脏样本(b;n=96)中分离的dna的甲基化水平的结果。
36.图2示出了根据预后将肝细胞癌样本(n=140)分类为甲基化组1(mg1;n=81)和甲基化组2(mg2;n=59),然后根据dna甲基化模式通过一致性聚类方法对每种样本进行分类的结果。
37.图3示出了鉴定甲基化组1(mg1)和甲基化组2(mg2)的无复发生存期(rfs)的结果。
38.图4示出了确认甲基化组1(mg1)和甲基化组2(mg2)中的变量重要性图(varimpplot)(a),然后选择参与甲基化组分类的前50个探针(b)的过程。
39.图5以曲线下面积(auc)值示出了根据参与甲基化组分类的探针数量确认肝癌预后预测效率的结果。
40.图6示出了鉴定正常肝脏样本(n)、甲基化组1(mg1)和甲基化组2(mg2)中参与甲基化组分类的探针甲基化水平的结果。
41.图7示出了使用参与甲基化组分类的探针对自主产生的肝癌验证队列数据进行分类的结果。
42.图8示出了使用参与甲基化组分类的探针对tcga(癌症基因组图谱)中发表的肝癌数据进行分类的结果。
具体实施方式
43.在下文中,将通过实施例更详细地描述一个或多个实施方案。然而,这些实施例用于说明一个或多个实施方案,并且本发明的范围不限于这些实施例。
44.实验方法
45.1.招募肝细胞癌患者
46.与本发明相关的研究得到了首尔国立大学医院审查委员会的批准,并获得了所有患者的书面同意。招募2011年至2016年在首尔国立大学医院外科接受肝细胞癌(以下简称hcc)切除术的184例患者,通过术后ct扫描证实镜下切缘干净,肿瘤完全切除。患者定期到医院就诊,并根据标准方案每3个月监测一次复发情况,所述标准方案包括血清肿瘤标志物、甲胎蛋白(afp)水平、对比ct扫描或磁共振成像(mri)。复发分类为早期复发(≤1年)和
晚期复发(>1年),不包括hcc切除术后1年内复发的患者。在医院病历表上确定人口统计学数据和临床病理特征,并在切除术后对患者进行随访直至死亡。排除1年内复发患者的原因是,在早期复发的情况下,hcc切除不正确或因肝癌以外的原因复发很常见,因此,它不符合本发明的目的,即发现涉及癌症治疗后在完全正常肝脏中复发新的癌症的标志物。
47.2.甲基化数据的生成
48.在182例hcc患者中,42例术后1年内出现hcc复发的患者被排除在分析之外。根据制造商的说明,使用maglisto
tm
5m基因组dna提取试剂盒(bioneer)从剩余140例患者的冷冻hcc样本中提取dna。根据制造商的标准方案使用500ng dna以及illumina iscan system(illumina,ca,美国)对每种样本进行illumina infinium humanmethylation epic 850k微珠芯片(illumina)分析。illumina infinium humanmethylation epic微珠芯片包含用于解析每个单核苷酸的850000或更多cpg位点的dna甲基化值。
49.3.dna甲基化数据处理
50.对于上述2中使用illumina infinium humanmethylation epic beadchip获得的数据,使用r平台中的minfi r包(https://cran.r
‑
project.org/bin/windows/base/;版本3.3.2)对甲基化数据进行预处理,这是个开放软件。具体地,处理hcc样本的原始idat文件,并校正背景校正和染料偏差。此后,测量甲基化和未甲基化探针的信号强度,从而将每个探针的dna甲基化值量化为0(未甲基化)至1(完全甲基化)范围内的b值。接下来对所有样本进行数据质量控制,并且140个样本全部通过质量控制标准。最后,为了减少批次效应问题,使用功能归一化方法对甲基化数据进行归一化。
51.4.dna甲基化值
52.将甲基化b值根据下面的公式1转换为m值用于统计分析。
53.[公式1]
[0054]
m值=log((b
‑
值)/(1
‑
(b
‑
值)))
[0055]
m值用于一致性聚类,b值用于热图可视化和箱线图生成。
[0056]
5.一致性聚类
[0057]
使用consensusclusterplus(https://bioconductor.org/packages/release/bioc/vignettes/consensusclusterplus/inst/doc/consensusclusterplus.pdf)进行一致性聚类以鉴定hcc亚组。将前3000个最易变的探针(中值绝对偏差(mad))(mad 3k)用作输入。
[0058]
6.预后标志物的选择
[0059]
随机森林方法基于为非线性效应建模而开发的机器学习算法。发明人使用r平台的随机森林r包发现了能够区分具有良好和不良预后的hcc组的探针,并将hcc肿瘤样本随机分为4个训练队列和1个测试队列进行五倍交叉验证。作为执行的结果,发明人建立了随机森林模型,其中通过混淆矩阵(预测样本vs观察样本)预测每组的预期比率,并根据曲线下面积(auc)、敏感性和特异性值确定每个模型的性能。
[0060]
本发明人考虑到基尼指数、探针杂质的整体减少以及通过随机森林测量的可变重要性的点图来测量每个探针的重要性值。结果,本发明人发现了能够将hcc样本分类为甲基化组1和2(下文中,分别描述为mg1和mg2)的前50个探针(图4)。下文中,在比较1个探针和前50个探针的性能效率时,通过考虑auc值确认了效率最高的探针的最少数量。
[0061]
7.基因本体论和通路分析
[0062]
本发明人使用innatedb(http://www.innatedb.com/)进行了各种路径搜索和基因本体论分析。在innatedb中,使用了基因本体论生物过程、kegg(京都基因与基因组百科全书)和reactome资源。显著的截断标准是p值<0.05。
[0063]
9.统计分析
[0064]
使用pearson's chi(χ2)检验或fisher精确检验对分类变量进行统计分析。中位无复发生存期(rfs)采用kaplan
‑
meyer法计算,不同组间比较采用对数秩检验。
[0065]
实验结果
[0066]
1.患者选择和基本特征
[0067]
182例hcc患者中,42例术后1年内出现hcc复发的患者被排除在分析之外。从对140个hcc样本的甲基化鉴定结果可以看出,hcc样本在样本之间表现出较大的甲基化变化,而正常组织则表现出几乎恒定的甲基化(图1)。
[0068]
2.mg1和mg2甲基化谱的比较
[0069]
由根据dna甲基化模式对140个hcc样本进行分类的结果可以证实它们被分为甲基化组1(n=81)和组2(n=59)(以下分别描述为mg1和mg2)(图2中的a)。此外,mg2的甲基化谱与非肿瘤性肝组织的甲基化谱差异最大,而mg1的甲基化谱与非肿瘤性肝组织的甲基化谱相似(图2中的b)。可见mg1的无复发生存期(rfs)较长,而mg2的无复发生存期较短,从而导致预后较差(图3)。
[0070]
这种表观遗传变化与信号转导、mapk(促有丝分裂原激活蛋白激酶)和g蛋白偶联受体(gpcr)信号通路有关(表1)。当发明人基于该组的go(基因本体论)系统解释一系列基因时,前50个探针与分解代谢、神经递质分泌、分子功能调控、wnt信号通路、细胞迁移调控、mrna代谢、小gtp酶介导的信号转导、基因表达调控和细胞凋亡过程调控相关。
[0071]
[表1]
[0072]
[0073][0074]
3.预测hcc复发的标志物的选择
[0075]
使用变量重要性图(varimpplot)发现了能够将hcc样本分类为mg1和mg2的前50个探针(图4)。以下,在根据探针数量比较性能效率的同时,考虑auc(曲线下面积)值选择具有最大效率的探针的最少数量。由结果可以看出,当探针数量为三个或更多时,肝癌复发预测的准确度收敛于1(图5和表2)。下面表2中描述的字母分别表示准确度(accu.)、灵敏度(sen.)和特异性(spec.)。
[0076]
[表2]
[0077]
[0078][0079]
基于上表2的结果,最终选择了下表3中描述的探针,其中探针按重要性值由高到低的顺序进行记载。
[0080]
[表3]
[0081]
[0082][0083]
表3中描述的探针的序列在下表4中记载。
[0084]
[表4]
[0085][0086]
*在基因组序列中,[cg]表示相关cg标志物靶向的甲基化位点
[0087]
3.hcc复发预测标志物验证
[0088]
由使用随机森林方法鉴定表3中描述的探针的甲基化水平的结果可以确认,正常肝组织(绿色)中的甲基化水平接近100%并且处于恒定水平,以及mg1(红色)显示出与正常肝组织相似的甲基化水平,而mg2(灰色)的甲基化水平与正常肝组织相比约为50%或更低(图6;x轴是选择的标志物,y轴是表示甲基化水平的b值)。
[0089]
此外,将140个hcc样本随机分为训练数据和验证数据,并且根据再次用表3中的探针进行测试的测试结果,可以确认现有的mg1再次归类为mg1,并且现有的mg2也再次归类为mg2(表5)。
[0090]
[表5]
[0091][0092]
本发明人自主生成的肝癌队列数据通过表3中的探针进行验证。结果可以证实,肝癌队列数据根据甲基化水平被分为两组(pre_mg1和pre_mg2),且pre_mg2的复发率较高(图7中的a)。此外,由确认rfs的结果可以确认pre_mg1的rfs明显长于pre_mg2的rfs(p=0.0136;图7中的b)。该结果意味着用本发明的探针可以成功预测肝癌的复发。
[0093]
此外,根据用表3中的探针以同样的方式验证tcga(癌症基因组图谱)肝癌数据(一个开放的数据库)的验证结果,可以确认肝癌患者被分为两组(pre_mg1和pre_mg2)(图8中的a),并且这些组具有不同的预后(图8中的b)。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。