基于pma高通量测序和picrust的抗生素抗性基因及潜在宿主细胞的鉴定方法
技术领域
1.本发明属于环境样品中新兴基因型污染物及其宿主细菌的鉴定技术领域,具体涉及一种环境样品中抗生素抗性基因(antibiotic resistance genes,args)及其潜在活性宿主细菌的鉴定方法。
背景技术:
2.抗生素被广泛应用于人类或动植物疾病预防/治疗及动物养殖中,中国是世界上生产和消耗抗生素最多的国家之一。抗生素在生物体内的代谢率较低(10%~40%),绝大多数会以原药或代谢产物的形式通过粪便/尿液排出体外,对环境和人体健康构成了巨大的潜在危害。由抗生素诱导的细菌抗药性等问题已经引起社会各界的广泛重视,其中抗生素抗性基因(antibiotic resistance genes,args)已经成为当下研究的热点。
3.args是一种新型的基因污染物,其比传统的化学污染更加难以研究和控制,且可在环境中持续残留、菌群间迁移、转化和传播对生态环境的危害极大。世界卫生组织(who)已将args作为21世纪威胁人类健康的最重大挑战之一。args在各个环境介质中广泛分布,如活性污泥、土壤、生物膜、水体、沉积物、空气介质等,且可以在微生物菌属之间实现垂直传播或水平转移,并且携带args的宿主细菌(多重耐药性细菌)可以同时抵抗多种抗生素,它们的传播和扩散可能导致更广泛的疾病和感染。因此,args在各种环境中的赋存特征、传播及削减等研究已成为环境污染控制领域研究的热点问题。
4.实时荧光定量pcr(qpcr)、高通量qpcr(ht
‑
qpcr)和宏基因组测序是研究不同环境样品中args赋存特征的常用方法。但均具有一些不足:
5.(1)qpcr费时、费力且需要特定的质粒标准品(需要一系列繁杂的分子生物学过程获得),往往限制了其在定量所有args方面的使用;
6.(2)ht
‑
qpcr在通量方面比qpcr有大幅的提升,但是依旧依赖于已知的args引物,在探索环境中未知args方面有一定不足。
7.(3)利用宏基因组测序结合抗性基因数据库(ardb和card)对args进行比对注释,可以较全面的获得args的丰度数据信息。但是,宏基因组测序价格昂贵,如想获取大尺度时间和空间范围内环境样品中args的赋存特征将花费大量的经费。
8.因此,需要开发一种可以全面且经济的鉴定环境样品中args分布特征的方法。虽然可以通过基于高通量测序的功能预测软件(如picrust,phylogenetic investigation of communities by reconstruction of unobserved states)可以获得基因注释信息。但是前人研究主要基于基因组dna,获得的是“死
‑
活”微生物的群落数据,而活性微生物在args的传播方便具有重要作用。因此,非常有必要挖掘args的活性宿主细菌。叠氮溴化丙锭(propidium monoazide,pma)是一种具有高亲和力的光敏反应dna结合染料,可选择性与胞内dna发生交联反应,将其与分子生物学手段相结合,可有效的抑制死菌的dna扩增信号,从而实现活微生物的检测。综上,提出“基于pma高通量测序技术结合picrust”的方法可以实
现全面、低成本的args信息及其活性宿主细菌的鉴定,使我们对环境中args的污染和传播有更深入和系统的理解,为其控制和削减提供依据。
技术实现要素:
9.本发明目的在于提供一种全面、经济的环境样品中args及其潜在活性宿主细菌的鉴定方法。
10.具体步骤包括:
11.1)环境样品采集:
12.依据不同环境样品(活性污泥、空气/颗粒物、土壤、生物膜等)的采样方法及要求进行样品采集。
13.2)环境样品pma处理:
14.在提取环境样品基因组dna之前,采用叠氮溴化丙锭(pma)对环境样品进行预处理,主要包括:pma试剂配制与储存;pma预处理;pma光交联操作;
15.其中,pma(2mm)试剂配制与储存如下:取1mg pma溶于980μl milliq,冰箱避光保存(
‑
20℃);pma预处理步骤如下:将2mm pma试剂解冻,准确量取一定体积的pma溶液与一定量的环境样品混合,使pma的终浓度维持在3
‑
4μg/ml,充分混匀后,避光培养15min,每隔4
‑
5min颠倒混匀,使pma最大限度进入受损细胞;pma光交联操作:将培养的环境样品置于蓝光交联装置(460nm)或将样品置于冰上,选用500w卤素灯曝光,时间为18min,距光源距离为20cm进行交联操作,之后进行离心操作(10000g,5min)。
16.3)pma
‑
高通量测序:
17.取一定量经步骤2)处理的环境样品,使用dna提取试剂盒,提取基因组dna,并利用微量紫外分光光度计测定其浓度,进而使用1%脂糖凝胶电泳检测抽提基因组dna的质量;选取细菌16s v3
‑
v4区进行高通量测序分析;测序分析主要包括:根据样品数量,设计合成引物接头、pcr扩增和产物纯化、pcr产物定量和均一化、测序文库制备及库建、上机测序和下机分析;
18.4)活性非致病菌和致病菌的注释:
19.通过去除测序原始序列中的低质量序列和嵌合体进行后续分析;有效序列经均一化处理后,进行物种注释,得到环境样品的微生物群落组成信息;基于病原菌毒力因子数据库(virulence factor database,vfdb,http://www.mgc.ac.cn/vfs/main.htm)提供的病原菌菌属信息从测序结果中将潜在致病菌筛选出来,从而实现活性非致病菌和致病菌的鉴定。
20.其中,vfdb中的致病菌属包括:acinetobacter、aeromonas、anaplasma、bacillus、bartonella、bordetella、brucella、burkholderia、campylobacter、chlamydia、clostridium、corynebacterium、coxiella、enterococcus、escherichia、francisella、haemophilus、helicobacter、klebsiella、legionella、listeria、mycobacterium、mycoplasma、neisseria、pseudomonas、rickettsia、salmonella、shigella、staphylococcus、streptococcus、vibrio和yersinia。
21.5)args鉴定:
22.基于picrust对高通量测序获得的细菌16s序列进行分析;通过四个脚本命令
(normalize_by_copy_number.py,predict_metagenomes.py,categorize_by_function.py和metagenome_contributions.py),得到(kegg)orthologs(kos)数据。进而,依据args对应的特定kos从上述数据中将args挑出来,实现对环境样品不同类型抗生素对应args的鉴定。
23.其中,不同类型抗生素args的kos如下:aac(k18816);aac3
‑
vi(k19277);aac(6
′
)
‑
ii(k18815);aac(6
′
)
‑
iy(k18816);aaca/aphd(k00663);aacc(k19277);aacc1(k03395);aacc4(k00663);aacc2(k00662);aada1(k00984);aade(k05593);spcn(k18844);ampc(k01467,k20319,k20320);bla1(k17836);blacmy
‑2–
1(k19096);blactx
‑
m
‑
14(k17836);blactx
‑
m
‑
15(k18767);blashv
‑
11(k18699);blatem
‑
1(k18698);blaz(k18766);cepa(k17836);cfxa(k01624);pbp2x(k12556);pbp5(k18149);pena(k03587);acrf(k18142);adea/cmea(k03585);mexd(k18296);catb3(k00638);cmla1(k18552);cmx(k18553);emrb/qaca(k03446);mexe(k19586);mexf(k19585);oprj(k08721);yidy/mdtl(k08163);basr(k07771);qnra/b/c/d/s(k18555);qepa(k08167);oqxa(k19586);oqxb(k19585);oqxb(k19594);erea(k06880);erm(34)/(35)/(36),ermb/c/f/x(k00561);ermk(k03543);lmra(k18939);lnua(k19545);mdta(k07799);mefa(k08217);mphb(k06979);vgab(k18231);vgb(k18235);sul1(k18974);sul2(k18824);tetl/k(k08168);tetm/o/w/p/q/s/t(k18220);teta(k08151);tetg/h/j(k08151);tetk(k08168);tetv(k18215);tetx(k18221);vanhb(k18347);vanrb(k18344);vansb(k18345);vantg(k18348);vanwb(k18346);vanxb(k08641);vanyb(k07260);vanj(k18353);vanw(k18346);vans(k18351);vank(k18354);vrar(k07694);acrr(k03577);catb8(k00638);emrd(k08154);fosx(k21252);marr(k03712);mdte/yhiu(k18898);mtrcde(k18991);putitive multidrug gene(k07799);emrb/qaca(k07786);qacedelta1(k18975);qach(k03297);sdeb(k19585);emrk(k07797);ttga(k03585);ttgb(k18324);ycee/mdtg(k08161);ycel/mdth(k08162);bcr/tcab(k07552);lmrp(k08152);blt(k08153);tpo1(k08157);mdr1/flr1/caf5(k08158);atr1(k08165);yebq(k08169);norb/norc(k08170);yitg/ymfd/yfmo(k08221);tc.smr3(k09771);ebra(k11814);ebrb(k11815);mdto(k15547);mdtd(k18326);lmrs(k18934);sdrm(k18935);mdea(k18936);bmr(k19578);marc(k05595);marb(k13630);ykkc(k18924)。
24.6)args潜在活性宿主细菌鉴定:
25.经过步骤3)、4)和5)后,得到环境样品中活性非致病菌、潜在致病菌和args数据,进一步通过网络分析确定args的潜在活性宿主细菌。网络分析包括:采用spearman等级相关系数非参数相关方法(spearmans’rank correlation coefficients,srcc)对活性非致病菌、潜在致病菌和args数据进行相关性分析,进一步筛选srcc≥0.6,p<0.05的结果,通过网络分析进行可视化分析,得到args的潜在宿主细菌。
26.本发明的有益效果为:本发明提供的args及其活性宿主细菌(非致病菌和致病菌)的鉴定方法,可以全面且经济的确定大尺度范围的大批量环境样品中args的赋存特征及传播风险;同时,基于上述结果筛选出典型且具有代表性的环境样品,利用其它常用的方法(如qpcr、ht
‑
qpcr和宏基因组测序)进行args的定量分析。综合分析上述结果,可以为不同环境中args的削减及控制提供坚实的基础数据和生物学依据,具有非常重要的意义。
附图说明
27.下面结合附图对本发明的具体实施方式做进一步详细说明。
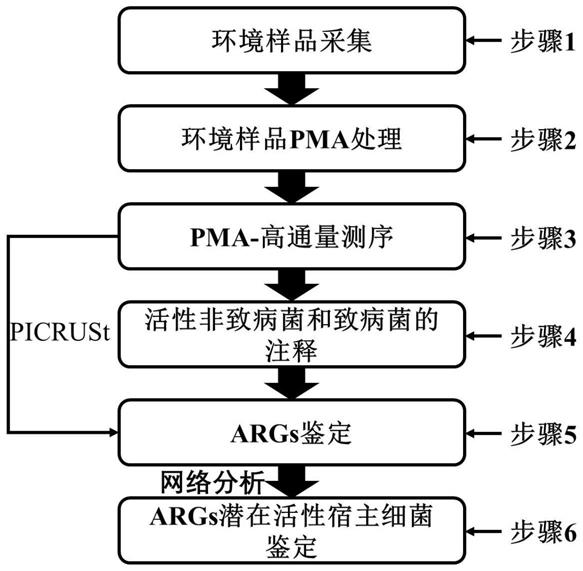
28.附图1:环境样品args及其活性宿主细胞鉴定示意图;
29.附图2:不同环境样品中args鉴定结果;
30.附图3:不同环境样品中args的潜在活性宿主细胞鉴定结果。
具体实施方式
31.:为了使本发明的技术方案更加清楚明白,下面结合具体实例对本发明进行详细说明。但本发明并不限于以下实施例。
32.实施例1
33.1.环境样品
34.环境样品为取自北京市某再生污水处理厂曝气池的活性污泥。
35.2.环境样品pma处理
36.在提取环境样品基因组dna之前,采用叠氮溴化丙锭(pma)对活性污泥进行预处理,主要包括:pma试剂配制与储存;pma预处理;pma光交联操作。其中,pma(2mm)试剂配制与储存如下:取1mg pma溶于980μl milliq,冰箱避光保存(
‑
20℃);pma预处理步骤如下:将2mm pma试剂解冻,准确量取一定体积的pma溶液与一定量的活性污泥混合,使pma的终浓度维持在3
‑
4μg/ml,充分混匀后,避光培养15min,每隔4
‑
5min颠倒混匀,使pma最大限度进入受损细胞;pma光交联操作:将培养的环境样品置于蓝光交联装置(460nm)或将样品置于冰上,选用500w卤素灯曝光,时间为18min,距光源距离为20cm进行交联操作,之后进行离心操作(10000g,5min)。
37.3.pma
‑
高通量测序
38.取一定量经步骤2)处理的活性污泥样品,使用dna提取试剂盒,按照标准流程提取基因组dna,并利用微量紫外分光光度计测定其浓度,进而使用1%脂糖凝胶电泳检测抽提基因组dna的质量。选取细菌16s v3
‑
v4区进行高通量测序分析。测序分析在测序公司完成,主要包括:根据样品数量,设计合成引物接头、pcr扩增和产物纯化、pcr产物定量和均一化、测序文库制备及库建、上机测序和下机分析。
39.4.活性非致病菌和致病菌的注释
40.通过去除测序原始序列中的低质量序列和嵌合体进行后续分析。有效序列经均一化处理后,进行物种注释,得到活性污泥的微生物群落组成信息。基于病原菌毒力因子数据库(virulence factor database,vfdb,http://www.mgc.ac.cn/vfs/main.htm)提供的病原菌菌属信息从测序结果中将潜在致病菌筛选出来,从而实现活性非致病菌和致病菌的鉴定。其中,vfdb中的致病菌属包括:acinetobacter、aeromonas、anaplasma、bacillus、bartonella、bordetella、brucella、burkholderia、campylobacter、chlamydia、clostridium、corynebacterium、coxiella、enterococcus、escherichia、francisella、haemophilus、helicobacter、klebsiella、legionella、listeria、mycobacterium、mycoplasma、neisseria、pseudomonas、rickettsia、salmonella、shigella、staphylococcus、streptococcus、vibrio和yersinia。
41.5.args鉴定
42.基于picrust对高通量测序获得的细菌16s序列进行分析,通过四个脚本命令(normalize_by_copy_number.py,predict_metagenomes.py,categorize_by_function.py和metagenome_contributions.py),得到(kegg)orthologs(kos)数据。进而,依据args对应的特定kos从上述数据中将args筛选出来,实现对活性污泥样品不同类型抗生素对应args的鉴定。
43.其中,不同类型抗生素args的kos如下:aac(k18816);aac3
‑
vi(k19277);aac(6
′
)
‑
ii(k18815);aac(6
′
)
‑
iy(k18816);aaca/aphd(k00663);aacc(k19277);aacc1(k03395);aacc4(k00663);aacc2(k00662);aada1(k00984);aade(k05593);spcn(k18844);ampc(k01467,k20319,k20320);bla1(k17836);blacmy
‑2–
1(k19096);blactx
‑
m
‑
14(k17836);blactx
‑
m
‑
15(k18767);blashv
‑
11(k18699);blatem
‑
1(k18698);blaz(k18766);cepa(k17836);cfxa(k01624);pbp2x(k12556);pbp5(k18149);pena(k03587);acrf(k18142);adea/cmea(k03585);mexd(k18296);catb3(k00638);cmla1(k18552);cmx(k18553);emrb/qaca(k03446);mexe(k19586);mexf(k19585);oprj(k08721);yidy/mdtl(k08163);basr(k07771);qnra/b/c/d/s(k18555);qepa(k08167);oqxa(k19586);oqxb(k19585);oqxb(k19594);erea(k06880);erm(34)/(35)/(36),ermb/c/f/x(k00561);ermk(k03543);lmra(k18939);lnua(k19545);mdta(k07799);mefa(k08217);mphb(k06979);vgab(k18231);vgb(k18235);sul1(k18974);sul2(k18824);tetl/k(k08168);tetm/o/w/p/q/s/t(k18220);teta(k08151);tetg/h/j(k08151);tetk(k08168);tetv(k18215);tetx(k18221);vanhb(k18347);vanrb(k18344);vansb(k18345);vantg(k18348);vanwb(k18346);vanxb(k08641);vanyb(k07260);vanj(k18353);vanw(k18346);vans(k18351);vank(k18354);vrar(k07694);acrr(k03577);catb8(k00638);emrd(k08154);fosx(k21252);marr(k03712);mdte/yhiu(k18898);mtrcde(k18991);putitive multidrug gene(k07799);emrb/qaca(k07786);qacedelta1(k18975);qach(k03297);sdeb(k19585);emrk(k07797);ttga(k03585);ttgb(k18324);ycee/mdtg(k08161);ycel/mdth(k08162);bcr/tcab(k07552);lmrp(k08152);blt(k08153);tpo1(k08157);mdr1/flr1/caf5(k08158);atr1(k08165);yebq(k08169);norb/norc(k08170);yitg/ymfd/yfmo(k08221);tc.smr3(k09771);ebra(k11814);ebrb(k11815);mdto(k15547);mdtd(k18326);lmrs(k18934);sdrm(k18935);mdea(k18936);bmr(k19578);marc(k05595);marb(k13630);ykkc(k18924)。
44.6.args潜在活性宿主细菌鉴定:
45.经过步骤3)、4)和5)后,得到活性污泥样品中活性非致病菌、潜在致病菌和args数据,进一步通过网络分析确定args的潜在活性宿主细菌。网络分析包括:采用spearman等级相关系数非参数相关方法(spearmans’rank correlation coefficients,srcc)对活性非致病菌、潜在致病菌和args数据进行相关性分析,进一步筛选srcc≥0.6,p<0.05的结果,通过网络分析进行可视化分析,得到args的潜在宿主细菌。
46.实验1的基于高通量测序和picrust的环境样品中抗生素抗性基因及其潜在宿主细胞的鉴定方法如图1所示;基于得到kos数据并依据本方法提及的args对应的特定kos,将活性污泥样品中可能存在的args筛选出来如图2所示;对活性非致病菌、潜在致病菌和args数据建立的网络分析如图3所示。
47.图2是通过pma预处理的活性污泥样品,经高通量测序与args数据集比对存在的不
同种类的args的含量情况;从图2中可以看出,含量在各个数量级(甚至在超痕量级,常量级等)均能筛选得出活性污泥样品中存在不同种类的args,其中检测出10种氨基糖苷类args(含量在101‑
103数量级),12种氟喹诺酮类args(含量在101‑
105数量级),5中beta
‑
内酰胺类args(含量在101‑
104数量级),3中磺胺类args(含量在101‑
102数量级),8中mlsb类args(含量在101‑
104数量级),5种四环素类args(含量在101‑
104数量级),5中万古素类args(含量在101‑
103数量级),12中多药耐药性蛋白类args(含量在101‑
104数量级)以及11中未分类args(含量在101‑
104数量级)。
48.图3是通过筛选得到的活性污泥样品中args,与活性非致病菌,活性潜在致病菌建立的网络分析。从图3中可以看出,通过基于pma高通量测序和picrust的环境样品中抗生素抗性基因及其潜在宿主细胞的鉴定方法,根据活性细菌与args的连接度大小(>6),共鉴定出7中潜在的致病菌,包括acinetobacter,aeromonas,bacillus,burkholderia,mycobacterium,streptococcus和yersinia,18种活性非致病菌,如hyphtomicrobium,haliangium,stenotropltobacter,ferruginibacter等。
49.分析可知,使用基于高通量测序和picrust的环境样品中抗生素抗性基因及其潜在宿主细胞的鉴定方法能够得到活性污泥样品中存在的不同种类的args,且确定潜在的宿主细胞。
50.通过参考示范性实施例,本发明的目的和功能以及用于实现这些目的和功能的方法将得以阐明。然而,本发明并不受限于以上所公开的示范性实施例;可以通过不同形式来对其加以实现。说明书的实质仅仅是帮助相关领域技术人员综合理解本发明的具体细节。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。