variation,cnv)的生物标志物预测卵巢癌同源重组缺陷(homologous recombination deficiency,hrd)的方法及应用。
7.为实现上述目的,本发明解决其技术问题所采用的技术方案如下:
8.一种预测卵巢癌同源重组缺陷的基于基因组拷贝数变异的生物标志物,所述生物标志物为基于染色体片段5q13.2、8q24.2或19q12中任意一种的cnv。
9.进一步地,所述生物标志物为5q13.2的删失、8q24.2的扩增或者19q12的扩增中的任意一种。
10.本发明还提供所述的生物标志物在预测卵巢癌患者同源重组缺陷中的应用。
11.本发明还提供所述的生物标志物在制备预测卵巢癌患者同源重组缺陷的生物标志物中的应用。
12.另外,本发明提供所述的生物标志物的产品在制备预测卵巢癌患者同源重组缺陷产品中的应用。
13.本发明还提供了一种预测卵巢癌同源重组缺陷的方法,其特征在于,采用上述的生物标志物进行预测。
14.进一步地,包括以下步骤:
15.s1.收集卵巢癌患者的手术切除样本或组织活检样本,包括肿瘤组织和癌旁组织;
16.s2.对步骤s1所取得的样本进行dna测序,得到相应的测序文件;
17.s3.利用gistic 2.0对dna测序文件进行分析,得到肿瘤全基因组的cnv图谱;
18.s4.基于染色体片段5q13.2、8q24.2或19q12的cnv预测卵巢癌的hrd状态:如果5q13.2的gistic值为
‑
1(单拷贝删失)或
‑
2(纯合性删失),或8q24.2的gistic值为1(低水平扩增)或2(高水平扩增),或19q12的gistic值为0(二倍体正常拷贝),则判断患者存在同源重组缺陷;若5q13.2、8q24.2的giscitc值为0(二倍体正常拷贝),而19q12的gistic值为1(低水平扩增)或2(高水平扩增)则判断患者无同源重组缺陷,gistic值由gistic 2.0计算得到。
19.另外,本发明还提供基于基因组拷贝数变异的生物标志物在预测泛癌同源重组缺陷的方法,所述方法主要步骤如下:
20.s1.收集癌症患者的手术切除样本或组织活检样本(包括肿瘤组织和癌旁组织)。
21.s2.对s1所取得的样本进行dna测序,得到相应的测序文件。
22.s3.利用gistic 2.0对dna测序文件进行分析,得到肿瘤全基因组的cnv面貌。
23.s4.基于染色体片段8q24.2的扩增或基因myc、ndrg1的扩增预测肿瘤的hrd状态:如果8q24.2扩增或基因myc扩增或基因ndrg1扩增,则判断患者存在同源重组缺陷;其余情况则判断患者无同源重组缺陷。
24.与现有技术相比,本发明的有益效果是:
25.1.本发明丰富了亚染色体和基因层面的同源重组缺陷生物标志物,还为跨肿瘤研究患者的hrd状态提供了一个新的视角。与现有技术相比,拷贝数变异对于同源重组缺陷的预测更加稳定,不易受到回复突变的影响。
26.2.本发明所提供的基于拷贝数变异预测卵巢癌同源重组缺陷的方法目前尚未见报道,可以实现特异有效预测并具有良好的准确性,这为卵巢癌的同源重组缺陷状态评估和预测提供了依据和基础。对于及时采取有效的临床措施、制定个体化的诊治方案,最终提
高卵巢癌患者生存率具有重要的价值和意义。
附图说明
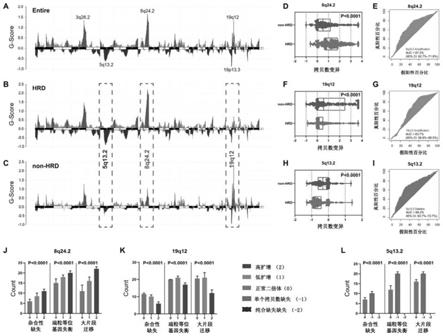
27.图1为本发明实施例1的基因组拷贝数变异的富集分数、拷贝数值在hrd组和非hrd组之间的差异以及拷贝数变异与hrd评分的关系。其中,a为来自tcga
‑
ov队列的所有卵巢癌患者的基因组拷贝数变异的富集分数,b、c分别为hrd组(b)和非hrd组(c)中cnv的富集度评分,d、f、h为通过gistic 2.0计算的8q24.2(d)、19.12(f)和5q13.2(h)的拷贝数值在hrd组和非hrd组之间的差异,e、g、i为8q24.2扩增(e)、19q12扩增(g)和5q13.2缺失(i)在预测hrd/非hrd方面的表现,j、k、l为8q24.2(j)、19q12(k)和5q13.2(l)的拷贝数变异与hrd评分的三个组成指标loh、tai和lst之间的关系。
28.图2为基于拷贝数变异生物标志物预测卵巢癌同源重组缺陷的方法在澳大利亚卵巢癌研究(the australian ovarian cancer study,aocs)队列中的验证。其中,a为aocs队列的所有卵巢癌患者基因组cnv的富集程度,b
‑
g为根据brca1/2突变、brca1启动子甲基化和其他同源重组修复基因突变(包括rad51c、chek2、brip1、pten),将aocs队列中的卵巢癌患者分为hrd组和非hrd组,b、d、f为通过gistic 2.0计算得到8q24.2(b)19q12(d)和5q13.2(f)的拷贝数值在hrd组和非hrd组之间的差异。c、e、g为8q24.2扩增(c)、19q12扩增(e)和5q13.2缺失(g)在预测hrd/非hrd中的表现。
29.图3为实施例2基于拷贝数变异的泛癌同源重组缺陷预测方法的应用。其中,a为tcga pan
‑
cancer队列中33种肿瘤的平均hrd评分和hrd频率(≥42为临界值),b为9种平均hrd评分最高的肿瘤中8q24.2、19q12扩增和5q13.2缺失的cnv富集程度,c为所有tcga肿瘤中hrd组和非hrd组8q24.2拷贝数值的小提琴图,d为7种肿瘤中hrd组和非hrd组8q24.2的拷贝数值。
30.图4为实施例3基因myc和ndrg1的扩增与泛癌同源重组缺陷的关联。其中,a为tcga中位于8q24.2的基因扩增频率,b为不同肿瘤中myc的变异类型和频率,c为myc的扩增与肿瘤的平均hrd评分的线性回归图,d为不同肿瘤中ndrg1的变异类型和频率,e为ndrg1的扩增与肿瘤的平均hrd评分的线性回归图。
31.图5为本发明研究设计的流程图。
具体实施方式
32.为了更好的说明本发明的目的、技术方案和优点,下面将结合附图和具体实施例对本发明作进一步说明,但下述的实例仅仅是本发明的简易例子,并不代表或限制本发明的权利保护范围,本发明的保护范围以权利要求书为准。
33.实施例1
34.一种基于拷贝数变异来预测卵巢癌同源重组缺陷方法的应用
35.本发明研究的流程如图5所示,实施例1将来自tcga
‑
ov队列的587名卵巢癌患者纳入该研究,其临床特征和dna测序数据来自https://www.cancer.gov/tcga。
36.根据hrd
‑
score(hrd评分)将587名患者分入hrd组(hrd评分≥42)和非hrd组(hrd评分<42),hrd评分是杂合性丢失(loss of heterozygosis,loh)、大片端迁移(large
‑
scale state transition,lst)和端粒等位基因不平衡(telomeric allelic imbalance,
tai)的未加权总和。
37.使用gistic 2.0测量肿瘤全基因组亚染色体水平的cnv图谱,tcga
‑
ov队列的segment文件(记录基因组各位点的拷贝数)被用作gsitic 2.0的输入,以量化每个肿瘤基因组中染色体片段的cnv状态,结果如图1a所示,卵巢癌患者的染色体片段3q26.2、8q24.2和19q12显著扩增,而染色体片段5q13.2和19p13.3则显著缺失。
38.进一步比较hrd组和non
‑
hrd组cnv的状态,如图1b,1c所示,结果表明与non
‑
hrd组相比,hrd组的5q13.2显著缺失、8q24.2显著扩增,而non
‑
hrd组的19q12显著扩增。
39.基于上述结果,比较了染色体片段5q13.2、8q24.2和19q12的拷贝数值在hrd组和non
‑
hrd组中的差异,并探究5q13.2、8q24.2或19q12的拷贝数值是否为hrd状态的显著预测因子。结果发现,hrd组的8q24.2的扩增(图1d,p<0.0001)和5q13.2的缺失(图1h,p<0.0001)显著高于非hrd组,auc值分别为67.3%(图1e)和68.2%(图1i)。而非hrd组的19q12的扩增值显著高于hrd组(图1f,p<0.0001),auc值为63.7%(图1g)。
40.综上可得,5q13.2、8q24.2或19q12的cnv具有卵巢癌同源重组缺陷状态预测潜力,如果5q13.2的gistic值为
‑
1(单拷贝删失)或
‑
2(纯合性删失),或8q24.2的gistic值为1(低水平扩增)或2(高水平扩增),或19q12的gistic值为0(二倍体正常拷贝),则判断患者存在同源重组缺陷;若5q13.2、8q24.2的gistic值为0(二倍体正常拷贝),而19q12的gistic值为1(低水平扩增)或2(高水平扩增)则判断患者无同源重组缺陷(gistic值由gistic 2.0计算得到)。
41.为了证实这三个片段的cnv对hrd的预测价值,本发明探讨了5q13.2、8q24.2和19q12的缺失或扩增与hrd
‑
core的三个组成部分loh、tai和lst之间的关系。
42.结果表明,8q24.2的扩增程度(图1j,p<0.0001)和5q13.2片段的缺失程度(图1l,p<0.0001)越大,loh、tai和lst的计数越多。19q12片段的扩增与loh、tai和lst呈负相关,其差异主要体现在高水平扩增,而二倍体正常拷贝与低水平扩增之间无明显差异。(图1k,p<0.0001),这与图1d
‑
i的结果一致。
43.为了进一步明确5q13.2、8q24.2和19q12三个片段的缺失或扩增可以作为预测卵巢癌hrd特异性生物标志物,我们在澳大利亚卵巢癌研究(the australian ovarian cancer study,aocs)队列中对上述三个基因拷贝数变异的标志物进行外部验证。
44.通过对aocs队列的所有卵巢癌患者基因组cnv的富集程度分析,我们发现在aocs队列中8q24.2和19q12存在显著扩增,在所有拷贝数扩增片段中排名靠前,同时5q13.2片段亦存在显著缺失(图2a)。此外,我们还观察到aocs队列中hrd组的8q24.2扩增(图2b,p<0.0001)、19q12扩增(图2d,p=0.0034)和5q13.2缺失(图2f,p=0.0056)显著高于非hrd组,auc分别为75.0%(图2c)、68.9%(图2e)和67.9%(图2g)
45.因此,本实例中确认了5q13.2、8q24.2或19q12的cnv作为卵巢癌同源重组缺陷的生物标志物。
46.实施例2
47.一种基于拷贝数变异的生物标志物预测泛癌同源重组缺陷的应用
48.对来自tcga pan
‑
cancer队列的含33种癌症类型的10635例患者的dna测序结果分析,将cnv对于卵巢癌同源重组缺陷的预测作用推广到其他癌种。
49.计算每种肿瘤的平均hrd分数,得到不同肿瘤中同源重组缺陷状态的分布,发现卵
巢浆液性囊腺癌(ov)、子宫癌肉瘤(ucs)、肺鳞癌(lusc)、肉瘤(sarc)、食道癌(esca)的患者。胃腺癌(stad)、膀胱尿道癌(blca)、乳腺癌(brca)和肺腺癌(luad)的hrd分数主要较高,而甲状腺癌(thca)和急性骨髓性白血病(aml)患者的hrd分数最低(图3a)。这与先前研究认为的卵巢癌和乳腺癌中较高的同源重组缺陷频率一致,并证实了同源重组缺陷的普遍性。
50.为了研究cnvs是否可用于泛癌的同源重组缺陷检测,探讨了9种hrd得分最高的肿瘤中8q24.2的扩增、19q12的扩增和5q13.2的缺失的cnv频率(图3b)。可以发现,在这些癌症中,8q24.2扩增普遍富集(肉瘤除外),而19q12扩增最常发生在卵巢癌和胃腺癌中,但5q13.2缺失只体现在卵巢癌中,说明8q24.2的扩增能够跨肿瘤类型预测同源重组缺陷,而5q13.2的缺失是卵巢癌同源重组缺陷特有的特征。
51.参照实施例1对卵巢癌患者进行分组的方法,将10,635例患者根据hrd评分分入hrd组和非hrd组。重点研究染色体片段8q24.2,发现在tcga pan
‑
cancer队列中,与非hrd患者相比,hrd患者的该片段拷贝数值明显较高(图3c,p<0.0001),这在肺鳞癌(p<0.0001)、膀胱尿道癌(p=0.0102)、肺腺癌(p=0.0035)、乳腺癌(p<0.0001)和胃腺癌(p<0.0001)分别得到证实(图3d)。这些结果共同为8q24.2的cnv对不同癌症类型的同源重组缺陷的预测作用提供了重要证据。
52.因此,本实例中确认了8q24.2的cnv作为泛癌同源重组缺陷的生物标志物。
53.实施例3
54.基因myc和ndrg1的扩增与泛癌同源重组缺陷的关联
55.对tcga pan
‑
cancer队列中8q24.2的基因扩增频率进行了分析,以寻求基因与hrd的相关性。
56.如图4a所示,8q24.2内的基因在肿瘤中扩增率较高,包括致癌基因myc和ndrg1。
57.鉴于致癌基因与治疗和肿瘤生物学的紧密联系,统计了所有tcga肿瘤中myc和ndrg1的变异类型及其频率,发现myc和ndrg1的主要变异类型正好是扩增,myc和ndrg1的变异频率最高的肿瘤都是卵巢癌,而在甲状腺癌中的变异频率较低,这与图3a中卵巢癌的hrd评分最高,而甲状腺癌的hrd评分较低相一致(图4b,d)。
58.利用泛癌数据研究了myc和ndrg1的扩增频率与hrd评分之间的关系,结果显示:myc和ndrg1的扩增与hrd评分明显相关(myc:rho=0.6809,p=0.0001;ndrg1:rho=0.6453,p=0.0004;图4c,e)。
59.因此,本实例提示8q24.2扩增片段中myc和ndrg1的扩增可能作为泛癌同源重组缺陷的生物标志物。
60.以上各实施例仅用以说明本技术的技术方案,而非对其限制;本技术领域的普通技术人员应当理解:其依然可以对前述各实施例所记载的技术方案进行修改,或者对其中部分或者全部技术特征进行等同替换;这些修改或者替换也应视为本发明的保护范围。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。