一种基于ngs的结直肠癌基因突变检测分析方法
技术领域
1.本发明属于基因检测技术领域,具体涉及一种基于ngs的结直肠癌基因突变检测分析方法。
背景技术:
2.根据结直肠癌分子标志物临床检测专家共识,对于所有mcrc患者,均推荐在综合治疗前行常规分子标志物检测(ras基因突变、braf基因突变、 msi状态/mmr蛋白表达),根据结果制定个体化治疗方案;推荐对于所有临床怀疑林奇综合征的患者,检测msi状态/ mmr蛋白表达进行遗传筛查。
3.目前检测结直肠癌常见的方式是:(1)一代测序检测相关若干结直肠癌相关基因热点;(2)ngs小panel针对结直肠癌相关若干基因进行靶向扩增/杂交捕获结合测序检测。如申请号为cn202110013800.7的文件公开了一种结直肠癌基因突变位点的检测方法及其试剂盒,通过pcr引物设计使得最终pcr扩增体系中具有至少140重位点,然后对产物进行信号收集,同时队血清、血浆、外周血、胸腔积液、体液或者组织来源的dna进行突变检测比对,来综合评估患者的基因组变异情况。该方法有检测周期长、样本要求高等缺陷。
技术实现要素:
4.针对上述存在的问题,本发明的目的是提供一种基于ngs的结直肠癌基因突变检测分析方法,根据结直肠癌及相关病症相关的基因和位点对样本进行杂交捕获并测序,特异性针对该实验设计研发的数据分析流程分析样本下机数据,获取其中的基因突变信息(点突变、插入缺失、拷贝数变异及染色体易位)及微卫星稳定性相关信息并自动化生成检测报告,从而对突变状况及潜在靶向用药位点作出全面评估及指导。
5.为了实现上述目的,本发明的技术方案如下:一种基于ngs的结直肠癌基因突变检测分析方法,包括以下步骤:s1、采用探针捕获的方法对正常样本和标准突变样本提取的dna进行建库和测序,得到原始样本fq数据;s2、对s1步骤得到的原始样本fq数据通过fastp预处理,除去低质量reads及接头污染reads;s3、对步骤s2得到的样本数据通过bwa mem比对至参考基因组,对原始bam文件去重,然后使用gatk best practice对bam文件进行碱基质量重校正,得到供变异检测的样本数据bam文件;s4、分别采用mutect2工具和lumpy软件对s3步骤得到的样本数据进行体细胞突变检测和融合基因检测:在体细胞突变检测中通过构建panel of normals过滤无意义/良性胚系变异,在融合基因检测中通过以正常样本数据为基线去除背景变异;最后通过cnvkit软件检测拷贝数变异,得到snv/indel/sv/cnv等变异类型的vcf结果文件及过滤后的变异列表;
s5、通过msisensor-pro软件,以正常样本数据为基础,检测各个样本的msi状态,其中结果大于等于10%即为msi-h,小于10%为msi-l;s6、分析步骤s4、s5中得到的变异结构以msi信息,生成检测报告。
6.作为本发明的进一步优选,s1步骤中探针序列的设计包括:确定目标捕获区域并得到bed文件,根据bed文件通过catch软件设计多个探针序列,将设计的探针序列通过blastn软件和参考基因组比对,找出多处比对到参考基因组的序列去除,得到探针序列。
7.作为本发明的进一步优选,目标捕获取区域的确定根据数据库和指南中关于结直肠癌相关基因和关键区域得到。相关的基因(包含结直肠癌密切相关22个基因)的确定来源于:数据库和指南包括oncokb数据库、nccn指南和csco指南)中关于肺癌/结直肠癌/乳腺癌等实体瘤相关基因、化疗相关位点、hrd相关基因、林奇综合征相关基因。
8.综上所述,本发明具有以下有益效果:本发明提供的检测分析方法囊括了结直肠癌及相关病症的绝大部分基因和位点,并且可在缺少正常对照组织的情况下准确评估患者msi信息,相比传统结直肠癌检测方法具有样本要求低、操作便捷、检测费用适中和检测范围广(包含snv/indel/cnv/sv/msi等变异类型)等优点。
具体实施方式
9.一种基于ngs的结直肠癌基因突变检测分析方法,具体包括以下步骤:(1)根据目标基因确定目标捕获区域,生成捕获区域bed文件。
10.(2)根据bed文件和人类hg19参考基因组,通过catch软件设计目标区域探针序列(参考文献:metsky h c ,siddle k j ,gladden-young a , et al. capturing sequence diversity in metagenomes with comprehensive and scalable probe design[j]. nat biotechnol, 2019:160-168.);其中,catch软件运行方法如下:nohup design.py
ꢀ‑
pl 120
ꢀ‑
l 60
ꢀ‑
m 5
ꢀ‑
e 5
ꢀ‑
o crc.fasta&& \makeblastdb
ꢀ‑
in crc.fasta
ꢀ‑
out blast/crc.fasta
ꢀ‑
dbtype nucl && \echo
ꢀ‑
e "qseqid\tsseqid\tpident\talignment_length\tmismatch\tgapopen\tqstart\tqend\tsstart\tsend\tevalue\tbitscore" 》blast/crc.probe.blastn.txtblastn
ꢀ‑
num_threads 10
ꢀ‑
query probe/crc.probe.fasta
ꢀ‑
db blast/crc.fasta
ꢀ‑
evalue 1e-50
ꢀ‑
max_target_seqs 1000
ꢀ‑
outfmt 6 》》blast/crc.probe.blastn.txt && \perl filter_mulialign_probe.pl blast/crc.probe.blastn.txt probe/crc.probe.fasta probe/crc.uniq.probe.fasta设计出的探索序列通过blastn软件和hg19比对,找出多处比对到hg19的序列去除,得到探针序列如表1所示。
[0011]
(3)对正常样本和标准突变样本采用探针捕获的方法对正常样本和标准突变样本提取的dna进行建库和测序。
[0012]
(4)将得到的原始样本fq数据根据正常样本和标准突变样本进行拆分。
[0013]
(5)对所有的原始样本fq数据通过fastp预处理,除去低质量reads及接头污染
reads;预处理的方法参考文献为:chen s ,zhou y ,chen y , et al. fastp : an ultra-fast all-in-one fastq preprocessor.2018。
[0014]
(6)对步骤(5)得到的样本数据通过bwa mem比对至hg19,对原始bam文件去重,然后使用gatk best practice对bam文件进行碱基质量重校正,得到供变异检测的样本数据bam文件。
[0015]
(7)对步骤(6)得到的各个样本进行如下处理:a.采用mutect2工具检测体细胞突变(snv/indel):建立正常样本的数据集作为本底过滤(pon, panel of normals),分析并过滤无意义/良性的胚系变异;b.lumpy软件检测融合基因:以正常样本作为基线来去除背景变异,具体检测方法参考文献:layer r m ,hall i m ,quinlan a r . lumpy: a probabilistic framework for structural variant discovery[j].2012;c.最后通过cnvkit软件检测拷贝数变异,得到snv/indel/sv/cnv等变异类型的vcf结果文件及过滤后的变异列表。
[0016]
(8)通过msisensor-pro软件在正常样本数据基础上检测各个样本的msi状态(参考文献为:jia p ,yang x ,guo l , et al. msisensor-pro: fast, accurate, and matched-normal-sample-free detection of microsatellite instability[j]. 基因组蛋白质组与生物信息学报:英文版, 2020(1):7),其中结果大于等于10%即为msi-h,小于10%为msi-l。
[0017]
(9)通过实验室内部积累的结直肠癌临床相关突变解读数据集及报告程序,读取信息分析步骤7-8中变异结果及msi信息,自动生成临床检测报告。
[0018]
以上4-9步骤分析流程均已封装成自动化流程,运行时仅需提供下机文件fq列表即能自动化运行程序并输出结果。
[0019]
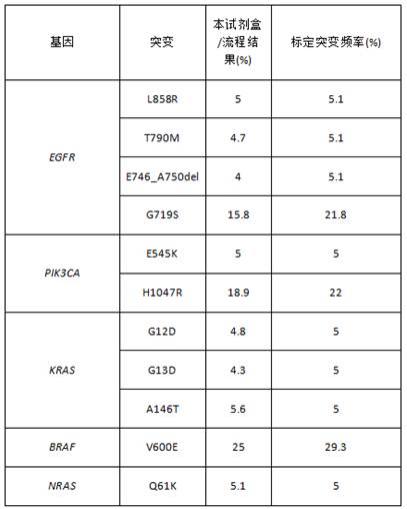
选取一例突变标准品:实验室编号s0-1(菁良,#gw-ogtm003,肿瘤snv 5% gdna标准品),采用上述实验分析步骤,得到的突变列表如下所示:
如结果所示,本发明采用的分析方法能检出标准品中所有致病性突变,检出率达到100%,且测得突变频率较为接近,近5%的较低频突变也能有效测出。
[0020]
本文中所描述的具体实施例仅仅是对本发明精神作举例说明。本发明所属技术领域的技术人员可以对所描述的具体实施例做各种各样的修改或补充或采用类似的方式替代,但并不会偏离本发明的精神或者超越所附权利要求书所定义的范围。
[0021]
表1
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。