一种识别hla
‑
a*02:01/e6
29
‑
38
的tcr及其应用
技术领域
1.本发明属于生物技术领域,具体涉及一种识别hla
‑
a*02:01/e6
29
‑
38
的tcr及其应用。
背景技术:
2.人乳头状瘤病毒(human papillomavirus,hpv)是一种嗜上皮性病毒,易感染人类表皮和粘膜鳞状上皮,并与多种癌症(宫颈癌、肛门癌、阴茎癌、阴道癌、外阴癌和头颈癌等)的发生发展相关(lancet oncol,2010,11,781;clin cancer res,2019,25,1486),在过去十多年中,因hpv感染相关的头颈癌的死亡率持续上升(ca cancer j clin,2021,71,7)。hpv病毒有几十种亚型(virology,2015,476,341),上述肿瘤主要与hpv16感染相关(j natl cancer inst,2015,107,djv086)。hpv16编码的e6蛋白作为病毒生命周期的主要蛋白之一,可通过多种作用机制诱导肿瘤发生与发展,例如抑制抑癌蛋白p53和prb,阻遏细胞凋亡(cancer sci,2007,98,1505);增强端粒酶活性使宿主细胞永生化(virus res,2017,231,50);诱导人类组织相容性抗原(hla)表达缺失(clin immunol,2005,115,295),有利于肿瘤细胞逃避宿主固有免疫应答从而导致肿瘤细胞的发生、发展、浸润、转移。hpv16
‑
e6属于肿瘤特异性抗原,只在相关肿瘤组织中特异性表达,不在正常组织中表达(nat rev cancer,2002,2,342;clin immunol,2005,115,295),因此在以hpv16
‑
e6为靶点的免疫治疗中能降低其脱靶率,并提高治疗的有效性和安全性(papillomavirus res,2018,5,46),是hpv相关癌症治疗的理想靶点。
3.t细胞受体基因工程改造的t细胞(t cell receptor gene engineered t cells,tcr
‑
t)疗法是将肿瘤抗原特异性的tcr基因转导到正常t细胞中,能够增强或重新赋予该t细胞识别肿瘤抗原的能力,特异地靶向和杀伤肿瘤细胞(science,2016,352,1337;cells,2020,9),是当前过继性细胞治疗中重要的治疗方法(immunol rev,2014,257,56)。目前开展的相关临床试验,已经证明了tcr
‑
t细胞疗法在治疗黑色素瘤、滑膜细胞肉瘤、骨髓瘤等恶性肿瘤中有显著疗效(science,2006,314,126;j clin oncol,2011,29,917;nat med,2015,21,914;blood,2017,130,1985;cancer discov,2018,8,944)。截至2021年8月,在clinicaltrials.gov网站上已有461项与tcr
‑
t相关的临床登记记录,预示着tcr
‑
t在肿瘤免疫治疗中具有巨大的潜能和开发价值。目前临床(nct02280811、nct03197025、nct03578406)已开展针对hpv16
‑
e6靶点的tcr
‑
t细胞治疗,其有效性和安全性已得到初步验证(clin cancer res,2015,21,4431;j clin oncol,2017,35,3009;j clin oncol,2019,37,2759),为hpv相关癌症的治疗提供一条新的途径。随着相关技术的发展和研究的不断深入,tcr
‑
t疗法将逐渐向高效、低毒及可操控的方向发展,在提高疗效和安全性的同时也能带来临床应用的便捷性,从而为更多肿瘤患者带来治愈的希望。
技术实现要素:
4.本发明提供一种有效识别hla
‑
a*02:01/e6
29
‑
38
靶点的tcr,采用该tcr改造的t细胞
通过高效识别hla
‑
a*02:01/e6
29
‑
38
靶点对肿瘤细胞进行特异性杀伤。
5.本发明的技术方案之一为:一种tcr,所述的tcr的α链可变区的cdr1、cdr2和cdr3分别包含如seq id no:1、seq id no:2和seq id no:3所示的氨基酸序列;
6.或者,分别包含如seq id no:7、seq id no:8和seq id no:9所示的氨基酸序列;
7.或者,分别包含如seq id no:1、seq id no:2和seq id no:15所示的氨基酸序列。
8.较佳地,所述tcr的β链可变区的cdr1、cdr2和cdr3分别包含如seq id no:4、seq id no:5和seq id no:6所示的氨基酸序列;
9.或者,分别包含如seq id no:10、seq id no:11和seq id no:12所示的氨基酸序列;
10.或者,分别包含如seq id no:16、seq id no:17和seq id no:18所示的氨基酸序列。
11.在本发明一具体实施方案中,所述α链可变区的cdr1、cdr2和cdr3分别包含如seq id no:1、seq id no:2和seq id no:3所示的氨基酸序列,且所述β链可变区的cdr1、cdr2和cdr3分别包含如seq id no:4、seq id no:5和seq id no:6所示的氨基酸序列。
12.在本发明另一具体实施方案中,所述α链可变区的cdr1、cdr2和cdr3分别包含如seq id no:7、seq id no:8和seq id no:9所示的氨基酸序列,且所述β链可变区的cdr1、cdr2和cdr3分别包含如seq id no:10、seq id no:11和seq id no:12所示的氨基酸序列。
13.在本发明另一具体实施方案中,所述α链可变区的cdr1、cdr2和cdr3分别包含如seq id no:1、seq id no:2和seq id no:15所示的氨基酸序列,且所述β链可变区的cdr1、cdr2和cdr3分别包含如seq id no:16、seq id no:17和seq id no:18所示的氨基酸序列。
14.本发明中所述tcr的α链和/或β链优选还包含框架区;其中:
15.所述α链的框架区来源于种系trav、traj以及trac,其中所述trav优选trav14或者trav17,所述traj优选traj44或者traj49;
16.所述β链的框架区来源于种系trbv、trbd、trbj和trbc,所述trbv优选trbv14、trbv28或者trbv20,所述trbd优选trbd1,所述trbj优选trbj1
‑
6、trbj1
‑
4或者trbj2
‑
7,所述trbc优选trbc1或者trbc2。
17.在本发明一较佳实施方案中,所述tcr的α链可变区含有如seq id no:19、seq id no:21或者seq id no:23所示的氨基酸序列。
18.在本发明另一较佳实施方案中,所述tcr的β链可变区含有如seq id no:20、seq id no:22或者seq id no:24所示的氨基酸序列。
19.在本发明一具体实施方案中,所述tcr的α链可变区含有如seq id no:19所示的氨基酸序列,且所述tcr的β链可变区含有如seq id no:20所示的氨基酸序列。
20.在本发明另一具体实施方案中,所述α链可变区含有如seq id no:21所示的氨基酸序列,且所述β链可变区含有如seq id no:22所示的氨基酸序列。
21.在本发明另一具体实施方案中,所述α链可变区含有如seq id no:23所示的氨基酸序列,且所述β链可变区含有如seq id no:24所示的氨基酸序列。
22.本发明中所述tcr的tcrα链较佳地还包含恒定区,所述tcrα链的恒定区优选来源于人或鼠种系。
23.本发明中所述tcr的tcrβ链较佳地还包含恒定区,所述tcrβ链的恒定区优选来源
于人或鼠种系。
24.来源于人种系的tcrα链的恒定区优选含有如seq id no:13所示的序列。
25.来源于鼠种系的tcrα链的恒定区优选含有如seq id no:27所示的氨基酸序列。
26.来源于人种系的tcrβ链的恒定区优选含有如seq id no:14或者seq id no:25所示的序列。
27.来源于鼠种系的tcrβ链的恒定区优选含有如seq id no:28所示的氨基酸序列。
28.此外,本发明中所述的tcrα链还可包括膜外区和跨膜区;较佳地,所述的tcrα链还包括胞内序列。
29.所述tcrβ链也还可包括膜外区和跨膜区;较佳地,所述的tcrβ链还包括胞内序列。
30.本发明的技术方案之二为:一种分离的核酸,其编码如本发明技术方案之一所述的tcr。
31.本发明的技术方案之三为:一种包含如技术方案之二所述核酸的载体,所述的载体优选慢病毒载体;所述的核酸在单个开放阅读框中,或者在两个不同的开放阅读框中分别编码tcrα链和tcrβ链。
32.本发明的技术方案之四为:一种含有如技术方案之二所述的核酸或者如技术方案之三所述的载体的细胞;较佳地,所述的细胞为t细胞或者干细胞,所述的t细胞优选cd8
t细胞。
33.本发明的技术方案之五为:一种提呈如技术方案之一所述的tcr的分离的或非天然存在的细胞,所述的细胞优选t细胞。
34.本发明的技术方案之六为:一种药物组合物,其含有如技术方案之一所述的tcr或者技术方案之四所述的细胞;较佳地,所述的药物组合物还包含药物上可接受的载体。
35.本发明的技术方案之七为:一种如技术方案之一所述tcr、如技术方案四所述的细胞或者技术方案之六所述的药物组合物在制备防治hpv16表达相关的肿瘤的药物中的应用;较佳地,所述的肿瘤包括宫颈癌、口咽癌、阴道癌、肛门癌及阴茎癌等。
36.在符合本领域常识的基础上,上述各优选条件,可任意组合,即得本发明各较佳实例。
37.本发明所用试剂和原料均市售可得。
38.本发明的积极进步效果在于:
39.本发明的tcr与pmhc(hla
‑
a*02:01/tihdiilecv)的亲和力高,k
d
值可达4.2e
‑
06m。对hla
‑
a*02:01
/hpv16
‑
e6
靶细胞(a375 e6
‑
1b3)有特异性的杀伤作用,且随着效靶比的增加杀伤作用增强;而对其他两个非双阳性靶细胞无明显杀伤作用。此外,gfp转导的t细胞对a375 e6
‑
1b3无明显杀伤作用。
附图说明
40.图1为hpv16
‑
e6
29
‑
38
抗原特异性双阳单克隆cd8
t细胞的分选过程。
41.图2a和图2b为e63 tcr复性后的阴离子交换层析和sds
‑
page电泳图。
42.图3a和图3b为e63 tcr复性后的凝胶过滤层析和sds
‑
page电泳图。
43.图4a和图4b为hla
‑
a*02:01/β2m/tihdiilecv复性后的阴离子交换层析和sds
‑
page电泳图;其中,分子量大的条带为hla
‑
a*02:01,分子量小的条带为β2m,tihdiilecv多
肽分子量太小,sds
‑
page上面看不到条带。
44.图5a和图5b为hla
‑
a*02:01/β2m/tihdiilecv复性后的凝胶过滤层析和sds
‑
page电泳图。
45.图6为e6
‑
pmhc生物素化后的gel shift图。
46.图7为e63 tcr亲和力测试结果图。
47.图8为e63 tcr慢病毒感染cd8
t细胞的阳性率结果图;其中,e6con为阳性tcr对照组,gfp为阴性对照组。
48.图9a和图9b为e63 tcr对负载e6
29
‑
38
或ny
‑
eso
‑1157
‑
165
多肽的t2细胞的inf
‑
γ释放图。
49.图10e63 tcr对肿瘤细胞系的inf
‑
γ释放图。
50.图11为肿瘤细胞系ldh特异性杀伤实验。
51.图12为不同来源c区对e63 tcr配对率影响的结果图。
52.图13a和图13b为e63 tcr对22例不同hla
‑
a型健康人pbmc的inf
‑
γ释放图。
53.图14为e63 tcr
‑
t动物实验的肿瘤生长曲线。
54.图15为e63 tcr
‑
t细胞给药26天后小鼠的瘤体情况。
具体实施方式
55.下面通过实施例的方式进一步说明本发明,但并不因此将本发明限制在所述的实施例范围之中。下列实施例中未注明具体条件的实验方法,按照常规方法和条件,或按照商品说明书选择。
56.另:以下实施例中,若没有特殊说明,所用细胞系均购自atcc。
57.实施例1抗原特异性αβ
‑
tcr克隆和基因序列鉴定
58.hpv16
‑
e6
29
‑
38
(tihdiilecv)抗原特异性cd8
t细胞tcr基因克隆采用的方法、试剂和耗材,主要参考curr.protoc.immunol.2002,7,1;plos one.2011,6,e27930;onco immunology.2016,5,e1175795;j vis exp.2011,8,3321;j immunol methods.2006,310,40;plos one.2014,9,e110741及其引用文献。通过免疫磁珠负筛选法从hla
‑
a*02:01基因型健康志愿者的pbmc中分离得到cd8
t细胞后,用负载hpv16
‑
e6
29
‑
38
肽段的ebv
‑
b细胞刺激cd8
t细胞,然后采用pe标记的hla
‑
a*02:01/hpv16
‑
e6
29
‑
38
四聚体及apc标记的抗cd8抗体对t细胞进行双染色,流式分选得到双阳性t细胞,并将该t细胞扩增培养至一定数量后再次进行分选(图1)。如此经过2轮的刺激培养和分选后,采用有限稀释法对双阳t细胞进行单克隆培养。增殖后的单克隆t细胞通过hla
‑
a*02:01/hpv16
‑
e6
29
‑
38
四聚体和抗cd8抗体双染色进行流式检测并分选得到hpv16
‑
e6
29
‑
38
抗原特异性单克隆t细胞。
59.采用quick
‑
rna
tm
miniprep试剂盒(zymo research,产品号r1050)对获得单克隆t细胞的总rna进行提取,并通过smarter race cdna amplification kit(clontech,产品号634923)对rna进行逆转录获得cdna。然后以cdna为模板,通过pcr扩增目标基因后连接至puc19载体,热激法转化e.coli
‑
dh5α,涂板培养过夜后,挑取单克隆菌落进行鉴定和测序,测序获得的基因序列在imgt数据库进行比对分析。共获得3个hla
‑
a*02:01/hpv16
‑
e6
29
‑
38
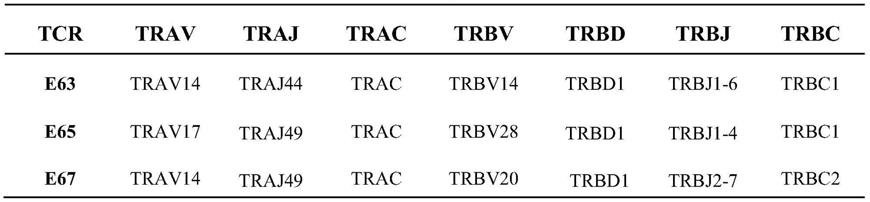
抗原特异性tcr,分别命名为e63、e65和e67 tcr,其tcrα和β链基因型见表1。
60.表1.tcrα和β链的基因型
[0061][0062]
e63 tcr的α链全长序列:
[0063][0064]
e63 tcr的β链全长序列:
[0065][0066]
e65的α链:
[0067][0068][0069]
e65的β链:
[0070][0071]
e67的α链:
[0072][0073]
e67的β链:
[0074][0075]
上述序列中:下划线标记序列为信号肽区,黑色加粗标记序列为vα(α链的可变区)或者vβ(β链的可变区)、灰色标记序列为cα(α链的恒定区)或者cβ(β链的恒定区)、斜体下划线标记序列为跨膜胞内区,加粗且下划线标记的序列为cdr序列。
[0076]
依据imgt数据库规则,e6 tcr v区的特有关键序列cdr区氨基酸序列见表2:
[0077]
表2.e6 tcr v区α和β链cdr区氨基酸序列
[0078][0079]
实施例2 e63 tcr基因的表达和纯化
[0080]
e63 tcr的α链和β链基因采用nco i/not i酶切位点分别连接到pet28a载体,热激
法转化e.coli
‑
bl21(de3),涂板培养过夜后,挑取单克隆菌落至lb培养基中,37℃振荡培养至od
600
=0.6
‑
0.8,加入终浓度为1mm的iptg诱导目的蛋白表达,37℃继续培养3h后,6000rpm离心10min收集菌体。
[0081]
用裂解液(含0.5%triton x
‑
100的1
×
pbs)重悬菌体,进行超声破碎后,12000rpm离心20min。弃上清,用裂解液重悬沉淀直至无肉眼可见的颗粒,12000rpm离心10min,重复以上操作2
‑
3次后,用6m盐酸胍溶液溶解沉淀,12000rpm离心10min后收集上清,上清即为纯化后的包涵体。采用bca法对包涵体进行定量。
[0082]
将20mg e63 tcrα链包涵体和15mgβ链包涵体分别稀释于5ml的6m盐酸胍溶液中,然后将tcrα链、tcrβ链依次缓慢加入到预冷的复性缓冲液中(science 1996,274,209;j.mol.biol.1999,285,1831;protein eng.2003,16,707),4℃下持续搅拌30min。将溶液加入透析袋中,置于10倍体积预冷的去离子水中,搅拌透析8
‑
12h后,置于预冷的透析液(ph 8.1,20mm tris
‑
hcl)于4℃透析8
‑
12h,重复2
‑
3次。
[0083]
将透析袋中的溶液取出,高速离心10min去除沉淀和气泡后,通过hitrap q hp(5ml)进行阴离子交换层析,用洗脱液(0
‑
2m nacl,20mm tris ph 8.1)线性洗脱(图2a)。分段收集含有目标蛋白组分的洗脱峰,浓缩后取样进行非还原性sds
‑
page电泳(图2b),结果显示目标蛋白纯度未满足要求,需进一步纯化。采用superdex 75 10/300对浓缩后的蛋白样品进行凝胶过滤层析(图3a),取样进行非还原性和还原性sds
‑
page电泳检测(图3b),结果显示非还原性电泳泳道除45kda附近有一条带,无其他明显杂带;还原性电泳泳道有两条带,分别是e63 tcr的α链和β链,其纯度满足后续实验需求。
[0084]
实施例3生物素化抗原肽
‑
mhc(pmhc)制备
[0085]
pmhc的复性和纯化,按照nih tetramer core facility的方法进行制备。按照在线protocols所述,将hpv16
‑
e6
29
‑
38
多肽溶液、β2m和hla
‑
a*02:01包涵体溶液依次加入复性缓冲液(0.1m tris
‑
hcl,0.4m l
‑
arginine,2mm edta,0.5mm氧化性谷胱甘肽和5mm还原性谷胱甘肽,0.2mm pmsf),4℃搅拌过夜,第二天早上和晚上分别再加入同量的hla
‑
a*02:01包涵体溶液,4℃搅拌1
‑
3天后,在10倍体积透析液(ph 8.1,20mm tris
‑
hcl)中透析3次。将透析后的蛋白样品用hitrap q hp(5ml)进行阴离子交换层析,用洗脱液(0
‑
2m nacl,20mm tris ph 8.1)线性洗脱,收集和合并洗脱峰(图4a),采用还原性sds
‑
page电泳分析,清晰可见hla
‑
a*02:01和β2m两条带(图4b),而hpv16
‑
e6
29
‑
38
多肽分子量太小,胶图看不到条带。对含有pmhc组分的洗脱峰进行浓缩,经凝胶过滤层析(superdex 75 10/300)进一步纯化(图5a),然后用还原性sds
‑
page电泳检测,从电泳图上可知,得到纯度更好的pmhc复合体(图5b)。用重组酶bira对pmhc复合体进行生物素化(protein expr.purif.2012,82,162;j.bacteriol.2012,194,1113.),然后加入链霉亲和素(sa)进行反应验证,反应体系按照nih tetramer core facility的方法进行制备和gel shift纯度鉴定。从gel shift电泳图来看(图6),e6
‑
pmhc复合体的生物素化制备成功。
[0086]
实施例4亲和力测试
[0087]
biacore是一种基于表面等离子共振(surface plasmon resonance,spr)技术检测亲和力的仪器。在本实验中,使用biacore t200,首先将生物素化的pmhc偶联在cm5芯片上,然后检测其与不同tcr的结合解离常数,计算出k
d
值。据此测试e63 tcr与pmhc(hla
‑
a*02:01/tihdiilecv)的亲和力,详见表3和图7。
[0088]
表3.e6 tcr亲和力k
d
值
[0089][0090][0091]
需说明的是:如本领域人员所知,spr技术是当前测定亲和力最常用、可靠的方法之一,但是涉及到蛋白定量、芯片新旧程度、仪器状态等,不同批次间的实验,会有一定的误差,误差值甚至可达3~5倍;而本发明为使用了同一蛋白定量、同一芯片以及同一仪器所进行的同批次实验,因此各数据之间可用于进行亲和力大小的比较,但具体数值并不构成对本发明保护范围的限制。
[0092]
实施例5 tcr慢病毒制备和转导cd8
t细胞
[0093]
1)tcr慢病毒包装
[0094]
采用第三代慢病毒包装系统(invitrogen,plenti6/v5 directional topo
tm
cloning kit,产品号k495510)包装含有编码目的tcr基因的慢病毒。将包装质粒pmdlg/prre(addgene,产品号k12251)、prsv
‑
rev(addgene,产品号12253)、pmd2.g(addgene,产品号12259)分别与含目的基因的穿梭质粒如plenti
‑
e63、plenti
‑
e6con(该tcr基因序列来源于专利号us9822162b2)、plenti
‑
gfp(阴性对照)等按质量比4:2:1:1进行混匀,通过转染试剂pei
‑
max(polyscience,产品号23966
‑
1)瞬时转染处于对数生长期的293t细胞。转染48
–
50h后收集含有慢病毒的培养基上清液,经离心和0.45μm滤器去除细胞碎片后,用配有ultracel
‑
50滤膜的amicon ultra
‑
15离心过滤器(merck millipore,产品号ufc905096)将上清液进行浓缩。对浓缩后的样品进行慢病毒滴度测定,步骤参照p24 elisa(clontech,产品号632200)试剂盒说明书。
[0095]
2)tcr慢病毒转导cd8
t细胞
[0096]
从健康志愿者的pbmc中分离得到cd8
t细胞,用含有10%fbs和100iu/ml il
‑
2的rpmi 1640完全培养基接种于48孔板中,每孔1
×
106个细胞,并加入抗cd3/cd28抗体偶联磁珠用于刺激活化cd8
t细胞,置于细胞培养箱培养过夜。刺激过夜后,按moi=5的比例加入e63、e6con或gfp慢病毒,32℃,900g离心感染1h。感染完毕后去除慢病毒感染液,继续培养细胞3天,用磁铁去除抗cd3/cd28抗体偶联磁珠。此后每两天细胞计数一次,更换或加入新鲜完全培养基,将细胞密度维持在1
‑2×
106cells/ml。在细胞培养第9天,通过hla
‑
a*02:01/hpv16
‑
e6
29
‑
38
四聚体及抗cd8抗体双染色,对t细胞进行流式检测和阳性率分析。结果如图8所示,e63 tcr
‑
t细胞的阳性率为48.4%,e6con tcr
‑
t细胞阳性率为59%,gfp tcr
‑
t细胞阳性率为67.4%。
[0097]
实施例6 e63 tcr体外功能特异性分析—elispot法检测负载多肽t2细胞的inf
‑
γ释放
[0098]
本实施例通过elispot试验分析e63 tcr在负载特异性或非特异性多肽的t2细胞刺激下inf
‑
γ因子的释放情况。本实施例的效应细胞是实施例5中的经e63、e6con和gfp慢
病毒转导的cd8
t细胞。本实施例的靶细胞是负载不同浓度多肽的t2细胞,将t2细胞分别与7个梯度浓度(10
‑
11
、10
‑
10
、10
‑9、10
‑8、10
‑7、10
‑6、10
‑5m)hpv16
‑
e6
29
‑
38
多肽或10
‑6m的ny
‑
eso
‑1157
‑
165
多肽混匀,置于37℃培养箱孵育4h后,离心,1
×
pbs洗涤1次,用含10%fbs的rpmi 1640培养基重悬细胞,用于下一步铺板。根据human inf
‑
γelispot set试剂盒说明书(bd biosciences,产品号551849),执后续的实验操作。将4
×
103个双阳性效应细胞/孔和4
×
104个靶细胞/孔加入elispot孔板,每孔培养体系200μl,将孔板置于细胞培养箱孵育过夜。孵育结束后按照试剂盒说明书进行洗涤,然后加入bcip/nbt溶液显影5
–
15min后,用去离子水洗涤孔板,最终倒扣孔板,使板在室温下自然干燥。用酶联免疫斑点分析仪(6000pro
‑
fβ,bio
‑
sys)对孔板进行分析。
[0099]
结果如图9a所示,负载hpv16
‑
e6
29
‑
38
肽段的t2细胞强烈刺激e63 tcr
‑
t细胞释放inf
‑
γ且呈多肽浓度依赖性,其趋势与阳性对照e6con一致。当负载浓度大于10
‑7m,e63和e6con tcr
‑
t细胞释放inf
‑
γ均达到峰值,且两者之间无显著差异。特异性识别ny
‑
eso
‑1157
‑
165
的1g4 tcr具有显著活性,而e63和e6con tcr
‑
t细胞对负载非特异性ny
‑
eso
‑1157
‑
165
的t2细胞均无明显活性(图9b)。负载e6或ny
‑
eso
‑
1肽段的t2细胞都无法刺激gfp tcr
‑
t细胞释放inf
‑
γ因子。综上所述,e63 tcr功能与阳性对照e6con接近,且对hla
‑
a*02:01/hpv16
‑
e6
29
‑
38
的识别具有特异性。
[0100]
实施例7构建e6转基因a375单克隆细胞
[0101]
为了构建hla
‑
a*02:01
/hpv16
‑
e6
双阳靶细胞系以用于e63 tcr功能验证,将e6全肽段基因通过实施例5的方法连接到相应的穿梭质粒,并进行慢病毒包装,然后转导到a375肿瘤细胞(hla
‑
a*02:01
/hpv16
‑
e6
‑
),初步得到染色体整合有e6基因的a375多克隆细胞。为了得到能稳定表达e6基因的a375单克隆细胞,通过有限稀释法对a375 e6多克隆细胞在96孔板中进行分离培养,每孔含有0.5
–
1个候选细胞。待候选细胞扩增完毕,采用荧光定量pcr方法测定细胞内e6基因拷贝数。如表4所示,编号为1b3的a375 e6单克隆细胞e6基因拷贝数是19.23,该细胞将用于e63 tcr的体外功能验证和动物实验。
[0102]
表4.a375 e6
‑
1b3细胞e6基因拷贝数检测结果
[0103][0104][0105]
hpv16
‑
e6全长序列:
[0106]
mhqkrtamfqdpqerprklpqlctelqttihdiilecvyckqqllrrevydfafrdlcivyrdgnpyavcdkclkfyskiseyrhycyslygttleqqynkplcdllircincqkplcpeekqrhldkkqrfhnirgrwtgrcmsccrssrtrretql(seq id no:26)
[0107]
上述序列中:下划线标记序列为hpv16
‑
e6
29
‑
38
肽段。
[0108]
实施例8 e63 tcr体外功能验证—elispot法检测肿瘤细胞系的inf
‑
γ释放
[0109]
本实施例通过elispot试验分析e63 tcr在不同肿瘤细胞系刺激下inf
‑
γ因子的释放情况。本实施例的效应细胞是实施例5中的经e63、e6con和gfp慢病毒转导的cd8
t细胞。本实施例的肿瘤靶细胞分别是a375、a375 e6
‑
1b3、caski、c33a、siha和hela细胞。按实施例6所述,依次将4
×
103个阳性效应细胞/孔和4
×
104个肿瘤靶细胞/孔加入elispot孔板,
每孔培养体系200μl,将孔板置于细胞培养箱孵育过夜。孵育结束后,参照实施例6的方法对elispot孔板进行洗涤和显影,最终用酶联免疫斑点分析仪对孔板进行分析。
[0110]
结果如图10所示,e63和e6con tcr
‑
t细胞对双阳肿瘤靶细胞a375 e6
‑
1b3展现出强烈刺激活性,两者释放inf
‑
γ因子的能力相当。其它肿瘤细胞均无法刺激e63与e6con tcr
‑
t细胞释放inf
‑
γ。gfp tcr
‑
t细胞对所有肿瘤靶细胞均无明显活性。
[0111]
实施例9 e63 tcr体外功能验证—肿瘤细胞系ldh特异性杀伤
[0112]
采用定量测定靶细胞裂解后释放的ldh来评估效应细胞杀伤靶细胞的功能,具体实验方案参考文献eur.j immunol.1993,23,3217。本实施例的效应细胞是实施例5中的经e63、e6con和gfp慢病毒转导的cd8
t细胞。本实施例的肿瘤靶细胞是a375和a375 e6
‑
1b3细胞。依次将3
×
104个双阳性效应细胞/孔和1
×
104个肿瘤靶细胞/孔加入96孔圆底板,每孔培养体系200μl,细胞在铺板时均置换成仅含5%fbs的rpmi 1640培养基,将孔板置于细胞培养箱中培养24h。根据cytotoxnon
‑
radioactive cytotoxicity assay试剂盒说明书(promega,产品号g1780),在培养基空白孔与肿瘤靶细胞最大自释放孔中加入裂解液,置于细胞培养箱中孵育45min。孵育结束后,每孔取50μl上清与50μl ldh检测液混匀,室温避光孵育30min后,加入终止液,并于490nm波长下读板。按照计算公式对数据进行处理分析:细胞毒性百分比%,即ldh释放百分比%=(实验组释放量
–
肿瘤细胞自释放量
–
tcr
‑
t细胞自释放量)/(肿瘤细胞最大释放量
‑
肿瘤细胞自释放量)*100%。计算时,各组ldh释放量数值均减去培养基本底光吸收值。
[0113]
结果如图11所示,e63和e6con tcr
‑
t细胞对双阳肿瘤靶细胞a375 e6
‑
b3展现出明显的杀伤作用,两者细胞毒性百分比接近,分别是35.56
±
5.86%和34.93
±
4.72%。e63和e6con tcr
‑
t细胞对a375无明显杀伤作用,与阴性对照gfp tcr
‑
t细胞相似。
[0114]
实施例10 e63 tcr体外功能验证—采用人源、鼠源c区氨基酸序列的e63、e6con tcr
‑
t细胞阳性率检测
[0115]
e63 tcrα链和β链采用了人源c区的氨基酸序列,而阳性对照e6con tcr的α链和β链采用了鼠源c区的氨基酸序列。为了比较不同来源的c区氨基酸序列对e63 tcr在t细胞内表达的影响,将e63和e6con tcr的c区互换,最后得到4种不同的tcr组合。这四个tcr分别转导到cd8
t细胞,采用hla
‑
a*02:01/hpv16
‑
e6
29
‑
38
四聚体及抗cd8抗体双染色,对t细胞进行流式检测和阳性率分析。
[0116]
如图12所示,同是人源c区序列,e63的阳性率可达70.5%,而e6con的阳性率为27.1%,比e63低43.4%。当c区替换成鼠源氨基酸序列,e63的阳性率为85.7%,增加了15.2%;同是鼠源c区的e6con,其阳性率为78.1%,比人源c区的e6con增加了51%。由此可见,人源或鼠源c区对e63 tcr在t细胞内的表达率影响较小,说明e63tcr的α链、β链具有较好的配对效率。
[0117]
e63 tcr(鼠源c区)的α链序列:
[0118][0119]
e63 tcr(鼠源c区)的β链序列:
[0120][0121]
e6con tcr(人源c区)的α链序列:
[0122][0123]
e6con tcr(人源c区)的β链序列:
[0124][0125]
上述序列中:黑色标记序列为信号肽和vα(α链的可变区)或者vβ(β链的可变区)、灰色标记序列为cα(α链的恒定区)或者cβ(β链的恒定区)、斜体下划线标记序列为跨膜胞内区。
[0126]
实施例11 e63 tcr体外功能验证—健康人pbmc特异性inf
‑
γ释放
[0127]
为排查e63 tcr的安全性,采用实施例6的elispot法,对22例健康人的pbmc进行安全性排查。22例健康人pbmc(图13a)包含4例hla
‑
a*02:01分型,18例非hla
‑
a*02:01分型。如图13b所示,inf
‑
γ释放反应显示e63 tcr
‑
t细胞与健康人的pbmc无明显反应。
[0128]
实施例12 e63 tcr动物实验验证—e6转基因a375黑色素瘤异源移植
[0129]
本实施例的实验动物种系是b
‑
ndg小鼠(百奥赛图江苏基因生物技术有限公司,spf级),雌性,4~6周龄。试验期间,实验动物安置在spf级动物中心,所有技术指标均符合gb14925
‑
2010屏障环境技术要求。本研究中的动物将食用质量符合国家标准并在有效期内的饲料,其饮用水经动物饮用纯水系统过滤除菌处理或高温高压灭菌处理,由动物饮水瓶
自由摄取。实验动物均适应性饲养1周后再进行后续实验。
[0130]
选择适应性观察合格的b
‑
ndg小鼠皮下接种a375 e6
‑
1b3肿瘤细胞,调整细胞密度为1
‑2×
107个/ml,每只接种0.2ml。接种后每天进行观察,在平均瘤体积为100mm3左右时分为3个组,即model control组、e63 tcr
‑
t组和gfp tcr
‑
t组,每组8只动物。e63 tcr
‑
t组按照剂量4
×
108个阳性t细胞/kg给药,gfp tcr
‑
t组给予相同剂量的t细胞,模型对照组动物给予等体积的溶媒。各组小鼠给药均为尾静脉注射。各组给药后腹腔注射il
‑
2,5万iu/只,连续给药5天。分组当天测量瘤径,之后每3天用游标卡尺测量肿瘤的长径(a)及短径(b),按公式1/2
×
a
×
b2计算肿瘤体积(tumor volume),绘制肿瘤生长曲线。给药26天后安乐死动物,取瘤体进行拍照。
[0131]
各组小鼠的肿瘤生长曲线和瘤体详见图14和图15,e63 tcr
‑
t细胞能有效杀伤肿瘤细胞a375 e6
‑
1b3、抑制肿瘤的生长。gfp tcr
‑
t组的肿瘤生长曲线和model control组相比无明显变化。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。