一种评估黄鳍鲷种群遗传多样性的ssr标记引物和方法
技术领域
1.本发明属于分子生物学技术领域,具体涉及一种基于黄鳍鲷转录组测序获得的评估黄鳍鲷种群遗传多样性的ssr标记引物、方法及其在群体遗传多样性分析中的应用。
背景技术:
2.黄鳍鲷(acanthopagrus latus)属于鲈形目鲷科棘鲷属鱼类。黄鳍鲷的分布贯穿印度西太平洋,从波斯湾沿着印度海岸到菲律宾,从澳大利亚到日本,在我国主要分布广东、福建、广西等沿海城市,是一种名贵的海水鱼,也是我国咸淡水养殖的重要对象。近年来,由于人类的过度捕捞、水体污染、种质资源的保护意识不强等因素,国内的黄鳍鲷数量急剧下降。此外,关于黄鳍鲷遗传多样性的研究在近十年来尚未有新的研究进展,所以对我国黄鳍鲷的遗传多样性研究是大势所趋。我国黄鳍鲷主要产地的资源状况为其人工养殖及品种培育提供理论依据,为分子标记辅助育种打下基础。
3.简单重复序列(simple sequence repeats,ssr),即微卫星标记。微卫星标记是由核心序列和侧翼序列组成,核心序列由1
‑
6个核苷酸组成单元,单元重复次数不低于5次的简单重复序列。ssr广泛分布于真核生物基因组和转录组中,因此成为真核生物基因组水平遗传多样性评估的有效方法。ssr是共显性标记,可直接反应物种的遗传信息。由于在dna复制过程中滑链导致错配概率大,所以ssr相比于双等位基因标记snp(single nucleotide polymorphism)和aflp(amplified fragment length polymorphism)具有更高的多态性和杂合性。微卫星标记两端的侧翼序列在亲缘关系比较近的物种基因组中具有保守性,所以某一物种已经开发的微卫星标记可以应用到近缘物种的相关研究,即ssr具有通用性,这一特性大大的减少了开发微卫星标记的工作量。微卫星标记分布广泛、共显性标记、通用性等优点,所以ssr广泛应用于亲缘关系鉴定、群体遗传结构研究、遗传连锁图谱构建等方面的研究。
技术实现要素:
4.本发明的目的在于提供一种评估黄鳍鲷种群遗传多样性的ssr标记引物,该引物扩增稳定,多态性强,杂合度高。
5.本发明的目的还在于提供一种评估黄鳍鲷群体遗传多样性的方法。
6.本发明的最后一个目的在于提供上述引物或方法在黄鳍鲷种群遗传多样性分析、品种鉴定、遗传图谱构建或分子辅助育种方面的应用。
7.本发明的上述第一个目的是通过以下技术方案来实现的:一种评估黄鳍鲷种群遗传多样性的ssr标记引物,所述引物包括8对,分别为引物1、引物2、引物3、引物4、引物5、引物6、引物7和引物8,其核苷酸序列分别如seq id no:1~16所示。
8.本发明的上述第二个目的是通过以下技术方案来实现的:一种评估黄鳍鲷群体遗传多样性的方法,包括以下步骤:
9.(1)黄鳍鲷微卫星标记开发
10.取黄鳍鲷脑、肾、肝脏、性腺和肌肉的新鲜组织,提取组织rna,进行转录组测序,从下机数据中提取高质量序列,使用软件misa对转录组序列的编码区进行搜索,寻找微卫星标记;
11.(2)黄鳍鲷多态性微卫星标记筛选
12.用步骤(1)预测的微卫星标记,通过pcr扩增,电泳检测扩增产物大小,收集数据,用软件分析ssr的遗传学参数,筛选得到上述的10对特异性扩增、高多态性的ssr标记引物;
13.(3)黄鳍鲷遗传多样性分析
14.收集黄鳍鲷群体样本,提取个体dna,分别利用转录组开发的步骤(2)中的8对多态性ssr标记引物对黄鳍鲷群体进行pcr扩增,毛细管电泳进行基因分型,读取毛细管电泳数据,利用软件fstat2.9.3分析部分ssr标记分别在黄鳍鲷群体中的等位基因数(a)、期望杂合度(he)、观测杂合度(ho)、近交系数(f)和哈代温伯格平衡显著性(p),利用软件cervus计算各ssr标记在所有个体的等位基因数(na)、观测杂合度(ho)、期望杂合度(he)和多态信息含量(pic),利用mega软件的upgma算法对黄鳍鲷群体进行聚类分析。
15.在上述评估黄鳍鲷群体遗传多样性的方法中:
16.优选的,步骤(1)中采用trizol
‑
氯仿法提取组织rna。
17.优选的,步骤(1)中在转录组编码区开发微卫星标记。
18.优选的,步骤(2)中筛选8对微卫星引物的过程是:根据转录组测序预测的微卫星标记,合成引物,并分别对6~10个黄鳍鲷个体进行pcr扩增,筛选出扩增稳定、多态性强、杂合度高的8对微卫星引物。
19.更佳的,步骤(2)中筛选8对微卫星引物的过程是:根据转录组测序预测的微卫星标记,合成引物,并分别对8个黄鳍鲷个体进行pcr扩增,筛选出扩增稳定、多态性强、杂合度高的8对微卫星引物。
20.优选的,步骤(2)中pcr扩增时,反应体系为10μl:2
×
pcrmix 5μl,浓度为10μmol/l的正、反引物各0.4μl,浓度为100ng/μl的dna模板1μl,ddh2o 3.2μl。
21.优选的,步骤(2)中pcr反应程序设定为:94℃预变性5min,然后94℃30s,退火温度48
‑
60℃30s,72℃30s,共30个循环,最后72℃延伸10min。
22.优选的,步骤(3)中在8对多态性ssr标记引物的5’端标记fam荧光基团。
23.优选的,步骤(3)采用abi 3730xl进行基因型分型,利用gene mapper v4.0读取个体等位基因大小(bp),利用软件fstat2.9.3分析8对ssr标记引物分别在黄鳍鲷群体中的等位基因数(a)、期望杂合度(he)、观测杂合度(ho)、近交系数(f)和哈代温伯格平衡显著性(p)。
24.本发明的上述第三个目的是通过以下技术方案来实现的:上述的引物或上述方法在黄鳍鲷种群遗传多样性分析、品种鉴定、遗传图谱构建或分子辅助育种方面的应用。
25.与现有技术相比,本发明具有以下优点:
26.(1)本发明基于转录组数据开发的ssr标记引物,该开发的ssr标记引物具有特异性扩增、跨物种通用性、高度多态性、共显性且易于检测等优点,同时这些ssr标记均来自黄鳍鲷转录组编码区,能够直接反应基因组的遗传信息,从而更好的分析黄鳍鲷群体的遗传多样性,丰富了黄鳍鲷的ssr标记资源库;
27.(2)本发明开发的ssr分子标记可用于黄鳍鲷种质鉴定、遗传多样性分析等领域,
为今后黄鳍鲷种质资源调查、开发和保护提供理论基础,并为我国黄鳍鲷主要产地的资源状况、人工养殖及品种培育提供理论依据,为分子标记辅助育种打下基础。
附图说明
28.图1是实施例1中六个黄鳍鲷群体聚类图。
具体实施方式
29.下面结合具体实施例进一步说明本发明的应用方法。下述实施例和附图仅用于示例性说明,不能理解为对本发明的限制。除非特别说明,下述实施例中使用的试剂原料为常规市购或商业途径获得的生化试剂原料,使用的实验仪器均为实验室常规仪器,除非特别说明,下述实施例中使用的方法和设备为本领域常规使用的方法和设备。
30.实施例1
31.本实施例提供的评估黄鳍鲷种群遗传多样性的ssr标记引物及评估黄鳍鲷群体遗传多样性的方法,通过以下方法获得:
32.(1)提取黄鳍鲷的脑、性腺、肝脏、肌肉和头肾5个组织的rna,并等量混合后,反转录合成第一条cdna合成,pcr扩增合成双链cdna,构建不同大小的cdna文库,经纯化、筛选后,使用pacbio sequel平台进行测序,从下机数据中提取高质量的ccs(circular consensus sequence)序列,移除引物和barcode、poly(a)和连环结构,得到全长非嵌合序列(full
‑
length non
‑
chimeric,flnc)。对相似的flnc reads进行聚类,合并成一个完整的isoform;
33.(2)采用misa软件对转录组的所有isoform进行搜索,寻找ssr位点。设置的参数为:二碱基、三碱基、四碱基、五碱基、六碱基重复单元的重复次数分别为6、5、4、4、4次;
34.(3)采用primer3软件进行ssr引物设计,引物设计参数为引物序列长度18
‑
27bp,pcr扩增产物长度100
‑
400bp,gc含量57%
‑
62%;
35.(4)提取8个黄鳍鲷总dna,pcr扩增鉴定ssr的特异性及多态性;
36.反应体系为10μl:2
×
pcr mix 5μl,正反引物(10μmol/l)各0.4μl(每对引物单独使用),dna模板(100ng/μl)1μl,ddh2o 3.2μl。
37.pcr扩增程序为:94℃预变性5分钟,然后94℃变性30秒,58℃退火30秒,72℃延伸30秒,共35个循环,最后72℃延伸10分钟。通过1%琼脂糖凝胶电泳淘汰无条带、无主带、无单一条带的ssr。剩下的ssr用8%非变性聚丙烯酰胺凝胶电泳检测多态性,硝酸银进行染色,银染显色;
38.(5)将上述步骤初步筛选的多态性ssr标记引物的正向5’端标记fam荧光基团,合成荧光标记引物;
39.(6)用一个群体去扩增步骤(5)的ssr分子标记引物,利用软件cervus3.0计算ssr在所有个体的等位基因数(na)、观测杂合度(ho)、期望杂合度(he)、多态信息含量(pic),筛选等位基因数多、期望杂合度和多态信息含量高的ssr标记引物;
40.(7)利用步骤(6)扩增稳定、多态性好的ssr标记引物对黄鳍鲷群体进行遗传多样性分析:收集我国多个海域的6个黄鳍鲷群体,共235个个体,提取其dna为模板进行扩增,将得到的荧光pcr扩增产物进行毛细管电泳基因分型,读取毛细管电泳数据,通过ssr标记在
不同群体中出现的等位基因频率计算杂合度、遗传距离等,从而描述群体的遗传结构和确定种群的遗传变异,并对黄鳍鲷群体进行聚类。
41.其中,步骤(1)提黄鳍鲷组织rna的方法具体步骤为:
42.1)取适量组织在液氮环境中充分研磨后再转移至1.5ml离心管,加入1ml trizol,立即充分混匀;
43.2)将混匀的组织在室温放置10min,使充分裂解;
44.3)加入200μl氯仿,充分震荡混匀,4℃离心,12000g 10min;
45.4)取上层水相,加入等体积的酚:氯仿(25:24),充分混匀,4℃离心,12000g 10min;
46.5)取上层水相,加入等体积氯仿,充分混匀,4℃离心,12000g 10min;
47.6)取上层水相,加入等体积异丙醇,
‑
20℃静置1小时,4℃离心,12000g 10min;
48.7)弃上清,加入1ml 75%乙醇,洗涤沉淀,4℃离心,8000g 5min,弃上清;
49.8)重复上一步;
50.9)短暂离心,移液器吸干乙醇,真空干燥2
‑
4min;
51.10)加20
‑
50μl rnase
‑
freewater,室温溶解10min,混匀后瞬时离心;
52.11)将黄鳍鲷所有组织的rna样品混成一管,稀释后使用。
53.其中,步骤4提dna利用天根生化科技有限公司的海洋动物dna提取试剂盒,按照试剂盒说明书步骤。
54.其中,步骤(4)的非变性聚丙烯酰胺凝胶电泳具体步骤为:
55.(4.1)将方板和耳朵板对齐固定在配套的制胶架上,旋紧制胶架底座两侧的螺旋,然后用长尾夹夹紧两玻璃板左右两侧,达到密封效果。
56.(4.2)将配置好的40ml 8%聚丙烯酰胺凝胶溶液注入两玻璃板之间,待液面到达耳朵板最高处时插入梳子,在未凝固之前应随时观察是否渗漏,静置至完全凝固。
57.(4.3)胶凝固之后,将玻璃板从制胶架取下,固定到电泳槽两侧,旋紧螺旋,旋紧程度不宜太紧,太紧容易导致胶变形,也不宜太松,太松导致电泳槽上方的缓冲液漏到下方。在电泳槽上方倒入0.5
×
的tbe缓冲液,在点样孔加入2μl的pcr产物,在中间及最左边胶孔分别点50bp dna ladder和pbr322dna maker。
58.(4.4)盖上电泳槽的上盖,先用电压200v跑10min,再用电压600v跑lh20min。电泳结束后断开电源,将胶取出后至agno3染色液中染色5min,随后将凝胶放清水漂洗10s,接着将凝胶转移到显色液中显色,直至观察到清晰条带出现,拍照记录。
59.其中,步骤(7)收集的黄鳍鲷材料均取自黄鳍鲷尾鳍、背鳍或者胸鳍,活体取下后保存在95%的乙醇中。6个黄鳍鲷群体分别采自广东珠海(zh;44尾)、广东阳江(yz;41尾)、广东大亚湾(dyw;36尾),福建漳州(zz;30尾),福建厦门(xm;38尾)和广西防城港(fcg;46尾)。
60.具体结果如下:
61.(1)ssr标记的开发和筛选
62.从黄鳍鲷转录组ssr数据中挑选2碱基>10重复的微卫星标记80个;3碱基>7重复83个;4碱基>6重复10个,pcr产物大小介于80bp到500bp之间,总共获得173个微卫星标记;并对8个黄鳍鲷个体进行pcr扩增,筛选出扩增稳定、多态性强、杂合度高的8对引物。
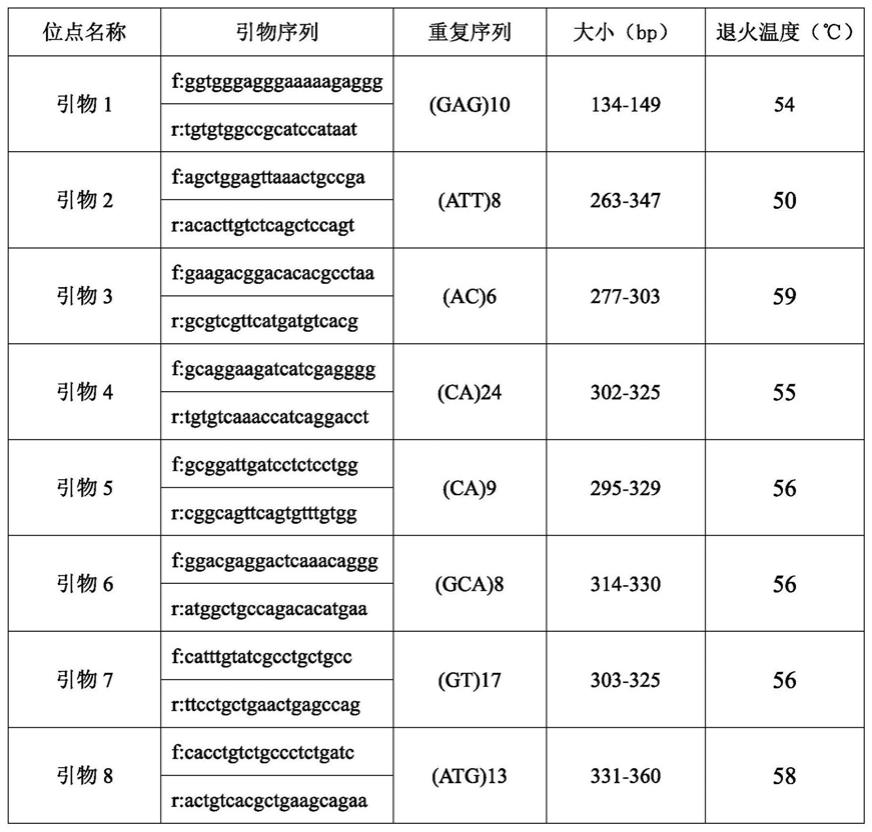
63.本发明筛选的8对微卫星标记引物为:引物1、引物2、引物3、引物4、引物5、引物6、引物7和引物8,具体如下表1所示。
64.表1 黄鳍鲷8对ssr标记引物信息
[0065][0066]
用8对引物分别对235个个体进行pcr扩增分析,通过软件进行统计分析,结果如表2所示。
[0067]
每个ssr标记的等位基因数(a)在7
‑
32个之间,平均每个ssr标记有17.1个等位基因,引物8扩增出的等位基因数最多,为32个,引物1等位基因数最少,为7个。
[0068]
观测杂合度位于0.592
‑
0.889,平均值为0.740;期望杂合度位于0.622
‑
0.932,平均值为0.823,观测杂合度明显低于期望杂合度,说明存在杂合度缺失。ssr位点的多态信息含量位于0.576
‑
0.933,平均值为0.803。
[0069]
表2 8个ssr在235尾黄鳍鲷个体中的遗传学参数
[0070]
[0071][0072]
(2)群体遗传多样性评估
[0073]
利用已获得的8个多态性高的优良ssr标记,对6个黄鳍鲷群体(广东珠海、广东阳江、广东大亚湾、福建漳州、福建厦门和广西防城港)进行遗传多样性分析。
[0074]
分析结果如表3所示,防城港群体遗传多样性要高于厦门群体,等位基因数分别为7.625和7,期望杂合度分别为0.765和0.741;阳江群体具有最高的遗传多样性,等位基因数和期望杂合度分别为8.25和0.778,大亚湾、漳州、珠海群体的等位基因数分别为7、7.125和6.5,期望杂合度分别为0.730、0.740、和0.717。
[0075]
表3 8个ssr在6个黄鳍鲷群体中的统计遗传学信息
[0076]
[0077]
[0078][0079]
a:等位基因数目;he:期望杂合度;h0:观测杂合度;f:近交系数;p:哈代温伯格平衡显著性。
[0080]
通过软件arlequin 3.11分析得到的pairwise fst值总体显示各群体之间都出现了高度的遗传分化,珠海群体和其他5个群体总体分化程度最大,平均fst达0.531,其中和阳江群体遗传分化程度最大(fst=0.630);阳江群体和漳州群体之间的遗传分化程度最小(fst=0.259),大亚湾群体和厦门群体之间的遗传分化程度较小(fst=0.367)。
[0081]
表4 六个群体基于8个微卫星标记引物的遗传分化系数fst(对角线下)及其群体遗传距离(对角线上)
[0082][0083]
mega通过upgma法聚类分析,六个黄鳍鲷群体聚类图如图1所示,图1中的结果显示6个群体分成六大簇:珠海群体、防城港群体独自成为一簇,漳州和阳江群体遗传分化程度最低聚成一个分支,大亚湾和厦门群体遗传分化程度较低聚成一个分支。
[0084]
以上结果表明,本发明中微卫星标记可以准确评估黄鳍鲷多个群体之间的遗传多样性,对研究黄鳍鲷的生物多样性和系统地理学具有重要作用。
[0085]
以上所述仅是本发明的非限定实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进,这些都视为本发明的保护范围。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。