1.本发明属于微生物与基因工程领域,具体涉及一种聚酮化合物骨架及其后修饰物的生物合成基因簇及其应用。
背景技术:
2.快速生长型非结核分枝杆菌(rgm,rapidly growing mycobacteria)是指除结核分枝杆菌复合群和麻风分枝杆菌以外的快速生长类型分支杆菌。rgm可以引起肺部病变、淋巴结炎、皮肤软组织感染和骨骼系统病变等疾病,统称为快速生长分枝杆菌病。近年来,快速生长分枝杆菌病(rgm)的患病率迅速上升,且对于目前的临床抗菌药物普遍呈耐药性。广州市胸科医院分离到的180株rgm的药敏检测发现总体耐药率为100%,除了对克拉霉素和硫酸阿米卡星耐药率低于40%以外,对其他10种一二线抗菌药物的耐药率普遍高于80%。rgm患病率迅速上升,耐药性强,治疗困难,寻找新的治疗药物已成为临床重大需求和抗菌药物研究的难点之一。
3.由于rgm对于不同结构类别和抗菌机制的药物几乎全部耐药,从现有药物靶点和抗生素结构衍生物寻找抗rgm药物的困难较大。
4.tubelactomicin是一种聚酮类天然化合物,于2000年被发现,属于16元环内酯抗生素,由土壤放线菌诺卡氏菌的发酵培养液中分离获得。tubelactomicin a对快速生长型分枝杆菌显示出强大活性。tubelactomicina对草分枝杆菌(mic为0.2μg/ml),牛分枝杆菌(mic为0.1μg/ml),耻垢分枝杆菌(mic为0.1μg/ml)和偶发分枝杆菌(mic为0.78μg/ml)等rgm表现出强大的抗菌活性。在100mg/kg静脉给药量的动物实验中,tubelactomicin a也表现出良好的体内安全性。tubelactomicin a与其它抗结核抗生素无交叉耐药性,表明其对快速生长型分枝杆菌的生长抑制作用具有新的机理。
5.诺卡氏菌产生的tubelactomicin类化合物包含tubelactomicin a、tubelactomicin b、tubelactomicin d、tubelactomicin e组分。其中tubelactomicin b、tubelactomicin d、tubelactomicin e组分为tubelactomicin a生物合成途径中的中间体。tubelactomicin d和tubelactomicin e组分也具有一定的抗rgm活性。
6.组合生物合成是近20年发展起来的一种以药物发现为中心,通过基因工程改造来挖掘和生物合成新型复杂的化合物,获取新型抗生素的新方法。对于化学手段难以合成或者合成起来代价昂贵的复杂生物大分子化合物,组合生物合成技术尤其适用于这些化合物。通过生物信息学对比分析,确定基因簇中各个模块或者结构域的功能,结合微生物遗传学手段,将这些模块或者结构域进行进行排列组合,就可能产生不同功能基团的新型人为改造的天然产物。
7.聚酮类化合物是由细菌、真菌、放线菌或植物产生,以小分子羧酸为前体,经酰基转移酶和聚酮合成酶pks催化合成大环骨架,再经由一系列后修饰操作形成的一类数目庞大、结构复杂的天然次级代谢产物。目前发现的pks根据结构和机理上的差异主要分为三
类:ⅰ型pks、ⅱ型pks、ⅲ型pks。相较于其他聚酮合成酶,ⅰ型pks是目前研究最深入,最广泛的一类聚酮合成酶,整个pks运行类似于工厂流水线车间,非重复使用的各个催化功能域依次执行识别、加载、加工处理等一系列操作,聚酮链的生物合成顺序与结构功能域一一对应。ⅰ型pks主要包括酮基合成酶(keto-synthase,ks)、酰基转移酶(acyl transferase,at)、酰基载体蛋白(acyl carrier protein,acp)、烯醇还原酶(enoyl reductase,er)、脱氢酶(dehydratase,dh)、酮基还原酶(ketoreducatse,kr)、转甲基酶(methyltransferase,mt)、硫化氢解酶(sulfhydrolase,sh)硫酯酶(thioesterase,te)等酶的功能域。聚酮碳链的构建时,需要各种催化结构域组合成不同模块参与反应,使得每个碳单位发生不同程度的还原,从而合成结构特异多样的聚酮化合物,这意味着聚酮化合物的结构具有一定的可塑性,聚酮合酶pks所具有的可塑性使人们可以通过组合生物合成手段获取获得新结构或新活性的化合物。
技术实现要素:
8.本发明首次对诺卡氏菌基因组进行了测序及分析,基于三代测序数据,对诺卡氏菌的655473条序列进行组装,得到初步组装结果;之后,将质量过滤的二代测序数据比对到组装结果上,对组装结果进一步校正,经过拼接组装及校正,得到了诺卡氏菌的基因组信息,一共含有10224019个碱基,基因组中总gc碱基百分比含量为65.47%。
9.完成组装后,对编码基因进行了预测,预测的编码基因数目为9106个,预测的编码基因总长度为9074577bp,最短基因为90bp,最长基因为71547bp,所有预测基因的总gc碱基百分比含量为65.90%。其中,基因组中含有133个碳水化合物结合模块基因(carbohydrate-binding modules,cbm)、349个编码糖基转移酶(glycosyl transferases,gt)的基因、173个编码糖苷水解酶(glycoside hydrolases,gh)的基因、88个编码碳水化合物酯酶(carbohydrate esterases,ce)的基因、2个编码多糖裂解酶(polysaccharide lyases,pl)的基因及36个辅助活动基因(auxiliary activities,aa)。进一步的,预测了诺卡氏菌种20个pks基因,从众多候选pks基因种最终确证了与tubelactomicin聚酮链的合成有关的聚酮合成酶基因簇。
10.本发明具体技术方案如下:
11.一种聚酮化合物骨架的生物合成基因簇,包括:
12.编码酰基转移酶(tub a)的基因,所述酰基转移酶(tub a)的氨基酸序列如seq id no:2所示;
13.编码聚酮合成酶1(tub b)的基因,所述聚酮合成酶1(tub b)的氨基酸序列如seq id no:3所示;
14.编码聚酮合成酶2(tub c)的基因,所述聚酮合成酶2(tub c)的氨基酸序列如seq id no:4所示;
15.编码聚酮合成酶3(tub d)的基因,所述聚酮合成酶3(tub d)的氨基酸序列如seq id no:5所示;
16.编码聚酮合成酶4(tub e)的基因,所述聚酮合成酶4(tub e)的氨基酸序列如seq id no:6所示;
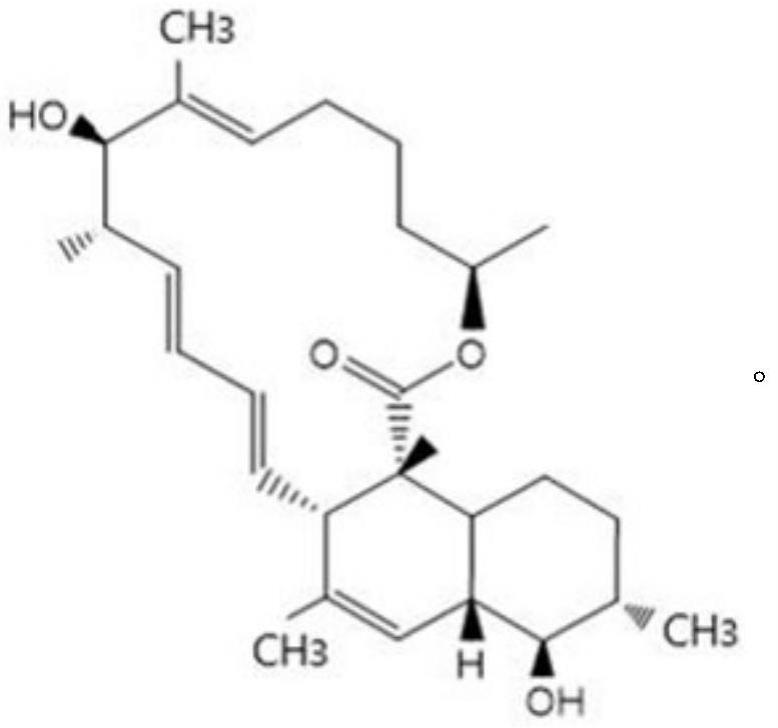
17.所述聚酮化合物骨架具有如下结构:
[0018][0019]
本发明所述生物合成基因簇中的各基因对应于诺卡氏菌的基因的第8079-8083位基因,如表1所示。
[0020]
表1聚酮化合物骨架的生物合成基因簇信息
[0021][0022]
上述各酶包含的功能域如图1所示。
[0023]
上述聚酮化合物骨架的生物合成基因簇中,编码酰基转移酶的基因序列为seq id no:1的序列第501~17702位所示的序列;编码聚酮合成酶1的基因序列为seq id no:1的序列第17719~22716位所示的序列;编码聚酮合成酶2的基因序列为seq id no:1的序列第22724~34054位所示的序列;编码聚酮合成酶3的基因序列为seq id no:1的序列第34178~49867位所示的序列;编码聚酮合成酶4酶的基因序列为seq id no:1的序列第49894~64248位所示的序列。
[0024]
一个具体的方案,上述聚酮化合物骨架的生物合成基因簇序列为seq id no:1的序列第501~64248位所示的序列。
[0025]
本发明另一目的在于提供一种聚酮化合物生物合成基因簇,包括本发明所述的聚酮化合物骨架的生物合成基因簇以及后修饰基因中的一种或几种,所述后修饰基因包括硫酯酶、细胞色素p450酶、铁氧化还原蛋白。
[0026]
一个具体的方案,所述聚酮化合物为tubelactomicin a、tubelactomicin d或tubelactomicin e,所述后修饰基因包括编码硫酯酶(tub f)、一个或多个细胞色素p450酶(tub g)和铁氧化还原蛋白(tub h)的基因,硫酯酶氨基酸序列如seq id no:7所示,细胞色素p450酶氨基酸序列如seq id no:8所示铁氧化还原蛋白氨基酸序列如seq id no:9所示。
[0027]
聚酮化合物骨架(tubelactomicin b)以及tubelactomicin a、tubelactomicin d或tubelactomicin e的生物合成途径如图2所示。
[0028]
上述后修饰基因对应于诺卡氏菌的基因的第8084-8086位基因,如表2所示。
[0029]
表2聚酮化合物骨架的后修饰基因信息
[0030][0031]
本发明在对上述生物合成基因簇的功能研究中发现,细胞色素p450酶基因能够促进聚酮化合物骨架环化的形成,进而提高tubelactomicin a、tubelactomicin d或tubelactomicin e的产率。
[0032]
上述聚酮化合物的生物合成基因簇中,编码硫酯酶的基因序列为seq id no:1的序列第66224~66985位所示的序列;编码细胞色素p450酶的基因序列为seq id no:1的序列第67135~68322位所示的序列;编码铁氧化还原蛋白的基因序列为seq id no:1的序列第68335~68529位所示的序列。
[0033]
一个具体的方案,tubelactomicin a的生物合成基因簇序列如seq id no:1所示。
[0034]
本发明另一目的在于提供本发明所述的聚酮化合物骨架和/或聚酮化合物的生物合成基因簇在构建聚酮化合物重组表达系统中的应用。可通过对聚酮化合物骨架或聚酮化合物生物合成基因簇中的功能基因进行基因重组和/或基因敲除和/或基因置换,或者将其与一种或几种其它生物合成酶基因和/或生物调节基因联合构建聚酮化合物重组表达系统。
[0035]
本发明另一目的在于提供一种tubelactomicin a重组表达系统,包含本发明所述的聚酮化合物生物合成基因簇和一个或多个细胞色素p450酶基因的共表达载体,或者包含表达本发明所述的的聚酮化合物生物合成基因簇的载体和表达细胞色素p450酶基因的载体。
[0036]
本发明另一目的在于提供一种tubelactomicin a的生物合成方法,包括如下步骤:
[0037]
(1)构建包含seq id no:1所示的聚酮化合物生物合成基因簇和一个或多个p450酶基因的重组表达工程菌;
[0038]
(2)种子培养:
[0039]
种子培养基:2%半乳糖,2%糊精,1%甘油,1%大豆蛋白胨,0.5%玉米浆,0.2%硫酸铵,0.2%碳酸钙;
[0040]
将步骤(1)构建的重组表达工程菌接种种子培养基,初始ph值7.4,180rpm转速,30℃培养3天;
[0041]
(3)发酵培养:
[0042]
发酵培养基:4%可溶性淀粉,1%黄豆饼粉,0.5%yeast extract,0.4%碳酸钙;
[0043]
将步骤(2)种子菌液接种发酵培养基,初始ph值7.4,7%接种量,180rpm转速,30℃培养6天。
[0044]
(4)分离纯化发酵液,得到tubelactomicin a。
[0045]
优选的,纯化步骤包括:
[0046]
(1)调节发酵液ph值1.0-3.0,优选2.0,加入hp20树脂(树脂事先使用甲醇溶胀过
夜),吸附1-5h,优选2.5h,使用甲醇浸泡hp20,将浸出液浓缩,用二氯甲烷萃取,收集二氯甲烷相。
[0047]
(2)硅胶柱层析,洗脱条件:依次使用二氯甲烷洗脱2个柱体积,使用体积比20:1的二氯甲烷:甲醇混合液洗脱5个柱体积。收集20:1的二氯甲烷:甲醇混合液的洗脱组分,浓缩得到tubelactomicin a。
[0048]
(3)可进一步使用hplc进行检测洗脱液纯度。
[0049]
hplc分析色谱柱为amchemteq aci-c18,4.6
×
250mm,柱温为30℃,流速为1ml/min,检测波长为238nm,流动相为a相:1
‰
甲酸水溶液,b相乙腈,洗脱梯度如表3所示。
[0050]
表3洗脱梯度
[0051][0052]
tubelactomicin a的保留时间为24.9min。
[0053]
本发明优点:
[0054]
本发明首次解析并确证了tubelactomicin生物合成有关的基因簇,所述聚酮合成酶基因簇通过基因重组/基因敲除/基因置换,得到tubelactomicin及其衍生物,并可以进一步与其它生物合成酶基因和生物调节基因共同构建tubelactomicin或其衍生物重组表达系统,用于提高tubelactomicin的产量或者用于获得新的tubelactomicin衍生物。
附图说明
[0055]
图1为聚酮化合物骨架的生物合成基因簇各酶包含的功能域示意图。
[0056]
图2为聚酮化合物骨架(tubelactomicin b)以及tubelactomicin a、d或e的生物合成途径。
[0057]
图3为tubelactomicin a hplc色谱图。
[0058]
图4为tub d单交换敲除基因片段电泳图(m:marker;1:tub d单交换敲除基因片段)。
[0059]
图5为poj260-tub d单交换重组质粒及电泳图(图5a:poj260-tub d单交换重组质粒图;
[0060]
图5(b):poj260-tub d单交换重组质粒电泳图)。
[0061]
图6为poj260-tub d单交换重组质粒酶切电泳图。
[0062]
图7为poj260-tub d单交换突变株电泳图。
[0063]
图8为野生型dsm 44638以及poj260-tub d单交换突变株表达tubelactomicin a。
[0064]
图9为tub g基因片段pcr验证电泳结果(1:tub基因片段;2:marker)。
[0065]
图10为pset152-tub g回补质粒及电泳图(图10a为pset152-tub g回补质粒示意图;图10b为pset152-tub g回补质粒pcr电泳图。1~3:pset152-tub g回补质粒;m:marker)。
[0066]
图11为pset152-tub g回补质粒酶切电泳图(1:pset152-tub g回补质粒酶切产物;m:marker)。
[0067]
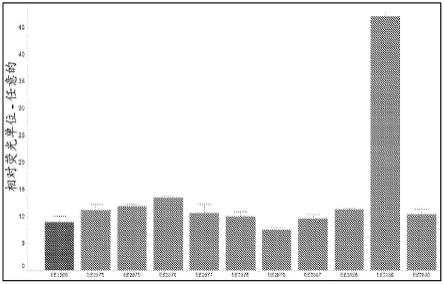
图12为野生型dsm 44638以及dsm 44638-tub g倍增突变株表达tubelactomicin a。
具体实施方式
[0068]
以下通过实施例说明本发明的具体步骤,但不受实施例限制。
[0069]
在本发明中所使用的术语,除非另有说明,一般具有本领域普通技术人员通常理解的含义。
[0070]
下面结合具体实例并参照数据进一步详细描述本发明。应理解,这些实施例只是为了举例说明本发明,而非以任何方式限制本发明的范围。
[0071]
在以下实施例中,未详细描述的各种过程和方法是本领域中公知的常规方法。
[0072]
实施例1使用诺卡氏菌发酵制备tubelactomicin a
[0073]
(1)菌种发酵
[0074]
按照2%半乳糖,2%糊精,1%甘油,1%大豆蛋白胨,0.5%玉米浆,0.2%硫酸铵,0.2%碳酸钙与去离子水配制种子培养基,调ph值至7.4,于121℃,高压蒸汽灭菌20min。接入诺卡氏菌nocαrdia vinacea dsm44638(购于德国微生物菌种保藏中心dsmz),180rpm转速,30℃培养3天;
[0075]
按照4%可溶性淀粉,1%黄豆饼粉,0.5%yeast extract,0.4%碳酸钙去离子水配制发酵培养基,将种子菌液接种发酵培养基,初始ph值7.4,7%接种量,180rpm转速,30℃培养6天。
[0076]
(2)发酵液分离纯化
[0077]
将培养的发酵液离心,取上清液调ph值到2左右,加入hp20树脂(树脂事先使用甲醇溶胀过夜),于摇床震荡吸附2.5h左右后,取出,静置沉淀树脂,将沉淀的树脂使用去离子水清洗,保留树脂。加入甲醇。将甲醇浸出液浓缩,用二氯甲烷萃取,收集二氯甲烷相。使用硅胶柱层析,依次使用二氯甲烷洗脱2个柱体积,使用体积比20:1的二氯甲烷:甲醇混合液洗脱5个柱体积。收集20:1的二氯甲烷:甲醇混合液的洗脱组分,浓缩得到tubelactomicin a。
[0078]
(3)使用hplc进行检测洗脱液,纯度。
[0079]
hplc分析色谱柱为amchemteq aci-c18,4.6
×
250mm,柱温为30℃,流速为1ml/min,检测波长为238nm,进样量根据处理方法和可能的含量选择10~25μl,流动相为1
‰
甲酸水和乙腈洗脱梯度如表4所示。
[0080]
表4洗脱梯度
[0081]
[0082]
将纯化的样品送样进行lc-ms检测,样品质谱结果显示为一个分子质量为487.3085的加氢峰,其分子式为c29h42o6,这与tubelactomicin a分子式相符合。
[0083]
进一步检测tubelactomicin a对耻垢分枝杆菌atcc607的最小抑菌浓度(mic)。结果显示,tubelactomicin a对atcc607有良好的抑菌活性,其mic值测量值为0.125μg/ml。将tubelactomicin a纯品使用氘代丙酮溶解后,进行400兆nmr测定,结果如下表5所示。
[0084]
表5.tubelactomicin a的1h and 13c nmr数据
[0085]
[0086][0087]
实施例2聚酮化合物的生物合成基因簇与tubelactomicin a的相关性验证
[0088]
验证策略:采用单交换同源重组法,敲降诺卡氏菌dsm44638菌株聚酮化合物的生物合成基因簇中编码聚酮合成酶3(tub d)的基因(诺卡氏菌dsm44638菌株全基因组第8082位基因),考察对tubelactomicin a生物合成的影响。
[0089]
(1)设计pcr扩增用引物序列如下:
[0090]
poj260-tub d_dan_f:5
’‑
cgacggccagtgccaagcttgaaccggttgtggtggtgg-3’(seq id no:10);
[0091]
poj260-tub d_dan_r:5
’‑
tatgacatgattacgaattcttcggcagtgtctcgtggc-3’(seq id no:11)。
[0092]
以诺卡氏菌dsm44638基因组为模板,使用prime star dna聚合酶进行pcr扩增,pcr反应程序如下:step 1:98℃,10s,step 2:62℃,5s,step 3:72℃,70s,以上三步重复30次,step 4:72℃,5min。
[0093]
对pcr扩增产物进行1%琼脂糖凝胶电泳分析,并回收扩增产物。pcr产物凝胶电泳图如图4所示(m:marker;1:tub d单交换敲除基因片段)。从图4中可以看出,扩增得到1100bp的tub d基因片段,结果符合预期。
[0094]
(2)酶切和连接
[0095]
将步骤(1)获得tub d基因条带进行琼脂糖凝胶电泳分离,切胶回收,测量胶回收浓度。将质粒poj260(bierman m,logan r,o
′
brien k,seno et,rao rn,schoner be(1992)plasmid cloning vectors for the conjugal transfer of dna from escherichia coli to streptomyces spp.gene 116:43
–
49)使用handⅲ酶和ecorⅰ酶进行酶切。将酶切体系放置于37℃水浴锅,反应30min,将反应后的体系进行琼脂糖电泳,在凝胶成像仪的紫外照射下进行切胶,回收酶切产物。
[0096]
将tub d扩增产物和质粒poj260的酶切产物采用infusion重组连接酶进行连接,获得poj260-tub d单交换重组质粒。质粒示意图如图5a所示,电泳结果如图5b所示(1~3为:poj260-tub d单交换重组质粒,m:marker)。
[0097]
将poj260-tub d单交换重组质粒使用xhoⅰ/hindⅲ进行酶切,酶切得到2890bp和1633bp的两条条带(酶切电泳图如图6所示),结果符合预期。将酶切进行测序,测序结果正
确。
[0098]
(3)转化、筛选并鉴定
[0099]
将步骤(2)获得的poj260-tub d单交换重组质粒与诺卡氏菌dsm44638菌体混合,将混合物通过电击转化后,涂在含有安普霉素(100μg/ml)的ms琼脂培养基上,10天后,长出的菌落阳性即为转化子。
[0100]
挑取阳性菌体作为模板,使用引物:pks-p3:5
’‑
tcggattcgactccctcacc-3’(seq id no:12)/rv-m:5
’‑
gagcggataacaatttcacacagg-3’(seq id no:13)进行菌落pcr,pcr程序为:step 1:98℃,10s,step 2:62℃,5s,step 3:72℃,90s,以上三步重复30次,step 4:72℃,5min。
[0101]
将扩增产物进行电泳,扩增出1274bp的基因片段,符合预期大小(如图7所示,泳道1:dsm 44638野生株;2:poj260-tub d单交换质粒;3:dsm 44638-tub d突变株;m:marker)。
[0102]
经pcr验证正确的突变株命名为dsm 44638-tub d,将dsm 44638-tub d突变菌株在不含抗生素的种子培养基中培养,并转接到发酵培养基进行发酵。发酵液经过1:1甲醇处理和浓缩处理后,进行hplc检测发酵液中的tubelactomicin a的发酵单位(发酵及分离纯化方法参照实施例1),结果如图8所示。结果显示突变株dsm 44638-tub d失去产tubelactomicin a的能力。结果表明,本发明所述的聚酮化合物骨架的生物合成基因簇是tubelactomicin a生物合成所必需的。
[0103]
实施例3高产tubelactomicin重组表达菌株的构建
[0104]
(1)设计pcr扩增用引物序列如下:
[0105]
pset_tub g_f:5
’‑
cgataagcttggatcattttgtccccaccgatagatagtc-3’(seq id no:14);
[0106]
pset_tub g_r:5
’‑
ggctgcaggtcgactcgagagaaaacagttgtcctgaataag-3’(seq id no:15)。
[0107]
以诺卡氏菌dsm44638基因组为模板,使用prime star dna聚合酶进行pcr扩增,pcr反应条件为step 1:98℃,10s,step 2:62℃,5s,step 3:72℃,70s,以上三步重复30次。扩增得到大小为1409bp;包含tub g完整基因的dna片段(如图9所示),结果符合预期。
[0108]
(2)酶切和连接
[0109]
将步骤(1)tub g扩增的目的条带进行琼脂糖凝胶电泳分离,切胶回收,测量胶回收浓度。将模板质粒pset152使用xbalⅰ内切酶和bamhⅰ内切酶进行酶切。将20μl的酶切体系放置于37℃水浴锅,反应30min,将反应后的体系进行琼脂糖电泳,在凝胶成像仪的紫外照射下进行切胶,回收酶切产物,线性模板条带大小为6195bp,结果符合预期。
[0110]
将tub g扩增产物和模板质粒pset152的酶切产物采用infusion重组连接酶进行连接,获得pset152-tub g回补质粒。质粒示意图如图10a所示,电泳结果如图10b所示(1~3为:pset152-tub g回补质粒,m:marker)。
[0111]
使用xhoⅰ/ncoⅰ对成功整合的pset152-tub g回补质粒进行酶切,酶切得到3927bp、2678bp和1222bp的三条条带(酶切电泳图如图11所示),结果符合预期。将酶切验证正确的送样测序,测序结果正确。
[0112]
(3)转化、筛选并鉴定
[0113]
将步骤(2)获得的pset152-tub g回补质粒与诺卡氏菌dsm 44638菌体混合,将混
合物通过电击转化后,涂在含有安普霉素(100μg/ml)的ms琼脂培养基上,10天后,长出的菌落阳性即为转化子。
[0114]
挑取具有安普霉素抗性的阳性转化子作为模板,使用pset152质粒上的通用引物序列rv-m:5
’‑
gagcggataacaatttcacacagg-3’(seq id no:16)以及tub g基因末端序列设计引物pset_tub g_jd_f1(5
’‑
attgcattcgggtcagggga-3’(seq id no:17),进行菌落pcr,pcr程序同实施例2。将扩增产物进行电泳,扩增得到797bp的基因片段,符合预期结果。将此条带测序后,结果与预期一致。将该菌株命名为dsm 44638-tub g。
[0115]
dsm 44638-tub g表达株采用实施例1的方法发酵、分离、纯化,并计算tubelactomicin a的产量,结果如图12所示。结果显示,dsm 44638-tub g相对于野生型提高了约30%。结果表明,tub g基因(p450)能够提高野生型菌dsm 44638 tubelactomicin a的产量。
再多了解一些
本文用于创业者技术爱好者查询,仅供学习研究,如用于商业用途,请联系技术所有人。