本发明涉及一种重组单纯疱疹病毒,它具有能够表达癌细胞靶向结构域与HVEM胞外结构域的融合蛋白的表达盒;以及其用途。
背景技术
迄今为止,在癌症的治疗中,已经广泛地使用手术疗法、抗癌化学疗法、放射疗法等,但这些疗法中的大部分疗法都有副作用,治疗效果不全,还有如癌症复发和转移的问题。因此,不断地需要开发新的有效的癌症疗法,近年来,抗癌免疫疗法已经得到快速的发展,例如溶瘤病毒、嵌合抗原受体T(CAR-T)细胞疗法等。
在抗癌免疫疗法中,溶瘤病毒是一种这样的病毒,它的特征是通过操纵活病毒的基因并在癌细胞中选择性地进行增殖来溶解癌细胞,它在正常的细胞中的增殖会受到限制。通过癌细胞的溶解而释放的病毒能够不断地感染周围的癌细胞,从而提供了连续而协同的治疗作用。此外,溶瘤病毒还能够通过在溶解癌细胞的过程中释放免疫原性肿瘤抗原来刺激人体的免疫应答,从而增加抗癌作用。而且,还能够通过人工操纵,使细胞因子、趋化因子等得到表达来增强这样的抗癌作用。
目前开发的溶瘤病毒可以分成10个或更多个类型,包括腺病毒、单纯疱疹病毒(HSV)、痘苗病毒等。其中,HSV是一种包膜二十面体病毒粒子,它含有152kb大小的线性双链DNA,分成HSV-1型和HSV-2型。HSV有许多非必要的基因,而且它的基因组规模庞大,这便于操纵或转运外部的基因,并且它的复制周期短,此外,HSV还具有高感染效率,理想地,能够通过对与细胞附着和感染有关的糖蛋白的简单操纵,表现出提高的癌细胞靶向效率。
T-VEC(拉他莫基(talimogene laherparepvec),产品名:Imlygic)在2015年10月被US FDA批准,它是一种使用HSV-1的针对恶性黑色素瘤的抗癌药物(溶瘤病毒治疗剂)。T-VEC是一种减毒HSV-1病毒,其中ICP34.5和ICP47基因缺失,从而减弱了病原性,并且还表达GM-CSF(粒细胞巨噬细胞集落刺激因子),从而促进人免疫应答。然而,T-VEC有着局限性,因为病毒的增殖因一些基因的损失而受到限制,使得它的治疗功效低。
HSV是一种具有包膜的病毒,通过包膜中存在的gD、gB、gH/gL和gC糖蛋白复杂的相互作用,HSV进入了细胞。首先,当gB和gC连接于细胞表面上的3-O-S HS(3-O-硫酸化的硫酸乙酰肝素)时,gD结合于如下细胞受体中的至少一个:HVEM(疱疹病毒进入中介体,HveA)、粘连蛋白-1(HveC)和粘连蛋白-2(HveB),因此诱导病毒与细胞膜之间的融合,借此HSV进入细胞(Hiroaki Uchida等人,《受疱疹病毒进入中介体(HVEM)限制的单纯疱疹病毒1型突变病毒的产生:表达HVEM的细胞的抗性和拯救粘连蛋白-1识别的突变的鉴定(Generation of Herpesvirus Entry Mediator(HVEM)-restricted Herpes Simplex Virus Type 1Mutant Viruses:Resistance of HVEM-expressing Cells and Identification of Mutations That Rescue nectin-1Recognition)》.J.Virol.2009年4月;83(7):2951-61)。
在HSV的细胞受体当中,HVEM属于肿瘤坏死因子受体蛋白家族(TNFR家族),主要在T/B淋巴细胞、巨噬细胞和DC细胞、感觉神经元和粘膜上皮细胞中表达(Shui J.W.,Kronenberg M.2013.Gut Microbes 4(2):146-151),但已知它在多种肿瘤组织如B/T淋巴瘤、黑色素瘤、结肠直肠癌、肝细胞癌、乳腺癌、卵巢浆液性腺癌、肾透明细胞癌和胶质母细胞瘤中大量表达(Pasero C.等人,Curr.Opin.Pharmacol.2012.2(4):478-85;Malissen N.等人,ONCOIMMUNOLOGY 2019,VOL.8(12):e1665976)。HVEM具有四个CRD(半胱氨酸富集结构域),这些CRD是TNFR家族的特征,四个CRD中的两个与HSV的gD连接,因此诱导HSV-1和HSV-2进入细胞(Sarah A Connolly等人,《对疱疹病毒进入中介体HveA(HVEM)上存在的单纯疱疹病毒糖蛋白D结合位点的基于结构的分析(Structure-based Analysis of the Herpes Simplex Virus Glycoprotein D Binding Site Present on Herpesvirus Entry Mediator HveA(HVEM))》.J.Virol.2002年11月;76(21):10894-904)。
如本发明的发明人所报告的,制造出CEA(癌胚抗原)的scFv(单链可变片段)与HVEM(一种HSV细胞表面受体)胞外结构域的双特异性融合蛋白(CEAscFv-HveA),并在用所述双特异性融合蛋白和HSV治疗表达CEA的细胞系时,融合蛋白充当衔接蛋白,以诱导HSV靶向对应的细胞系并感染(第10-0937774号韩国专利和第8318662号美国专利)。
当编码用作衔接蛋白的融合蛋白(CEAscFv-HveA)的基因可表达地插入到HSV基因组中以便在感染上HSV的细胞中表达融合蛋白时,确定所述融合蛋白用以诱导HSV由此靶向表达CEA的细胞系并感染,而且,不是CEA的scFv,而是HER2的scFv(人表皮生长因子受体2)与HVEM胞外结构域的融合蛋白(HER2scFv-HveA)同等地诱导表达HER2的细胞系的靶向和感染,因此产生了本发明。
技术实现要素:
本文公开了一种重组HSV,其含有能够表达衔接蛋白的表达盒,所述衔接蛋白是癌细胞靶向结构域与HVEM胞外结构域的融合蛋白。
此外,本文还公开了一种用于抗癌治疗的药物组合物,其包含以上提及的重组HSV作为活性成分。
此外,本文还公开了一种治疗或预防癌症(肿瘤)的方法,其包括向受试者如患者施用有效量的含有以上提及的重组HSV的药物组合物。
下文将提供本发明的其它或具体方面。
具体实施方式
本发明涉及一种重组HSV(单纯疱疹病毒),它能够表达衔接蛋白,所述衔接蛋白是癌细胞靶向结构域(其可以与本文中的癌症靶向结构域互换使用,或者可以表示成配体,取决于本说明书的上下文)与HVEM胞外结构域的融合蛋白。
当本发明的重组HSV感染靶细胞,即癌细胞,并进入靶细胞时,HSV会增殖,并且作为融合蛋白的衔接蛋白在细胞中表达,在细胞溶解后与增殖的HSV病毒粒子一起被释放到细胞外,或者当衔接蛋白含有前导序列时,甚至在病毒粒子因细胞溶解而被释放前不断地释放。被释放到细胞外的衔接蛋白用于诱导HSV病毒粒子由此感染周围的表达被衔接蛋白的癌细胞靶向结构域识别的靶分子的癌细胞,或者提高感染效率。
一般说来,重组HSV是与野生型HSV相比,通过引入人工突变(通过一些核酸序列的缺失、取代或插入)进行基因操纵,以便能够失去或改变某些功能或者表达感兴趣的靶蛋白的HSV。在本发明中,重组HSV是通过在不抑制HSV的增殖下将表达衔接蛋白的盒(即,将衔接蛋白基因与使它能表达的启动子序列和多聚腺苷酸化信号序列可操作地连接的构建体)引入(即插入)到HSV基因组中而能够在感染它的癌细胞中表达衔接蛋白的HSV。重组病毒生产技术,例如病毒的基因操纵和病毒粒子的产生,是本领域中众所周知的,可以参考论文[Sandri-Goldin RM等人,《α疱疹病毒:分子和细胞生物学(Alpha Herpesviruses:Molecular and Cellular Biology)》,Caister Academic Press,2006]、论文[Robin H Lachmann,《基于单纯疱疹病毒的载体(Herpes simplex virus-based vectors)》,Int J Exp Pathol.2004年8月;85(4):177-190]等。本说明书中引用的所有文献,包括以上的文献,都被认为是本说明书的一部分。
具体地说,除了被操纵用于表达衔接蛋白之外,本发明的重组HSV还可以被操纵成只通过HVEM受体作为进入受体,而不通过粘连蛋白-1,才能进入细胞中。在本发明的以下实例中,HSV包膜糖蛋白gD的序列被操纵成允许HSV只通过HVEM受体才能进入细胞中。明确地说,gD位置222处的精氨酸(R)和gD位置223处的苯丙氨酸(F)分别经天冬酰胺(N)和异亮氨酸(I)取代,因此改变了gD的功能。由此改变了gD功能的重组HSV只通过HVEM(HveA)受体才能进入宿主细胞中(Hiroaki Uchida等人《受疱疹病毒进入中介体(HVEM)限制的单纯疱疹病毒1型突变病毒的产生:表达HVEM的细胞的抗性和拯救粘连蛋白-1识别的突变的鉴定(Generation of Herpesvirus Entry Mediator(HVEM)-restricted Herpes Simplex Virus Type 1Mutant Viruses:Resistance of HVEM-expressing Cells and Identification of Mutations That Rescue nectin-1Recognition)》.J.Virol.2009年4月;83(7):2951-61)。已知HVEM(HveA)受体在多种肿瘤组织如B/T淋巴瘤和黑色素瘤中大量表达(Pasero C.等人,Curr.Opin.Pharmacol.2012.2(4):478-85;Malissen N.等人,ONCOIMMUNOLOGY 2019,VOL.8(12):e1665976)。
此外,还可以使本发明的重组HSV突变,以使得对于HSV增殖(即,生存和复制)来说不需要的非必需基因缺失或不表现出这些基因的功能(即,转录或翻译中断)。非必需基因的具体实例可包括UL3基因(例如GenBank登录号AFE62830.1)、UL4基因(例如GenBank登录号AFE62831.1)、UL14基因(例如GenBank登录号AFE62841.1)、UL16基因(例如GenBank登录号AFE62843.1)、UL21基因(例如GenBank登录号AFE62848.1)、UL24基因(例如GenBank登录号AFE62851.1)、UL31基因(例如GenBank登录号AFE62859.1)、UL32基因(例如GenBank登录号AFE62860.1)、US3基因(例如GenBank登录号AFE62891.1)、UL51基因(例如GenBank登录号AFE62880.1)、UL55基因(例如GenBank登录号AFE62884.1)、UL56基因(例如GenBank登录号AFE62885.1)、US2基因(例如GenBank登录号AFE62890.1)、US12基因(例如GenBank登录号AFE62901.1;ICP47基因)、LAT基因(例如GenBank登录号JQ673480.1)、gB基因(例如介于GenBank登录号GU734771.1的52996与55710之间的序列)、gL基因(例如GenBank登录号AFE62828.1)、gH基因(例如GenBank登录号AFE62849.1)、gD基因(例如GenBank登录号AFE62894.1)等等。
有关HSV的非必需基因的具体信息,可以参考论文[DM Knipe和PM Howley(编辑)Fields virology(第2卷)Lippincott Williams&Wilkins,Philadelphia,Pa.2001.第2399-2460页)]、论文[Subak-Sharpe J.H.,Dargan D.J.《HSV分子生物学:单纯疱疹病毒分子生物学的综合方面(HSV molecular biology:general aspects of herpes simplex virus molecular biology)》Virus Genes,1998,16(3):239-251]、论文[Travis J.Taylor和David M.Knipe,《单纯疱疹病毒复制室的蛋白质组学:细胞DNA复制、修复、重组和染色质重塑蛋白与ICP8的关联(Proteomics of Herpes Simplex Virus Replication Compartments:Association of Cellular DNA Replication,Repair,Recombination,and Chromatin-Remodeling Proteins with ICP8)》,J.Virol.2004年6月;78(11):5856-5866]等等。
本发明的重组HSV可以是重组HSV-1病毒、重组HSV-2病毒或HSV-1/HSV-2嵌合病毒(即,基因组含有源自于HSV-1的DNA和源自于HSV-2的DNA两者的重组HSV),优选地是重组HSV-1病毒,更优选地是源自于HSV-1KOS病毒株的重组HSV-1。HSV-1KOS病毒株可从ATCC(目录号VR-1493TM)获得,并且在GenBank登录号JQ673480.1中完整分析和表示病毒株的整个基因组序列(Stuart J.Macdonald等人《单纯疱疹病毒1病毒株KOS的基因组序列(Genome Sequence of Herpes Simplex Virus 1Strain KOS.)》J.Virol.2012年6月;86(11):6371-2)。
HSV-1病毒基因组由152kb双链线性DNA构成,编码总共84个基因,包含两个彼此连接的片段,即长片段(L区)和短片段(S区)。长片段(L区)占基因组约82%,短片段(S区)占基因组约18%,长片段和短片段通过两个作为交界区的IRL(中间反向重复序列)相连,并且在每个片段的末端处存在TRL(末端反向重复区段)。L区(UL)包含56个UL1-UL56基因和10个基因(UL8.5、9.5、10.5、12.5、15.5、20.5、26.5、27.5、43.5、49.5),S区(US)包含12个US1-US12和2个基因(US1.5、8.5),作为交界区的两个IRL包含4个基因(ICP4、ICP34.5、ICP0和LAT)。
在本发明中,表达衔接蛋白的盒被配置成衔接蛋白基因与使衔接蛋白基因能表达的启动子序列和作为转录终止信号序列的多聚腺苷酸化信号序列可操作地连接。这里,“可操作地连接”意指使表达的衔接蛋白基因能转录及/或翻译的连接。举例来说,当任何启动子影响与之连接的衔接蛋白基因的转录时,该启动子可操作地连接于衔接蛋白基因。
通常,启动子是具有控制一种或多种基因转录的功能的核酸序列,它位于基因转录起始位点的上游(5’侧)并包括依赖DNA的RNA聚合酶的结合位点、转录起始位点、转录因子结合位点等。在真核来源的情况下,启动子包括TATA盒,在转录起始位点上游(相对于转录起始位点( 1),通常位于位置-20至-30);CAAT盒(相对于转录起始位点,通常位于位置-75);增强子;转录因子结合位点等。
只要启动子能够表达与之连接的靶基因,所有的组成型启动子(一直诱导基因表达)、诱导型启动子(响应于特定的外界刺激,诱导靶基因的表达)、组织特异性启动子(诱导特定的组织或细胞中的基因表达)、非组织特异性启动子(诱导所有的组织或细胞中的基因表达)、内源性启动子(源自于病毒感染细胞)和外源性启动子(源自于除病毒感染细胞以外的细胞)都可以使用。本领域中已知许多启动子,可以在其中选择性地使用适当的启动子。举例来说,有用的启动子是CMV(巨细胞病毒)启动子、RSV(劳氏肉瘤病毒(Rous sarcoma virus)启动子、HSV(单纯疱疹病毒)TK(胸苷激酶)启动子、腺病毒晚期启动子、痘苗病毒75K启动子、SV40启动子、金属硫蛋白启动子、CD45启动子(造血干细胞特异性启动子)、CD14启动子(单核特异性启动子)和癌细胞特异性启动子(肿瘤特异性启动子),例如生存素(Survivin)、中期因子(Midkine)、TERT、CXCR4等。具体地说,当使用癌细胞特异性启动子时,只诱导癌细胞中衔接蛋白的表达,由此遏制正常细胞中的衔接蛋白的表达,从而提高了本发明的重组HSV的安全性。
除了启动子之外,表达衔接蛋白的盒还被配置成包括转录终止信号序列,转录终止信号序列是用作多聚(A)添加信号(多聚腺苷酸化信号)以提高转录的完整性和效率的序列。本领域中已知许多转录终止信号序列,可以在其中选择性地使用适当的序列,例如SV40转录终止信号序列、HSV TK(单纯疱疹病毒胸苷激酶)转录终止信号序列等等。
在不抑制HSV的增殖下使表达衔接蛋白的盒可表达地插入到HSV基因组中,并且这种插入可以在HSV基因组没有缺失的情况下进行,或者可以插入到缺失了HSV基因组中的一些或所有非必需基因的基因座中。当在HSV基因组没有缺失下插入表达衔接蛋白的盒时,它可以插入到基因之间。插入基因座的优选实例包括介于UL3与UL4基因之间的基因座、介于UL26与UL27基因之间的基因座、介于UL37与UL38基因之间的基因座、介于UL48与UL49基因之间的基因座、介于UL53与UL54基因之间的基因座、介于US1与US2基因之间的基因座等。
当表达衔接蛋白的盒被插入到缺失了HSV基因组中的一些或所有非必需基因的基因座中时,缺失的非必需基因可以是如上文例示的任何非必需基因。
在本发明的重组HSV中,衔接蛋白的癌细胞靶向结构域是特异性地识别和结合于作为靶细胞的癌细胞的靶分子的位点,被癌细胞靶向结构域识别的靶分子是癌细胞表面上存在的任何抗原或任何受体。
抗原或受体优选是只在癌细胞中表达或者与正常细胞相比在癌细胞中过表达的抗原或受体。抗原或受体的实例可以包括例如以下的靶分子:EGFRvIII(表皮生长因子受体变体III),在胶质母细胞瘤中表达;EGFR(表皮生长因子受体),在未分化甲状腺癌、乳腺癌、肺癌、胶质瘤等中过表达;转移抑制素(metastin)受体,在乳头状甲状腺癌等中过表达;基于ErbB的受体酪氨酸激酶,在乳腺癌等中过表达;HER2(人表皮生长因子受体2),在乳腺癌、膀胱癌、胆囊癌、胆管细胞癌、食管胃交界部癌等中过表达;酪氨酸激酶-18-受体(c-Kit),在肉瘤样肾癌等中过表达;HGF受体c-Met,在食管腺癌等中过表达;CXCR4或CCR7,在乳腺癌等中过表达;内皮素-A受体,在前列腺癌中过表达;PPAR-δ(过氧化物酶体增殖物激活受体δ),在直肠癌等中过表达;PDGFR-α(血小板衍生生长因子受体α),在卵巢癌等中过表达;CD133,在肝癌、多发性骨髓瘤等中过表达;CEA(癌胚抗原),在肺癌、结肠直肠癌、胃癌、胰腺癌、乳腺癌、直肠癌、结肠癌、甲状腺髓样癌等中过表达;EpCAM(上皮细胞粘附分子),在肝癌、胃癌、结肠直肠癌、胰腺癌、乳腺癌等中过表达;GD2(双唾液酸神经节苷脂),在神经母细胞瘤等中过表达;GPC3(磷脂酰肌醇蛋白聚糖3),在肝细胞癌等中过表达;PSMA(前列腺特异性膜抗原),在前列腺癌等中过表达;TAG-72(肿瘤相关糖蛋白72),在卵巢癌、乳腺癌、结肠癌、肺癌、胰腺癌等中过表达;GD3(双唾液酸神经节苷脂),在黑色素瘤等中过表达;HLA-DR(人白细胞抗原-DR),在血癌、实体癌等中过表达;MUC1(粘蛋白1),在晚期实体癌等中过表达;NY-ESO-1(纽约食管鳞状细胞癌1),在晚期非小细胞肺癌等中过表达;LMP1(潜伏膜蛋白1),在鼻咽肿瘤等中过表达;TRAILR2(肿瘤坏死因子相关溶解诱导配体受体),在肺癌、非霍奇金淋巴瘤(non-Hodgkin’s lymphoma)、卵巢癌、结肠癌、结肠直肠癌、胰腺癌等中过表达;VEGFR2(血管内皮生长因子受体2)和HGFR(肝细胞生长因子受体),在肝细胞癌等中过表达。此外,癌症干细胞的表面抗原,例如CD44、CD166等,也可以是靶分子。本领域中已知与正常细胞相比在癌细胞中过表达的许多靶分子,除了上文列出的实例之外,其它的靶分子可以参考论文[Anne T Collins等人《致瘤性前列腺癌干细胞的未来鉴定(Prospective Identification of Tumorigenic Prostate Cancer Stem Cells).Cancer Res.2005年12月1日;65(23):10946-51]、论文[Chenwei Li等人《胰腺癌干细胞的鉴定(Identification of Pancreatic Cancer Stem Cells).Cancer Res.2007年2月1日;67(3):1030-7]、论文[Shuo Ma等人《实体瘤CAR-T细胞疗法的当前进展(Current Progress in CAR-T Cell Therapy for Solid Tumors)》.Int.J.Biol.Sci.2019年9月7日;15(12):2548-2560]、论文[Dhaval S.Sanchala等人《溶瘤单纯疱疹病毒疗法:大步迈向选择性靶向癌细胞(Oncolytic Herpes Simplex Viral Therapy:A Stride Toward Selective Targeting of Cancer Cells)》.Front Pharmacol.2017年5月16日;8:270]等。
具体地说,在本发明中,靶分子优选是HER2或CEA。
本发明的重组HSV的衔接蛋白所靶向的靶细胞是具有本发明的衔接蛋白的癌细胞靶向结构域所靶向的靶分子的任何癌细胞。癌细胞可以是任何类型的癌瘤,例如食管癌、胃癌、结肠直肠癌、直肠癌、口腔癌、咽癌、喉癌、肺癌、结肠癌、乳腺癌、宫颈癌、子宫内膜癌、卵巢癌、前列腺癌、睾丸癌、黑色素瘤、膀胱癌、肾癌、肝癌、胰腺癌、骨癌、结缔组织癌、皮肤癌、脑癌、甲状腺癌、白血病、霍奇金病(Hodgkin’s disease)、淋巴瘤、多发性骨髓瘤、血癌等。
在本发明中,除了具有与靶分子特异性结合的能力的完全抗体之外,衔接蛋白的细胞靶向结构域还可以是抗体衍生物或抗体类似物。抗体衍生物是完全抗体的片段,它包括至少一个具有与靶分子特异性结合的能力的抗体可变区;或者是修饰抗体。抗体衍生物的实例可以包括抗体片段,例如Fab、scFv、Fv、VhH、VH、VL等;多价或多特异性修饰抗体,例如Fab2、Fab3、微型抗体、双功能抗体、三功能抗体、四功能抗体、双scFv等;诸如此类。抗体类似物是像抗体一样,具有与靶分子特异性结合的能力,但在结构方面与抗体不同,一般分子量比抗体低的人工肽或多肽。抗体类似物的实例可以包括ABD、Adhiron、亲和体(affibody)、affilin类、affimer类、α抗体类(alphabody)、抗运载蛋白(anticalin)、犰狳重复蛋白、centyrin类、DARPin类、fynomer类、孔尼茨区(Kunitz region)、pronectin、重复体类(repebody)等。
本领域中已经累积了相当多的关于抗体、抗体衍生物、抗体类似物和它们的产生的论文,这些论文的实例包括论文[Renate Kunert和David Reinhart,《重组抗体制造的发展(Advances in recombinant antibody manufacturing)》.Appl.Microbiol.Biotechnol.2016年4月;100(8):3451-61]、论文[Holliger P,Hudson P.J.,《工程化抗体片段和单结构域的崛起(Engineered antibody fragments and the rise of single domains)》,Nat.Biotechnol.2005年9月;23(9):1126-36]、论文[Xiaowen Yu等人,《作为结合搭配物,除了抗体以外:抗体模拟物在生物分析中的作用(Beyond Antibodies as Binding Partners:The Role of Antibody Mimetics in Bioanalysis)》,Annual Review of Analytical Chemistry,2017,10:293-320]、论文[Abdul Rasheed Baloch等人,《抗体模拟物:有望与动物来源抗体互补的试剂(Antibody mimetics:promising complementary agents to animal-sourced antibodies)》,Critical Reviews in Biotechnology,2016,36:268-275]等等。
在本发明中,衔接蛋白的细胞靶向结构域优选地是scFv(单链可变片段)。scFv是指一种单链抗体,其中免疫球蛋白的重链可变区(VH)与轻链可变区(VL)经由短的连接肽连接。在scFv中,VH的C端连接于VL的N端,或者VL的C端连接于VH的N端。在scFv中,连接肽可以是具有任何长度和任何序列的连接肽,只要它不干扰重链和轻链的固有三维结构并使重链和轻链能在空间上彼此相邻,由此具有与靶分子特异性结合的能力即可。考虑到柔韧性、溶解度、对蛋白质水解的抗性等,连接子优选地由选自例如Ser、Gly、Ala、Thr等氨基酸中的至少一个构成,并且长度是1-30个氨基酸,优选地3-25个氨基酸,并且更优选地8-20个氨基酸。
在本发明中,scFv靶向的靶分子是HER2或CEA。明确地说,针对HER2的scFv优选地被配置成SEQ ID NO:1的VH和SEQ ID NO:2的VL以VH、连接肽和VL的顺序经由连接肽连接(即,VH的C端经由连接肽连接于VL的N端),针对CEA的scFv优选地被配置成SEQ ID NO:3的VL和SEQ ID NO:4的VH以VL、连接肽和VH的顺序经由连接肽连接(即,VL的C端经由连接肽连接于VH的N端)。这里,针对HER2的scFv的连接肽优选地包含SEQ ID NO:5的氨基酸序列,并且针对CEA的scFv的连接肽优选地包含SEQ ID NO:6的氨基酸序列。
在本发明中,除了在以下实例中使用的SEQ ID NO:7的HveA82(包含前导序列的HveA82序列在SEQ ID NO:8中表示)之外,HVEM的胞外结构域可以是SEQ ID NO:9的HveA87(包含前导序列的HveA87序列在SEQ ID NO:10中表示)、SEQ ID NO:11的HveA102(包含前导序列的HveA102序列在SEQ ID NO:12中表示)或SEQ ID NO:13的HveA107(包含前导序列的HveA107序列在SEQ ID NO:14中表示),在第10-0937774号韩国专利和第8318662号美国专利(这些文献被认为是本说明书的一部分)中公开。如第10-0937774号韩国专利中证实的,HveA87、HveA102和HveA107分别比HveA82多5个、20个和25个氨基酸,所有这些都可以用作衔接蛋白的HSV受体。
在本发明中,连接序列可以插入癌细胞靶向结构域与HVEM胞外结构域之间,并且该连接序列可以是具有任何长度和任何序列的连接子,只要它不抑制衔接蛋白的每个结构域的功能即可。优选地,连接序列包含四种氨基酸Ser、Gly、Ala和Thr中的至少一种氨基酸,并且它的长度可以是1-30个氨基酸,优选地3-25个氨基酸,更优选地8-20个氨基酸。
本发明的衔接蛋白还可以按NH2/癌细胞靶向结构域/HVEM胞外结构域/COOH的顺序或与之相反的顺序来配置。当连接肽插入中间时,衔接蛋白可以按NH2/癌细胞靶向结构域/连接肽/HVEM胞外结构域/COOH的顺序或与之相反的顺序来配置。
本发明的衔接蛋白可以被配置成:前导序列还可以连接于它的N端,具体地说,在呈NH2/癌细胞靶向结构域/连接肽/HVEM胞外结构域/COOH顺序的衔接蛋白构型中,连接于癌细胞靶向结构域的N端(当使用scFv时VH或VL的N端);或在呈NH2/HVEM胞外结构域/连接肽/癌细胞靶向结构域/COOH顺序的衔接蛋白构型中,连接于HVEM胞外结构域的N端。前导序列是一种用于诱导衔接蛋白在靶细胞中表达并将衔接蛋白释放到细胞外的序列,也可以省略掉,因为衔接蛋白只在溶解靶细胞和释放HSV以后才会用于诱导HSV感染相邻的靶细胞。
在本发明中,为了便于克隆,当针对靶分子的scFv用作细胞靶向结构域时,与任何限制酶位点对应的氨基酸可以插入VH与VL之间,当连接肽布置在VH与VL之间时,插入VH或VL与连接肽之间;可以插入scFv与HVEM之间,当连接肽布置在scFv与HVEM之间时,插入scFv与连接肽之间,或连接肽与HVEM之间。举例来说,限制酶EcoRI起作用的EF(碱基顺序:GAATTC)或BamHI起作用的GS(碱基顺序:GGATCC)可以如以下实例中所示来插入。
在本发明中,为了表达单独或任何组合的因子以诱导或增强对癌细胞的免疫应答,重组HSV可以被配置成将对应因子的基因插入到HSV基因组中。这类因子可以被操纵成表达细胞因子、趋化因子、免疫检查点拮抗剂(例如抗体、抗体衍生物或抗体类似物,特别是scFv)、能够诱导免疫细胞(T细胞或NK细胞)激活的共刺激因子、能够抑制遏制对癌细胞的免疫应答的TGFβ功能的拮抗剂(例如抗体、抗体衍生物或抗体类似物,特别是scFv)、能够降解实体瘤微环境的硫酸乙酰肝素蛋白聚糖的乙酰肝素酶、能够抑制血管生成受体VEGFR-2(VEGF受体-2)的功能的拮抗剂(例如抗体、抗体衍生物或抗体类似物,特别是scFv)等等。
作为细胞因子,举例来说,例如IL-2、IL-4、IL-7、IL-10、IL-12、IL-15、IL-18、IL-24等白细胞介素、例如IFNα、IFNβ、IFNγ等干扰素、例如TNFα等肿瘤坏死因子和例如GM-CSF、G-CSF等集落刺激因子可以单独使用或以其中的两种或更多种细胞因子的任何组合使用,以便在重组HSV中表达。
作为趋化因子,举例来说,CCL2(C-C基元趋化因子配体2)、CCL5(RANTES)、CCL7、CCL9、CCL10、CCL12、CCL15、CCL19、CCL21、CCL20和XCL-1(X-C基元趋化因子配体1)可以单独使用或组合使用以便在重组HSV中表达。
作为免疫检查点抗体,针对PD-1(程序性细胞死亡1)、PD-L1(程序性细胞死亡配体1)、PD-L2(程序性细胞死亡配体2)、CD27(分化簇27)、CD28(分化簇28)、CD70(分化簇70)、CD80(分化簇80)、CD86(分化簇86)、CD137(分化簇137)、CD276(分化簇276)、KIR(杀伤细胞免疫球蛋白样受体)、LAG3(淋巴细胞活化基因3)、GITR(糖皮质激素诱导的TNFR相关蛋白)、GITRL(糖皮质激素诱导的TNFR相关蛋白配体)和CTLA-4(溶细胞性T淋巴细胞相关抗原-4)的拮抗剂可以单独使用或组合使用以便在重组HSV中表达。
作为共刺激因子,CD2、CD7、LIGHT、NKG2C、CD27、CD28、4-1BB、OX40、CD30、CD40、LFA-1(淋巴细胞功能相关抗原-1)、ICOS(诱导型T细胞共刺激物)、CD3γ、CD3δ和CD3ε可以单独使用或组合使用以便在重组HSV中表达。
在本发明中,重组HSV可以被操纵成表达前药激活酶,所述前药激活酶将前药转化成对癌细胞有毒的药物。前药激活酶的实例可以包括胞嘧啶脱氨酶,其将作为前药的5-FC(5-氟胞嘧啶)转化成作为药物的5-FU(5-氟尿嘧啶);大鼠细胞色素P450(CYP2B1),其将作为前药的CPA(环磷酰胺(cyclophosphamide))转化成作为药物的PM(磷酰胺氮芥(phosphoramide mustard));羧酸酯酶,其将作为前药的伊立替康(irinotecan,SN-38150)转化成作为药物的SN-38;细菌硝基还原酶,其将作为前药的BC1954转化成作为DNA交联剂的4-羟胺151;从大肠杆菌分离的PNP(嘌呤核苷磷酸化酶),其将作为前药的6-甲基嘌呤-2’-脱氧核糖核苷转化成作为药物的6-甲基嘌呤等等。
此外,在本发明中,重组HSV可以被操纵成表达TRAIL(TNF相关溶解诱导配体)。已知TRAIL通过结合于它的在癌细胞中过表达的受体来诱导癌细胞的溶解(Kaoru Tamura等人《多机制性肿瘤靶向溶瘤病毒克服了脑肿瘤中的抗性(Multimechanistic Tumor Targeted Oncolytic Virus Overcomes Resistance in Brain Tumors)》.Mol.Ther.2013年1月;21(1):68-77)。
关于因子或前药活化酶用于诱导或增强这些免疫应答的更多细节,可以参考论文[Michele Ardolino等人,《癌症免疫疗法中的细胞因子治疗(Cytokine treatment in cancer immunotherapy)》,J.Oncotarget,Oncotarget.2015年8月14日;6(23):]、论文[Bernhard Homey等人《趋化因子:用于癌症免疫疗法的药剂(Chemokines:Agents for the Immunotherapy of Cancer)》.Nat Rev Immunol.2002年3月;2(3):175-84]、论文[Marianela Candolfi等人,《评估促细胞凋亡转基因与Flt3L组合用于多形性胶质母细胞瘤(GBM)的免疫刺激基因疗法中(Evaluation of proapoptotic transgenes to use in combination with Flt3L in an immune-stimulatory gene therapy approach for Glioblastoma multiforme(GBM)),J.FASEB J.,2008,22:107713]、论文[Danny N Khalil等人《癌症治疗的未来:免疫调节、CAR和组合免疫疗法(The Future of Cancer Treatment:Immunomodulation,CARs and Combination Immunotherapy)》.Nat Rev Clin Oncol.2016年5月;13(5):273-90]、论文[Paul E Hughes等人《用于治疗癌症的靶向疗法和检查点免疫疗法组合(Targeted Therapy and Checkpoint Immunotherapy Combinations for the Treatment of Cancer.Trends Immunol)》.2016年7月;37(7):462-476]、论文[Cole Peters,Samuel D.Rabkin,《设计疱疹病毒为溶瘤形式(Designing herpes viruses as oncolytics)》,Mol.Ther Oncolytics.2015;2:15010]等等。
在本发明中,如上述衔接蛋白中,诱导或增强免疫应答的因子或前药活化酶被配置成在不抑制HSV的增殖下将它们基因的表达盒(即,它们的基因与使它们能够表达的启动子序列和多聚腺苷酸化信号序列可操作地连接的构建体)插入到HSV基因组中。这种插入可以在HSV基因组没有缺失的情况下进行,或者可以插入到缺失了HSV基因组中的一些或所有非必需基因的基因座中。这里,在HSV基因组没有缺失的情况下插入时,可以插入基因之间,例如,优选的插入基因座是介于UL3与UL4基因之间、介于UL26与UL27基因之间、介于UL37与UL38基因之间、介于UL48与UL49基因之间、介于UL53与UL54基因之间以及介于US1与US2基因之间。当插入到缺失了非必需基因的基因座中或在非必需基因没有缺失的情况下插入到基因中时,可以从如上所述的任何非必需基因中选择这种非必需基因。
本发明的另一方面涉及一种用于抗癌治疗的药物组合物,其含有上述重组HSV作为活性成分。
本发明的药物组合物对表达靶分子的癌瘤具有抗癌作用,所述靶分子被重组HSV所表达的衔接蛋白的靶向结构域靶向。癌瘤的实例如上文关于靶分子所描述。
具体地说,优选地,本发明的组合物对具有表达CEA或HER2的肿瘤细胞的癌瘤具有抗癌作用。表达CEA的肿瘤细胞的实例包括结肠直肠癌细胞、胃癌细胞、肺癌细胞、乳腺癌细胞、直肠癌细胞、结肠癌细胞和肝癌细胞,表达HER2的肿瘤细胞的实例包括乳腺癌细胞、卵巢癌细胞、胃癌细胞、肺癌细胞、头颈癌细胞、骨肉瘤细胞、多形性胶质母细胞瘤细胞和唾液腺肿瘤细胞。
在本发明中,抗癌作用包括溶解癌细胞、降低癌细胞活力、因遏制癌细胞增殖而抑制或延迟癌症的病理性症状、抑制或延迟这类病理性症状的发作、抑制癌转移和抑制癌症复发。
除了重组HSV作为活性成分之外,本发明的药物组合物还可以包括重组衔接分子。重组衔接分子意指通过重组过程产生一种融合蛋白,所述融合蛋白具有如重组HSV所表达的衔接蛋白中的,或更精确地说,如癌细胞靶向结构域与HVEM胞外结构域的融合蛋白中的癌细胞靶向结构域。这里,具有如重组HSV所表达的衔接蛋白中的癌细胞靶向结构域意指,当HSV所表达的衔接蛋白的癌细胞靶向结构域靶向的靶分子是HER2时,衔接分子的癌细胞靶向结构域靶向的靶分子也是HER2。使用重组过程产生感兴趣的靶蛋白的方法通常包括制备能够表达靶蛋白的表达载体并将表达载体转化到宿主细胞如大肠杆菌、酵母或动物细胞(CHO细胞、NSO细胞、BHK细胞、Sp2细胞或HEK-293细胞)中,随后进行培养,然后分离靶蛋白,使用重组过程产生感兴趣的靶蛋白的方法是本领域中众所周知的(Sambrook等人,Molecular Cloning,A Laboratory Manual,Cold Spring Harbor Laboratory Press,(2001))。具体地说,关于本发明中所用的重组衔接分子的产生,可以参考第10-0937774号韩国专利和第8318662号美国专利。本发明的药物组合物还包括这类重组衔接分子,因此,当作为本发明的活性成分的重组HSV和重组衔接分子一起施用于患者时,重组HSV的初始感染效率将得到有效提高。
此外,本发明的药物组合物可以与批准的抗癌剂组合使用或呈与批准的抗癌剂的混合物形式使用。抗癌剂的实例可以包括对癌细胞表现出细胞毒性的任何抗癌剂、任何细胞因子药物、任何抗体药物、任何免疫检查点抑制剂药物和任何细胞治疗剂(对于car-T细胞疗法或car-NK细胞疗法),例如代谢拮抗剂、烷基化剂、拓扑异构酶拮抗剂、微管拮抗剂和植物来源的生物碱。它们的具体实例可以包括紫杉酚(taxol)、氮芥(nitrogen mustard)、伊马替尼(imatinib)、奥沙利铂(oxaliplatin)、吉非替尼(gefitinib)、硼替佐米(bortezomib)、舒尼替尼(sunitinib)、卡铂(carboplatin)、顺铂(cisplatin)、利妥昔单抗(rituximab)、埃罗替尼(erlotinib)、索拉非尼(sorafenib)、IL-2药物、IFN-α药物、IFN-γ药物、曲妥珠单抗(trastuzumab)、博纳吐单抗(blinatumomab)、伊匹单抗(ipilimumab)、派姆单抗(pembrolizumab)、纳武单抗(nivolumab)、阿特珠单抗(atezolizumab)、度伐鲁单抗(durvalumab)、贝伐单抗(bevacizumab)、西妥昔单抗(cetuximab)、替沙仑赛(tisagenlecleucel,Kymriah)、阿基仑赛(axicabtagene ciloleucel,Yescarta)等等。除了例示的抗癌剂之外,本领域中已知的其它的抗癌剂也可以(不限于)与本发明的药物组合物组合或呈与本发明的药物组合物的混合物形式使用。
本发明的药物组合物可以包括药学上可接受的载体或赋形剂,因此通过本领域中已知的典型方法制备成口服制剂或肠胃外制剂形式,取决于施用途径。
这类药学上可接受的载体或赋形剂不会削弱药物的活性或特性,本身对人体没有毒性,它的实例可包括乳糖、右旋糖、蔗糖、山梨糖醇、甘露糖醇、淀粉、阿拉伯胶、磷酸钙、海藻酸盐、明胶、硅酸钙、微晶纤维素、聚乙烯吡咯烷酮、纤维素、水(例如盐水和无菌水)、糖浆、甲基纤维素、羟基苯甲酸甲酯、羟基苯甲酸丙酯、滑石、硬脂酸镁、矿物油、林格氏溶液(Ringer’s solution)、缓冲剂、麦芽糖糊精溶液、丙三醇、乙醇、右旋糖苷、白蛋白以及它们的任何组合。具体地说,当本发明的药物组合物被配制成液体溶液形式时,适当的载体或赋形剂可包括盐水、无菌水、林格氏溶液、缓冲盐水、白蛋白注射溶液、右旋糖溶液、麦芽糖糊精溶液、丙三醇和乙醇,它们可以单独使用或组合使用。必要时,可以添加和使用其它典型的药物添加剂,例如抗氧化剂、缓冲剂和抑菌剂。
当本发明的药物组合物被制备成口服制剂时,它可以制成片剂、药片、胶囊、酏剂、悬浮液、糖浆、糯米纸等形式,当被制备成肠胃外制剂,特别是注射液时,它可以制成单位剂量安瓿或多剂量形式。本发明的药物组合物还可以制成溶液、悬浮液、片剂、丸剂、胶囊、持续释放制剂等形式。
本发明的药物组合物可以根据制药领域中的典型方法配制成适合施用于患者身体的单位剂型,并且可以使用本领域中通常使用的任何施用方法通过口服施用途径或肠胃外施用途径施用,例如皮肤、病灶内、静脉内、肌内、动脉内、髓内、鞘内、心室内、经肺、经皮、皮下、腹膜内、鼻内、消化道内、局部、舌下、阴道内和直肠途径。
本发明的药物组合物的剂量(有效量)可以视如下因素而变化:配制方法、施用模式、患者年龄、体重和性别、病理状态、饮食、施用时间、施用途径、排泄率和反应敏感性,宜由本领域技术人员考虑这些因素来决定。在一个优选的实施例中,本发明的药物组合物被制备成呈单位剂型的注射液。当制备成单位剂型的注射液时,每单位剂量的本发明的药物组合物包括的重组HSV的量可以在102-1014pfu,尤其是104-1011pfu范围内。
本发明的又一方面涉及一种治疗或预防癌症(肿瘤)的方法,所述方法包括向受试者如患者施用有效量的含有如上所述的重组HSV的药物组合物。
所述治疗癌症的方法通过溶解和杀死具有重组HSV的衔接蛋白的癌细胞靶向结构域所靶向的靶分子的癌细胞使得治疗癌症成为可能。因此,本发明的治疗方法可以应用于具有这类靶分子的任何癌瘤。具体地说,本发明的治疗方法优选应用于表达CEA或HER2的癌瘤。
本发明的治疗方法可以(不限于)与如上所述的其它癌症治疗方法组合使用。举例来说,可以在施用本发明的组合物之前或之后,或以与本发明的组合物组合同时施用的方式,使用如上例示的细胞毒性抗癌剂、细胞因子药物、抗体药物、免疫检查点抑制剂药物、细胞治疗剂(car-T细胞疗法或car-NK细胞疗法)、放射疗法、外科手术等。
在本发明的治疗方法中,有效量是当本发明的药物组合物施用到受试者如患者,待续基于医学专家建议的施用期时,使本发明的药物组合物施用后表现出预期医疗作用,例如癌症治疗或预防作用的量等。如上所述,这样的有效量宜由本领域的技术人员如医学专家等依据患者的年龄、体重和性别、病理状态等来决定,如上所述。
在本发明的治疗方法中,药物组合物优选地以肠胃外施用模式,例如病灶内(肿瘤内)、静脉内、肌内、动脉内施用等,呈注射到患者的注射液形式等施用。
如上所述,根据本发明,可以提供一种重组HSV,其含有能够表达细胞靶向结构域与HVEM胞外结构域的融合蛋白的表达盒;以及其用途。
当重组HSV感染靶细胞,即癌细胞,并进入靶细胞时,HSV会增殖,并且作为融合蛋白的衔接蛋白在细胞中表达,在细胞溶解后与增殖的HSV病毒粒子一起释放到细胞外,或者当衔接蛋白含有前导序列时,甚至在病毒粒子因细胞溶解而被释放前不断地释放。释放到细胞外的融合蛋白用于诱导HSV病毒粒子感染周围的表达被癌细胞靶向结构域识别的靶分子的癌细胞,或者提高感染效率。
附图简单说明
图1示意性示出了KOS-37BAC的基因组结构和通过Cre-Lox系统切除BAC;
图2示意性示出了作为HVEM限制的HSV-1的KOS-gD-R222N/F223I病毒的基因组结构;
图3示意性示出了作为HVEM限制的表达EmGFP的HSV-1的KOS-EmGFP-gD-R222N/F223I病毒的基因组结构;
图4示出了HVEM限制的表达EmGFP的HSV-1的荧光表达以及具有HVEM受体的细胞的特异性感染的结果;
图5示意性示出了表达HER2scFv-HveA衔接蛋白的KOS-HER2scFv-HveA-EmGFP-gD/R222N/F223I病毒的基因组结构、表达HveA-HER2scFv衔接蛋白的KOS-HveA-HER2scFv-EmGFP-gD/R222N/F223I病毒的基因组结构和表达CEAscFv-HveA衔接蛋白的KOS-CEAscFv-HveA-EmGFP-gD/R222N/F223I病毒的基因组结构;
图6示出了HER2scFv-HveA衔接蛋白、HveA-HER2scFv衔接蛋白和CEAscFv-HveA衔接蛋白的整个序列以及相应的序列;
图7示出了用表达HER2scFv-HveA衔接蛋白的KOS-HER2scFv-HveA-EmGFP-gD/R222N/F223I病毒特异性感染表达HER2的细胞的结果;
图8示出了用表达HER2scFv-HveA衔接蛋白的KOS-HER2scFv-HveA-EmGFP-gD/R222N/F223I病毒特异性溶解表达HER2的细胞的结果;
图9示出了用野生型病毒(HSV-1KOS)和表达HER2scFv-HveA衔接蛋白的KOS-HER2scFv-HveA-EmGFP-gD/R222N/F223I病毒(Her2衔接蛋白)溶解表达HER2的细胞的比较结果;
图10示出了用表达CEAscFv-HveA衔接蛋白的KOS-CEAscFv-HveA-EmGFP-gD/R222N/F223I病毒特异性感染表达CEA的细胞的结果;
图11示出了用作为HVEM限制的表达EmGFP的HSV-1的KOS-EmGFP-gD-R222N/F223I病毒(gD/NI)和用表达CEAscFv-HveA衔接蛋白的KOS-CEAscFv-HveA-EmGFP-gD/R222N/F223I病毒特异性感染表达CEA的细胞的比较结结果;
图12示出了用表达HER2scFv-HveA衔接蛋白的KOS-HER2scFv-HveA-EmGFP-gD/R222N/F223I病毒和表达HveA-HER2scFv衔接蛋白的KOS-HveA-HER2scFv-EmGFP-gD/R222N/F223I病毒特异性感染的比较结果;以及
图13示出了表达HER2scFv-HveA衔接蛋白的病毒的衔接蛋白的胞外表达结果和病毒感染传播至周围癌细胞的观测结果。
特定实施例的描述
通过以下实例,将更好地了解本发明。然而,不应将这些实例视为限制本发明的范围。
<实例1>HVEM限制的HSV-1的产生
HSV-1基因由约152kb大基因构成,因此使用KOS-37BAC(GenBank登录号MF156583)(Gierasch W.W.等人J.Virol.Methods.2006.135:197-206)将外源基因插入或将突变引入到特定的基因座中。HSV-1KOS病毒株是一种主要用于实验室中的HSV-1病毒株,因为它具有众所周知的特征并且适用于研究基因功能和病源学(Smith KO.Proc.Soc.Exp.Biol.Med.1964.115:814-816)。KOS-37BAC是通过将BAC质粒插入到KOS基因中制成的,它能够通过DH10B细菌(Invitrogen)的转化在细菌水平下克隆(Gierasch W.W.等人;J.Virol.Methods.2006.135:197~206)。在KOS-37BAC中,细菌人工染色体(BAC)与两侧的LoxP位点一起插入到HSV-1KOS基因组的UL37与UL38之间的基因座中。这被设计成在后续程序中使用Cre-Lox系统去除BAC基因。图1中示出了示意图。
为了制造HVEM限制的HSV-1(其只通过HVEM细胞受体进入细胞),制造出gD-R222N/F223IHSV-1病毒,其中HSV-1gD氨基酸序列(GenBank登录号ASM47818,SEQ ID NO:15)的位置222处的精氨酸(R)和位置223处的苯丙氨酸(F)分别经天冬酰胺(N)和异亮氨酸(I)取代。
通过突变制造的gD-R222N/F223I HSV-1病毒只通过HVEM(HveA)而不是粘连蛋白-1作为细胞进入受体才能感染宿主细胞(Uchida H等人,J.Virol.2009.83(7):2951-2961)。
图2中示意性示出了HVEM限制的HSV-1的基因组结构。
通过根据制造商的方案,使用反选择BAC修饰试剂盒(GeneBridges公司)将R222N/F223I突变引入到KOS-37BAC的gD位点中来制造gD-R222N/F223I HSV-1病毒。
明确地说,含有KOS-37BAC的大肠杆菌克隆用pRed/ET质粒转化,所述质粒表达能够执行同源重组功能的RecE和RecT(Muyrers J.P.等人;Nucleic Acids Res.1999.27(6):1555-1557)。使用一组同源区引物(正向引物gD-rpsL For:SEQ ID NO:16,反向引物gD-rpsL Rev:SEQ ID NO:17)来制造gD-rpsL-neo/kan盒,包括将突变引入gD中的基因座。gD-rpsL-neo/kan盒由以下构成:rpsL基因,它是赋予链霉素(streptomycin)敏感性的选择性标志物;和在插入基因座处的gD同源区;和赋予卡那霉素(kanamycin)抗性的neo/kan基因。当插入gD-rpsL-neo/kan盒时,制造出通过rpsL基因而对链霉素抗生素具有敏感性和通过neo/kan基因而具有卡那霉素抗性的大肠杆菌。在通过添加L-阿拉伯糖(Sigma-Aldrich)至含有KOS-37BAC和pRedET的大肠杆菌克隆而激活pRed/ET的功能来诱导RecE和RecT表达以便能够进行同源重组(Muyrers JP等人;Nucleic Acids Res.1999.27(6):1555-1557)之后,用200ng制造的gD-rpsL-neo/kan盒转化。通过同源重组,将gD-rpsL-neo/kan盒插入到KOS-37BAC的gD基因座中。gD-rpsL-neo/kan插入到KOS-37BAC中的大肠杆菌具有卡那霉素抗性,但链霉素抗性被rpsL基因阻挡。这是针对从卡那霉素培养基选择其中插入了gD-rpsL-neo/kan的大肠杆菌推测的,并进行插入基因的最后步骤。在通过添加L-阿拉伯糖(Sigma-Aldrich)至含有KOS 37-BAC gD-rpsL-neo/kan克隆的大肠杆菌而激活pRed/ET的功能来诱导RecE和RecT表达以便能够进行同源重组之后,用100pmol R222N_F223I_突变体转化,R222N_F223I_突变体是编码在gD的位置222和223处经N和I取代的gD的寡核苷酸(SEQ ID NO:18)。基于激活被rpsL阻挡的链霉素抗性同时用插入的寡核苷酸替换存在的gD-rpsL-neo/kan盒的原理,在链霉素培养基中选择候选物(Heermann R.等人,Microb.Cell Fact.2008.14:doi:10.1186)。在所选的候选物中,使用DNA制备方法分离DNA(Horsburgh BC等人,Methods Enzymol.1999.306:337-352),并通过PCR(聚合酶链式反应)和DNA测序来证实gD的相应位置222和223处的N和I取代。
然后,为了产生病毒,使用大型构建体DNA纯化试剂盒(Macherey-Nagel),提取完整的KOS-BAC-gD-R222N/F223I DNA,接着使用Lipofectamine 2000试剂(Invitrogen)用1μg DNA转染2×105Cre-Vero-HVEM细胞。然后,使用含有100U/ml青霉素(penicillin)/100μg/ml链霉素(Welgene)和10%FBS(胎牛血清,Welgene)的DMEM(达尔伯克改良伊格尔培养基(Dulbecco’sModified Eagle’s Medium))(Welgene)进行细胞培养。Cre-Vero-HVEM细胞系是一种通过将HVEM基因插入到Cre-Vero细胞系中来诱导HVEM蛋白表达的细胞系(Gierasch等人;J.Virol.Methods.2006.135:197~206)。使用Cre-Vero-HVEM的理由是可以使用细胞的Cre重组酶去除KOS 37BAC的BAC基因,而且由于HVEM过表达,故有效地感染KOS-gD-R222N/F223I病毒,因此容易大量生产病毒。在DNA转染之后3-4天,证实形成了斑点,接着收集含有病毒的细胞,进行冻融过程三次(Gierasch W.W.等人;J.Virol.Methods.2006.135:197~206)并进行超声波处理,最终获得了KOS-gD-R222N/F223I病毒。
<实例2>HVEM限制的表达EmGFP的HSV-1的产生
将能够表达EmGFP(翠绿荧光蛋白)的表达盒插入到以上实例1中制造的KOS-gD-R222N/F223I病毒的基因UL26/UL27基因座中。使用EmGFP作为标志物,便于观测病毒的产生和感染。使用pCDNA6.2-GW/EmGFP-miR质粒(Invitrogen)制造EmGFP盒。
图3中示意性示出了HVEM限制的表达UL26/27-EmGFP的HSV-1的基因组结构。
为了表达EmGFP,将使用巨细胞病毒的基因启动子和作为HSV TK(单纯疱疹病毒胸苷激酶)的多聚腺苷酸化信号的tkpA的pCMV-EmGFP-tkpA插入到KOS-BAC-gD-R222N/F223I中。
根据制造商的方案,使用如以上实例1中的反选择BAC修饰试剂盒(GenBridges.公司)进行所有插入方法。
明确地说,将含有KOS-BAC-gD-R222N/F223I基因组的大肠杆菌克隆用pRed/ET质粒转化,所述质粒表达能够执行同源重组功能的RecE和RecT(Muyrers JP等人;Nucleic Acids Res.1999.27(6):1555-1557)。使用一组同源区引物(正向引物UL26/27-rpsL_For:SEQ ID NO:19,反向引物UL26/27-rpsL_Rev:SEQ ID NO:20)来制造UL26/27-rpsL-neo/kan盒,包括将靶基因引入UL26与UL27之间的基因座。向含有KOS-BAC-gD-R222N/F223I和pRed/ET的克隆添加L-阿拉伯糖(Sigma-Aldrich),因此诱导同源重组,然后用200ng制造的UL26/27-rpsL-neo/kan盒转化。通过同源重组,将UL26/27-rpsL-neo/kan盒插入到KOS-BAC-gD-R222N/F223I的UL26/27基因座中。插入了UL26/27-rpsL-neo/kan的大肠杆菌具有卡那霉素抗性,但链霉素抗性被rpsL基因阻挡。这是针对从卡那霉素培养基选择的其中插入了UL26/27-rpsL-neo/kan的大肠杆菌推测的,并进行插入基因的最后步骤。
向含有UL26/27-rpsL-neo/kan盒的大肠杆菌添加激活pRed/ET功能的L-阿拉伯糖(Sigma-Aldrich),因此诱导同源重组,然后用200ng的UL26/27-tkpA-EmGFP-pCMV盒转化。使用pCDNA6.2-GW/EmGFP-miR质粒(Invitrogen)作为模板、正向引物UL26-tkpA_For(SEQ ID NO:21)和反向引物UL27-pCMV_Rev(SEQ ID NO:22)制造UL26/27-tkpA-EmGFP-pCMV盒。
基于激活被rpsL阻挡的链霉素抗性同时用插入的UL26/27-tkpA-EmGFP-pCMV替换存在的UL26/27-rpsL-neo/kan盒的原理,在链霉素培养基中选择候选物(Heermann R等人,Microb.Cell Fact.2008.14:doi:10.1186)。使用DNA制备方法(Horsburgh BC等人,Methods Enzymol.1999.306:337-352)从所选的候选物中分离DNA。通过EcoRI和XhoI处理和PCR(聚合酶链式反应)证实tkpA-EmGFP-pCMV引入UL26/27中,并通过PCR产物的测序鉴定确切的基因序列。
针对荧光蛋白的正常表达和病毒的产生进行一项实验。使用大型构建体DNA纯化试剂盒(Macherey-Nagel),提取完整的KOS-BAC-EmGFP-gD-R222N/F223I DNA,接着使用Lipofectamine2000试剂(Invitrogen)用1μg DNA转染2×105Cre-Vero-HVEM细胞。转染3天后,使用荧光显微镜观察EmGFP的荧光表达,并通过Cre-Vero-HVEM细胞的斑点形成观察病毒的产生。在证实形成了斑点后,收集含有病毒的细胞,进行冻融过程三次(Gierasch WW等人;J.Virol Methods.2006.135:197~206)并进行超声波处理,因此获得了KOS-EmGFP-gD-R222N/F223I病毒。
为了感染KOS-EmGFP-gD-R222N/F223I病毒和它的荧光表达,使用无HVEM的细胞系(J1和J-粘连蛋白)和表达HVEM的细胞系(J-HVEM)。J1细胞是缺乏作为病毒HSV-1受体的HVEM和粘连蛋白-1的幼仓鼠肾细胞系(Petrovic B.等人,2017.PLoS Pathog.19;13(4):e1006352)。J-粘连蛋白和J-HVEM细胞系是在J1细胞中分别过表达粘连蛋白-1和HVEM的细胞系(Petrovic B.等人,2017.PLoS Pathog.19;13(4):e1006352)。在含有100U/ml青霉素/100μg/ml链霉素(Welgene)和10%FBS(胎牛血清,Welgene)的DMEM(Welgene)中培养每个细胞系。将1×104个细胞用以上获得的KOS-EmGFP-gD-R222N/F223I病毒以10MOI(感染复数)感染,并在24小时后,使用荧光显微镜观察荧光蛋白的表达和病毒的感染(Baek H.J.等人,Mol.Ther.2011.19(3):507-514)。
结果在图4中示出,上部和下部的图像是分别使用荧光显微镜和光学显微镜获取的。参看图4的上部荧光显微镜图像,可以看到J1细胞系和J-粘连蛋白细胞系未被感染,只有J-HVEM细胞系被感染。
基于以上结果,证实了按照预期,容易通过荧光蛋白的表达观察KOS-EmGFP-gD-R222N/F223I病毒的感染,并且只使用HVEM,而不是粘连蛋白-1作为细胞进入受体来进入细胞成为可能。
<实例3>表达HER2scFv-HveA衔接蛋白、HveA-HER2scFv衔接蛋白和CEAscFv-HveA衔接蛋白的KOS-EmGFP-Gd-R222N/F223I病毒的产生
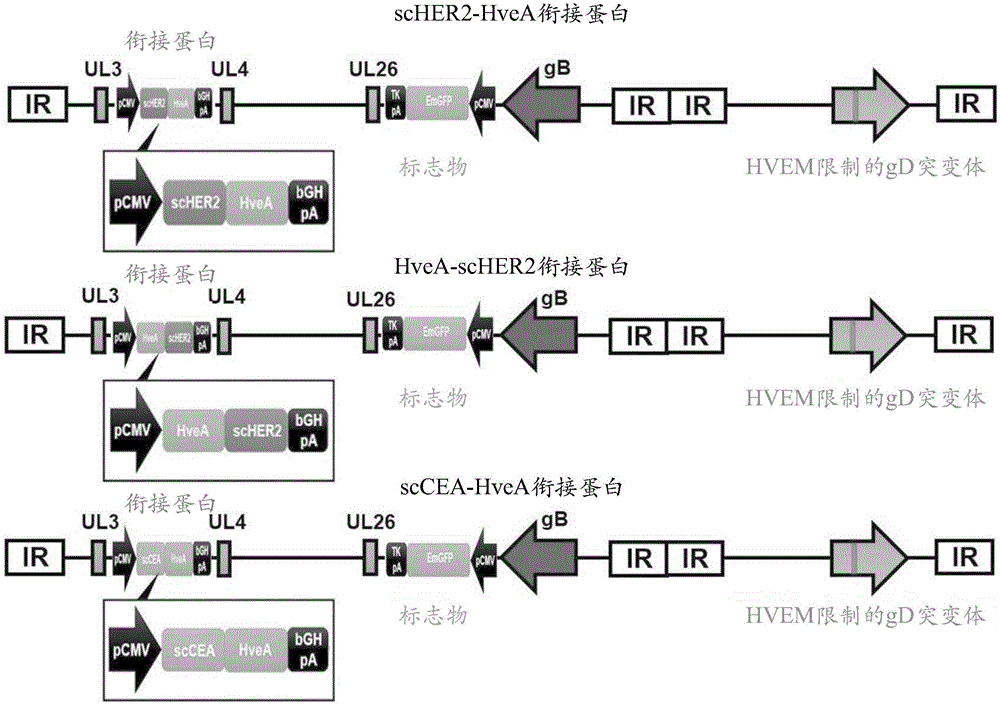
将表达HER2scFv-HveA衔接蛋白的盒、表达HveA-HER2scFv衔接蛋白的盒和表达CEAscFv-HveA衔接蛋白的盒每一者插入到实例2中制造的其中插入了EmGFP表达盒(tkpA-EmGFP-pCMV)的KOS-EmGFP-gD-R222N/F223I病毒的基因UL3/UL4基因座中。
图5示意性示出了其中插入了作为表达HER2scFv-HveA衔接蛋白的盒的pCMV-HER2scFv-HveA-bGHpA的KOS-HER2scFv-HveA-EmGFP-gD/R222N/F223I病毒基因组、其中插入了作为表达HveA-HER2scFv衔接蛋白的盒的pCMV-HveA-HER2scFv-bGHpA的KOS-HveA-HER2scFv-EmGFP-gD/R222N/F223I病毒基因组和其中插入了作为表达CEAscFv-HveA衔接蛋白的盒的pCMV-CEAscFv-HveA-bGHpA的KOS-CEAscFv-HveA-EmGFP-gD/R222N/F223I病毒基因组。此外,图6还示出了HER2scFv-HveA衔接蛋白、HveA-HER2scFv衔接蛋白和CEAscFv-HveA衔接蛋白的整个序列以及相应的序列。这里,HER2的scFv被配置成SEQ ID NO:1的VH和SEQ ID NO:2的VL经由SEQ ID NO:5的连接肽连接,CEA的scFv被配置成SEQ ID NO:3的VL和SEQ ID NO:4的VH经由SEQ ID NO:6的连接肽连接,并且在HER2scFv-HveA衔接蛋白和CEAscFv-HveA衔接蛋白中HveA是不包括前导序列的SEQ ID NO:7的HveA82,并且在HveA-HER2scFv衔接蛋白中是包括前导序列的SEQ ID NO:8的Hve82。同样地,在HER2scFv-HveA衔接蛋白和CEAscFv-HveA衔接蛋白中,SEQ ID NO:33的前导序列包括在N端中,即在HER2scFv的VH和CEAscFv的VL的前面。
在HER2或CEA的scFv序列和NH2-GGGGS序列(HveA序列的连接序列)之后,添加EF(碱基序列:GAATTC),它是容易克隆的限制酶EcoRI位点,并且在HveA序列和NH2-GGGGS序列(Her2的scFv序列的连接序列)之后,添加GS(碱基序列:GGATCC),它是容易克隆的限制酶BamHI位点,并且还是可以从衔接蛋白中除去的序列。bGHpA是bGH-多聚A(牛生长激素多聚腺苷酸化)信号序列。本实例中所用的HER2scFv-HveA衔接蛋白全长的氨基酸序列和基因序列分别在SEQ ID NO:23和SEQ ID NO:24中表示,HveA-HER2scFv衔接蛋白全长的氨基酸序列和基因序列分别在SEQ ID NO:25和SEQ ID NO:26中表示,并且CEAscFv-HveA衔接蛋白全长的氨基酸序列和基因序列分别在SEQ ID NO:27和SEQ ID NO:28中表示。
根据制造商的方案,使用如实例1和2中的反选择BAC修饰试剂盒(GeneBridges.公司)插入表达HER2scFv-HveA衔接蛋白的盒、表达HveA-HER2scFv衔接蛋白的盒和表达CEAscFv-HveA衔接蛋白的盒。
明确地说,将含有实例2中制造的KOS-BAC-EmGFP-gD-R222N/F223I基因组的大肠杆菌克隆用pRed/ET质粒转化,所述质粒表达能够执行同源重组功能的RecE和RecT(Muyrers JP等人;Nucleic Acids Res.1999.27(6):1555-1557)。使用一组同源区引物(正向引物HSV-1_UL3/4-rpsL-neo_for:SEQ ID NO:29,反向引物HSV-1_UL3/4-rpsL-neo_rev:SEQ ID NO:30)来制造UL3/4-rpsL-neo/kan盒,包括将靶基因引入UL3与UL4之间的基因座。向含有KOS-BAC-EmGFP-gD-R222N/F223I和pRedET的克隆添加L-阿拉伯糖(Sigma-Aldrich),因此诱导同源重组,然后用200ng的以上制造的UL3/4-rpsL-neo/kan盒转化。通过这类同源重组,将UL3/4-rpsL-neo/kan盒插入到KOS-BAC-EmGFP-gD-R222N/F223I的UL3/4基因座中。插入了UL3/4-rpsL-neo/kan的大肠杆菌具有卡那霉素抗性,但链霉素抗性被rpsL基因阻挡。这是针对从卡那霉素培养基选择其中插入了UL3/4-rpsL-neo/kan的大肠杆菌推测的,并进行插入靶基因的最后步骤。
向含有UL3/4-rpsL-neo/kan盒的大肠杆菌添加L-阿拉伯糖(Sigma-Aldrich),激活pRed/ET的功能,因此诱导同源重组,接着用200ng的UL3/4-pCMV-Her2scFv-HveA-bGHpA盒、UL3/4-pCMV-HveA-Her2scFv-bGHpA盒和UL3/4-pCMV-CEAscFv-HveA-bGHpA盒中的每一个盒转化。使用pCDNA3.1-HER2scFv-HveA质粒、pCDNA3.1-HveA-HER2scFv质粒和pCDNA3.1-CEAscFv-HveA质粒作为模板,使用正向引物HSV-1_UL3/4-HM_pCMV_For(SEQ ID NO:31)和反向引物UL3/4_bGH_poly_R(SEQ ID NO:32)制造UL3/4-pCMV-Her2scFv-HveA-bGHpA盒、UL3/4-pCMV-HveA-Her2scFv-bGHpA盒和UL3/4-pCMV-CEAscFv-HveA-bGHpA盒(Baek H.J.等人,Mol.Ther.2011.19(3):507-514)。
基于激活被rpsL阻挡的链霉素抗性同时用以上插入的UL3/4-pCMV-Her2scFv-HveA-bGHpA、UL3/4-pCMV-HveA-Her2scFv-bGHpA和UL3/4-pCMV-CEAscFv-HveA-bGHpA替换通常插入的UL3/4-rpsL-neo/kan盒的原理,在链霉素培养基中选择候选物(Heermann R等人,Microb Cell Fact.2008.14:doi:10.1186)。使用DNA制备方法(Horsburgh BC等人,Methods Enzymol.1999.306:337-352)从所选的候选物中分离DNA。通过限制酶EcoRI和XhoI处理以及PCR(聚合酶链式反应)证实UL3/4-pCMV-Her2scFv-HveA-bGHpA、UL3/4-pCMV-HveA-Her2scFv-bGHpA和UL3/4-pCMV-CEAscFv-HveA-bGHpA引入到UL3/4中,并通过PCR产物的测序来鉴定确切的基因序列。
使用大型构建体DNA纯化试剂盒(Macherey-Nagel),提取完整的KOS-BAC-Her2scFv-HveA-EmGFP-gD-R222N/F223I、KOS-BAC-HveA-Her2scFv-EmGFP-gD-R222N/F223和KOS-BAC-CEAscFv-HveA-EmGFP-gD/R222N/F223I DNA,接着使用Lipofectamine 2000试剂(Invitrogen)用1μg DNA转染2×105Cre-Vero-HVEM细胞。在转染3天后,使用荧光显微镜观察EmGFP的荧光表达和细胞的斑点形成。在证实形成了斑点后,收集含有病毒的细胞,进行冻融过程三次(Gierasch WW等人;J.Virol Methods.2006.135:197~206)并进行超声波处理,因此获得了KOS-Her2scFv-HveA-EmGFP-gD-R222N/F223I病毒、KOS-HveA-Her2scFv-EmGFP-gD-R222N/F223I病毒和KOS-CEAscFv-HveA-EmGFP-gD/R222N/F223I病毒。
<实例4>使用表达衔接蛋白的溶瘤病毒靶向表达HER2或CEA的癌细胞
<实例4-1>使用表达HER2scFv-HveA衔接蛋白的溶瘤病毒靶向表达HER2的癌细胞
为了评估通过使用实例3中制造的表达HER2scFv-HveA衔接蛋白的KOS-Her2scFv-HveA-EmGFP-gD-R222N/F223I病毒表达HER2scFv-HveA衔接蛋白是否诱导周围癌细胞的病毒感染或在感染之后溶解,进行以下的实验。
实验中使用的细胞系是不表达HER2的细胞系(J1、CHO-K1、MDA-MB-231)和表达HER2的细胞系(J-HER2、CHO-HER2、SK-OV-3)。中国仓鼠卵巢细胞系CHO-K1和CHO-HER2(KurokiM等人,J.Biol.Chem.1991.74:10132-10141)是使用含有100U/ml青霉素/100μg/ml链霉素(Welgene)和10%FBS(胎牛血清)的HaM’s F-12K培养基(Welgene)来培养的,并且J1和J-HER2(Petrovic B等人,2017.PLoS Pathog.19;13(4):e1006352)、乳腺癌细胞系MDA-MB-231(ATCC,HTB-26)和卵巢癌细胞系SK-OV-3(ATCC,HTB-77)是使用含有100U/ml青霉素/100μg/ml链霉素(Welgene)和10%FBS的DMEM来培养的。
对于HER2特异性病毒感染,将1×104J细胞系以10MOI、1.5×104CHO细胞系以1MOI和1×104SK-OV-3和MDA-MB-231细胞系以0.1MOI感染实例3中制造的表达HER2scFv-HveA衔接蛋白的病毒。在90分钟后,培养基替换为新鲜的培养基以去除残余的早期病毒和HER2scFv-HveA衔接蛋白。在感染3天后,使用荧光显微镜通过荧光表达观察每个细胞系的病毒感染(Baek H.J.等人,Mol.Ther.2011.19(3):507-514)。
结果在图7中示出,左侧和右侧的图像分别使用光学显微镜和荧光显微镜获取。参看图7右侧的荧光显微镜图像,可以证实,作为表达HER2的细胞系,CHO-Her2、J-Her2和SK-OV-3特异性地被感染。HSV-1感染通路的作用机制是将gB和gC附着至细胞并通过gD进入细胞。表达HER2scFv-HveA的gD R222N/F223I突变病毒引起缺乏HVEM和粘连蛋白-1的CHO-K1细胞系轻微感染的原因在于,除了gD的功能之外,轻微感染还通过gB和gC的细胞附着进行(Baek H.J.等人,Mol.Ther.2011.19(3):507-514)。
并且,作为表达HER2的细胞系,CHO-Her2、J-Her2和SK-OV-3尽管缺乏HVEM受体但仍首先感染上病毒。关于这一点的原因是,用于感染的在实例3中制造的表达HER2scFv-HveA衔接蛋白的病毒含有痕量的与病毒gD结合的HER2scFv-HveA衔接蛋白或在Cre-Vero-HVEM细胞产生病毒期间表达的HER2scFv-HveA衔接蛋白。
为了观察由病毒引起的溶解,将1.5×104CHO-K1和CHO-Her2细胞系以1MOI感染表达HER2scFv-HveA衔接蛋白的病毒,并将1×104SK-OV-3和MDA-MB-231细胞系以2MOI感染表达HER2scFv-HveA衔接蛋白的病毒。在感染3天后,使用光学显微镜观察每个细胞系。结果在图8中示出。在作为表达HER2的细胞系的CHO-Her2和SK-OV-3的情况下,可以看到细胞数目比作为不表达HER2的细胞系的CHO-K1和MDA-MB-231的细胞数目低得多。
并且,将作为表达HER2的细胞系的1×104SK-OV-3细胞以2MOI感染HVEM限制的疱疹病毒(gD/NI)和表达HER2scFv-HveA衔接蛋白的病毒(Her2衔接蛋白)中的每一种,并在第1天、第2天、第3天、第4天和第5天通过使用Alamar蓝(Sigma)的细胞染色观察到溶解。结果在图9中示出。可以看到表达HER2scFv-HveA衔接蛋白的病毒(Her2衔接蛋白)对诱导溶解的作用比通用的疱疹病毒(HSV-1KOS)高得多。
<实例4-2>使用表达CEAscFv-HveA衔接蛋白的溶瘤病毒靶向表达CEA的癌细胞
为了评估通过使用实例3中制造的表达CEAscFv-HveA衔接蛋白的KOS-CEAscFv-HveA-EmGFP-gD/R222N/F223I病毒表达CEAscFv-HveA是否诱导周围癌细胞的病毒感染,进行以下的实验。
实验中使用的细胞系是不表达CEA的细胞系(CHO-K1)和表达CEA的细胞系(CHO-CEA、MKN45)。中国仓鼠卵巢细胞系CHO-K1和CHO-HER2(Kuroki M等人,J.Biol.Chem.1991.74:10132-10141)是使用含有100U/ml青霉素/100μg/ml链霉素(Welgene)和10%FBS的HaM’s F-12K培养基(Welgene)来培养的,并且胃癌细胞系MKN45(JCRB、JCRB0254)是使用含有100U/ml青霉素/100μg/ml链霉素(Welgene)和10%FBS的RPMI-1640培养基来培养的(Baek H.J.等人,Mol.Ther.2011.19(3):507-514)。
关于CEA特异性病毒感染,将1.5×104CHO-K1和CHO-CEA细胞以10MOI感染病毒。在90分钟后,培养基替换为新鲜的培养基以去除残余的早期病毒和CEAscFv-HveA衔接蛋白。在72小时后,使用荧光显微镜观察每个细胞系的病毒感染程度。
结果在图10中示出,左侧和右侧的图像分别使用光学显微镜和荧光显微镜获取。参看图10右侧的荧光显微镜图像,CHO-K1细胞系很少被感染,观察到CHO-CEA细胞系被感染。
表达CEAscFv-HveA的gD R222N/F223I突变病毒引起缺乏HVEM和粘连蛋白-1的CHO-K1细胞系轻微感染的原因在于,如上所述,除了gD的功能之外,轻微感染还通过gB和gC的细胞附着进行(Baek H.J.等人,Mol.Ther.2011.19(3):507-514)。
并且,为了证实表达CEA的癌细胞的特异性感染,将6×104MKN45细胞系以1MOI感染在实例2中制造的KOS-EmGFP-gD-R222N/F223I病毒(gD/NI,对照)和在实例3中获得的KOS-CEAscFv-HveA-EmGFP-gD-R222N/F223I(scCEA-HveA)。在90分钟后,培养基替换为新鲜的培养基以去除残余的早期病毒和CEAscFv-HveA衔接蛋白。在72小时后,使用荧光显微镜观察每个细胞系的病毒感染程度。
结果在图11中示出,左侧和右侧的图像分别使用光学显微镜和荧光显微镜获取。参看右侧的图像,可以看到MKN45癌细胞特异性地感染上KOS-CEAscFv-HA-EmGFP-gD-R222N/F223I病毒,而没有感染上作为对照的KOS-EmGFP-gD-R222N/F223I病毒。
<实例5>表达衔接蛋白的溶瘤病毒的感染活性视HER2衔接蛋白的结构改变而变
为了评估通过使用实例3中制造的具有不同的衔接蛋白结构的表达HER2scFv-HveA衔接蛋白的KOS-Her2scFv-HveA-EmGFP-gD-R222N/F223I病毒和表达HveA-HER2scFv衔接蛋白的KOS-HveA-HER2scFv-EmGFP-gD-R222N/F223I病毒表达HER2scFv-HveA衔接蛋白或HveA-HER2scFv衔接蛋白,感染活性是否视衔接蛋白结构而变,进行以下的实验。
实验中使用的细胞系是不表达HER2的CHO-K1细胞系和表达HER2的CHO-HER2细胞系。中国仓鼠卵巢细胞系CHO-K1和CHO-HER2(Kuroki M等人,J.Biol.Chem.1991.74:10132-10141)是使用含有100U/ml青霉素/100μg/ml链霉素(Welgene)和10%FBS(胎牛血清)的HaM’s F-12K培养基(Welgene)来培养的。
为了评估病毒感染的活性取决于衔接蛋白结构的变化,将1.5×104CHO-K1和CHO-HER2细胞系以1MOI感染在实例3中制造的表达HER2scFv-HveA衔接蛋白的病毒和表达HveA-HER2scFv衔接蛋白的病毒。在90分钟后,培养基替换为新鲜的培养基以去除残余的早期病毒和HER2scFv-HveA和HveA-HER2scFv衔接蛋白。在感染3天后,将细胞用VP16染色,VP16是一种对病毒的早期基因转录来说不可或缺的蛋白质,并使用荧光显微镜观察具有不同的衔接蛋白结构的表达衔接蛋白的病毒的感染情况(Baek H.J.等人,Mol.Ther.2011.19(3):507-514)。
参看图12右侧的图像,通过VP16染色示出了CHO-HER2细胞感染具有不同的衔接蛋白结构的表达衔接蛋白的病毒的程度,表达HER2scFv-HveA和HveA-HER2scFv衔接蛋白的病毒的感染水平相仿。在表达衔接蛋白的系统中,证实了感染程度不会因HveA的位置而不同,并证实了对于两种结构来说,HER2特异性感染活性都较高。DAPI(4’,6-二脒基-2-苯基吲哚)用作通过核酸染色的VP16染色的比较观察指标。
表达HER2scFv-HveA或HveA-HER2scFv的gD R222N/F223I突变病毒引起缺乏HVEM和粘连蛋白-1的CHO-K1细胞系轻微感染的原因在于,如上所述,除了gD的功能之外,轻微感染还通过gB和gC的细胞附着进行(Baek H.J.等人,Mol.Ther.2011.19(3):507-514)。
并且,作为表达HER2的细胞系,CHO-Her2尽管缺乏HVEM受体但仍首先感染上病毒。关于这一点的原因是,用于感染的在实例3中制造的表达HER2scFv-HveA或HveA-HER2scFv衔接蛋白的病毒含有痕量的与病毒gD结合的HER2scFv-HveA或HveA-HER2scFv衔接蛋白或在Cre-Vero-HVEM细胞产生病毒期间表达的HER2scFv-HveA。
<实例6>表达衔接蛋白的病毒的衔接蛋白的胞外表达和病毒传播到周围癌细胞的观察
为了证实细胞中由实例3中制造的表达HER2scFv-HveA衔接蛋白的KOS-Her2scFv-HveA-EmGFP-gD-R222N/F223I病毒表达的HER2scFv-HveA衔接蛋白释放到细胞外,进行以下的实验。
实验中使用的细胞系是Vero-HVEM细胞系(Gierasch等人;J.Virol.Methods.2006.135:197~206)。Vero-HVEM细胞系是使用含有100U/ml青霉素/100μg/ml链霉素(Welgene)和10%FBS(胎牛血清,Welgene)的DMEM(达尔伯克改良伊格尔培养基)(Welgene)来培养的。将2.0×105Vero-HVEM细胞以0.1MOI感染KOS-Her2scFv-HveA-EmGFP-gD-R222N/F223I和作为对照的KOS-EmGFP-gD-R222N/F223I病毒。在90分钟后,培养基替换为不含FBS的新鲜的培养基以去除残余的早期病毒和HER2scFv-HveA衔接蛋白。48小时后,收集培养基。通过蛋白质印迹法(Western blotting)测量蛋白质的表达程度,以证实所收集的培养基中HER2scFv-HveA衔接蛋白的表达。
结果在图13顶部示出。如图13顶部的结果所示,在从感染不含衔接蛋白的KOS-EmGFP-gD-R222N/F223I病毒(gD/NI)的组获得的培养基中没有检测到衔接蛋白的表达,但在从感染KOS-Her2scFv-HveA-EmGFP-gD-R222N/F223I病毒(Her2scFv-HveA)的组获得的培养基中检测到释放到细胞外的衔接蛋白。
基于以上结果证实,按照预期,插入的衔接蛋白的表达和它释放到细胞外通过KOS-Her2scFv-HveA-EmGFP-gD-R222N/F223I病毒的细胞内感染有效地进行。
此外,为了观察病毒感染是由于细胞溶解,另外还由于衔接蛋白释放到细胞外而传播至周围癌细胞,进行以下的实验。
实验中使用的细胞系是SK-OV-3细胞系。作为卵巢癌细胞系,SK-OV-3细胞系是使用含有100U/ml青霉素/100μg/ml链霉素(Welgene)和10%FBS(胎牛血清,Welgene)的DMEM(达尔伯克改良伊格尔培养基)(Welgene)来培养的。
然后,将1×104SK-OV-3细胞系稀释并以0.01MOI感染KOS-Her2scFv-HveA-EmGFP-gD-R222N/F223I病毒。病毒稀释和感染的原因在于观察到病毒感染传播至周围癌细胞。使用荧光显微镜观察病毒感染和传播。
结果在图13底部示出。如图13底部的结果所示,在感染24小时后,观察到一组感染上KOS-Her2scFv-HveA-EmGFP-gD-R222N/F223I病毒。然后,在过了48小时和72小时后,在第一感染组中观察到病毒感染迅速传播至周围癌细胞。
基于以上结果,按照预期,通过KOS-Her2scFv-HveA-EmGFP-gD-R222N/F223I病毒的细胞内感染,插入的衔接蛋白被释放到细胞外并与周围癌细胞表面上的抗原结合,并证实了由于细胞溶解而释放的病毒靶向与癌细胞结合的衔接蛋白或使用与病毒结合的衔接蛋白将感染传播到表达靶分子的周围癌细胞的模式。
尽管已经出于说明的目的公开了本发明的优选实施例,但本领域的技术人员将理解,在不脱离如随附权利要求书中公开的本发明的范围和精神下可以进行多种修改、添加和替代。
SEQUENCE LISTING
<110> 吉赛尔美德公司
韩国原子力医学院
<120> 具有能够表达由癌细胞靶向区与HVEM胞外结构域形成的融合蛋白的表达盒的重组单纯疱疹病毒和其用途
<130> PF-B2725
<140> PCT/KR2020/011224
<141> 2020-08-24
<150> KR 10-2019-0103317
<151> 2019-08-22
<150> KR 10-2020-0072978
<151> 2020-06-16
<160> 33
<170> PatentIn version 3.5
<210> 1
<211> 120
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> HER2 VH
<400> 1
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asn Ile Lys Asp Thr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Arg Ile Tyr Pro Thr Asn Gly Tyr Thr Arg Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ser Arg Trp Gly Gly Asp Gly Phe Tyr Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 2
<211> 107
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> HER2 VL
<400> 2
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Val Asn Thr Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln His Tyr Thr Thr Pro Pro
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 3
<211> 107
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> CEA VH
<400> 3
Asp Ile Gln Met Thr Gln Ser Pro Ala Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Thr Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Gln Gly Lys Ser Pro Gln Leu Leu Val
35 40 45
Tyr Asn Ala Lys Ala Leu Ser Glu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Gln Phe Ser Leu Arg Ile Asn Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Gly Asp Tyr Tyr Cys Gln His His Tyr Asn Ser Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 4
<211> 107
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> CEA VL
<400> 4
Asp Ile Gln Met Thr Gln Ser Pro Ala Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Thr Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Gln Gly Lys Ser Pro Gln Leu Leu Val
35 40 45
Tyr Asn Ala Lys Ala Leu Ser Glu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Gln Phe Ser Leu Arg Ile Asn Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Gly Asp Tyr Tyr Cys Gln His His Tyr Asn Ser Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 5
<211> 15
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> HER2 Linker
<400> 5
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10 15
<210> 6
<211> 14
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> CEA Linker
<400> 6
Gly Ser Thr Ser Gly Ser Gly Lys Ser Ser Glu Gly Lys Gly
1 5 10
<210> 7
<211> 82
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> HveA 82
<400> 7
Leu Pro Ser Cys Lys Glu Asp Glu Tyr Pro Val Gly Ser Glu Cys Cys
1 5 10 15
Pro Lys Cys Ser Pro Gly Tyr Arg Val Lys Glu Ala Cys Gly Glu Leu
20 25 30
Thr Gly Thr Val Cys Glu Pro Cys Pro Pro Gly Thr Tyr Ile Ala His
35 40 45
Leu Asn Gly Leu Ser Lys Cys Leu Gln Cys Gln Met Cys Asp Pro Ala
50 55 60
Met Gly Leu Arg Ala Ser Arg Asn Cys Ser Arg Thr Glu Asn Ala Val
65 70 75 80
Cys Gly
<210> 8
<211> 120
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> Sp.-HveA 82
<400> 8
Met Glu Pro Pro Gly Asp Trp Gly Pro Pro Pro Trp Arg Ser Thr Pro
1 5 10 15
Arg Thr Asp Val Leu Arg Leu Val Leu Tyr Leu Thr Phe Leu Gly Ala
20 25 30
Pro Cys Tyr Ala Pro Ala Leu Pro Ser Cys Lys Glu Asp Glu Tyr Pro
35 40 45
Val Gly Ser Glu Cys Cys Pro Lys Cys Ser Pro Gly Tyr Arg Val Lys
50 55 60
Glu Ala Cys Gly Glu Leu Thr Gly Thr Val Cys Glu Pro Cys Pro Pro
65 70 75 80
Gly Thr Tyr Ile Ala His Leu Asn Gly Leu Ser Lys Cys Leu Gln Cys
85 90 95
Gln Met Cys Asp Pro Ala Met Gly Leu Arg Ala Ser Arg Asn Cys Ser
100 105 110
Arg Thr Glu Asn Ala Val Cys Gly
115 120
<210> 9
<211> 87
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> HveA87
<400> 9
Leu Pro Ser Cys Lys Glu Asp Glu Tyr Pro Val Gly Ser Glu Cys Cys
1 5 10 15
Pro Lys Cys Ser Pro Gly Tyr Arg Val Lys Glu Ala Cys Gly Glu Leu
20 25 30
Thr Gly Thr Val Cys Glu Pro Cys Pro Pro Gly Thr Tyr Ile Ala His
35 40 45
Leu Asn Gly Leu Ser Lys Cys Leu Gln Cys Gln Met Cys Asp Pro Ala
50 55 60
Met Gly Leu Arg Ala Ser Arg Asn Cys Ser Arg Thr Glu Asn Ala Val
65 70 75 80
Cys Gly Cys Ser Pro Gly His
85
<210> 10
<211> 125
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> Sp-HveA87
<400> 10
Met Glu Pro Pro Gly Asp Trp Gly Pro Pro Pro Trp Arg Ser Thr Pro
1 5 10 15
Arg Thr Asp Val Leu Arg Leu Val Leu Tyr Leu Thr Phe Leu Gly Ala
20 25 30
Pro Cys Tyr Ala Pro Ala Leu Pro Ser Cys Lys Glu Asp Glu Tyr Pro
35 40 45
Val Gly Ser Glu Cys Cys Pro Lys Cys Ser Pro Gly Tyr Arg Val Lys
50 55 60
Glu Ala Cys Gly Glu Leu Thr Gly Thr Val Cys Glu Pro Cys Pro Pro
65 70 75 80
Gly Thr Tyr Ile Ala His Leu Asn Gly Leu Ser Lys Cys Leu Gln Cys
85 90 95
Gln Met Cys Asp Pro Ala Met Gly Leu Arg Ala Ser Arg Asn Cys Ser
100 105 110
Arg Thr Glu Asn Ala Val Cys Gly Cys Ser Pro Gly His
115 120 125
<210> 11
<211> 102
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> HveA102
<400> 11
Leu Pro Ser Cys Lys Glu Asp Glu Tyr Pro Val Gly Ser Glu Cys Cys
1 5 10 15
Pro Lys Cys Ser Pro Gly Tyr Arg Val Lys Glu Ala Cys Gly Glu Leu
20 25 30
Thr Gly Thr Val Cys Glu Pro Cys Pro Pro Gly Thr Tyr Ile Ala His
35 40 45
Leu Asn Gly Leu Ser Lys Cys Leu Gln Cys Gln Met Cys Asp Pro Ala
50 55 60
Met Gly Leu Arg Ala Ser Arg Asn Cys Ser Arg Thr Glu Asn Ala Val
65 70 75 80
Cys Gly Cys Ser Pro Gly His Phe Cys Ile Val Gln Asp Gly Asp His
85 90 95
Cys Ala Ala Cys Arg Ala
100
<210> 12
<211> 140
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> Sp-HveA102
<400> 12
Met Glu Pro Pro Gly Asp Trp Gly Pro Pro Pro Trp Arg Ser Thr Pro
1 5 10 15
Arg Thr Asp Val Leu Arg Leu Val Leu Tyr Leu Thr Phe Leu Gly Ala
20 25 30
Pro Cys Tyr Ala Pro Ala Leu Pro Ser Cys Lys Glu Asp Glu Tyr Pro
35 40 45
Val Gly Ser Glu Cys Cys Pro Lys Cys Ser Pro Gly Tyr Arg Val Lys
50 55 60
Glu Ala Cys Gly Glu Leu Thr Gly Thr Val Cys Glu Pro Cys Pro Pro
65 70 75 80
Gly Thr Tyr Ile Ala His Leu Asn Gly Leu Ser Lys Cys Leu Gln Cys
85 90 95
Gln Met Cys Asp Pro Ala Met Gly Leu Arg Ala Ser Arg Asn Cys Ser
100 105 110
Arg Thr Glu Asn Ala Val Cys Gly Cys Ser Pro Gly His Phe Cys Ile
115 120 125
Val Gln Asp Gly Asp His Cys Ala Ala Cys Arg Ala
130 135 140
<210> 13
<211> 107
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> HveA107
<400> 13
Leu Pro Ser Cys Lys Glu Asp Glu Tyr Pro Val Gly Ser Glu Cys Cys
1 5 10 15
Pro Lys Cys Ser Pro Gly Tyr Arg Val Lys Glu Ala Cys Gly Glu Leu
20 25 30
Thr Gly Thr Val Cys Glu Pro Cys Pro Pro Gly Thr Tyr Ile Ala His
35 40 45
Leu Asn Gly Leu Ser Lys Cys Leu Gln Cys Gln Met Cys Asp Pro Ala
50 55 60
Met Gly Leu Arg Ala Ser Arg Asn Cys Ser Arg Thr Glu Asn Ala Val
65 70 75 80
Cys Gly Cys Ser Pro Gly His Phe Cys Ile Val Gln Asp Gly Asp His
85 90 95
Cys Ala Ala Cys Arg Ala Tyr Ala Thr Ser Ser
100 105
<210> 14
<211> 145
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> Sp-HveA107
<400> 14
Met Glu Pro Pro Gly Asp Trp Gly Pro Pro Pro Trp Arg Ser Thr Pro
1 5 10 15
Arg Thr Asp Val Leu Arg Leu Val Leu Tyr Leu Thr Phe Leu Gly Ala
20 25 30
Pro Cys Tyr Ala Pro Ala Leu Pro Ser Cys Lys Glu Asp Glu Tyr Pro
35 40 45
Val Gly Ser Glu Cys Cys Pro Lys Cys Ser Pro Gly Tyr Arg Val Lys
50 55 60
Glu Ala Cys Gly Glu Leu Thr Gly Thr Val Cys Glu Pro Cys Pro Pro
65 70 75 80
Gly Thr Tyr Ile Ala His Leu Asn Gly Leu Ser Lys Cys Leu Gln Cys
85 90 95
Gln Met Cys Asp Pro Ala Met Gly Leu Arg Ala Ser Arg Asn Cys Ser
100 105 110
Arg Thr Glu Asn Ala Val Cys Gly Cys Ser Pro Gly His Phe Cys Ile
115 120 125
Val Gln Asp Gly Asp His Cys Ala Ala Cys Arg Ala Tyr Ala Thr Ser
130 135 140
Ser
145
<210> 15
<211> 369
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> gD ASM47818
<400> 15
Lys Tyr Ala Leu Ala Asp Ala Ser Leu Lys Met Ala Asp Pro Asn Arg
1 5 10 15
Phe Arg Gly Lys Asp Leu Pro Val Leu Asp Gln Leu Thr Asp Pro Pro
20 25 30
Gly Val Arg Arg Val Tyr His Ile Gln Ala Gly Leu Pro Asp Pro Phe
35 40 45
Gln Pro Pro Ser Leu Pro Ile Thr Val Tyr Tyr Ala Val Leu Glu Arg
50 55 60
Ala Cys Arg Ser Val Leu Leu Asn Ala Pro Ser Glu Ala Pro Gln Ile
65 70 75 80
Val Arg Gly Ala Ser Glu Asp Val Arg Lys Gln Pro Tyr Asn Leu Thr
85 90 95
Ile Ala Trp Phe Arg Met Gly Gly Asn Cys Ala Ile Pro Ile Thr Val
100 105 110
Met Glu Tyr Thr Glu Cys Ser Tyr Asn Lys Ser Leu Gly Ala Cys Pro
115 120 125
Ile Arg Thr Gln Pro Arg Trp Asn Tyr Tyr Asp Ser Phe Ser Ala Val
130 135 140
Ser Glu Asp Asn Leu Gly Phe Leu Met His Ala Pro Ala Phe Glu Thr
145 150 155 160
Ala Gly Thr Tyr Leu Arg Leu Val Lys Ile Asn Asp Trp Thr Glu Ile
165 170 175
Thr Gln Phe Ile Leu Glu His Arg Ala Lys Gly Ser Cys Lys Tyr Ala
180 185 190
Leu Pro Leu Arg Ile Pro Pro Ser Ala Cys Leu Ser Pro Gln Ala Tyr
195 200 205
Gln Gln Gly Val Thr Val Asp Ser Ile Gly Met Leu Pro Arg Phe Ile
210 215 220
Pro Glu Asn Gln Arg Thr Val Ala Val Tyr Ser Leu Lys Ile Ala Gly
225 230 235 240
Trp His Gly Pro Lys Ala Pro Tyr Thr Ser Thr Leu Leu Pro Pro Glu
245 250 255
Leu Ser Glu Thr Pro Asn Ala Thr Gln Pro Glu Leu Ala Pro Glu Asp
260 265 270
Pro Glu Asp Ser Ala Leu Leu Glu Asp Pro Val Gly Thr Val Ala Pro
275 280 285
Gln Ile Pro Pro Asn Trp His Ile Pro Ser Ile Gln Asp Ala Ala Thr
290 295 300
Pro Tyr His Pro Pro Ala Thr Pro Asn Asn Met Gly Leu Ile Ala Gly
305 310 315 320
Ala Val Gly Gly Ser Leu Leu Ala Ala Leu Val Ile Cys Gly Ile Val
325 330 335
Tyr Trp Met His Arg Arg Thr Arg Lys Ala Pro Lys Arg Ile Arg Leu
340 345 350
Pro His Ile Arg Glu Asp Asp Gln Pro Ser Ser His Gln Pro Leu Phe
355 360 365
Tyr
<210> 16
<211> 60
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> gD-rpsL For
<400> 16
cccaggccta ccagcagggg gtgacggtgg acagcatcgg gatgctgccc ggcctggtga 60
<210> 17
<211> 88
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> gD-rpsL Rev
<400> 17
ccggcgatct tcaagctgta tacggcgacg gtgcgctggt tctcggggat tcagaagaac 60
tcgtcaagaa ggcgtgatgg cgggatcg 88
<210> 18
<211> 369
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> gD R222N_F223I
<400> 18
Lys Tyr Ala Leu Ala Asp Ala Ser Leu Lys Met Ala Asp Pro Asn Arg
1 5 10 15
Phe Arg Gly Lys Asp Leu Pro Val Leu Asp Gln Leu Thr Asp Pro Pro
20 25 30
Gly Val Arg Arg Val Tyr His Ile Gln Ala Gly Leu Pro Asp Pro Phe
35 40 45
Gln Pro Pro Ser Leu Pro Ile Thr Val Tyr Tyr Ala Val Leu Glu Arg
50 55 60
Ala Cys Arg Ser Val Leu Leu Asn Ala Pro Ser Glu Ala Pro Gln Ile
65 70 75 80
Val Arg Gly Ala Ser Glu Asp Val Arg Lys Gln Pro Tyr Asn Leu Thr
85 90 95
Ile Ala Trp Phe Arg Met Gly Gly Asn Cys Ala Ile Pro Ile Thr Val
100 105 110
Met Glu Tyr Thr Glu Cys Ser Tyr Asn Lys Ser Leu Gly Ala Cys Pro
115 120 125
Ile Arg Thr Gln Pro Arg Trp Asn Tyr Tyr Asp Ser Phe Ser Ala Val
130 135 140
Ser Glu Asp Asn Leu Gly Phe Leu Met His Ala Pro Ala Phe Glu Thr
145 150 155 160
Ala Gly Thr Tyr Leu Arg Leu Val Lys Ile Asn Asp Trp Thr Glu Ile
165 170 175
Thr Gln Phe Ile Leu Glu His Arg Ala Lys Gly Ser Cys Lys Tyr Ala
180 185 190
Leu Pro Leu Arg Ile Pro Pro Ser Ala Cys Leu Ser Pro Gln Ala Tyr
195 200 205
Gln Gln Gly Val Thr Val Asp Ser Ile Gly Met Leu Pro Asn Ile Ile
210 215 220
Pro Glu Asn Gln Arg Thr Val Ala Val Tyr Ser Leu Lys Ile Ala Gly
225 230 235 240
Trp His Gly Pro Lys Ala Pro Tyr Thr Ser Thr Leu Leu Pro Pro Glu
245 250 255
Leu Ser Glu Thr Pro Asn Ala Thr Gln Pro Glu Leu Ala Pro Glu Asp
260 265 270
Pro Glu Asp Ser Ala Leu Leu Glu Asp Pro Val Gly Thr Val Ala Pro
275 280 285
Gln Ile Pro Pro Asn Trp His Ile Pro Ser Ile Gln Asp Ala Ala Thr
290 295 300
Pro Tyr His Pro Pro Ala Thr Pro Asn Asn Met Gly Leu Ile Ala Gly
305 310 315 320
Ala Val Gly Gly Ser Leu Leu Ala Ala Leu Val Ile Cys Gly Ile Val
325 330 335
Tyr Trp Met His Arg Arg Thr Arg Lys Ala Pro Lys Arg Ile Arg Leu
340 345 350
Pro His Ile Arg Glu Asp Asp Gln Pro Ser Ser His Gln Pro Leu Phe
355 360 365
Tyr
<210> 19
<211> 74
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> UL26/27-rpsL_For
<400> 19
gcgtgggggg gaggaaatcg gcactgacca agggggtccg ttttgtcacg tcagaagaac 60
tcgtcaagaa ggcg 74
<210> 20
<211> 74
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> UL26/27-rpsL_Rev
<400> 20
aacacataaa ctcccccggg tgtccgcggc ctgtttcctc tttcctttcc ggcctggtga 60
tgatggcggg atcg 74
<210> 21
<211> 74
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> UL26-tkpA_For
<400> 21
gcgtgggggg gaggaaatcg gcactgacca agggggtccg ttttgtcacg gcctcagaag 60
ccatagagcc cacc 74
<210> 22
<211> 74
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> UL27-pCMV_Rev
<400> 22
aacacataaa ctcccccggg tgtccgcggc ctgtttcctc tttcctttcc tatacgcgtt 60
gacattgatt attg 74
<210> 23
<211> 351
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> HER2scFv-HveA adaptor
<400> 23
Met Ser Val Pro Thr Gln Val Leu Gly Leu Leu Leu Leu Trp Leu Thr
1 5 10 15
Gly Ala Arg Cys Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asn
35 40 45
Ile Lys Asp Thr Tyr Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Arg Ile Tyr Pro Thr Asn Gly Tyr Thr Arg Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys
85 90 95
Asn Thr Ala Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr Tyr Cys Ser Arg Trp Gly Gly Asp Gly Phe Tyr Ala Met Asp
115 120 125
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr
145 150 155 160
Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile
165 170 175
Thr Cys Arg Ala Ser Gln Asp Val Asn Thr Ala Val Ala Trp Tyr Gln
180 185 190
Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ser Ala Ser Phe
195 200 205
Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Arg Ser Gly Thr
210 215 220
Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr
225 230 235 240
Tyr Tyr Cys Gln Gln His Tyr Thr Thr Pro Pro Thr Phe Gly Gln Gly
245 250 255
Thr Lys Val Glu Ile Lys Gly Gly Gly Gly Ser Glu Phe Leu Pro Ser
260 265 270
Cys Lys Glu Asp Glu Tyr Pro Val Gly Ser Glu Cys Cys Pro Lys Cys
275 280 285
Ser Pro Gly Tyr Arg Val Lys Glu Ala Cys Gly Glu Leu Thr Gly Thr
290 295 300
Val Cys Glu Pro Cys Pro Pro Gly Thr Tyr Ile Ala His Leu Asn Gly
305 310 315 320
Leu Ser Lys Cys Leu Gln Cys Gln Met Cys Asp Pro Ala Met Gly Leu
325 330 335
Arg Ala Ser Arg Asn Cys Ser Arg Thr Glu Asn Ala Val Cys Gly
340 345 350
<210> 24
<211> 1053
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HER2scFv-HveA adaptor
<400> 24
atgagtgtgc ccactcaggt cctggggttg ctgctgctgt ggcttacagg tgccagatgt 60
gaggtgcagc tggttgaatc tggcggagga ctggttcagc ctggcggatc tctgagactg 120
tcttgtgccg ccagcggctt caacatcaag gacacctaca tccactgggt ccgacaggcc 180
cctggcaaag gacttgaatg ggtcgccaga atctacccca ccaacggcta caccagatac 240
gccgactctg tgaagggcag attcaccatc agcgccgaca ccagcaagaa caccgcctac 300
ctgcagatga acagcctgag agccgaggac accgccgtgt actactgttc tagatgggga 360
ggcgacggct tctacgccat ggattattgg ggccagggca ccctggtcac agtttctagc 420
ggaggcggag gttctggcgg cggaggaagt ggtggcggag gctctgatat ccagatgaca 480
cagagcccca gcagcctgtc tgcctctgtg ggagacagag tgaccatcac ctgtagagcc 540
agccaggacg tgaacacagc cgtggcttgg tatcagcaga agcctggcaa ggcccctaag 600
ctgctgatct acagcgccag ctttctgtac agcggcgtgc ccagcagatt cagcggctct 660
agaagcggca ccgacttcac cctgaccata agcagtctgc agcccgagga cttcgccacc 720
tactactgtc agcagcacta caccacacct ccaaccttcg gacagggcac caaggtggaa 780
atcaagggtg gtggcggttc agaattcctg ccgtcctgca aggaggacga gtacccagtg 840
ggctccgagt gctgccccaa gtgcagtcca ggttatcgtg tgaaggaggc ctgcggggag 900
ctgacgggca cagtgtgtga accctgccct ccaggcacct acattgccca cctcaatggc 960
ctaagcaagt gtctgcagtg ccaaatgtgt gacccagcca tgggcctgcg cgcgagccgg 1020
aactgctcca ggacagagaa cgccgtgtgt ggc 1053
<210> 25
<211> 369
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> HveA-HER2scFv adaptor
<400> 25
Met Glu Pro Pro Gly Asp Trp Gly Pro Pro Pro Trp Arg Ser Thr Pro
1 5 10 15
Arg Thr Asp Val Leu Arg Leu Val Leu Tyr Leu Thr Phe Leu Gly Ala
20 25 30
Pro Cys Tyr Ala Pro Ala Leu Pro Ser Cys Lys Glu Asp Glu Tyr Pro
35 40 45
Val Gly Ser Glu Cys Cys Pro Lys Cys Ser Pro Gly Tyr Arg Val Lys
50 55 60
Glu Ala Cys Gly Glu Leu Thr Gly Thr Val Cys Glu Pro Cys Pro Pro
65 70 75 80
Gly Thr Tyr Ile Ala His Leu Asn Gly Leu Ser Lys Cys Leu Gln Cys
85 90 95
Gln Met Cys Asp Pro Ala Met Gly Leu Arg Ala Ser Arg Asn Cys Ser
100 105 110
Arg Thr Glu Asn Ala Val Cys Gly Gly Gly Gly Gly Ser Gly Ser Glu
115 120 125
Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
130 135 140
Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asn Ile Lys Asp Thr Tyr
145 150 155 160
Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala
165 170 175
Arg Ile Tyr Pro Thr Asn Gly Tyr Thr Arg Tyr Ala Asp Ser Val Lys
180 185 190
Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr Leu
195 200 205
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ser
210 215 220
Arg Trp Gly Gly Asp Gly Phe Tyr Ala Met Asp Tyr Trp Gly Gln Gly
225 230 235 240
Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
245 250 255
Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
260 265 270
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
275 280 285
Gln Asp Val Asn Thr Ala Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys
290 295 300
Ala Pro Lys Leu Leu Ile Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val
305 310 315 320
Pro Ser Arg Phe Ser Gly Ser Arg Ser Gly Thr Asp Phe Thr Leu Thr
325 330 335
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
340 345 350
His Tyr Thr Thr Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
355 360 365
Lys
<210> 26
<211> 1107
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HveA-HER2scFv adaptor
<400> 26
atggagcctc ctggagactg ggggcctcct ccctggagat ccacccccag aaccgacgtc 60
ttgaggctgg tgctgtatct caccttcctg ggagccccct gctacgcccc agctctgccg 120
tcctgcaagg aggacgagta cccagtgggc tccgagtgct gccccaagtg cagtccaggt 180
tatcgtgtga aggaggcctg cggggagctg acgggcacag tgtgtgaacc ctgccctcca 240
ggcacctaca ttgcccacct caatggccta agcaagtgtc tgcagtgcca aatgtgtgac 300
ccagccatgg gcctgcgcgc gagccggaac tgctccagga cagagaacgc cgtgtgtggc 360
ggtggtggcg gttcaggatc cgaggtgcag ctggttgaat ctggcggagg actggttcag 420
cctggcggat ctctgagact gtcttgtgcc gccagcggct tcaacatcaa ggacacctac 480
atccactggg tccgacaggc ccctggcaaa ggacttgaat gggtcgccag aatctacccc 540
accaacggct acaccagata cgccgactct gtgaagggca gattcaccat cagcgccgac 600
accagcaaga acaccgccta cctgcagatg aacagcctga gagccgagga caccgccgtg 660
tactactgtt ctagatgggg aggcgacggc ttctacgcca tggattattg gggccagggc 720
accctggtca cagtttctag cggaggcgga ggttctggcg gcggaggaag tggtggcgga 780
ggctctgata tccagatgac acagagcccc agcagcctgt ctgcctctgt gggagacaga 840
gtgaccatca cctgtagagc cagccaggac gtgaacacag ccgtggcttg gtatcagcag 900
aagcctggca aggcccctaa gctgctgatc tacagcgcca gctttctgta cagcggcgtg 960
cccagcagat tcagcggctc tagaagcggc accgacttca ccctgaccat aagcagtctg 1020
cagcccgagg acttcgccac ctactactgt cagcagcact acaccacacc tccaaccttc 1080
ggacagggca ccaaggtgga aatcaag 1107
<210> 27
<211> 346
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> CEAscFv-HveA
<400> 27
Met Ser Val Pro Thr Gln Val Leu Gly Leu Leu Leu Leu Trp Leu Thr
1 5 10 15
Gly Ala Arg Cys Asp Ile Gln Met Thr Gln Ser Pro Ala Ser Leu Ser
20 25 30
Ala Ser Val Gly Asp Thr Val Thr Ile Thr Cys Arg Ala Ser Glu Asn
35 40 45
Ile Tyr Ser Tyr Leu Ala Trp Tyr Gln Gln Lys Gln Gly Lys Ser Pro
50 55 60
Gln Leu Leu Val Tyr Asn Ala Lys Ala Leu Ser Glu Gly Val Pro Ser
65 70 75 80
Arg Phe Ser Gly Ser Gly Ser Gly Thr Gln Phe Ser Leu Arg Ile Asn
85 90 95
Ser Leu Gln Pro Glu Asp Phe Gly Asp Tyr Tyr Cys Gln His His Tyr
100 105 110
Asn Ser Pro Tyr Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Gly
115 120 125
Ser Thr Ser Gly Ser Gly Lys Ser Ser Glu Gly Lys Gly Gln Ile Gln
130 135 140
Leu Val Gln Ser Gly Pro Glu Leu Lys Lys Pro Gly Glu Thr Val Lys
145 150 155 160
Ile Ser Cys Lys Ala Ser Gly Tyr Ser Phe Thr Asn Asp Gly Ile Asn
165 170 175
Trp Val Lys Gln Ala Pro Gly Lys Gly Phe Lys Tyr Met Gly Trp Ile
180 185 190
Asn Thr Ile Thr Gly Glu Pro Thr Tyr Thr Glu Asp Phe Lys Gly Arg
195 200 205
Phe Ala Phe Ser Leu Glu Thr Ser Ala Ser Thr Ala Tyr Leu Gln Ile
210 215 220
Asn Asn Leu Lys Asp Glu Asp Thr Ala Thr Phe Phe Cys Ala Lys Gly
225 230 235 240
Thr Gly Thr Ser Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser
245 250 255
Ala Gly Gly Gly Gly Ser Glu Phe Leu Pro Ser Cys Lys Glu Asp Glu
260 265 270
Tyr Pro Val Gly Ser Glu Cys Cys Pro Lys Cys Ser Pro Gly Tyr Arg
275 280 285
Val Lys Glu Ala Cys Gly Glu Leu Thr Gly Thr Val Cys Glu Pro Cys
290 295 300
Pro Pro Gly Thr Tyr Ile Ala His Leu Asn Gly Leu Ser Lys Cys Leu
305 310 315 320
Gln Cys Gln Met Cys Asp Pro Ala Met Gly Leu Arg Ala Ser Arg Asn
325 330 335
Cys Ser Arg Thr Glu Asn Ala Val Cys Gly
340 345
<210> 28
<211> 1038
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> CEAscFv-HveA adaptor
<400> 28
atgagtgtgc ccactcaggt cctggggttg ctgctgctgt ggcttacagg tgccagatgt 60
gacatccaga tgactcagtc tccagcctcc ctttctgcat ctgtgggaga cactgtcacc 120
atcacatgtc gagcaagtga gaacatttat agttatttag catggtatca gcagaaacag 180
ggaaaatctc ctcagctcct ggtctataat gcaaaggcct tatcagaagg tgtgccgtca 240
aggttcagtg gcagtggatc aggcacacag ttttctctga ggatcaacag cctgcagcct 300
gaagattttg gggattatta ctgtcaacat cattataatt ctccttatac gttcggaggg 360
gggaccaaac tggaaataaa gggctccacc tccgggtctg gtaaatcttc cgagggcaag 420
ggccagatcc agttggtgca gtctggacct gagctgaaga agcctggaga gacagtcaag 480
atctcctgca aggcttctgg ttattccttc acaaacgatg gaataaactg ggtgaagcag 540
gctccaggaa agggttttaa gtacatgggc tggataaaca ccatcactgg agagccaaca 600
tatactgaag acttcaaggg gcggtttgcc ttctctttgg aaacctctgc cagcactgcc 660
tatttgcaga tcaacaacct caaagatgag gacacggcta catttttctg tgcaaagggg 720
actgggacga gcgcttactg gggccaaggg actctggtca ctgtctctgc tggtggtggc 780
ggttcagaat tcctgccgtc ctgcaaggag gacgagtacc cagtgggctc cgagtgctgc 840
cccaagtgca gtccaggtta tcgtgtgaag gaggcctgcg gggagctgac gggcacagtg 900
tgtgaaccct gccctccagg cacctacatt gcccacctca atggcctaag caagtgtctg 960
cagtgccaaa tgtgtgaccc agccatgggc ctgcgcgcga gccggaactg ctccaggaca 1020
gagaacgccg tgtgtggc 1038
<210> 29
<211> 79
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HSV-1_UL3/4-rpsL-neo_for
<400> 29
taaataacac ataaatttgg ctggttgttt gttgtcttta atggaccgcc cgcaaggcct 60
ggtgatgatg gcgggatcg 79
<210> 30
<211> 78
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HSV-1_UL3/4-rpsL-neo_rev
<400> 30
taggatcccg gccggatcgc gctcgtcacc cgacactgaa acgccccccc cccctcagaa 60
gaactcgtca agaaggcg 78
<210> 31
<211> 79
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HSV-1_UL3/4-HM_pCMV_For
<400> 31
taaataacac ataaatttgg ctggttgttt gttgtcttta atggaccgcc cgcaatatac 60
gcgttgacat tgattattg 79
<210> 32
<211> 78
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> UL3/4_bGH_poly_Rev
<400> 32
taggatcccg gccggatcgc gctcgtcacc cgacactgaa acgccccccc ccccgcctca 60
gaagccatag agcccacc 78
<210> 33
<211> 20
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 信号序列HER2(Signal seq. HER2)
<400> 33
Met Ser Val Pro Thr Gln Val Leu Gly Leu Leu Leu Leu Trp Leu Thr
1 5 10 15
Gly Ala Arg Cys
20
本文用于企业家、创业者技术爱好者查询,结果仅供参考。