脱氨酶介导的dna中n
4-甲基胞嘧啶的单碱基分辨率定位分析方法
技术领域
1.本发明涉及生物技术领域,具体涉及一种脱氨酶介导的dna中n
4-甲基胞嘧啶的单碱基分辨率定位分析方法。
背景技术:
2.n
4-甲基胞嘧啶(4mc)是一种dna甲基化修饰,主要存在于极度嗜热菌和部分嗜常温菌dna中,在真核生物特别是哺乳动物中目前还没有报道。迄今为止,已经发现了多种细菌dna的4mc甲基转移酶,这些甲基转移酶能够与限制性内切酶结合形成序列特异性限制
‑ꢀ
修饰系统。宿主dna中的4mc碱基在识别特定基序的限制性内切酶和保护宿主基因组方面起着非常重要的作用。尽管4mc能够通过液相色谱、液相色谱串联质谱(lc-ms)等技术高灵敏地被检测和定量,但它们在基因组dna中的定位信息一直是一个挑战。建立4mc的测序技术有助于对其生物学功能进行更加深入的研究。
3.目前对于dna中4mc的单碱基分辨率测序方法有两类,一种是基于传统亚硫酸氢盐测序的分析方法,另一种为单分子实时测序(smrt)方法。亚硫酸氢盐测序是一种广泛使用的dna中5-甲基胞嘧啶(5mc)单碱基分辨率测序方法,由于5mc对亚硫酸氢盐转化具有严格的抗性,而且5mc广泛分布于各物种基因组中,因此标准亚硫酸氢盐测序并不适用于区分5mc和4mc。基于此,有人开发了一种4mc-tet辅助的亚硫酸氢盐测序(4mc-tab-seq) 方法。在4mc-tab-seq方法中,5mc被tet蛋白氧化为5-羧基胞嘧啶,后者经亚硫酸氢盐处理后脱氨,并在测序中读作胸腺嘧啶。在4mc-tab-seq方法中,胞嘧啶和5mc都被测序读为胸腺嘧啶,而只有4mc位点被解读为胞嘧啶,因此,实现了对整个基因组中4mc的特异性检测。然而,基于亚硫酸氢盐的策略需要苛刻的化学脱氨条件,从而导致高达99.9%的dna 被降解。与二代测序方法不同的是,smrt技术能够以单核苷酸分辨率直接检测修饰碱基,并已被用于dna中4mc的检测。然而,与常用的二代测序技术相比,smrt测序成本更高, dna用量更大,测序错误率相对较高。
4.人载脂蛋白b mrna编辑催化亚基3a(a3a)家族酶的成员能够在单链dna中将胞嘧啶转化为尿嘧啶,并在病毒感染和癌症发生中具有非常关键的功能。研究表明,a3a对dna 中的胞嘧啶和5mc具有高效的脱氨活性,且两种胞嘧啶衍生物5-羟甲基胞嘧啶(5hmc)和 5-醛基胞嘧啶(5fc)都可以被高效脱氨。a3a的脱氨活性不依赖于序列特异性,因此能够对全基因组上所有胞嘧啶进行脱氨基。在此基础上,有研究开发了用于5hmc测序的a3a辅助的定位分析方法,即ace-seq和amd-seq。目前,还没有不依赖于亚硫酸氢盐处理、高选择性、操作简单的4mc定位分析方法。
技术实现要素:
5.本发明的目的在于提供一种胞嘧啶脱氨酶介导的dna中n
4-甲基胞嘧啶(4mc)单碱基分辨率定位分析方法,即一种不依赖于亚硫酸氢盐处理、高选择性、操作简单的4mc定位
分析方法。
6.本发明的目的通过下述技术方案实现:
7.一种脱氨酶介导的dna中n
4-甲基胞嘧啶的单碱基分辨率定位分析方法,包括如下步骤:
8.(1)对待检dna进行变性处理。
9.(2)经变性处理的dna用胞嘧啶脱氨酶进行脱氨处理。
10.(3)脱氨后的dna样品进行pcr扩增。
11.(4)将pcr扩增产物进行测序,待检dna中的n
4-甲基胞嘧啶在测序中被读为胞嘧啶,正常的胞嘧啶和5-甲基胞嘧啶在测序中被读为胸腺嘧啶。
12.步骤(1)中,dna变性处理的方法优选为:在0~60%dmso存在下,将dna在90~ 98℃高温下孵育2~30分钟后转入冰水浴中。
13.步骤(2)中,所述的胞嘧啶脱氨酶优选为氨基酸序列如seq id no.1所示的a3a蛋白。 dna进行脱氨处理的方法优选为:dna在含2-(n-吗啉)乙磺酸(mes)10~30mm、tritonx-100 0.05~0.5%、ph 5.5~7.5、a3a蛋白1~10μm的反应体系中,于30~45℃反应0.5~ 4小时,反应结束后再在75~98℃变性2~30分钟使a3a蛋白灭活。
14.步骤(4)中,若pcr扩增得到的是特定序列的dna片段,扩增产物可以直接进行sanger 测序;若pcr扩增形成dna文库,则将扩增产物进行高通量测序。
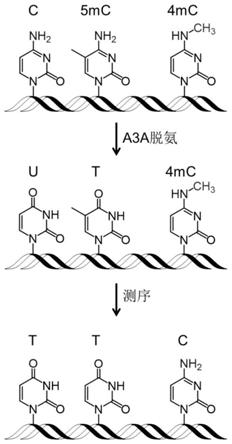
15.本发明方法的原理见图1,胞嘧啶脱氨酶a3a蛋白对dna中正常的胞嘧啶(c)和5
‑ꢀ
甲基胞嘧啶(5mc)进行脱氨基,随后在后续聚合酶链式反应扩增形成胸腺嘧啶(t),而目标分析物n
4-甲基胞嘧啶(4mc)能够抵抗胞嘧啶脱氨酶a3a的脱氨作用,因此在随后的聚合酶链式反应中扩增为胞嘧啶。这样,只有n
4-甲基胞嘧啶(4mc)修饰才能在最终的测序中被读为胞嘧啶(c)。
16.本发明方法通过a3a蛋白对dna中的胞嘧啶和5-mc脱氨基,4mc不脱氨,无需亚硫酸氢盐处理直接测序获得4mc的单碱基分辨率定位信息。
17.本发明的优点及有益效果如下:
18.1.本发明中,方法操作简便,无需复杂的样品前处理。
19.2.本发明无需利用衍生化试剂对分析物进行化学标记,极大地缩短了分析时间。
20.3.本发明无需亚硫酸氢盐处理,反应条件温和,不会造成dna的大量降解。
21.4.本发明dna用量小,仅需100ng的基因组dna即可对4mc进行定位分析。
22.5.本发明涉及的胞嘧啶脱氨酶对胞嘧啶和5mc具有高效的脱氨效率(97%),而对4mc 的脱氨基效率很低(3%),有利于4mc的定位分析。
23.6.本发明无需采用昂贵的第三代测序平台,只需采用下一代测序平台技术即可完成4mc 的单碱基分辨率定位分析。
24.7.本发明涉及的方法能够广泛应用于原核生物dna中4mc修饰位点与相关甲基化酶保守序列的研究,并有助于对4mc的生物学功能进行更加深入的研究。
附图说明
25.图1为本发明示意图。本发明中,4mc能够抵抗a3a的脱氨作用,而c与5mc能够被 a3a转化为u和t。因此,在测序时,只有4mc能够被读成c。
26.图2a为本发明中利用lc-ms/ms检测人工合成的不含修饰的215bp dna链脱氨基处理前后dc含量对比图。
27.图2b为本发明中利用lc-ms/ms检测人工合成的含有4mc修饰的215bp dna链脱氨基处理前后4mdc含量对比图。
28.图2c为本发明中利用lc-ms/ms检测人工合成的含有5mc修饰的215bp dna链脱氨基处理前后5mdc含量对比图。
29.图3为本发明中对含胞嘧啶、4mc或5mc的215bp dna链经a3a蛋白处理后,分别利用液相色谱串联质谱和单克隆测序检测到的胞嘧啶、4mc和5mc的脱氨率。
30.图4a为本发明中人工合成的不含修饰的215bp dna链经a3a蛋白处理前后,sanger 测序的读出结果,其中每个c经a3a脱氨后均突变为t。
31.图4b为本发明中人工合成的含5mc修饰的215bp dna链经a3a蛋白处理前后,sanger 测序的读出结果,其中每个5mc经a3a脱氨后均突变为t。
32.图4c为本发明中人工合成的含4mc修饰的215bp dna链经a3a蛋白处理前后,sanger 测序的读出结果,其中每个4mc经a3a脱氨后均未突变,仍读作c。
33.图5为本发明方法对全基因组4mc甲基化测序文库构建流程示意图。
34.图6为本发明方法对全基因组4mc甲基化测序文库构建过程中使用的接头及引物序列。
35.图7为应用本发明中方法对耐辐射奇异球菌全基因组4mc进行测序,平行样品1、2和 3检测出来的各4mc位点甲基化率的相关性。
具体实施方式
36.在一些实施方案中,本发明脱氨酶介导的dna中n
4-甲基胞嘧啶单碱基分辨率定位方法,包括下述步骤:
37.(1)配制高浓度的a3a反应缓冲液的储存液:200mm 2-(n-吗啉)乙磺酸(mes),1%的triton x-100,ph为5.5-7.5。
38.(2)表达并纯化a3a蛋白。
39.(3)从生物样品中提取dna,用rnasea酶将dna中的rna去除。
40.(4)取100ng提取的dna,超声进行片段化处理,片段化的dna进行末端修复和3’端加da处理,随后进行接头连接。连接后的dna进行纯化。
41.(5)在步骤(4)纯化的dna中加入2~12μl的dmso,加水至16μl,在90~98℃水浴中孵育2~30分钟,随后立即转移至冰水浴中猝火。加入2μl步骤(1)配制的a3a反应缓冲液,加入2μl的a3a蛋白(终浓度1~10μm),30~45℃下反应0.5~4小时。反应结束后,将样品加热至75~98℃变性2~30分钟使蛋白失活。
42.(6)进行第一次聚合酶链式反应,使用与脱氨后的接头互补配对的引物(p5-pre,p7-pre,如图6),对脱氨后的dna进行扩增。纯化扩增后的dna,去除多余的引物。
43.(7)以步骤(6)中纯化后的dna为模板,使用高通量测序平台所用引物(p5-universal 引物和p7-index引物,如图6),进行第二次聚合酶链式反应。扩增产物进行琼脂糖凝胶电泳,切胶纯化,得到dna文库。
44.(8)将纯化后的dna文库进行高通量测序。
45.本发明所使用的胞嘧啶脱氨酶a3a蛋白(ncbi genbank:eaw60278.1)的氨基酸序列如seq id no.1所示。
46.以下实施例用于进一步说明本发明,但不应理解为对本发明的限制,其他的任何未背离本发明的精神实质与原理下所作的改变、修饰、替代、组合、简化,均应为等效的置换方式,都包含在本发明的保护范围之内。
47.若未特别指明,实施例中所用的技术手段(包括核酸的提取、酶解以及聚合酶链式反应等)为本领域技术人员所熟知的常规手段。
48.实施例1.利用人工合成的含不同修饰的215bp dna链区分胞嘧啶、5-甲基胞嘧啶和n4‑ꢀ
甲基胞嘧啶
49.为了验证本发明方法的准确性,首先合成了一段215bp含不同修饰的dna链,与a3a 蛋白反应,通过质谱和sanger测序验证脱氨率。
50.a.215bp dna链的合成方法
51.在50μl的反应体系中,加入0.5ng puc19 dna(takara生物技术股份有限公司),5μl 10
×
反应缓冲液,2.5u taq dna聚合酶(takara),2μl 10μm正向引物(序列为 5
′‑
gagtgagtgagggaggaag-3
′
),2μl 10μm反向引物(序列为 5
′‑
ccactcacaattccacacaacatac-3
′
),1μl datp、dgtp、ttp和dctp(浓度均为 2.5mm)。当合成5mc或4mc修饰的dna链时,以5mdctp或4mdctp取代dctp即可。
52.聚合酶链式反应(pcr)程序为:(1)95℃变性2min;(2)95℃变性1min;(3)52℃退火2min;(4)72℃延伸3min;(2)-(4)步重复30次;72℃延伸7min,放置4℃进行贮存。对pcr产物利用试剂盒进行回收纯化(omega bio-tek inc.,norcross,ga,usa)。利用微量分光光度计对合成的dna浓度进行定量。
53.合成的215bp dna序列如seq id no.2所示。
54.b.人工合成的dna链进行脱氨反应
55.往人工合成的dna链中加入2μl的dmso,加水至16μl,在95℃水浴中孵育10分钟,随后立即转移至冰水浴中猝火。加入2μl步骤(1)配制的a3a反应缓冲液,加入2μl的 a3a蛋白(终浓度6μm),37℃下反应2小时。反应结束后,将样品加热至95℃变性5分钟使蛋白失活。
56.c.液相色谱串联质谱分析c、5mc和4mc修饰的脱氨效率
57.将a3a处理后的dna酶解为核苷,利用液相色谱串联质谱分析a3a处理前后dc、5mdc 和4mdc含量的变化情况。如图2a-2c所示,a3a处理后c和5mc均脱氨,而4mc含量没有显著降低。如图3所示,c和5mc的脱氨效率达到97%,而4mc脱氨率仅为3%。
58.d.sanger测序分析c、5mc和4mc修饰的脱氨效率
59.液相色谱串联质谱技术只能分析c、5mc和4mc修饰的整体水平,而sanger测序能够对单个位点上c、5mc和4mc的脱氨情况进行分析。在对三种人工合成的dna链进行a3a 处理后,利用上述步骤a中的引物进行pcr扩增,扩增产物直接进行sanger测序。图4a-4b 显示所有位点上的c和5mc在a3a处理后均读为t,而图4c表明所有位点上4mc仍读作 c。此外,还通过单克隆测序对不同位点的脱氨率进行了定量分析。如图3所示,单克隆测序结果显示c完全脱氨,5mc仅1.2%未脱氨,而4mc只有3.0%脱氨。
60.除了对4mc修饰位点进行鉴定外,本发明还能够对单个位点上4mc的水平进行定量分析。首先混合不同比例含4mc和c的dna链,随后进行a3a处理和pcr扩增,单克隆测序表明
各个4mc位点上c的读出率与理论值一致,不同含量4mc测量值与理论值线性关系良好(r2=0.999)。
61.以上结果表明,4mc在a3a处理后仍读作c,和c与5mc会被a3a脱氨因而读作t,所以,测序读出的c位点即为4mc修饰位点,基于此,该方法能够应用至生物样品dna中 4mc的定位分析。
62.实施例2.脱氨酶介导的dna中4mc单碱基定位方法用于耐辐射奇异球菌全基因组 4mc位点的鉴定
63.耐辐射奇异球菌是一种特殊的细菌,它对电离辐射、紫外线、氧化和干燥等致命影响具有极强的抵抗能力。有研究表明,4mc在维持耐辐射奇异球菌基因组稳定性方面起着重要作用。目前用于鉴定4mc的方法主要基于亚硫氢盐转化或第三代测序技术,然而,这些技术都存在其各自的缺点。比如,亚硫酸氢盐转化能够对dna造成大量的降解,而三代测序的准确性一直是个问题。因此,利用上述建立的方法,对耐辐射奇异球菌基因组dna中特定位点4mc修饰进行了鉴定。
64.为了探索是否在全基因组水平上4mc的分布及序列保守性,本发明采用建立的4mc单碱基定位方法分析了耐辐射奇异球菌所有两条染色体和两个质粒中4mc的分布情况。对全基因组4mc甲基化测序文库构建流程见图5。具体包括如下步骤:
65.a.提取耐辐射奇异球菌全基因组,超声将dna碎裂成200-400bp片段,利用试剂盒对 dna进行末端修复和3’端加da(翌圣生物科技股份有限公司)。
66.b.随后进行接头连接,采用t4 dna连接酶(yeasen)根据制造商推荐的条件进行连接。连接后的dna通过kapa纯化磁珠(roche)进行纯化。
67.c.接着利用实施例1中的方法对连接产物进行脱氨基与灭活处理。
68.d.脱氨后的dna首先使用p5-pre引物、p7-pre引物和q5u热启动超保真dna聚合酶(neb)进行扩增,配制如下反应:
[0069][0070][0071]
扩增程序为:
[0072][0073]
e.扩增产物通过kapa纯化磁珠(roche)进行纯化。
[0074]
f.进行第二次pcr。配制如下反应体系:
[0075][0076]
扩增程序为:
[0077][0078]
g.pcr产物采用低熔点琼脂糖凝胶电泳分离,切胶纯化450-650bp dna。最后将纯化后的dna于illumina novaseq 6000平台进行高通量测序(novogene co.,ltd.,china)。
[0079]
上述过程中所用接头与引物序列见图6。
[0080]
本发明方法重现性好,三组平行样品测得的含量相关性良好(皮尔逊相关系数大于0.98,如图7),使用本发明脱氨酶介导的dna中4mc单碱基定位方法发现了1586个4mc位点,其中,约35.6%的4mc位点位于其保守序列“ccgcgg”中。其中,4mc修饰于第二个胞嘧啶处。处于“ccgcgg”基序中的4mc位点甲基化程度普遍较高,含量约为50-90%(平均值为70%)。而处于该基序外的4mc位点的甲基化率绝大部分低于50%(平均值为23%)。该结果与文献报道一致(li,s.j.et al.frontier in microbiology 2019,10),虽然文献中利用
单分子实时测序(smrt)技术也鉴定出了耐辐射奇异球菌中4mc的保守基序“ccgcgg”,但是相比于本发明所采用的高通量测序平台,smrt测序价格高昂,不利于大批量样品的分析。上述结果表明本发明方法准确度高,能够用于其它物种dna中4mc的定位分析。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。
