与小麦粒长主效qtl连锁的snp分子标记kasp
‑
be
‑
kl
‑
sau2及应用
技术领域
1.本发明涉及作物分子遗传育种技术领域,特别是涉及与小麦粒长主效qtl连锁的snp分子标记kasp
‑
be
‑
kl
‑
sau2及应用。
背景技术:
2.普通小麦(triticum aestivum l.2n=6x=42)是温带地区的主要作物之一,为人类的饮食贡献了必需的氨基酸、矿物质和维生素,以及有益的膳食纤维。由于全球气候变化、人口增长和耕地面积的大量减少,只有提高作物产量潜力,才能满足未来粮食需求。因为粮食产量构成因素,如每穗可育小穗数、千粒重和单位面积有效分蘖数,通常表现出比粮食产量更高的遗传力,针对这些因素进行改良是提高小麦籽粒产量潜力的重要途径。
3.现代育种模式下小麦品种间遗传多样性的降低导致小麦改良的遗传基础日益狭窄。与祖先小麦品种相比,现代种质库中籽粒性状的表型变异明显减少。小麦的单株籽粒产量由每株穗数、每穗粒数和粒重构成。前人的研究表明粒长、粒宽以及粒厚都与粒重呈显著相关,并且可以直接影响小麦产量和品质。因此,在育种计划中增加籽粒长度可能是增加粒重的有效策略。小麦粒长是复杂的受多基因控制的数量性状,且对环境因素也很敏感。小麦基因组庞大而复杂,重复核苷酸序列较多,且缺乏注释基因组序列。分子标记辅助育种,不依赖于表型选择,即不受环境、基因互作、基因与环境互作等因素的影响,而是直接对基因型进行选择,因而能大大提高育种效率。
4.snp(single nucleotide polymorphism)标记是指在核苷酸序列中,某一处特定的位置单核苷酸位点a、t、g或c发生变异,使dna序列改变而出现多态性标记。snp在植物基因组中分布广泛、数量较多,基因的编码区和非编码区均存在。对于基因组较简单的生物来说,snp常被用于全基因组扫描,构建高密度遗传图谱,进行重要性状的qtl检测和分析;对于基因组较复杂的小麦来说,snp常常被用于跟基因芯片结合使用,例如9k、55k和660k snp芯片,用来检测目标性状相关的基因组区域,促进育种进程。
5.竞争性等位基因特异性pcr(kompetitive allele
‑
specific pcr,kasp)技术就是现代随着科技发展而出现的一种新型snp检测技术,它的工作原理是对snp位点进行特异性引物设计,通过snp的特异性来进行标记分型和检测,可应用于广泛的基因组dna,它的master mix通常是在普通pcr的基础上,使用由两种特殊荧光基团和猝灭基团做互补探针而构成的。而在之前也有一些分子标记,例如ssr、aflp等,这些在成本、标记密度、实验流程复杂程度、结果准确性等方面有一些弊端;kasp技术具有通量高、结果准确、成本低、操作简单、无需电泳经检测等特点,很好地解决了其他标记在应用上的缺点。
6.此前部分学者对小麦粒长进行了qtl定位,在小麦中几乎所有的21条染色体上均有控制粒长的qtl被检测到过。然而目前与小麦粒长性状相关且可用于实际分子育种的紧密连锁的分子标记却并不多。因此研究获得有关粒长的qtl或基因,利用分子生物学技术,选择合适的粒长小麦植株,对于育种工作中产量的提升是非常有必要的。
技术实现要素:
7.本发明的目的是提供与小麦粒长主效qtl连锁的snp分子标记kasp
‑
be
‑
kl
‑
sau2及应用,以解决上述现有技术存在的问题,该分子标记kasp
‑
be
‑
kl
‑
sau2与粒长qtl qkl.sau
‑
be
‑
4a极显著相关,呈现紧密连锁标记特征,用于分子标记辅助选择的准确性高,可显著提高不同环境下粒长较长小麦品种的选择鉴定效率,且成功率高。
8.基于以上目的,申请人利用小麦品种
‘
bls1’为父本,以小麦品系
‘
99e18’为母本杂交,得到杂种f1,f1代单株自交获得f2,在f2使用单粒传法加代到f3代,获得含有238个单株的群体,构成遗传作图群体。对f3群体粒长进行表型鉴定,将混合群体分离分析法(bsa,bulk segregant analysis)与小麦660k snp芯片结合,分析2个亲本和2个极端性状混池的差异snp。通过以下步骤,在两个相对极端混合池中各选取30个极端株系:(1)ble18的kl表型数据在三种环境中的排列顺序为先降后升。分别取3个环境中最大值和最小值的40个株系,记录其编号。(2)计算并记录三个环境每个株系表型数据的平均值。(3)对(1)和(2)得到的编号进行交集筛选,最终选取最大值和最小值各30个株系。采用ctab法提取粒长最短的30个株系(每株系10粒)、粒长最长的30个株系(每株系10粒)和2个亲本的等量dna用于小麦660k snp芯片分析,并对该群体进行图谱构建,从而定位粒长qtl。小麦660k芯片是由中国农业科学院作物研究所的贾继增课题组开发的一款小麦多倍体二倍体化的高密度snp芯片,共含有630518个snp标记。与90k芯片相比,660k芯片中snp的数量显着增加,是适合于一般的种质资源多样性分析、一般的遗传作图、新基因发现、比较基因组分析与品种注册与鉴别等的snp芯片,能服务于接下来的标记开发和精细定位工作。
9.根据660k snp芯片数据,在目标染色体的候选区段内开发kasp标记对群体进行基因分型,利用joinmap4.0构建遗传图谱。结合群体的粒长表型数据,用qtl icimapping 4.0中的完备区间作图法(inclusive composite interval mapping,icim),设置阀值lod≥2.5的条件下,用2020年3个生态点及3个生态点粒长的blup(最佳线性无偏预测,best linear unbiased prediction)值来检测qtl,在4a染色体短臂上的2cm区间定位出稳定表达的小麦粒长主效qtl qkl.sau
‑
be
‑
4a,开发分子标记,最终得到标记kasp
‑
be
‑
kl
‑
sau2与粒长qtl qkl.sau
‑
be
‑
4a紧密连锁。
10.本发明所述的小麦粒长qtl qkl.sau
‑
be
‑
4a,来自父本
‘
bls1’,该qtl位于小麦染色体4a短臂上,在refseqv2.0基因组版本的物理位置为101.7
‑
109.2mbp。本发明提供了上述小麦粒长qtl qkl.sau
‑
be
‑
4a在调控小麦籽粒性状中的应用。所述小麦粒长qtl qkl.sau
‑
be
‑
4a显著增加小麦籽粒长度,平均lod值为5.06,解释约10.87
‑
19.30%的表型变异。
11.为实现上述目的,本发明提供了如下方案:
12.本发明提供一种与小麦粒长qtl qkl.sau
‑
be
‑
4a连锁的snp分子标记kasp
‑
be
‑
kl
‑
sau2,其多态性为c/t,所述kasp
‑
be
‑
kl
‑
sau2与小麦粒长qtl qkl.sau
‑
be
‑
4a共定位于小麦4a染色体短臂上,且位于qtl qkl.sau
‑
be
‑
4a区间内。
13.本发明提供一种引物组,包括用于扩增所述kasp
‑
be
‑
kl
‑
sau2的两条特异性引物和一条通用引物,两条所述特异性性引物的核苷酸序列如seq id no:4
‑
5所示,所述通用引物的核苷酸序列如seq id no:6所示。
14.本发明一种检测小麦全基因或其基因片段的芯片,包括所述的kasp
‑
be
‑
kl
‑
sau2
或所述的引物组。
15.本发明还提供一种检测小麦全基因或其基因片段的试剂盒,包括所述的kasp
‑
be
‑
kl
‑
sau2或所述的引物组。
16.本发明化提供一种所述的kasp
‑
be
‑
kl
‑
sau2或所述的引物组或所述的芯片或所述的试剂盒的应用,用于如下任一项应用中:
17.(1)筛选具有合适粒长的小麦品种或品系;
18.(2)调控小麦粒长性状;
19.(3)提高小麦产量;
20.(4)小麦粒长基因遗传分析或遗传精细定位。
21.本发明还提供一种筛选含有粒长qtl qkl.sau
‑
be
‑
4a的小麦株系方法,包括以下步骤:
22.以待测植株样品的基因组dna作为模板,利用所述的引物组对模板进行荧光定量pcr扩增,利用扩增结果进行基因型分型。
23.优选的是,荧光定量pcr扩增反应体系为:7.5μl master mix、2.1μl混合引物、6ng模板dna、双蒸水加至总量为15μl;其中,所述混合引物是由如seq id no:4
‑
seq id no:6所示引物均按照10ng/μl的浓度,分别加入120μl、120μl和300μl,并添加460μl ddh2o进行混合后制备而成。
24.优选的是,荧光定量pcr扩增反应程序为:94℃预变性15min;94℃变性20s、60℃复性/延伸50s,共8个循环;94℃变性20s、55℃复性/延伸60s,共28个循环。
25.本发明还提供所述的方法在小麦分子育种、培育转基因小麦和小麦种质资源改良中应用。
26.本发明还提供一种小麦kasp
‑
be
‑
kl
‑
sau2分子标记或粒长基因qtl qkl.sau
‑
be
‑
4a在调控小麦粒长性状中的应用。
27.本发明公开了以下技术效果:
28.(1)本发明首次公开了来自小麦
‘
bls1’的粒长qtl qkl.sau
‑
be
‑
4a,位于小麦4a染色体短臂上,显著增加小麦粒长。该qtl在小麦产量(调控粒长)育种中具有较高的利用价值。
29.(2)本发明公开了基于荧光定量pcr平台精确检测小麦
‘
bls1’的粒长qtl qkl.sau
‑
be
‑
4a的分子标记kasp
‑
be
‑
kl
‑
sau2,检测准确高效、扩增方便稳定。
30.(3)本发明公开的分子标记kasp
‑
be
‑
kl
‑
sau2与粒长qtl qkl.sau
‑
be
‑
4a极显著相关,呈现紧密连锁标记特征,用于分子标记辅助选择的准确性高,可显著提高适应不同环境的小麦较长籽粒品种的选择鉴定效率,且成功率高。
31.(4)本发明公开了位于小麦4a染色体上的与小麦粒长连锁的分子标记kasp
‑
be
‑
kl
‑
sau2,该分子标记是小麦4a染色体短臂上粒长qtl qkl.sau
‑
be
‑
4a的侧翼标记,连锁度高。该标记可用来检测小麦4a染色体上的粒长qtl,快速筛选具有该位点的植株,进而方便进行高产小麦的分子辅助育种。
32.(5)本发明提供的分子标记kasp
‑
be
‑
kl
‑
sau2与小麦4a上的粒长qtl qkl.sau
‑
be
‑
4a紧密连锁,可用来对小麦粒长这一性状进行定位,从而在育种过程中淘汰较短籽粒的植株,提高育种工作效率,并为小麦粒长基因的研究提供基础。
附图说明
33.为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
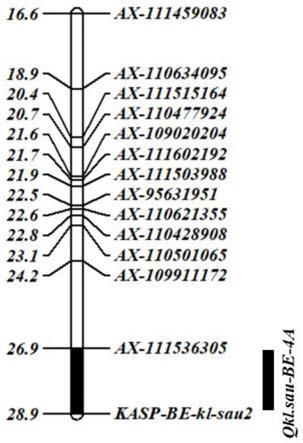
34.图1为本发明实施例1中小麦粒长qtl qkl.sau
‑
be
‑
4a在4a染色体上的定位;
35.图2为本发明实施例1中,小麦
‘
bls1
’ב
99e18’f3验证群体的株系植株分子标记qkl.sau
‑
be
‑
4a检测的荧光读值结果;其中,fam(圆形,
‘
99e18’)荧光为有较短籽粒的株系,hax(正方形,
‘
bls1’)荧光为较长籽粒株系;黑色菱形荧光为空白对照;
36.图3为本发明实施例2中,小麦
‘
bls1
’ב
sumai3’f3验证群体的株系植株分子标记qkl.sau
‑
be
‑
4a检测的荧光读值结果;其中,fam(圆形,
‘
sumai3’)荧光为有较短籽粒的株系,hax(正方形,
‘
bls1’)荧光为较长籽粒株系;黑色菱形荧光为空白对照。
具体实施方式
37.现详细说明本发明的多种示例性实施方式,该详细说明不应认为是对本发明的限制,而应理解为是对本发明的某些方面、特性和实施方案的更详细的描述。
38.应理解本发明中所述的术语仅仅是为描述特别的实施方式,并非用于限制本发明。另外,对于本发明中的数值范围,应理解为还具体公开了该范围的上限和下限之间的每个中间值。在任何陈述值或陈述范围内的中间值以及任何其他陈述值或在所述范围内的中间值之间的每个较小的范围也包括在本发明内。这些较小范围的上限和下限可独立地包括或排除在范围内。
39.除非另有说明,否则本文使用的所有技术和科学术语具有本发明所述领域的常规技术人员通常理解的相同含义。虽然本发明仅描述了优选的方法和材料,但是在本发明的实施或测试中也可以使用与本文所述相似或等同的任何方法和材料。本说明书中提到的所有文献通过引用并入,用以公开和描述与所述文献相关的方法和/或材料。在与任何并入的文献冲突时,以本说明书的内容为准。
40.在不背离本发明的范围或精神的情况下,可对本发明说明书的具体实施方式做多种改进和变化,这对本领域技术人员而言是显而易见的。由本发明的说明书得到的其他实施方式对技术人员而言是显而易见的。本技术说明书和实施例仅是示例性的。
41.关于本文中所使用的“包含”、“包括”、“具有”、“含有”等等,均为开放性的用语,即意指包含但不限于。
42.以下实施例中小麦品系
‘
99e18’由四川省农科院作物所朱华忠研究员提供,四川农业大学小麦所保存;
‘
bls1’由孟加拉国库尔拉大学ahsan habib教授提供,四川农业大学小麦所保存;
‘
sumai3’由四川农业大学小麦所保存和提供。
43.实施例1小麦粒长qtl qkl.sau
‑
be
‑
4a及其分子标记kasp
‑
be
‑
kl
‑
sau2的获得
44.(1)利用小麦品系
‘
99e18’为母本,以小麦品种
‘
bls1’为父本杂交,得到杂种f1,f1代单株自交获得f2,在f2使用单粒传法到f3代,获得含有238个单株的重组自交系,构成遗传作图群体。
45.(2)f3群体粒长表型鉴定:小麦成熟期对f3群体株系进行收获,脱粒,烘干并进行
籽粒性状表型鉴定,每个株系进行30个籽粒长度重复值的测量,并得出平均值,代表该株系的粒长。
46.(3)运用混合群体分离分析法(bsa),构建两个亲本池,并在f3群体中选取两组分别具有30个极端粒长差异的株系分别构建混池。
47.(4)660k snp芯片分析
48.a)dna提取:用ctab法提取亲本
‘
99e18’、
‘
bls1’和f3群体植株dna。
49.b)使用超微量分光光度计对提取的dna进行质量检测,合格后送样至公司进行基因型分析,在本研究中双亲和作图群体的基因型分析由北京博奥晶典生物技术有限公司(http://www.capitalbio tech.com)与贾继增课题组合作开发的660k snp芯片完成,该芯片市售可得。
50.c)通过筛选两个极端混池之间有差异的snp,并将这些差异snp位点整合到小麦的21条染色体上,结果表明,在4a染色体上含量最多,因此初步确定在4a染色体上可能存在着控制粒长的位点。并且在4a染色体90
‑
160mbp之间差异snp的集中度最高,将这一段被选为候选区段。
51.d)连锁图谱的构建:根据660k snp芯片数据,在目标染色体的候选区段及其周围,开发可用的kasp标记对ble18群体进行基因分型,利用joinmap4.0构建遗传图谱。结合群体的粒长表型数据,用qtl icimapping 4.0中的完备区间作图法,设置阀值lod≥2.5的条件下,用2020年共3个生态点及3个生态点粒长的blup(最佳线性无偏预测,best linear unbiased prediction)值来检测qtl,定位出小麦粒长qtl qkl.sau
‑
be
‑
4a,并计算qtl qkl.sau
‑
be
‑
4a的位置和分子标记之间的遗传距离。
52.e)粒长位点的比较以及分子标记的获得:huang等人利用多对近等基因系对3个与粒重相关的qtl(qgw.nau
‑
2d、qgw.nau
‑
4b和qgw.nau
‑
5a)进行研究,验证了这3个qtl对粒长、粒宽和粒厚均有影响(huang et al.2015,theoretical and applied genetics,128:2437
‑
2445)。brinton等人利用charger
×
badger对粒长相关qtl有更深入的研究,在检测到主效qtl的同时对qtl的分子机制进行了一定的研究。其检测到一个位于5a的主效qtl,该qtl通过增加种皮细胞的长度从而增加粒长,达到增加粒重的目的(brinton et al.2017,new phytologist,215:1026
‑
1038)。zhang等人也发现水稻osgs3的同源基因tags
‑
d1与普通小麦的粒重和粒长有关,tags
‑
d1位于7ds染色体上,可以增加粒长和粒重(zhang et al.2014,molecular breeding,34:1097
‑
1107)。su利用重组自交系分析了7al上一个与粒长相关的qtl,发现一个snp与其紧密连锁(su et al.2016,molecular breeding 36:15)。位于4a上的qtl很少且都与qkl.sau
‑
be
‑
4a的距离很远,这表明qkl.sau
‑
be
‑
4a是一个新的稳定的qtl。
53.为了进一步获得与粒长qtl qkl.sau
‑
be
‑
4a紧密连锁的分子标记,利用660k snp芯片数据定位结果对侧翼标记进行物理定位并筛选位于区间内的基因。开发获得高效的kasp分子标记,需要如下多个步骤:
54.(i)设计扩增特定小麦基因型目标同源染色体(4a)候选基因序列的引物。虽然目前已有六倍体小麦
‘
中国春’参考基因组,但是因为小麦进化过程中可能有染色体结构变异(ma j,stiller j,wei y,zheng y
‑
l,devos km,j,liu c(2014)extensive pericentric rearrangements in the bread wheat(triticum aestivum l.)genotype
“
chinese spring”revealed from chromosome shotgun sequence data.genome biol evol6:3039
‑
3048),不同小麦基因型的基因序列结构和多态性位点差异可能较大,要高效、快速、低成本地获得特定小麦基因型的基因序列,最简单的方法便是同源序列克隆。而因为小麦具有三个部分同源染色体组,要分离获得特异于其中某个同源染色体上的序列具有很大难度(bagge m,xia x,l
ü
bberstedt t(2007)functional markers in wheat.curr opin plant biol 10:211
‑
216),需要通过设计特异于某个染色体组上的引物。在熟练掌握比较基因组学技术、生物信息学技术的基础上,对六倍体小麦的供体二倍体亲本乌拉尔图小麦和节节麦、四倍体野生二粒小麦和六倍体
‘
中国春’等参考基因组进行序列截取、比对、分析,获得特异于某个染色体上的多态性位点,从而设计扩增目标区域的特异引物。在设计好引物后,需依靠比较基因组学技术对引物特异性、退火温度、扩增长度等进一步分析,从而确定引物的可用性。
55.(ii)利用特异引物对目标小麦基因型进行扩增。在进行扩增时,需要凭借熟练的分子生物学技术对扩增条件进行优化,进一步克隆测序。以获得目标区域的基因序列。
56.(iii)目标同源染色体候选基因序列多态性位点的获得。在获得两个亲本候选基因序列后,需要进一步对序列进行详细分析,检测是否有多态性位点,如果没有,则需要重新回到第i步,选择其他可能的候选区域进行分离克隆。
57.(iv)多态性位点上下游kasp引物的设计。在获得多态性位点之后,需要设计kasp特异引物。由如前所述,小麦为异源六倍体植物,则仍然要凭借熟练的生物信息学技术对abd染色体序列进行分析,从而获得特异于目标染色体的kasp引物。
58.(v)kasp引物扩增条件的优化。引物合成之后,需要凭借经验进一步对引物扩增条件进行调试优化,以便能达到在亲本之间能够显著区分的效果。
59.综上所述,虽然kasp标记技术已经广泛应用于二倍体物种,但是要想在六倍体小麦中获得高效的kasp标记时,对本领域技术人员而言绝非易事。
60.最终经过多次kasp标记开发,引物设计和扩增,共设计kasp引物7对,最终得到标记kasp
‑
be
‑
kl
‑
sau2(多态性为c/t)与粒长qtl qkl.sau
‑
be
‑
4a紧密连锁(表1)。
61.表1 kasp引物序列
62.63.[0064][0065]
设计的7对kasp引物中最终得到了1个分子标记kasp
‑
be
‑
kl
‑
sau2,其与粒长qtl qkl.sau
‑
be
‑
4a紧密连锁,结果见图1
‑
2。
[0066]
实施例2分子标记kasp
‑
be
‑
kl
‑
sau2在选择控制粒长qtl qkl.sau
‑
be
‑
4a上的应用
[0067]
(1)利用较长粒长的普通小麦品
‘
bls1’为父本,普通小麦品系
‘
sumai3’为母本构建f3群体,在后代株系中选择183个株系。
[0068]
(2)对所获得的183个株系进行kasp
‑
be
‑
kl
‑
sau2标记检测,具体方法为:提取183个株系的dna;将其作为模板,以分子标记kasp
‑
be
‑
kl
‑
sau2的特异性引物对为引物进行pcr扩增并进行荧光读值,所述引物为:
[0069]
fam标签上引物:(下划线部分为fam标签序列)5
’‑
gaaggtgaccaagttcatgctccagccggcaggtggaatgcc
‑3’
(seq id no:4)
[0070]
hex标签上引物:(下划线部分为hex标签序列)5
’‑
gaaggtcggagtcaacggattccagccggcaggtggaatgct
‑3’
(seq id no:5)
[0071]
通用下游引物:5
’‑
aaggggaaggtcagctgctg
‑3’
(seq id no:6)
[0072]
pcr扩增的扩增体系为:7.5μl master mix、混合引物2.1μl、6ng模板dna、双蒸水加至总量为15μl,其中,混合引物是由seq id no:4
‑
seq id no:6所示引物均按照10ng/μl的浓度,分别加入120μl,120μl和300μl并添加ddh2o 460μl进行混合后制备而成。同时需添
加至少3个独立的以双蒸水代替dna模板的空白。
[0073]
pcr扩增的反应程序为:94℃预变性15min;94℃变性20s、60℃复性/延伸50s,共8个循环;94℃变性20s、55℃复性/延伸60s,共28个循环;完成后进行荧光读值。
[0074]
荧光读值结果如图3所示,将检测到与
‘
suami3’一致的fam荧光的植株基因型记为b,为粒长较短的株系,同
‘
bls1’一样表现为hax荧光的植株基因型记为a,为粒长较长的株系。各个株系基因型与粒长的表型值如表2所示。
[0075]
表2
‘
bls1
’ב
sumai3’f3群体kasp
‑
be
‑
kl
‑
sau2基因型与表型对应结果
[0076]
[0077][0078]
由表2可见,与含有粒长qtl qkl.sau
‑
be
‑
4a的
‘
bls1’类型相同的植株平均粒长为8.3mm,极显著高于与
‘
sumai3’类型的植株平均粒长(7.9mm)。实际结果与预期结果一致,说明本发明的粒长qtl qkl.sau
‑
be
‑
4a确实有显著增加粒长的作用;同时本发明的分子标记kasp
‑
be
‑
kl
‑
sau2可以用于跟踪鉴定粒长qtl qkl.sau
‑
be
‑
4a。
[0079]
以上所述的实施例仅是对本发明的优选方式进行描述,并非对本发明的范围进行限定,在不脱离本发明设计精神的前提下,本领域普通技术人员对本发明的技术方案做出的各种变形和改进,均应落入本发明权利要求书确定的保护范围内。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。