1.本发明涉及海洋碳汇技术领域,具体涉及一种用于海洋微型生物碳汇过程监测的高准确度功能基因芯片制备方法。
背景技术:
2.自工业革命以来,人类活动大量排放二氧化碳,导致气候变化已经成为最大的全球性环境问题,严重威胁着人类社会可持续发展。我国作为世界上最大的碳排放国之一,率先提出碳中和日程,但我国现阶段仍处于经济高度增长的阶段,仍位于发展中国家的状态,这就要求我国实现碳中和目标时要将“减排”和“增汇”并行。2019年第25届联合国气候变化大会首次将“海洋议题”纳入会议议程,旨在提高各国对海洋与气候变化关系的认知能力,并鼓励各国将海洋纳入应对气候变化的途径中。我国作为海洋大国,海洋自然条件优越且空间资源优渥,包括海岸线漫长、海岸带资源丰富且领海面积辽阔等特点。其中,我国海岸线总长度超过32600千米,纵跨热带、亚热带、温带等多个气候带,领海面积约300万平方公里,约占领土面积的31.25%。现已有研究证明海洋碳库为陆地碳库的20倍,为大气碳库的50倍,海洋作为地球上最大的碳库,被誉为全球气候变化的重要“调节器”,每年可吸收约排放入大气中co2的30%,海洋碳汇潜力巨大,因此,解析海洋碳汇链条关键过程,挖掘海洋负排放潜力,是助力我国实现“碳中和”目标的重要途径之一。
3.人们多年来主要将研究聚焦于关键的海洋储碳机制——生物泵(biological pump,bp),该理论指出,无机碳通过光合作用等作用被转化为有机碳,但近三十年的研究积累结果表明,其所导致的颗粒有机碳(poc)仅有约初级生产力的0.1%可以到达海底埋藏,而其中绝大部分poc则被转化为co2重新返回大气中。实际上,海洋中的有机碳主要存在形式为溶解有机碳(dissolved organic carbon,doc),而其中约95%的doc以生物难以利用的惰性溶解有机碳(recalcitrant dissolved organic carbon,rdoc)形式存在。由我国科学家提出的海洋微型生物碳泵(microbial carbon pump,mcp)指出,海洋微生物通过生理生态与生物地球化学过程,将有机碳从可利用的活性态转化为不可利用的惰性态rdoc,使其可在海底封存周期可达数千年。mcp一经提出便得到国际同行的广泛关注和认可,被称为“巨大碳库的幕后推手”。mcp强调微型生物是形成rdoc的主要贡献者,微型生物个体虽小但生物量极大、生物多样性高,已有大型海洋生态系统实验体系研究证明微生物碳泵效率极高,在不到一年的时间中可将浮游植物产生的活性doc转化为与深海中rdoc十分相近的化合物。但由于海洋生态系统和碳汇形成过程的复杂性以及现有知识体系的局限性,决定了海洋碳汇核查技术研发的难度,迄今国际上尚未确定微型生物驱动海洋碳汇链条关键过程的关键类群及功能基因,我国典型海区碳指纹基因情况也尚未明晰。
4.我国碳市场作为全球配额交易量第二大的市场,与“海洋碳汇”相比,“林业碳汇”发展已经较成熟,包括林木碳汇量的测定,检测等。“林业碳汇”已经进入碳交易市场并已成功交易多起,逐步实现中国林业碳汇生态效益价值化。对于“海洋碳汇”的发展,目前对海岸带“蓝色碳汇”的计量与检测的研究已经启动,主要涉及红树林、海藻床等,但评价标准、核
查标准等方面仍然欠缺,现阶段仍基本处于空白状态。海洋生态系统复杂,各不同的微型生物代表类群所产生的doc代谢活性不同,不同相对活性doc转化为不尽相同类型的rdoc。现阶段,基因芯片为探索海洋微型生物有机碳代谢特征有效且富有前景的手段之一,且基因芯片只包含研究者所感兴趣的部分关键基因,使后续的分析更加的简单、省时且经济有效。且随着下一代测序技术(ngs,next generation sequencing)的发展,测序获得的数据量呈指数增长,后续数据分析的简便性和有效性成为人们关注的重点之一。传统的聚合酶链式反应(pcr,polymerase chain reaction)对引物的选择以及扩增要求较高,而仅针对研究者感兴趣的功能基因进行引物设计十分困难,尤其是对于复杂环境中的样本,最终扩增结果的准确性不易把握,实验的可重复性较低,综合以上因素,基因芯片则表现出其在研究海洋这类复杂环境、解析碳汇链条关键过程方面特有的优势。现有的基因芯片主要包括高通量qpcr基因芯片和功能基因芯片,高通量qpcr基因芯片无法将微生物群落结构和其生态系统功能相联系,功能基因芯片不仅可以将物种和功能联系,且更适用于研究来自复杂环境的样本,但专门针对海洋碳汇过程的功能基因芯片至今仍未被研发。
技术实现要素:
5.为解决现有技术存在的不足,本发明提供了一种用于海洋碳汇的高准确度功能基因芯片制备方法。
6.本发明的技术方案为:
7.一种用于海洋碳汇的高准确度功能基因芯片制备方法,包括如下步骤:
8.步骤1:构建碳汇功能基因数据库。
9.步骤2:获取海区碳汇过程关键功能基因数据集。
10.步骤3:设计寡核苷酸探针。
11.步骤4:合成功能基因芯片。
12.进一步的,步骤1的具体步骤为:
13.1)获取参与海洋碳汇过程的微型生物关键基因家族的基础核苷酸和蛋白序列,通过序列比对将通过以序列相似性30%作为阈值的部分序列纳入种子数据库。
14.2)将1)中的种子数据库与公共数据库进行融合,将种子数据库与公共同源数据库进行序列比对,使用参数为e值1e
‑
5,全局序列相似性30%以获取属于目标基因组的同源序列,提取比对结果符合阈值的序列,将其纳入种子数据库,对结合后的数据库去冗余,形成碳汇功能基因数据库。
15.进一步的,步骤2的具体步骤为:对我国海区进行航次调查与现场采样,获得不同深度的海水与沉积物样品,对海水样品进行海水滤膜过滤,提取滤膜与沉积物样品dna以获得微生物群落总dna并进行宏基因组测序获得海洋宏基因组数据集,与碳汇功能基因数据库比对,以获得海区碳汇过程关键功能基因数据集。
16.进一步的,步骤3中寡核苷酸探针由针对单一序列的序列特异性探针和针对多条序列的群组特异性探针合成,所述序列特异性探针和群组特异性探针通过条件筛选获取最终的探针。
17.更进一步的,单一序列的序列特异性探针的筛选条件为:针对单一序列的序列特异性探针与非目标序列之间:与序列相似度不高于90%
‑
95%,不超过20个连续碱基相同,
吉布斯自由能不低于
‑
32kcal/mol。
18.进一步的,群组特异性探针的筛选条件为:针对多条序列的群组特异性探针与非目标序列之间:序列相似度不高于90%
‑
95%,不超过20个连续碱基相同,吉布斯自由能不低于
‑
32kcal/mol;同时属于同一组的特异性探针之间:序列相似度高于96%
‑
98%,超过35个连续的碱基保持一致,吉布斯自由能不高于
‑
60kcal/mol。
19.进一步的,步骤4的具体步骤为:选择一定数量的碳汇功能基因探针,在探针理论杂交温度在42℃
‑
45℃,按照一定坐标位置将其排列在芯片上,将被标记的若干靶核苷酸序列与mcpchip特定位点上的探针杂交,通过探针原位合成或使用阵列机器人将探针附着于固相表面,制作合成功能基因芯片mcpchip。
20.本发明所达到的有益效果为:
21.本发明通过宏基因组大数据挖掘,更加全面的涵盖海洋碳汇关键功能基因家族,以减轻因缺少相关基因参考而导致的假阴性问题。
22.本发明区分公共同源基因,减轻因现有公共基因数据库中同源基因家族混杂而导致的假阳性问题。
23.本发明在探索海洋碳汇过程中,基于海洋宏基因组数据集,以经济有效的方法获得准确度高、可靠性强的数据结果。
附图说明
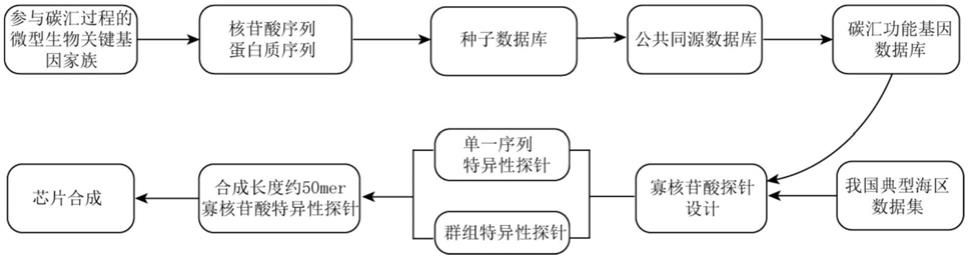
24.图1是本发明一种用于海洋碳汇的高准确度功能基因芯片制备方法的流程图。
具体实施方式
25.为便于本领域的技术人员理解本发明,下面结合附图说明本发明的具体实施方式。
26.本发明基于海洋宏基因组数据集,提供一种用于解析海洋碳汇高准确度功能基因芯片的制备方法,制备方法流程请参考图1,具体包括以下步骤:
27.步骤1:构建高准确度的碳汇功能基因数据库
28.通过基因组大数据挖掘,明确参与海洋碳汇过程的微型生物关键基因家族,获得相应的核苷酸和蛋白序列。碳汇功能基因数据库包括碳固定、碳排放、有机物合成、有机物降解、有机物转化等多个与碳汇过程相关的子过程,同时整合现有功能基因芯片体系中与海洋微型生物介导的碳汇过程紧密相关的功能基因。最终覆盖碳基因家族将大于2,500个,包含基因序列大于100,000条。为使本发明的技术方案更加清晰,特此对该步骤进行进一步详细说明。
29.(a)通过基因组大数据挖掘,明确参与海洋碳汇过程的微型生物关键基因家族,获得基础的核苷酸和蛋白序列,后利用序列比对工具(如diamond和usearch)将获得的核苷酸和蛋白序列进行筛选过滤,将通过以序列相似性30%作为阈值的部分序列纳入种子数据库。
30.(b)将种子数据库与公共数据库(如arcog、cog、kegg、eggnog、genbank refseq等)进行融合,将种子数据库与公共同源数据库进行序列比对,使用参数为e值1e
‑
5,全局序列相似性30%以获取属于目标基因组的同源序列,以降低现有数据库中所普遍存在较高假阳
性的问题。提取比对结果符合阈值的序列,将其纳入种子数据库,对结合后的数据库去冗余,形成碳汇功能基因数据库mcpbase。
31.步骤2:获取我国典型海区碳汇过程关键功能基因数据集
32.对我国典型海区(如渤海贝藻养殖区,黄海浒苔爆发区,南海珊瑚礁区等海区)进行航次调查与现场采样,获得不同深度的海水与沉积物样品,对海水样品进行滤膜过滤,提取滤膜和沉积物dna以获得微生物群落总dna并进行宏基因组测序获得海洋宏基因组数据集,与碳汇功能基因数据库mcpbase比对,获得我国典型海区碳汇过程关键功能基因数据集。
33.步骤3:设计寡核苷酸探针
34.由于样本存在复杂性高,功能基因序列同源性较高等问题,本发明将设计两种特异性探针核苷酸序列——针对单一序列的序列特异性探针和针对多条序列的群组特异性探针。最终合成的寡核苷酸探针长度约50mer。
35.基于探针设计工具和算法(如commoligo),同时将序列相似度、允许连续碱基长度、吉布斯自由能等条件考虑在内,并以0.1作为全局匹配的阈值,将待选的寡核苷酸探针进行排序,以选择最优的对象作为最终的探针。本发明中,寡核苷酸探针的筛选标准包括以下条件:
36.(a)针对单一序列的序列特异性探针与非目标序列之间:与序列相似度不高于90%
‑
95%,不超过20个连续碱基相同,吉布斯自由能不低于
‑
32kcal/mol。
37.(b)针对多条序列的群组特异性探针与非目标序列之间:序列相似度不高于90%
‑
95%,不超过20个连续碱基相同,吉布斯自由能不低于
‑
32kcal/mol;同时属于同一组的特异性探针之间:序列相似度高于96%
‑
98%,超过35个连续的碱基保持一致,吉布斯自由能不高于
‑
60kcal/mol。
38.步骤4:芯片合成
39.选择一定数量的碳汇功能基因探针,在探针理论杂交温度在42℃
‑
45℃,按照一定坐标位置将其排列在芯片上,将被标记的若干靶核苷酸序列与mcpchip特定位点上的探针杂交,通过探针原位合成或使用阵列机器人将探针附着于固相表面,制作合成功能基因芯片mcpchip。
40.以上所述的本发明实施方式,并不构成对本发明保护范围的限定。任何在本发明的精神和原则之内所作的修改、等同替换和改进等,均应包含在本发明的权利要求保护范围之内。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。