1.本发明涉及单细胞测序领域,特别是涉及微生物单细胞全基因组扩增及测序文库构建方法。
背景技术:
2.长期以来,因缺乏强有力的研究方法,微生物在很大程度上仍是一个黑箱。由于绝大多数微生物无法在现有实验条件下纯培养,对微生物组的发掘及研究主要依赖与宏基因组学方法。宏基因组学以环境样本中微生物群体基因组作为研究对象,本质上不具备单细胞的分辨率,无法获得单一物种的独立数据,而对于未知微生物的充分挖掘往往需要获取该种微生物的全基因组信息。因此,主流宏基因组学方法在研究较为稀有的微生物种群时捉襟见肘;开发高效的、具有微生物单细胞分辨率的微生物组研究方法,特别是大规模全基因组扩增及测序文库构建方法,对于推动微生物组学的研究具有重要价值。
3.近年来,高通量单细胞测序技术的飞速发展正逐渐将生命科学推向单细胞时代。单细胞转录组测序、甲基化测序等先进方法学的成功建立,使得科研人员能够在单细胞层面分析基因表达,表观遗传等信息。此类方法学主要依赖于高通量微流控技术(特别是微液滴技术)来高效地进行单细胞测序文库制备。然而,在微生物研究领域,单细胞分析并不普及,相关方法学的开发也较为困难。以全基因组扩增及测序文库构建为例,由于微生物基因组较小(kbp-mbp级别),使用聚合酶链反应(polymerase chain reaction,pcr)、多重置换扩增(multiple displacement amplification,mda)等方法并不能获得高质量的扩增产物,往往会伴随着高扩增偏倚和大量基因组片段丢失。此外,还存在着如何进行单基因组标记等测序文库制备的难题。
技术实现要素:
4.鉴于以上所述现有技术的缺点,本发明的目的在于提供微生物单细胞全基因组扩增及测序文库构建方法。
5.本发明第一方面提供一种微生物单细胞全基因组的扩增方法,至少包括如下步骤:
6.(1)将微生物单细胞捕获于单一微腔室中,获得多个独立包含微生物单细胞的微腔室:
7.(2)在包含微生物单细胞基因组及扩增反应体系的微腔室中,进行微生物单细胞全基因组扩增,获得包含微生物单细胞全基因组扩增产物。
8.本发明第二方面提供微生物单细胞全基因组的测序文库构建方法,包括如下步骤:
9.a.将前述微生物单细胞全基因组的扩增方法获得的包含微生物单细胞全基因组扩增产物的微腔室中的扩增产物片段化,获得包含片段化扩增产物的微腔室;
10.b.将步骤a获得的微腔室中的片段化扩增产物与条形码片段在单一微腔室中连
接,获得微生物单细胞全基因组的测序文库,所述测序文库中,所述微生物单细胞全基因组处于微腔室中,且每个微腔室中有且仅有单一微生物单细胞全基因组。
11.如上所述,本发明的微生物单细胞全基因组扩增及测序文库构建方法,具有以下有益效果:
12.a.本项发明将可为探索微生物暗物质提供新的重要方法。
13.b.本发明中的微生物单细胞全基因组扩增方法将通过大幅压缩反应体系体积、提高反应体系内底物的相对浓度、隔绝外界以及交叉污染,提高微生物单细胞全基因组扩增质量和提高基因组测序覆盖率。
14.c.本发明中,测序文库构建方法中,通过在独立微腔室内将微生物单细胞全基因组扩增产物片段与携带条形码序列的微球或微液滴融合,并在pcr或ligase(连接酶)作用下,完成微生物单细胞全基因组扩增产物的索引化步骤;该方法可大幅提高测序效率和基因组的测序覆盖率。
附图说明
15.图1:本发明一实施例的微生物单细胞全基因组测序文库构建方法流程图。
16.图2:包裹有单个大肠杆菌或单个枯草芽孢杆菌的微液滴(有箭头)。
17.图3:mda反应后,包含有单菌全基因组扩增产物的微液滴(绿色荧光表示里面含有单个大肠杆菌或枯草芽孢杆菌的全基因组扩增产物。
18.图4:包含有单菌全基因组扩增产物的聚丙烯酰胺水凝胶微球。
19.图5:含单菌全基因组扩增产物片段的聚丙烯酰胺水凝胶微球。
20.图6:未经磁珠分选的文库与经磁珠分选后的文库的bioanalyzer分析图。
21.图7:索引序列与对应reads数量的分布。
22.图8:测序数据参照大肠杆菌和枯草芽孢杆菌基因组进行比对的结果。
具体实施方式
23.以下通过特定的具体实例说明本发明的实施方式,本领域技术人员可由本说明书所揭露的内容轻易地了解本发明的其他优点与功效。本发明还可以通过另外不同的具体实施方式加以实施或应用,本说明书中的各项细节也可以基于不同观点与应用,在没有背离本发明的精神下进行各种修饰或改变。
24.在进一步描述本发明具体实施方式之前,应理解,本发明的保护范围不局限于下述特定的具体实施方案;还应当理解,本发明实施例中使用的术语是为了描述特定的具体实施方案,而不是为了限制本发明的保护范围;在本发明说明书和权利要求书中,除非文中另外明确指出,单数形式“一个”、“一”和“这个”包括复数形式。
25.当实施例给出数值范围时,应理解,除非本发明另有说明,每个数值范围的两个端点以及两个端点之间任何一个数值均可选用。除非另外定义,本发明中使用的所有技术和科学术语与本技术领域技术人员通常理解的意义相同。除实施例中使用的具体方法、设备、材料外,如本技术领域的技术人员对现有技术的掌握及本发明的记载,还可以使用与本发明实施例中所述的方法、设备、材料相似或等同的现有技术的任何方法、设备和材料来实现本发明。
26.除非另外说明,本发明中所公开的实验方法、检测方法、制备方法均采用本技术领域常规的分子生物学、生物化学、染色质结构和分析、分析化学、细胞培养、重组dna技术及相关领域的常规技术。
27.本发明一实施例的微生物单细胞全基因组的扩增方法,至少包括如下步骤:
28.(1)将微生物单细胞捕获于单一微腔室中,获得多个独立包含微生物单细胞的微腔室:
29.(2)在包含微生物单细胞基因组及扩增反应体系的微腔室中,进行微生物单细胞全基因组扩增,获得包含微生物单细胞全基因组扩增产物。
30.在微腔室中进行扩增可以大幅度的压缩反应体系的体积,同时可以提高反应体系内底物的相对浓度。例如以20μl体系在pcr管中进行单细胞的全基因组扩增与在单个微液滴中进行全基因组扩增,液滴的体积大概是nl级别,因此可以大幅的压缩反应的体积,同时一个微生物单细胞的基因组在pcr管中与在单个液滴中的相对浓度是不同的。
31.本项发明中,可通过利用各种技术,将目标微生物单细胞捕获在独立微腔室(皮升至纳升级别)中,以备进行后续实验。
32.可选的,所述微腔室的形式可选自但不限于微液滴、微球、半透膜体系、脂质体体系、微孔板或离心管。
33.所述微腔室的体积大小为皮升至纳升数量级。
34.在一种实施方式中,所述微腔室内含有微腔室基质,所述微腔室基质可选自但不限于聚丙烯酰胺、聚乙二醇或琼脂糖。
35.可选的,步骤(1)可在微流控液滴生成芯片中进行。具体的,将油、微生物单细胞裂解液、微生物单细胞悬浮液泵入微流控液滴生成芯片的流道中混合获得包含微生物单细胞全基因组的微液滴。
36.所述微腔室的体积大小为皮升至纳升数量级。
37.步骤(2)中,所述形成包含微生物单细胞基因组及扩增反应体系的微腔室的方式选自以下任一:
38.i.对单一微腔室中所包含的微生物单细胞进行原位裂解,获得包含有微生物单细胞的基因组的微腔室,将扩增体系加入含有微生物单细胞基因组的微腔室中,形成所述包含微生物单细胞基因组及扩增反应体系的微腔室;
39.ii.在所述包含微生物单细胞的微腔室内加入扩增反应体系,形成所述包含微生物单细胞及扩增反应体系的微腔室,在所述微腔室内进行对微生物单细胞进行原位裂解和扩增,形成所述包含微生物单细胞基因组及扩增反应体系的微腔室。
40.微生物单细胞的裂解操作可选自以下任一种方法但不局限于以下方法:
41.1)酶裂解法,即向微生物单细胞中加入裂解酶,进行相应的化学反应裂解细胞,释放胞内dna。裂解酶可以为但不局限于:labiase裂解酶,溶葡球菌酶,鸡蛋蛋白来源溶菌酶,人源溶菌酶,或消化肽酶等。
42.2)碱裂解法,即向微生物单细胞中加入碱,在特定碱性条件(ph>7且ph≤14)下,配合适合温度,破裂细胞,释放胞内dna。碱可以为但不局限于:koh或naoh等。
43.3)热休克法,即将微生物单细胞反复冻融,引起细胞的溶胀,致使细胞结构破碎,释放胞内dna;
44.4)超声波处理法,即利用超声加热方法,致使细胞破碎,释放胞内dna。
45.步骤(2)中,对微生物单细胞进行原位全基因组扩增,需将扩增反应体系(反应试剂)与包含微生物单细胞全基因组的微腔室融合。
46.可选的,所述扩增反应体系包封于微腔室中。
47.在一种实施方式中,将扩增反应体系与包含微生物单细胞全基因组的微腔室融合的方法可选自以下任一种方法但不局限于以下实验方法:
48.将扩增反应体系注射入包含微生物单细胞全基因组的微腔室中或将扩增反应体系与包含微生物单细胞全基因组的微腔室融合。
49.在一种实施方式中,可通过微流控液滴生成芯片将扩增反应体系注射入微液滴中或将扩增反应体系与微液滴融合。
50.在一种实施方式中,在适宜温度下进行0.01-999小时的扩增。扩增后,即可获得可用于下游实验的高质量的微生物单细胞全基因组扩增产物。
51.在一种实施方式中,步骤(2)中,扩增方法可选自以下任一种方法但不局限于以下扩增方法:
52.1)pcr(polymerase chain reaction),利用聚合酶链反应进行微生物单细胞的扩增。
53.2)mda(multiple displacement amplification),利用随机引物六聚体和聚合酶与dna模板结合并进行扩增。
54.所述聚合酶可以为φ29dna聚合酶。
55.3)malbac(multiple annealing and looping-based amplification cycles),对微生物单细胞全基因组进行多次退火环状循环扩增。
56.4)lianti(linear amplification via transposon insertion),通过插入转座子对微生物单细胞全基因组进行线性扩增。
57.对于pcr反应,可以无需酶裂解法,碱裂解法,热休克法或超声波处理法等方式进行裂解,pcr反应的变性温度相当于在包含微生物单细胞的微腔室中进行了原位裂解,即可使细胞裂解释放胞内dna,同时,关于pcr反应的引物,可以设计为对微生物单细胞内的某一特异性片段进行的扩增包含但不限制于对微生物的16s全长或者可变区的扩增,或设计为多对随机引物实现对微生物单细胞基因组内的全基因扩增。
58.如图1所示,本发明一实施例的微生物单细胞全基因组的测序文库构建方法,包括如下步骤:
59.a.将前述微生物单细胞全基因组的扩增方法获得的包含微生物单细胞全基因组扩增产物的微腔室中的扩增产物片段化,获得包含片段化扩增产物的微腔室;
60.b.将步骤a获得的微腔室中的片段化扩增产物与条形码片段在单一微腔室中连接,获得微生物单细胞全基因组的测序文库,所述测序文库中,所述微生物单细胞全基因组处于微腔室中,且每个微腔室中有且仅有单一微生物单细胞全基因组。
61.步骤(2)中,所述扩增引物选自随机引物或区域特异性引物。
62.在一种实施方式中,包含片段化扩增产物的微腔室中采用如下方法获得:使所述的微生物单细胞全基因组扩增产物的微腔室中包含有水凝胶组分,在适宜凝固的条件下,进行凝固,获得包含微生物单细胞全基因组扩增产物的水凝胶微腔室;将包含微生物单细
胞全基因组扩增产物的水凝胶微腔室与片段化反应体系融合,获得包含片段化扩增产物的水凝胶微腔室。
63.水凝胶为三维网络结构凝胶,凝胶的聚集态既非完全的固体也非完全的液体。固体的行为是一定条件下可维持一定的形状与体积,液体行为是溶质可以从水凝胶中扩散或渗透。故片段化扩增产物可负载于水凝胶的网格中,液态的扩增体系或者片段化反应体系可以渗入到水凝胶微球中与片段化扩增产物进行反应。
64.可选的,水凝胶试剂中包含丙烯酰胺单体和过硫酸铵。
65.在一种实施方式中,所述适宜凝固的条件包括但不局限于uv照射、温度改变或ph环境改变等。
66.所述水凝胶试剂的注入可以在步骤(1)-(3)任一步骤进行,能够保证获得包含片段化扩增产物的水凝胶微球即可。
67.在一种实施方式中,所述水凝胶微球选自聚丙烯酰胺水凝胶微球。
68.在一种实施方式中,可以通过微流控液滴生成芯片进行水凝胶试剂的注入。
69.在一种实施方式中,步骤a中,片段化的方法选自以下任一种但不局限于以下方法:
70.1)超声法,利用超声波,打断微生物单细胞基因组扩增产物。
71.2)离心剪切法,利用离心产生剪切力,使微生物单细胞基因组扩增产物片段化。
72.3)酶切法,包括但不局限于以下方法:
73.a.利用dsdna fragmentase,对微生物单细胞基因组扩增产物进行剪切,并使其片段化;
74.b.利用一种转座酶,该转座酶插入到微生物单细胞基因组扩增产物中,并将其片段化。
75.转座酶包括但不局限于tn5转座酶。tn5转座酶目前已商业化,可利用包含tn5转座酶的试剂盒(包括但不局限于nextera xt dna library preparation kit、truepreptm dna library prep kit v2 for illumina等)。
76.优选的,步骤b中,将包含片段化扩增产物的微腔室与包含条形码序列的微球、扩增反应体系或连接酶反应体系或杂交反应体系共包埋于同一微腔室中,使包含条形码序列的微球上的条形码脱落,进行扩增或连接或杂交反应,使所述片段化扩增产物与条形码片段连接。
77.每种条形码微球上连接的条形码序列不同。使得同一个微液滴中的包含片段化扩增产物均来自同一个单细胞,且连接相同的条形码;即不同的微液滴中的包含片段化扩增产物均来自不同的单细胞,且连接不同的条形码,使得测序时得以标记区分。
78.使包含条形码序列的微球上的条形码脱落的方法为现有技术,可以采用但不局限于化学键(例如二硫键)切割和紫外光切的方法,例如,可以加入dtt(二硫苏糖醇)使二硫键断开试剂使条形码脱落到微液滴中进行反应。
79.无需电极融合即可完成索引化。
80.连接方法可以为pcr、连接酶连接(ligation)或杂交(hybridization)等技术。
81.所述测序文库可以添加上可被测序平台识别的接头,该添加接头步骤,可以在完成索引化步骤后进行,亦可在索引化的过程中,同时添加至基因组片段上。
82.接头序列包含但不局限于illumina测序平台通用的p5和p7接头序列、自主设计的接头序列等。
83.接头序列添加完成后,需对测序文库进行大小选择和文库纯化,选择方法包括但不局限于:磁珠、凝胶电泳等方法,文库纯化方法包括但不局限于使用商用纯化试剂盒等方法。
84.文库构建及测序后,可进行可识别单个基因组的宏基因组学分析。
85.在一种实施方式中,所述微生物单细胞全基因组的测序文库构建方法的构建过程在微流控液滴生成芯片中进行。
86.本发明所述的微流控液滴生成芯片是为了能够实现微液滴或微球的融合。将实验所需制剂泵至微流控液滴生成芯片的入口,汇聚并形成层流,之后再与油相汇聚,即可形成单分散的微滴。
87.所述微流控液滴生成芯片为现有技术,例如可根据https://mccarrolllab.org/dropseq/进行构建(macosko e,basu a ,satija r,et al.highly parallel genome-wide expression profiling of individual cells using nanoliter droplets[j].cell,2015,161(5):1202-1214)。本发明的创新点在于本发明所述的方法,本领域技术人员获知本发明所述的方法后,无需创造性劳动,即可利用微流控液滴生成芯片进行操作。
[0088]
实施例1大肠杆菌和芽孢杆菌的混合样本的微生物单细胞全基因组扩增及测序文库制备将大肠杆菌(escherichia coli)和枯草芽孢杆菌(bacillus subtilis)分别置于lb(luria-bertani)培养液中培养,测量600nm的吸光值,利用细菌浓度与吸光值的关系分别推算出大肠杆菌和枯草芽孢杆菌的数目(od600=1,对应菌量为109cfu/ml)。大肠杆菌菌液和枯草芽孢杆菌菌液分别经2000g离心5min去除上清液,加入pbs缓冲液清洗后再次离心去除上清液,以无菌1xpbs溶液(ph值为7.2),使用前利用紫外照射半小时)回溶并稀释成2.4
×
107cfu/ml(即为2.4
×
107microbial single cell/ml)。将新制成的大肠杆菌菌液和枯草芽孢杆菌菌液1:1(v/v)混匀,即得实验所需的混菌悬液。
[0089]
制备裂解液:所用裂解液为repli-g single cell kit(qiagen,germany)试剂盒中所提供的裂解液。
[0090]
将混菌悬液和裂解液输入至微流控液滴生成芯片中,生成包裹有单个大肠杆菌或者单个枯草芽孢杆菌的微液滴,并将微液滴收集至1.5ml无菌离心管内。将收集到单菌微液滴,置于65℃下,培养10分钟,以实现细菌的裂解并充分释放胞内dna,此时微液滴中包含有单个大肠杆菌或单个枯草芽孢杆菌的dna。
[0091]
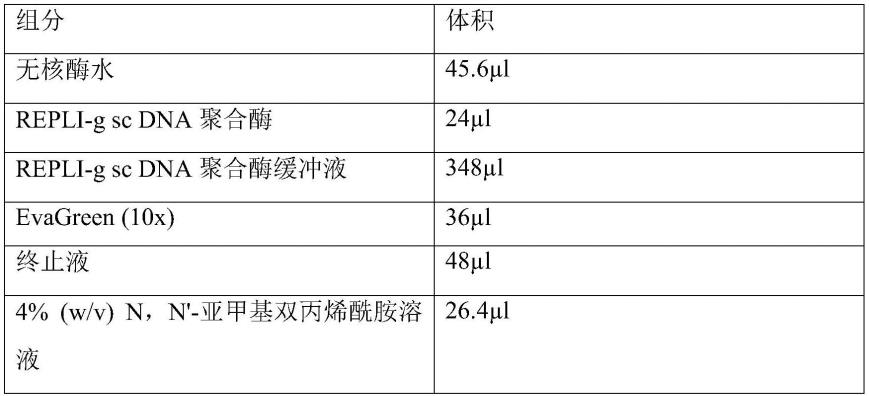
将包含有单个大肠杆菌或单个枯草芽孢杆菌dna的微液滴,与包含有mda反应试剂(配方如表1所示)的微液滴(预先通过微流控液滴生成芯片,生成包含有mda反映实际的微液滴),在电场作用下1:1融合后,生成新的微液滴(包含单个细菌的dna和mda反应试剂),并收集至1.5ml无菌离心管内,置于30℃下反应8小时后,65℃下反应15分钟终止扩增反应,即可获得包含有单菌全基因组扩增产物的微液滴(如图3所示)。
[0092]
表1 mda反应试剂配比表
[0093][0094]
将包含有单菌全基因组扩增产物的微液滴与聚丙烯酰胺水凝胶微液滴(预先通过微流控液滴生成芯片,生成水凝胶微液滴:丙烯酰胺单体的浓度为30%w/v,过硫酸铵的浓度为3.3%w/v),在电场的作用下1:1融合,并将新生成的微液滴收集于1.5ml无菌离心管内,置于室温下过夜,使其成胶,即得包含有单菌全基因组扩增产物的水凝胶微球(如图4所示)。收集到的水凝胶微球,经过破乳清洗步骤,即可进行单菌全基因组扩增产的片段化操作。
[0095]
将包含有单菌全基因组扩增产物的聚丙烯酰胺水凝胶微球加入片段化反应体系(如表2所示)中,并置于55℃下反应2小时,即可获得包含单菌全基因组扩增产物片段的聚丙烯酰胺水凝胶微球(如图5所示)。
[0096]
表2片段化反应体系
[0097][0098]
将包含有单菌全基因组扩增产物片段的水凝胶微球与包含条形码序列的微珠和pcr反应试剂的微液滴,在电场作用下1:1融合,加入dtt试剂,使得条形码序列从条形码序列的微珠上脱落到微液滴中,通过pcr反应(反应体系如表3所示,反应条件如表4所示)完成单菌全基因组扩增产物片段的索引化步骤。在此索引化步骤中,已为基因组片段添加上illumina测序平台可识别的p5和p7接头,因此,测序文库构建已完成。
[0099]
条形码序列的微珠的制备方法为:通过applied biosystems,3900dna合成仪对微球表面进行修饰,合成所需要的条形码序列。
[0100]
表3索引化反应体系
[0101]
组分体积
2%sds2μlenhancer(4x)25μlstabilizer(5x)20μlevagreen(20x)10μlprimer fl1276μlbarcode-15-3-666μltae20μl无核酶水9μl
[0102]
表4索引化pcr反应条件
[0103][0104]
对构建完成的测序文库利用vahts dna clean beads(诺唯赞)进行大小筛选和纯化,根据生产商提供的实验流程,筛选出大小在200-600bp范围内的文库并将其纯化,利用bioanalyzer对测序文库进行质量控制(图6),确定无误后,该纯化后的测序文库,即可进行二代测序。文库由解码(上海)生物医药科技有限公司(以下称“解码”)利用dnbseq-t7平台进行双端150bp测序。
[0105]
测序完成后,从解码的dnbseq-t7平台获得的原始数据fastq文件,首先对其进行质控去除测序质量低于q20的数据,再根据索引序列将质控后的测序数据利用bbtools程序进行分组。理论上,每一个索引序列代表单个细胞。索引序列与对应reads数量的分布如图7所示。分组后,利用bowtie2 v2.4.4分别对每组的测序数据参照大肠杆菌和枯草芽孢杆菌基因组进行比对,用krona可视化kraken的比对结果。
[0106]
比对结果如图8,图8中的结果表示每个液滴里面就有且只有一个细菌基因组的信息。结果表明,本发明方法获得的测序数据基因组片段丢失量低,准确率高。
[0107]
以上的实施例是为了说明本发明公开的实施方案,并不能理解为对本发明的限制。此外,本文所列出的各种修改以及发明中方法、组合物的变化,在不脱离本发明的范围和精神的前提下对本领域内的技术人员来说是显而易见的。虽然已结合本发明的多种具体优选实施例对本发明进行了具体的描述,但应当理解,本发明不应仅限于这些具体实施例。事实上,各种如上所述的对本领域内的技术人员来说显而易见的修改来获取发明都应包括在本发明的范围内。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。
