新型冠状病毒cd8 t细胞表位肽及其用途
技术领域
1.本发明属于生物技术领域,并且特别涉及用于新型多肽疫苗的研发的新型冠状病毒cd8 t细胞表位肽及其用途。
背景技术:
[0002][0003]
t
‑
scan利用基于报告细胞的抗原肽表达文库与人的cd8
t细胞孵育,以鉴定可以无偏见地激活cd8
t细胞的多肽。报告细胞(edc,抗原肽呈递细胞)过表达hla亚型和感兴趣病原体蛋白质组中的表位多肽。此外,edc还含有颗粒酶b(gzb)的表达框和修饰的红外线荧光蛋白(ifp)序列,该蛋白仅在被gzb切割后才发出荧光。当呈现相关表位的edc激活cd8
t细胞时,引起gzb分泌以诱导相关edc凋亡,该 edc中修饰的ifp将被裂解从而激活荧光信号。基于流式细胞仪对ifp 阳性的edc进行分选,然后进行下一代测序,可以鉴定分选出edc中富集的表位。
技术实现要素:
[0004]
本发明从中国汉族人群covid
‑
19康复患者的四种流行hla亚型 (hla
‑
a*02:01、hla
‑
a*02:07、hla
‑
a*11:01和hla
‑
a*24:02)中鉴定了约49个表位肽。验证了人类冠状病毒中保守的几种新型表位,进一步支持了预存的细胞免疫。重要的是,本发明验证了在新兴的英国菌株、南非菌株或巴西菌株中突变的三种表位肽,同时发现这些突变导致表位与hla复合物结合的能力下降。我们的数据提供了新近出现的sars
‑
cov
‑
2变种逃避细胞免疫的证据,这可能有助于增加这些变种的传播性和致病性。
[0005]
具体地,本发明提供了以下技术方案:
[0006]
一方面,本发明提供新型冠状病毒cd8 t细胞表位肽,其特征在于,其氨基酸序列如seq id no:1
‑
49中任一项所示或与根据seq idno:1
‑
49中任一项所示序列相比具有一个或多个(优选2个或3个)保守氨基酸突变(优选置换、插入或缺失)的氨基酸序列。
[0007]
另一方面,本发明提供编码多肽表位的核酸序列,其编码上述的新型冠状病毒cd8 t细胞表位肽。
[0008]
另一方面,本发明提供重组载体,其包含上述的编码多肽表位的核酸序列。
[0009]
在一些实施方案中,所述重组载体包括但不限于腺病毒载体、腺相关病毒载体、慢病毒载体和痘病毒载体。
[0010]
在一些实施方案中,所述重组载体选自腺病毒载体、腺相关病毒载体、慢病毒载体和痘病毒载体。
[0011]
另一方面,本发明提供重组蛋白,其包含上述的新型冠状病毒cd8 t细胞表位肽。
[0012]
在一些实施方案中,所述重组蛋白的50%以上的序列为上述的新型冠状病毒cd8 t细胞表位肽。
[0013]
另一方面,本发明提供宿主细胞,其包含上述的编码多肽表位的核酸序列或导入
了上述的重组载体。
[0014]
在一些实施方案中,所述宿主细胞选自来自个体的任何原代细胞,优选hek293t细胞。
[0015]
另一方面,本发明提供疫苗组合物或试剂盒或免疫原性组合物,其包含上述的新型冠状病毒cd8 t细胞表位肽或上述的编码多肽表位的核酸序列或上述的重组载体,和药学上可接受的载体和/或辅料。
[0016]
另一方面,本发明提供抗体或抗原结合片段,其由上述的新型冠状病毒cd8 t细胞表位肽制备而成。
[0017]
另一方面,本发明提供pmhc复合物,其包含上述的新型冠状病毒 cd8 t细胞表位肽。
[0018]
另一方面,本发明提供药物组合物,其包含上述的抗体或抗原结合片段,和药学上可接受的载体和/或辅料。
[0019]
另一方面,本发明提供上述的新型冠状病毒cd8 t细胞表位肽或上述的编码多肽表位的核酸序列或上述的重组载体或上述的重组蛋白或上述的宿主细胞在制备新型冠状病毒疫苗或抗体或检测试剂中的用途。
[0020]
另一方面,本发明提供诱导对新型冠状病毒的免疫应答的方法,其包括施用有效量的上述的新型冠状病毒cd8 t细胞表位肽或上述的编码多肽表位的核酸序列或上述的重组载体或上述的重组蛋白,所述方法用于非诊断或治疗目的。
[0021]
另一方面,本发明提供刺激、引发和/或扩增cd8 t细胞的(优选体外)方法,其包括施用有效量的上述的新型冠状病毒cd8 t细胞表位肽或上述的编码多肽表位的核酸序列或上述的重组载体或上述的重组蛋白,所述方法用于非诊断或治疗目的。
[0022]
有益效果
[0023]
本发明将t
‑
scan技术应用于sars
‑
cov
‑
2的表位筛选中,验证了人类冠状病毒中保守的几种新型表位,进一步支持了预存的细胞免疫。这些序列为今后新冠多肽疫苗研发提供基础。
[0024]
本发明发现的新冠病毒t细胞表位,增进了对病毒与人类宿主相互作用机制的认识。这些序列信息可以被用于诊断试剂盒开发和新型治疗药物研发。
附图说明
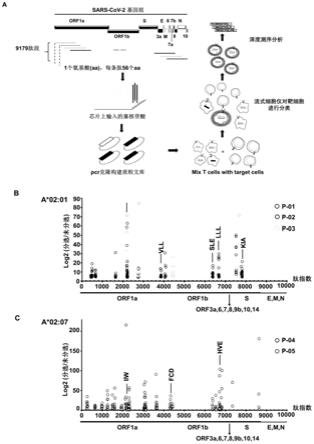
[0025]
图1示出通过t
‑
scan方法对covid
‑
19康复期患者的cd8
t细胞表位进行分析。(a)整个编码区范围内的sars cov2抗原库的设计以及t
‑
scan表位鉴定筛选的概述图。我们设计并合成了9179条多肽序列,覆盖sars
‑
cov
‑
2全基因组文库。通过分子克隆及慢病毒感染的方法构建了稳定呈递多肽文库的靶细胞文库,选用细胞为293t细胞。将构建的靶细胞文库和covid
‑
19康复者的cd8 t细胞共孵育12小时,被cd8 t 细胞识别的靶细胞报告基因被激活。用流式细胞仪来筛选靶细胞,提取基因组dna,通过深度测序的方法来分析富集的靶细胞所呈递的抗原表位序列。(b
‑
e)10名covid
‑
19康复期(三例为hla
‑
a*02:01基因型,两例为hla
‑
a*02:07基因型,三例为hla
‑
a*11:01基因型,两例为hla
‑
a*24:02基因型)的t
‑
scan筛选数据,x轴显示出设计的编码区范围的sars
‑
cov2抗原库的肽指数,y轴代表与相应的未分选edc库相比,分选靶细胞中每种肽的富集倍数。每个圆圈代表在t
‑
scan筛选中被显
著富集的sars
‑
cov2开放阅读框的56个氨基酸的肽段。基于肽富集指数(y轴>4)和netmhc 4.0服务器预测评分 (http://www.cbs.dtu.dk/services/netmhc/),我们鉴定了49条高置信度表位 (seq id no:1
‑
49)。
[0026]
图2示出鉴定出的cd8
t细胞保守表位的验证。(a)不同冠状病毒株之间鉴定的保守cd8 t细胞表位的比对。比对表明,所鉴定的保守表位与sars
‑
cov和其他四个先前存在的导致感冒的人类冠状病毒 (oc43、hku1、nl63、229e)具有高度同源性。从比对图可看出,序列同源性高达近80%(相同氨基酸数/总氨基酸数=7/9)。(b)对vll (seq id no:2)、sle(seq id no:3)、lll(seq id no:4)三个表位的mhc四聚体染色验证,这些表位在covid
‑
19恢复期和健康人对照中既存的人冠状病毒之间是保守的,因此可能介导针对新冠的预存免疫。我们体外纯化了携带apc荧光标签的mhc四聚体,分别能够呈递vll (seq id no:2)、sle(seq id no:3)、lll(seq id no:4)多肽。通过将mhc
‑
多肽四聚体和pbmc孵育,我们可以检测到能够特异性识别多肽的cd8 t细胞的比例。如covid
‑
19康复者p
‑
02的pbmc里面分别有0.028%、0.025%、0.022%的cd8 t细胞分别识别vll、sle、 lll多肽。h
‑
01和h
‑
02是健康志愿者,可以看到来自健康志愿者h
‑
01 的pbmc里面也有一部分cd8 t细胞可以识别vll、sle、lll多肽,比例分别是0.011%、0.012%、8.8e
‑
3%,说明了这些保守性高的多肽可能帮助建立了covid
‑
19的预存免疫。健康志愿者h
‑
02的pbmc中识别vll、sle、lll的比例较低甚至检测不到,反映出预存免疫在人群中的差异性。(c)hla
‑
a*02:01基因型对应的2条抗原表位[sle(seqid no:3),lll(seq id no:4)]和hla
‑
a*02:07基因型对应的2条抗原表位[fcd(seq id no:17),hve(seq id no:18)]的胞质内ifn
‑
γ细胞因子染色验证。将来自covid
‑
19康复者或者健康志愿者的pbmc 细胞和多肽共孵育5小时后,检测胞质内ifn
‑
γ的含量。x轴代表ifn
‑
γ的表达,y轴代表cd8的表达,可以看到和未处理组相比,加入多肽后表达ifn
‑
γ的cd8 t细胞的比例上升(第二象限),说明多肽刺激相应的cd8 t细胞的应答。比如covid
‑
19康复者p
‑
12的pbmc,未处理组cd8 t细胞能够分泌ifn
‑
γ的细胞比例是0,加入sle(seq idno:3)或者lll(seq id no:4)多肽后,分泌ifn
‑
γ产生应答的cd8 t细胞分别有0.39%和0.16%。p
‑
07为covid
‑
19康复者,h
‑
03为健康志愿者,加入多肽fcd(seq id no:17)或者hve(seq id no:18) 后,来自p
‑
07的pbmc分别有0.20%和0.47%的cd8 t细胞分泌ifn
‑ꢀ
γ,比未处理组高(0.062%);来自h
‑
03的pbmc分别有0.93%和0.58%的cd8 t细胞分泌ifn
‑
γ,fcd组比未处理组高(0.58),hve组和未处理组持平。(d)hla
‑
a*11:01基因型yic(seq id no:36)和sqd (seq id no:37)两条表位的胞质内ifn
‑
γ细胞因子染色验证。在康复患者p
‑
14中,与未处理组相比,加入yic(seq id no:36)和sqd (seq id no:37)多肽后表达ifn
‑
γ的cd8 t细胞的比例有所增加上升,阳性百分比分别由3.88e
‑
3升高为0.034%和0.065%,说明表位肽能刺激cd8 t细胞的活化。同时在健康人h
‑
04中,与未处理组相比,加入yic(seq id no:36)多肽后表达ifn
‑
γ的cd8 t细胞的阳性百分比由0.053%上升高为0.16%,进一步证实了yic(seq id no:36)多肽的保守性,表明健康人体内有预先免疫存在的可能性。
[0027]
图3示出包含sars
‑
cov
‑
2变异株中突变位点的cd8 t细胞表位的验证。(a)三个含有全球流行的sars
‑
cov
‑
2变种的代表性突变位点 (y145
‑
、k417/n或k417/t、i2230w)的表位[gvy(seq id no:49)、 kia(seq id no:48)、iiw(seq id no:1)],这些突变可能影响了新的病毒变种的感染力和传播力。(b)胞质内ifn
‑
γ细胞因子染色的方法验证抗原表位:
iiwflllsv(iiw)(seq id no:1)。来自covid
‑
19康复者p
‑
11和p
‑
12的pbmc,加入iiw(seq id no:1)多肽后,分泌ifn
‑ꢀ
γ产生应答的cd8 t细胞分别有0.33%和0.40%,比未处理组cd8 t细胞能够分泌ifn
‑
γ的细胞比例高(分别是0.11%和0.05%),来自covid
‑ꢀ
19康复者p
‑
04和p
‑
20的pbmc,加入iiw(seq id no:1)多肽后,分泌ifn
‑
γ产生应答的cd8 t细胞分别有0.11%和0.63%,比未处理组 cd8 t细胞能够分泌ifn
‑
γ的细胞比例高(分别是0.02%和0.06%)。 (c
‑
d)mhc四聚体染色和ifn
‑
γ胞质内染色的方法验证抗原表位: kiadynykl(kia)(seq id no:48)。我们体外纯化了携带apc荧光标签的mhc四聚体,呈递kia多肽。通过将mhc
‑
kia四聚体和 pbmc孵育,我们可以检测到能够特异性识别kia的cd8 t细胞的比例。如covid
‑
19康复者p
‑
01、p
‑
09、p
‑
10、p
‑
11、p
‑
12的pbmc里面分别有5.5e
‑
3%、0.048%、6.2e
‑
3%、4.8e
‑
3%、7.3e
‑
3%的cd8 t细胞能够识别kia。(e)ifn
‑
γ胞质内染色的方法验证的结果与mhc四聚体染色检测的验证结果呈正相关,验证的抗原表位:gvyyhknnk(gvy) (seq id no:49)。在新冠康复病人p
‑
07、p
‑
13、p
‑
15、p
‑
16、p
‑
17、 p
‑
18、p
‑
19中,使用pearson相关计算四聚体染色阳性比例和ifn
‑
γ阳性比例的相关系数r,横坐标为gvy(seq id no:49)四聚体染色阳性细胞的比例,纵坐标为ifn
‑
γ阳性比例,从图中可看出二者具有正相关性。(f
‑
i)抗原表位携带的点突变对cd8 t细胞抗原响应能力的影响。vaccinated donors为接种sars
‑
cov
‑
2疫苗(coronavac)第一针后2
‑ꢀ
3周的志愿者。可以看到接种了疫苗的志愿者产生了对kia seq idno:48)、iiw(seq id no:1)、gvy seq id no:49)、nyn(seqid no:47)多肽响应的cd8 t细胞,而且这些cd8 t细胞对携带点突变的mut kia、mut iiw、mut gvy、mut nyn应答能力减弱,提示这些点突变可能造成了cd8 t细胞介导的细胞免疫逃逸。
[0028]
图4.hla
‑
a*02:01与kia肽复合物和hlaa*24:02与nyn肽复合物的x射线晶体结构。(a
‑
b)显示hla
‑
a*02:01和kia表位之间相互作用的结构概述(a)和电子密度图(b),显示kia表位和hla
‑‑
a*02:01 w167相互作用的电子云密度,表明两者之间有很强的相互作用。如果将 k突变为n/t则可能会破坏这种强相互作用。(c
‑
d)hla
‑
a*24:02和 nyn表位的结构概述和相应的电子密度图(d),从图中可看出l452的疏水侧链埋在hla
‑
a*24:02表面的一个口袋中。(e
‑
f)当与kia/gvy肽或相应的突变种肽一起孵育时,纯化a*02:01
‑
β2m
‑
肽复合物和a*11:01
‑ꢀ
β2m
‑
肽复合物的色谱图,横坐标表示洗脱体积,纵坐标表示od
280
的紫外吸收值。图中显示突变的多肽无法有效结合hla分子形成四聚体复合物,即突变体蛋白在分子筛约85ml处无明显蛋白被洗脱下来。(g)来自t
‑
scan分析的4个已鉴定表位的总结,其中包含最近流行的sars
‑ꢀ
cov
‑
2变体中的突变位点。%rank和结合强度由netmhc4.0预测。强结合%rank<=0.5,弱结合0.5%<rank<=2。
具体实施方式
[0029]
为使本发明的目的、技术方案和优点更加清楚明白,以下结合具体实施例,并参照附图,对本发明作进一步的详细说明。
[0030]
本发明中所涉及的所有健康人或covid
‑
19康复患者均签署了知情同意书。
[0031]
实施例1:细胞系
[0032]
在含有10%(v/v)fbs,100单位/ml青霉素和0.1mg/ml链霉素的 dmem(life technologies)中培养hek293t细胞。在含有10%(v/v) fbs,100单位/ml青霉素,0.1mg/ml
链霉素和60u/mlil
‑
2(sigma)的rpmi(lifetechnologies)中培养pbmc细胞(外周血单核细胞)。
[0033]
实施例2:外周血单核细胞的分离及扩增
[0034]
edta抗凝管用于收集康复患者和正常人的血液。使用密度为1.077g/ml的ficoll分层溶液用于分离外周血单核细胞(pbmc)。在室温下,将3ml全血轻轻加入等体积的ficoll分层溶液的15ml锥形管中。400g离心30分钟。从ficoll
‑
血浆界面小心吸出含有pbmc的浑浊相,转移至15ml无菌离心管中,加满dmem培养基,500g离心15min,弃上清。1mldmem重悬细胞团块,取出10微升细胞并在显微镜下计数。最后,用新赛美无血清细胞冷冻保存液(苏州新赛美生物科技有限公司,catno.c40100)以107个细胞每毫升重悬细胞,并保存在
‑
80℃冰箱中。复苏细胞,在24孔板的每个孔中播种100万个pbmc,用dynabeadshumant激活剂cd3/cd28(lifetechnologies)按照说明书进行刺激。使用cd8
记忆t细胞分离试剂盒(milenyi,130
‑
094
‑
412)按说明书从pmbc细胞中分离cd8
记忆t细胞。为了扩增,将cd8
记忆t细胞培养在rpmi加入10%(v/v)fbs,100单位/ml青霉素(lifetechnologies),0.1mg/ml链霉素(lifetechnologies),60u/mlil
‑
2(sigma)和0.1μg/ml抗cd3抗体(okt3,ebioscience)中。
[0035]
实施例3:慢病毒生产
[0036]
慢病毒是通过转染hek293t细胞产生的。对于6孔细胞板,使用neofecttmdna转染试剂(catno.tf201201)将1.5μg慢病毒转移载体,1.5μgpax2质粒(addgene,#35002)和1μgpmd2.g质粒(addgene,#12259)共转染到6孔细胞板的hek293t细胞中。转染后48h收集慢病毒上清液,并通过0.30μm的滤膜过滤。为了建立细胞系,将慢病毒上清液在8μg/ml聚丙烯存在下添加到细胞中。
[0037]
实施例4:sars
‑
cov
‑
2肽库的设计和生成
[0038]
为了产生一套完整的潜在sars
‑
cov
‑
2抗原,我们在每个orf中平铺了56个氨基酸的片段,其中55个氨基酸重叠在相邻的肽段之间,共有9179个肽片段。使用非稀有人类密码子将肽段反向翻译。每个片段由一个核苷酸序列编码。
[0039]
用以下引物扩增文库,所述引物包含编码bp重组位点的突出端(小写为载体部分序列,大写为所扩增文库的引物序列):
[0040]
cmv_bpf:
[0041]5’
ggggacaagtttgtacaaaaaagcaggctcaggaattctgagctcg(seqidno:50)
[0042]
cmv_bpr:
[0043]5’
ggggaccactttgtacaagaaagctgggtcagctagttaagctcgagcacccga(seqidno:51)
[0044]
使用bp克隆酶(thermo)将扩增的文库克隆到pdonr221载体(gateway
tm
pdonr
tm
221载体购自invitrogen,货号为12536017)中,然后使用gateway克隆将其转染到phagecmvnflaghadestires慢病毒载体(由哈佛大学stephenj.elledge教授的实验室提供,参考文献kulaetal.cell2019)中。在所有克隆步骤中均保持150倍的文库覆盖率。
[0045]
建立慢病毒表达文库,并以0.5的moi感染携带报告系统的293t细胞,该报告系统含有能稳定表达phagecmvicadcr,phagecmvifpgzb和phagecmvhla的载体(由哈佛大
学stephen j.elledge教授的实验室提供,参考文献kula et al.cell 2019),载体上通过分子克隆引入hla序列,分别是hla
‑
a*02:01(seq id no:53),hla
‑
a*02:07 (seq id no:55),hla
‑
a*11:01(seq id no:54)和hla
‑
a*24:02 (seq id no:56),每种hla质粒都建立一个细胞文库。转导后48小时开始,以1μg/ml嘌呤霉素选择细胞3天。
[0046]
实施例5:sars
‑
cov
‑
2多肽筛选
[0047]
对于每种hla基因型,将表达sars
‑
cov
‑
2文库的2000万个 hek293t细胞(2100x库表示)与恢复期的covid
‑
19患者或健康人的 400万个hla基因型匹配记忆cd8
t细胞共培养12h,之后通过流式细胞仪(bd,influx)对那些ifpgzb阳性靶细胞进行了分选。提取收集的靶细胞的基因组dna,并将其用于制备深度测序样品,并通过测序比对鉴定多肽表位的富集程度。然后对筛选得到的多肽序列在冠状病毒中进行序列比对,发现有一些多肽较为保守,比对结果如图2a所示,从比对图可看出,这些序列与其他冠状病毒的同源性高达近80%(相同氨基酸数/总氨基酸数=7/9)。
[0048]
结果
[0049]
基于肽富集指数(y轴>4)和netmhc 4.0服务器预测评分 (http://www.cbs.dtu.dk/services/netmhc/),我们鉴定了49条高置信度表位 (seq id no:1
‑
49)。
[0050]
筛选到的表位序列如下(括号内为多肽名称,即前三个氨基酸的排列):
[0051]
1.iiwflllsv(iiw)(seq id no:1)
[0052]
2.vllsvlqql(vll)(seq id no:2)
[0053]
3.slenvafnv(sle)(seq id no:3)
[0054]
4.lllddfvei(lll)(seq id no:4)
[0055]
5.llddfveii(lld)(seq id no:5)
[0056]
6.tmadlvyal(tma)(seq id no:6)
[0057]
7.fvaaifyli(fva)(seq id no:7)
[0058]
8.aifylitpv(aif)(seq id no:8)
[0059]
9.ylitpvhvm(yli)(seq id no:9)
[0060]
10.klneeiaii(kln)(seq id no:10)
[0061]
11.flalcadsi(fla)(seq id no:11)
[0062]
12.sliystaal(sli)(seq id no:12)
[0063]
13.ylatalltl(yla)(seq id no:13)
[0064]
14.ilsplyafa(ils)(seq id no:14)
[0065]
15.klvnkflal(klv)(seq id no:15)
[0066]
16.alweiqqvv(alw)(seq id no:16)
[0067]
17.fcdlkgkyv(fcd)(seq id no:17)
[0068]
18.hvetfypkl(hve)(seq id no:18)
[0069]
19.wlptgtllv(wlp)(seq id no:19)
[0070]
20.kldgfmgri(kld)(seq id no:20)
[0071]
21.ympyfftll(ymp)(seq id no:21)
[0072]
22.qlffsyfav(qlf)(seq id no:22)
[0073]
23.llflmsftv(llf)(seq id no:23)
[0074]
24.flpgvysvi(flp)(seq id no:24)
[0075]
25.ylyltfylt(yly)(seq id no:25)
[0076]
26.flahiqwmv(fla)(seq id no:26)
[0077]
27.wmvmftplv(wmv)(seq id no:27)
[0078]
28.fleyhdvrv(fle)(seq id no:28)
[0079]
29.qlemeltpv(qle)(seq id no:29)
[0080]
30.clhvvgpnv(clh)(seq id no:30)
[0081]
31.flkkdapyi(flk)(seq id no:31)
[0082]
32.wldmvdtsl(wld)(seq id no:32)
[0083]
33.nllkdcpav(nll)(seq id no:33)
[0084]
34.svkglqpsv(svk)(seq id no:34)
[0085]
35.elpdefvvv(elp)(seq id no:35)
[0086]
36.yicgfiqqk(yic)(seq id no:36)
[0087]
37.sqdlsvvsk(sqd)(seq id no:37)
[0088]
38.vvcteidpk(vvc)(seq id no:38)
[0089]
39.asmpttiak(asm)(seq id no:39)
[0090]
40.ttiakntvk(tti)(seq id no:40)
[0091]
41.yilftrffy(yil)(seq id no:41)
[0092]
42.qvvnvvttk(qvv)(seq id no:42)
[0093]
43.vvttkialk(vvt)(seq id no:43)
[0094]
44.alctfllnk(alc)(seq id no:44)
[0095]
45.aimqlffsy(aim)(seq id no:45)
[0096]
46.lliimrtfk(lli)(seq id no:46)
[0097]
47.nynylyrlf(nyn)(seq id no:47)
[0098]
48.kiadynykl(kia)(seq id no:48)
[0099]
49.gvyyhknnk(gvy)(seq id no:49)
[0100]
其中seq id no:1的多肽预测表明同时结合hla
‑
a*02:01和hla
‑ꢀ
a*02:07,seq id no:2
‑
16和seq id no:48的多肽对应hla
‑ꢀ
a*02:01,seq id no:17
‑
35的多肽对应hla
‑
a*02:07,seq id no:36
‑ꢀ
46和seq id no:49的多肽对应hla
‑
a*11:01,seq id no:47的多肽对应hla
‑
a*24:02。
[0101]
s蛋白中的一个表位iiwflllsv(iiw)发生在三种变种中的一种以上。该表位在经过验证的表位列表中,在康复期引发cd8
t细胞反应。我们的数据支持这种突变可能提供逃避细胞免疫的额外优势。此外,我们的分析已经确定了几个在人类冠状病毒中高度保守的表位,特别是在orf1a和orf1b中。这些表位可以支持人们预先存在的免疫力,部分解释了人群之间疾病严重程度的差异。重要的是,包含这些表位序列的疫苗可能因此引发广泛的抗病毒细胞反应。
[0102]
实施例6:胞浆内细胞因子染色(ics)来测量cd8 t细胞的激活
[0103]
pbmc在含有20%fbs,100单位/ml青霉素和0.1mg/ml链霉素的 rpmi中培养。解冻后,在60u/ml il
‑
2和0.5μg/ml抗cd28存在下,将pbmc于37℃下用10ug/ml的表位多肽处理3
天,然后进行胞质内细胞因子抗体染色。
[0104]
鉴于sars
‑
cov
‑
2变异的日益流行和关注,我们重点研究了在分析中确定的在b.1.1.7、b.1.351和p.1变异中突变的四个表位。对于ics测试,每孔在带有v型底部的96孔细胞板中接种了20万个pbmc。将候选肽(5μg/ml)添加至pbmc,并在10μg/ml布雷菲德菌素a(bdbiosciences)存在下于37℃刺激5h。加入150μlpbs洗涤细胞,以1000g离心,5分钟后,用live/dead可固定死细胞染色试剂盒(biolegend,catno.423105)和抗cd3(biolegend,brilliantviolet510),抗cd4(biolegend,percp/cyanine5),抗cd8(bdpharmingen,alexafluor647)抗体染色细胞。在4℃放置30分钟。然后用pbs洗涤细胞两次,并在4℃下每孔加入100ulbd固定/通透溶液(bdcytofix/cytoperm,目录号554714)。然后用1x透化缓冲液(bdcytofix/cytoperm,目录号554714)洗涤细胞,并在室温下用抗ifn
‑
γ(biolegend,pe)染色细胞15分钟。用1x通透缓冲液洗涤细胞两次,并用pbs重悬细胞,然后通过流式细胞仪(bdlsrfortessasorp)进行分析。
[0105]
结果
[0106]
对于b.1.1.7、b.1.351和p.1三种突变株而言,gvy(seqidno:49)在b.1.1.7、b.1.351中存在突变位点;kia(seqidno:48)在b.1.1.7、b.1.351和p.1三种突变中均有涉及;nyn(seqidno:47)和iiw(seqidno:1)在b.1.1.7中都存在突变位点(图3a)。胞质内ifn
‑
γ细胞因子染色的方法验证抗原表位:iiwflllsv(iiw)(seqidno:1)显示,来自covid
‑
19康复者p
‑
11和p
‑
12的pbmc,加入iiw(seqidno:1)多肽后,分泌ifn
‑
γ产生应答的cd8 t细胞分别有0.33%和0.40%,比未处理组cd8 t细胞能够分泌ifn
‑
γ的细胞比例高(分别是0.11%和0.05%),来自covid
‑
19康复者p
‑
04和p
‑
20的pbmc,加入iiw(seqidno:1)多肽后,分泌ifn
‑
γ产生应答的cd8 t细胞分别有0.11%和0.63%,比未处理组cd8 t细胞能够分泌ifn
‑
γ的细胞比例高(分别是0.02%和0.06%)(图3b)。
[0107]
hla
‑
a*02:01基因型对应的2条抗原表位[sle(seqidno:3),lll(seqidno:4)]和hla
‑
a*02:07基因型对应的2条抗原表位[fcd(seqidno:17),hve(seqidno:18)]的胞质内ifn
‑
γ细胞因子染色验证结构显示,将来自covid
‑
19康复者或者健康志愿者的pbmc细胞和多肽共孵育5小时后,检测胞质内ifn
‑
γ的含量;x轴代表ifn
‑
γ的表达,y轴代表cd8的表达,可以看到和未处理组相比,加入多肽后表达ifn
‑
γ的cd8 t细胞的比例上升(第二象限),说明多肽刺激相应的cd8 t细胞的应答;covid
‑
19康复者p
‑
12的pbmc,未处理组cd8 t细胞能够分泌ifn
‑
γ的细胞比例是0,加入sle(seqidno:3)或者lll(seqidno:4)多肽后,分泌ifn
‑
γ产生应答的cd8 t细胞分别有0.39%和0.16%;p
‑
07为covid
‑
19康复者,h
‑
03为健康志愿者,加入多肽fcd(seqidno:17)或者hve(seqidno:18)后,来自p
‑
07的pbmc分别有0.20%和0.47%的cd8 t细胞分泌ifn
‑
γ,比未处理组高(0.062%);来自h
‑
03的pbmc分别有0.93%和0.58%的cd8 t细胞分泌ifn
‑
γ,fcd组比未处理组高(0.58),hve组和未处理组持平(图2c)。
[0108]
hla
‑
a*11:01基因型yic(seqidno:36)和sqd(seqidno:37)两条表位的胞质内ifn
‑
γ细胞因子染色验证结果显示,在康复患者p
‑
14中,与未处理组相比,加入yic(seqidno:36)和sqd(seqidno:37)多肽后表达ifn
‑
γ的cd8 t细胞的比例有所增加上升,阳性百分比分别由3.88e
‑
3升高为0.034%和0.065%,说明表位肽能刺激cd8 t细胞的活化。同时在健康人h
‑
04中,与未处理组相比,加入yic(seqidno:36)多肽后表达ifn
‑
γ的cd8
t细胞的阳性百分比由 0.053%上升高为0.16%,进一步证实了yic(seq id no:36)多肽的保守性,表明健康人体内有预先免疫存在的可能性(图2d)。
[0109]
实施例7:phla四聚体的制备
[0110]
编码生物素亲和蛋白的序列(bap:glndifeaqkiewh)(seq idno:52)通过gs接头通过pcr连接到i类hla
‑
a*α链的胞外段(残基 25
‑
300)的c末端。将扩增的pcr产物在具有sal i和not i限制性位点的t7启动子的控制下克隆到由实验室经pet30a(novagen,货号 69909,序列请见https://www.addgene.org/vector
‑
database/2576/)改造的 v1载体中(sal
‑
insert
‑
noti
‑
his6
‑
stop,t7 promoter)。α链以包涵体的形式在大肠杆菌中表达,并且使用镍亲和层析通过6
×
his进一步纯化溶于8 m尿素中的变性α蛋白。同时,重组蛋白β2m
‑
mbp(麦芽糖结合蛋白) 在大肠杆菌中表达,通过用tev酶消化并进一步纯化获得可溶性β2m蛋白。为了重折叠,将3mgβ2m加入200ml冷重折叠缓冲液中(5mm还原l
‑
谷胱甘肽、5mm氧化l
‑
谷胱甘肽、2mm edta、5%甘油、1%叠氮化钠、0.4ml
‑
精氨酸hcl,0.1m tris
‑
hcl,ph 8.0),并在4℃下搅拌 2小时。接下来,将2mg表位肽和12mgα链的混合物滴到重折叠缓冲液中,并在4℃下温育72h。用浓缩管(截留分子量,30kda)将重折叠的蛋白质混合物浓缩至5ml体积,然后通过120ml superdex
tm 200 16/600色谱柱(cytiva hiload)进行纯化。在120ml凝胶过滤中,约85 ml洗脱出phla单体部分。使用bira生物素试剂盒(bira
‑
500, avidity)获得生物素化的phla复合物,并通过24ml superdex 200 10/30色谱柱(ge healthcare)进一步纯化。然后将生物素化的phla复合物与apc偶联的链霉亲和素(bioss,cat no.bs
‑
0437p
‑
apc)偶联以形成四聚体,该四聚体用于对covid
‑
19患者的pbmc进行染色。
[0111]
实施例8:mhc四聚体染色法
[0112]
解冻pbmc细胞团或分离的记忆cd8
t细胞,用预热的rpmi
‑
10% fbs培养基洗涤,并以100万个细胞/孔的量接种于v型底部96孔板中。沉淀细胞并用含1%fbs的pbs和相应的四聚体重悬,终浓度为10 μg/ml,并在37℃孵育15分钟,然后添加fitc偶联的抗人cd8抗体 (biolegend,cat no.344703)和live/dead可固定死细胞染色试剂盒 (biolegend,cat no.423105),并在室温下再孵育15分钟。将染色的细胞沉淀并洗涤3次,然后通过流式细胞仪(bd lsrfortessa sorp)进行分析。
[0113]
结果
[0114]
对vll(seq id no:2)、sle(seq id no:3)、lll(seq idno:4)三个表位进行mhc四聚体染色验证,这些表位在covid
‑
19恢复期和健康人对照中既存的人冠状病毒之间是保守的,因此可能介导针对新冠的预存免疫。我们体外纯化了携带apc荧光标签的mhc四聚体,分别能够呈递vll(seq id no:2)、sle(seq id no:3)、lll (seq id no:4)多肽。通过将mhc
‑
多肽四聚体和pbmc孵育,我们可以检测到能够特异性识别多肽的cd8 t细胞的比例。covid
‑
19康复者p
‑
02的pbmc里面分别有0.028%、0.025%、0.022%的cd8 t细胞分别识别vll(seq id no:2)、sle(seq id no:3)、lll(seq id no:4) 多肽。h
‑
01和h
‑
02是健康志愿者,可以看到来自健康志愿者h
‑
01的 pbmc里面也有一部分cd8 t细胞可以识别vll(seq id no:2)、 sle(seq id no:3)、lll(seq id no:4)多肽,比例分别是0.011%、 0.012%、8.8e
‑
3%,说明了这些保守性高的多肽可能帮助建立了covid
‑ꢀ
19的预存免疫。健康志愿者h
‑
02的pbmc中识别vll(seq idno:2)、sle(seq id no:3)、lll(seq id no:4)的比例较低甚至检测不到,反映出预存免疫在人群中的差异性。
[0115]
实施例9:复合物的结晶、数据收集和结构测定
[0116]
hla
‑
a*02:01
‑
kia复合物在含有26%peg 4000,0.2m乙酸钠和 0.1m tris
‑
hcl[ph 9.0]的溶液中结晶。hla
‑
a*24:02
‑
nyn复合物在含有 0.1m hepes 8.1和22%peg 8000的溶液中结晶。分别在储液中加入 10%和18%的甘油(v/v)保护晶体,用于x射线衍射数据收集。衍射数据是在上海同步辐射装置(ssrf)的19u处采集的。数据用xds处理,结构用 phenix程序套件中实现的phaser进行分子置换(mr)确定(ref)。以已发表的hla
‑
a*02:01结构(pdb id:5hhn)hla
‑
a*24:02结构(pdb id: 2bck)作为初始模型。使用coot和phenix进行结构构建和优化。通过 molprobity服务器和rcsb adit服务器对晶体结构进行验证。最后使用 pymol程序(delano scientific llc,san carlos,ca)显示结构。
[0117]
结果
[0118]
kia表位和hla
‑‑
a*02:01 w167相互作用的电子云密度,表明两者之间有很强的相互作用。如果将k突变为n/t则可能会破坏这种强相互作用(图4a和图4b)。从hla
‑
a*24:02和nyn表位的结构概述和相应的电子密度图中可看出l452的疏水侧链埋在hla
‑
a*24:02表面的一个口袋中(图4c和4d)。
[0119]
本发明的序列表
[0120]
seq id no:1新型冠状病毒cd8 t细胞表位肽序列
[0121]
iiwflllsv
[0122]
seq id no:2新型冠状病毒cd8 t细胞表位肽序列
[0123]
vllsvlqql
[0124]
seq id no:3新型冠状病毒cd8 t细胞表位肽序列
[0125]
slenvafnv
[0126]
seq id no:4新型冠状病毒cd8 t细胞表位肽序列
[0127]
lllddfvei
[0128]
seq id no:5新型冠状病毒cd8 t细胞表位肽序列
[0129]
llddfveii
[0130]
seq id no:6新型冠状病毒cd8 t细胞表位肽序列
[0131]
tmadlvyal
[0132]
seq id no:7新型冠状病毒cd8 t细胞表位肽序列
[0133]
fvaaifyli
[0134]
seq id no:8新型冠状病毒cd8 t细胞表位肽序列
[0135]
aifylitpv
[0136]
seq id no:9新型冠状病毒cd8 t细胞表位肽序列
[0137]
ylitpvhvm
[0138]
seq id no:10新型冠状病毒cd8 t细胞表位肽序列
[0139]
klneeiaii
[0140]
seq id no:11新型冠状病毒cd8 t细胞表位肽序列
[0141]
flalcadsi
[0142]
seq id no:12新型冠状病毒cd8 t细胞表位肽序列
[0143]
sliystaal
[0144]
seq id no:13新型冠状病毒cd8 t细胞表位肽序列
[0145]
ylatalltl
[0146]
seq id no:14新型冠状病毒cd8 t细胞表位肽序列
[0147]
ilsplyafa
[0148]
seq id no:15新型冠状病毒cd8 t细胞表位肽序列
[0149]
klvnkflal
[0150]
seq id no:16新型冠状病毒cd8 t细胞表位肽序列
[0151]
alweiqqvv
[0152]
seq id no:17新型冠状病毒cd8 t细胞表位肽序列
[0153]
fcdlkgkyv
[0154]
seq id no:18新型冠状病毒cd8 t细胞表位肽序列
[0155]
hvetfypkl
[0156]
seq id no:19新型冠状病毒cd8 t细胞表位肽序列
[0157]
wlptgtllv
[0158]
seq id no:20新型冠状病毒cd8 t细胞表位肽序列
[0159]
kldgfmgri
[0160]
seq id no:21新型冠状病毒cd8 t细胞表位肽序列
[0161]
ympyfftll
[0162]
seq id no:22新型冠状病毒cd8 t细胞表位肽序列
[0163]
qlffsyfav
[0164]
seq id no:23新型冠状病毒cd8 t细胞表位肽序列
[0165]
llflmsftv
[0166]
seq id no:24新型冠状病毒cd8 t细胞表位肽序列
[0167]
flpgvysvi
[0168]
seq id no:25新型冠状病毒cd8 t细胞表位肽序列
[0169]
ylyltfylt
[0170]
seq id no:26新型冠状病毒cd8 t细胞表位肽序列
[0171]
flahiqwmv
[0172]
seq id no:27新型冠状病毒cd8 t细胞表位肽序列
[0173]
wmvmftplv
[0174]
seq id no:28新型冠状病毒cd8 t细胞表位肽序列
[0175]
fleyhdvrv
[0176]
seq id no:29新型冠状病毒cd8 t细胞表位肽序列
[0177]
qlemeltpv
[0178]
seq id no:30新型冠状病毒cd8 t细胞表位肽序列
[0179]
clhvvgpnv
[0180]
seq id no:31新型冠状病毒cd8 t细胞表位肽序列
[0181]
flkkdapyi
[0182]
seq id no:32新型冠状病毒cd8 t细胞表位肽序列
[0183]
wldmvdtsl
[0184]
seq id no:33新型冠状病毒cd8 t细胞表位肽序列
[0185]
nllkdcpav
[0186]
seq id no:34新型冠状病毒cd8 t细胞表位肽序列
[0187]
svkglqpsv
[0188]
seq id no:35新型冠状病毒cd8 t细胞表位肽序列
[0189]
elpdefvvv
[0190]
seq id no:36新型冠状病毒cd8 t细胞表位肽序列
[0191]
yicgfiqqk
[0192]
seq id no:37新型冠状病毒cd8 t细胞表位肽序列
[0193]
sqdlsvvsk
[0194]
seq id no:38新型冠状病毒cd8 t细胞表位肽序列
[0195]
vvcteidpk
[0196]
seq id no:39新型冠状病毒cd8 t细胞表位肽序列
[0197]
asmpttiak
[0198]
seq id no:40新型冠状病毒cd8 t细胞表位肽序列
[0199]
ttiakntvk
[0200]
seq id no:41新型冠状病毒cd8 t细胞表位肽序列
[0201]
yilftrffy
[0202]
seq id no:42新型冠状病毒cd8 t细胞表位肽序列
[0203]
qvvnvvttk
[0204]
seq id no:43新型冠状病毒cd8 t细胞表位肽序列
[0205]
vvttkialk
[0206]
seq id no:44新型冠状病毒cd8 t细胞表位肽序列
[0207]
alctfllnk
[0208]
seq id no:45新型冠状病毒cd8 t细胞表位肽序列
[0209]
aimqlffsy
[0210]
seq id no:46新型冠状病毒cd8 t细胞表位肽序列
[0211]
lliimrtfk
[0212]
seq id no:47新型冠状病毒cd8 t细胞表位肽序列
[0213]
nynylyrlf
[0214]
seq id no:48新型冠状病毒cd8 t细胞表位肽序列
[0215]
kiadynykl
[0216]
seq id no:49新型冠状病毒cd8 t细胞表位肽序列
[0217]
gvyyhknnk
[0218]
seq id no:50扩增文库的引物cmv_bp f
[0219]5’
ggggacaagtttgtacaaaaaagcaggctcaggaattctgagctcg
[0220]
seq id no:51扩增文库的引物cmv_bp r
[0221]5’
ggggaccactttgtacaagaaagctgggtcagctagttaagctcgagcacccga
[0222]
seq id no:52编码生物素亲和蛋白的序列bap
[0223]
glndifeaqkiewh
[0224]
seq id no:53hla
‑
a*02:01氨基酸序列
[0225]
mnsvdgshsmryfftsvsrpgrgeprfiavgyvddtqfvrfdsda asqrmeprapwieqegpeywdgetrkvkahsqthrvdlgtlrgy ynqseagshtvqrmygcdvgsdwrflrgyhqyaydgkdyialk edlrswtaadmaaqttkhkweaahvaeqlraylegtcvewlrr ylengketlqrtdapkthmthhavsdheatlrcwalsfypaeitlt wqrdgedqtqdtelvetrpagdgtfqkwaavvvpsgqeqrytch vqheglpkpltlrwepgsglndifeaqkiewhaaalehhhhhh
[0226]
seq id no:54hla
‑
a*11:01氨基酸序列
[0227]
mnsvdgshsmryfytsvsrpgrgeprfiavgyvddtqfvrfdsda asqrmeprapwieqegpeywdqetrnvkaqsqtdrvdlgtlrgy ynqsedgshtiqimygcdvgpdgrflrgyrqdaydgkdyialned lrswtaadmaaqitkrkweaahaaeqqraylegrcvewlrryl engketlqrtdppkthmthhpisdheatlrcwalgfypaeitltwq rdgedqtqdtelvetrpagdgtfqkwaavvvpsgeeqrytchvq heglpkpltlrwelgsglndifeaqkiewhaaalehhhhhh
[0228]
seq id no:55hla
‑
a*02:07氨基酸序列
[0229]
mnsvdgshsmryfftsvsrpgrgeprfiavgyvddtqfvrfdsda asqrmeprapwieqegpeywdgetrkvkahsqthrvdlgtlrgy ynqseagshtvqrmcgcdvgsdwrflrgyhqyaydgkdyialk edlrswtaadmaaqttkhkweaahvaeqlraylegtcvewlrr ylengketlqrtdapkthmthhavsdheatlrcwalsfypaeitlt wqrdgedqtqdtelvetrpagdgtfqkwaavvvpsgqeqrytch vqheglpkpltlrwepgsglndifeaqkiewhaaalehhhhhh
[0230]
seq id no:56hla
‑
a*24:02氨基酸序列
[0231]
mnsvdgshsmryfstsvsrpgrgeprfiavgyvddtqfvrfdsda asqrmeprapwieqegpeywdeetgkvkahsqtdrenlrialry ynqseagshtlqmmfgcdvgsdgrflrgyhqyaydgkdyialk edlrswtaadmaaqitkrkweaahvaeqqraylegtcvdglrr ylengketlqrtdppkthmthhpisdheatlrcwalgfypaeitlt wqrdgedqtqdtelvetrpagdgtfqkwaavvvpsgeeqrytch vqheglpkpltlrwepgsglndifeaqkiewhaaalehhhhhh
[0232]
seq id no:57β2m氨基酸序列
[0233]
vdmiqrtpkiqvysrhpaengksnflncyvsgfhpsdievdllkng eriekvehsdlsfskdwsfyllyyteftptekdeyacrvnhvtlsq pkivkwdrdm
[0234]
以上所述的具体实施例,对本发明的目的、技术方案和有益效果进行了进一步详细说明,应理解的是,以上所述仅为本发明的具体实施例而已,并不用于限制本发明,凡在本发明的精神和原则之内,所做的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。