一种m13噬菌体单链dna的制备方法
技术领域
1.本发明涉及生物技术领域,尤其涉及一种m13噬菌体单链dna的制备方法。
背景技术:
2.dna折纸术(dna origami)是dna纳米技术的一个重要分支。该方法发明于2006年(nature 2006),即利用上百条短单链dna作为“订书钉”,辅助折叠一条长达数千碱基的噬菌体基因组单链dna(又称脚手架链),并自组装形成预先设计的结构。其中m13噬菌体所携带的单链dna为最常用的脚手架链之一。m13噬菌体是一种丝状噬菌体,内有一个环状单链dna分子,含dna复制和噬菌体增殖所需的遗传信息。其中m13mp系列对野生型m13加以改造,插入了多克隆位点和lacz基因,可容纳外源dna300-400bp,可用于制备dna测序时用的单链模板和核酸探针。
3.m13噬菌体颗粒是丝状的,只感染f (含f质粒,能产生性菌毛)的大肠杆菌。感染宿主后通常不裂解宿主细胞,而是从感染的细胞中分泌出噬菌体颗粒,宿主细胞仍能继续生长和分裂。
4.现有技术制备m13噬菌体单链dna时,主要采用聚乙二醇法沉淀m13病毒颗粒,然后利用乙醇沉淀法、苯酚抽提法等有机试剂提取m13 单链dna,在表达方面,无法通过设备线性放大从而产业化且表达水平较低;在提取方面,通常需要大量使用有机试剂且常常需要-20℃预冷,增加了对生产车间在环保、防爆等方面的需求,使得工业放大成本和风险大增。
技术实现要素:
5.有鉴于此,本发明提供了一种m13噬菌体单链dna的制备方法。该方法大大提高了m13噬菌体单链dna的产率,降低了ssdna的生产成本,适于规模化制备dna纳米折纸载体,有效地提高了经济效益。
6.为了实现上述发明目的,本发明提供以下技术方案:一种m13 噬菌体单链dna的制备方法,包括:培养宿主菌至od值为10~20,按照moi 0.1~10接入m13噬菌体进行发酵培养;将共培养的培养物离心,取上清液超滤换液后进行聚乙二醇沉淀;将沉淀物裂解、纯化,获得m13噬菌体单链dna。
7.本发明中,对m13噬菌体的具体种类没有特殊限定,本领域常见的m13噬菌体均可。在具体实施例中,具体为m13mp18噬菌体。
8.本发明中,所述宿主菌为大肠杆菌,在具体实施例中具体为jm109菌株。
9.一些具体实施例中,所述培养宿主菌至od值为15,接入m13噬菌体。
10.一些具体实施例中,所述moi 为0.1。
11.本发明中,所述发酵培养的培养基为含有5~15mm mg
2
和 10~15mm ca
2
的 2
×
yt培养基。
12.一些具体实施例中,所述发酵培养的培养基为含有15mm mg
2
和 15mm ca
2
的 2
×
yt培养基。
13.本发明中,所述发酵培养的条件为:温度37℃,ph值7.00,搅拌速率150~650rpm,溶氧值do20~30%;发酵培养的时间为4h~5h。
14.本发明中,所述聚乙二醇沉淀包括:取上清液超滤换液后,加入聚乙二醇和nacl,使聚乙二醇终浓度为4wt%-10wt%,nacl终浓度为0.5mmol/l,静置、离心;所述裂解采用碱进行裂解;所述纯化为柱层析纯化,采用的纯化柱为为sephacryls-500hr、sepharose6ff或captocore700,优选为captocore700;将共培养的培养物离心,取上清液超滤换液后进行聚乙二醇沉淀;将沉淀物裂解、纯化。
15.本发明提供一种m13噬菌体单链dna的制备方法,包括:培养宿主菌至od值为10~20,按照moi0.1~10接入m13噬菌体进行发酵培养;将共培养的培养物离心,取上清液超滤换液后进行聚乙二醇沉淀;将沉淀物裂解、纯化,获得m13mp18噬菌体单链dna。该方法大大提升了m13噬菌体单链dna的表达水平,弥补了现有技术m13噬菌体单链dna的表达水平低的缺点。本发明降低了m13噬菌体单链dna生产成本,提高了m13噬菌体单链dna的经济效益,为其在dna纳米折纸载体的推广应用奠定基础,对规模化制备dna纳米折纸载体、提高经济效益具有重要意义。
附图说明
16.图1示不同侵染时机对噬菌体生长的影响;图2示不同侵染复数(moi)对噬菌体生长的影响;图3示不同补料条件对噬菌体生长的影响;图4示captocore700柱层析纯化后的色谱图。
具体实施方式
17.本发明提供了一种m13噬菌体单链dna的制备方法。本领域技术人员可以借鉴本文内容,适当改进工艺参数实现。特别需要指出的是,所有类似的替换和改动对本领域技术人员来说是显而易见的,它们都被视为包括在本发明。本发明的方法及应用已经通过较佳实施例进行了描述,相关人员明显能在不脱离本发明内容、精神和范围内对本文的方法和应用进行改动或适当变更与组合,来实现和应用本发明技术。
18.如无特殊说明,本发明采用的试材皆为普通市售品,皆可于市场购得。
19.下面结合实施例,进一步阐述本发明:实施例1m13mp18噬菌体和宿主菌共发酵的发酵参数的优化-侵染时机1目的:比较不同侵染时机对噬菌体在5l生物反应器上生长情况的影响,并确定最优侵染时机。
20.2材料与方法2.1培养基2
×
yt培养基:大豆蛋白胨1.6%、酵母提取物1%、nacl0.5%,ph7.0。
21.2.2实验仪器2.2.1恒温摇床:上海一恒thi-300c
2.2.2 超净工作台:苏净安泰sw-cj-2fd2.2.3 ph计:梅特勒fe28 le4382.2.4 超微量光度计:denovix ds-11 2.2.5 电子天平秤:上海越平3-0.012.2.6 高压灭菌锅:上海博迅yxq-100a2.2.7 5l发酵罐:上海高机b1of-6005b1g2.3试验菌株2.3.1 宿主菌:大肠杆菌jm109 escherichia coli (migula) castellani and chalmers,购自于美国菌种保藏中心(atcc),编号为53323,储存人为crop genetics international n.v.,基因型为f' trad36 proa prob laciq delta(lacz)m15 delta(lac-proab) supe44 hsdr17 reca1 gyra96 thi-1 enda1 rela1 e14
‑ꢀ
lambda-。
22.2.3.2 噬菌体:m13 mp182.4试验方法2.4.1 共发酵主要实验参数:侵染时机:jm109大肠杆菌的od值为10 od、15 od和20od2.4.2 共发酵其他参数:2.4.2.1 侵染复数(moi):0.12.4.2.1 温度:37℃2.4.2.2 搅拌速率:150~650 rpm2.4.2.3 ph值:7.002.4.2.4 溶氧值(do):25%
±
5%温度、搅拌转数、ph值参数通过在发酵罐控制系统界面设置参数,自动调节。通气量由手动调节阀门,通过流量计来确定,为2.0~8.0l/min。培养期间,通过调整发酵罐搅拌转数和通气量。发酵过程中通过自动控制补酸碱量,使罐内ph值保持在设定值。每间隔一小时取样检测菌液od600值,并按照《分子克隆实验指南》上所述提取方法提取m13 ssdna,记录相关数据。
23.3试验结果结果见表1和图1。
24.表1 不同侵染时机对噬菌体生长的影响发酵时长1h2h3h4h5hod101.294.288.839.4210.15od155.2215.0818.8319.8121.18od205.7711.4111.4211.63-注:表中数值为累计ssdna产率,单位为mg/l,即折合每升发酵液所产ssdna量。
25.4结果分析如表1与图1所示,侵染时机为jm109大肠杆菌的od值15 od时,累计ssdna产率明显高于10od和20od组,故选取侵染时机为od15为最优。
26.实施例2 m13 mp18噬菌体和宿主菌共发酵的发酵参数的优化-侵染复数(moi)1目的:比较不同侵染复数(moi)对噬菌体在5l生物反应器上生长情况的影响,并
确定最优侵染复数(moi)。
27.2材料与方法2.1培养基2
×
yt培养基:大豆蛋白胨1.6%、酵母提取物1%、nacl 0.5%,ph7.0。
28.2.2 实验仪器2.2.1 恒温摇床:上海一恒thi-300c2.2.2 超净工作台:苏净安泰sw-cj-2fd2.2.3 ph计:梅特勒fe28 le4382.2.4 超微量光度计:denovix ds-11 2.2.5 电子天平秤:上海越平3-0.012.2.6 高压灭菌锅:上海博迅yxq-100a2.2.7 5l发酵罐:上海高机b1of-6005b1g2.3试验菌株2.3.1 宿主菌:大肠杆菌jm109 escherichia coli (migula) castellani and chalmers,购自于美国菌种保藏中心(atcc),编号为53323,储存人为crop genetics international n.v.,基因型为f' trad36 proa prob laciq delta(lacz)m15 delta(lac-proab) supe44 hsdr17 reca1 gyra96 thi-1 enda1 rela1 e14
‑ꢀ
lambda-。
29.2.3.2 噬菌体:m13 mp182.4试验方法2.4.1 共发酵主要实验参数:侵染复数(moi):0.1、1、5、102.4.2 共发酵其他参数:2.4.2.1 侵染时机:jm109大肠杆菌的od值为15 od2.4.2.2 温度:37℃2.4.2.3 搅拌速率:150~650 rpm2.4.2.4 ph值:7.002.4.2.5 溶氧值(do):25%
±
5%温度、搅拌转数、ph值参数通过在发酵罐控制系统界面设置参数,自动调节。通气量由手动调节阀门,通过流量计来确定,为2.0~8.0l/min。培养期间,通过调整发酵罐搅拌转数和通气量。发酵过程中通过自动控制补酸碱量,使罐内ph值保持在设定值。每间隔一小时取样检测菌液od600值,并按照《分子克隆实验指南》上所述提取方法提取m13 ssdna,记录相关数据。
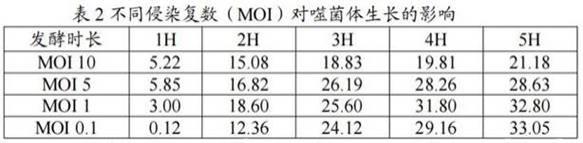
30.试验结果注:表中数值为累计ssdna产率,单位为mg/l,即折合每升发酵液所产ssdna量
4结果分析如表2与图2数据所示,侵染复数为0.1和1时,累计ssdna产率明显高于5和10组,0.1和1两组产率基本相同,但考虑到moi降低一个量级后,噬菌体种子库建库规模将大大减小,故选取moi值为0.1为最优。
31.实施例3 m13 mp18噬菌体和宿主菌共发酵的发酵参数的优化-补料条件1目的:比较不同补料条件对噬菌体在5l生物反应器上生长情况的影响,并确定最优补料条件。
32.2材料与方法2.1试剂2.1.1 2
×
yt培养基:大豆蛋白胨1.6%、酵母提取物1%、nacl 0.5%,ph7.0。
33.2.1.2 mg
2
离子2.1.3 ca
2
离子2.2 实验仪器2.2.1 恒温摇床:上海一恒thi-300c2.2.2 超净工作台:苏净安泰sw-cj-2fd2.2.3 ph计:梅特勒fe28 le4382.2.4 超微量光度计:denovix ds-11 2.2.5 电子天平秤:上海越平3-0.012.2.6 高压灭菌锅:上海博迅yxq-100a2.2.7 5l发酵罐:上海高机b1of-6005b1g2.3试验菌株2.3.1 宿主菌:大肠杆菌jm109 escherichia coli (migula) castellani and chalmers,购自于美国菌种保藏中心(atcc),编号为53323,储存人为crop genetics international n.v.,基因型为f' trad36 proa prob laciq delta(lacz)m15 delta(lac-proab) supe44 hsdr17 reca1 gyra96 thi-1 enda1 rela1 e14
‑ꢀ
lambda-。
34.2.3.2 噬菌体:m13 mp182.4试验方法2.4.1 共发酵的主要实验参数:详见表32.4.2 共发酵的其他参数:2.4.2.1 侵染时机:jm109大肠杆菌的od值为15 od2.4.2.2 侵染复数(moi):0.12.4.2.3 温度:37℃2.4.2.4 搅拌速率:150~650 rpm2.4.2.5 ph值:7.002.4.3.6 溶氧值(do):25%
±
5%温度、搅拌转数、ph值参数通过在发酵罐控制系统界面设置参数,自动调节。通气量由手动调节阀门,通过流量计来确定,为2.0~8.0l/min。培养期间,通过调整发酵罐搅拌转数和通气量。发酵过程中通过自动控制补酸碱量,使罐内ph值保持在设定值。每间隔一小时取样检测菌液od600值,并按照《分子克隆实验指南》上所述提取方法提取m13 ssdna,记
录相关数据。
35.3试验结果表3 不同侵染复数(moi)对噬菌体生长的影响实验编号12345补培养基
‑‑
3l2
×
yt3l2
×
yt3l2
×
ytmgcl2浓度-15mm5mm15mm15mmcacl2浓度-15mm10mm10mm15mm1h0.120.070.034.800.072h12.3612.9813.1721.8411.503h24.1222.6334.0539.5625.974h29.1625.9641.7044.9449.785h33.0532.2850.2055.2058.42注:表中数值为累计ssdna产率,单位为mg/l,即折合每升发酵液所产ssdna量4结果分析如表3与图3所示,实验编号5所用的补料组合 ssdna产率最高。
36.结合以上实验结果,可以确定本发明制备噬菌体单链dna的最佳方案为实验编号为5的方案:侵染时机为jm109大肠杆菌od值15 、侵染复数(moi)为0.1、补料条件为:含15mmol/lmg
2
、15mmol/l的ca
2
的2
×
yt培养基。
37.实施例4 m13 单链dna的柱层析法纯化1 目的:使用capto core 700柱层析对裂解后的m13 单链dna进行纯化,去除残留的宿主蛋白和内毒素,取代传统的乙醇沉淀法。
38.2 材料与方法2.1 试剂和耗材2.1.1 层析柱:capto core 7002.1.2 流动相:20mm tris-hcl,150mm nacl,ph 8.02.1.3 试剂:2.1.3.1 pbb2(50 ml量)0.4g naoh0.5g sds加水至50ml2.1.3.2 pbb3(50 ml量)12.3g醋酸钠或14.72g醋酸钾5.75ml冰醋酸加水至50ml2.1.3.3 75%乙醇(-20℃预冷)2.1.3.4 无水乙醇(-20℃预冷)2.2 实验仪器2.2.1 离心机:赛默飞 lynx 40002.2.2 纯化仪:赛普 sdl 100
2.3 实验材料:经peg法、碱裂解法粗提后的m13 单链dna2.4 试验方法2.4.1 peg法提取m13颗粒2.4.1.1 方法详见《分子克隆实验指南第4版》(上册)方案8 聚乙二醇沉淀法制备m13噬菌体单链dna,p24-p26。
39.2.4.2 碱裂解法提取m13单链dna2.4.2.1 按pbb2:m13病毒颗粒溶液=1:2的比例加入pbb2溶液,轻柔搅拌均匀后,室温下分别放置3min。
40.2.4.2.2 按pbb3:m13病毒颗粒溶液=1:1.5的比例加入pbb3溶液,轻柔搅拌均匀,2~8℃静置10min。
41.2.4.2.3 离心,16500g,30min,4℃,弃沉淀。若上清较为浑浊,再离心一次,16500g,15min,4℃。收集上清液。
42.2.4.3 对照组:乙醇沉淀法2.4.3.1 方法详见《分子克隆实验指南第4版》(上册)方案8 聚乙二醇沉淀法制备m13噬菌体单链dna,p24-p26。
43.2.4.4 实验组:capto core 700柱层析法2.4.4.1 将裂解后的m13单链dna上清液上样于capto core 700层析柱。
44.2.4.4.2 根据260nm吸光值收集流穿液,该流穿液为目的物。
45.2.4.4.3 紫外定浓度,扫描波段:230~300nm。 并检测宿主蛋白及内毒素残留水平。
46.3 试验结果3.1层析结果层析结果见图4。
47.3.2 m13单链dna回收率及杂质去除水平表4
组别m13单链dna收率宿主蛋白去除率内毒素水平对照组:乙醇沉淀法62.1s.8%0.00705≤x<0.07052实验组:本发明柱层析法85.5�.4%0.00041≤x<0.00408
4结果分析结果表明,从收率及杂质残留水平等方面看,本发明柱层析法均优于传统的乙醇沉淀法,且由于免除的乙醇的使用,对未来生产车间的建设极大程度降低了难度。
48.以上仅是本发明的优选实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。
再多了解一些
本文用于创业者技术爱好者查询,仅供学习研究,如用于商业用途,请联系技术所有人。