一种dna信息存储方法及dna信息储存载体
技术领域
1.本发明涉及生物信息学领域,尤其是涉及一种dna信息存储方法及dna信息储存载体。
背景技术:
2.当今世界数据的指数级增长对存储技术提出了重大挑战,人类步入信息化社会的脚步已无法停歇,大数据时代要求我们创造更密集、更便宜的存储技术。研究统计表明2020年全球数字档案达到44万亿千兆字节(gb),比2013年增加10倍,到2040年,如果所有数据使用闪存芯片即时访问,那么所需微芯硅片将是预期产量的10-100倍。研究人员认为,如果信息可以像大肠杆菌基因一样密集包裹,那么大约1公斤dna可以满足全世界额存储需求。作为携带基因信息的生物材料,dna被认为是一种极具吸引力的数据存储介质,可以提供极高的存储密度、耐用性以及可靠的信息复制能力,在信息存储方面具有极大的潜力。
3.质粒载体通常带有一个或一个以上的选择性标记基因(如抗生素抗性基因)和一个人工合成的含有多个限制性内切酶识别位点的多克隆位点序列,并去掉了大部分非必需序列,使分子量尽可能减少,以便于基因工程操作。质粒具有良好的稳定性、可扩增性和便于体内体外存储的特性,由此可见,质粒载体是dna信息存储的完美载体,通过质粒载体可以实现dna信息的复制及长期保存。
4.随着dna写入(合成)和读取(测序)技术的发展,dna信息存储技术得以快速发展。dna信息存储技术一般由四个步骤组成:一是信息编码,即将数字信息编码为dna序列信息;二是信息写入,即通过dna合成或编辑将编码后的序列信息写入dna分子中;三是信息保存,即通过一定的方式将合成的dna序列在体内/体外进行长期保存,以便随时检索及选择性信息读取;四是信息解码,即通过测序等方式读取dna序列信息,并将序列信息转换为数字信息。
5.现阶段常用的dna单链合成法为柱式合成法和高通量合成法,其中高通量合成法方法分为affymetrix公司光刻掩模法、agilent和twist公司的喷墨打印法和custom array公司(已被南京金斯瑞收购)的电化学阵列合成法,微软研究院提出使用高密度纳米级电极孔扩展dna数据存储,高通量合成的主要原则是降低单个序列生成所需的面积。高通量合成法与传统的柱式法合成相比,可并行合成105个不同寡核苷酸链,价格降低了3-4个数量级,更适用于较大规模的dna存储。但受化学合成法副产物的限制,这一方法合成的dna链长一般也不超过200碱基,其中喷墨的方法可能能合成更长的链。
6.现阶段常用的dna双链合成法为基于连接反应的组装(ligation-based assembly)和基于聚合酶链式反应的组装(pcr-based assembly)。寡核苷酸直接由化学法合成,并且在5’端进行磷酸化。在进行连接反应组装时,构成dna片段或dna全长的所有寡核苷酸在连接酶缓冲液中与连接酶混合在一起,进行变性、退火和连接。其中,连接反应温度是由寡核苷酸退火条件和连接酶反应条件共同决定的。这种变性和退火、连接过程称为连接酶链式反应(ligase chain reaction,lcr),是可以不断重复很多轮的,类似聚合酶链式
反应的循环。为了得到足够数量的全长基因产物,连接反应之后一般会跟随一步pcr扩增反应(在连接产物中加入特异性基因末端引物)。聚合酶链式反应(pcr)已经成为基于寡聚核苷酸的基因合成方案中最常用的一种。通过一步pcr组装的方法,可以将一组寡核苷酸混合物一步组装成为基因长度dna,但这种方法得到的pcr产物通常是一组长度不一的dna。另外一种基于pcr的两步法组装方法,首先将基因拆分成一系列亚库(subpools/subassembly reactions),分别组装成为片段,再将这些片段混合,最终组装成为完整基因。
7.然而现有的dna存储技术仅仅通过dna单链或双链的单一形式进行存储,无法满足多样化信息存储的需求,且存储的信息容量有限,不利于信息储量的扩增。
8.需要说明的是,在上述背景技术部分公开的信息仅用于对本技术的背景的理解,因此可以包括不构成对本领域普通技术人员已知的现有技术的信息。
技术实现要素:
9.本发明的目的在于解决使dna能够储存多样化信息的问题,提供一种dna信息存储方法。
10.为实现上述目的,本发明采用以下技术方案:
11.一种dna信息存储方法,包括以下步骤:
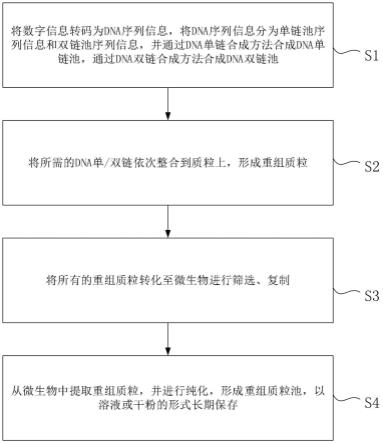
12.s1、将数字信息转码为dna序列信息,将dna序列信息分为单链池序列信息和双链池序列信息,并通过dna单链合成方法合成dna单链池,通过dna双链合成方法合成dna双链池;
13.s2、将所需的dna单/双链依次整合到质粒上,形成重组质粒;
14.s3、将所有的重组质粒转化至微生物进行筛选、复制;
15.s4、从微生物中提取重组质粒,并进行纯化,形成重组质粒池,以溶液或干粉的形式长期保存。
16.在一些实施例中,所述dna序列信息所存储信息的格式包括文本、图片、音频和视频。
17.在一些实施例中,步骤s2中,所述将dna单/双链整合到质粒上的方式为传统酶连方式或无缝克隆。
18.在一些实施例中,步骤s2中,所述将dna单/双链整合到质粒上的具体步骤为:
19.s21、将单链序列扩增为双链:若需整合的dna链为单链,则将单链序列扩增为双链;若需整合的dna链为双链,则跳过该步骤;
20.s22、获取目的片段:选择dna链整合到质粒载体上的具体位置及对应的酶切位点,设计出相应的同源臂序列,将同源臂序列扩增到dna链上获得目的片段;
21.s23、线性化质粒载体:在单/双链待插入的位置对质粒载体线性化处理;
22.s24、构建重组质粒:将目的片段与线性化载体连接,形成重组质粒。
23.进一步地,步骤s22中,目的片段与载体连接过程使用同源重组方法或传统的酶连方法。
24.在一些实施例中,步骤s3中,所述的重组质粒转化包括感受态细胞的制备及转化。
25.在一些实施例中,步骤s3中,所述筛选过程包括重组菌株的培养和阳性克隆的筛选。
26.在一些实施例中,步骤s4中,所述微生物为原核生物或真核生物,所述原核生物为大肠杆菌或芽孢杆菌,所述真核生物为酵母菌。
27.在一些实施例中,步骤s4中,所述提取重组质粒的方法为试剂盒抽提质粒法或碱抽提质粒法。
28.本发明还公开了一种dna信息储存载体,所述载体通过上述的方法获得。
29.本发明具有如下有益效果:
30.本发明的dna信息存储方法信息存储稳定性强且形式多样化,通过将dna信息的形式分为单链和双链的设置,可根据需要将不同形式的链整合到质粒上,能够解决dna信息的多样化存储的问题,从而有效提高信息存储的多样性。通过形成重组质粒池,以溶液或干粉的形式长期保存,使dna信息存储具有更强的稳定性。
附图说明
31.图1是本发明实施例中的一种dna信息存储方法的流程图;
32.图2为本发明基于dna单双链的可拓展信息存储方法的重组质粒池构建示意图;
33.图3为本发明基于dna单双链的可拓展信息存储方法的总流程示意图;
34.图4为本发明实施例中载体puc57的质粒图谱;
35.图5为本发明实施例中质粒与dna片段重组的流程示意图。
具体实施方式
36.以下对本发明的实施方式做详细说明。应该强调的是,下述说明仅仅是示例性的,而不是为了限制本发明的范围及其应用。
37.需要说明的是,当元件被称为“固定于”或“设置于”另一个元件,它可以直接在另一个元件上或者间接在该另一个元件上。当一个元件被称为是“连接于”另一个元件,它可以是直接连接到另一个元件或间接连接至该另一个元件上。另外,连接既可以是用于固定作用也可以是用于耦合或连通作用。
38.需要理解的是,术语“长度”、“宽度”、“上”、“下”、“前”、“后”、“左”、“右”、“竖直”、“水平”、“顶”、“底”“内”、“外”等指示的方位或位置关系为基于附图所示的方位或位置关系,仅是为了便于描述本发明实施例和简化描述,而不是指示或暗示所指的装置或元件必须具有特定的方位、以特定的方位构造和操作,因此不能理解为对本发明的限制。
39.此外,术语“第一”、“第二”仅用于描述目的,而不能理解为指示或暗示相对重要性或者隐含指明所指示的技术特征的数量。由此,限定有“第一”、“第二”的特征可以明示或者隐含地包括一个或者更多该特征。在本发明实施例的描述中,“多个”的含义是两个或两个以上,除非另有明确具体的限定。
40.有鉴于此,本发明实施例的目的在于针对现有技术中存在的dna信息存储形式单一、存储容量有限等问题,本发明实施例提供了一种dna信息存储方法。通过将数字信息转码为dna序列信息,再将dna序列信息按照一定的方式分为dna单链池序列信息和dna双链池序列信息,并通过生物合成的方式构建出相应的dna单/双链池,dna单链池/dna双链池是许多dna单/双链(信息链)所构成的集合;然后将其中的dna单/双链依次整合到质粒载体上,待所有单/双链序列整合完毕后,将全部重组质粒转化至微生物体内进行筛选、复制,最后
通过质粒提取及纯化形成重组质粒池,以质粒干粉或溶液的形式进行长期保存。本方法构建的存储信息体系明显优于现有的dna信息存储体系,信息存储稳定性强且形式多样化,可根据需要将若干dna单/双链依次整合到同一质粒载体上;信息存储容量的扩增方便快捷,可通过后续重组质粒的构建与添加进行扩容;信息存储更加系统化,便于进行信息的筛选及检索;开拓了dna作为信息存储介质的应用潜力,本发明实施例的信息存储技术可用于数字信息存储、数字图书馆、信息加密传输等领域。
41.本发明实施例中扩容具体原理如下:转化至微生物体内的重组质粒通过质粒抽提的相关方法即可完成提取,以质粒干粉或溶液的形式进行存储,形成信息存储池。因此,信息存储容量与重组质粒的种类及数量正相关,根据本技术方法不断新建重组质粒,即可实现信息存储量的扩大;
42.本发明实施例中便于进行信息的筛选及检索的原理如下:对于体外存储的信息池——质粒,更便于对质粒进行各种生物操作,如酶切、dna片段插入等,也可通过dna探针设计等方式对质粒上的信息片段进行检索,并可直接用于测序分析,因此更加便于进行信息池的信息筛选与检索,远远优于传统的dna“冷数据”存储的方法。
43.常用的质粒载体类型如:puc57、puc57-kan、puc57-simple、puc57-mini、puc18、puc19等;
44.dna合成公司也会有常用的商业化载体,不同公司之间的商业化载体略有出入,整体差别不大,金斯瑞公司提供的150余种商业化载体如表1。
45.表1
46.47.另外质粒载体也可根据不同的需求在原有载体的基础上进行设计改造,形成新的载体。
48.参考图1和图3,本发明实施例提供了一种基于dna单双链的可拓展信息存储方法,包括以下步骤:
49.s1、将存储信息的二进制编码通过算法转码为dna序列信息,将dna序列信息通过一定的方式分为单链池序列信息和双链池序列信息,最后通过柱式合成法或高通量合成法合成dna单链池,通过基于连接反应的组装方法或基于聚合酶链式反应的组装方法合成dna双链池;
50.s2、通过传统酶连、无缝克隆等方式将所需的dna单/双链依次整合到质粒上,形成重组质粒;
51.s3、将所有的重组质粒转化至微生物进行筛选、复制;
52.s4、从微生物中提取重组质粒,并进行纯化,形成重组质粒池,以溶液或干粉的形式长期保存;
53.解码步骤:
54.s5、将重组质粒池溶解后进行酶切,酶切产物纯化回收后,通过测序等方式读取dna序列信息,并将序列信息解码为数字信息。
55.本发明实施例中,所述存储信息包括文本、图片,音频和视频等。
56.本发明实施例中,所述算法包括霍夫曼编码、逗号编码、交替编码和喷泉码,但不限于以上四种编码。
57.参考图2,步骤s1中,所述通过现阶段的dna合成方法分别合成单双链,dna单链合成方法包括但不限于柱式合成法、芯片合成法、酶促合成法,dna双链合成方法包括但不限于连接酶拼接法(lcr)、聚合酶循环组装法(pca),本发明实施例所述dna单链合成方法为半导体芯片合成法,合成的单链池为12k或92k芯片,每条单链为飞摩级别,单链池总量为2—10微克,合成周期为1—2周,该合成方法控制准确性高,在封闭空间内合成,受外界因素小。在一些实施方案中,本发明实施例所述dna双链合成方法为基于pcr的两步组装方法,该方法合成的基因序列保真度较高(错误率:1.2error/kb),成本低,适于合成长链基因序列、高g/c含量dna序列和具有复杂二级结构的dna序列等,图2中的双链1既可以指由单链扩增出的双链,又可以指本身即为双链的dna信息片段;只有双链的形式才便于用本方法整合至质粒上,编号为双链1、2、3
……
n等用于区别整合到质粒上的不同的信息链。
58.步骤s2中,所述将dna单/双链整合到质粒上的方式包括但不限于传统酶连方式、无缝克隆等,具体步骤为:
59.s21、pcr扩增为双链:若需整合的dna链为单链,则通过引物设计及pcr扩增后,将单链序列扩增为双链,并对pcr产物进行回收纯化;若需整合的dna链为双链,则跳过该步骤;
60.s22、pcr获取目的片段:选择dna链整合到质粒载体上的具体位置及对应的酶切位点,设计出相应的同源臂序列,通过pcr法将同源臂序列扩增到dna链上获得目的片段,对pcr产物进行回收纯化;
61.s23、线性化质粒载体:在单链(双链2)待插入的位置采用单酶切、双酶切或pcr方式对质粒载体线性化处理,对反应产物进行纯化回收;
62.s24、构建重组质粒:将目的片段通过酶连反应试剂盒或无缝克隆试剂盒等与线性化载体连接,形成重组质粒;
63.s25、重复步骤s21-s24将所有的dna单/双链整合到质粒载体上。
64.本发明实施例中,所述的质粒载体包括但不限于puc57(重组大肠杆菌),所述将dna单/双链整合到质粒上的方式包括但不限于传统酶连方式、无缝克隆等,质粒载体线性化的方式包括但不限于双酶切方式,还包括单酶切、pcr方式等,目的片段与载体连接过程包括但不限于传统的酶连方法,同源重组方法等。
65.步骤s3中,所述将所有的重组质粒转化至微生物进行筛选、复制。其中,重组质粒转化包括感受态细胞的制备及转化过程,转化方法包括但不限于电转化法、化学转化法。鉴定与筛选过程包括重组菌株的培养和阳性克隆的筛选。其中感受态细胞是微生物细胞的一种特殊状态,如大肠杆菌的感受态细胞。
66.本发明实施例中,所述微生物可以为原核生物或真核生物。其中,所述原核生物包括但不限于大肠杆菌或芽孢杆菌、农杆菌、乳酸菌、链球菌等,所述真核生物包括但不限于酵母菌、哺乳动物(如猴肾细胞)、昆虫(如家蚕细胞)、真菌等。
67.步骤s4中,所述从微生物中提取重组质粒,进行纯化后形成重组质粒池,其中提取质粒的方法包括但不限于试剂盒抽提质粒法、碱抽提质粒法。
68.步骤s5中,所述测序方式包括但不限于第一代测序方法、第二代测序方法、第三代测序方法。所述解码方法与编码方法相对应即可,进一步恢复出原来的文字、图片及视频等原始信息。
69.本发明实施例还提出了一种dna信息储存载体,由上述的方法获得。
70.实施例
71.本实施例中将数字信息通过霍夫曼算法转码为dna序列,再将总的dna序列按照一定的方式分为若干单链(短链)序列及双链(长链)序列,然后将一条单链序列及其对应的一条双链序列依次整合到同一载体质粒puc57的不同位置,整合后puc57如图4所示,待所有单双链序列整合完毕后,将全部质粒转化至大肠杆菌体内进行筛选、复制,最后通过质粒提取及纯化形成重组质粒池,以质粒干粉的形式进行长期保存。
72.1、dna单双链的合成
73.将存储信息的二进制编码通过霍夫曼算法转码为总的dna序列,将总的dna序列通过一定的方式分为单链序列双链序列,通过半导体芯片法合成dna单链,其中合成的单链池为12k芯片,每条单链为飞摩级别,长度为170nt左右,单链池总量为2—10微克;通过基于pcr的两步组装法合成dna双链序列,考虑到合成时间和成本,双链序列的长度为5000bp左右。
74.2、dna单链序列扩增为双链序列
75.进行反向引物设计,通过pcr方式将单链序列扩增为双链2,对pcr产物进行纯化和回收,具体步骤如下:
76.1)通过琼脂凝胶电泳尽可能将目的dna片段与其它片段分开,用干净的手术刀片将含目的dna片段的凝胶切下,放入1.5ml离心管中,称重;
77.2)根据胶块的重量和浓度,按每100mg琼脂糖加300μl的比例加入bufferb2(溶胶用溶液),如琼脂糖胶块不足100mg则用水补充至100mg;
78.3)将离心管置于50℃水浴5-10min,间或混匀,直至胶块完全溶化;
79.4)将溶化好的溶液全部移入吸附柱,8000g离心30sec。倒掉收集管中的液体,将吸附柱放入同一个收集管中;
80.5)向吸附柱中加入500μl wash solution(洗涤液),9000g离心30sec。倒掉收集管中的液体,将吸附柱放入同一个收集管中;
81.6)重复步骤5)一次;
82.7)将空吸附柱和收集管放入离心机,9000g离心1min;
83.8)在吸附膜中央加入15-40μl elution buffer(一种洗脱液),室温静置1-2min,9000g离心1min。将所得到的dna溶液置于-20℃保存或后续实验。
84.3、dna序列-同源臂序列的连接
85.选择质粒载体为puc57,单链(双链2)序列插入至酶切位点zraι、aatιι之间,双链序列插入至酶切位点sacι、bsmι之间,根据dna序列及对应插入位置的碱基序列设计出前向同源臂序列及反向同源臂序列,再通过pcr法将同源臂序列扩增至相应的dna序列前后两端,最后对pcr产物进行纯化回收,具体方法同上,其中pcr扩增的反应流程如下:
86.首先,将10
×
pcr缓冲液、上游引物、下游引物、模板dna和4dntp混合液加入0.5ml的塑料离心管中,加灭菌水至适当的反应体系,具体pcr反应体系各试剂体积表2所示。
87.表2
[0088][0089]
注意:试剂在冰上融化,50μl体系混匀,在pcr仪上设置程序并进行反应。然后,将上述液体混合,短暂离心后,置95℃加热变性5min;接着加入适量taq dna聚合酶,混匀;再加入两三滴液体矿物油,使之覆盖在反应液上,以防蒸发;再短暂离心后,将反应管置于pcr仪中按一定反应条件作热循环,具体pcr反应条件如表3所示:
[0090]
表3
[0091]
[0092]
pcr产物经1%琼脂糖电泳110v,30min;利用eastep(总rna提取试剂盒)凝胶及pcr回收试剂盒,对pcr产物进行回收纯化,具体操作方法同上。
[0093]
4、质粒载体线性化
[0094]
用限制性核酸内切酶zraι、aatιι对载体puc57进行双酶切,酶切反应体系如表4所示,将获得的60μl酶切反应产物进行凝胶电泳,条件电压110v,时间30min,电泳结束后,酶切反应产物60μl进行纯化回收,具体操作方法同上。
[0095]
表4
[0096]
试剂体积质粒puc572μgzrai2μlaatii2μl10
×
green buffer6μl去离子水补加到60μl
[0097]
5、重组质粒1构建
[0098]
将带有同源臂序列的双链2通过seamless assembly cloning kit无缝克隆
[0099]
试剂盒与线性化载体连接,线性化载体建议使用20-60ng,目的片段建议是载体的3倍摩尔量,重组反应体系如表5所示,可将重组质粒1放-20℃保存。
[0100]
表5
[0101]
试剂体积载体3μl目的片段1μl2
×
seamless mix5μl去离子水1μl
[0102]
6、重组质粒1线性化
[0103]
参考图5,用限制性核酸内切酶saci、bsmi对重组质粒1进行双酶切,将获得的60μl酶切反应产物进行凝胶电泳,条件电压110v,时间30min,电泳结束后,酶切反应产物60μl进行纯化回收,具体操作方法同上。
[0104]
7、重组质粒构建
[0105]
将带有同源臂序列的双链通过seamless assembly cloning kit无缝克隆试剂盒与线性化载体连接,形成最终的重组质粒,重组反应体系同上,可将重组质粒放-20℃保存。
[0106]
8、重复步骤1——7直至将所有的单双链整合到质粒载体上。
[0107]
9、重组质粒转化至大肠杆菌
[0108]
该步骤包括大肠杆菌电转化感受态细胞制备及重组质粒电转化过程,具体操作如下:
[0109]
感受态细胞制备:
[0110]
1)从-70℃中拿出冻存的dh5α菌株,不待融化,迅速用牙签(接种环)挑取一些冰渣,在lb培养板(amp-单磷酸腺苷)上划线,37℃培养过夜;
[0111]
2)挑取1个单菌落,放入100ml的盛放于250ml三角锥瓶中的lb液体培养基(培养细菌的常用液体培养基)(amp-)中,37℃,220rpm摇菌过夜;
[0112]
3)对上述菌液测量od值(光密度)在0.4-0.5之间时,取出锥瓶,转入50ml离心管,置冰上,4℃,3000rpm,10minuts离心收集细胞,将上清液倒尽;
[0113]
4)加入10ml预先冰冷的tss(总悬浮固体)溶液在冰上重悬细胞。勿使细胞下沉,迅速将感受态细胞悬液分装为100ul/管,丢入液氮速冻,并转移到-70℃冰箱储存备用。
[0114]
重组质粒电转化:
[0115]
1)将保存于-70℃的感受态细胞取出放置于冰上解冻,并立即加入5ul连接产物,并在冰上放置60min。
[0116]
2)42℃热激90sec,立刻冰浴2min。
[0117]
3)向转化后的细菌中加入780ullb液体培养基和20ul 1m葡萄糖储液。室温下,150rpm温和摇菌30min,37℃200rpm摇菌30min。
[0118]
10、筛选与鉴定:
[0119]
该步骤包括重组菌株的培养和阳性克隆的筛选,具体操作如下:
[0120]
重组菌株的培养:
[0121]
1)将复苏后的重组细菌在常温下,4000rpm离心5min,吸取800ul澄清lb液体培养基;
[0122]
2)用200ul黄枪头,小心吹打离心后的细菌,使成悬液,并将其加入到预先冻存的lb平板,amp ,含x-gal和iptg;
[0123]
3)用涂布棒小心涂布均匀,在超净工作台中放置,半开盖,待菌液完全被吸收,约10分钟,倒置于37℃生化培养箱中培养12-18h。
[0124]
阳性克隆的筛选:
[0125]
1)取出培养基,在冰箱中4℃放置数小时,直到显现蓝色菌斑;
[0126]
2)用灭菌牙签挑取白色菌斑,在10ul反应体系(载体测序引物与扩增cdna特异引物组合)pcr管内蘸洗1-2次,然后再在事先标记好的培养皿的方格区域内接种4-5个菌落(注意pcr管和培养皿方格标记一致)牙签回收灭菌,重复利用;
[0127]
3)培养菌的同时,做菌落pcr。菌落pcr做完,1.2%的琼脂糖凝胶进行电泳,根据目的条带大小确定阳性克隆菌斑并判断dna插入的方向,并在对应的培养皿的方格上做标记;
[0128]
4)挑选已标记的阳性菌斑,放入800ul lb液体培养基(amp )中于37℃300rpm摇菌数小时;
[0129]
5)扩大培养的菌液亚克隆测序。
[0130]
11、重组质粒提取与纯化
[0131]
该步骤包括质粒提取与dna的纯化过程,形成重组质粒池,以溶液或干粉的形式长期保存。质粒提取实验流程如下:
[0132]
质粒快提试剂盒参照说明书,具体操作如下:
[0133]
1)收集菌体,1分钟,10000rpm;
[0134]
2)根据菌体量加入相应体积的rb溶液,已经按照说明书要求加入相应体积的rnasea,将菌体吹打混匀;
[0135]
3)加入与rb溶液等体积的lb溶液,轻轻地上下颠倒混匀6~10次,静置4分钟;
[0136]
4)向步骤“3)”中加入相应体积的nb溶液,轻轻地上下颠倒混匀6~10次,静置2分钟;
[0137]
5)离心5分钟,12000rpm;
[0138]
6)将全部上清小心吸取转入试剂盒中吸附柱,离心1分钟,12000rpm,弃掉吸附柱配套底部收集管中废液。若上清过多,可分次注入吸附柱,每次最大容量为800μl;
[0139]
7)向吸附柱中加入650μl溶液wb(bufferwb一种洗脱液),已经按照说明书要求加入相应体积的无水乙醇,离心1分钟,12000rpm,弃掉吸附柱配套底部收集管中废液;
[0140]
8)再次离心2分钟,12000rpm,弃掉收集管及废弃液;
[0141]
9)将吸附柱放置于无菌1.5ml离心管上,向吸附柱中心滤膜小心滴加提前70℃预热的20~40μl超纯水,室温静置1分钟,然后离心1分钟,10000rpm;
[0142]
10)离心回收的20-40μl质粒再次注入同一吸附柱,离心1分钟,10000rpm;
[0143]
11)将回收的质粒放入-20℃冰箱储备。
[0144]
dna的纯化实验流程如下:
[0145]
1)切凝胶:电泳结束后,利用凝胶成像仪,可以看到琼脂糖凝胶中有含目的dna片段的亮条带,用干净的刀片迅速切下凝胶;
[0146]
2)凝胶称重:将胶块放入已经高压灭菌过的1.5ml离心管中,称取重量,并计算出胶块的重量,注意:离心管已称重;
[0147]
3)溶解胶:准备好65℃的水浴锅,算出凝胶重每10mg加入膜结合液10μl,将其放入水浴锅中溶解;
[0148]
4)结合:试剂盒中微量纯化柱插入收集管,将溶解后的凝胶混合液吸入微量纯化柱内,于室温高速离心机16000g离心1min,将微量纯化柱下面收集管中的废液弃掉,注意不要用手触碰到微量纯化柱;
[0149]
5)洗涤:加入700μl膜清洗液于微量纯化柱中,膜清洗液已加入乙醇,于室温高速离心机16000g离心1min。将微量纯化柱下面收集管中的废液弃掉,注意不要用手触碰到微量纯化柱;
[0150]
6)二次洗涤:加入500μl膜清洗液于微量纯化柱中,膜清洗液已加入乙醇,于室温高速离心机16000g离心5min。将微量纯化柱下面收集管中的废液弃掉,注意不要用手触碰到微量纯化柱;
[0151]
7)洗脱:小心取出微量纯化柱,禁止任何物品与微量纯化柱接触,将柱子转移到已经高压灭菌好的1.5ml离心管中。向微量纯化柱中加入50μl无核酸酶水,室温放置1min后,高速离心机16000g离心1min,将离心管中收集的dna水溶液保存在-20℃。
[0152]
12、信息读取
[0153]
将重组质粒池溶解后进行酶切,酶切产物纯化回收后,通过dna测序方法读取dna序列信息,并将序列信息解码为数字信息。
[0154]
由上述技术方案可知,本发明实施例提供了一种基于dna单双链的可拓展信息存储方法。通过将数字信息转码为dna序列信息,再将dna序列信息按照一定的方式分为dna单链池序列信息和dna双链池序列信息,并通过生物合成的方式构建出相应的dna单/双链池,然后将其中的dna单/双链依次整合到质粒载体上,待所有单/双链序列整合完毕后,将全部重组质粒转化至微生物体内进行筛选、复制,最后通过质粒提取及纯化形成重组质粒池,以质粒干粉或溶液的形式进行长期保存。本方法构建的存储信息体系明显优于现有的dna信息存储体系,信息存储稳定性强且形式多样化,可根据需要将若干dna单/双链依次整合到
同一质粒载体上;信息存储容量的扩增方便快捷,可通过后续重组质粒的构建与添加进行扩容;信息存储更加系统化,便于进行信息的筛选及检索;开拓了dna作为信息存储介质的应用潜力,本方法的信息存储技术可用于数字信息存储、数字图书馆、信息加密传输等领域。
[0155]
dna单链一般链长较短,可以实现少量信息的存储,如标题、目录、标记等,dna双链的链长则可以达到5000bp以上,可以实现大量信息的存储,如图片、音视频文件等;质粒是dna的良好载体,具有稳定性、可扩增性和便于体内体外存储的特性,是dna信息存储的完美载体,通过质粒载体可以实现dna信息的复制及长期保存。pcr技术可将dna单链扩增为双链,再通过无缝克隆技术即可将全部dna信息链整合到质粒上,实现dna信息的多样化稳定存储。
[0156]
转化至微生物体内的重组质粒通过质粒抽提的相关方法即可完成提取,以质粒干粉或溶液的形式进行存储,形成信息存储池。因此,信息存储容量与重组质粒的种类及数量正相关,根据本技术方法不断新建重组质粒,即可实现信息存储量的扩大。
[0157]
对于体外存储的信息池——质粒,更便于对质粒进行各种生物操作,如酶切、dna片段插入等,也可通过dna探针设计等方式对质粒上的信息片段进行检索,并可直接用于测序分析,因此更加便于进行信息池的信息筛选与检索,远远优于传统的dna“冷数据”存储的方法。
[0158]
本实施例方法涉及生物信息学领域,针对dna数字信息存储需要大量专业领域知识的情况,提出一种可拓展的、形式多样的dna信息存储方法。
[0159]
本实施例方法中dna载体为质粒,信息存储稳定性强,dna信息的形式为单链和双链,可根据需要将不同形式的链整合到质粒上,有效提高信息存储的多样性。
[0160]
本实施例方法dna信息存储容量的扩大简单快捷,通过后续重组质粒的构建与添加,能够有效提高信息存储容量。
[0161]
本实施例方法的信息存储更加规范化、系统化,便于进行后续的信息筛选与检索。
[0162]
本发明实施例解决的痛点问题包括:
[0163]
dna信息存储稳定性低;
[0164]
dna信息存储形式单一,不具有多样性;
[0165]
dna信息存储容量有限,不便于扩容;
[0166]
dna信息存储系统化低,不便于信息检索与筛选。
[0167]
本实施例方法与现有技术的差异:
[0168]
dna信息存储的形式更加稳定且多样化;
[0169]
dna信息存储的重组质粒的制备流程更加系统化、规范化,提高效率;
[0170]
dna信息存储容量的扩大方便快捷,通过重组质粒的构建即可实现;
[0171]
本实施例的方法打破了dna信息作为“冷数据”进行存储的传统,可以在重组质粒池中实现信息的添加,也为后续实现信息的筛选和随机检索等提供必要的条件。
[0172]
具体应用场景:
[0173]
dna信息存储技术可用于数字信息存储、数字图书馆、dna硬盘、u盘等领域。
[0174]
可以实现常规的数字信息的写入与读出,可以提供给科研院所、研发机构,商业公司等科研项目。
[0175]
可以降低对微芯硅片的需求,使用该技术构建数字档案、数字信息库、数字图书馆等,可以提供给政府部门、图书馆、博物馆、档案管理机构等。
[0176]
可以实现即时访问,使用该技术构建dna硬盘、u盘等dna存储器,可以提供给通讯公司、科技公司等。
[0177]
本发明实施例拥有巨大的商业前景,对于数字信息存储行业有以下市场价值:
[0178]
1)本发明实施例可以用于有特殊需求的数字信息的存储,极大提高存储密度和时间;
[0179]
2)本发明实施例可以用于制造传统硬盘等存储器的替代品,满足通讯公司及科技公司的特定需求。
[0180]
本实施例在数字档案应用上的市场规模:我国数字化档案资源达2243万gb;
[0181]
在数字信息存储领域,可使用本技术的部门有全国各级国家综合档案馆、博物馆、图书馆等,实现全新的dna数字存储。
[0182]
简述该技术的发展规划。
[0183]
本发明实施例未来可应用至dna存储器、dna云端数据库、dna数字图书馆等,在全球范围内处于遥遥领先水平。
[0184]
本发明实施例通过信息保存及传输的极高安全性、稳定性,数据存储的先进性、前沿性等优势均可占据市场。
[0185]
以上内容是结合具体/优选的实施方式对本发明所作的进一步详细说明,不能认定本发明的具体实施只局限于这些说明。对于本发明所属技术领域的普通技术人员来说,在不脱离本发明构思的前提下,其还可以对这些已描述的实施方式做出若干替代或变型,而这些替代或变型方式都应当视为属于本发明的保护范围。在本说明书的描述中,参考术语“一种实施例”、“一些实施例”、“优选实施例”、“示例”、“具体示例”、或“一些示例”等的描述意指结合该实施例或示例描述的具体特征、结构、材料或者特点包含于本发明的至少一个实施例或示例中。在本说明书中,对上述术语的示意性表述不必须针对的是相同的实施例或示例。而且,描述的具体特征、结构、材料或者特点可以在任一个或多个实施例或示例中以合适的方式结合。在不相互矛盾的情况下,本领域的技术人员可以将本说明书中描述的不同实施例或示例以及不同实施例或示例的特征进行结合和组合。尽管已经详细描述了本发明的实施例及其优点,但应当理解,在不脱离专利申请的保护范围的情况下,可以在本文中进行各种改变、替换和变更。
再多了解一些
本文用于创业者技术爱好者查询,仅供学习研究,如用于商业用途,请联系技术所有人。