一种与多鳞鱚性别性状相关的qtl位点的构建方法
技术领域
1.本发明属于鱼类分子标记开发及其应用研究领域,具体涉及一种与多鳞鱚性别性状相关的qtl位点的构建方法。
背景技术:
2.多鳞鱚(sillago sihama)隶属鲈形目、鱚科、鱚属,为热带印度-西太平洋浅海鱼类,广泛分布于我国沿海地区。该鱼肉质细嫩,味道鲜美,不饱和脂肪酸含量十分丰富,营养价值高,深受消费者喜爱,具有重要的经济价值。该物种曾在我国近海捕捞作业中占有相当重要的位置,但由于过度捕捞,自然资源已面临枯竭,近年来市场售价持续上升,已从2003年的20~30元/公斤上升到目前的100~110元/公斤。
3.本技术发明人所在课题组早期突破了多鳞鱚全人工繁育技术,实现了其全人工规模化育苗,为下一步多鳞鱚良种选育奠定了基础。然而,多鳞鱚养殖苗种缺乏定向选育,存在因近亲繁殖导致的生长速度减慢、抗逆性能下降等性状退化风险,严重限制了其养殖业的健康发展,亟需开展分子遗传育种研究,解析重要经济性状相关遗传机制。近年来,学界在多鳞鱚缺氧、盐度的耐受性,群体遗传学,人工繁殖,基因组学等方面进行了探索研究,但尚未见分子标记开发及相关应用报道。
4.基于分子标记技术构建遗传连锁图谱是实现重要经济性状qtl定位和分子标记辅助育种有效途径。高通量测序技术简化了遗传标记分型方式,降低了实验成本,使得在众多水产养殖物种中通过构建高密度遗传图谱进行性状qtl精细定位成为可能。借助简化基因组测序技术,目前共有30多种鱼类构建了高密度遗传连锁图谱,并开展了经济性状qtl定位研究。多鳞鱚具有性别生长二态性,在同等养殖条件下,雌鱼生长速度快于同龄雄鱼,但目前尚未开展多鳞鱚性别性状等重要经济性状的分子标记鉴定和应用研究。因此,借助连锁图谱定位多鳞鱚性别性状具有重要的现实意义。
技术实现要素:
5.本发明的目的在于提供一种与多鳞鱚性别性状相关的qtl位点的构建方法,该方法利用获得的带有基因序列的snp分子标记,构建获得多鳞鱚高密度遗传连锁图谱,实现对多鳞鱚性别性状qtl定位,且定位位点稳定性好,使用范围广,在多鳞鱚性别性状分子标记辅助育种中具有良好的应用前景。
6.本发明的上述目的可以通过以下技术方案来实现:一种与多鳞鱚性别性状相关的qtl位点的构建方法,包括以下步骤:
7.(s1)选取性成熟多鳞鱚雌雄个体为亲本,父母本个体杂交产生f1代全同胞家系,提取各样品的基因组dna;
8.(s2)利用gbs技术对父母本以及161份f1代个体进行基因分型,并对snp标记过滤,共获得107406个多态性snp位点,接着以已知染色体水平的参考基因组为参考,根据标记的物理位置信息将其归并成对应的连锁群,去除非染色体上的标记,在每个遗传位置的标记
中取完整度最高(缺失率最低)的标记作为作图标记,最终获得4735个snp标记用于构建整合图谱;
9.(s3)利用步骤(s2)中的4735个筛选出来的snp标记构建遗传图谱;
10.(s4)对步骤(s1)中的父母本及f1代全同胞家系群体进行性别性状表型鉴定;
11.(s5)分析步骤(s3)构建的遗传图谱和步骤(s4)的性别性状表型鉴定结果,使用r/qtl对性别性状进行区间作图,得到与性别性状相关的qtl位点,共得出3个与性别性状相关的qtl位点,三个qtl位点均位于第18号染色体上,分别命名为qsex-18-1、qsex-18-2和qsex-18-3。
12.在上述与多鳞鱚性别性状相关的qtl位点的构建方法中:
13.优选的,步骤(s2)中对snp标记过滤时,去除分型比例低于30%的位点,去除杂合比例大于75%的位点,按理论比对标记位点的基因型比例进行卡方检验,p值小于0.001的位点视为严重偏分离位点并去除,保留分离类型为lmxll、nnxnp和hkxhk的标记,共获得107406个多态性snp位点。
14.本发明中提供的多鳞鱚snp分子标记,所述分子标记共计4735个,分布于24个连锁群上,具有如下所示的snp分子标记分布位点以及具有如下所示的多态性标签及连锁群上的分布。
15.进一步的,步骤(s2)中4735个snp标记具有如下所示的多态性标签及连锁群上的分布:
16.连锁群编号snp数量遗传长度(cm)平均遗传距离(cm)lg1232112.7660.486lg220399.5160.490lg322286.5210.390lg421883.4480.383lg5203100.4280.495lg619994.0020.472lg720399.1420.488lg8230114.2760.497lg920183.4560.415lg1016880.4600.479lg1120197.9990.488lg1218882.5410.439lg13227106.2700.468lg1419889.9410.454lg1521181.6090.387lg1618979.4690.420lg1716780.7540.484lg1822392.0190.413lg1916568.7310.417lg2021298.2250.463
lg2118073.9450.411lg2217496.4660.554lg2313165.0590.497lg2419087.7600.462total47352154.8030.455
17.作为本发明的一种优选的实施方式,本发明所述分子标记共计4735个,分布于24个连锁群上,所述snp分子标记是按照以下方法获得的:挑选体型良好的性成熟雌雄个体为亲本,父母本杂交产生f1代全同胞家系,提取各样品的基因组dna;利用gbs(genotyping-by-sequencing)技术分别对父母本以及f1代个体进行基因分型;随后对snp标记进行过滤,去除分型比例低于30%的位点,去除杂合比例大于75%的位点,按理论比对标记位点的基因型比例进行卡方检验,p值小于0.001的位点视为严重偏分离位点并去除,保留分离类型为lmxll、nnxnp和hkxhk的标记;共获得107406个多态性snp位点;以已知染色体水平的基因组为参考,根据标记的物理位置信息将其归并成对应的连锁群,去除非染色体上的标记,在每个遗传位置的标记中取完整度最高(缺失率最低)的标记作为作图标记,最终获得用于构建遗传图谱的4735个snp标记。
18.这些多鳞鱚snp分子标记还可以进一步在构建多鳞鱚遗传连锁图谱和性别性状定位方面应用。
19.优选的,步骤(s3)中使用perl-svg模型构建遗传图谱。
20.优选的,步骤(s4)中通过解剖性腺对步骤(s1)中的父母本及f1代全同胞家系群体进行性别性状表型鉴定。
21.优选的,步骤(s5)中使用r/qt1对性别性状进行区间作图,扫描步长为1cm,使用lod=4.0做为阈值筛选qtl。
22.优选的,步骤(5)中各qtl位点的区间如下:
23.qtl连锁群markerpeak position区间(cm)qsex-18-1lg18lg17_5959092.761080-12.886qsex-18-2lg18lg17_532224926.3837318.714-28.224qsex-18-3lg18lg17_806990138.6570728.531-48.475。
24.这些qtl位点还可以进一步的在多鳞鱚性别性状定位的遗传图谱、多鳞鱚性别性状分子标记辅助育种方面应用。
25.可以采用与性别性状相关的qtl位点分布在由上述多鳞鱚snp分子标记构建的多鳞鱚遗传图谱上,所述qtl位点分布如上所述。
26.本发明具有以下有益效果:本发明基于gbs技术筛选获得大量带有基因序列的snp标记,利用这些标记位点构建了多鳞鱚首张高密度遗传连锁图谱,并对多鳞鱚性别性状进行定位研究,获得了与多鳞鱚性别性状关联的分子标记,且定位位点稳定性好,适用范围广,在多鳞鱚性别性状分子标记辅助育种中具有良好的应用前景。
附图说明
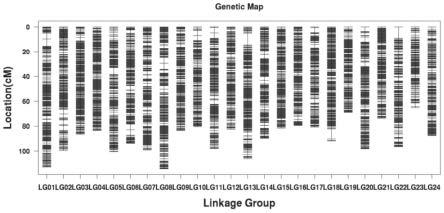
27.图1为实施例1中构建的多鳞鱚高密度snp分子遗传连锁图谱;
28.图2为实施例1中与多鳞鱚遗传连锁图谱中性别性状qtl定位的lod值曲线图。
具体实施方式
29.下面结合附图和具体实施例对本发明进行详细说明,但不应理解为本发明的限制。下述实施例中的实验方法,如无特殊说明,均为常规方法,下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。
30.实施例1
31.本实施例提供的与多鳞鱚性别性状相关的qtl位点的构建方法,包括以下步骤:
32.(s1)挑选体型良好的性成熟雌雄个体为亲本,父母本个体杂交产生f1代全同胞家系,采用常规试剂盒提取各样品的基因组dna;
33.试验在广东海洋大学东海岛养殖基地进行。挑选体型良好的性成熟雌雄个体为亲本,一对一繁殖构建全同胞家系。2018年6月进行人工繁殖,后将孵化的幼鱼移入室内水泥池进行培育,2019年6月挑选161尾f1代全同胞家系幼鱼及其父母本用于构建遗传图谱;同时,解剖f1代幼鱼观察性腺确定其性别。2019年6月采集选取的161尾f1代个体及父母本鳍条,提取基因组dna。用nanodrop 2000型分光光度计检测dna质量。
34.(s2)利用gbs技术对父母本以及161份f1代个体进行基因分型,并对snp标记过滤,去除分型比例低于30%的位点,去除杂合比例大于75%的位点,按理论比对标记位点的基因型比例进行卡方检验,p值小于0.001的位点视为严重偏分离位点并去除,保留分离类型为1mxll、nnxnp和hkxhk的标记,共获得107406个多态性snp位点;接着以已知染色体水平的参考基因组为参考,根据标记的物理位置信息将其归并成对应的连锁群,去除非染色体上的标记,在每个遗传位置的标记中取完整度最高(缺失率最低)的标记作为作图标记,最终获得4735个snp标记用于构建整合图谱。具体操作如下:
35.利用gbs技术共测序两个亲本和161个后代,质检合格的基因组dna被两种限制性内切酶ecori和niaiii(new england biolabs,ipswich,ma)消化,然后进行末端修复,加a-尾,并使用mltra
tm
dna library prep kit库(neb,usa)添加illumina测序接头。300-500bp的dna片段进行pcr扩增富集,最后使用ampure xp系统(beckman coulter,brea,ca,usa)对pcr产物进行纯化,使用agilent 2100生物分析仪(agilent,santa clara,ca)对测序文库进行检测,并使用实时pcr进行文库定量。测序在novaseq 6000测序仪上进行,采用pe150测序策略。
36.用fastp(0.18.0版本)对测序后的原始数据(raw data)进行过滤,过滤标准如下:1)去除含有未知核苷酸(n)≥10%的reads;2)去除phred质量评分≤20的碱基≥50%的reads;3)删除含接头的reads。过滤后的clean reads用于组装分析。统计结果显示,两个亲本的平均raw data为1.70g bp,161个子代样本中平均每个个体的raw data为550m bp。过滤后两个亲本的平均clean data为1.60g bp,161个子代样本中平均每个个体的clean data为534m bp,测序质量高(q20≥98%,q30%≥94%),样本gc分布正常(平均43%)。过滤后获得657.88m clean reads,reads长度为142bp。将测序数据与参考基因组进行比对,比对结果显示,所有reads与参考多鳞鱚基因组sraprjna642704的平均比对率为98%以上,可用于后续的变异检测及相关分析。
37.使用比对软件bwa(版本0.7.12)采用mem算法将过滤后的reads比对到参考基因组
(sraprjna642704)上,比对参数为-k 32-m;比对完之后结果使用软件picard(版本1.129)进行标记。使用变异检测软件gatk(版本3.4-46)进行群体snp检测,snp标记过滤标准为:去除分型比例低于30%的位点,去除杂合比例大于75%的位点,按理论比对标记位点的基因型比例进行卡方检验,p值小于0.001的位点视为严重偏分离位点并去除,保留分离类型为1mxll、nnxnp和hkxhk的标记。最终该项目共获得多态性位点107406个。接着以已知染色体水平的参考基因组为参考,根据标记的物理位置信息将其归并成对应的连锁群,去除非染色体上的标记,在每个遗传位置的标记中取完整度最高(缺失率最低)的标记作为作图标记,最终获得4735个snp标记用于构建整合图谱。
38.(s3)利用步骤(s2)的4735个筛选出来的snp标记构建遗传图谱,所述分子标记共计4735个,分布于24个连锁群上,总长度为2154.803cm,平均遗传距离为0.455cm(表1)。对于有染色体水平参考基因组的物种,根据标记的物理位置信息将其归并成对应的连锁群,并去除非染色体上的标记。我们在每个遗传位置的标记中取出一个完整度最高(缺失率最低)的标记作为作图标记,如果最高完整度条件下有多个标记,取第一个作为代表。随后使用joinmap4.1划分连锁群,对划分好的连锁群采用最大释然法进行排序,对排序后的结果进行校正,然后再使用回归算法进行排序,使用perl svg模型获得整合图谱(图1)。
39.表1 snp多态性标签及连锁群上的分布
[0040][0041][0042]
(s4)对步骤(s1)的父母本及f1代全同胞家系杂交群体进行性别性状表型鉴定:通过解剖性腺观察确定双亲及其后代个体性别,使用r/qtl的scanone()函数对性别性状进行区间作图(interval mapping,im),扫描步长为1 cm,使用lod=4.0做为阈值筛选qtl。得
到与性别性状相关的qtl位点在遗传连锁图谱上的lod曲线图,如图2所示。这些与性别性状相关的qtl位点,在双亲整合的遗传图谱上共检测到3个与性别性状关联的稳定qtl位点,如图2所示,图2为实施例1中与多鳞鱚遗传连锁图谱中性别性状qtl定位的lod值曲线图。共得出3个与性别性状相关的qtl位点,三个标记均位于第18号染色体上,分别命名为qsex-18-1、qsex-18-2和qsex-18-3。各位点的区间及解释的贡献率见表2。
[0043]
表2性别性状qtl相关信息
[0044][0045]
本发明基于gbs技术筛选获得大量带有基因序列的snp标记,利用这些标记位点构建了多鳞鱚高密度遗传连锁图谱,并对多鳞鱚性别性状进行定位研究,获得了与多鳞鱚性别性状相关的分子标记,该发明在多鳞鱚性别性状分子标记辅助育种中具有良好的应用前景。
[0046]
显然,本领域的技术人员可利用本发明进行各种改动和变型而不脱离本发明的精神和范围。这样,倘若本发明的这些修改和变型属于本发明权利要求及其等同技术的范围之内,则本发明也意图包含这些改动和变型在内。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。