1.本发明涉及物种鉴别技术领域,尤其是涉及一种对海南黄花梨及其近缘极易混淆种进行树种识别的方法及应用。
背景技术:
2.红木作为一类珍贵的木材资源,是经长期自然形成的经过归类的木材商品名,而不是树木分类学上某个树种的名称。随着国民经济的快速增长,人们对木质材料的追求不论质还是量都越来越高,红木市场近几年更是火爆异常,需求量极大的增长,而优质正宗的红木类资源非常短缺,于是以次充好、以假乱真的现象十分普遍。2000年,国家标准gb/t 18107-2000规定《红木》确定了5属8类33个树种。降香黄檀(dalbergia odorifera t.chen)俗称海南黄花梨被定为唯一的黄檀属-香枝木类,是我国特有珍贵红木树种。市场上还有另外一种黄檀属木材,材色、密度及构造与降香黄檀极为相似,也作为黄花梨在市场上流通,因其产地主要为越南,市场上俗称越南香枝(dalbergia tonkinensis prain)或越南黄花梨,它与降香黄檀同属但产地不同,品性悬殊、价格相差甚远,市场上常有人以越南香枝木假冒降香黄檀,这让消费者甚至部分专业鉴定人员难以识别,以致消费者、经营者均可能蒙受极大经济损失。采取与现代科学鉴察手段相结合的方法,寻找简便、高效、准确的方法来鉴别红木显得迫在眉睫。
3.有鉴于此,特提出本发明。
技术实现要素:
4.本发明的目的之一在于提供一种对海南黄花梨及其近缘极易混淆种进行树种识别的方法,以至少解决现有技术中存在的技术问题之一。
5.本发明的目的之二在于提供上述对海南黄花梨及其近缘极易混淆种进行树种识别的方法的应用。
6.为了实现本发明的上述目的,特采用以下技术方案:
7.本发明提供了一种对海南黄花梨及其近缘极易混淆种进行树种识别的方法,包括:
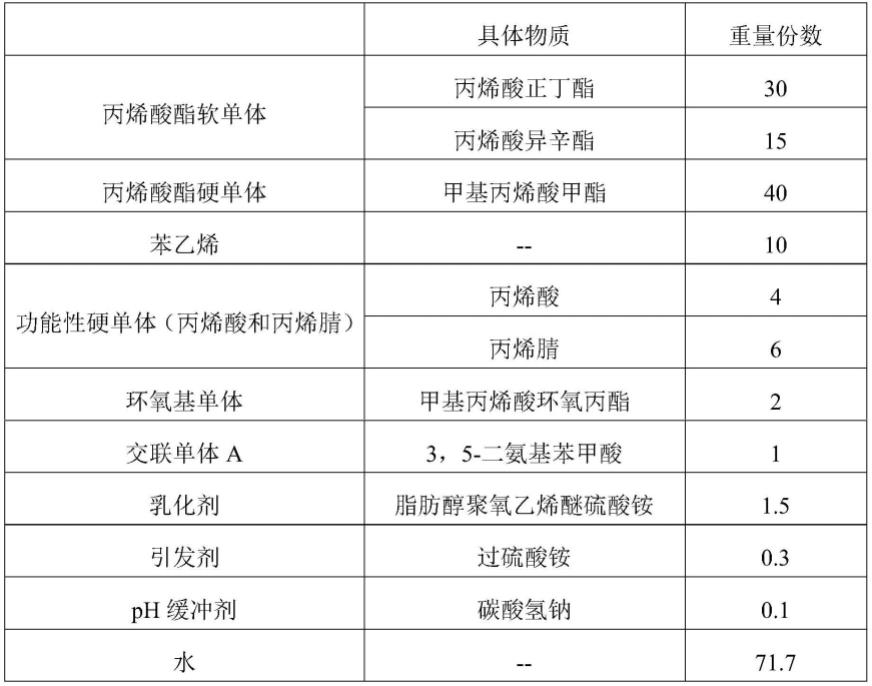
8.提取待鉴定树种的总dna,进行测序与序列拼接和组装,得到待鉴定树种的全叶绿体基因组序列;将所述待鉴定树种的全叶绿体基因组序列与标准参照序列比对并构建系统进化树;根据待鉴定树种与标准参照序列支的聚集结果,实现对海南黄花梨及其近缘极易混淆种的树种识别。
9.进一步的,所述标准参照序列包括海南黄花梨mt644131、越南黄花梨mt644133、巴西黑黄檀mt644130、黑黄檀mt644128、巴里黄檀mt644134、奥式黄檀mt644132、黄檀mt644129和mt644135交趾黄檀。
10.进一步的,取待鉴定树种的叶片,研磨后提取总dna;
11.优选地,通过qiagen dneasy plant maxi kit试剂盒提取待鉴定树种的总dna。
12.进一步的,将所述鉴定树种的总dna进行高通量测序,获取一定读长的片段序列;
13.优选地,应用illumina二代测序平台进行高通量测序;
14.优选地,所述一定读长的片段序列为8.0~10.0gb数据量的片段序列;
15.优选地,还包括按照n bases》10%和low-quality bases≤5》50%标准进行数据过滤的步骤,得到6.0~8.0gb数据量的片段序列。
16.进一步的,拼接时使用trimmomatic对所述片段序列进行质控;
17.优选地,利用bwa v0.7.17-r1188软件将所述片段序列与ncbi叶绿体数据库进行比对,然后使用picard v2.20.3程序筛选叶绿体序列;
18.优选地,筛选出的叶绿体序列通过spades v3.14.0以默认参数进行从头组装,并将输出的gfa文件导入到bandage v0.8.1中,得到待鉴定树种的全叶绿体基因组序列;
19.进一步的,还包括通过质体基因组注释软件对所述待鉴定树种的全叶绿体基因组序列进行基因结构注释的步骤。
20.进一步的,比对后去除空缺和比对模糊区域的序列,然后将得到的核苷酸序列进行串联并构建系统进化树;
21.优选地,使用mafft v7.464软件进行比对,使用trimal v1.4去除空缺和比对模糊区域的序列;
22.优选地,使用iq-tree v2.0构建系统进化树。
23.进一步的,用于比对和构建系统进化树的序列为待鉴定树种的保守蛋白编码基因最长的cds序列。
24.进一步的,当所述聚集结果为待鉴定树种与标准参照序列聚为一支,且支持率大于50%时,实现海南黄花梨及其近缘极易混淆种的树种识别。
25.此外,本发明还提供了上述的方法在鉴别海南黄花梨树种中的应用。
26.与现有技术相比,本发明具有如下有益效果:
27.本发明提供的对海南黄花梨及其近缘极易混淆种进行树种识别的方法,以叶绿体全基因组为基准进行鉴别,相较于单个或多个片段而言,可以提供更多的变异位点,实现精准鉴别。并且,在dna水平上进行的树种识别不易受地域、生长期及生长环境等的影响,具有较高的稳定性。此外,只需要有合格的dna样品和计算机操作平台即可开展工作,操作简单,成本低廉,应用本发明提供的方法可建立标准化的操作流程,具有广阔的应用前景。
附图说明
28.为了更清楚地说明本发明具体实施方式或现有技术中的技术方案,下面将对具体实施方式或现有技术描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图是本发明的一些实施方式,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
29.图1为本发明实施例提供的利用叶绿体全基因组序列构建的系统进化树;其中,8种不同颜色分布代表4个黄檀属类群及4个近缘极易混淆种,分支点上的数字代表支持率(%),低于50%不标注。
具体实施方式
30.除非本文另有定义,连同本发明使用的科学和技术术语应具有本领域普通技术人员通常理解的含义。术语的含义和范围应当清晰,然而,在任何潜在不明确性的情况下,本文提供的定义优先于任何字典或外来定义。在本技术中,除非另有说明,“或”的使用意味着“和/或”。此外,术语“包括”及其他形式的使用是0非限制性的。
31.一般地,连同本文描述的细胞和组织培养、分子生物学、免疫学、微生物学、遗传学以及蛋白和核酸化学和杂交使用的命名法和其技术是本领域众所周知和通常使用的那些。除非另有说明,本发明的方法和技术一般根据本领域众所周知,且如各种一般和更具体的参考文献中所述的常规方法来进行,所述参考文献在本说明书自始至终引用和讨论。酶促反应和纯化技术根据制造商的说明书、如本领域通常实现的或如本文所述来进行。连同本文描述的分析化学、合成有机化学以及医学和药物化学使用的命名法、以及其实验室程序和技术是本领域众所周知和通常使用的那些。
32.下面将结合实施例对本发明的技术方案进行清楚、完整地描述,显然,所描述的实施例是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
33.1、降香黄檀与东京黄檀植物特征对比:
34.降香黄檀,俗称海南黄花梨,花梨木,属蝶形花科(papilionaceae)黄檀属(dalbergia)植物。自然分布主要在我国海南岛西部及西南部平原或丘陵地区,北部琼山也有零星分布。降香黄檀为常绿半落叶乔木树种,其分布区域常年气温较高,年平均气温23~25℃,立地条件要求一般,在陡坡、山脊、裸露岩石、干旱瘦瘠地均能种植生长。
35.东京黄檀,俗称越南黄花梨,亦属于蝶形花科黄檀属植物。分布于越南各地,从北部的永福、北宁、华平、北江、广宁、宁平、河西、河内到南部的庆和、富安、嘉莱、广义、同奈等省均有分布,以北部较多,但也有认为主要在中部高地(西原地区)的嘉莱省。东京黄檀为常绿中等乔木,常见散落在海拔500米以下常绿湿润次生林中,适应深厚土壤层的平缓地带,喜光,萌发能力强。
36.将二者的主要植物形态学特征归纳总结如下表1,其二者在植物形态特征的差异主要表现在小叶的数量和花朵的雄雌花比例,且差异不显著,肉眼鉴别难度较大。
37.表1.海南黄花梨与越南黄花梨形态特征对比
[0038][0039]
2、降香黄檀与东京黄檀木材解剖结构特征对比:
[0040]
国家标准gb/t 18107~2000《红木》中规定,香枝木是蝶形花科黄檀属,材色红褐
至深红褐色,具有辛辣香味的一类商品材的名称。目前,红木市场上降香黄檀和东京黄檀木在气味、颜色、纹理、密度和结构上极为相似,区别微小,主要表现为:1)木材密度:降香黄檀密度高,东京黄檀密度低;2)木材颜色:降香黄檀颜色深,东京黄檀颜色浅;3)木材气味:降香黄檀香味浓,东京黄檀香味淡;4)木纹结构(“鬼脸”):降香黄檀鬼脸花纹显著,东京黄檀鬼脸花纹不显著。近年来,围绕着降香黄檀与东京黄檀树种的学术鉴定主要集中在二者的木材解剖结构对比,主要不同也仅仅表现在在管孔大小、轴向薄壁组织类型和木射线类型上存在较为显著的构造差异见。
[0041]
3、降香黄檀与东京黄檀木材化学成分对比:
[0042]
近年来,学者试图利用气质联用的方法来鉴定木材的种类。学者研究发现:降香黄檀与越南香枝两材种的化学成分差异显著,其中可挥发性成分差异不明显,无法以此准确区分材种,而脂溶性成分种类及含量差异显著,可准确区分两材种。
[0043]
综上三个鉴别维度所述,传统的植物分类鉴定方法很难区分降香黄檀和东京黄檀,从本质上把握其二者真正的生物学本质差异。基于此,本发明的发明人创造性地提供了一种基于叶绿体全基大组序列对海南黄花梨和越南黄花梨及其近缘极易混淆种进行鉴定的方法,该方法针对利用常规方法难以鉴别海南黄花梨和越南黄花梨及其近缘极易混淆种的问题,建立了一种基于叶绿体全基因组序列的树种识别方法,包括:
[0044]
提取待鉴定树种的总dna,进行测序与序列拼接和组装,得到待鉴定树种的全叶绿体基因组序列;将所述待鉴定树种的全叶绿体基因组序列与标准参照序列比对并构建系统进化树;根据待鉴定树种与标准参照序列支的聚集结果,实现对海南黄花梨及其近缘极易混淆种的树种识别。
[0045]
叶绿体是植物特有的进行光合作用的细胞器,具有自身独立的基大组。叶绿体基因组具有基大组小、结构稳定、编码区较密集、基因种类和数量较稳定等特点,因此易于测序、组装和比对。
[0046]
本发明以叶绿体全基因组为基准进行鉴别,相较于单个或多个片段而言,可以提供更多的变异位点,实现精准鉴别。并且,在dna水平上进行的树种识别不易受地域、生长期及生长环境等的影响,具有较高的稳定性。此外,只需要有合格的dna样品和计算机操作平台即可开展工作,操作简单,成本低廉,应用本发明提供的方法可建立标准化的操作流程,具有广阔的应用前景。
[0047]
可选地,所述标准参照序列包括海南黄花梨mt644131、越南黄花梨mt644133、巴西黑黄檀mt644130、黑黄檀mt644128、巴里黄檀mt644134、奥式黄檀mt644132、黄檀mt644129和mt644135交趾黄檀。
[0048]
确立上述标准参照序列的方法为,每个种从海南乐东尖峰国家热带林木种质资源库采集一株性状优良的植株,经鉴定无误后,测序获得其叶绿体基因组序列。上述8个标准参照序列已提交至genbank保存,科研工作者可免费下载直接用于鉴定。
[0049]
在一些优选的实施方式中,取待鉴定树种的叶片,研磨后提取总dna;
[0050]
优选地,通过qiagen dneasy plant maxi kit试剂盒提取待鉴定树种的总dna,方便、快捷,提取率高。
[0051]
作为优选,将所述鉴定树种的总dna应用illumina二代测序平台进行高通量测序,获取8.0~10.0gb数据量的片段序列。
[0052]
优选地,还包括按照n bases》10%和low-quality bases(≤5)》50%标准进行数据过滤的步骤,得到6.0~8.0gb干净数据(clean reads),避免了测序错误导致的叶绿体序列多态性,从而获得高质量的准确的叶绿体序列。
[0053]
在一些优选的实施方式中,进行序列拼接和组装时,使用trimmomatic对上述原始数据进行质控,利用对二代测序数据进行比对的金标准软件bwa v0.7.17-r1188软件将所有生成的序列与ncbi叶绿体数据库进行比对,然后使用picard v2.20.3程序筛选叶绿体序列。筛选出的叶绿体序列通过spades v3.14.0以默认参数进行从头组装,避免了通常使用的有参组装方法造成的序列结构性变异减少的影响,并将输出的gfa文件导入到bandage v0.8.1中,生成每个物种的最终叶绿体基因组,并通过质体基因组注释软件pga对其进行基因结构注释。
[0054]
需要说明的是,进行序列拼接和组装时,将修整过的片段序列匹配(map)到参考基因组上时,为确保准确性,极少数测序深度小于100
×
的区域通过pcr扩增和测序补全从而获得全叶绿体基因组序列。
[0055]
其中,以越南黄花梨绿体基因组(genbank登录号mn202179)为参考基因组。
[0056]
在一些优选的实施方式中,比对后去除空缺和比对模糊区域的序列,然后将得到的核苷酸序列进行串联并构建系统进化树;
[0057]
其中,使用了兼具比对速度和准确度的mafft v7.464软件进行比对,使用trimal v1.4去除空缺和比对模糊区域的序列,使用运算速度更快的iq-tree v2.0构建系统进化树。
[0058]
需要说明的是,用于比对和构建系统进化树的序列为待鉴定树种的保守蛋白编码基因最长的cds序列。
[0059]
对于待鉴定树种与标准参照序列支的聚集结果,当所述聚集结果为待鉴定树种与标准参照序列聚为一支,且支持率大于50%时,实现海南黄花梨及其近缘极易混淆种的树种识别。
[0060]
常规鉴定手段通常是形态学或是its区的序列多态性来鉴定和区分物种,但近缘物种常具有相似的性状表型,因此准确性不高。在区分物种的方法中,测序分析待比较物种的全基因组是最有效可靠的方法,但成本高、耗时长、难度大,故一般选用物种相对特异的its区来从dna层面区分物种,本方法也是在dna层面,但能提供更多有效信息,如通过对叶绿体全长序列进行比对,获得每个位点的比对信息,从而基于物种或分支特有的单核苷酸多态性位点和插入、缺失位点,更准确地鉴别近源物种。
[0061]
下面通过实施例对本发明作进一步说明。如无特别说明,实施例中的材料为根据现有方法制备而得,或直接从市场上购得。
[0062]
实施例1
[0063]
1、dna提取和测序
[0064]
本实施例采集了八种黄檀的新鲜叶片进行dna提取。植物叶片材料在中国乐东黎族自治县尖峰镇中国林业科学院热带林业研究所试验站(北纬18.69度,东经108.79度)采集。分别为:43年生的降香黄檀(dalbergia odorifera,原产于中国海南);13年生的交趾黄檀(dalbergia cochinchensis,来自泰国萨拉布里baan subprik,muak-lek,saraburi,thailand);5年生东京黄檀(dalbergia tonkinensis来至于越南凉山so’n,
vietnam);10年生奥氏黄檀(dalbergia oliveri,引种于柬埔寨菩萨pursat,cambodia);8年生巴里黄檀(dalbergia bariensis,引种于泰国孔敬khon kaen,thailand);35年生黑黄檀(dalbergia fusca,引种于中国云南;2年生巴西黑黄檀(dalbergia nigra,引种于巴西库里蒂巴curitiba,brazil);以黄檀(dalbergia hupeana,原产于江苏省南京市)为外参树种。
[0065]
采集海南黄花梨及其近缘极易混淆种的新鲜叶片,将100mg左右的样品加入液氮研磨充分的样品,迅速转入2ml离心管中,加入500μl裂解液1,65℃水浴20分钟,每隔5分钟轻轻震荡几次。12000g/min离心2分钟。将上清液转移到1.5ml离心管。加入200μl裂解液2,轻摇2min,使之充分混匀。在室温下以12000
×
g离心1分钟。将上清液转移至1.5ml离心管。添加500μl缓冲液,涡流5s。将裂解液装入mb旋转柱,并以12000
×
g离心1分钟。将离心液并重复上述步骤1次,以确保所有裂解液均已通过mb旋转柱。将mb旋转柱放入干净的2ml收集管。将650μl洗脱液1添加到mb自旋柱中。以12000
×
g离心1分钟。倒掉离心液,并将mb旋转柱放回相同的2毫升收集管中。将650μl洗脱液2添加到mb自旋柱中。以12000
×
g离心1分钟。倒掉离心液,并将mb旋转柱放入相同的2毫升收集管中。在高达16000
×
g的条件下离心2分钟。小心地将mb旋转柱放入新的1.5ml收集管中。向白色滤膜中心添加50~100μl eb缓冲液。静置10min。12000
×
g离心1分钟。丢弃mb旋转柱。洗脱液即为dna。
[0066]
经检测合格的总dna样品分别应用illumina二代测序平台进行高通量测序,每个样品获取数据量为8.0~10.0gb。并通过trimmomatic按照n bases》10%和low-quality bases≤5》50%标准进行数据过滤的步骤,得到6.0~8.0gb数据量的片段序列。
[0067]
利用bwa v0.7.17-r1188软件基于参数
‘
bwa mem-t 16-m cp_plant_ref.fa r1.fq.gz r2.fq.gz》cp.sam’,将所述片段序列与ncbi叶绿体数据库进行比对,然后使用picard v2.20.3程序基于参数
‘
picard samtofastq i=cp.bam f=cp_r1.fastq f2=cp_r2.fastq’,筛选叶绿体序列,将筛选出的叶绿体序列通过spades v3.14.0以默认参数进行从头组装,并将输出的gfa文件导入到bandage v0.8.1中,得到每个样本的全叶绿体基因组序列。
[0068]
通过质体基因组注释软件pga对每个样本的全叶绿体基因组序列进行基因结构注释。基于注释信息,提取每个样本以及标准参照序列中相同基因的cds序列,使用mafft v7.464软件以默认参数进行比对,并通过trimal v1.4基于参数
‘‑
fasta-gappyout’,去除空缺和比对模糊区域的序列,然后将得到的不同基因的核苷酸序列比对文件进行串联并使用iq-tree v2.0基于参数
‘‑
b 1000
‑‑
seed 12345-t 20’,构建系统进化树。
[0069]
当所选样本与标准参照序列聚为一支,且支持率大于50%时,实现海南黄花梨及其近缘极易混淆种的树种识别。
[0070]
最后应说明的是:以上各实施例仅用以说明本发明的技术方案,而非对其限制;尽管参照前述各实施例对本发明进行了详细的说明,本领域的普通技术人员应当理解:其依然可以对前述各实施例所记载的技术方案进行修改,或者对其中部分或者全部技术特征进行等同替换;而这些修改或者替换,并不使相应技术方案的本质脱离本发明各实施例技术方案的范围。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。