根瘤菌trna衍生的小rna及其调控植物结瘤的用途
1.优先权
2.本专利申请与2019年5月16日提交的美国临时专利申请序列号62/848,672相关并要求其优先权益,其内容在此通过引用整体并入本公开中。
3.政府支持声明
4.本发明是在2015-67013-22811和2018-67013-27425项的政府资助下完成的,这两项资助是由美国农业部国家食品与农业研究所(usda-nifa)授予的。政府对本发明享有一定的权利。
技术领域
5.本公开涉及提高豆科植物的结瘤效率并且用于将共生固氮能力扩展到非豆科植物。具体地,根瘤菌trna衍生的小rna片段(trf)及它们对引发大豆根瘤的影响提供了增强植物固氮能力的替代靶标和机制。
背景技术:
6.本部分所介绍的方面可能有助于更好地理解本公开。因此,这些陈述应据此解读,并且不应被理解成承认什么是或不是现有技术。
7.豆科植物是诸如大豆、苜蓿、三叶草、豌豆、豆角、扁豆、羽扇豆、牧豆树和花生的植物,它们的根部与细菌(特别是根瘤菌科)形成共生关系。根瘤菌脂-壳寡糖根结瘤(nod)因子是诱导使得根瘤形成的植物反应的关键信号分子。一旦感知植物黄酮类化合物,根瘤菌会分泌nod因子,诱导根毛在所述细菌周围卷曲,随后形成感染痕迹,使所述细菌能够穿透根部的皮层细胞形成根瘤。
8.豆科植物中的结瘤提供了可利用氮进入生物圈的主要渠道。更具体地说,所述植物以光合作用产生的三磷酸腺苷(atp)的形式为所述细菌提供营养和能量来源。作为回报,所述细菌将大气中的元素氮固定为氨,这是一种可被植物消化的可用形式的氮。这种称为共生固氮(snf)的相互作用很重要,因为它使所述植物宿主能够获得大气中的氮,从而为所述植物提供丰富的氮源。snf存在于被称为豆科植物根瘤的特殊根部器官中,是农业和自然生态系统中氮的主要来源。由于氮是最常限制绿色植物生长的养分,优化其应用是优化植物产量的关键。
9.由于可以通过固氮获得氮,豆科植物的蛋白质含量通常高于其他植物科。高蛋白质含量使豆科植物成为人类食物和动物饲料最重要的粮食作物之一。此外,豆科植物用于轮作实践,从而通过snf增加土壤的氮含量,以备未来生长季节使用,并减少所需的肥料量。这对种植者来说具有成本效益,并且可以减少氮径流。
10.为了优化豆科作物产量,重要的是提高结瘤效率和固氮效率,从而保持植物中足够的氮含量。因此,需要一种有效且具有成本效益的方法来改善豆科植物的结瘤,从而提高植物的产量潜力。
技术实现要素:
11.本发明提供转基因豆科植物、植物根部和植物细胞。在至少一个示例性实施方案中,此类豆科植物、植物根部和植物细胞包含至少一种突变以表达编码具有发夹结构的根瘤菌衍生rna的多核苷酸构建体,所述根瘤菌衍生rna在植物根部体内被切割成人工小rna片段(即“人工trf”)。所述人工小rna片段/trf下调或抑制作为结瘤负调控因子的豆科植物、植物根部或植物细胞的一个或多个靶基因。以此方式,与从相应接种的野生型豆科植物、植物根部或植物细胞产生的根瘤相比,所述转基因豆科植物、植物根部或植物细胞在接种根瘤菌时产生的根瘤增加了。在某些实施方案中,所述豆科植物、植物根部或植物细胞是大豆,并且所述人工小rna片段具有的核苷酸序列选自由以下组成的组:seq id nos:1-3,seq id nos:50-70和seq id nos:1-3、seq id nos:50-70的基本同源核酸序列。在至少一个示例性实施方案中,所述人工小rna片段的核苷酸序列是seq id no:1、seq id no:2、seq id no:3,或seq id nos:1-3的基本同源核酸序列。在替代实施方案中,所述豆科植物、植物根部或植物细胞是埃特里根瘤菌,并且所述人工小rna片段的核苷酸序列是seq id nos:34-49或seq id nos:34-49的基本同源核酸序列。
12.在其他实施方案中,所述至少一种突变还可包括所述豆科植物、植物根部或植物细胞的所述一个或多个靶基因中的至少一个的第二突变。就这样,所述第二突变可以被设计来下调或抑制所述一个或多个靶基因。此类靶基因可包含作为结瘤机制的负调控因子的任何宿主植物基因,例如,通过非限制性示例,gmrhd3a、gmrhd3b、gmham4a、gmham4b、gmlrx5,或前述任一项的直系同源物。还应理解的是,前述基因的一种或任何组合也可包含该第二突变。例如,所述第二突变可以在gmrhd3a和gmrhd3b上,或在gmham4a和gmham4b上。另外地或备选地,所述第二突变可以包括缺失。就这样,可以删除所有所述靶基因,或者一个或多个所述靶基因可以被删除,而其他靶基因只是被抑制或未修饰。
13.本发明还提供了增强豆科植物根部结瘤活性的方法。此类方法可包括抑制或下调宿主植物根部或细胞的一个或多个靶基因的表达,每种所述靶基因是所述宿主中结瘤的负调控因子,并用至少一种根瘤菌接种或已接种经修饰的植物宿主根以引发结瘤。在至少一个实施方案中,所述靶基因可以是以下中的一个或多个:rhd3、ham4、lrx5、rhd3的直系同源物、ham4的直系同源物和lrx5的直系同源物和/或所述至少一种根瘤菌的菌种或菌株选自以下组成的组:大豆慢生根瘤菌、埃特里根瘤菌、苜蓿中华根瘤菌、豌豆根瘤菌、拟山黄麻根瘤菌和百脉根根瘤菌。在至少一个示例性实施方案中,所述宿主根部或细胞是大豆,并且每个靶基因选自以下组成的组:gmrhd3a、gmrhd3b、gmham4a、gmham4b和gmlrx5。
14.抑制或下调步骤可以包括引入和表达编码根瘤菌衍生rna的第一多核苷酸构建体,所述根瘤菌衍生rna用于在所述植物根部中产生下调至少一个所述靶基因的一个或多个人工小rna片段(“人工trf”)。在至少一个实施方案中,此类根瘤菌衍生rna可具有发夹结构,在体内被切割以在所述植物根部中产生人工trf。此外,每个所述人工trf可以具有的核苷酸序列选自由以下组成的组:seq id nos:1-3、seq id nos:34-70,或seq id nos:1-3或seq id nos:34-70的基本同源核酸序列。更进一步地,每个所述人工trf可以具有的核苷酸序列为seq id no:1,seq id no:2,seq id no:3,或seq id no:1、seq id no:2或seq id no:3的基本同源核酸序列。另外地或备选地,所述抑制或下调步骤可包括构建crispr-cas9载体以沉默一个或多个所述靶基因并在所述宿主植物根部或细胞中表达所述载体。在至少
一个示例性实施方案中,所述宿主根或细胞是大豆,并且每个靶基因选自以下组成的组:gmrhd3a、gmrhd3b、gmham4a、gmham4b和gmlrx5。在又一个实施方案中,其中所述靶基因是gmrhd3a、gmrhd3b或两者,所述crispr-cas9载体包含seq id nos:74和75;在所述靶基因是gmham4a、gmham4b或两者的情况下,所述crispr-cas9载体包含seq id nos:76和77;和/或在至少一个靶基因是gmlrx5的情况下,所述crispr-cas9载体包含seq id nos:78和79。在至少一个示例性实施方案中,所述抑制或下调步骤包括:引入和表达编码根瘤菌衍生rna的第一多核苷酸构建体,所述根瘤菌衍生rna在体内被切割以在所述植物根部中产生一个或多个人工小rna片段;以及在所述宿主植物根部或细胞中表达crispr-cas9载体,所述crrispr-cas9载体构建为沉默一个或多个以下宿主基因:gmrhd3a、gmrhd3b、gmrhd3a/gmrhd3b、gmham4a、gmham4b、gmham4a/gmham4b、gmlrx5。
15.所述方法的其他实施方案通过将第二多核苷酸构建体引入所述宿主植物根部或细胞并表达来抑制或下调所述一个或多个宿主基因的表达,其中所述第二多核苷酸构建体选择性地抑制所述一个或多个靶基因的表达。本公开的某些方法可以另外地或备选地包括所述至少一种根瘤菌,其包含表达第三多核苷酸构建体的根瘤菌微生物的一个或多个菌种或菌株,如与相应的野生型根瘤菌微生物中转移核糖核酸衍生片段(trf)的生成相比,所述第三多核苷酸构建体增强了一个或多个trf的生成。
16.在本公开中还提供了新型构建体,在这些构建体的实施方案中,其编码折叠成发夹结构并在体内切割成人工小核糖核酸分子的核糖核酸转录本,当所述人工小核糖核酸分子在豆科植物根部中表达时,增强根瘤形成。在至少一个非限制性实施方案中,所述人工小核糖核酸分子具有的核苷酸序列选自由以下组成的组:seq id nos:1-3、34-49和50-70。此类新型构建体可用于本公开的任何所述转基因植物、植物根部、植物细胞和方法中。
17.更进一步,某些实施方案提供了一种根瘤菌微生物,与相应的野生型根瘤菌微生物中转移核糖核酸衍生片段(trf)的生成相比,所述根瘤菌微生物表现出选择性扩增一个或多个trf的生成。例如,此类根瘤菌微生物可表达增强生成一个或多个trf的多核苷酸构建体,并且在至少一个实施方案中,此类trf可以抑制或下调宿主生物的至少一个靶基因(例如之前描述的靶基因)的表达。在至少一个实施方案中,所述宿主生物的所述至少一个靶基因选自以下组成的组:rhd3、ham4、lrx5或它们的直系同源物。在又一个实施方案中,所述宿主生物是大豆,并且每个所述至少一个靶基因选自由以下组成的组:gmrhd3a、gmrhd3b、gmham4a、gmham4b和gmlrx5。某些其他实施方案提供包含本文所述的根瘤菌微生物的一个或多个菌种或菌株的化合物,其中所述trf抑制或下调宿主植物根部的至少一个靶基因的表达并且将所述化合物配制在接种剂组合物中。
18.序列表的简要说明
19.seq id no:1是大豆慢生根瘤菌衍生trf的核酸序列:
20.cgaucccuugugcgcccacca;
21.seq id no:2是大豆慢生根瘤菌衍生trf的核酸序列:
22.cgauucccuucacccgcucca;
23.seq id no:3是大豆慢生根瘤菌衍生trf的核酸序列:
24.uggggaaugguguaacgguag;
25.seq id no:4是靶向rhd3(rhd3_psd_f)的寡核苷酸人工核酸序列:
26.attgaggtgaagttggctgaatg;
27.seq id no:5是靶向rhd3(rhd3_psd_b)的寡核苷酸人工核酸序列:
28.aaccattcagccaacttcacctc;
29.seq id no:6是靶向ham4(ham4_psc_f)的寡核苷酸人工核酸序列:
30.attgcctgtggggatgagggaac;
31.seq id no:7是靶向ham4(ham4_psc_b)的寡核苷酸人工核酸序列:
32.aacgttccctcatccccacaggc;
33.seq id no:8是靶向lrx5(lrx5_psc_f)的寡核苷酸人工核酸序列:
34.attgttgaaactgctctacgaac;
35.seq id no:9是靶向lrx5(lrx5_psc_b)的寡核苷酸的人工核酸序列:
36.aacgttcgtagagcagtttcaac;
37.seq id no:10是野生型大豆慢生根瘤菌衍生trf的核酸序列:
38.caaatcctatccccgcaacca;
39.seq id no:11是野生型大豆慢生根瘤菌衍生trf的核酸序列:
40.caaatcctctcgctccgacca;
41.seq id no:12是野生型大豆慢生根瘤菌衍生trf的核酸序列:
42.caatcctgtctggcagcacca;
43.seq id no:13是野生型大豆慢生根瘤菌衍生trf的核酸序列:
44.cgaatccagctgccccgacca;
45.seq id no:14是野生型大豆慢生根瘤菌衍生trf的核酸序列:
46.cgaatccaggttccccagcca;
47.seq id no:15是野生型大豆慢生根瘤菌衍生trf的核酸序列:
48.cgaatcccactctctccgcca;
49.seq id no:16是野生型大豆慢生根瘤菌衍生trf的核酸序列:
50.cgaatccctccctctccgcca;
51.seq id no:17是野生型大豆慢生根瘤菌衍生trf的核酸序列:
52.cgaatcctgccactccgacca;
53.seq id no:18是野生型大豆慢生根瘤菌衍生trf的核酸序列:
54.cgaatcgtgtcgggtgcgcca;
55.seq id no:19是野生型大豆慢生根瘤菌衍生trf的核酸序列:
56.cgagcccacccaactgtacca;
57.seq id no:20是野生型大豆慢生根瘤菌衍生trf的核酸序列:
58.cgagccctgccactcctgcca;
59.seq id no:21是野生型大豆慢生根瘤菌衍生trf的核酸序列:
60.cgagtcatcccgggatcgcca;
61.seq id no:22是野生型大豆慢生根瘤菌衍生trf的核酸序列:
62.cgagtccctccgagcgcacca;
63.seq id no:23是野生型大豆慢生根瘤菌衍生trf的核酸序列:
64.cgatcccgtctggctccacca;
65.seq id no:24是野生型大豆慢生根瘤菌衍生trf的核酸序列:
66.cgatccctgtcgtgcccacca;
67.seq id no:25是野生型大豆慢生根瘤菌衍生trf的核酸序列:
68.cgatcccttgtgcgcccacca;
69.seq id no:26是野生型大豆慢生根瘤菌衍生trf的核酸序列:
70.cgattcccttcacccgctcca;
71.seq id no:27是野生型大豆慢生根瘤菌衍生trf的核酸序列:
72.cgattcccttcgcccgctcca;
73.seq id no:28是野生型大豆慢生根瘤菌衍生的trf的核酸序列:
74.cgattccgcccctgggcacca;
75.seq id no:29是野生型大豆慢生根瘤菌衍生trf的核酸序列:
76.gcgctcgtggcggaactggta;
77.seq id no:30是野生型大豆慢生根瘤菌衍生trf的核酸序列:
78.gcgggtgtagctcagtggtag;
79.seq id no:31是野生型大豆慢生根瘤菌衍生trf的核酸序列:
80.tcctcggtagctcagcggtag;
81.seq id no:32是野生型大豆慢生根瘤菌衍生trf的核酸序列:
82.tggggaatggtgtaacggtag;
83.seq id no:33是野生型大豆慢生根瘤菌衍生trf的核酸序列:
84.ttggtagacgcaagggactta;
85.seq id no:34是野生型埃特里根瘤菌衍生trf的核酸序列:
86.caaatccctcctccgctacca;
87.seq id no:35是野生型埃特里根瘤菌衍生trf的核酸序列:
88.caagtcttcccgggcccacca;
89.seq id no:36是野生型埃特里根瘤菌衍生trf的核酸序列:
90.cgagtccaggttccccagcca;
91.seq id no:37是野生型埃特里根瘤菌衍生trf的核酸序列:
92.agtaggtcagagcagaggaat;
93.seq id no:38是野生型埃特里根瘤菌衍生trf的核酸序列:
94.agtaggtcagagcagaggaat;
95.seq id no:39是野生型埃特里根瘤菌衍生trf的核酸序列:
96.agtaggtcagagcagaggaat;
97.seq id no:40是野生型埃特里根瘤菌衍生trf的核酸序列:
98.caagtcttcccgggcccacca;
99.seq id no:41是野生型埃特里根瘤菌衍生trf的核酸序列:
100.cgaatcactccactccgacca;
101.seq id no:42是野生型埃特里根瘤菌衍生trf的核酸序列:
102.cgaatccggcccggggagcca;
103.seq id no:43是野生型埃特里根瘤菌衍生trf的核酸序列:
104.cgagccctgctgcccctgcca;
105.seq id no:44是野生型埃特里根瘤菌衍生trf的核酸序列:
106.cgagtctctcatcgcccacca;
107.seq id no:45是野生型埃特里根瘤菌衍生trf的核酸序列:
108.gattttcattctgtaaagagg;
109.seq id no:46是野生型埃特里根瘤菌衍生trf的核酸序列:
110.gattttcattctgtaaagagg;
111.seq id no:47是野生型埃特里根瘤菌衍生trf的核酸序列:
112.gtaggtcagagcagaggaatc;
113.seq id no:48是野生型埃特里根瘤菌衍生trf的核酸序列:
114.gtaggtcagagcagaggaatc;
115.seq id no:49是野生型埃特里根瘤菌衍生trf的核酸序列:
116.gtaggtcagagcagaggaatc;
117.seq id no:50是大豆慢生根瘤菌衍生trf的核酸序列:
118.caaauccuauccccgcaacca;
119.seq id no:51是大豆慢生根瘤菌衍生trf的核酸序列:
120.caaauccucucgcuccgacca;
121.seq id no:52是大豆慢生根瘤菌衍生trf的核酸序列:
122.caauccugucuggcagcacca;
123.seq id no:53是大豆慢生根瘤菌衍生trf的核酸序列:
124.cgaauccagcugccccgacca;
125.seq id no:54是大豆慢生根瘤菌衍生trf的核酸序列:
126.cgaauccagguuccccagcca;
127.seq id no:55是大豆慢生根瘤菌衍生trf的核酸序列:
128.cgaaucccacucucuccgcca;
129.seq id no:56是大豆慢生根瘤菌衍生trf的核酸序列:
130.cgaaucccucccucuccgcca
131.seq id no:57是大豆慢生根瘤菌衍生trf的核酸序列:
132.cgaauccugccacuccgacca;
133.seq id no:58是大豆慢生根瘤菌衍生trf的核酸序列:
134.cgaaucgugucgggugcgcca;
135.seq id no:59是大豆慢生根瘤菌衍生trf的核酸序列:
136.cgagcccacccaacuguacca;
137.seq id no:60是大豆慢生根瘤菌衍生trf的核酸序列:
138.cgagcccugccacuccugcca;
139.seq id no:61是大豆慢生根瘤菌衍生trf的核酸序列:
140.cgagucaucccgggaucgcca;
141.seq id no:62是大豆慢生根瘤菌衍生trf的核酸序列:
142.cgagucccuccgagcgcacca;
143.seq id no:63是大豆慢生根瘤菌衍生trf的核酸序列:
144.cgaucccgucuggcuccacca;
145.seq id no:64是大豆慢生根瘤菌衍生trf的核酸序列:
146.cgaucccugucgugcccacca;
147.seq id no:65是大豆慢生根瘤菌衍生trf的核酸序列:
148.cgauucccuucgcccgcucca;
149.seq id no:66是大豆慢生根瘤菌衍生trf的核酸序列:
150.cgauuccgccccugggcacca;
151.seq id no:67是大豆慢生根瘤菌衍生trf的核酸序列:
152.gcgcucguggcggaacuggua;
153.seq id no:68是大豆慢生根瘤菌衍生trf的核酸序列:
154.gcggguguagcucagugguag;
155.seq id no:69是大豆慢生根瘤菌衍生trf的核酸序列:
156.uccucgguagcucagcgguag;
157.seq id no:70是大豆慢生根瘤菌衍生trf的核酸序列:
158.uugguagacgcaagggacuua;
159.seq id no:71是野生型大豆慢生根瘤菌trna:
160.gggcgcauagcucagcgggagagcguucccuucacacgggagagguccaagguucgaucccuugugcgcccacca;
161.seq id no:72是野生型大豆慢生根瘤菌trna:
162.gcggguguagcucaaugguagagcagcagccuuccaagcugaauacgaggguucgauucccuucacccgcucca;
163.seq id no:73是野生型大豆慢生根瘤菌trna:
164.uggggaaugguguaacgguagcacaacagacucugacucuguuugucuugguucgaauccagguuccccagcca;
165.seq id no:74是用于过表达构建体rhd3b(rhd3b_cds_f)的引物的人工核酸序列:gcgcgtcgacatggcaaatagtgagacttgttg;
166.seq id no:75是用于过表达构建体rhd3b(rhd3b_cds b)的引物的人工核酸序列:gcgctctagactactcatcttttaatggacttgc;
167.seq id no:76是用于过表达构建体ham4a(ham4a_cds_f)的引物的人工核酸序列:ttgcctcgagatgagagttcccgttccctcatcc;
168.seq id no:77是用于过表达构建体ham4a(ham4a_cds_b)的引物的人工核酸序列:gcttggtaccctaacagcgccatgctgacgtggca;
169.seq id no:78是用于过表达构建体lrx5(lrx5_cds_f)的引物的人工核酸序列:gcgcgtcgacatgatgatgatgatgaagaagaagg;
170.seq id no:79是用于过表达构建体lrx5(lrx5_cds_b)的引物的人工核酸序列:gcaatctagactaataaaagggtggtggtggaggc。
171.除上述内容外,上述序列以计算机可读形式(编码在一起提交的文件中)提供,并通过引用并入本文。根据37c.f.r.
§
1.821(f),以计算机可读形式记录的信息与上面提供的
书面序列表相同。
附图说明
172.根据以下对本公开各种示例性实施方案的详细描述,所公开的实施方案和本文包含的其他特征、优点和方面,以及实现它们的问题将变得显而易见。结合附图将更好地理解此类详细描述,其中:
173.图1是调控大豆结瘤从而调控根瘤数的机制的示意图,其中如本文所述,标记为x的线表示基于植物的结瘤负调控,标记为y的线表示基于根瘤菌的结瘤正调控(aon=结瘤的自动调控;non=结瘤的氮调控;ric1和ric2=氮抑制clavata3/胚胎周围区域相关(cle)肽1和2);
174.图2显示了与根瘤菌和根瘤中根瘤菌trf的大小、位置和丰度相关的图形数据,分图a至分图d表示自由生存的根瘤菌(usda110菌株)(
●
)以及由usda110(
■
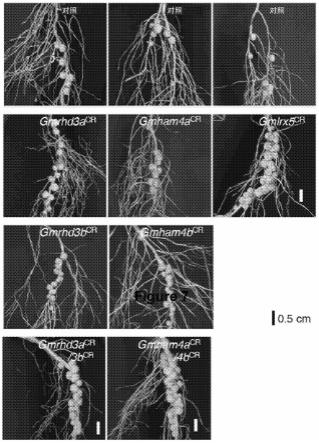
和
▲
)诱导的大豆根瘤中根瘤菌trf的大小分布和相对丰度的数据,分图e至分图h表示usda110诱导的大豆根瘤中21-nt trf沿根瘤菌trna的位置和丰度的数据,频率反映了21-nt trf沿单个trna的每个位点的相对覆盖率,并且分图i至分图k示出了设置在发夹结构中的三种野生型根瘤菌trna的结构和由相应的trna产生的三种trf的物理位置,如黑线之间所示(分别是bj-trf001/seq id no:1、bj-trf002/seq id no:2和bj-trf003/seq id no:3),其中分图i具有seq id no:71,分图j具有seq id no:72,并且分图k具有seq id no:73;
175.图3a是基于trna序列(每个命名格式为“bj(根瘤菌菌种的缩写)
”‑“
通过特定trna拷贝转移的氨基酸
”‑“
通过识别多个密码子转移相同氨基酸的trna顺序
”‑“
(condo)”)制备的大豆慢生根瘤菌基因组菌株usda110k中trna系统进化关系的邻接树图,与节点相邻的数字表示1000次重复测试的步长值;
176.图3b和图3c示出了与根瘤菌和根瘤中根瘤菌trf的大小分布和丰度相关的图形数据,x轴表示范围为18-24nt的主要根瘤菌trf的大小,y轴表示大小不同的所述trf的相对丰度;在所有情况下,下方图线代表自由生存的根瘤菌(菌株usda110)中根瘤菌trf的大小分布和相对丰度,而标记为a和b的图线分别代表大豆10-dpi根瘤和20-dpi根瘤菌中根瘤菌trf的大小分布和相对丰度;
177.图4a和图4b显示了与大豆根瘤中21-nt trf沿根瘤菌trna的物理位置和丰度相关的数据,其中10-dpi根瘤和20-dpi根瘤是由usda 110诱导的,示出的频率反映了所述21-nt trf沿单个trna的每个位点的相对覆盖率,每个x轴代表大豆慢生根瘤菌usda110中trna的核苷酸位置,每个y轴表示沿所述trna由所述21-nt trf覆盖的单个核苷酸的频率,所述21-nt trf衍生自usda110诱导的所述10-dpi根瘤或20-dpi根瘤中的trna;
178.图5a显示的图形数据说明了,通过茎环定量rt-pcr测量的所述三种trf在自由生存的大豆慢生根瘤菌(b.j.)usda110(1)、10天和20天接种后(dpi)根瘤(分别为2和3)中丰度,其值显示成三个生物学重复的平均值
±
se(一个设定为“1”,其他的相应调整),星号表示p《0.01的显著性水平(学生t检验);
179.图5b显示的图形数据说明了,通过定量rt-pcr测量的,已识别的trf靶基因在10天和20天的根瘤(分别为2和4)以及未接种的大豆根部(1和3)中的表达,其值显示成三个生物学重复的平均值
±
se(一个设定为“1”,其他的相应调整),星号表示p《0.01显著性水平(学
生t检验);
180.图5c显示了大豆根瘤中三种根瘤菌trf富集后的凝胶,其中显示的丰度相对于通过茎环rt-pcr检测的大豆慢生根瘤菌基因bll631的转录本在自由生存的大豆慢生根瘤菌菌株usda110、10-dpi根瘤和20-dpi根瘤中的丰度,使用不添加逆转录酶的20-dpi rna样本的反应作为阴性对照;单个trf的茎环rt-pcr产物的特异性通过对凝胶上加载的不同样本的扩增片段和等量总rna进行测序来确认,以使得根瘤菌rrna和大豆rrna的不同大小和相对丰度可视化;
181.图6a至图6c显示了本公开的三种已识别的trf、它们的假定靶转录本,以及在所述20-dpi根瘤中检测到的切割位点和频率(用箭头和比率表示);
182.图6d显示了如本文识别的人工小rna的序列,即asrna001(seq id no:1)和asrna001
*
,以及bj-trf001靶位点,并且图6e显示的图形数据说明了,在转化后20小时,与asr001
*-转基因根原生质体中的表达相比,靶宿主基因gmrhd3a/3b在大豆asr001-转基因根部原生质体中的表达降低,其中相对于gmcos4的基因表达水平通过qrt-pcr检测,并显示为三个生物学重复的平均值
±
标准误差(在每组中,一个样本的表达水平设置为1,其他样本的表达水平进行相应调整;
*
和
**
分别表示p《0.05和p《0.01的显著性差异(t检验);
183.图6e显示了根据本公开识别的根瘤菌trf靶基因的mrna内的切割位点,其中20-dpi根瘤和未接种根部中,所述5种大豆基因的mrna内切割位点的分布(x轴)以及切割时间(y轴)通过对rlm-race产物进行测序检测;指向方框内核苷酸之间位点的点代表大豆基因的切割位点,所述大豆基因的5'utr和3'utr(标记为u)、内含子(标记为i)和外显子(标记为e)中的一些、它们的mrna和已识别的trf靶位点由各自的标记、比对和箭头表示;
184.图7a显示了位点特异性crispr-cas9编辑gmrhd3a/3b、gmham4a/4b和gmlrx5产生的毛状根部突变序列,用于编辑的每个基因的野生型外显子序列和相应的氨基酸序列显示在每组的顶部,用于设计被引导rna的原始间隔区相邻基序(pam)加下划线,并且由crispr-cas9产生的核苷酸缺失、插入和替换的每种都位于方框内(
□
);星号表示共有核苷酸;
185.图7b,分图a至分图c显示了所述转基因根部中图7a的根瘤菌trf靶标和所述靶标的crispr-cas9-编辑形式的图形描述,其中显示的所述野生型(wt)trf靶标和crispr-cas9编辑形式的靶标相对于gmelf1b的基因表达水平通过qrt-pcr检测,并显示为三个技术性重复的平均值
±
s.e.,所述转基因根部样本与图8a中描述的相同,均在28dpi时收集,对照是用于培育crispr-cas9敲除的空载体的转基因根部,并且在每组中,一个样本的表达水平设置为1,其他样本的表达水平进行相应调整(
“‑
1”和
“‑
2”表示具有使用相同引导rna由crisr-cas9编辑的相同基因或一对重复基因的两个毛状根部,每个具有三个技术性重复;
186.图8a显示了五个基因(gmrhd3a、gmrhd3b、gmham4a、gmham4b和gmlrx5)的每个基因和所述两个重复基因对(gmrhd3a/gmrhd3b和gmham4a/gmham4b)两个拷贝的突变体根部的照片,与所述对照相比,每种敲除情况的根瘤数均增加;
187.图8b显示了图8a中举例说明的突变体和对照的根瘤数图,所有数据点均由点表示并显示为箱线图,显示了接种后28天收集的三个生物学重复(每个重复有12株植物)95%至5%的间隔(星号表示p《0.01(学生t测试)的显著性水平);显示为三个独立实验(每个实验n=12)的平均值
±
标准差;
188.图9a显示了被工程化以过表达本公开的三种trf靶标(gmrhd3b、gmham4a和
gmlrx5)的转基因根部的照片,所有突变体相对于对照产生的根瘤数减少;图9b显示了图9a中举例说明的突变体和对照的根瘤数图(所有数据点由点表示并显示为箱线图,显示了接种后28天收集的三个生物学重复(每个重复为12株植物)95%至5%的间隔(星号表示p《0.01的显著性水平(学生t-检验);显示为三个独立实验(每个实验n=12)的平均值
±
标准差);并且图9c显示了与空载体转基因对照相比,毛状根部中根瘤菌trf靶标过表达相关的图形数据,
**
表示显著性差异(t检验,p《0.01),其中相对于gmelf1b的基因表达水平由qrt-pcr检测,显示为三个生物学重复的平均值
±
误差,并且在每组中,一个样本的表达水平设置为1,其他样本的表达水平进行相应调整;
189.图10a显示的图形数据与以下情况相关:与空载体转基因对照(c)中根瘤菌trf靶标表达相比,抑制根瘤菌trf功能的sttm毛状根部(s)中根瘤菌trf靶标表达增强,其中相对于gmelf1b的基因表达水平通过qrt-pcr检测,显示为三个生物学重复的平均值
±
s.e.,所有样本均在28dpi收集;图10b显示了转基因根部的照片,其被工程化以通过sttm使单个trf沉默;图10c显示了图10a中所示根部的根瘤数图,所有数据点由点表示并显示为箱线图,显示了接种后28天收集的三个生物学重复(每个重复为12株植物)的95%至5%的间隔;并显示为三个独立实验(每个实验n=12)的平均值
±
标准差);并且图10d显示了sttm转基因根部和根瘤中与根瘤菌trf以及识别的靶基因表达相关的图形数据,其中所述trf和大豆基因的表达水平相对于gmelf1b的表达水平通过qrt-pcr检测,显示为三个生物学重复的平均值
±
s.e.,对照是用于培育所述sttm trf沉默根部的空载体的转基因根部,所有样本在28dpi时采集(图10a、图10c和图10d,星号表示p《0.01的显著性水平(学生t测试),并且在图10a和图10d的每组中,一个样本的表达水平设置为1,其他样本的表达水平进行相应调整);
190.图11a和图11b显示了在根瘤菌感染大豆根毛的早期所述根瘤菌trf丰度及其靶基因的表达,图11a显示的图形数据表示了,与通过茎环rt-qrt-pcr检测到的自由生存的根瘤菌(c)的trf相比,相对于根毛(未标记)中根瘤菌bll631转录本的根瘤菌trf的富集,图11b示出的图形数据显示了,与通过qrt-pcr检测的未接种根毛中的表达相比,根毛中相对于gmelf1b的根瘤菌trf靶标的表达降低(图11a和图11b中的值显示为三个生物学重复的平均值
±
s.e.,一个样本的值设置为1,其他样本的值进行相应调整;星号表示在p《0.05(t-测试)的显著性差异;
191.图12显示的图形数据与过表达和sttm转基因毛状根部中根毛数和长度相关,其中分图a显示了6dpi时过表达所述根瘤菌trf靶基因的大豆毛状根部与用所述空载体转化的毛状根部之间的比较,并且分图b显示了6dpi时抑制所述根瘤菌trf功能的大豆sttm毛状根部与用所述空载体转化的毛状根部之间的比较(数值显示为三个独立实验(每个实验,n=25)的平均值
±
s.d.;
192.图13显示的数据与大豆中根瘤菌trf及其靶标对早期根瘤菌感染的调控相关,分图a和分图b显示了过表达所述根瘤菌trf靶标的根毛和阴性对照(分图a)之间的形态差异以及抑制所述根瘤菌trf功能的sttm根毛和阴性对照(分图b)之间的形态差异,并且分图c显示的图形数据与如分图a和分图b所示样本中带有感染灶的变形根毛和卷曲根毛的定量相关(数值显示为三个生物学重复的平均值
±
sd(每个重复n=25个毛状根部),星号表示p《0.05的显著性水平(学生t测试);
193.图14a至图14c显示的图形数据与通过劫持宿主rnai机制的根瘤菌trf引导基因调
控相关,图14a示出的数据与在28dpi时用茎环qrt-pcr在amir-trf001(2)和amir-trf003(3)转基因根部及各自的空载体转基因根部(1)中测量的人工mirna的丰度相关,图14b显示了在与图14a所示的相同样本中用qrt-pcr测量的所识别的trf/人工mirna靶标的表达(图14a和图14b中的数值,其中一个样本设定为“1”,其他样本进行相应调整,显示为三个生物学重复的平均值
±
se,星号表示p《0.01(学生t测试)的显著性水平),并且图14c显示的箱线图表示与图14a中所述相同样本中的根瘤数,由点表示的所有数据点,显示三个生物重复(每个重复为12株植物)的95%至5%的间隔;
194.图14d示出的示意图表示了,根据本公开的方法产生人工mirna的人工mirna基因的结构,其中这种产生的mirna与两种野生型根瘤菌trf序列相同;
195.图15,分图a是gfp-trf靶位点构建体的示意图,其中w1-w3代表三种野生型转基因根部,m1-m3代表根据本公开制备的三种突变型转基因根部,每个方框(
□
)内的核苷酸代表切割位点,下划线和星号表示突变;分图b显示的图形数据由定量图15的分图d中所示的gfp信号产生,其中使用imagej软件测量了所述gfp信号,误差线表示12个图像gfp信号的s.d.;分图c显示的图形数据与以下相关:在用usda11接种后24小时(hpi),gfp融合基因的表达水平相对于野生型融合基因转基因根部中gmefl1b的表达水平降低,其中gfp转录本丰度通过qrt-pcr测量,并且一个样本的数值设定为1,其他样本的数值进行相应调整(数值显示为三个生物学重复的平均值
±
s.e.,一个样本的数值设定为1,其他样本的数值进行相应调整,bj 表示24hpi接种根部,bj-表示未接种的根部,并且柱形条上方的星号表示p《0.01显著性水平(t-检验));并且分图d显示的照片为接种usda110 24小时后“gfp-trf靶位点”融合基因(w1至w3)和“gfp突变的trf靶位点”(m1至m3)的转基因根部中gfp活性,bj-和bj 分别表示未接种根部和接种根部;
196.图16显示的凝胶说明了三种trf与接种28天后从三个实验重复中检测到的根瘤中大豆gmagolb的关联性,“ ”和
“‑”
分别表示由myc抗体免疫沉淀的gmagolb-myc融合蛋白相关部分和未经myc抗体孵育的根瘤裂解物;
197.图17和图18显示的图形数据与沿大豆种群中五个根瘤菌靶中每一个的核苷酸多样性(π)相关,其中x轴代表大豆参考基因组(品种,williams 82,版本2)中五个大豆基因中每一个的核苷酸位置,y轴代表699个大豆品种中单个核苷酸位置的多样性水平,箭头指向五个基因内的trf靶位点;
198.图19a至图19c说明了四个根瘤菌群中三种根瘤菌trna的序列保守性和趋异性,图19a显示val-l-trna(cac),图19b显示gly-l-trna(ucc),图19c显示gln-l-trna(cug);和
199.图20a和图20b显示了常见豆类根瘤中21-nt trf沿根瘤菌trnas的分布和相对丰度数据的图形描述,每个x轴代表埃特里根瘤菌菌株ce3 pmp604的trna的核苷酸位置,每个y-轴表示沿trna的单个核苷酸的频率,这些核苷酸由ce3 pmp604诱导的常见豆类根瘤中trna衍生的21-nt trf覆盖。
200.尽管本公开易于进行各种修改和替代形式,但它们的示例性实施方案在附图中以示例方式示出并且在此进行详细描述。
具体实施方式
201.出于利于理解本公开原理的目的,将会参考附图中示出的实施方案并且将使用特
定措辞来描述这些实施方案。然而,应当理解的是,这些实施方案的描述并不旨在限制本公开的范围。相反,本公开旨在覆盖可包括在由所附权利要求限定的本技术精神和范围内的替代方案、修改方案和等同方案。如前所述,虽然可以在一个或多个优选实施方案中说明和描述该技术,但是它们的构成、系统和方法可以包括许多不同的配置、形式、材料和附件。
202.在以下描述中,对许多具体细节进行阐述以便于透彻理解本公开。可以在没有部分或全部这些特定细节的情况下实现特定示例,并且应当理解的是,本公开不限于特定的生物系统,其当然可以变化。
203.除非另有定义,本文使用的所有技术术语和科学术语与相关领域技术人员通常理解的含义相同。尽管在实施或测试本技术主题中可以使用与本文所述的方法和材料相似或等效的任何方法和材料,但本文描述了优选的方法和材料。为了便于理解本发明,下面定义了许多术语。在此引用的所有出版物、专利和专利申请,无论是上文还是下文,均通过引用整体并入本文,只要该参考文献与本文提供的教导不矛盾。此外,如在本说明书和所附权利要求书中所使用的,除非内容另有明确规定,否则单数形式“一个”(a)、“一个”(an)和“所述”包括所指对象的复数。因此,例如,提及“一个trna”包括两个或多个trna的组合;提及“细菌”包括细菌的混合物等。
204.此外,除非另有特别说明,否则术语“约”可以允许数值或范围存在一定程度的可变性,例如,在规定值或百分比范围边界的正负10%以内、5%以内或1%以内,以及单位值的正负1.0单位,例如,约1.0是指从0.9到1.1的数值范围。术语“基本上”可以允许数值或范围存在一定程度的可变性,例如在规定值或规定范围边界的90%以内、95%以内或99%以内。
205.术语“植物”包括整株植物、植物器官(例如,叶、茎、花、根、生殖器官、胚胎及它们的部分等)、幼苗、种子和植物细胞及其后代。虽然本公开中主要关注的植物种类涉及豆科植物,但应当理解的是,本发明的技术和概念不限于任何特定类别的高等植物,并且本文的方法通常与适用于转化技术和/或表现出结瘤或类似共生、跨界机制的高等植物类别一样广泛。
206.本文所指的“豆科植物”是豆科(fabaceae)(或豆科(leguminosae))的任何成员,当被根瘤菌微生物感染时,可以形成根瘤。
207.如本文所用,术语“转基因植物”或“转基因植物根部”或“转基因植物细胞”是指植物、农杆菌诱导的植物根部和植物细胞,它们的dna序列通常不会转录成rna或翻译成蛋白质(“表达的”),包括但不限于可能不正常存在的基因,或人们希望引入未转化植物中但希望进行基因工程或改变表达的任何其他基因或dna序列。预期在某些情况下,本发明转基因植物的基因组将通过稳定引入所述转基因而增加;然而,在其他情况下,所引入的基因将取代内源序列。转基因植物/根部/细胞包括从本公开最初转化的植物或细胞再生的植物/根部/细胞以及来自本文所述转化植物后代或杂交的后代转基因植物。
208.本文所指的“根瘤菌微生物”可包括在豆科植物根瘤中逐渐形成后能够固氮的任何微生物。
209.如本文所用,“接种”是指使豆科植物(包括豆科植物种子或根部)与根瘤菌微生物接触的任何方法或过程。在一些实施方案中,接种可包括将所述根瘤菌微生物施用于所述植物生长的土壤。在其他情况下,接种可包括将所述根瘤菌微生物施用于植物根部的一部
分。
210.此外,如本文所用,术语“过表达”(当与基因结合使用时)和“上调”具有相关领域普通技术人员理解的含义,包括(但不限于)过表达或错误表达可能导致突变表型和/或造成大量靶蛋白或小rna片段表达的野生型基因产物。“下调”或“下调的”可以互换使用,指的是与既定水平(例如,相应的野生型基因的水平)相比,标记物(例如基因、核酸、代谢物、转录本、蛋白质或多肽)的水平降低。
211.本公开中的术语“敲除”、“消除”和“缺失”可互换使用,是指细胞基因组的靶基因序列的任何添加或丢失,从而完全去除所述靶基因介导的蛋白质表达。
212.如本文所用,“crispr-cas9”是指由与所述靶基因组互补结合的sgrna(引导rna)和通过同时结合所述sgrna和所述靶基因组来切割基因组基因的cas9蛋白组成的系统。因此,当所述sgrna载体和cas9载体同时在细胞中临时表达时,会产生sgrna和cas9蛋白以改变被靶基因序列,从而使所述被靶基因在cas9结合位点发生修饰,并可能破坏所述基因的功能。
[0213]“核酸”是指单链或双链形式的脱氧核糖核苷酸或核糖核苷酸及它们的聚合物,以及它们的互补物。所述术语包括含有已知核苷酸类似物或修饰的骨架残基或键的核酸,它们是合成的、天然存在的和非天然存在的,具有与参考核酸相似的结合特性,并且以与所述参考核苷酸相似的方式代谢。
[0214]
术语“多肽”、“肽”和“蛋白质”在本文中可互换使用,以指氨基酸残基的聚合物,多肽或多肽、肽或融合多肽的片段。所述术语适用于其中一个或多个氨基酸残基是相应天然存在氨基酸的人工化学模拟物的氨基酸聚合物,以及天然存在的氨基酸聚合物和非天然存在的氨基酸聚合物。
[0215]
术语“氨基酸”是指天然存在的氨基酸和合成的氨基酸,以及以与相应天然存在氨基酸类似的方式发挥作用的氨基酸类似物和氨基酸模拟物。天然存在的氨基酸是由遗传密码编码的氨基酸,以及那些后来被修饰的氨基酸,例如羟脯氨酸、γ-羧基谷氨酸和o-磷酸丝氨酸。氨基酸类似物是指与天然存在的氨基酸具有相同基本化学结构(即与氢、羧基、氨基和r基团(例如,高丝氨酸、正亮氨酸、甲硫氨酸亚砜、甲硫氨酸甲基锍)结合的碳)的化合物。此类类似物具有修饰的r基团(例如正亮氨酸)或修饰的肽骨架,但保留与天然存在的氨基酸相同的基本化学结构。氨基酸模拟物是指结构不同于氨基酸的一般化学结构,但以类似于天然存在的氨基酸的方式起作用的化合物。氨基酸在本文中可以通过它们通常已知的三个字母符号或通过iupac-iub生物化学命名委员会推荐的一个字母符号来提及。同样地,核苷酸可以用它们普遍接受的单字母代码来提及。
[0216]
如本文所用,术语“调控元件”在其最广泛的上下文中意指并包括具有基因调控活性的多核苷酸分子,即能够影响转录或翻译可操作连接的可转录多核苷酸分子的多核苷酸分子。事实上,调控元件包含一系列核苷酸,这些核苷酸决定特定基因是否、何时以及在何种水平上表达。诸如启动子、前导序列、内含子和转录终止区的调控元件是具有基因调控活性的多核苷酸分子,其在活细胞中基因的整体表达中起不可或缺的作用。启动子可以衍生自经典的真核基因组基因,包括(但不限于)通常用于实现准确转录起始的tata盒,带有或不带有ccaat盒序列和额外的调控或控制元件(即上游激活序列、增强子和沉默子)或者可能是经典原核基因的转录调控序列。术语“促进”在本文中还可用于描述赋予、激活或增强
核酸分子在细胞、组织或器官中表达的合成分子或融合分子,或衍生物。启动子可以包含一个或多个特定调控元件的额外拷贝,以进一步增强核酸分子的表达和/或改变核酸分子的空间表达和/或时间表达,或赋予特定细胞或组织(例如分生组织、叶、根部、胚胎、花、种子或果实)表达核酸分子(即组织特异性启动子)。在本发明的语境文中,启动子优选是植物可表达的启动子序列,这意味着所述启动子序列(包括添加到其中或包含在其中的任何额外的调控元件)至少能够诱导、赋予、激活或增强在植物细胞、组织或器官中的表达。然而,在非植物细胞例如细菌、酵母细胞、昆虫细胞和动物细胞中也起作用或仅起作用的启动子不排除在本发明之外。
[0217]
如本文所用,术语“可操作地连接”是指并置,其中如此描述的组分处于使得它们以预期方式起作用的关系中。例如,第一多核苷酸分子(例如启动子)与第二可转录的多核苷酸分子(例如所关注基因)“可操作地连接”,其中所述多核苷酸分子的排列方式使得所述第一多核苷酸分子影响所述第二多核苷酸分子的功能。所述两个多核苷酸分子可以是也可以不是单个连续多核苷酸分子的一部分,并且可以相邻也可以不相邻。例如,如果启动子调控所关注的基因在细胞中的转录,则所述启动子与所关注的基因可操作地连接。
[0218]
术语“分离的”是指材料从原始环境(例如,如果它是自然存在的,则是指自然环境)中移除。例如,存在于活生物体中的天然存在的多肽不是分离的,但是与天然系统中的一些或全部共存材料分开的相同多肽是分离的。术语“纯化”并不要求绝对纯度;相反,它旨在作为相对定义。换言之,“纯化的”表示分子在基本上不存在其他相同类型分子的情况下存在。如本文所用,术语“纯化的”优选意指存在至少75重量%,更优选至少85重量%,更优选至少95重量%,最优选至少98重量%的相同类型的分子。
[0219]
术语“丰度”和“含量”在本文中可互换使用。“扩增”是指多核苷酸序列被拷贝并因此扩展成大量多核苷酸分子的任何方式,例如通过逆转录、聚合酶链反应以及连接酶链反应。
[0220]
如本文所用,化学化合物的“类似物”是例如在结构上类似于另一种化合物,但不一定是异构体的化合物(例如,5-氟尿嘧啶是胸腺嘧啶的类似物)。
[0221]
如本文所用,术语“多核苷酸”是指任何长度的核苷酸(核糖核苷酸或脱氧核糖核苷酸)的聚合形式。该术语仅指分子的一级结构,因此包括双链和单链dna和rna。它还包括已知类型的修饰,例如本领域已知的标记、甲基化、“封端”、用类似物替换一个或多个天然存在的核苷酸、核苷酸间修饰(例如具有不带电荷的键和具有带电荷的键的修饰、含有侧链部分(例如蛋白质)的修饰、带有嵌入剂的修饰、含有螯合剂的修饰、含有烷化剂的修饰、具有修饰键的修饰);以及多核苷酸的未修饰形式。
[0222]
如本文所用,术语“表达盒”是指包含与控制序列可操作地连接的至少一个编码序列的分子,所述控制序列包括在合适的宿主细胞中转录所述编码序列的克隆拷贝和翻译mrna所需的所有核苷酸序列。表达盒可以包括但不限于克隆载体、专门设计的质粒、病毒或病毒颗粒。所述表达盒可以还包括用于在宿主细胞中自主复制的复制起点、可选择标记物、各种限制性位点、高拷贝数的潜力和强启动子。
[0223]“载体”是包含分离的核酸的物质组合物,可用于在细胞之间转移基因序列。因此,所述术语包括克隆媒介物和表达媒介物,以及病毒载体。许多载体是本领域已知的,包括但不限于线性多核苷酸、与离子化合物或两亲化合物相关的多核苷酸、质粒和病毒。因此,所
述术语“载体”包括自主复制的质粒或病毒。所述术语还应解释为包括促进向细胞转移或递送核酸的非质粒化合物和非病毒化合物,例如聚赖氨酸化合物、脂质体等。病毒载体的示例包括但不限于腺病毒载体、腺相关病毒载体、逆转录病毒载体、重组病毒载体等。非病毒载体的示例包括但不限于脂质体、dna的多胺衍生物等。
[0224]“互补的”是指两条核酸链的区域之间或同一核酸链的两个区域之间序列互补性的广义概念。众所周知,第一核酸区域的腺嘌呤残基能够与反平行于所述第一区域的第二核酸区域的残基(如果所述残基是胸腺嘧啶或尿嘧啶的话)形成特定的氢键(“碱基配对”)。如本文所用,术语“互补的”或“互补性”用于指代通过碱基配对规则相关的多核苷酸(即核苷酸序列)。例如,序列“a g t”与序列“t c a”互补。类似地,众所周知,第一核酸链的胞嘧啶残基能够与反平行于所述第一链的第二核酸链的残基(如果所述残基是鸟嘌呤的话)碱基配对。如果当两个区域以反平行方式排列,第一区域的至少一个核苷酸残基能够与第二区域的残基碱基配对时,则核酸的所述第一区域与相同或不同核酸的所述第二区域互补。优选地,所述第一区域包括第一部分并且所述第二区域包括第二部分,由此,当所述第一部分和所述第二部分以反平行方式排列时,所述第一部分的核苷酸残基的至少约50%,优选至少约75%,至少约90%或至少约95%能够与所述第二部分的核苷酸残基碱基配对。更优选地,所述第一部分的所有核苷酸残基能够与所述第二部分中的核苷酸残基碱基配对。
[0225]
对于序列比较,通常将一个序列作为参考序列,与测试序列进行比较。当使用序列比较算法时,将测试序列和参考序列输入计算机,必要时指定子序列坐标,并指定序列算法程序参数。可以使用默认程序参数,也可以指定替代参数。然后,所述序列比较算法基于所述程序参数计算所述测试序列相对于所述参考序列的序列同一性百分比。
[0226]
在两个或多个核酸或多肽序列的语境中,术语“相同的”或百分比“同一性”是指两个或多个序列或子序列,它们当在比较窗口或指定区域上进行比较和比对以获得最大对应时,使用序列比较算法或通过手动比对和目视检查进行测量,在指定区域内是相同的或具有相同(即,至少约50%、65%、75%、85%、95%、99%或更多)同一性的特定百分比的氨基酸残基或核苷酸。所述定义还指测试序列的互补,当所述测试序列与所述参考序列具有基本的同一性时,所述测试序列具有基本的序列或子序列互补性。所述定义还指测试序列的互补,当所述测试序列与参考序列具有基本的同一性时,所述测试序列具有基本的序列或子序列互补性。
[0227]“基本同源核酸序列”是指与参考核酸序列相对应的核酸序列,其中所述相应序列编码的肽与所述参考核酸序列编码的肽具有基本相同的结构和功能;例如,仅存在不会显著影响所述肽功能的氨基酸变化。核酸序列的基本同一性可以通过比较两个序列的序列同一性来确定,例如通过物理/化学方法(即杂交)或通过计算机算法的序列比对。
[0228]
至于氨基酸序列,本领域技术人员会认识到,对核酸、肽、多肽或蛋白质序列的个别替换、删除或添加会改变、添加或删除所编码的序列中单个氨基酸或一小部分氨基酸,是“保守修饰的变体”,其中所述改变会使得氨基酸被化学上相似的氨基酸取代。提供功能相似氨基酸的保守置换表是本领域公知的。这种保守修饰的变体是本发明的多态性变体、种间同源物和等位基因的补充,但不排除这些变体。
[0229]
如本文所用,“化合物”是指在本公开方法中使用的蛋白质、多肽、分离的核酸或其他试剂。
[0230]“编码”是指多核苷酸中特定核苷酸序列(例如基因、cdna或mrna)的固有特性,用作生物过程中合成其他聚合物和大分子的模板,这些聚合物和大分子具有确定的核苷酸序列(即rrna、trna和mrna)或确定的氨基酸序列以及由此产生的生物学特性。因此,如果转录和翻译对应于该基因的mrna会在细胞或其他生物系统中产生蛋白质,则所述基因编码所述蛋白质。编码链(其核苷酸序列与所述mrna序列相同,通常在序列表中提供)和非编码链(用作转录基因或cdna的模板)都可以称为编码该基因或cdna的蛋白质或其他产物。
[0231]“片段”是包含至少一个氨基酸的氨基酸序列的一部分,或包含至少一个核苷酸的核酸序列的一部分。
[0232]
如本文所用,“功能性”生物分子是表现出其特征的性质或活性的形式的生物分子。例如,功能性酶是表现出所述酶所表征的特征催化活性的酶。
[0233]
如本文所用,短语“表达水平”是指可用于将测定结果与所关注基因或蛋白质的表达水平相关联的任何测量或测定。此类测定包括测量mrna的水平、蛋白质水平等,并且可以通过诸如诺慎印迹分析和蛋白质印迹分析、结合测定、免疫印迹等的测定来进行。所述表达水平可以包括表达率,并且可以根据存在的mrna或蛋白质的实际量来测量或讨论。此类测定与过程或系统结合以存储和处理信息,有助于量化表达水平、信号等并将所述信息数字化以用于比较表达水平。
[0234]
本公开的新型转基因植物/根部/细胞广泛地针对与野生型相比表现出根瘤形成增强的转基因根部(即豆科植物的根部),以及系统。此外,本公开提供了与根瘤菌衍生的转移rna(trna)衍生的片段(trf)相同的人工小rna,当其应用于植物和/或在植物中过度表达时,通过抑制所述植物的先天机制来上调根瘤形成。本公开还提供了用于控制植物根部上根瘤数的方法,在至少一个实施方案中,所述方法利用本文所述的本发明转基因根部来有利地影响作物产量。
[0235]
为了提高植物的产量潜力,重要的是提高结瘤效率和固氮效率。许多豆科植物可以通过与固氮根瘤菌的共生关系,利用大气二氮(n2)气来满足它们对氮的需求。具体来说,大气中的n2被转化为氨(nh3),然后被吸收为氨基酸、核苷酸和其他细胞成分,如维生素、黄酮和激素。豆科植物固定气态氮的能力使它们成为理想的农业生物,因为它们对氮肥的需求减少了。
[0236]
根瘤菌-豆科植物共生的建立取决于这两个伙伴之间对信号分子的识别。在感知植物黄酮类化合物后,根瘤菌合成并分泌脂壳多糖寡聚糖,即所谓的nod因子(nf),根部nod因子受体(nfr)可感知所述nod因子以引发根瘤菌感染,并在发生共生固氮(snf)的地方下形成根瘤。更具体地说,所述nf引发根毛卷曲,这从围绕所述细菌卷曲的根毛最尖端开始,然后是感染痕迹的发展,为所述细菌渗透到根部皮质细胞中以形成根瘤提供了途径。根瘤是植物种中相对不同的器官,实质上代表了受控微生物侵入根部。与人类肠道类似,所述植物提供了特定微生物可以繁殖的环境。复杂的是对结瘤发育进行遗传控制,本文提供的数据显示对从芽到根部运输小rna(srna)的依赖性。
[0237]
由于snf是资源密集型的,豆科植物已经进化出许多机制,例如结瘤的自动调控(aon)和结瘤的氮调控,以控制形成的根瘤数。虽然之前已经说明过真菌小rna介导的与植物的相互作用,但除此之外,之前关于任何细菌srna是否和/或如何介导真核生物中的生物过程知之甚少。事实上,传统观念是,只有宿主植物居于主导地位。本公开利用了由本研究
人员确定的新机制,通过该机制所述过程由两个共生伙伴控制;细菌确实在通过使用细菌衍生的转移rna(trna)衍生的片段(trf)调控根瘤数中发挥作用。
[0238]
trna是细胞中最保守和最丰富的rna种类之一;它们的功能是在核糖体中将信使rna(mrna)解码为蛋白质。trna具有独特的三叶草状结构,包含三个发夹环,末端连接特定的氨基酸,用于将所述mrna中的密码子翻译成转移到生长蛋白质末端的氨基酸。在trna生物生成期间中,或在特定条件下,裂解会产生trf。事实上,在原核生物和真核生物中都发现了trf,并且在历史上它们被认为是随机降解产物,因为它们经常在应激细胞或病毒感染的细胞中积累。这类短的非编码rna片段的长度小于约30个核苷酸(nt)碱基(更具体地为约17至约22个核苷酸),几乎没有已知的功能,其中很大一部分衍生自在成熟或前体trna的5'或3'末端进行精确加工。
[0239]
特别令人关注的是,一些类似于微rna(mirna)的trf与argonaute(ago)蛋白结合,表明它们使用类mirna机制来调控基因表达。与之前认为的宿主对结瘤的负调控不同,本公开提出的研究确立了trf是结瘤的正调控因子,其通过rna干扰(rnai)介导的沉默途径抑制逆转录转座子。如图1所示,经由严格控制根瘤数而建立的snf与宿主植物生长之间的共生平衡,是通过结瘤潜在的负调控途径和正调控途径的相互联系来实现的。
[0240]
可以预计的是,在识别trf敲掉的靶基因及它们与植物根部中根瘤形成相关的功能后,通过crispr-cas基因编辑或其他方式调控这些靶基因的活性可以产生固氮效率增强的稳定转基因植物。本文的示例性实施方案适用于大豆根部,但只要识别了所述靶基因,其他适合此类基因调控/crispr-cas编辑的植物也是可以考虑的。
[0241]
最近的一项研究报告称,小鼠干细胞中的内源trf能够阻断ltr-逆转录转座子的逆转录,进一步证明了trf通过rna干扰作为调控性小rna的作用;然而,在目前所描述的研究之前,尚不清楚trf是否可以靶向基因。相反,传统研究倾向于通过过滤掉trna和rrna来关注mirna和sirna,而本研究举例说明了trf在跨界相互作用中的重要性。
[0242]
也许更具体地说,如下文详细描述的,根据srna测序数据,从根瘤菌大豆慢生根瘤菌中识别出多个trf。这些trf然后在植物中富集,并用于识别大豆(glycine max)基因组中的57个靶基因。这些基因中的三种——gmrhd3、gmham4和gmlrx5,它们分别是拟南芥基因根毛缺陷3(rhd3)、多毛分生组织4(ham4)和富含亮氨酸的重复序列类伸展蛋白5(lrx5)的直系同源物,直接参与根瘤发育,包括根毛的发育,并作为代表性样本进行了进一步研究。然后确定所述细菌产生的trf被加载到大豆ago1蛋白中,随后以序列同源性依赖的方式与靶基因相互作用。
[0243]
如本文所示,从最初的根瘤菌感染到根瘤器官形成,检测到根瘤菌trf下调了所识别的靶宿主基因。尽管它们的丰度相对较低,但在自由生存的根瘤菌培养物中仍可检测到一些根瘤菌trf。因此,在根瘤菌与宿主相互作用时,所述trf很可能在所述根瘤菌细胞中迅速积累并迁移到宿主细胞中,在那里它们调控宿主基因的表达。除了详细研究的三种根瘤菌trf外,预计还有其他trf靶向备选生长素受体和外排载体、ring/u-box蛋白和蛋白激酶,它们也与结瘤有关(表2)。因此,trf在调控宿主结瘤中的功能性作用比原先想象的要重要和广泛得多。识别这种细菌调控宿主植物根瘤数的新型细菌机制是共生生物学和细菌-真核生物相互作用领域的重大进展。
[0244]
这些新发现使得对一个或两个共生伙伴进行转基因修饰,以利用新发现的跨界信
号传递机制并赋予转基因基因型和/或转基因表型,从而促进和增强根瘤形成(与野生型相比)。虽然本公开聚焦于作为实验系统的豆科植物-根瘤菌共生伙伴,但应当理解的是,本发明的概念不限于此,并且可以使用任何菌种或菌株的细菌,其中此类细菌涉及与宿主生物的跨界共生关系,因为根据本文提供的数据,显然经由trf进行跨界交流和/或调控在进化方面是保守的。
[0245]
例如但不限于,本公开的化合物、原理和方法可适用于其他宿主-细菌共生关系,例如哺乳动物肠道微生物群及其宿主(无论是人类、动物还是其他生物)。所述哺乳动物肠道代表了由大量常驻共生细菌组成的复杂生态系统,这些细菌与所述宿主的免疫系统保持稳态。类似于豆科植物和固氮细菌之间的所述共生关系,所述宿主不仅耐受这些细菌,而且已经进化到在消化、免疫发育和功能的各个方面需要这种定植。虽然传统上已知微生物群(例如但不限于多形类杆菌)提供关键信号(其促进免疫细胞和组织成熟以防止病原体感染和/或导致非感染性免疫疾病),但迄今为止,这种信号传递的确切机制仍然未知。基于本文提供的数据,可以预想的是,可能适用如本文所公开的trf信号传递到宿主基因的原理,并且因此可以类似于本文结合豆科植物和固氮细菌所述的方式加以利用。
[0246]
在本公开的至少一个实施方案中,提供了转基因植物、植物根部和/或植物细胞,它们被工程化以抑制或下调一个或多个结瘤相关宿主基因的表达。这可以通过以下方式实现:(1)对所述宿主植物进行基因工程化以直接下调或完全抑制一个或多个靶基因,和/或(2)通过工程化所述宿主植物细胞/根部以产生与根瘤菌衍生trf相同的人工小rna(即表达一个或多个编码人工小rna片段的rna前体的多核苷酸序列,这些片段与衍生自野生型根瘤菌的trf相同)来抑制这些基因。实际上,本公开的所述转基因植物/植物根部/植物细胞可以包含一种或多种突变,它们针对编码根瘤菌衍生trf和/或直接抑制/下调所述植物的靶基因。作为此类转基因植物,植物细胞或根部已减少所识别的靶基因的表达(即通过根瘤菌trf介导的类mirna转录后调控和/或经由直接抑制或下调所述靶基因本身),与野生型相比,任何由此产生的相关转基因根部将表现出根瘤形成的增强。
[0247]
在至少一个示例性实施方案中,对豆科植物、植物根部或植物细胞进行修饰以表达编码根瘤菌衍生rna的多核苷酸构建体,使得人工小rna片段从中切割并在所述植物根部和/或细胞中产生。所述多核苷酸构建体可以配置为编码单个人工小rna(靶向单个宿主基因)或小rna的组合(靶向宿主基因的组合,例如gmrhd3a/gmrhd3b或gmham4a/gmham4b)。以这种方式,植物本身产生人工trf(与野生型根瘤菌在接种时产生的trf相同),其下调作为本文结瘤的负调控因子的所靶向宿主(其自身)基因。以这种方式,与从相应的野生型豆科植物、植物根部或植物细胞产生的根瘤数相比,所述转基因豆科植物、植物根部或植物细胞能够产生的根瘤数增加了。重要的是,以下实施例证实了所得小rna片段与野生型根瘤菌产生的trf相同,并且此类“人工trf”在所述宿主根部/细胞中以与根瘤菌衍生trf相同的方式起作用。
[0248]
另外地或备选地,所述植物、植物根部或植物细胞可以被修饰以直接下调或甚至沉默所述植物宿主的一个或多个作为结瘤的负调控因子的基因(即所述“靶基因”或“宿主基因”)。例如,这可以通过选择性抑制所述一个或多个宿主基因(使用本领域已知的遗传修饰技术)来实现。
[0249]
此外,crisr-cas9或类似方法可用于敲除或删除所述植物/根部/细胞的所靶向基
因。例如,在至少一个实施方案中,可以构建crispr-cas9载体(例如但不限于,利用选自seq id nos:4-9(见表1)的一对或多对引物)并在所述宿主植物、植物根部或植物细胞中表达,使得所述植物/根部/细胞的一个或多个所靶向基因被沉默。
[0250]
所述靶基因可以包括表2和表4中列出的任何基因(包括rhd3、ham4和/或lrx5)。备选地,所述靶基因可包含前述基因的直系同源物,包括但不限于用作本文公开研究的代表性靶基因的以下大豆(glycine max)基因:gmrhd3a、gmrhd3b、gmham4a、gmham4b和gmlrx5。如本文所示,这些基因被某些trf靶向,虽然通常这些基因的功能未知,但本公开确定它们实际上是豆科植物结瘤的负调控因子。在至少一个示例性实施方案中,所靶向的基因包含一个或多个以下基因或基因组合:gmrhd3a、gmrhd3b、gmrhd3a/gmrhd3b、gmham4a、gmham4b、gmham4a/gmham4b、gmlrx5。
[0251]
在至少一个实施方案中,转基因植物/植物根部/细胞可包含在gmrhd3a和gmrhd3b处的第一突变(即下调这两个基因)和在gmlrx5处的第二突变。如上所述,这可以是导致所述植物/根部/细胞表达一个或多个多核苷酸序列的补充或替代,所述多核苷酸序列编码根瘤菌衍生trf的rna前体。事实上,本领域技术人员将理解,宿主基因结瘤负调控因子的任何组合可以被靶向以实现抑制/下调,无论是否结合根瘤菌衍生trf的表达。
[0252]
用编码此类化合物的多核苷酸序列、载体或表达盒转化细胞的方法是本领域技术人员众所周知的,选择性扩增或抑制宿主基因的方法同样如此。植物、植物根部和植物细胞可以通过以下方式进行转化:例如电穿孔、农杆菌转化、工程化植物病毒复制子、电泳、显微注射、微弹轰击、微激光束诱导的细胞壁穿孔或只需在有或没有聚乙二醇(peg)的情况下温育。
[0253]
本公开的trf可以是通过抑制根瘤形成的宿主根瘤中rnai介导的沉默途径靶向和沉默或下调被靶向的宿主基因(即,宿主结瘤负调控因子)的任何trf。因此,根瘤中trf丰度的扩增阻碍了植物的结瘤抑制机制,从而促使了根瘤形成的增强。在至少一个实施方案中,本文的trf包括表2和表4(下文)中列出的任何trf,或其他能够下调作为结瘤负调控因子的植物宿主基因表达的其他trf。因此,本文所述的多核苷酸构建体可包含与此类trf相关的任何序列。然而,在示例性实施方案中,所述trf包含一个或多个衍生自以下三种trna的主要trf产物:val-t-trna(cac)、gly-1-trna(ucc)和gln-1-trna(cug)(分别称为bj-trf001(seq id no:1)、bj-trf002(seq id no:2)和bj-trf003(seq id no:3),并如图2中分图i至分图k中所示)。因此,所述多核苷酸构建体可以包含seq id no:1-3,或seq id no:1-3的基本同源核酸序列。
[0254]
例如但不限于,此类多核苷酸构建体可以编码所识别的序列以及与其互补序列,从而产生具有发夹结构的前体rna,与根瘤菌衍生trf相同的人工小rna最终从所述前体rna中被切割。图14d示出了,根据本公开使用编码seq id no:1及其互补序列
*
(左)和seq id no:3及其互补序列
*
(右)的构建体制备的这种前体rna的至少一个示例性实施方案。由此产生的rna随后在植物中被切割以产生与野生型根瘤菌衍生trf相同的所需的人工小rna。
[0255]
虽然本公开提供了特定多核苷酸序列,但对本领域普通技术人员来说显而易见的是,由于遗传密码的冗余,所公开的人工trf和/或其rna前体可以由多个多核苷酸序列编码。本领域受过训练的人员完全有能力创造出这些替代的rna序列和dna序列,这些序列具有不实质性影响本发明人工trf(即能够进行跨界交流并在植物根部引起结瘤的trf)的生
物活性的氨基酸取代、缺失、添加或插入。保留诱导结瘤能力的所述人工trf片段也包括在此定义中。本公开所述的多核苷酸可包括载体和表达盒。所述载体和表达盒可包含与编码所述前体rna和其相关人工trf的多核苷酸序列可操作连接的转录控制序列。
[0256]
本公开的其他实施方案包括与相应的野生型细菌微生物相比上调一个或多个trf生成的工程细菌微生物。在本公开的至少一个实施方案中,提供了一种细菌菌种或菌株,其被工程化从而表现出一个或多个trf表达的上调(与在野生型相应菌种或菌株的这种生成相比)。所述一个或多个扩增的trf可以是能够与宿主生物的至少一个靶基因进行跨界交流和/或信号传递的任何trf。
[0257]
例如但不限于,所述根瘤菌微生物可以表达多核苷酸构建体,所述构建体增强微生物生成一个或多个trf,例如本公开的表2和表4中确定的trf。所述多核苷酸构建体可以被配置为上调单个trf(靶向单个宿主基因)或trf的组合(靶向宿主基因的组合,例如gmrhd3a/gmrhd3b或gmham4a/gmham4b)的表达。
[0258]
在至少一个示例性实施方案中,所述宿主生物是豆科植物、植物根部或植物细胞,例如大豆(例如,glycine max)并且可以包含先前描述的所述转基因豆科植物、植物根部或植物细胞或野生型豆科植物。在任何情况下,细菌衍生trf都能够抑制或下调所述宿主生物根瘤中至少一个基因靶标的表达。就这样,例如,所述一个或多个trf可以是,通过根瘤中rnai介导的沉默途径靶向和沉默或抑制靶宿主的trf,所述路径被设计以抑制根瘤形成。
[0259]
当所述宿主生物是豆科植物时,所述细菌微生物可以是固氮细菌的任何菌株或种,例如但不限于根瘤菌微生物,例如大豆慢生根瘤菌usda 110。此类根瘤菌微生物的其他非限制性示例包括其他大豆慢生根瘤菌菌株(例如,例如usda 123和138)、埃特里根瘤菌、苜蓿中华根瘤菌、豌豆根瘤菌、拟山黄麻根瘤菌、百脉根根瘤菌和其他相容的根瘤菌种。
[0260]
还公开了包含本文所述细菌微生物的一个或多个菌种或菌株的化合物。在至少一个实施方案中,将所述化合物配制成包含如前所述的工程细菌微生物的接种剂组合物。所述化合物可以包含本公开所述的工程细菌微生物的单一菌种/菌株或所述化合物中不同菌种和/或菌株的细菌微生物的组合。工程细菌可以另外地与野生型细菌菌株或菌种组合,或者所述化合物内的所有细菌可以被遗传修饰(具有相同或不同的突变)以扩增如本文所述的一个或多个trf的表达。
[0261]
所述接种剂组合物可包含载体或添加剂。所用的载体或添加剂将取决于所述接种剂组合物的性质。例如,所述接种剂组合物的形式可以是液体组合物、固体组合物(例如粉末、丸粒组合物或颗粒组合物)、种子包衣,或适合于所需应用的任何其他形式。在至少一个实施方案中,所述接种剂组合物包括液体组合物,并且包括适合于溶解或悬浮所述化合物的溶剂。
[0262]
本公开所述的接种剂组合物可适用以任何合适的方式应用于豆科植物、植物根部或细胞。例如,所述接种剂组合物可适作为种子包衣施用,作为固体组合物或液体组合物施用至植物的叶子或根部,或作为固体组合物或液体组合物在播种豆科植物之前、期间或之后施用至土壤。一系列有用的载体或添加剂对本领域技术人员来说是显而易见的,并且可以包括,例如:一种或多种树胶(包括黄原胶)、粘土基载体或泥炭基载体、一种或多种养分(包括碳源或氮源)、一种或多种抗真菌剂或抗菌剂、一种或多种种子包衣剂、一种或多种润湿剂等。
[0263]
增强豆科植物根部结瘤活性的方法可以在被工程化以抑制或下调所识别靶基因的转基因植物/植物根部/植物细胞中实施,以及在应用工程细菌微生物和/或接种剂的非转基因植物中实施。此外,本公开还提供了同时利用两种方法的方法——即,将trf生成扩增的工程细菌微生物和/或接种剂应用于本公开的转基因植物/植物根部/植物细胞。
[0264]
在至少一个实施方案中,这种方法包括抑制或下调宿主植物根部或细胞的一个或多个靶基因的表达,并用至少一种根瘤菌接种或已经接种经修饰的植物宿主根部或细胞以引起结瘤。所述接种步骤可根据已知方法并使用常规微生物菌种和菌株进行。在一个实施方案中,可以损伤所述根部,以使细菌细胞更快、更容易地穿透所述根部;然而,损伤所述根部不是必需的。
[0265]
所述靶基因可包含本文所述的任一靶基因,并且在至少一个示例性实施方案中,包含rhd3、ham4、lrx5、rhd3的直系同源物、ham4的直系同源物和/或lrx5的直系同源物。也许更具体地,在又一个示例性实施方案中,所述豆科植物根部是大豆,每个宿主基因可以包含gmrhd3a、gmrhd3b、gmham4a、gmham4b或gmlrx5。
[0266]
所述至少一种根瘤菌的菌种或菌株同样可以包括在宿主植物根部或细胞中引发结瘤的任何微生物,包括例如大豆慢生根瘤菌、埃特里根瘤菌、苜蓿中华根瘤菌、豌豆根瘤菌、拟山黄麻根瘤菌、百脉根根瘤菌等。
[0267]
下调或抑制所述宿主植物根部或细胞的所述一个或多个靶基因表达的步骤,可以包括引入和表达编码根瘤菌衍生rna的第一多核苷酸构建体,所述rna是如前所述在植物根部中产生一个或多个人工trf的前体。在至少一个示例性实施方案中,所述rna可以具有如图14d所示的发夹结构。所述第一多核苷酸构建体可以编码seq id nos:1-3、34-70,或与前述任一项基本同源的任何核酸序列的一种或多种。此外,所述第一构建体还可以编码与上述序列互补的序列(类似于图14d中所示,与所得发夹结构相关)。
[0268]
另外地或备选地,下调或抑制所述宿主植物根部或细胞的所述一个或多个靶基因的表达可包括构建如本文所述的crispr-cas9载体,并在所述宿主植物根部或细胞中表达所述载体,其中该载体被设计成使在本文中所识别的一个或多个靶宿主基因沉默。在至少一个示例性实施方案中,所述crispr载体可以被设计为使以下基因的一个或多个沉默:gmrhd3a、gmrhd3b、gmrhd3a/gmrhd3b、gmham4a、gmham4b、gmham4a/gmham4b和gmlrx5。
[0269]
更进一步,不是经由所述crispr-cas9方法使宿主基因沉默,下调或抑制所述宿主植物根部或细胞的所述一个或多个靶基因表达的步骤可以包括,抑制所述一个或多个被靶向的宿主基因的表达。这种抑制表达可以通过本领域当前或下文已知的任何合适方法来实现,并且在至少一个实施方案中,可以包括在所述宿主根部或细胞中表达多核苷酸构建体,所述多核苷酸构建体编码用于下调或抑制所述一个或多个宿主基因表达的转录因子。此外,这种方法可以涉及将具有工程细菌微生物的所述化合物和/或组合物直接应用于本文所述的转基因根部或转基因植物的根部、植物根部或植物细胞。在一个实施方案中,可以损伤所述根部以使细菌细胞更快、更容易地穿透所述根部。然而,使所述根部损伤不是必需的。
[0270]
由于本公开的发明化合物、植物/根部/细胞和方法增强豆科植物根瘤形成的能力得到了证实,应用本文所述概念可以显著减少氮基肥料的使用,在不降低产量的情况下降低农业的经济成本和环境成本。参考以下非限制性实施例进一步描述本公开。
[0271]
材料和方法
[0272]
对于此处描述的实施例,使用了以下材料和方法。
[0273]
植物及根瘤菌材料
[0274]
大豆(glycine max)栽培品种williams 8在温室中光照16h/8h处于黑暗条件下生长,用于rna分离和毛状根部转化,以及根部和根瘤组织收集。williams 82也用于以大豆根部制备原生质体。大豆慢生根瘤菌菌株usda110在修饰的阿拉伯糖葡萄糖酸盐(mag)培养基中培养并用于大豆根部接种,所述培养基每升由1.3ghepes、1.1gmes、1g酵母提取物、1gl-阿拉伯糖、1g d-葡萄糖酸钠盐、0.22gkh2po4和0.25gna2so4组成,ph为6.6。
[0275]
大豆种子在无菌蛭石砂混合物(3:1)中发芽7天,然后接种根瘤菌培养物或用不含所述根瘤菌的mag培养基(对照组)处理。收集接种后10天及20天(dpi或dpi)的接种根部和未接种根部在同一阶段的最大根瘤用于rna分离。按照林(lin)等人,《比较转录组学揭示的10种近等基因系的大豆对病原体大豆疫霉菌的分子反应》(molecular response to the pathogen phytophthora sojae among ten soybean near isogenic lines revealed by comparative transcriptomics),《bmc基因组学》(bmc genomics),15:18(2014)提出的方案,从根部中分离根毛细胞。
[0276]
dna和rna分离、pcr和测序
[0277]
基因组dna分离、聚合酶链反应(pcr)、逆转录-pcr(rt-pcr)、定量rt-pcr(qrt-pcr)和dna片段测序按照以下文献如前所述进行:平(ping)等人,《dt2是一种指定大豆半决定性的功能获得性mads域因子基因》(dt2 is a gain-of-function mads-domain factor gene that specifies semideterminacy in soybean),《植物细胞》(plant cell),26:2831-2842(2014)。在qrt-pcr中,大豆基因gmelf1b(glyma.02g276600)或gmcons4(glyma.12g020500)被用作内部参考,以量化使用三个生物学重复所识别的根瘤菌trf靶标的相对表达水平。
[0278]
进行茎环rt-pcr以评估根瘤菌trf的相对丰度。茎环rt-pcr对单个trf的特异性通过扩增片段的测序得到证实。大豆慢生根瘤菌usda110中的bll631基因被用作内部参考,以量化使用三个生物学重复通过茎环rt-pcr所识别的trf的相对丰度。用于各种pcr和扩增片段测序的引物是使用本领域已知的策略和技术开发的,并列在以下文章的补充材料中:雷恩(ren)等人,《根瘤菌trna衍生的小rna是调控植物调节的信号分子》(rhizobial trna-derived small rnas are signal molecules regulating plant modulation),《科学》(science)365,919-922(2019)(包括与其相关的所有补充材料,“雷恩等人的文章”),其全部内容与表1中列出的用于crispr-cas9和扩增研究的特异性引物一起通过引用并入本文。
[0279]
表1.用于crispr-cas9和扩增研究的引物
[0280][0281][0282]
srna-seq和mrna-seq&数据分析
[0283]
使用trizol试剂(加利福尼亚州的英杰公司/生命科技公司(invitrogen/life technologies))从植物根部、根瘤和根瘤菌中分离出总rna。小rna-seq(srna-seq)和mrna文库分别使用truseq小rna试剂盒和truseq链状mrna试剂盒(加利福尼亚州的照明公司(illumina))根据制造商的方案分别构建,并使用hiseq2500(加利福尼亚州的照明公司(illumina))在普渡大学基因组学核心实验室(印第安纳州西拉斐特)测序。
[0284]
使用fastq/a短读预处理工具fastx-toolkit(版本0.0.14,http://hannonlab.cshl.edu/fastx_toolkit/)处理原始srna-seq数据,以去除接头序列和低质量的读数。使用参数为
“‑
v 0.”的bowtie(版本1.0.0)将长度超过16nt所处理的读数与大豆参考基因组序列(版本2,phytozome)以及大豆叶绿体和线粒体序列进行比较。随后使用bowtie将未映射到这些大豆序列的读数与大豆慢生根瘤菌usda110参考基因组序列进行比较。完美映射到所述usda110基因组序列而没有任何错配的读数被进一步标注,那些完美匹配usda110的转移rna(trna)序列(http://gtmadb.ucsc.edu/gtrnadb2/)被认为是根瘤菌trna衍生片段(trf)。独特根瘤菌trf在每个文库的相对丰度被标准化为计数/百万读数(cpm)。在至少一个根瘤srna-seq文库中相对丰度高于100cpm的独特根瘤菌trf(bj-trf)被用于识别根瘤菌trf的大豆基因靶点。
[0285]
原始mrna-seq数据也使用fastx-toolkit进行处理,以去除接头序列和低质量读数。使用tophat2映射工具(版本2.1.1)将每个文库的已处理读数映射到所述大豆参考基因组(版本2,phytozome),参数允许每个读数有一个错配。使用bedtools软件(版本2.27.1)基于唯一映射到相应基因的读数量确定单个大豆基因的相对表达水平。使用“edger”(版本3.22.5)定义比较样本之间的差异表达基因(deg),调整后的错误发现率(fdr)控制的q值《0.05。通过qrt-pcr进一步验证了deg的子集。
[0286]
序列比对和系统发育分析
[0287]
使用以下文献描述的程序进行邻接法系统发育树的序列比对和构建:牛(niu)等人,《在转基因拟南芥中表达人工微rna赋予病毒抗性》(expression of artificial micrornas in transgenic arabidopsis thaliana confers virus resistance),《自然
生物技术》(nat.biotechnol),24:1420-1428(2006)。所述系统发育树使用evolview(一种系统发育树的在线可视化工具(版本2))进行可视化。
[0288]
trf靶基因预测和靶位点验证
[0289]
psrna target,一种植物小rna靶分析服务器,用于预测根瘤菌trf的靶基因。当由服务器确定的期望值不超过3时,并且备选基因在所述根部样本和根瘤样本之间表现出差异表达时,预测的trf靶标被认为是最可能的备选基因。根据制造商的方案,使用gene racer rlmrace试剂盒(马萨诸塞州的英杰(invitrogen)公司)进行5'rna连接酶介导的cdna末端快速扩增(rlm-race)以从所述根瘤菌trf靶标中识别mrna的切割位点。所述cdna样本通过巢式pcr扩增。pcr产物被克隆到pgem-t简易载体(威斯康星州的普罗梅格(promega)公司)中,每个靶位点的40个克隆在普渡大学基因组学核心实验室(印第安纳州的西拉斐特)进行测序以确定所述trf靶标的转录本中的切割位点。
[0290]
质粒构建和大豆毛状根部转化
[0291]
psc1载体用于按照以下文献中描述的方案开发grna-cas9表达载体:罗登(rueden)等人,《imagej2:imagej用于下一代科学图像数据》(imagej2:imagej for the next generation of scientific image data),bmc生物信息学(bmc bioinformatics),18:529(2017)。用于编辑所述五个大豆基因gmrhd3a/3b、gmham4a/4b和gmlrx5的sgrna是使用crispr-p(一种基于网络的引导式rna设计工具)设计的。用于本研究的引物对经过退火(在95℃持续5分钟,然后冷却至室温)以形成二聚体,然后单独整合到lgu i酶切的pscl载体中。所述sgrna靶向所述基因的外显子,如图7所示。
[0292]
所述pbin438载体用于在花椰菜花叶病毒35s启动子控制下开发大豆基因gmrhd3b、gmham4a和gmlrx5的过表达构建体。所述gmrhd3b和gmlrx5编码序列(cds)分别通过表1中列出的所述引物扩增,然后使用sal i/xba i酶整合到pbin438中,并且使用xho i/kpn i酶将gmham4a cds插入pbin438。
[0293]
所述pegad载体用于形成三个sttm构建体,每个构建体都使大豆毛状根部中的根瘤菌trf沉默。基于所述三种21-nt根瘤菌trf设计了单独sttm模块。将含有所述sttm根瘤的引物对退火成二聚体,然后使用age i/bam hi酶单独整合到pegad中。雷恩(ren)等人的文章中列出了用于质粒构建的所有引物。
[0294]
如前所述进行大豆毛状根部转化,稍作修改。将1周龄大豆幼苗的下胚轴注射发根农杆菌k599,并用塑料盖使其保持在高湿度(》90%)下,直到注射部位的毛状根部发育到5-10cm长。然后将原幼苗主根部剪断,将所述毛状根部浸入水中约5天,之后将具有毛状根部的植株移植到装有灭菌蛭石和沙子混合物(3∶1)的盆中。在植株移植到盆中两天后,所述植物根部接种大豆慢生根瘤菌菌株usda110(每盆20ml悬浮液(od=0.05))。通过对各个质粒构建体的独特序列进行pcr识别转基因根部,并通过pcr片段的测序进一步验证。crispr-cas9诱导的敲除毛状根部是通过对扩增的基因片段进行测序来识别的,这些基因片段包含克隆到所述pgem-t简易载体(威斯康星州的普罗梅格(promega)公司)中的设计引导rna的靶位点。只有在所述靶位点具有两个等位基因突变的毛状根部被认为是用于表型研究的敲除毛状根部。
[0295]
根瘤数的表型
[0296]
在28dpi时对转基因毛状根部和对照上的根瘤进行计数。在将6dpi毛状根部的根
毛区剪下约1cm的根段并用蒸馏去离子水洗涤,然后用乙醇:乙酸(3∶1)固定两小时。检查所固定根部的根毛形态(包括变形根尖和膨胀根尖或波浪状生长和卷曲(感染灶的形成)),以及根毛数和长度,并用nikon eclipse ti2显微镜(纽约州尼康(nikon)公司)拍照。
[0297]
实施例1
[0298]
跨界交流验证
[0299]
为了解决trf是否参与跨界交流,大豆-根瘤菌共生伙伴被用作实验系统。具体是,大豆(glycine max))和根瘤菌(大豆慢生根瘤菌)作为共生伙伴进行了研究。鉴于细菌中缺乏ago修饰的rnai机制,研究的目的是识别衍生自调控大豆基因的根瘤菌trna的trf。大豆慢生根瘤菌菌株usda110、由usda110诱导的10天和20天的大豆(品种,williams 82)根瘤,以及未接种的williams 82根部的小rna和mrna使用上述方案进行分离和测序,并且识别了由在根瘤菌培养物中或在根瘤菌诱导的根瘤中根瘤菌产生的假定trf。
[0300]
所有50种根瘤菌trna在根瘤菌(usda110菌株)培养物、10天和20天的大豆(品种williams 82)根瘤中都产生了trf,其中大多数trf的大小在18到24nt之间,并且丰度变化很大(参见图2a和图3a以及表2)。总体而言,所述根瘤中的trf明显多于所述根瘤菌培养物中的trf,其中主要衍生自trna的3'末端的21-nt trf最为丰富(见图2a和图3a至图3c)。
[0301]
预测大豆基因组中共有57个大豆基因是25个不同的21或22nt根瘤菌trf的靶标,相对丰度》100拷贝/百万根瘤菌小nra读数(表2,宿主靶序列是作为srna-seq列出序列的互补序列)。
[0302]
表2.衍生自大豆慢生根瘤菌的根瘤菌trf及其大豆中的潜在靶标
[0303]
[0304]
[0305][0306]
共有68个大豆基因在根瘤中的表达也至少减少了1/2(与未接种的根部相比),这些基因被定义为25个根瘤菌trna的36个独特trf的靶标(表2)。这些trf在无根瘤大豆组织的小rna文库中均未发现(ncbi登录号gse58779;表3(基于以下文献的代表性数据:阿里吉特(arikit)等人,《大豆小rna图谱从数百个编码基因中识别出定相sirna》(an atlas of soybean small rnas identifies phased sirnas from hundreds of coding genes),《植物细胞》(plant cell)26:4584-4601(2014),其全部内容以引用方式并入本文),常规上也未预测trf靶向根瘤菌基因。
[0307]
表3.大豆不同组织中存在/不存在大豆慢生根瘤菌衍生trf
[0308][0309]
参考表2,大豆基因gmrhd3a/gmrhd3b、gmham4a/gmham4b和gmlrx5分别是拟南芥根毛缺陷3(rhd3)、多毛分生组织4(ham4)和富含亮氨酸的重复序列类伸展蛋白5(lrx5)的直系同源物,它们被识别用于进一步研究,部分原因是这些拟南芥基因对根毛和植物发育很重要。事实上,这些大豆基因被预测为三种根瘤菌trf的靶标,这三种根瘤菌被称为bj-trf001、bj-trf002和bj-trf003,是分别衍生自三咎trna的主要产物:val-l-trna(cac)、gly-l-trna(ucc)和gln-l-trna(cug)(见图2,分图i至分图k)。
[0310]
与未接种的根瘤菌培养物(即自由生存(野生型)大豆慢生根瘤菌菌株usda110中的大豆慢生根瘤菌基因bll631的转录本)相比,在所述根瘤中三种识别的根瘤菌trf的富集通过茎环rt-qpcr测定进一步验证(见图5a和图5c)。具体而言,检测在10-dpi和20-dpi时的根瘤中这三种根瘤菌trf的丰度(图5a中x轴上分别标记为2和3)。在不添加逆转录酶的情况下20-dpi rna样本的反应作为阴性对照(图5a中x轴上标记为1)。
[0311]
然后通过测序扩增的片段来确认单个trf的茎环rt-pcr产物的特异性。将不同样本的等量总rna加载到凝胶上,以显示根瘤菌rrna和大豆rrna的不同大小和相对丰度。正如预期的那样,与未接种的根部相比,根瘤中它们的备选基因靶标的表达降低(见图5b)。
[0312]
实施例2
[0313]
靶基因在转录本中的切割位点和通过trf的抑制
[0314]
为了确认所识别的大豆基因是所述三种根瘤菌trf的靶标,通过rna连接酶介导的cdna末端5'快速扩增(rlm-race)识别了这些基因在转录本中的切割位点。如果这些大豆基因表达的降低是由所识别的根瘤菌trf通过类mirna转录后调控造成的,那么这些基因的mrna就会在预测的trf靶位点发生切割。
[0315]
通过检查这一点,数据证实,这些基因的mrna实际上在20天根瘤中所预测的trf靶位点被准确切割,而这些位点在未接种的根部中没有被切割(见图6a至图6c和图6e)。这清楚地证实这些trf通过类mirna转录后调控机制抑制所述靶基因的表达。
[0316]
重要的是,在本研究之前,这些位点中没有一个与之前识别的大豆小rna互补,或者预测为被它们靶向。事实上,大豆mir171k是被预测靶向gmham4a/gmham4b的唯一小rna,但主要在未接种的根部中表达(每百万读数为9.38个)而不是根瘤中(每百万读数为0.27个),因此它不太可能是造成根瘤中所观察到抑制gmham4a/gmham4b的原因。因此,数据表明,事实上,新识别的根瘤菌trf与所述大豆基因之间存在跨界交流,从而证实所述根瘤菌trf确实参与了结瘤的调控。
[0317]
为了理解单个trf在抑制其靶标的重要性,合成了两种人工小rna,即asrna001(与bj-trf001相同)和asrna001
*
(与asrna001互补)(图6d),并分别将其转化为在无菌组织培养中生长的大豆(品种williams 82)根部制备的原生质体。与asrna001
*
转化原生质体中的gmrhd3a和gmrhd3b转录本相比,在所述asrna001转化原生质体中检测到gmrhd3a和gmrhd3b转录本显著地或大量地减少(图6e),证实了单个21-nt根瘤菌trf足以在转录后抑制其靶标。
[0318]
实施例3
[0319]
trf靶基因突变引发根瘤
[0320]
迄今为止,尚不清楚gmrhd3a/gmrhd3b、gmham4a/gmham4b和gmlrx5在大豆中的潜在功能以及它们的同源物在其他豆科植物中的潜在功能。由于这些基因被如先前所述仅在大豆根瘤中局部检测到根瘤菌trf下调,因此考虑这些基因与结瘤有关。为了确定根瘤中抑制gmrhd3a/gmrhd3b、gmham4a/gmham4b和gmlrx5表达是否实际上与结瘤相关,根部突变经由crispr-cas9(cr)(图7a和图7b)通过针对每个所述五种基因和每个所述两个复制基因对的两个拷贝的毛根转化,然后接种根瘤菌菌株usda110产生。对照是用于cr敲除的空载体的转基因根部。
[0321]
在所有情况下,所编辑基因的表达比所述空载体转基因对照的表达产生更多的根
瘤。此外,双突变体(gmrhd3a/gmrhd3b和gmham4a/gmham4b)产生的根瘤最多。图8a显示了每个突变体根部和所述对照根部的照片,所有突变体都通过cr敲除已识别的trf靶标,使得根瘤数显著增加。图8b还显示了所得根瘤数的图形表示。
[0322]
实施例4
[0323]
trf靶基因的过度表达减少了根瘤数
[0324]
还检查了根瘤中gmrhd3b、gmham4a和gmlrx5的过度表达对结瘤的影响。制备的转基因根部分别通过花椰菜花叶病毒(camv)35s启动子过表达gmrhd3b、gmham4a或gmlrx5,这增加了此类基因(即trf靶标)的表达。从图9a至图9c中所见,与所述对照非转基因根部相比,这些基因的过度表达使得所述基因过度表达根部中的根瘤数显著减少。结合本文提供的其他研究的数据,这证实了这些基因是结瘤负调控因子。
[0325]
实施例5
[0326]
根瘤菌trf通过切割靶基因的转录本直接作用于结瘤
[0327]
为了检查单个根瘤菌trf通过从其靶标切割转录本而对结瘤的直接影响,生成了转基因短串联模拟(sttm)大豆根部以沉默所述三种根瘤菌trf中的每一种。如图10a所示,在完成本文所述的sttm方案后,所述三种trf中每一种的表达几乎被完全阻断;因此,所识别基因的表达水平显著增加。图10d显示了在去除根瘤的sttm毛状根部和根瘤(对照(c)和sttm根部/根瘤(s))中根瘤菌trf丰度的减少和大豆基因表达的增加。
[0328]
如图10b和图1oc所示,与所述空载体转基因对照的根瘤数相比,所述sttm根部中的根瘤数显著减少。正如预期的那样,拟三种trf的相对丰度也降低了,并且所识别靶标的表达增加了,从而证实这些trf是结瘤正调控因子并通过抑制相关靶基因起作用。考虑到这些根瘤菌trf与整个基因组中任何其他大豆基因的转录本缺乏序列互补性,因此所述trf对根瘤形成的影响很可能直接源于对其靶标的抑制表达。
[0329]
实施例6
[0330]
根瘤菌感染期间根瘤菌trf和所述宿主细胞中靶基因相互作用
[0331]
由于根瘤菌感染是引发根瘤的关键步骤,因此检测了所述五种大豆靶基因的trf介导调控如何影响根瘤菌感染。然后考察了在接种usda110后早期大豆根毛中所述五种trf靶标的表达模式。
[0332]
在检测的所有五个时间点,靶向这些基因的三种trf丰度从6hpi增加到72hpi(图11a)。相比之下,与未接种的根毛相比,所有五种基因在接种的根毛中的表达水平均表现出显著降低(图11b)。所有五种基因也显示出从6hpi到72hpi表达的降低。这些数据证实了在根瘤菌感染期间,根瘤菌trf与所述宿主细胞中其靶基因之间确实发生了相互作用。
[0333]
实施例7
[0334]
根部表型与靶基因表达和根瘤菌trf沉默相关
[0335]
在根瘤菌感染早期对接种的根毛中宿主基因表达进行分析后,检测了其中gmrhd3a、gmham4a和gmlrx5被所述35s启动子过表达的接种根毛的形态,以及其中靶向gmrhd3a/gmrhd3b、gmham4a/gmham4b和gmlrx5的所述三种根瘤菌trf中的每一种都被sttm沉默的接种根毛形态(这两项研究都使用载体转基因根部作为对照)。对于每种毛状根部,在成熟区切割、固定并且在显微镜下检查1cm的片断。
[0336]
在所述三种gmrhd3a、gmham4a和gmlrx5过表达根部的每一种与所述对照之间,或
在trf沉默sttm根部与所述对照之间,未观察到根毛数和根毛长度的显著性差异(图12);然而,与各自的对照相比,变形和卷曲根毛的比例在三种过表达根部的每一种和sttm根部中都显著降低(图13,分图a至分图c)。据记载,豆科植物根部中根瘤菌nod因子感知可诱导结瘤基因的表达,并使得捕获根瘤菌形成根瘤的根毛卷曲。鉴于此,目前所述的数据证实根毛卷曲和根瘤数均受根瘤菌trf的正向调控,并且根瘤菌trf促进根瘤菌感染。
[0337]
实施例8
[0338]
验证根瘤菌trf机制
[0339]
为了更好地了解根瘤菌trf调控结瘤的机制并确认某些构建体的表达可以在植物体内产生所需的“人工trf”,通过用根瘤菌trf001及其互补trf001
*
或用trf003及其互补trf003
*
替换大豆mir172a前体mir172a的mir172a序列和mir172a
*
序列来构建两种人工mirna前体(amir-trf001和amir-trf003)。图14d说明了这种人工mirna基因的结构。在所述35s启动子的控制下,amir-trf001和amir-trf003(与bj-trf001和bj-trf003相同/互补)单独在williams 82毛状根部中表达,从而在所述转基因根部中产生人工mirna amir-tfr001和amir-tfr003(参见图14a,对照数据标记为c,转基因根数据标记为x)。
[0340]
如图14b所示,与空载体转基因对照(数据标记为c)的情况相比,所述amir-tfr001和amir-tfr003靶标gmrhd3a/3b和gmlrx5(数据标记为x)的表达显著降低。同样地,如图14c中所示,在所述amir-trf001和amir-trf003转基因根部(数据标记为x)中比在各自的对照(数据标记为c)中产生了更多的根瘤。这些结果证实了所述人工mirna/trf序列确实使得在根部中生成所需的人工trf,并直接抑制所识别的宿主基因靶标以促进结瘤。
[0341]
为了确定人工mirna/trf介导的基因调控所需的序列互补程度,两组融合基因通过以下方式制备,即将dna片段的每个21碱基对(bp)(对应于所述三种已识别trf靶位点(野生型)的每种),以及在靶植物基因(突变型)所检测的切割位点处具有4-bp修饰的dna片段的每个21-bp添加到绿色荧光蛋白(gfp)基因的编码序列。然后在williams 82毛状根部中于35s启动子的控制下分别表达所述融合基因(参见图15,分图a)。
[0342]
如图15分图b和分图d所示,在接种usda110后约24小时,“野生型”根部(w1-w3)中gfp活性显著降低,而在“突变型”根部(m1-m3)中没有观察到gfp活性的变化。gfp转录本的相对丰度也与gfp活性一致(参见图15,分图c)。突变型植物基因不被所述trf切割,这有力地证明了本文所述的trf切割机制。因此,这些结果表明所述“野生型”融合基因通过其mrna在整合的“靶位点”与所述根瘤菌trf的碱基配对而受到负调控。
[0343]
实施例9
[0344]
通过agol途径的trf作用
[0345]
在拟南芥中,ago1是rna诱导的沉默复合物的组分,所述复合物可介导mirna引导的靶mrna切割。ago1的表达响应于几种植物病毒感染而上调,并且已显示其是造成mirna途径中互补靶mrna的翻译抑制或切割的原因。
[0346]
为了确定所述根瘤菌trf是否通过大豆中agol的功能对应物起作用,拟南芥agol的两种大豆直系同源物中的一种(gmagolb)与myc表位标签融合并在williams 82的毛状根部中表达,所述gmagolb在大豆根瘤中的转录本比另一种(gmagola)的转录本更丰富。然后所述融合蛋白通过来自usda110诱导的20天根瘤的myc抗体免疫沉淀。
[0347]
如图16所示,所有三种根瘤菌trf在被所述myc抗体免疫沉淀的gmagolb-myc相关
级分中均检测到,但在无所述抗体培养的根瘤裂解物中未检测到。此数据证明了,这些根瘤菌trf劫持了大豆ago1,转而催化在所述宿主细胞中trf引导的靶标mrna的切割。
[0348]
有趣的是,回到实施例6,所述trf介导的宿主基因表达调控实际上是在根瘤菌感染的早期阶段检测到的。在接种usda110后6至72小时的所有五个时间点,与未接种的根毛相比,接种的根毛中三种trf的丰度增加(图11a),而它们靶标的表达降低(图11b)。此数据证实了trf实际上采用类似mirna的机制来调控基因表达。
[0349]
实施例10
[0350]
不同豆科植物-根瘤菌伙伴中三种根瘤菌trf、trf靶位点及其对应序列的序列保守性
[0351]
为了确定根瘤菌trf介导的宿主基因调控在进化相关的豆科植物中是否保守,还分析了四种豆科植物(大豆、普通豆(菜豆)、蒺藜苜蓿和百脉根)和12个根瘤菌种的序列数据,以及大豆种群与相容的共生相互作用有关的gmrhd3a/gmrhd3b、gmham4a/gmham4b和gmlrx5序列。如图17和图18所示,在699个大豆品种中,未发现所述五个基因的三种trf靶位点的序列变异。此外,在8个大豆慢生根瘤菌菌株中,在各自根瘤菌trna内的三种trf位点没有检测到序列变异。图19a至图19c列出了保留所述三种trf靶位点cac、ucc和cug的细菌菌株/菌种,粗直黑条表示不同的根瘤菌群。
[0352]
相比之下,所述靶位点的序列在四种豆科植物中存在差异(数据未显示)。苜蓿中华根瘤菌(sm)、百脉根根瘤菌(ml)、大豆慢生根瘤菌(bj)和埃特里根瘤菌(re),是在科学研究中常用于分别接种蒺藜苜蓿(mtru)、百脉根(ljap)、大豆(gmax)和菜豆(pvul)的相容根瘤菌菌种。评估了来自四个根瘤菌菌种的对应于bj-trf001、bj-trf002和bj-trf003的根瘤菌dna序列(称为trf直系同源物)和来自四种豆科植物种的对应于三种trf靶位点的序列(靶位点直系同源物)。确定了任何trf直系同源物和任何靶位点直系同源物之间最高水平的序列互补性,psrnatart预测的只有(1)bj-trf001和gmax&pvul的序列互补性,(2)bj-trf002和gmax的序列互补性,以及(3)bj-trf003和gmax的序列互补性足以进行mirna引导切割。具体是,在其他三种豆科植物中没有发现gmlrx5的直系同源物。三种根瘤菌trf序列在各自trna中的对应物也显示出种间差异(数据未显示)。
[0353]
然而,虽然普通大豆中的pvrhd3(gmrhd3a/3b的直系同源物)确实具有与gmrhd3a/3b相同的trf001靶位点,但埃特里根瘤菌(普通大豆的相容共生伙伴)没有衍生出trf001的大豆慢生根瘤菌val-l-trna(cac)。使用来自埃特里根瘤菌菌株诱导的普通大豆根瘤的小rna数据,确定38种埃特里根瘤菌trna以产生21-nt trf。如图20a和图20b所示,这些trf主要衍生自所述trna的3'末端。
[0354]
预测10种不同的21-nt trf(每种在常见大豆植物根瘤中的相对丰度》100计数/百万根瘤菌小rna读数)靶向14种常见的大豆基因,包括编码蛋白激酶的基因、gras转录因子,以及被认为参与结瘤调控的类apetala2转录因子(见表4)。
[0355]
表4.埃特里根瘤菌的根瘤菌trf及其在常见大豆中的潜在靶标
[0356][0357][0358]
然而,常见大豆中这14种推定的埃特里根瘤菌trf靶标中没有一个是大豆中25个被靶向的大豆慢生根瘤菌trf靶标的直系同源物(见表2)。
[0359]
结果证实了,虽然使用细菌trf靶向宿主基因似乎是保守的,但所述trf及其靶基因可能因种而异。事实上,基于本文提供的数据,上表3的根瘤菌衍生序列和配对的埃特里根瘤菌trf基因靶标可以与本文所述的大豆慢生根瘤菌trf/基因靶标相关的相同方式利用。这些发现与本文介绍的其他研究相结合,突出了在结瘤形成过程中发生的调控网络的复杂性。
[0360]
因此,本文提供的数据表明,所述根瘤菌trf是大豆根瘤菌感染和根瘤形成的正调控因子,在平衡植物生长和共生方面发挥重要作用。除了本文确定的根瘤菌trf外,预计表2中确定的其他根瘤菌trf会靶向注释为编码生长素受体和外排载体、ring/u-box蛋白和蛋白激酶的大豆基因,这可能会影响结瘤。此外,数据证实了,这种跨界交流在共生伙伴中很常见,但根瘤菌trf-宿主基因相互作用的节点似乎是多种多样的。这些发现展示了基因组编辑对于增强豆科植物结瘤能力的有效性。
[0361]
虽然已经相当详细地描述了化合物、转基因植物/植物根部/植物细胞及其方法的各种实施方案,但这些实施方案仅作为非限制性示例而提供。根据本公开,对本领域普通技术人员而言,本文描述的实施方案的许多变化和修改将是显而易见的。因此,本领域技术人员将理解,在不脱离本公开范围的情况下,可以进行各种改变和修改,并且可以用等效物替代其要素。事实上,本公开并非详尽无遗或限制性太强。本公开的范围由所附权利要求及其等效物限定。
[0362]
此外,在描述代表性实施方案时,本公开可能已经将方法和/或过程呈现为特定的步骤序列。然而,就所述方法或过程不依赖于本文阐述的特定步骤顺序而言,所述方法或过程不应限于所描述的特定步骤顺序。本领域普通技术人员将理解,其他步骤顺序也是可能
的。因此,此处公开步骤的特定顺序不应被解释为对权利要求的限制。此外,针对方法和/或过程的权利要求不应限于按所写顺序执行其步骤,因此本领域技术人员可以容易地理解,所述顺序可以变化并且仍然保持在本公开的精神和范围内。
[0363]
因此,本说明书和所附权利要求将涵盖,基于本公开对本领域普通技术人员显而易见的所有修改和改变。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。