一种位于14号染色体的大豆高蛋白含量相关的分子标记和鉴定高蛋白含量大豆的方法
1.本技术为申请号202110583739.x、申请日2021年5月24日、发明名称“一种大豆高蛋白含量相关的分子标记和鉴定高蛋白含量大豆的方法”的分案申请。
技术领域
2.本发明属于生物技术领域,具体涉及一种位于14号染色体的大豆高蛋白含量相关的分子标记和鉴定高蛋白含量大豆的方法。
背景技术:
3.大豆有着丰富的营养成分,蛋白质在40%左右。大豆蛋白质中含有8种人体必需氨基酸,人们可以通过食用大豆用以补充所需的营养物质,并且可以预防人体的心血管疾病,大豆同时是重要的油料作物,可以被加工为食用油,可以满足人们的饮食需求,同时主要由五种脂肪酸组成,而脂肪酸可以预防心脏病、癌症等。随着人们的生活水平日益提高,越来越多的人更多的注重于食用健康与食物的营养价值,因此对大豆的需求很大,但我国的大豆更多的是依赖于从其他国家进口,所以我国急需提高大豆蛋白及培育高蛋白、高油大豆品种,满足人们的日常所需。
4.大豆子粒的蛋白是品质相关性状,是比较复杂的数量性状,受到多个基因控制,一直以来受到遗传特性和育种方法的限制,传统方法过于缓慢,随着科技的不断进步,分子辅助选择被提出,在传统杂交育种方法的基础上利用分子标记与决定目标性状的基因紧密连锁,通过对标记的选择来实现对性状的选择,大幅度提高育种效率,实现定向改良大豆品种的作用即可以选择出高蛋白的大豆品种。
技术实现要素:
5.本发明的目的是为了快速和准确的筛选高蛋白优质大豆品种,本发明提供了一种大豆高蛋白含量相关的分子标记,所述分子标记的核苷酸序列为snp1,所述snp1的序列为大豆1号染色体上50.84mb-50.87mb位置的的核苷酸序列,且gm01号染色体的第50861576核苷酸位点为a或c。
6.在一种实施方式中,扩增snp1的引物为gm01_50861576-f和gm01_50861576-r,所述gm01_50861576-f的核苷酸序列如seq id no.15或如seq id no.16所示;所述gm01_50861576-r的核苷酸序列如seq id no.17所示。
7.本发明还提供了一种大豆高蛋白含量相关的分子标记,所述分子标记的核苷酸序列为snp2,所述snp2的序列为大豆6号染色体上38.49mb-47.89mb位置的的核苷酸序列,且gm06号染色体的第44869874核苷酸位点为a或g。
8.在一种实施方式中,扩增snp2的引物为gm06_44869874-f和gm14_16525645-r,所述gm06_44869874-f的核苷酸序列如seq id no.51或如seq id no.52所示;所述gm06_44869874-r核苷酸序列如seq id no.53所示。
9.本发明还提供了一种大豆高蛋白含量相关的分子标记,所述分子标记的核苷酸序列为snp3,所述snp3的序列为大豆14号染色体上16.13mb-16.66mb位置的的核苷酸序列,且gm14号染色体的第16525645核苷酸位点为a或t。
10.在一种实施方式中,扩增snp3的引物为gm14_16525645-f和gm14_16525645-r,所述gm14_16525645-f的核苷酸序列如seq id no.84或如seq id no.85所示;所述gm14_16525645-r核苷酸序列如seq id no.86所示。
11.本发明还提供了一种snp1、snp2和snp3分子标记在制备鉴定高蛋白含量的大豆的试剂盒中的应用,用(a)~(c)任一组引物扩增snp1、snp2和snp3分子标记:
12.(a)扩增snp1的上游引物的核苷酸序列如seq id no.15或如seq id no.16所示;扩增snp1的下游引物的核苷酸序列如seq id no.17所示;
13.(b)扩增权利snp2的上游引物的核苷酸序列如seq id no.51或如seq id no.52所示;扩增snp2的下游引物的核苷酸序列如seq id no.53所示;
14.(c)扩增snp3的上游引物的核苷酸序列如seq id no.84或如seq id no.85所示;扩增snp3的下游引物的核苷酸序列如seq id no.86所示。
15.本发明还提供了一种鉴定高蛋白含量大豆的方法,所述方法的具体步骤为:
16.(1)提取待检测大豆的dna;
17.(2)利用snp1分子标记的引物进行pcr反应,检测待检测品种的大豆为cc基因型则待检测品种的大豆为高蛋白含量的大豆,若为aa基因型则待检测品种的大豆为低蛋白含量的大豆。
18.本发明还提供了一种鉴定高蛋白含量大豆的方法,所述方法的具体步骤为:
19.(1)提取待检测大豆的dna;
20.(2)利用snp2分子标记的引物进行pcr反应,检测待检测品种的大豆为gg基因型则待检测品种的大豆为高蛋白含量的大豆,若为aa基因型则待检测品种的大豆为低蛋白含量的大豆。
21.本发明还提供了一种鉴定高蛋白含量大豆的方法,所述方法的具体步骤为:
22.(1)提取待检测大豆的dna;
23.(2)利用snp3分子标记的引物进行pcr反应,检测待检测品种的大豆为aa基因型则待检测品种的大豆为高蛋白含量的大豆,若为tt基因型为低蛋白含量的大豆。
24.有益效果:本研究利用643份经过全基因组重测序的资源群体结合其2年3次重复的大豆子粒贮藏物质的表型数据,利用分层评价的方法筛选出与大豆子粒蛋白、油分极显著相关的snp位点,采用snp分子标记技术中的kasp在151份大豆非测序极端蛋白资源材料及162份大豆非测序极端油分资源材料中进行验证,根据其分型结果及其表型数据,开发出与大豆蛋白、油分相关的分子标记,用以在生产中提前筛选高蛋白、高油分优质品种提供一种高速准确的方法。
附图说明
25.图1为2018、2019年资源测序材料蛋白、油分含量及blup分布直方图,其中,图a为2018年蛋白含量分布直方图,b是2019年蛋白含量分布直方图,c是2年蛋白blup分布直方图,横坐标是组别,纵坐标是频率;
26.图2为20条染色体上snp位点数目分布,其中,横坐标是染色体,纵坐标是snp个数;
27.图3为20条染色体上与蛋白相关的snp位点数目分布,其中,横坐标是染色体,纵坐标是snp个数;
28.图4为与蛋白相关的snp位点突变基因组对应等位基因与参考基因组对应表型效应值差值,其中,横坐标是组别,纵坐标是表型效应之差值;
29.图5为与蛋白相关snp位点的高蛋白优异单倍型与低蛋白单倍型表型均值,其中,横坐标是组别,纵坐标是蛋白质含量;
30.图6为snp标记在151个大豆极端蛋白资源材料中的kasp基因分型。
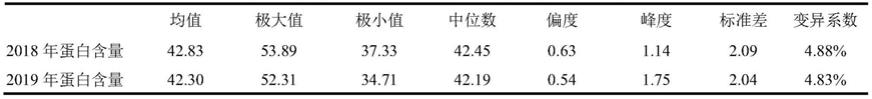
具体实施例
31.大豆蛋白含量的mqtl记载在文献qi et al.2018meta-analysis and transcriptome profiling reveal hub genes for soybean seed storage composition during seed development。
32.实施例1.
33.实验群体:选取643份东北地区大豆核心种质测序资源作为实验群体,于2018、2019年在吉林省农科院及东北农业大学向阳农场试验田中种植且3次重复,1m行长,每1行播撒20粒种子,播种的深度为3-4cm,田间管理方法同大田管理,收获后考察各个性状,选择长势一致的5株进行测量。在营养生长阶段,采取植株顶端最幼嫩处叶片用于提取dna,收获时每个株系随机取5株进行脱粒,用于测定蛋白质含量。
34.一、大豆子粒蛋白含量测定及处理:使用foss谷物分析仪(infratec1241)用大量法测出实验材料及验证材料的蛋白含量,该仪器利用近红外透射技术进行全谱扫描,可获得丰富的光谱信息,通过对比定标数据库从而获得精度非常高的蛋白含量表型数据。测量时要保证子粒在安全含水量范围内,每份单株子粒重复测量3次,取3次测量数据的平均值作为最终蛋白含量表型数据。利用microsoft office excel2013进行表型数据处理,取其平均值,利用spss统计分析2年3次重复的大豆子粒蛋白,包括显著性检验,频率分布直方图,计算均值等。通过r软件进行最佳线性无偏预测值(the best linear unbiased predictions,blup)的计算。
35.由表1可知,该群体1个品质相关性状两年均无较大变幅,变异系数在4%-5%之间,蛋白这个性状标准差较小,2年分别为2.09、2.04。通过分析峰度、偏度发现,该群体的1个品质相关性状的蛋白表现中等偏态分布,将2018、2019两年蛋白油分表型数据进行blup分析,并将2018、2019两年蛋白均值(图1中的a和b)及blup值利用spss软件做了正态分布图(图1中的c),从图中可看出测得的大豆子粒蛋白的两年数据和blup值均显示连续分布,而且分布趋势均很明显,从正态曲线上也能得知大豆子粒蛋白均呈现出正态分布。其次,从蛋白两年及其blup值的图中可以看出,blup值的峰值较2年数据较低一些,数据分布也较广且较均匀;这个品质性状的blup值分布特征也符合数量性状遗传特点,更适合利用blup值进行后续分析研究。
36.表1大豆测序材料2018、2019年蛋白品质性状的描述性分析
[0037][0038]
dna提取方法:取大豆3-4g新鲜叶片装入1.5ml离心管,再加入3颗3mm灭菌的小钢珠。将离心管浸于液氮中,应用组织研磨仪将冻干的叶片组织震荡成粉末。加入650-700μl预先在65℃水浴锅中水浴的ctab提取液,置于涡旋震荡仪上充分混匀,65℃水浴1小时,每15min颠倒混匀一次。加入等体积氯仿充分混匀,12,000rpm离心20min,吸取上清液注入新的离心管中,重新加入700μl氯仿,充分颠倒混匀后离心12,000rpm,20min,吸取上清匀速滴加入预先装有-20℃预冷异丙醇的离心管中,-20℃环境下保存20min。8,000rpm离心10min,将上清液倒入废液缸,分别用无水乙醇和75%乙醇洗涤底部团状dna。将离心管开盖置于超净工作台中吹干,加入灭菌水。用分光光度计(nanodrop)检测提取得到的dna的质量水平,利用琼脂糖凝胶电泳检测出dna浓度,统一稀释到工作液浓度20ng/μl。
[0039]
二、资源测序材料snp位点的分层评价:选取643份大豆资源群体进行重测序,20条染色体共获得snp位点53,946个,snp质量控制maf《0.05,杂合率《10%,平均每条染色体含有2,697个。其中chr18染色体中snp数量最多,有4,462个;chr11染色体中snp数量最少,有862个,其余染色体上的snp数目见图2。
[0040]
三、重要等位基因挖掘:(1)针对表型将材料进行分类,分别按照研究的性状不同即大豆蛋白,求其所有数据平均值及其标准差,将平均值分别加减标准差所得数据分别作为临界,高于平均值加标准差材料作为高蛋白。(2)把研究的该snp位点的测序结果根据是参考基因组的等位基因还是突变后基因组对应的等位基因统计分析,并且结合表型数据即蛋白、油分按照平均值加减一倍标准差作为标准将材料进行分类,取两端高于和低于该标准材料,将所统计得到的数据列入如下四联表如表2中,进行卡方检测:
[0041]
表2卡方分析的四联表
[0042][0043]
注:i为a/c/t/g,a
11
为高蛋白中有i等位基因的数量,a
21
为高蛋白中无i等位基因的数量,a
12
为低蛋白中有i等位基因的数量,a
22
为低蛋白中无i等位基因的数量,c1为高蛋白的总数,c2为高蛋白的总数,r1为有i等位基因的总数,r2为无i等位基因的总数,n为材料的总数。
[0044]
(3)原假设h0:蛋白含量的大小与i等位基因相互独立,ha:2个变数有关联。通过下列公式得到χ2结果,当所得到的χ2<χ2α时,同意h0成立;当所得到的χ2≥χ2α时,不同意h0成立,ha成立。因此根据得到的χ2值与α=0.001时的阈值针对研究的snp位点进行判断。
[0045][0046]
(4)重复(2)、(3)步骤,对蛋白性状的所有snp位点进行独立性检验,用以来判断对该研究性状的具体影响,本实验以alpha=0.001作为阈值,当所得到的结果对应的p<0.001时,则判断该snp位点为影响该性状的显著位点,进行后续研究,而p≥0.001时,则放
弃继续研究该位点。(5)对于(4)步骤所选出的显著位点进行下一步的表型效应值验证,在所有材料中对显著位点进行表型效应计算,通过以下公式:注:rate of change表示效应值,a等位基因表示该snp位点的参考基因组对应的等位基因,value a表示有着a样品的蛋白的均值,b等位基因表示该snp位点的突变基因组对应的等位基因,value b表示有着b样品蛋白含量的均值。
[0047]
结果:通过以蛋白表型的平均值加减一倍标准差为标准取两端蛋白表型及测序结果,将其进行重要等位基因挖掘,通过利用卡方分析方法,选择显著性alpha=0.001作为分层阈值,将分析所得到结果进行分层,当检验p值小于0.001时,我们将其认为是与蛋白相关的极显著的snp位点,其余p值大于0.001的相较之下效应相对小,选择意义甚小,因此得到与蛋白相关极显著位点个数为7,404,其中4号染色体上最多,达1,537个,其他染色体上数目见图3,为进一步挖掘好的位点因此针对其snp位点等位基因突变分别在高低表型中占比进行进一步限制,并且将其得到极显著位点与大豆蛋白mqtl结果相比较找到重合区间的位点,比较后得到的相关snp位点个数为147个,与蛋白相关的snp位点在14号染色体上最多,达48个。
[0048]
经卡方检测及与mqtl对比得到的关键snp位点突变前后的效应各不相同,有些是对蛋白含量起正向效应,有些为负向效应:如果含有某等位基因的蛋白含量平均值比所有资源的蛋白含量平均值高,那么该等位基因能够增加蛋白含量;相反,如果含有其突变等位基因的蛋白含量平均值比所有资源蛋白含量平均值低,那么该等位基因具有能够减少蛋白含量的作用。snp位点对蛋白含量的效应值有所不同(如图4),图中上方的点列出的是突变对应等位基因的表型效应值与参考基因组含有等位基因对应的表型效应值的差值为正值,即突变后蛋白含量增加,图中下方的点是突变对应等位基因的表型效应值与参考基因组对应等位基因的表型效应值的差值为负值,即突变后蛋白含量减少。
[0049]
在1号染色体上50.84mb-50.87mb的5个snp位点,其占高蛋白的比例为63.17-66.99%,其表型效应率在1.56%-2.44%;在3号染色体上36.79-38.93的34个snp位点,其占高蛋白的比例为60.19%-64.52%,其表型效应率在1.92%-2.59%;在5号染色体上40081658、40270960处的2个snp位点,其占高蛋白的比例分别为60.19%、61.17%,其表型效应率均为2.43%;在6号染色体上38.49-47.89的37个snp位点,其占高蛋白的比例为60.22%-73.79%,其表型效应率在1.65%-2.52%;在8号染色体上9.23-9.24的5个snp位点,其占高蛋白的比例为60.19%-64.08%,其表型效应率在1.30%-1.71%;在11号染色体上14918130处的snp位点,其占高蛋白的比例为60.19%,其表型效应率为2.29%;在13号染色体上1389378处的snp位点,其占高蛋白的比例为62.14%,其表型效应率2.51%;在14号染色体上16.13-16.66的48个snp位点,其占高蛋白的比例为60.22%-77.67%,其表型效应率在1.76%-2.69%;在19号染色体上42.65-42.75的4个snp位点,其占高蛋白的比例为62.14%-65.59%,其表型效应率在1.39%-1.76%;在20号染色体上33.67-33.99的9个snp位点,其占高蛋白的比例为60.19%-62.14%,其表型效应率在1.56%-2.29%(结果如表3)。以上大豆的染色体的信息是来自于网站:https://phytozome.jgi.doe.gov/pz/portal.html#!info?alias=org_gmax。
[0050]
表3筛选后的与蛋白相关的snp位点
[0051][0052]
四、蛋白相关snp位点单倍型分析:为了确定所得到的与蛋白相关snp位点与蛋白的关系,分析147个其变异位点的单倍型,将接近的位点分为一组共同分析,共得到46组,每组均产生不同单倍型,分析得到该单倍型在643份测序材料中所占比例,并计算出该单倍型的表型均值,最后分析得到分析14组位点存在高蛋白优异单倍型和低蛋白单倍型的蛋白表型均值有较大差异,能够较好的达到蛋白的分离见图5。
[0053]
在1号染色体上50836411(代表在染色体上的位置)、50838581、50840858、50854308、50861576处分析得到,高蛋白优异单倍型hap_1(tcccc)和低蛋白单倍型hap_4(tccca),高蛋白优异单倍型占34.8%,蛋白主要分布在41-48左右,低蛋白单倍型占1.5%,蛋白主要分布在40-43,达到明显差异;在3号染色体上36911977、36956744、36976313、37015622处分析得到,高蛋白优异单倍型hap_1(tactcatattac)和低蛋白单倍型hap_35(caaatgagccga),高蛋白优异单倍型占36.12%,蛋白主要分布在41-48左右,低蛋白单倍型占1.17%,蛋白主要分布在40-42,达到明显差异;在3号染色体上38222729处分析得到,高蛋白优异单倍型hap_4(acgcaatgtaga)和低蛋白单倍型hap_102(cgatggtaacat),高蛋白优异单倍型占42.31%,蛋白主要分布在41-47.5左右,低蛋白单倍型占1.17%,蛋白主要分布在39-40,达到明显差异;在5号染色体上40081658处分析得到,高蛋白优异单倍型hap_5(tagttccctctca)和低蛋白单倍型hap_12(taattccctctca),高蛋白优异单倍型占20.40%,蛋白主要分布在41-46左右,低蛋白单倍型占1.67%,蛋白主要分布在39-43,达到明显差异;在5号染色体上40270960处分析得到,高蛋白优异单倍型hap_1(ccaggttagccga)和低蛋白单倍型hap_10(ccgtgttagccga),高蛋白优异单倍型占23.91%,蛋白主要分布在41-48左右,低蛋白单倍型占1.00%,蛋白主要分布在36-40,达到明显差异;在6号染色体上44869874、45732460、46313677、46682433、47893908处分析得到,高蛋白优异单倍型hap_5(gtggcgcctg)和低蛋白单倍型hap_60(actatactcc),高蛋白优异单倍型占19.23%,蛋白主要分布在41-48左右,低蛋白单倍型占1.17%,蛋白主要分布在38-42,达到明显差异;在11号染色体上14918130处分析得到,高蛋白优异单倍型hap_3(aagtcagtagcaaatggca)和低蛋白单倍型hap_58(tgactctgaaaggggta tg),高蛋白优异单倍型占39.46%,蛋白主要分布在41-48左右,低蛋白单倍型占1.17%,蛋白主要分布在38-42,达到明显差异;在13号染色体上13893781处分析得到,高蛋白优异单倍型hap_1(aatggacaggagca)和低蛋白单倍型hap_27(aatggacagaagca),高蛋白优异单倍型占24.75%,蛋白主要分布在41-48左右,低蛋白单倍型占1.00%,蛋白主要分布在40-41,达到明显差异;在13号染色体上13893781处分析得到,高蛋白优异单倍型hap_1(aatggacaggagca)和低蛋白单倍型hap_27(aatggacagaagca),高蛋白优异单倍型占24.75%,蛋白主要分布在41-48左右,低蛋白单倍型占1.00%,蛋白主要分布在40-41,达到明显差异。
[0054]
五、验证群体:选取151份东北地区大豆核心非测序极端蛋白资源材料(表4),用于重要等位基因挖掘的验证,种植、管理、采样、收获方法同实验材料。
[0055]
表4 151份大豆非测序极端蛋白材料品种名称及蛋白含量
[0056][0057][0058]
snp位点的标记筛选及方法:kasp反应体系由混合引物,master mix和样本dna组成。根据分层评价得到的snp位点,通过本地blast提取snp位点上下游各50bp的碱基序列,利用primer 5.0软件设计kasp引物。每一个位点的引物皆由2条具有不同等位基因和荧光标签的特异性正向引物(f1/f2)以及1条公共的反向引物(r)构成,其中各组分主要配方分别是46μl的ddh2o,正向引物各12μl(100μmol
·
l-1)和反向引物30μl(100μmol
·
l-1),master mix来自于lgc公司。荧光标签fam:gaaggtgaccaagttcatgct(seq id no.1),荧光标签hex:gaaggtcggagtcaacggatt(seq id no.2),引物序列信息如表5。
[0059]
表5引物序列信息
[0060]
[0061][0062]
kasp反应所需组分加于384孔板,采用罗氏lightcycler480ⅱ实时荧光定量pcr仪并在反应终止后读取终端荧光信号,其pcr扩增程序:95℃,15min;95℃,20s;65℃,25s;go to step 2,10cycles,-0.8℃ per cycle;95℃,10s;57℃,1min;go to step 4,35cycles;4℃,∞。
[0063]
kasp分型验证:罗氏lightcycler480ⅱ得到分型结果,转置到excel软件,进行分析,计算该位点符合率,基本思路为:(1)根据不同的极端大豆蛋白、油分非测序材料,统计出每个引物在高蛋白、低蛋白材料或高油分、低油分材料中snp位点的参考基因组对应的等位基因及突变基因组对应的等位基因的个数及分布,构建符合率的四格表如表6所示:
[0064]
表6符合率的四格表
[0065][0066]
注:x、y为snp位点设计引物的kasp分型的基因型,a为非测序高蛋白材料分型结果中有x等位基因数量,b为非测序低蛋白材料分型结果中有x等位基因数量,c为非测序高蛋白材料分型结果中有y等位基因数量,d为非测序低蛋白材料分型结果中有y等位基因数量,m为非测序高蛋白材料总数,n为非测序低蛋白材料的总数。
[0067]
(2)原假设h0:含量的大小与x/y等位基因相互独立,ha:2个变数有关联。通过下列得到符合率p1、p2,当所得到的p1《p
α
或p2《p
α
时,同意h0成立;当所得到的p1≥p
α
且p2≥p
α
时,不同意h0成立,ha成立。故依据计算出来的p1、p2值与α=60%时的阈值对每个引物的结果进行判断。
[0068][0069]
(3)重复(1)、(2)步骤,对蛋白性状的所有引物分型结果进行独立性检验,用以来验证对该性状的影响,对于(3)步骤所得到的所有结果进行进一步表型效应验证,在所有材料中对主效位行表型效应计算,利用以下公式:
[0070][0071]
结果:利用蛋白的29个标记进行分型验证,利用kasp对极端蛋白非测序资源材料151份进行基因分型。与蛋白相关的标记最终20个分型成功,图6表示,1个kasp分型成功标记的的结果示意图,其中表示2种不同的纯合等位基因型(gg、aa),表示合基因型(ag)。表示蛋白相关snp标记的kasp验证结果,分析可知与蛋白相关的gm01_50861576标记在高蛋白材料中54份cc基因型,低蛋白材料中42份为aa基因型,分别占高蛋白、低蛋白材料为70.13%和56.76%,其表型效应值为3.31%;与蛋白相关的gm06_44869874标记在高蛋白材料中73份gg基因型,低蛋白材料中57份为aa基因型,分别占高蛋白、低蛋白材料为94.81%和77.03%,其表其表型效应值为8.34%;与蛋白相关的gm14_16525645标记在高蛋白材料中41份aa基因型,低蛋白材料中51份为tt基因型,分别占高蛋白、低蛋白材料为53.25%和68.92%,其表型效应值为2.24%,以上3个标记均能够成功分型且在高低蛋白中表现不同
的基因型,能够成功开发出snp标记。
[0072]
一种大豆高蛋白含量相关的分子标记,所述分子标记的核苷酸序列为snp1,所述snp1的序列为大豆1号染色体上50.84mb-50.87mb位置的的核苷酸序列,且gm01号染色体的第50861576核苷酸位点为a或c;
[0073]
一种大豆高蛋白含量相关的分子标记,所述分子标记的核苷酸序列为snp2,所述snp2的序列为大豆6号染色体上38.49mb-47.89mb位置的的核苷酸序列,且gm06号染色体的第44869874核苷酸位点为a或g。
[0074]
一种大豆高蛋白含量相关的分子标记,所述分子标记的核苷酸序列为snp3,所述snp3的序列为大豆14号染色体上16.13mb-16.66mb位置的的核苷酸序列,且gm14号染色体的第16525645核苷酸位点为a或t。
[0075]
实施例2.
[0076]
一、一种筛选高蛋白大豆的试剂盒:
[0077]
(a)扩增snp1的上游引物的核苷酸序列如seq id no.15或如seq id no.16所示;扩增snp1的下游引物的核苷酸序列如seq id no.17所示;
[0078]
(b)扩增snp2的上游引物的核苷酸序列如seq id no.51或如seq id no.52所示;扩增snp2的下游引物的核苷酸序列如seq id no.53所示;
[0079]
(c)扩增snp3的上游引物的核苷酸序列如seq id no.84或如seq id no.85所示;扩增snp3的下游引物的核苷酸序列如seq id no.86所示。
[0080]
筛选方法:选择未知大豆蛋白含量的样品,利用步骤一所述的筛选高蛋白大豆的试剂盒,进行pcr扩增程序:95℃,15min;95℃,20s;65℃,25s;go to step 2,10cycles,-0.8℃ per cycle;95℃,10s;57℃,1min;go to step 4,35cycles;4℃,∞。经过kasp分析,步骤如下:
[0081]
二、一种鉴定高蛋白含量大豆的方法,所述方法的具体步骤为:
[0082]
(1)提取待检测大豆的dna;
[0083]
(2)利用snp1分子标记的引物进行pcr反应,检测待检测品种的大豆为cc基因型则待检测品种的大豆为高蛋白含量的大豆,若为aa基因型则待检测品种的大豆为低蛋白含量的大豆;利用snp2分子标记的引物进行pcr反应,检测待检测品种的大豆为gg基因型则待检测品种的大豆为高蛋白含量的大豆,若为aa基因型则待检测品种的大豆为低蛋白含量的大豆;利用snp3分子标记的引物进行pcr反应,检测待检测品种的大豆为aa基因型则待检测品种的大豆为高蛋白含量的大豆,若为tt基因型为低蛋白含量的大豆。
[0084]
结果:检测未知的大豆蛋白含量的样品中大豆蛋白含量有大于42%,利用基因型为snp1标记检测为cc基因型,snp2标记检测为gg基因型,snp3标记检测为aa基因型,大豆高蛋白的含量与标记检测得到的基因型是相符的。大豆低蛋白的含量与标记检测得到的基因型是相符的。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。
