carcinoma.cancerresearch.2016;76(7):1954.
10.3.buck mj,raaijmakers lm,ramakrishnan s,et al.alterations in chromatin accessibility and dna methylation in clear cell renal cell carcinoma.oncogene.2013;33(41):4961-4965.
11.4.cohen,herbert t,f.j.mcgovern.renal-cell carcinoma.n.engl.j.med 353.23(2005):2477
12.5.feinberg,andrew p,tycko,et al.the history of cancer epigenetics[j].4(2):143-153.
[0013]
6.celik h,kramer a,challen g a.dna methylation in normal and malignant hematopoiesis[j].international journal of hematology,2016,103(6):617-626.
[0014]
7.samuel t keating,assam el-osta.epigenetics and metabolism[j].circulation research,2015,116(4):715-736.
[0015]
8.demetriou c a,van veldhoven k,relton c,et al.biological embedding of early-life exposures and disease risk in humans:a role for dna methylation[j].european journal of clinical investigation,2015,45(3):303-332.
[0016]
9.du q,luu p l,stirzaker c,et al.methyl-cpg-binding domain proteins:readers of the epigenome[j].epigenomics,2015:1-23.
[0017]
10.wei j h,haddad a,wu k j,et al.a cpg-methylation-based assay to predict survival in clear cell renal cell carcinoma[j].nature communications,2015,6:8699.
[0018]
11.butcher lm,beck s.probe lasso:a novel method to rope in differentially methylated regions with 450k dna methylation data.methods.2015;72:21-28.
[0019]
12.cha k,yi l,yi gs.discovering gene expression signatures responding to tyrosine kinase inhibitor treatment in chronic myeloid leukemia.bmc medical genomics.2016;9(1):29.
[0020]
13.elena c,elizabeth m,rao s,et al.inactivation of the von hippel-lindau tumor suppressor leads to selective expression of a human endogenous retrovirus in kidney cancer.oncogene.2011;30(47):4697-4706.。
技术实现要素:
[0021]
本发明的目的在于基于现有技术的基础与现状,提供一种dna甲基化生物标志物,尤其是与肾透明细胞癌预后相关的dna甲基化标志物。
[0022]
本发明通过试验观察在肾透明细胞癌中dna甲基化相关标记物的变化与所属疾患预后状况的关系,对肿瘤组织和邻近的正常组织之间的全基因组甲基化水平、差异性甲基化位点和差异性甲基化区域进行了比较分析,确定了与肾透明细胞癌预后相关的生物标志物,并建立了相关预后模型。
[0023]
具体的,
[0024]
本发明提供了与肾透明细胞癌预后相关的生物标志物及其筛选方法。基于tcga、geo数据库,分析了肾透明细胞癌的485578个cpg位点的dna甲基化情况;筛选了肾癌组织和邻近正常组织间的差异性cpg位点和差异性表达基因,并确定二者之间存在负性相关的关系;本发明利用lasso
–
cox回归模型,筛选获得5种与肾透明细胞癌预后相关的生物标志物,分别是tp73(cg00295572,cg07382920,cg20611911,cg01915516)和ctbp2(cg05749728)。
[0025]
在本发明的一个方面,提供了肾透明细胞癌和毗邻的正常组织间的全基因组dna对比分析,发现二者的全基因组dna甲基化情况存在差异,包括:
[0026]
(1)在肾透明细胞癌组织和正常组织间存在14980个cpg位点高甲基化表达(δβ≥0.14,p<0.05);
[0027]
(2)在肾透明细胞癌组织和正常组织间存在14922个cpg位点低甲基化表达(δβ≥0.14,p<0.05)。
[0028]
在本发明的另一个方面,提供了51对肾透明细胞癌组织样本和正常组织样本之间的甲基化差异,包括:
[0029]
(1)在这102个样本中存在显著差异的前500个cpg位点;
[0030]
(2)1号染色体和2号染色体上的cpg位点表现出最高的甲基化水平;
[0031]
(3)18号染色体和21号染色体上的cpg位点表现出最低的甲基化水平;
[0032]
(4)除了1号、11号、16号和22号染色体,其余染色体更多地表现出低甲基化水平;
[0033]
(5)4号、5号、13号和18号染色体的甲基化水平增高幅度超过两倍。
[0034]
本发明提供了上述基因在生理过程中的作用,包括正性调控蛋白质的聚合,负性调控丝氨酸/苏氨酸蛋白激酶的活动和细胞基质的黏附。
[0035]
在本发明的另一方面,提供了与上述基因相关的信号通路,包括p53信号通路、notch信号通路、mtor信号通路、tgf-β信号通路、erbb信号通路和内吞等。
[0036]
本发明分析显示,在肾透明细胞癌患者中存在7066个差异性甲基化区域(dmr)(p<0.05)。
[0037]
在本发明进一步提供了存在差异性甲基化区域的染色体,分别是1号染色体(9.9%),2号染色体(8.2%),5号染色体(7.5%),6号染色体(7.4%),7号染色体(6.9%)和11号染色体(6.2%)。
[0038]
在本发明进一步提供了差异性甲基化区域(dmr)在基因内的分布情况:在基因内部
–
4192dmrs(59%);在启动子区域
–
798dmrs(11.3%);在5’重叠区域
–
135dmrs(1.9%);转录起始点下游
–
996dmrs(14%);转录起始点上游
–
893dmrs(12%)。
[0039]
本发明提供了上述差异性甲基化区域(dmr)在生理过程中的作用,包括细胞分化、细胞间信息传递、系统发育和发育过程、神经系统发育、解剖结构形态形成和骨骼系统的发育。
[0040]
本发明进一步提供了与这些区域相关的信号通路,分别是磷脂酰肌醇信号系统、p53信号通路、notch信号通路、胆汁分泌、病毒性心肌炎、胰腺分泌、mtor信号通路、霍乱弧菌感染、erbb信号通路、缝隙连接和神经胶质瘤等。
[0041]
本发明对27例肾透明细胞癌患者的肿瘤组织和正常组织的基因表达谱进行分析,该27例患者中,包括13例肾透明细胞癌未发生转移的早期患者,和14例肾透明细胞癌已经发生远处转移的患者,并分别显示出3304和5056个差异基因表达(p<0.05);其中,相对于正
常组织,未发生转移的肾透明细胞癌患者的肿瘤样本中有1516个基因表达上调,1743个基因表达下调;发生远处转移的肾透明细胞癌患者的肿瘤样本中有2514个基因表达上调,2542个基因表达下调。
[0042]
本发明进一步分析显示在所述的27例肾透明细胞癌患者样本中,约有70%的差异性甲基化cpg位点与基因表达存在负性相关的关系。
[0043]
本发明利用cox比例风险回归模型分析了320名肾透明细胞癌患者中267个cpg位点的甲基化情况,其中有24个cpg位点和患者的总生存期(os)显著相关(p<0.05)。所述的24个cpg位点中有15个基因已注解,分别是ncor2,ctbp2,hdac1,scarb1,rptor,ccnd1,kcnma1,vegfa,cdkn1b,pip4k2a,slc9a3,pten,prkcb,slc4a4和inpp5a。
[0044]
本发明采用lasso回归模型和kaplan-meier生存分析,结果显示tp73(cg00295572,cg07382920,cg20611911,cg01915516)和ctbp2(cg05749728)该五个cpg位点与患者的生存期密切相关。
[0045]
本发明进一步利用lasso
–
cox回归模型分析与所述的5个cpg位点甲基化相关的危险系数,危险系数=(0.5745
×
cg00295572) (1.1764
×
cg07382920) (0.4687
×
cg20611911) (0.2034
×
cg01915516) (0.0394
×
cg05749728);分析结果显示,所述的5个cpg位点的危险系数和肾透明细胞癌患者的生存期密切相关,危险系数越高,患者的生存期越短。
[0046]
本发明提供了与肾透明细胞癌预后相关的生物标志物及其筛选方法。本发明通过试验观察在肾透明细胞癌中dna甲基化相关标记物的变化与所属疾患预后状况的关系,对肿瘤组织和邻近的正常组织之间的全基因组甲基化水平、差异性甲基化位点和差异性甲基化区域进行了比较分析,确定了与肾透明细胞癌预后相关的生物标志物,并建立了相关预后模型。本发明所述的dna甲基化相关标记物可用于制备评估肾透明细胞癌预后的制品。
附图说明
[0047]
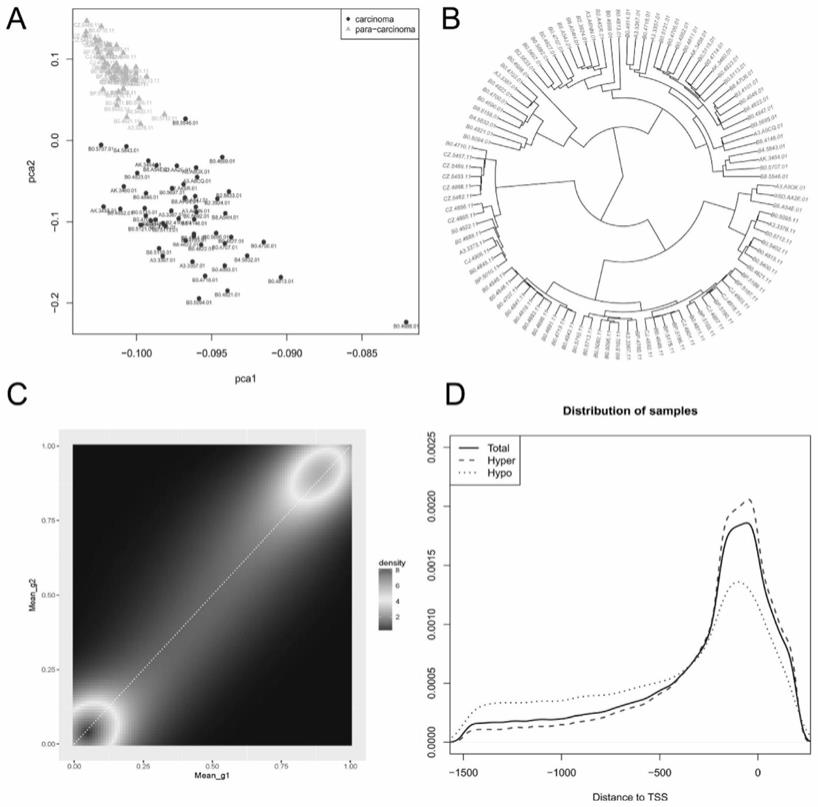
图1:ccrcc和邻,近正常组织的dna甲基化的情况,其中,
[0048]
(a)细胞内主要物质的450k甲基化芯片分析图,(b)450k甲基化芯片层次聚类分析图,红色代表ccrcc样本,蓝色代表相邻的正常组织样本,(c)ccrcc的全基因组的cpg甲基化水平始终低于邻近的正常组织,(d)在ccrcc中,低甲基化水平的cpg位点更多地分布在转录起始位点的上游或者是第一外显子区域内,高甲基化水平的cpg位点更多地分布在启动子区域(p<0.0001,威尔科克森秩和检验)。
[0049]
图2:ccrcc和邻近正常组织间差异性甲基化的特点,其中,
[0050]
(a)利用单变量分析选择出的500个cpg位点,火山图显示ccrcc和邻近正常组织甲基化水平的对比(n=102,humanmethylation450 platform),x轴代表生物学意义,y轴代表统计学意义(-log10 p),存在显著差异的前500个cpg位点的β>0.375,在肿瘤组织中有141个cpg位点(红色)的甲基化水平高于正常组织,有359个cpg位点(绿色)的甲基化水平低于正常组织,(b)使用t检验和调整后p≤0.05的ccrcc和正常组织样本的无监督聚类分析图,ccrcc组织样本总体呈现出低甲基化水平,红色区域代表高甲基化,蓝色区域代表低甲基化,(c)核型图显示了前500个存在显著差异的甲基化cpg位点。红色代表高甲基化,绿色代表低甲基化,每条染色体上的黑线区域代表全基因组被覆盖的区域,(d)ccrcc启动子内dna
的平均甲基化水平偏低。(e-f)由go和kegg数据库分别分析得到的散点图,富集值由某通路中差异性表达的基因的数目和总基因数目的比值得来,经p值校验后的q值范围为0~1,图中散点的颜色和形状分别代表了q值的范围和degs的数目,图中表示了前30条富集的信号通路。
[0051]
图3:ccrcc和正常组织间的差异性甲基化区域,其中,
[0052]
(a-b)51个蓝点和51个红点分别代表51份正常组织样本和51份肿瘤组织样本所在的基因区域,曲线代表了甲基化水平的平滑估计,其中蓝色代表肿瘤组织,红色代表正常组织,绿色横线所在的区域代表差异性甲基化区域(dmr),黑色曲线表示了正常组织和肿瘤组织总体水平上的差异,(c-d)由go和kegg数据库分析得到的散点图。
[0053]
图4:筛选的差异性表达的基因和27对ccrcc聚类分析的微阵列数据表,其中,
[0054]
(a)基于u133微阵列表达数据对基本组成成分的分析,(b)u133微阵列表达数据的层次聚类分析,红色代表ccrcc病人,蓝色代表毗邻的正常组织,(c-d)使用t检验和调整后p≤0.05的正常组织和ccrcc样本的无监督聚类分析图,红色区域代表过表达,蓝色区域代表低表达,(e-f)火山图中,两条竖线是2倍改变的边界线,水平线是具有统计学意义的边界(p<0.05),倍数改变≥2且有统计学意义的基因用红点表示,倍数改变≤1/2的基因用蓝点表示。
[0055]
图5:ccrcc中cpg甲基化和基因表达之间的联系,其中,
[0056]
(a-b)cpg甲基化和基因表达主要为负性相关的关系,在未发生转移和发生转移的ccrcc样本中分别存在70.4%和69.2%的差异性甲基化cpg位点。
[0057]
图6:五个cpg位点的相关分析,其中,
[0058]
(a-b)由lasso
–
cox回归分析得出的5个cpg位点,a,图中竖线所在的最佳值即为最小标准,b,24个cpg位点的lasso系数曲线,由lasso
–
cox回归模型得出的5个cpg位点——(cg00295572,cg07382920,cg20611911,cg01915516)和cg05749728的系数分别是0.5745,1.1764,0.4687,0.2034和0.0394,(c-h)ccrcc患者无复发生存率的kaplan
–
meier的生存曲线,其中:(c)cg00295572;(d)cg07382920;(e)cg20611911;(f)cg01915516;(g)cg05749728;(h)5个cpg的总影响。
具体实施方式
[0059]
下面结合具体实施实例,进一步阐释本发明,但本发明不局限于如下描述范围。
[0060]
实施例1、分析全基因组dna甲基化情况
[0061]
dna甲基化基础数据采集:从tcga数据库中下载320个肾透明细胞癌样本,样本包括以下信息:临床病理因素,如诊断时的年龄和阶段、淋巴结的状态以及人种和种族等:
[0062]
微阵列数据:利用美国国家生物技术中心(ncbi)的基因表达谱(geo,https://www.ncbi.nlm.nih.gov/geo/)对基因表达数据集(gse66272)进行分析。其中,gse66272包括27对肾透明细胞癌组织和其毗邻的正常组织(平台:gpl96 affymetrix human genome u133a array)。
[0063]
实施例2、分析ccrcc和邻近正常组织间差异性表达的cpg位点和差异性表达的基因之间的关系
[0064]
甲基化数据处理:使用tcga level-3数据库分析相关cpg位点的β值和cpg位点所
在位置、所在染色体;
[0065]
β值由(m/m u)计算得,其数值范围是从0到1,其中,m代表甲基化,u代表非甲基化,本发明移除了染色体x、y上的cpg探针以消除性别的差异,在cpg位点和基因水平检测102份样本的甲基化水平,102份样本包括51份原发肿瘤组织和51份正常组织;
[0066]
差异性甲基化位点分析:包括长度在10bp以内的cpg单核苷酸在内的所有探针全部移除,当原发肿瘤和正常组织样本之间β值大于等于0.14,p值小于0.05时,则认为该cpg位点的甲基化水平存在差异;
[0067]
差异性甲基化区域分析:使用minfi甲基化芯片分析疾病和正常组织之间的差异性甲基化区域,每个位点经过标准化处理后,建立其线性回归模型,随后通过基因群的系数来确定凸点;
[0068]
另一方面,通过线性模型来测定肿瘤组织和正常组织的甲基化水平差异的系数,随后,利用bump hunter确定在肿瘤组织和正常组织间甲基化数据有显著性差异的区域;
[0069]
聚类分析:基于分位数归一化数据来对主成分进行分析,比较两组之间的甲基化密度差异,用散点图表示;
[0070]
基因本体论和富集通路的分析:对于每个共表达簇中的蛋白编码基因,通过go分析方法论的r包分析生物过程中不同基因集的富集情况,利用超几何检验估计go富集的显著性;
[0071]
通过keggrest的r包分析每个集群的生物信息通路。
[0072]
实施例3、筛选出作为生物标志物的cpg位点,并分析其与生存期的关系
[0073]
lasso回归分析:对于高维度的数据,利用lasso进行基因的优化选择,以防止过度拟合,获得更高的诊断和预后价值,lasso通过消除多余变量来使系数值趋近于0。使用r软件3.0.3版本和“glmnet”软件包来进行lasso回归分析;
[0074]
数据分析:利用r软件对数据进行分析,显著性特征为两侧p值<0.05,通过平均值
±
标准差或中位数表示数据,在回归性分析中,p值由似然比检验计算得,利用t检验分析连续数据,卡方检验比较比例;
[0075]
利用lasso逻辑回归中的非零系数来确定候选的cpg位点;
[0076]
利用单因素cox比例风险回归模型初步筛选基因,然后进一步使用lasso方法筛选候选cpg位点中关于预后的标志物;
[0077]
利用kaplan
–
meier法分析变量和总生存期之间的关系,利用时序检验比较生存曲线。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。