1.本发明涉及细胞学领域,特别是涉及一种新型冠状病毒侵染细胞的筛选系统及其应用。
背景技术:
2.新型冠状病毒(英语:severe acute respiratory syndrome coronavirus 2,缩写:sars
‑
cov
‑
2),是一种具有包膜的正链单股rna病毒,属于冠状病毒科乙型冠状病毒属严重急性呼吸道综合征相关冠状病毒种。它的基因序列和sars病毒及mers病毒属于同一谱系但不同进化枝,是已知的第七种可感染人类的冠状病毒。病毒的宿主包括哺乳动物和禽类动物。该病毒可通过人类上呼吸道入侵人体,以多种细胞表面表达的ace2为受体达到感染;主要感染器官包括肺部、心脏、肾脏等多个主要器官。sars
‑
cov
‑
2从蛋白组成上类似sars
‑
cov和mers
‑
cov,基因组都由结构蛋白、非结构蛋白和辅助蛋白编码。据电镜观察,每个病毒颗粒平均可达到三十多个核蛋白 (rnp),其中结构蛋白包括含有受体结合域的s蛋白。最近的研究发现,人tmprss2,npr1,tmem30a等蛋白,可以作为新型冠状病毒进入细胞的辅助受体,促进新型冠状病毒s蛋白与人受体之间的结合介导的病毒侵染;此外,近来陆续有报道axl,cd147,ldlrad3和clec4g等也可以作为新型冠状病毒受体,独立于ace2,能直接与新型冠状病毒s蛋白结合,促进新型冠状病毒侵染细胞。新型冠状病毒侵染人体,除了病毒与细胞直接结合外,受新型冠状病毒感染的细胞还可以通过细胞
‑
细胞融合,形成合胞体的形式,达到在患者体内复制并扩散的目的。报道显示,目前的中和抗体,在拮抗以细胞
‑
细胞融合方式传播的能力,显著低于抑制病毒直接结合细胞。这提示,需要加强抑制新型冠状病毒通过合胞体形式扩散的药物研发。
3.目前已经陆续有报道检测新型冠状病毒介导细胞融合的方法,其中主要包括通过细胞融合后转录激活信号基因的表达来检测细胞的融合,但是由于转录激活通常需要耗费24
‑
48小时,因此筛选时间有待提高。此外,经典的受体拮抗剂筛选方法是同位素或荧光素等标记的配体结合测试法。但是研究发现,ace2等受体现有的拮抗剂或者抗体不具备抗新型冠状病毒作用。由于新型冠状病毒,尤其是变异新型冠状病毒强大的感染和传播能力,通过抗病毒侵染法大规模筛选新型冠状病毒侵染抑制剂显得不切实际。目前已经报道了多种模拟新型冠状病毒
‑
细胞融合的替代检测方法,例如:假新型冠状病毒
‑
细胞融合测试法和细胞
‑
细胞融合法。虽然这两种方法一定程度上满足了检测的需求,但是存在需求大量的新病毒粒子、过程繁琐冗长、耗时长等缺陷,不利于大规模用于药物筛选上。
4.dnae内含肽(intein)的剪接特性广泛应用于蛋白间的相互作用。将两个具有相互作用蛋白分别融合进dnae intein的两个剪接片段,再直接与报告蛋白(如可发光检测的荧光素酶、绿色荧光蛋白等)的c端和n端相连。两个蛋白相互作用导致内含肽相互靠近进而折叠显示出剪接活性,使两个分别在不同载体表达的无活性报告蛋白片段恢复完整的蛋白活性而发光,通过光检测而定量定性蛋白间相互作用。已报道用此方法来监测线粒体释放蛋白、核定位蛋白等。而目前,还没有将其用于新型冠状病毒相关蛋白检测中的报道。
技术实现要素:
5.本发明的目的是提供一种新型冠状病毒侵染细胞的筛选系统及其应用,以解决上述现有技术存在的问题,应用细菌dnae内含肽的自我剪切功能,以及新型冠状病毒包膜蛋白spik与人受体和辅助受体蛋白间相互作用可以诱导细胞融合的特点,结合pb转座系统,提供了一种可用于检测新型冠状病毒及其突变体与人新型冠状病毒受体以及辅助受体介导的细胞融合的模型,以及拮抗新型冠状病毒侵染细胞的药物筛选模型。
6.为实现上述目的,本发明提供了如下方案:
7.本发明提供一种新型冠状病毒侵染细胞的筛选系统,包括新型冠状病毒受体筛选细胞系和新型冠状病毒包膜蛋白spike筛选细胞系,其中,所述新型冠状病毒受体筛选细胞系和所述新型冠状病毒包膜蛋白spike筛选细胞系共孵育,通过检测细胞内报告蛋白活性,获得模拟新型冠状病毒侵染细胞的信息。
8.本发明还提供一种所述的新型冠状病毒侵染细胞的筛选系统的制备方法,包括以下步骤:
9.(1)将携带人新型冠状病毒受体蛋白基因、报告蛋白
‑
dnae内含肽融合基因以及抗性筛选基因的质粒转染至hek293细胞,获得新型冠状病毒受体筛选细胞系;
10.(2)将携带新型冠状病毒spike基因、报告蛋白
‑
dnae内含肽融合基因以及抗性筛选基因的质粒转染至hek293细胞,获得新型冠状病毒包膜蛋白spike筛选细胞系;
11.(3)将所述新型冠状病毒受体筛选细胞系和所述新型冠状病毒包膜蛋白spike筛选细胞系共孵育,通过检测细胞内报告蛋白活性,获得模拟新型冠状病毒侵染细胞的信息。
12.优选的是,具体包括以下步骤:
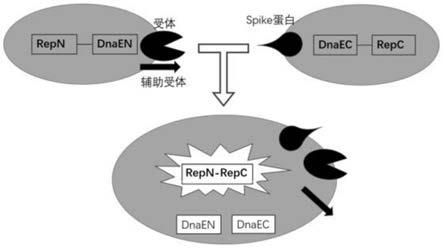
13.s1:将dnae内含肽基因的c端与报告基因的c端拼接,插入质粒1 中获得融合表达载体1;将dnae内含肽基因的n端与报告基因的n端拼接,插入质粒1中获得融合表达载体2;
14.s2:将c端携带t2a多肽的抗性筛选基因插入所述融合表达载体1 的n端,构建融合表达载体3;将c端携带t2a多肽的抗性筛选基因插入所述融合表达载体2的n端,构建融合表达载体4;
15.s3:将人新型冠状病毒的受体蛋白基因插入所述融合表达载体3,获得重组表达载体5;将人新型冠状病毒的辅助受体蛋白基因插入所述融合表达载体3,获得重组表达载体6;
16.s4:将新型冠状病毒株的spike基因或其突变体的基因插入重组表达载体4,获得重组表达载体7;
17.s5:将所述重组表达载体5和/或所述重组表达载体6,与质粒2共转染至hek293细胞,获得稳定表达的新型冠状病毒受体筛选细胞系;
18.s6:将所述重组表达载体7与质粒2共转染至hek293细胞,利用pb 转座系统,获得稳定表达的新型冠状病毒包膜蛋白spike筛选细胞系;
19.其中,所述报告基因为用于检测的有活性的酶基因或可发光的蛋白基因,所述报告基因的c端与报告基因的n端拼接即为报告基因;所述质粒 1为在哺乳动物细胞内高效表达蛋白的piggybac转座子载体,质粒2为piggybac转座酶质粒。
20.进一步地,质粒1为pb
‑
cmv
‑
mcs
‑
ef1α
‑
greenpuro cdna cloning andexpression vector质粒(#pb513b
‑
1,sbi公司产品)为基础进行改造;质粒2可自己构建,也选用商品化
的superpiggybactransposaseexpressionvector(#pb210pa
‑
1,sbi公司产品);所述报告基因为常规用于检测的有活性的酶基因或可发光的蛋白基因,包括各种发光蛋白(例如gfp)及其突变体,以及各种荧光素酶等。把报告基因一分为二,即报告基因n端和报告基因c端,这二部分均失去酶的活性或荧光,只有经内含肽把报告蛋白n端和报告蛋白c端重新拼接成完整的蛋白,才恢复原有的酶活性或荧光。新型冠状病毒受体筛选细胞系表达dnaec
‑
repc的重组载体时,新型冠状病毒包膜蛋白spike筛选细胞系选择含repn
‑
dnaen的重组载体;反之亦然。
21.优选的是,所述dnae内含肽基因的n端和c端分别来自通过密码子优化的蓝球藻(anacystisnidulans)r2pcc7942基因序列的n端和c端。
22.优选的是,所述报告基因为绿色荧光蛋白基因、海肾荧光素酶基因或萤火虫荧光素酶基因中任意一种。
23.优选的是,所述人新型冠状病毒受体包括ace2、axl、cd147、ldlrad3和clec4g中的一种或多种。
24.优选的是,所述人新型冠状病毒辅助受体包括tmprss2、npr1和tmem30a中的一种或多种。
25.优选的是,所述抗性基因为pac基因或bsr/bsd/bls基因。
26.进一步地,所述筛选系统的构建包括以下的步骤:
27.(1)将人源优化的dnae内含肽基因的n端(核苷酸序列见seqidno.1,氨基酸序列见seqidno.2)与报告基因的n段拼接,插入质粒pb
‑
cmv
‑
mcs
‑
ef1α
‑
greenpurocdnacloningandexpressionvector中的ef1α下游,并替换掉原有的greenpuro基因序列,获得融合表达载体1;将dnae内含肽基因的c端(核苷酸序列见seqidno.3,氨基酸序列见seqidno.4)与报告基因的c端拼接,插入质粒pb
‑
cmv
‑
mcs
‑
ef1α
‑
greenpurocdnacloningandexpressionvector中的ef1α下游,并替换掉原有的greenpuro基因序列,获得融合表达载体2;
28.(2)将c端携带t2a多肽的抗性筛选基因插入融合表达载体1和融合表达载体2中的n端,构建成融合表达载体3和融合表达载体4;
29.(3)将人新型冠状病毒受体ace2基因(核苷酸序列见seqidno.13,氨基酸序列见seqidno.14)或axl(核苷酸序列见seqidno.15,氨基酸序列见seqidno.16),cd147(核苷酸序列见seqidno.17,氨基酸序列见seqidno.18),ldlrad3(核苷酸序列见seqidno.19,氨基酸序列见seqidno.20)和clec4g(核苷酸序列见seqidno.21,氨基酸序列见seqidno.22)插入融合表达载体3的cmv启动子下游mcs区,获得重组表达载体5;将人新型冠状病毒辅助受体tmprssr2(核苷酸序列见seqidno.23,氨基酸序列见seqidno.24)或tmem30a(核苷酸序列见seqidno.25,氨基酸序列见seqidno.26)或npr1(核苷酸序列见seqidno.27,氨基酸序列见seqidno.28)基因插入融合表达载体3的cmv启动子下游mcs区,获得重组表达载体6;
30.(4)合成人源优化的新冠病毒spike基因(核苷酸序列见seqidno.29,氨基酸序列见seqidno.30)或其各型突变体基因插入融合表达载体4的的cmv启动子下游mcs区,获得重组表达载体7;
31.(5)将重组表达载体5和/或重组表达载体6,与piggybac转座酶质粒(按照常规方
法自己构建或用sbi公司产品#pb210pa
‑
1)以3:1的比例共转染至hek293细胞,抗性基因药物筛选7天,获得稳定表达的新冠病毒受体筛选细胞系;
32.(6)将重组表达载体7与piggybac转座酶质粒(按照常规方法自己构建或用sbi公司产品#pb210pa
‑
1)以3:1的比例共转染hek293细胞,抗性基因药物筛选7天,获得稳定表达的新型冠状病毒包膜蛋白spike筛选细胞系。
33.上述构建的筛选细胞系(构建原理如图1所示)是利用新型冠状病毒包膜蛋白spike与人新型冠状病毒受体和辅助受体蛋白间相互作用可以诱导细胞融合的原理,与所选用的细胞系来源无关,并不受报告基因类型的影响。
34.在人新型冠状病毒受体筛选细胞系中,新冠病毒受体和辅助受体蛋白在细胞膜上过量表达;抗性筛选基因,报告基因和dnae内含肽的融合片段在ef1a启动子下转录后,由t2a在蛋白水平切割,分别在细胞质内高效表达,并且报告基因片段没有任何活性。新型冠状病毒包装糖蛋白spike 基因筛选细胞系中,spike基因在转录翻译成前体蛋白s蛋白后,锚定在细胞膜上;报告蛋白和dnae的融合片段则在细胞质内高效表达,同样不具任何活性。当人新型冠状病毒受体筛选细胞系和新冠病毒包装糖蛋白 spike基因筛选细胞系混合培养时,spike蛋白与人辅助受体蛋白切割,促使s蛋白发生剪切形成s1和s2蛋白。s1作用于新型冠状病毒受体筛选细胞系的新型冠状病毒受体,诱导细胞膜接触后,s2蛋白促进细胞融合。细胞融合后,分别位于dnae内含肽的两个片段识别并发生自我剪接,使得报告蛋白发生拼接恢复完整活性。通过相应的检测工具,就可以检测到报告蛋白的活化程度,从而用来确定细胞的融合比例。
35.本发明还提供一种所述的筛选系统在高通量筛选s蛋白或新型冠状病毒受体或新型冠状病毒辅助受体拮抗剂中的应用。
36.优选的是,将新型冠状病毒受体筛选细胞系和新型冠状病毒包膜蛋白 spike筛选细胞系加入添加有待筛选药物的适用于hek293细胞的培养基 (dmem 10%fbs培养基)中,混合培养1
‑
2小时,培养产物加入与报告基因相对应的反应底物(如萤火虫报告蛋白,培养产物内加入d
‑
luciferin) 或直接检测,获得待测药物活性数据。如报告基因为可发光的蛋白基因,则只需检测荧光强度,可获得待测药物活性数据。
37.本发明公开了以下技术效果:
38.本发明构建的筛选系统是利用人源优化的dnae内含肽的自我剪接功能,并且蛋白表达水平和剪接功能均较高,可实现报告蛋白片断高效重组,形成完整功能蛋白,所以在细胞发生融合的数分钟到半小时就能检测到报告蛋白的活性。
39.本发明将人新型冠状病毒受体/新型冠状病毒spike基因,报告基因,抗性筛选基因构建至同一个转座子质粒,在转座酶的作用下,可以将其完整的整合入细胞基因组,并且通过优选的抗性基因,可以快速的筛选稳定细胞株。本发明构建稳定株的方法显著快于常规质粒通过g418抗性构建稳定株,获得大量稳定细胞株的时间从常规的2个月缩短至15天。
40.本发明可使用多种报告蛋白,即可以在常规的实验室,手工操作完成,也能利用高通量仪器平台、自动操作系统,根据需要进行实验方案的特定设计。在高通量检测系统中,可以通过标准荧光全自动定量绘图酶标仪定量检测流式细胞,多孔板或微量滴定板。这种方法能够高通量筛选新冠病毒受体和辅助受体作用的抗病毒药物,也可以高通量筛选能与新冠病毒的 spike基因编码的s蛋白作用的中和抗体药物,最后决定是否有合适的药物能
够拮抗细胞
‑
细胞融合作用。可见,本发明提供了一种快速的细胞水平高效筛选系统构建方法,利用该方法构建的系统检测抗新冠病毒药物,并且检测耗时短,操作简便,安全可靠。
附图说明
41.为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
42.图1为本发明新型冠状病毒侵染细胞的筛选系统构建的原理图;
43.图2为新冠病毒spike基因重组表达载体 pb
‑
cmv
‑
s
‑
ef1α
‑
purot2a
‑
dcrc的结构示意图;
44.图3为新冠受体ace2基因重组载体 pb
‑
cmv
‑
ace2
‑
ef1α
‑
purot2a
‑
rndn的结构示意图;
45.图4为新冠辅助受体tmprssr2重组载体pb
‑
cmv
‑ꢀ
tmprssr2
‑
ef1α
‑
purot2a
‑
rndn的结构示意图;
46.图5为新型冠状病毒受体筛选细胞系和新型冠状病毒包膜蛋白spike 筛选细胞系混合培养后的信号分析结果;
47.图6为不同浓度的新型冠状病毒中和抗体对由新型冠状病毒受体 ace2介导的细胞融合的抑制作用。
具体实施方式
48.现详细说明本发明的多种示例性实施方式,该详细说明不应认为是对本发明的限制,而应理解为是对本发明的某些方面、特性和实施方案的更详细的描述。
49.应理解本发明中所述的术语仅仅是为描述特别的实施方式,并非用于限制本发明。另外,对于本发明中的数值范围,应理解为还具体公开了该范围的上限和下限之间的每个中间值。在任何陈述值或陈述范围内的中间值以及任何其他陈述值或在所述范围内的中间值之间的每个较小的范围也包括在本发明内。这些较小范围的上限和下限可独立地包括或排除在范围内。
50.除非另有说明,否则本文使用的所有技术和科学术语具有本发明所述领域的常规技术人员通常理解的相同含义。虽然本发明仅描述了优选的方法和材料,但是在本发明的实施或测试中也可以使用与本文所述相似或等同的任何方法和材料。本说明书中提到的所有文献通过引用并入,用以公开和描述与所述文献相关的方法和/或材料。在与任何并入的文献冲突时,以本说明书的内容为准。
51.在不背离本发明的范围或精神的情况下,可对本发明说明书的具体实施方式做多种改进和变化,这对本领域技术人员而言是显而易见的。由本发明的说明书得到的其他实施方式对技术人员而言是显而易见的。本技术说明书和实施例仅是示例性的。
52.关于本文中所使用的“包含”、“包括”、“具有”、“含有”等等,均为开放性的用语,即意指包含但不限于。
53.实施例1一种新型冠状病毒侵染细胞的筛选系统的制备方法
54.(1)根据蓝球藻菌株anacystis nidulans r2(pcc7942)基因组dna 中dnae内含肽基因,通过expoptimizer软件进行密码子优化,通过 snapgene软件,拼装出anacystis nidulans r2 dnae
‑
c与renilla荧光素酶基因c端(rluc
‑
c)的融合基因(dnaec
‑
rlucc)(核苷酸序列见seq id no.5,氨基酸序列见seq id no.6),和anacystis nidulans r2 dnae
‑
n端与renilla 荧光素酶基因n端(rluc
‑
n)的融合基因(rlucn
‑
dnaen)(核苷酸序列见 seq id no.7,氨基酸序列见seq id no.8)。在此基础上,进一步在 dnaec
‑
rlucc和dnaen
‑
rlucn的n端插入携带t2a多肽的嘌呤霉素抗性基因,形成puro
‑
t2a
‑
dnaec
‑
rlucc片段(核苷酸序列见seq id no.9,氨基酸序列见seq id no.10),和puro
‑
t2a
‑
rlun
‑
dnaen片段(核苷酸序列见seqidno.11,氨基酸序列见seq id no.12)。由公司直接合成上述两片段,并通过无缝克隆技术,整合进入pb
‑
cmv
‑
mcs
‑
ef1α
‑
greenpuro表达载体的ef1α启动子后面,剔除原有质粒的greenpuro基因,分别形成 pb
‑
cmv
‑
mcs
‑
ef1α
‑
puro
‑
t2a
‑
dnaec
‑
rlucc重组质粒和 pb
‑
cmv
‑
mcs
‑
ef1α
‑
puro
‑
t2a
‑
rlun
‑
dnaen重组质粒。
55.(2)直接合成人ace2基因(核苷酸序列见seq id no.13,氨基酸序列见seq id no.14),tmprssr2基因(核苷酸序列见seq idno.23,氨基酸序列见seq id no.24),分别插入pb
‑
cmv
‑
mcs
‑
ef1α
‑
puro
‑
t2a
‑ꢀ
rlun
‑
dnaen重组质粒的mcs区,形成pb
‑
cmv
‑
ace2
‑
ef1α
‑
purot2a
‑
rndn (结构示意图如图3所示)和pb
‑
cmv
‑
tmprssr2
‑
ef1α
‑
purot2a
‑
rndn 重组质粒(结构示意图如图4所示)
56.(3)新型冠状病毒spike基因,通过expoptimizer软件进行密码子优化,并直接合成(核苷酸序列见seq idno.29,氨基酸序列见seq idno.30);插入pb
‑
cmv
‑
mcs
‑
ef1α
‑
puro
‑
t2a
‑
dnaec
‑
rlucc重组质粒的mcs 区,形成pb
‑
cmv
‑
s
‑
ef1α
‑
purot2a
‑
dcrc重组质粒(结构示意图如图2所示)。
57.(4)hek293细胞以3
×
105密度传代于6孔细胞培养板中,在含 10%胎牛血清的dmem培养基中,37℃,5%co2环境培养。第二天,一组细胞用lipofectamine转染试剂(invitrogen公司产品)共转染质粒1ug 的pb
‑
cmv
‑
ace2
‑
ef1α
‑
purot2a
‑
rlun
‑
dnaen和1ugpb
‑
cmv
‑
tmprssr2
‑
ef1α
‑
purot2a
‑
rlun
‑
dnae重组质粒以及0.25μg的 transposase expression vector(
#
pb210pa
‑
1,sbi公司产品),转染后继续培养24小时。随后加入2μg/ml的puromycin,筛选7天。
58.另一组细胞共转染1μg的 pb
‑
cmv
‑
hncovs
‑
ef1α
‑
puro
‑
t2a
‑
dnaec
‑
rlucc重组质粒以及0.25μg的 transposase expression vector,转染和筛选方法同上。
59.(5)筛选得到的稳定细胞株用胰酶消化共培养2小时,混合前培养基 dmem 10%fbs中加入待筛选的细胞融合拮抗药物。
60.(6)使用renilla
‑
lumi
tm plus海肾萤光素酶报告基因检测试剂盒(碧云天公司产品),按试剂盒要求,将与培养液等体积的检测试剂加入到细胞培养板内反应5分钟,用化学发光检测仪spectramax(molecular device 公司产品)测定荧光强度(延时2s,检测2s),结果如图3所示。由图5 中可知:只有表达稳定表达新冠受体的细胞系和表达spike蛋白的细胞系共孵育,细胞才能发生融合。不同浓度的新型冠状病毒中和抗体(义翘神州产品)对由spike蛋白介导的细胞融合的抑制作用结果见图6。从图6 中可以看出,中和抗体浓度越大,其对新型冠状病毒spike蛋白介导的细胞融合的抑制作用越强。
61.上述结果可知,spike、新型冠状病毒受体是研制抗新型冠状病毒药物的重要靶
位,本发明筛选系统可以简单、快速的用来高通量筛选spike蛋白、人新型冠状病毒受体或人新型冠状病毒辅助受体拮抗剂,为高效筛选抗新型冠状病毒中和抗体等新型药物提供了一条有效途径。
62.以上所述的实施例仅是对本发明的优选方式进行描述,并非对本发明的范围进行限定,在不脱离本发明设计精神的前提下,本领域普通技术人员对本发明的技术方案做出的各种变形和改进,均应落入本发明权利要求书确定的保护范围内。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。