1.本发明涉及微生物技术领域,具体涉及微生物益生性功能基因的检测方法及其应用。
背景技术:
2.益生菌是一类可改善宿主肠道菌群平衡的活性微生物,对宿主具有增强免疫机能、保护肠道黏膜、抑制致病菌和控制内毒素等有益功能,在临床治疗中也发挥着非常重要的作用。目前益生菌已被广泛应用于保健品、发酵食品、糖果、乳制品和牙膏等产品中。近年来,益生菌产业增长率较高,拥有巨大的市场空间。
3.随着益生菌的需求快速增加,筛选出具有益生性功能的菌株,并对其进行功能性分析,最终生产出一系列含益生菌的食品和保健品,成为近几年研究者关注的热点。因此,建立一种快速、准确的益生菌筛选模型进而确定其益生机理很有必要。传统的益生菌筛选方法是基于多种益生菌之间生理、生化指标的不同,利用益生菌的产酸、产气以及生长代谢方面等特性进行选择性培养,进而判断的。由于这些传统方法主要依赖于表型表达和生化技术,故存在着操作繁琐、检测周期长、辨识能力差等缺点。近年来,微生物全基因组测序技术的迅猛发展为在基因组水平上评价益生菌和潜在益生菌菌株的筛选提供了新技术。基因组的分析可以将目的菌株的基因与益生性功能联系起来,从而在分子水平上筛选出具有特定功效的益生菌菌株。然而,目前尚缺少有效的基因组水平的益生性功能基因和益生菌的检测方法。
技术实现要素:
4.本发明的目的是提供一种微生物益生性功能基因的检测方法,该方法能够在基因组水平分析、检测微生物所含有的益生性功能基因。本发明的另一目的是提供该方法的应用。
5.具体地,本发明提供以下技术方案:
6.本发明提供一种微生物益生性功能基因的检测方法,该方法包括如下步骤:构建益生性功能基因数据库,将待测微生物的所有蛋白的序列在所述数据库中进行blast比对,根据阈值确定所述待测微生物含有的益生性功能基因,其中,所述阈值包括blast比对的蛋白序列相似度、比对区域覆盖度。
7.优选地,以上所述的益生性功能基因数据库包括基础氨基酸和维生素合成代谢基因数据库、抗菌活性基因数据库、抗氧化基因数据库、胞外多糖合成基因数据库和降胆固醇基因数据库。
8.本发明所述的检测方法还包括获得待测微生物的全基因组序列的步骤。在获得全基因组序列后,将所有蛋白序列在所述数据库中进行blast比对。
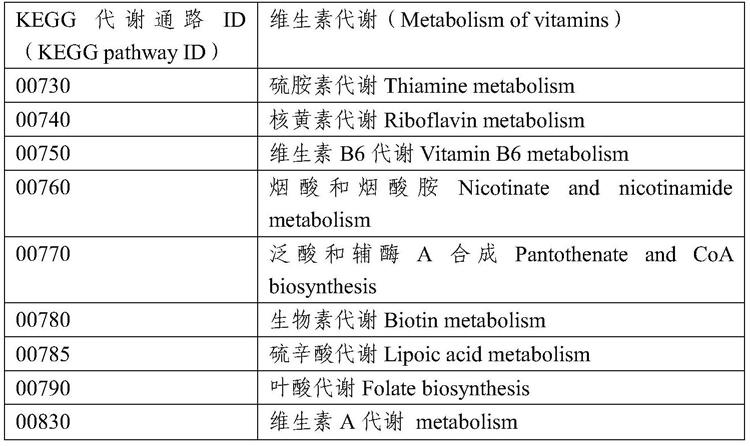
9.具体地,所述基础氨基酸和维生素合成代谢基因数据库包括kegg代谢通路(pathway)id为00730、00740、00750、00760、00770、00780、00785、00790、00830、00250、
00260、00270、00290、00300、00220、00330、00340、00350、00360、00380、00400的蛋白序列及其注释信息。
10.上述kegg代谢通路的信息如表1和表2所示。
11.表1维生素代谢相关通路
[0012][0013]
表2氨基酸代谢相关通路
[0014][0015][0016]
优选地,使用kobas
‑
3.0软件,对所述待测微生物的所有蛋白进行所述基础氨基酸和维生素合成代谢基因数据库中的kegg代谢通路注释,获得候选氨基酸和维生素代谢相关基因。
[0017]
进一步优选地,使用kobas
‑
3.0软件的annotate.py程序进行kegg代谢通路注释,所用到的参数如下:
‑
i蛋白序列
‑
s物种简写
‑‑
coverage=0.6
‑‑
evalue=1e
‑
10。
[0018]
具体地,所述抗菌活性基因数据库为从bactibase下载获得的细菌素数据库文件。
[0019]
作为本发明的优选方案,所述细菌素数据库文件包括229个细菌素的蛋白序列及其注释信息。
[0020]
对于抗菌活性基因检测,在blast比对结果中,筛选出蛋白序列相似度≥30%且比对区域覆盖度≥80%的结果,获得候选抗菌活性基因。
[0021]
具体地,所述抗氧化基因数据库包含uniprot蛋白数据库中注释信息中含有关键词superoxide dismutase(sod),glutathione peroxidase(gpx),catalase(cat),heme oxygenase1(ho1),glutathione reductase(gr),methionine synthase(meth)的蛋白序列及其注释信息。
[0022]
作为本发明的优选方案,所述抗氧化基因数据库包括过氧化氢酶基因47个,谷胱甘肽过氧化物酶基因7个,谷胱甘肽还原酶基因2个,血红素氧化酶基因11个、甲硫氨酸合成酶基因11个、超氧化物歧化酶基因122个。
[0023]
对于抗氧化基因的检测,在blast比对结果中,筛选出蛋白序列相似度≥60%且比对区域覆盖度≥80%的结果,获得候选抗氧化基因。
[0024]
具体地,所述胞外多糖合成基因数据库包括ncbi网站中以exopolysaccharide biosynthesis protein和tyrosine
‑
protein kinase为关键词收集的蛋白序列及其注释信息。
[0025]
优选包括27个胞外多糖合成基因及其编码蛋白序列和注释信息。
[0026]
对于胞外多糖合成基因的检测,在blast比对结果中,筛选出蛋白序列相似度≥30%且比对区域覆盖度≥80%的结果,获得候选胞外多糖合成基因。
[0027]
具体地,所述降胆固醇基因数据库包括从蛋白数据库uniprot中筛选出注释信息包含关键词“bile salt hydrolase”的蛋白序列及其注释信息。
[0028]
作为本发明的优选方案,降胆固醇基因数据库包括68个胆盐水解酶基因及其编码蛋白序列和注释信息。
[0029]
对于降胆固醇基因的检测,在blast比对结果中,筛选出蛋白序列相似度≥60%,且比对区域覆盖度≥80%的结果,获得候选降胆固醇基因。
[0030]
本发明的检测方法中,所述blast使用blast
‑
2.4.0 软件进行,参数设置为:
‑
evalue1e
‑
10
‑
num_threads 8
‑
max_target_seqs 3
‑
outfmt 6。
[0031]
本发明还提供所述微生物益生性功能基因的检测方法在益生菌筛选或益生菌功能评价中的应用。
[0032]
优选地,本发明所述的微生物为嗜酸乳杆菌(lactobacitlus acidophilus)。
[0033]
本发明的有益效果在于:本发明提供的微生物益生性功能基因的检测方法以待测微生物的所有基因和蛋白序列为基础,通过将蛋白序列与构建的益生性功能基因数据库进行blast比对,检测出与益生性功能相关的基因信息。该方法基于微生物全基因组测序数据,可从基因和蛋白序列水平上,快速检测出菌株中含有的基础氨基酸和维生素合成代谢基因、抗菌活性基因、抗氧化基因、胞外多糖合成基因和降胆固醇基因的数目和具体信息,获得菌株的益生基因信息,具有检测方便快捷、周期短、准确性较高的特点,为益生菌的评
价和潜在益生菌菌株的筛选提供高效的技术手段。
具体实施方式
[0034]
以下实施例用于说明本发明,但不用来限制本发明的范围。
[0035]
实施例1微生物益生性功能基因的检测方法
[0036]
本实施例提供一种微生物益生性功能基因的检测方法,该方法为以待测菌株的全基因组序列中的所有蛋白序列为对象,进行基础氨基酸和维生素代谢基因、抗菌活性基因、抗氧化基因、胞外多糖合成基因和降胆固醇基因的检测,具体包括如下步骤:
[0037]
1、基础氨基酸和维生素代谢基因的检测
[0038]
(1)使用kobas
‑
3.0软件的annotate.py程序,对待测微生物的所有基因进行kegg代谢通路注释,kegg代谢通路(pathway)id为00730、00740、00750、00760、00770、00780、00785、00790、00830、00250、00260、00270、00290、00300、00220、00330、00340、00350、00360、00380、00400。上述kegg代谢通路的信息如表1和表2所示,所用到的参数如下:
‑
i蛋白序列
‑
s物种简写
‑‑
coverage=0.6
‑‑
evalue=1e
‑
10;
[0039]
(2)根据步骤(1)得到的基因代谢通路信息和gff文件,筛选出与氨基酸和维生素代谢有关的基因,最终获得一个包含kegg通路id号、通路名称、基因数目、相关基因、相关基因在基因组上的位置等信息的列表。
[0040]
2、抗菌活性基因的检测
[0041]
(1)从bactibase网站上(http://bactibase.hammamilab.org/downloads.php)下载细菌素数据库文件,共含细菌素蛋白序列及其注释信息229条,作为抗菌活性基因检测数据库;
[0042]
(2)使用blast
‑
2.4.0 软件,将待测微生物的所有蛋白序列与步骤(1)构建的抗菌活性基因检测数据库进行比对,参数如下:
‑
evalue 1e
‑
10
‑
num_threads 8
‑
max_target_seqs 3
‑
outfmt 6;
[0043]
(3)筛选出蛋白序列相似度≥30%,且比对区域覆盖度≥80%的blast结果,最终获得一个包含检测到的抗菌活性基因在基因组上的位置信息、基因id、基因名称、属于分类、uniprot id等信息的列表。
[0044]
3、抗氧化基因的检测
[0045]
(1)参考乳杆菌属抗氧化相关基因(superoxide dismutase(sod),glutathione peroxidase(gpx),catalase(cat),heme oxygenase1(ho1),glutathione reductase(gr),methionine synthase(meth)),从蛋白数据库uniprot(https://www.uniprot.org/)中筛选出注释信息里含有“sod”、“gpx”关键词的蛋白。最终搜集到过氧化氢酶基因47个,谷胱甘肽过氧化物酶基因7个,谷胱甘肽还原酶基因2个,血红素氧化酶基因11个、甲硫氨酸合成酶基因11个、超氧化物歧化酶基因122个。将搜集到的这些蛋白序列及其注释信息作为抗氧化基因检测数据库。
[0046]
(2)使用blast
‑
2.4.0 软件,将待测微生物的所有蛋白序列与步骤(1)构建的抗氧化基因数据库做比对,参数如下:
‑
evalue 1e
‑
10
‑
num_threads 8
‑
max_target_seqs 3
‑
outfmt 6;
[0047]
(3)筛选出序列相似度≥60%,且比对区域覆盖度≥80%的blast结果,最终获得
一个包含检测到的抗氧化基因在基因组上的位置信息、基因id、相似度、e
‑
value值、score值、蛋白名称、基因名称、物种名称等信息的列表。
[0048]
4、胞外多糖合成基因的检测
[0049]
(1)以“exopolysaccharide biosynthesis protein”和“tyrosine
‑
protein kinase”为关键词,从ncbi网站上下载胞外多糖合成有关的蛋白;最终获得27个胞外多糖合成基因。将这些蛋白序列及其注释信息作为胞外多糖基因检测数据库。
[0050]
(2)使用blast
‑
2.4.0 软件,将待测微生物的所有蛋白序列与步骤(1)构建的胞外多糖基因数据库进行比对,参数如下:
‑
evalue 1e
‑
10
‑
num_threads 8
‑
max_target_seqs 3
‑
outfmt 6;
[0051]
(3)筛选出序列相似度≥30%,且比对区域覆盖度≥80%的blast结果,最终获得一个包含检测到的胞外多糖合成基因在基因组上的位置信息、基因id、相似度、e
‑
value值、score值、蛋白功能等信息的列表。
[0052]
5、降胆固醇基因的检测
[0053]
(1)从蛋白数据库uniprot(https://www.uniprot.org/)中筛选出注释信息里含有“bile salt hydrolase”关键词的蛋白。最终搜集到胆盐水解酶68个,并将这些蛋白及其注释信息作为降胆固醇基因检测数据库。
[0054]
(2)使用blast
‑
2.4.0 软件,将待测微生物的所有蛋白序列与步骤(1)构建的降胆固醇基因数据库进行比对,参数如下:
‑
evalue 1e
‑
10
‑
num_threads 8
‑
max_target_seqs 3
‑
outfmt 6。
[0055]
(3)筛选出序列相似度≥60%,且比对区域覆盖度≥80%的blast结果,最终获得一个包含检测到的抗氧化基因在基因组上的位置信息、基因id、相似度、e
‑
value值、score值、蛋白名称、基因名称、物种名称等信息的列表。
[0056]
实施例2益生性功能基因检测方法的应用
[0057]
1、嗜酸乳杆菌lactobacitlus acidophilus ncfm(atcc700396)的降胆固醇基因的检测
[0058]
下载lactobacitlus acidophilus ncfm(nc_006814.3)菌株的蛋白序列集,利用实施例1提供的微生物益生性功能基因的检测方法进行检测,检测结果如表3所示。
[0059]
表3利用实施例1的方法检测得到的lactobacitlus acidophilus ncfm的降胆固醇基因
[0060][0061]
根据文献报道(maire begley,colin h,cormac g m g.bile salt hydrolase activity in probiotics[j].applied and environmental microbiology,2006,1729
‑
1738.),lactobacitlus acidophilus ncfm共含有2个降胆固醇基因bsh基因(表4),其中,lba0892即为bsha。
[0062]
表4文献报道的lactobacitlus acidophilus ncfm的降胆固醇基因
[0063]
降胆固醇基因登录号lba1078(bshb)aac42923yp_193954lba0892aac42751yp_193782
[0064]
结果表明,利用本发明实施例1的方法得到的lactobacitlus acidophilus ncfm的降胆固醇基因的种类和数量与文献报道完全一致,证明本发明的方法具有较高的准确性。
[0065]
2、嗜酸乳杆菌lactiplantibacillus paraplantarum l
‑
zs9的抗菌活性基因的检测
[0066]
下载lactobacillus paraplantarum strain l
‑
zs9(nz_cp013130.1)的蛋白序列集。利用实施例1提供的菌益生性功能基因的检测方法进行检测,检测结果如表5所示。
[0067]
表5利用实施例1的方法检测得到的lactobacitlus acidophilus ncfm的降胆固醇基因
[0068]
query_idsubject_id%identitye
‑
valuescorenameclassuniprotidwp_003641979.1bac0221003.07e
‑
1348.5plantaricin
‑
aunclassifiedp80214wp_003643811.1bac0501002.92e
‑
2271.6plantaricin fclass iibp71469;q7akv8wp_033611266.1bac04696.972.22e
‑
1964.7plantaricin eunclassifiedp71470;q7akv7wp_033611278.1bac0271008.15e
‑
1552.8plantaricin jclass iibp71461;q7akw6wp_033611279.1bac0441001.57e
‑
1965.1plantaricin kunclassifiedp71460
[0069]
根据文献报道(张旭,赵斌,张香美,等.产细菌素乳酸菌的筛选及细菌素相关基因的分析[j].中国农业大学学报,2013,18(4).),lactiplantibacillus paraplantarum l
‑
zs9具有4个细菌素编码基因:plnef、plnjk、plna、plnn。
[0070]
结果表明,文献中报道的4个抗菌活性基因,除plnn外,利用本发明实施例1的方法均检测到。但在数据库中也未检索到plnn的序列。
[0071]
3、新分离菌株的益生性功能基因检测
[0072]
对分离得到的一株嗜酸乳杆菌(lactobacillus acidophilus)进行了基因组测序,获得各基因的蛋白序列。利用实施例1提供的微生物益生性功能基因的检测方法进行益生性功能基因的检测。基础氨基酸和维生素代谢基因的检测结果分别如表6、表7所述,抗菌活性基因、胞外多糖合成基因和降胆固醇基因的检测结果分别如表8、表9、表10所示。该菌株中未检测到抗氧化基因。
[0073]
表6利用实施例1的方法检测得到的氨基酸代谢基因
[0074][0075]
表7利用实施例1的方法检测得到的维生素代谢基因
[0076][0077]
表8利用实施例1的方法检测得到的抗菌活性基因
[0078]
query_idsubject_id%identitye
‑
valuescorenameclassuniprotidassembly_01158bac19053.1254.33e
‑
1143.5enterocin xbetaclass iibd7up04assembly_01165bac0241006.33e
‑
1451.6acidocin j1132βclass iibq9r499assembly_01392bac13437.8152.25e
‑
39131helveticin
‑
junclassifiedp22294
[0079]
表9利用实施例1的方法检测得到的胞外多糖合成基因
[0080][0081]
表10利用实施例1的方法检测得到的降胆固醇基因
[0082][0083]
虽然,上文中已经用一般性说明及具体实施方案对本发明作了详尽的描述,但在本发明基础上,可以对之作一些修改或改进,这对本领域技术人员而言是显而易见的。因此,在不偏离本发明精神的基础上所做的这些修改或改进,均属于本发明要求保护的范围。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。

