1.本发明属于生物技术领域,具体涉及一种控制水稻叶片结构的方法及应用。
背景技术:
3.水稻叶脉属于典型单子叶植物类平行脉,由叶片中央一条中脉(主脉)和中脉两侧数条大脉以及大脉之间多条小脉沿叶片纵向(轴)平行构成,这些平行脉之间通过连接脉在横向(轴)上连接,大脉也称作大维管束,小脉也称作小维管束。水稻叶脉由维管束鞘细胞包围,叶脉-维管束鞘细胞之间是叶肉细胞,叶肉细胞执行主要的光合作用(图1)。
4.光合作用为水稻的生长发育提供了物质来源和能量来源,也是的水稻结实后籽粒中积累淀粉和其它营养物质的代谢基础,提高光合作用的效率是水稻增产的关键基础。水稻是典型的c3植物,其光合效率受到暗反应(开尔文循环)过程中关键酶核酮糖-1,5-二磷酸羧化酶/加氧酶(rubisco)催化效率的严重制约。rubisco在叶肉细胞的叶绿体基质中催化co2与1,5-二磷酸核酮糖(rubp)生成2分子3-磷酸甘油酸(3-pga),进而再经过开尔文循环,利用光反应产出的atp中的化学能和nadph的还原力转化产生葡萄糖中。葡萄糖经过进一步的转换和运输,最后在水稻籽粒中形成淀粉,同时光合作用生产的c骨架也是水稻其它重要的有机物形成的基础。水稻的rubisco与其它c3植物的rubisco存在相同的制约光合效率的三个因素:首先rubisco是一种催化效率极低的酶,绝大多数种类的酶分子1秒钟可催化1000个以上的底物分子,但rubisco每秒钟仅固定大约3分子co2,由于其极低酶促效率,为满足光合作用的需要,c3植物叶片中的总蛋白中大约一半的蛋白是rubisco,这极大增加了植物对氮素的需求;其次,rubisco同时有固定co2的羧化酶和分解光合作用初始产物pga的加氧酶的活性,大田中午时刻由于高温或水分亏缺会导致c3植物气孔关闭,而气孔关闭使叶片内co2浓度大大降低,此时rubisco主要行使加氧酶的活性而消耗掉大量光合作用的有效产物pga,从而大大降低了叶片总的光合效率。第三,pga被rubisco氧化后生成的磷酸甘油醛有生物学毒性,需要通过光呼吸途径被代谢掉,而光呼吸会消耗大量的光反应产生的能量(atp)和还原力(nadph),理论计算和试验测定c3植物的光呼吸最终会导致损失至少30%光合产物。
5.自然界中演化出了高光效的c4植物,其固定co2由磷酸烯醇式丙酮酸羧化酶(pepc)在叶肉细胞中完成,pepc具有高催化效率,大约每秒催化3000-5000个分子co2发生羧化反应。pepc只有co2羧化酶活性而没有加氧酶的活性且催化效率远高于rubisco,使c4植物叶肉细胞中不在积累rubisco,这大大降低了c4植物进行羧化反应需要的羧化酶的需要,从而大大降低c4植物对n素的需求。c4植物co2固定(羧化反应)和还原(开尔文循环)在两种类型的细胞中完成:羧化反应在叶肉细胞中进行,产生的4碳化合物被主动转运到邻近的包裹叶脉(维管束)的维管束鞘细胞中,在维管束鞘细胞中4碳化合物脱羧释放co2,随后在维管束鞘细胞中完成开尔文循环,这样co2的就会在叶鞘细胞中得到浓缩,使得rubisco周围的co2浓度提高约数百倍而抑制了其加氧酶的活性,这样也就消除了光呼吸,从而使c4植物具有更高的光合效率,c4作物理论上比c3作物至少可以提高30%的产量。c4植物由于
仅需要少量的pepc即可满足co2羧化作用从而获得比c3植物更高的氮素利用率。c4植物中co2的浓缩机制使其在气孔关闭下仍然能维持一定的光合作用,这样c4植物也有更高水分利用效率。
6.将c4途径引入c3作物水稻是一条潜力极大提高光合效率的技术途径,同时还带来了更高水分利用率和氮素利用率。c4光合作用需要在两类细胞中(图1)完成,其中叶肉细胞仅具备固定co2的功能(由pepc完成),维管束鞘细胞则进行co2同化的开尔文循环,最终在维管束鞘细胞中形成淀粉,淀粉再经过复杂的转化和运输最终在籽粒中积累形成淀粉和其它营养物质。c4植物进化出c4光合途径的同时进化出了相适应高叶脉密度的特殊解剖学结构。禾本科c3光合类作物叶片中相邻的叶脉(维管束)之间,维管束鞘细胞与叶肉细胞的比例约为4:1(图1中上图),而c4光合类作物叶片中相邻的叶脉(维管束)之间,维管束鞘细胞与叶肉细胞的比例为1:1,这也是几乎所有的c4作物包括玉米,谷子和高粱等作物都备的特异性解剖学结构(图1中下图)。
7.目前科学家已经成功地把c4光合途径相关的各种酶导入到水稻中,但是没有能建立起有效的c4同化过程,造成这一结果的原因是没有在水稻叶片中同步构建c4类似的解剖学结构。这进一步的提示了c4水稻的成功不仅仅需要c4的代谢相关酶类,更需要与之配合的c4叶片解剖学结构。目前仅有很少报道控制叶片中叶脉密度基因,且水稻中还没有相关基因控制叶片形成类c4解剖学结构的基因的报道。
技术实现要素:
8.本发明所要解决的技术问题是如何在水稻叶片中同步构建与c4植物类似的解剖学结构。
9.为了解决现有技术存在的问题,本发明提供了一种调控植物叶片结构的方法。
10.本发明提供的调控植物叶片结构的方法,包括调控目的植物中蛋白质的活性和/或含量,或/和调控蛋白质的编码基因的表达量,来调控植物叶片结构,所述蛋白质是如下任一种的蛋白质:
11.a1)氨基酸序列是seq id no.3的蛋白质和氨基酸序列是seq id no.4的蛋白质组成的组合物;
12.a2)氨基酸序列是seq id no.3的蛋白质或氨基酸序列是seq id no.4的蛋白质;
13.a3)将a1)和a2)的蛋白质经过氨基酸残基的取代和/或缺失和/或添加得到的与a1)和a2)所示的蛋白质具有80%以上的同一性且具有调控植物叶片结构的蛋白质;
14.a4)在a1)或a2)的n末端或/和c末端连接蛋白质标签得到的融合蛋白质。
15.本发明还提供了培育叶片结构改变的植物的育种方法,包括调控目的植物中权利要求1中所述蛋白质的活性和/或含量,或/和调控前文所述蛋白质的编码基因的表达量,得到植物叶片结构改变的植物。
16.上述方法中,所述调控目的植物中前文所述蛋白质的活性和/或含量,或/和所述蛋白质的编码基因的表达量,包括向受体植物中导入包含抑制或降低或沉默前文所述蛋白质的编码基因表达的核酸分子的重组表达载体,得到植物叶片结构改变的目的植物;所述编码基因编码前文所述蛋白质。
17.上述方法中,所述蛋白质是如下任一种的蛋白质:
18.a1)氨基酸序列是seq id no.3的蛋白质和氨基酸序列是seq id no.4的蛋白质组成的组合物;
19.a2)氨基酸序列是seq id no.3的蛋白质或氨基酸序列是seq id no.4的蛋白质;
20.a3)将a1)和a2)的蛋白质经过氨基酸残基的取代和/或缺失和/或添加得到的与a1)和a2)所示的蛋白质具有80%以上的同一性且具有调控植物叶片结构的蛋白质;
21.a4)在a1)或a2)的n末端或/和c末端连接蛋白质标签得到的融合蛋白质。
22.上述方法中,所述蛋白质来源于水稻(oryza sativa)。
23.上述方法中,所述蛋白质的名称为osshr1。蛋白质osshr1的氨基酸序列具体可为seq id no.3,含有602个氨基酸。
24.上述方法中,所述蛋白质的名称为osshr2。蛋白质osshr2的氨基酸序列具体可为seq id no.4,含有603个氨基酸。
25.上述蛋白质可人工合成,也可先合成其编码基因,再进行生物表达得到。
26.上述蛋白质中,所述标签是指利用dna体外重组技术,与目的蛋白一起融合表达的一种多肽或者蛋白,以便于目的蛋白的表达、检测、示踪和/或纯化。所述标签可为flag标签、his标签、mbp标签、ha标签、myc标签、gst标签和/或sumo标签等。
27.本发明中,所述调控可为上调或增强或提高。所述调控也可为下调或减弱或降低。
28.上文中,所述调控蛋白质的编码基因表达的物质可为进行如下6种调控中至少一种调控的物质:1)在所述基因转录水平上进行的调控;2)在所述基因转录后进行的调控(也就是对所述基因的初级转录物的剪接或加工进行的调控);3)对所述基因的rna转运进行的调控(也就是对所述基因的mrna由细胞核向细胞质转运进行的调控);4)对所述基因的翻译进行的调控;5)对所述基因的mrna降解进行的调控;6)对所述基因的翻译后的调控(也就是对所述基因翻译的蛋白质的活性进行调控)。
29.本文中,所述调控所述蛋白质的编码基因的表达可为抑制或降低或下调所述编码基因表达。抑制或降低或下调所述编码基因表达可通过基因敲除或基因沉默实现。
30.所述基因敲除(gene knock-out)是指通过基因编辑技术使特定靶基因失活的现象。基因敲除通过dna序列的改变使特定靶基因失活,包括且不限于基于锌指核酸酶(zinc-finger nucleases,zfn),转录激活因子样效应因子核酸酶(transcription activator-like effector nucleases,talen)和crispr/cas系统,crispr(clustered regulatory interspaced short palindromic repeat)即成簇的规律间隔的短回文重复序列,是基因组中一个含有多个短重复序列的位点,cas9蛋白在rna的介导下能够对crrna
–
tracrrna识别的靶序列进行切割。
31.所述基因沉默是指在不损伤原有dna的情况下使基因不表达或低表达的现象。基因沉默以不改变dna序列为前提,使基因不表达或低表达。基因沉默可发生在两种水平上,一种是由于dna甲基化、异染色质化以及位置效应等引起的转录水平的基因沉默,另一种是转录后基因沉默,即在基因转录后的水平上通过对靶标rna进行特异性抑制而使基因失活,包括反义rna、共抑制(co-suppression)、基因压抑(quelling)、rna干扰(rnai)和微小rna(mirna)介导的翻译抑制等。
32.上述方法中,所述抑制或降低或沉默目的植物中所述蛋白质osshr1和osshr2的编码基因的表达量和/或活性可为利用基因突变、基因敲除、基因编辑或基因敲减技术使目的
植物基因组中所述蛋白质osshr1和osshr2的编码基因活性下降或失活。
33.本发明还提供了上述蛋白质或调控基因的表达物质或调控所述蛋白质活性或含量的物质在下述任一种中的应用:
34.u1)蛋白质或调控基因的表达物质或调控所述蛋白质活性或含量的物质在调控植物叶片结构中的应用;
35.u2)蛋白质或调控基因的表达物质或调控所述蛋白质活性或含量的物质在制备调控植物叶片结构的产品中的应用;
36.u3)蛋白或调控基因的表达物质或调控所述蛋白质活性或含量的物质在培育植物叶片结构改变的植物中的应用;
37.u4)蛋白质或调控基因的表达物质或调控所述蛋白质活性或含量的物质在制备培育植物叶片结构改变的植物的产品中的应用;
38.u5)蛋白质或调控基因的表达物质或调控所述蛋白质活性或含量的物质在植物育种中的应用;
39.所述蛋白质是如下任一种的蛋白质:
40.a1)氨基酸序列是seq id no.3的蛋白质和氨基酸序列是seq id no.4的蛋白质组成的组合物;
41.a2)氨基酸序列是seq id no.3的蛋白质或氨基酸序列是seq id no.4的蛋白质;
42.a3)将a1)和a2)的蛋白质经过氨基酸残基的取代和/或缺失和/或添加得到的与a1)和a2)所示的蛋白质具有80%以上的同一性且具有调控植物叶片结构的蛋白质;
43.a4)在a1)或a2)的n末端或/和c末端连接蛋白质标签得到的融合蛋白质。
44.上述应用中,所述调控基因的表达物质或调控所述蛋白质活性或含量的物质为与所述蛋白质相关的生物材料,所述生物材料为下述中的任一种:
45.b1)编码前文所述蛋白质的核酸分子;
46.b2)含有b1)所述核酸分子的表达盒;
47.b3)含有b1)所述核酸分子的重组载体、或含有b2)所述表达盒的重组载体;
48.b4)含有b1)所述核酸分子的重组微生物、或含有b2)所述表达盒的重组微生物、或含有b3)所述重组载体的重组微生物;
49.b5)含有b1)所述核酸分子的转基因植物细胞系、或含有b2)所述表达盒的转基因植物细胞系;
50.b6)含有b1)所述核酸分子的转基因植物组织、或含有b2)所述表达盒的转基因植物组织;
51.b7)含有b1)所述核酸分子的转基因植物器官、或含有b2)所述表达盒的转基因植物器官;
52.c1)抑制或降低或沉默前文所述蛋白质的编码基因表达的核酸分子;
53.c2)表达c1)所述核酸分子的编码基因;
54.c3)含有c2)所述编码基因的表达盒;
55.c4)含有c2)所述编码基因的重组载体、或含有c3)所述表达盒的重组载体;
56.c5)含有c2)所述编码基因的重组微生物、或含有c3)所述表达盒的重组微生物、或含有c4)所述重组载体的重组微生物;
57.c6)含有c2)所述编码基因的转基因植物细胞系、或含有c3)所述表达盒的转基因植物细胞系、或含有c4)所述重组载体的转基因植物细胞系;
58.c7)含有c2)所述编码基因的转基因植物组织、或含有c3)所述表达盒的转基因植物组织、或含有c4)所述重组载体的转基因植物组织;
59.c8)含有c2)所述编码基因的转基因植物器官、或含有c3)所述表达盒的转基因植物器官、或含有c4)所述重组载体的转基因植物器官。
60.上述应用中,其特征在于:b1)所述核酸分子为如下b1)或b2)所示的基因:
61.b1)编码链的编码序列是seq id no.1的cdna分子或dna分子;
62.b2)编码链的编码序列是seq id no.2的cdna分子或dna分子。
63.本文所述核酸分子可以是dna,如cdna、基因组dna或重组dna;所述核酸分子也可以是rna,如grna、mrna、sirna、shrna、sgrna、mirna或反义rna。
64.本领域普通技术人员可以很容易地采用已知的方法,例如定向进化或点突变的方法,对本发明的编码蛋白质osshr1和osshr2的核苷酸序列进行突变。那些经过人工修饰的,具有与本发明分离得到的蛋白质osshr1和osshr2的核苷酸序列75%或75%以上同一性的核苷酸,只要编码蛋白质osshr1和osshr2且具有蛋白质osshr1和osshr2功能,均是衍生于本发明的核苷酸序列并且等同于本发明的序列。
65.上述75%或75%以上同一性,可为80%、85%、90%或95%以上的同一性。
66.本文中,同一性是指氨基酸序列或核苷酸序列的同一性。可使用国际互联网上的同源性检索站点测定氨基酸序列的同一性,如ncbi主页网站的blast网页。例如,可在高级blast2.1中,通过使用blastp作为程序,将expect值设置为10,将所有filter设置为off,使用blosum62作为matrix,将gap existence cost,per residue gap cost和lambda ratio分别设置为11,1和0.85(缺省值)并进行检索以对氨基酸序列的同一性进行计算,然后即可获得同一性的值(%)。
67.本文中,所述80%以上的同一性可为至少80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的同一性。
68.本文所述载体是本领域技术人员公知的,包括但不限于:质粒、噬菌体(如λ噬菌体或m13丝状噬菌体等)、黏粒(即柯斯质粒)、ti质粒或病毒载体。
69.可用现有的植物表达载体构建含有osshr1和osshr2基因的重组表达载体。所述植物表达载体包括但不限于如双元农杆菌载体和可用于植物微弹轰击的载体等。所述植物表达载体还可包含外源基因的3'端非翻译区域,即包含聚腺苷酸信号和任何其它参与mrna加工或基因表达的dna片段。所述聚腺苷酸信号可引导聚腺苷酸加入到mrna前体的3'端,如包括但不限于农杆菌冠瘿瘤诱导(ti)质粒基因(如胭脂合成酶nos基因)、植物基因(如大豆贮藏蛋白基因)3'端转录的非翻译区均具有类似功能。
70.为了便于对转基因植物细胞或植物进行鉴定及筛选,可对所用植物表达载体进行加工,如加入包括但不限于可在植物中表达的编码可产生颜色变化的酶或发光化合物的基因(gus基因、荧光素酶基因等)、具有抗性的抗生素标记物(庆大霉素标记物、卡那霉素标记物等)或是抗化学试剂标记基因(如抗除草剂基因)等。从转基因植物的安全性考虑,可不加任何选择性标记基因,直接以逆境筛选转化植株。
71.上述e4)中,所述重组载体可为用植物基因编辑载体。所述植物基因编辑载体可为pylcrispr/cas9pubi-h载体。
72.作为一个具体实施例,所述重组载体为重组载体pcrispr-osshr1/2。所述重组载体pcrispr-osshr1/2是在pylcrispr/cas9pubi-h载体插入靶向于水稻基因shr1和shr2的sgrna-1(序列为5
’‑
cttcatccatgtaaagctgg-3’)、sgrna-2(序列为:5
’‑
acatgagctgctgcacgcgc-3’)和sgrna-3(序列为5
’‑
agcgcgtgcagcagctcatg-3’)的基因敲除载体。
73.本文所述微生物可为酵母、细菌、藻或真菌。其中,细菌可来自埃希氏菌属(escherichia),欧文氏菌(erwinia),根癌农杆菌属(agrobacterium)、黄杆菌属(flavobacterium),产碱菌属(alcaligenes),假单胞菌属(pseudomonas),芽孢杆菌属(bacillus)等。具体可为根癌农杆菌eha105。
74.所述重组微生物具体可为重组农杆菌eha105/pcrispr-osshr1/2。
75.上述方法或应用中,所述调控叶片结构可为使植物叶脉数量/密度增加和/或叶肉细胞数目/密度减少。
76.所述植物叶片结构改变可体现在如下中任一种:
77.1)所述转基因植物的叶片大脉数量增加;
78.2)所述转基因植物的叶片小脉数量增加;
79.3)所述转基因植物的大脉之间的小脉数量增加;
80.4)所述转基因植物的大脉和小脉之间的叶肉数量减少;
81.5)所述转基因植物的小脉之间的叶肉数量减少;
82.6)所述转基因植物的叶片大脉数量比所述受体植物高;
83.7)所述转基因植物的叶片小脉数量比所述受体植物高;
84.8)所述转基因植物的大脉之间的小脉数比所述受体植物高。
85.9)所述转基因植物的大脉和小脉之间的叶肉数量比所述受体植物少。
86.10)所述转基因植物的小脉之间的叶肉数量比所述受体植物少。
87.所述大脉在单子叶植物可为次于中(主)脉的次级脉。
88.所述小脉可为次于次级脉的细脉。
89.所述大脉和小脉与中(主)脉一起构成单子叶植物比如水稻的平行脉系统。
90.上述方法或应用中,所述植物是如下中的任一种:e1)双子叶植物或单子叶植物;
91.e2)禾本目植物;
92.e3)禾本科植物;
93.e4)稻属植物;
94.e5)水稻。
95.本文中所述的生物材料也属于本发明要求保护的范围。
96.本发明的osshr1和osshr2蛋白属于gras家族转录因子,二者的序列高度相似,蛋白的编码基因属于同源基因,具有控制水稻中改变叶片解剖学结构和产生类c4叶片解剖学结构的功能。通过crispr-cas9的方法下调或减弱或降低或敲除osshr1和/或osshr2基因表达并验证了其基因功能,结果发现下调或减弱或降低或敲除osshr1和/或osshr2基因表达可以增加大脉之间小脉的数量,同时可以减少叶脉间叶肉细胞的数量以及减少叶肉细胞的
密度,进而可以增加叶片的叶脉密度。本发明在创制c4水稻的应用中具有重要意义,为c4水稻的c4光合途径的引入提供了结构上的重要控制基因。本发明为c4水稻育种提供了重要的候选基因,为推动高光效水稻的创制提供了底盘材料。
附图说明
97.图1为经典的c3植物和c4植物叶片的解剖学结构示意图,上图为水稻的叶片横切,下图为玉米叶片横切,两类不同光合途径的作物叶片结构最显著差别是叶脉之间叶肉细胞的数目。
98.图2为徒手切片效果示意图,用于筛选转基因材料和叶脉数目以及叶肉细胞数目统计。
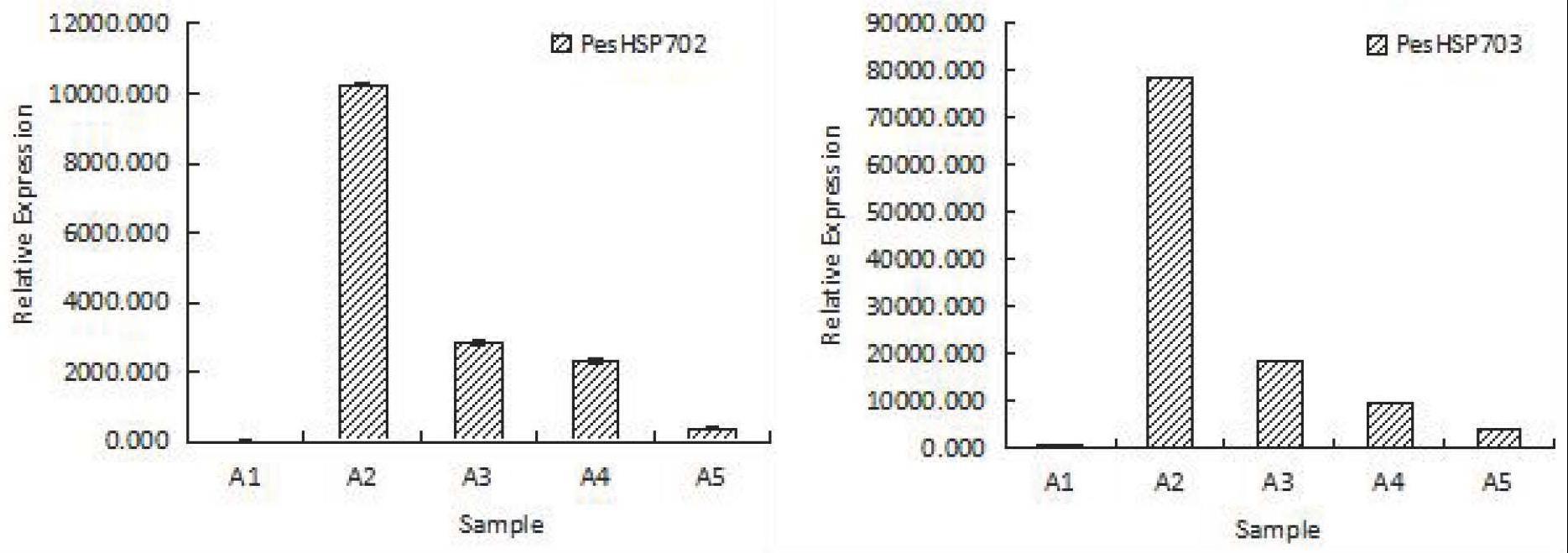
99.图3为敲除osshr1或/和osshr2转基因系的osshr1和osshr2基因的表达水平,采用定量pcr的方法分析基因的相对表达量。
100.图4为osshr1和osshr2基因敲除后植株形态。最左侧植株为对照日本晴,中间植株为转基因系1,右侧植株为转基因系2,图中白色标尺代表20厘米。
101.图5为osshr1和osshr2基因敲除后叶片形态。最左侧叶片为对照日本晴旗叶,中间植株为转基因系1旗叶,右侧植株为转基因系2旗叶,图中白色标尺代表15厘米。
102.图6为osshr1和osshr2基因敲除植株旗叶叶片的石蜡切片。图片左侧植株为对照日本晴旗叶叶片横切,中间植株为转基因系1-1旗叶叶片横切,右侧植株为转基因系1-2旗叶叶片横切,红色星芒标识叶肉细胞,图中黑色标尺代表100微米。
103.图7为敲除osshr1和osshr2植株旗叶叶片中叶脉数目和小脉间叶肉细胞数统计。左侧图为统计的旗叶中部横切小脉的数量,转基因系1和转基因系2比日本晴小脉数量明显增加(p《0.05),右侧图为统计的旗叶中部横切小脉间叶肉细胞数统计。转基因系1和基因系2比日本晴小脉间叶肉细胞数量显著减少(p《0.01)。
具体实施方式
104.下面结合具体实施方式对本发明进行进一步的详细描述,给出的实施例仅为了阐明本发明,而不是为了限制本发明的范围。以下提供的实施例可作为本技术领域普通技术人员进行进一步改进的指南,并不以任何方式构成对本发明的限制。
105.下述实施例中的实验方法,如无特殊说明,均为常规方法,按照本领域内的文献所描述的技术或条件或者按照产品说明书进行。下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。
106.以下实施例中的定量实验,如无特别说明,均设置三次重复实验。
107.下述实施例中的粳稻品种日本晴已记载于:goff,s.a.et al.,(2002).a draft sequence of the rice genome(oryza sativa l.ssp.japonica).science,296(5565),92
–
100,公众可以从中国农业科学院生物技术研究所获得,该生物材料只为重复本发明的相关实验所用,不可作为其它用途使用。
108.下述实施例中的载体pylcrispr/cas9pubi-h记载于:ma,x.et al.,(2015).a robust crispr/cas9 system for convenient,high-efficiency multiplex genome editing in monocot and dicot plants.molecular plant 8:1274-1284,公众可以从中
国农业科学院生物技术研究所获得,该生物材料只为重复本发明的相关实验所用,不可作为其它用途使用。
109.下述实施例中统计方法:采用统计学软件ssps内置的student’s t-test方法。样品数量为日本晴野生型对照,各转基因系1至少各统计11个单株。
110.实施例1、osshr1和osshr2基因的获得
111.1、osshr1基因的获得
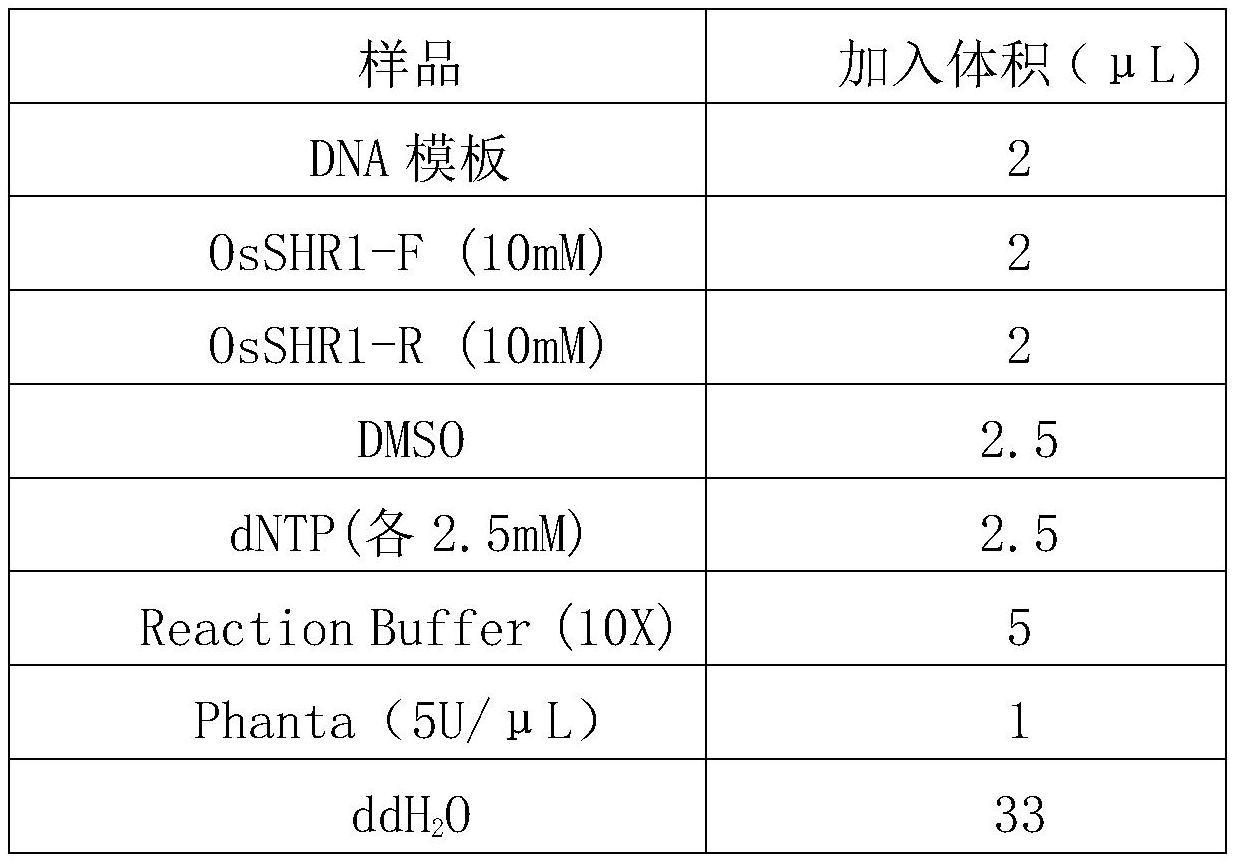
112.osshr1基因通过pcr方法扩增,高保真酶(phanta max super-fidelity dna polymerase)购自诺唯赞(vazyme)公司。以日本晴的萌发后7天叶片dna为模板,采用引物为:osshr1-f:5'-atggatacgctgtttaggttgg-3',osshr1-r:5'-tcatggccgccacgcgcttg-3'进行pcr扩增,pcr反应体系如表1所示,反应条件如表2所示,得到扩增产物(即osshr1基因)。osshr1基因在水稻品种日本晴的编码序列如seq id no.1所示的核苷酸序列,编码蛋白的氨基酸序列如seq id no.3所示。
113.表1.osshr1基因的pcr反应体系
[0114][0115][0116]
表2.osshr1基因的pcr反应条件
[0117][0118]
2、osshr2基因的获得
[0119]
osshr2基因通过pcr方法扩增,高保真酶(phanta max super-fidelity dna polymerase)购自诺唯赞(vazyme)公司。以日本晴的萌发后7天叶片来源的dna为模板,采用
引物为:osshr2-f:5'-atggataccctcttcaggttgg-3',osshr2-r:5'-tcatggcttccacgcgctcg-3'进行pcr扩增,pcr反应体系如表3所示,反应条件如表2所示,得到扩增产物(即osshr1基因)。osshr2基因在水稻品种日本晴的编码序列如seq id no.2所示的核苷酸序列,编码蛋白的氨基酸序列如seq id no.4所示。
[0120]
表3.osshr2基因的pcr反应体系
[0121]
样品加入体积(μl)dna模板2osshr2-f(10mm)2osshr2-r(10mm)2dmso2.5dntp(各2.5mm)2.5reactionbuffer(10x)5phanta(5u/μl)1ddh2o33总体积50
[0122]
表4.osshr1基因的pcr反应条件
[0123][0124][0125]
实施例2、降低表达植株的获得及表型鉴定
[0126]
1、降低表达植株的获得
[0127]
1)pcrispr-osshr1/2的构建
[0128]
pcrispr-osshr1/2构建如下:基于crispr-cas9的基因编辑系统,特异性地破坏目标基因使其不能表达功能正常的蛋白质。根据载体要求和osshr1和osshr2基因特点设计编辑的靶位点,这些位点分别是osshr1和osshr2基因单独或者共同特异位点,在整个水稻基因组中没有其配对序列。按表5列出引物,采用pcr方法扩增出各个包含靶位点的dna片段,再将片段在酶切-连接体系中将各针对目标靶点片段与载体骨架pylcrispr/cas9pubi-h连接获得pcrispr-osshr1/2敲除载体。
[0129]
pcrispr-osshr1/2敲除载体表达靶向于osshr1和osshr2基因的sgrna-1序列为:5
’‑
cttcatccatgtaaagctgg-3’,该sgrna的靶序列为:5
’‑
ccagctttacatggatgaag-3’位于seq id no.1的第282-301位,seq id no.2的第297-316位序列为:5
’‑
ccagttttacatggatgaag-3’,与sgrna-1序列(除第301位外,sgrna-1为g而seq id no.2为t)反向互补。
[0130]
pcrispr-osshr1/2敲除载体表达靶向于osshr1和osshr2基因的sgrna-2序列为:5
’‑
acatgagctgctgcacgcgc-3’,该sgrna的靶序列为:5
’‑
gcgcgtgcagcagctcatgt-3’位于seq id no.1的第627-646位和seq id no.2的第615-634位。
[0131]
pcrispr-osshr1/2敲除载体表达靶向于osshr1和osshr2基因的sgrna-3序列为:5
’‑
agcgcgtgcagcagctcatg-3’,该sgrna的靶序列5
’‑
agcgcgtgcagcagctcatg-3’位于seq id no.1的第626-645位和seq id no.2的第614-633位。
[0132]
敲除载体pcrispr-osshr1/2是在pylcrispr/cas9pubi-h载体限制性内切酶bsai位点之间插入一个敲除osshr1/2表达盒,该表达盒包含如下三个grna scaffold序列:其中间隔序列一位于seq id no.5的9024位至9046位,osu3启动子序列位于seq id no.5的9047位至9429位,包含靶位点序列为5
’‑
cttcatccatgtaaagctgg-3’(位于seq id no.5的9430位至9449位)的grna scaffold序列位于seq id no.5的第9430位至9525位;间隔序列二位于seq id no.5的第9526位至9581位,osu6a启动子序列位于seq id no.5的第9582位至10028位,包含靶位点序列为5
’‑
acatgagctgctgcacgcgc-3’(位于seq id no.5的第10029位至10048位)的grna scaffold序列位于seq id no.5的第10029位至10124位;间隔序列三位于seq id no.5的第10125位至10180位,osu6b启动子序列位于seq id no.5的第10181位至10513位,包含靶位点序列为5
’‑
agcgcgtgcagcagctcatg-3’(位于seq id no.5的第10514位至10533位)的grna scaffold序列位于seq id no.5的第10514位至10609位)以及隔序列四位于seq id no.5的第10610位至10642位的dna片段,保持pylcrispr/cas9pubi-h载体其他序列不变得到的重组质粒。经过序列测定确定连接正确,将重组质粒命名为重组载体pcrispr-osshr1/2,其核苷酸序列见seq id no.5和seq id no.6。重组载体pcrispr-osshr1/2是由17353bp组成的双链dna,一条链的第1-15000位的核苷酸序列是seq id no.5的1-15000位,第15001位-17353位的核苷酸序列是seq id no.6的1-2353位。
[0133]
表5.基因敲除所用引物序列
[0134][0135]
2)重组农杆菌的获得
[0136]
将步骤1获得的基因敲除载体pcrispr-osshr1/2通过电击的方法转入根癌农杆菌eha105(淼灵生物,货号:ccell32003)中,得到重组农杆菌,将该重组农杆菌命名为eha105/pcrispr-osshr1/2,用于转化水稻愈伤组织。
[0137]
3)水稻的遗传转化
[0138]
将日本晴成熟种子经过诱导培养基培养3周后(培养条件:32℃光照强度13230lx),挑选生长旺盛的愈伤作为转化的受体。用步骤2获得的重组菌eha105/pcrispr-osshr1/2分别侵染愈伤,25℃暗培养3天,然后在含有50mg/l潮霉素和400mg/l羧苄青霉素的筛选培养基上培养2周(培养条件:32℃,光照强度13230lx),将无农杆菌污染并有新鲜愈伤长出的个体用于分化培养(培养条件:32℃,光照强度13230lx),待分化出小苗,将其转至生根壮苗培养基上培养2周左右后(培养条件:32℃,光照强度13230lx)转田间种植,得到t0代pcrispr-osshr1/2转基因水稻植株,pcr鉴定转基因阳性植株株系,收获阳性植株获得t1代种子。
[0139]
4)osshr1/2基因突变纯合体的筛选
[0140]
将t1代种子种植获得t2代幼苗并进行如下检测。以t2代幼苗的基因组dna为模板,分别利用靶序列上游约260bp和下游约180bp附近设计的pcr引物扩增靶序列附近的左右的
dna序列,并对pcr产物进行测序以检测osshr1/2基因编辑方式,靶点pcr引物及测序引物见如下:正向引物为5
’‑
aggttggttagcctcca-3’,正向引物为5
’‑
gggctgagctcctggaac-3’。测序成功后通过crispr靶点编辑方式分析网站dsdecode(http://dsdecode.scgene.com/)并结合人工读峰图的方式与基因标准序列进行比对,对各个靶序列及其上下游序列的编辑方式进行分析。
[0141]
osshr1和osshr2基因突变纯合体突变经过测序分析,确定获得两个纯合突变系,分别命名为转基因系-1和转基因系-2。
[0142]
转基因系-1中,sgrna对应osshr1靶点之间大片段缺失353个碱基对(具体缺失位置为seq id no.1第293至646位),产生缺失、移码并提前终止的模板而造成截短和翻译错误的无功能蛋白,sgrna对应osshr2靶点附近分别插入g(seq id no.2第603位前)以及插入t(具体插入位置为seq id no.2第637位前),产生移码突变和提前终止的模板而造成截短和翻译错误的无功能蛋白。
[0143]
基因系-2中,sgrna对应osshr2靶点之间大片段缺失352个碱基对(具体缺失位置为seq id no.1第293至645位),产生缺失、移码并提前终止的模板而造成截短和翻译错误的无功能蛋白,sgrna对应osshr2靶点附近插入t(具体插入位置为seq id no.2第637位前),产生移码突变和提前终止的模板而造成截短和翻译错误的无功能蛋白。
[0144]
收获转基因系-1和转基因系-2的t2代植株所结的种子(t3代)用于后续实验。
[0145]
2、pcrispr-osshr1/2的转基因植株的表型初步鉴定
[0146]
对步骤一获得的t3代种子种植于并生长60天后获得t3代植株的叶片进行徒手切片初步筛选,标记叶片结构明显变化的株,观察植株生长状况。
[0147]
1)pcrispr-osshr1/2转基因植株的徒手切片初步筛选叶片结构改变的株系水合氯醛,乳酸,甲苯胺蓝,牙签,双面刀,载玻片均购自市场。
[0148]
a、样品
[0149]
将上述转基因水稻植株转基因以及野生型水稻(日本晴)的叶片用于徒手切片,将叶片中部切下1厘米长的叶段。实验重复2次,每次每个株系测定20株水稻。
[0150]
b、固定
[0151]
将切好的叶段样品块放入2ml离心管中,加入1.5ml新配制的卡诺固定液(无水乙醇:冰醋酸=3:1)进行固定。在室内通风橱中用真空仪器中抽真空直到叶片完全下沉,24小时后更换固定液一次。室温放置1周后徒手切片观察。
[0152]
c、切片和染色
[0153]
使用两个紧密贴合的双面刀片横切材料,将材料转移到脱色液中(饱和水合氯醛的乳酸溶液),50℃脱色液中4小时。将脱色的材料用牙签转移到清水中,然后将清水中的材料用小镊子夹起,在1%的甲苯胺蓝染料中染色10-20秒,再用清水中漂洗去除浮色。然后用牙签转移到载玻片上。
[0154]
d、显微镜观察
[0155]
使用显微镜进行材料的显微观察和并记录叶片结构变化的株系。
[0156]
结果表明:在pcrispr-osshr1/2的2个纯合转基因系中,各转基因材料中都存检查20株材料的叶片结构明显变化,最后明确pcrispr-osshr1/2两个转基因系进行细节观察和统计。
[0157]
3、基因敲除的转基因植株的叶片解剖结构表型鉴定和统计
[0158]
1)基因敲除植株的叶片石蜡切片
[0159]
将通过徒手切片观察明确的pcrispr-osshr1/2两个系进行进一步的观察和统计,具体实施是将各个转基因系种植于大田中,待植株开花进入灌浆期后20天,取旗叶的叶片进行石蜡切片以便细致观察叶片结构的变化。
[0160]
切片使用的冰醋酸,乙醇,二甲苯,粘片剂,番红,固绿,中性树胶,载玻片,盖玻片购自市场。
[0161]
a、样品
[0162]
取田间栽培pcrispr-osshr1/2上步骤2明确的各两个系开花后20天的旗叶,转基因系1和转基因系2各取样20个单株先徒手切片确定表型,徒手切片确定表型后,每个株系各取典型的两单株,每个单株各取2片旗叶用于切片。
[0163]
b、材料固定
[0164]
取旗叶叶片中段1-2厘米,用新配置的卡诺固定液(无水乙醇:冰醋酸=3:1)进行固定,室温真空泵抽气使样品完全沉没于固定液中。室温放置24小时,更换一次固定液,材料在固定液中长久保存。
[0165]
c、材料脱水
[0166]
固定后的材料用70%乙醇冲洗3次。70%乙醇脱水2小时,重复三次。75%,80%,85%,90%,100%,100%浓度乙醇梯次脱水,每次1小时。
[0167]
d、材料透明
[0168]
保存在100%乙醇中的材料使用不同梯度的乙醇/二甲苯透明液中透明,依次为100%乙醇,乙醇/二甲苯=3:1,乙醇/二甲苯=1:1,乙醇/二甲苯=1:3,100%二甲苯,100%二甲苯各1小时。
[0169]
e、材料浸蜡
[0170]
38℃左右将碎石蜡逐渐加入二甲苯中,最后到达体积比1:1为止,静止待石蜡完全溶解。将上述石蜡转移到58℃温箱中,让二甲苯完全挥发,然后分装到有材料的小烧杯中,然后58℃保温,4小时更换新石蜡,重复三次。
[0171]
f、材料包埋
[0172]
将浸蜡过的材料放置到小纸盒中,然后融化好的石蜡倒入,随后将小纸盒放入冷水中使石蜡尽快凝固。
[0173]
g、材料切片和展片
[0174]
根据需要将上述包埋有材料的蜡块用刀片修成梯形。保留蜡块完整和材料周边尽可能少保留石蜡。将修整好的蜡块粘在硬木块上(自制小木块,大约1厘米
×
2厘米
×
2厘米),随后将蜡块固定到切片机的载蜡器上。调整切片的角度,调整机器使切片厚度8微米。在干净的载玻片上各滴加一滴水和样品粘片剂后涂匀,将蜡带漂浮于该载玻片上,然后将玻片铺在42℃展片机上。
[0175]
h、溶蜡及复水
[0176]
切片置二甲苯中溶蜡至石蜡溶解干净,进入二甲苯:无水乙醇(1:1)5分钟,100%,95%,90%,85%,80%,70%,50%,35%乙醇,蒸馏水中复水各5分钟。
[0177]
i、染色及脱水
[0178]
1%番红水溶液染色约12小时,取出流水冲洗以去除多余的染料,随后经过35%,50%,70%乙醇脱水各5分钟。继续在0.1%固绿中染色约10秒,立即进入100%乙醇30秒,100%乙醇,无水乙醇:二甲苯(1:1),二甲苯各5分钟。
[0179]
j、封片
[0180]
上述载片室温吹干,镜检确定染色成功后,将一滴左右的封固剂滴到材料上,然后小心地盖上盖玻片,以后自然风干。
[0181]
k、显微镜观察以及拍照
[0182]
使用自带拍照系统的显微镜进行材料的显微观察和拍照。
[0183]
结果表明:与日本晴野生型比较,通过基因编辑的方法关闭基因osshr1和osshr2的表达,可以有效的改变水稻的叶片结构,表现为水稻叶片叶脉数目增加,各级脉之间的叶肉细胞数目明显减少,参见图6。野生型对照日本晴两个小脉之间典型存在8个细胞的间隔,而降低shr表达后,叶肉细胞减少到5个左右,基因敲除后更是减少到5个以下。
[0184]
2)pcrispr-osshr1/2转基因植株叶脉和叶肉细胞的统计
[0185]
对pcrispr-osshr1/2转基因两个转基因系取各至少11单株进行统计,对这些单株各级叶脉以及叶脉之间叶肉细胞的数目统计,统计表明叶脉数目增加和叶肉细胞数目减少都具有统计学上的下显著差异,结果见图7。采用student’s t-test,p《0.01为差异及其显著,p《0.1为差异显著。
[0186]
综上所述,osshr1蛋白和osshr2蛋白具有控制水稻叶片结构的功能,通过组成型启动子降低osshr1和osshr2基因表达,可以增加水稻叶片大脉之间小脉的数量,同时可以减少叶脉间叶肉细胞的数量以及减少叶肉细胞的密度,进而可以增加叶片的叶脉密度。
[0187]
以上对本发明进行了详述。对于本领域技术人员来说,在不脱离本发明的宗旨和范围,以及无需进行不必要的实验情况下,可在等同参数、浓度和条件下,在较宽范围内实施本发明。虽然本发明给出了特殊的实施例,应该理解为,可以对本发明作进一步的改进。总之,按本发明的原理,本技术欲包括任何变更、用途或对本发明的改进,包括脱离了本技术中已公开范围,而用本领域已知的常规技术进行的改变。
再多了解一些
本文用于创业者技术爱好者查询,仅供学习研究,如用于商业用途,请联系技术所有人。