1.本发明属于生物医药技术领域,具体而言,涉及一种构建单细胞测序文库构建的方法,更具体涉及一种单细胞转录组及染色质可及性双组学测序文库构建方法、所制备的测序文库及以及利用所述文库进行测序的方法。
背景技术:
2.单细胞转录组及染色质可及性双组学测序技术(本交底书后续简称为单细胞双组学测序技术)于2018年首次发表于science杂志(图2,sci-car-seq1),sci-car-seq采用组合索引的原理实现了对单个细胞的编码,然而由于只有两轮组合索引流程,理论通量只有万级。随后研究人员又开发出基于流式细胞分选技术(图3,sccat-seq2,2019,百级通量)及基于微流控(图4,snare-seq3,2019,万级通量)的单细胞双组学测序技术。提升通量与质量始终是技术发展的方向。由于组合索引技术可以提供极高通量的测序技术,研究人员基于此又开发出了百万级通量的单细胞染色质可及性及转录组双组学测序技术(图5,paired-seq4,2019;图6,share-seq5,2020)。目前市面上已有10x genomics公司发布的首个基于微流控技术的单细胞染色质可及性及转录组双组学测序试剂盒(chromium,2020)。在专利申请日前最新开发的技术可基于流式分选或微流控进行细胞编码(issac-seq6,2022)。
3.目前已有的单细胞双组学测序技术简介如下:
4.sci-car-seq1:先将细胞分散至孔板的不同的孔中,通过逆转录反应将mrna逆转录成cdna并使来自于同一个孔的细胞的cdna获得第一轮编码,再用转座子进行转座反应使来自于同一个孔的细胞的染色质开放位点带上第一轮编码,然后将所有细胞混合均匀再分散到新的孔板中进行裂解,取一部分裂解液进行cdna文库的扩增,并使cdna带上第二轮编码,另一部分裂解液进行染色质开放位点文库的扩增,并使染色质开放位点文库带上第二轮编码。最终通过识别两轮编码的组合来确定来自于同一个细胞的转录组文库及染色质开放位点文库(sci-car-seq技术流程示意图见图2)。
5.sccat-seq2:用激光将细胞分选到孔板中的孔中,每个孔有单个细胞。将空中的细胞裂解,分离细胞质与细胞核,并将细胞质进行逆转录反应构建转录组文库,同时将细胞核进行转座反应构建染色质开放位点文库(sccat-seq技术流程示意图见图3)。
6.snare-seq3:制备单个细胞核(单核)悬液,对单核进行tn5转座子转座反应,捕获染色质开放位点信息,通过微流控流程,将捕获磁珠、单核及splint引物包裹在一个微滴中,在微滴中进行反应,将染色质开放位点序列信息转移到磁珠上,并且利用磁珠的引物将mrna信息进行捕获。对文库进行扩增获得可供测序用的转录组文库及染色质开放位点文库(snare-seq流程示意图见图4)。
7.paired-seq4:将细胞均匀分散到不同的孔中,用转座及逆转录反应分别使同一个孔中的细胞中的染色质开放位点及转录组带有相同的编码,利用三轮组合索引的方法进行编码的连接,最后根据编码的组合来确定来自于同一个细胞的染色质开放位点及转录组序
列(paired-seq流程示意图见图5)。
8.shared-seq5:先将细胞进行转座,再用含有生物素标记的逆转录引物进行逆转录;利用组合索引的方式,将细胞均匀分散到不同的孔中进行编码连接再重复两次,最后可根据编码的组合识别来自于同一个细胞的染色质开放位点信息及转录组信息(share-seq技术流程示意图见图6)。
9.10x chromium单细胞多组学(atac 基因表达)试剂盒:制备单个细胞核(单核)悬液,对单核进行tn5转座子转座反应,捕获染色质开放位点信息,通过微流控流程,将捕获磁珠、单核及反应体系包裹在一个微滴中,在微滴中进行反应,同时给细胞的染色质开放位点及转录组文库进行编码。
10.issac-seq6:制备单个细胞核(单核)悬液,对单核进行tn5转座子转座反应,捕获染色质开放位点信息;接着进行逆转录反应捕获转录组信息,后续可选择通过微流控流程,将捕获磁珠、单核及反应体系包裹在一个微滴中,在微滴中进行反应,同时给细胞的染色质开放位点及转录组文库进行编码;或者也可以选择采用流式分选的方法,分选到孔板中进行编码连接。
11.然而目前所有的单细胞双组学测序技术中主要获得的是信使rna的3’端信息,无法保留信使rna较多的5’信息,这导致目前已有的单细胞双组学测序技术的使用存在一定限制。例如目前的双组学技术无法方便地对进行基因编辑筛选(crispr-screen)的商业化sgrna序列库进行测序,通过获得sgrna序列及基因编辑或基因调控后的转录组及染色质开放位点变化信息,可以便于进行转录调控网络及细胞信号转导网络等的研究。同时3’测序对tcr测序及bcr测序兼容有一定的限制,使得在免疫系统的单细胞研究中通过tcr及bcr信息进行溯源成为困难。而通过5’测序可以获得tcr及bcr信息,并对免疫细胞进行溯源,可以对免疫细胞的演化轨迹推测进行进一步修正,得到更加可靠的结果,对于免疫可塑性的研究十分重要。本公开所展示的技术原理可解决这些问题和其他相关需求。
技术实现要素:
12.本技术发明人设计了一种可捕获信使rna5’信息的高通量单细胞转录组及染色质可及性双组学测序建库技术。具体而言,通过以下项目所示技术方案解决了本领域中存在的技术问题。
13.1.一种单细胞转录物和染色质可及性双组学单细胞测序文库的构建方法,其包括以下步骤:
14.a)制备单细胞悬液;
15.b)获取染色质开放位点,其包括用引物二聚体和tn5转座酶组装而成的tn5转座子进行对所述细胞的染色质进行转座反应,对细胞的染色质开放位点进行切割,并将引物二聚体连接到染色质开放位点;
16.c)使用逆转录酶和逆转录引物对细胞的转录组进行逆转录反应,以获取单细胞的转录组信息;
17.d)使用模板转换寡聚物(tso)进行模板转换反应;
18.e)后续编码以得到带有细胞编码的转录组文库和染色质开放位点文库,所述后续编码包括根据需要采用不同的平台或流程对细胞进行单细胞分离或编码整合,其中来自同
一个细胞的转录组文库和染色质开放位点文库带有相同的细胞编码或细胞编码组合;
19.f)初始文库扩增,其包括对前面构建的文库进行扩增以增加可供使用的文库片段;
20.g)制备测序用染色质开放位点文库以及转录组文库,其包括从上一步扩增的文库分离出染色质开放位点文库和转录组文库,
21.其中所述模板转换寡聚物(图1所示)包含与逆转录酶的末端转移酶活性生成的序列
⑧
完全或部分互补的序列
⑤
,使得所述逆转录酶可以以模板转换寡聚物为模板继续进行序列合成,所述模板转换寡聚物还包含位于序列
⑤
的5’端的部分结构区
④
以及位于所述部分结构区
④
的5’端的用于后续编码整合的把手序列
③
,任选地,所述模板转换寡聚物还包含与部分结构区
④
互补以与模板转换寡聚物形成双链结构的序列
⑥
。
22.2.项目1所述的方法,其中步骤a)中的所述单细胞悬液不用或用固定剂,例如甲醛固定后用于后续步骤。
23.3.项目2所述的方法,其中所述固定后进行终止固定或者不终止固定而用于后续步骤。
24.4.项目1至3任一项所述的方法,其中所述引物二聚体、模板转换寡聚物、逆转录引物被修饰,例如被磷酸化修饰及生物素修饰。
25.5.项目1至4任一项所述的方法,其中所述图1序列
⑥
被3’磷酸化修饰,并且在后续反应中可被消除3’磷酸化修饰,例如,通过多聚核苷酸激酶反应。
26.6.项目1至5所述的方法,其中所述模板转换寡聚物附着到介质如珠子、磁化珠子、微孔、芯片、平板上或者处于游离状态。
27.7.项目1至6所述的方法,其中所述图1部分结构区
④
包括一个或多个功能区,所述功能区可选自并不限于独特分子识别信号(umi)、部分编码和双链互补序列等。
28.8.项目1至7所述的方法,其中所述图1序列
⑤
由dna、rna、锁核酸或其组合组成。
29.9.项目1至4所述的方法,其中所述图1序列
⑥
被5’磷酸化修饰。
30.10.项目1至9所述的方法,其中初始文库扩增通过pcr反应进行,所用引物包含编码结构、作为细胞编码组合中的一部分,和/或包含样本来源、实验批次、或细胞种类的编码。
31.11.项目1至10所述的方法,其还包括对分离后的染色质开放位点文库及转录组文库进行进一步的扩增及纯化。
32.12.项目1至11所述的方法,其还包括对分离后的染色质开放位点文库添加测序通用接头和/或对分离后的转录组文库添加测序通用接头。
33.13.项目1至12所述的方法,其中所述转录组包括但不限于编码t细胞受体(tcr)、b细胞受体(bcr)的mrna和/或crispr系统中的guide rna,所述转录组文库包括tcr文库、bcr文库和/或grna文库。
34.14.通过项目1至13所述的方法制备的单细胞转录组和染色质可及性双组学测序文库。
35.在本文中将本发明技术命名为ustc-v-seq,其采用新型编码(barcode)连接方式,可以将编码连接在染色质开放位点或/和转录组上并保留转录组的5’信息,并可以将来自于同一个细胞的染色质开放序列片段及转录组序列上带有相同的编码组合。同时由于存在
特殊序列,可以从获得的转录组文库进一步富集grna文库及bcr或/和tcr文库序列,完成相应的grna测序、bcr测序及tcr测序。基于此,ustc-v-seq可同时获得来自于同一个细胞的染色质可及性信息及转录组信息并可兼容grna信息(若含)、bcr序列信息(若含)以及tcr序列信息(若含)的获取。
36.本发明中模板转换的设计具有独创性。在一般的知识背景下,大多数人会认为在进行模板转换反应中新合成的互补链会补平模板转换链,其他技术的流程图或示意图中也会默认补平,理论上编码是无法连接在模板转换寡聚物上的。但这种共识并不是完全正确的,在我们的实验中发现,即使是单链的模板转换寡聚物,在进行完转座反应后,编码也可以连接在模板转换链上,并经过多次实验验证可行。背后的机理目前还没有验证,但推测可能是由于不完全反应导致模板转换寡聚物并没有被补平,所以编码整合把手位点得以与编码进行连接,从而实现对细胞的编码。加入双链也是希望能进一步阻挡模板转换反应的进行,在实际情况下,编码可以整合到单链模板转换寡聚物或双链模板转换寡聚物的编码整合把手位点上。
附图说明
37.图1.转录组的获取示意图。
38.图2.sci-car-seq技术流程示意图。
39.图3.sccat-seq技术流程示意图。
40.图4.snare-seq流程示意图。
41.图5.paired-seq流程示意图。
42.图6.share-seq技术流程示意图。
43.图7.后续编码的整合示意图。
44.图8.细胞编码效果图。
45.图9.转录组数据基因比对图。
46.图10.染色质开放位点与转录组数据具相关性。
具体实施方式
47.为使本发明的目的、技术方案和优点更加清楚明白,以下结合具体实施例,并参照附图,对本发明作进一步的详细说明。除非在本文中特别说明,本技术中的术语具有本领域技术人员通常理解的含义。
48.定义
49.编码(barcode):本文所述的编码(barcode、barcoding或index)或编码组合是指由核酸构成的不同碱基序列,例如atcg及tacg为两种不同编码。
50.细胞:哺乳动物(如人、鼠)行驶生命活动的基本组成元件。本公开中的细胞不只局限于细胞整体,还另外指代包括其他细胞组成部分,如细胞核、线粒体等。例如单细胞悬液也可以是单细胞核悬液。
51.染色质:细胞核内由dna、组蛋白、非组蛋白及少量rna组成的线性复合结构。其基本原件为dna缠绕在组蛋白上形成的核小体。
52.染色质可及性:即评价某段dna是否缠绕在组蛋白上。一般情况下,染色质有两种
情况:1)dna紧紧缠绕在核小体上,称为关闭的dna;2)dna为缠绕在核小体上,呈裸露状态,称开放的dna。
53.染色质可及性文库:由染色质开放位点的序列构成的测序文库。
54.染色质可及性测序(atac-seq):一种2012年斯坦福大学开发的一款,用于检测生物样本(》500细胞)染色质可及性情况的测序技术。
55.基因组:即生物体全dna序列,由atcg四种碱基有序排列组成。人、鼠等主要哺乳动物的基因组已经全部测序完成。
56.基因:基因(遗传因子)是产生一条多肽链或功能rna所需的全部dna序列。一个基因一般是基因组上一段或多段dna。
57.转录组:transcriptome,也称为“转录物组”,广义上指细胞所能转录出的所有rna的综合,包括信使rna(mrna),核糖体rna(rrna),转运rna(trna)及非编码rna;狭义上指细胞所能转录出的所有信使rna(mrna)。本交底书中即采用特有的定义,即所有符合建库需求的rna,包含但不限于mrna及crispr系统中的grna等。
58.抗原(antigen):是指能引起抗体生成的物质,是任何可诱发免疫反应的物质。
59.抗体(antibody):是指机体由于受到抗原刺激而产生的具有保护作用的蛋白质。
60.b细胞受体(bcr,b cell receptor):位于b细胞表面的负责特异性识别和结合抗原的分子,本质是膜表面的免疫球蛋白。
61.t细胞受体(tcr,t cell receptor):位于t细胞表面的特异性受体,负责识别由主要组织相容性复合体(mhc)所提呈的抗原;但与b细胞受体不同,无法识别游离抗原。
62.b细胞受体/t细胞受体测序(bcr-seq/tcr-seq):靶向于b细胞受体/t细胞受体序列的测序技术。
63.转录因子:一种可识别特定dna序列模式(motif)并结合在dna上的蛋白质,可以启动或调节基因的表达。
64.单细胞染色质可及性测序(scatac-seq):用于检测单个细胞染色质可及性的测序方法。
65.单细胞转录组测序(scrna-seq):用于检测单个细胞转录组的测序方法。
66.单细胞多组学测序:用于检测单个细胞多个维度信息的测序文库构建方法,比如同时获得来自于同一个细胞的转录组、染色质可及性及/或蛋白组信息等双组学或更多组学信息的测序文库构建方法(如sci-car、paired-seq、shared-seq、10x chromium、cite-seq、reap-seq等)。
67.本文所述的“模板转换寡聚物(tso)”为可以被命名为其他名称或无名称,其实质为一段单链或双链核酸序列,这段序列可以附着包括但不限于珠子、磁化珠子、微孔、芯片、平板等介质上,也可以处于游离状态。模板转换寡聚物(tso)可包含包括但不限于编码、独特分子识别信号(unique molecular identifier,umi)及后续编码整合把手位点等功能区域。
68.本文所述的“模板转换反应”通常指在逆转录酶的作用下,以模板转换寡聚物为模板继续进行序列合成的反应,生成可与模板转换寡聚物互补或部分互补的序列。
69.单细胞悬液的制备
70.非游离状态的组织或/和游离状态的组织(如血液)可通过各种途径包括但不限于
切割、研磨及酶解等制备为单细胞悬液。所述组织可以是健康状态下的组织或疾病状态下的组织,并且所述组织的存在状态包括但不限于新鲜组织、冷冻组织、切片组织等。在一些实施方式中,可能需要提取细胞核,提取环节包括但不限于使用研磨、透化剂处理、流式细胞术分选等。
71.单细胞悬液的固定
72.在一些实施方式中,单细胞悬液可以不进行固定也可以进行后续流程。在一些实施方式中,单细胞悬液中细胞浓度需要进行测定并采用合适的固定体系(含有固定剂)以达到理想固定效果。可以使用的固定剂包括但不限于醛类如甲醛、戊二醛和多聚甲醛,醇类如甲醇、乙醇,以及丙酮等,并且可以以任何合适的水平或浓度使用固定剂。
73.在一些实施方式中,固定后可进行固定终止,可以使用包括但不限于任何合适的水平或浓度的甘氨酸及牛血清白蛋白或其他试剂或方式等进行固定终止。在一些实施方式中,可不终止固定直接进行后续反应。
74.染色质开放位点的获取
75.可以使用设计的引物二聚体与tn5转座酶孵育,组装成可以用于转座的tn5转座子。配制相应的tn5转座体系,并使用tn5转座子进行转座反应,对细胞的染色质开放位点进行切割,将引物二聚体连接到染色质开放位点。引物二聚体上通常含有后续编码整合的把手(handle)序列。
76.设计引物二聚体的方法是本领域中公知的。在一些实施方式中,引物二聚体可以根据需要自己进行设计或调整,本文中只提供一种可实施的方案举例,并不限制本发明所使用的序列。
77.tn5转座酶可以是自己合成或纯化获得的,也可以是从供应商购买的,包括但不限于例如vazyme s601-01。引物与转座酶的孵育体系可以是自己进行配制的或者是商业化的。孵育反应及转座反应的反应条件可以进行调整。
78.在一些实施方式中,引物二聚体的任何组分可以进行修饰,这种修饰包括但不限于磷酸化修饰及生物素修饰。
79.转录组信息的获取
80.可以使用逆转录引物对细胞进行逆转录反应,逆转录反应完成后,可于42℃进行孵育一段时间,并进一步使用模板转换寡聚物(tso)进行模板转换反应。模板转换寡聚物可以为逆转录产物提供后续编码整合的把手(handle)序列。
81.模板转换反应的结果是将模板转换寡聚物、模板转换寡聚物的互补产物或部分模板转换寡聚物的互补产物整合到文库序列上。
82.在一些实施方式中,“模板转换反应”可以采用包括但不限于逆转录反应、连接反应及dna聚合反应等替代。在一些实施方式中,逆转录酶可使用包括但不限于基于小鼠白血病病毒(mmlv)的逆转录酶及其衍生的逆转录酶。在一些实施方式中,逆转录引物可以进行修饰,这种修饰包括但不限于生物素修饰。
83.42℃下孵育的时间可以根据需要调整。也可以不进行42℃下的孵育。通常,42℃下的孵育可以在一定程度上提高产率。
84.模板转换寡聚物包括但不限于经修饰或非修饰的双链模板转换寡聚物或经修饰或非修饰的单链模板转换寡聚物。例如为了避免双链tso在逆转录酶的作用下进行延伸导
致编码连接位点被封闭,同时可以保证双链tso的连接效率,可以通过对双链模板转换寡聚物进行包括但不限于3’磷酸化修饰及锁核酸修饰等。
85.在一些实施方式中,双链模板转换寡聚物(tso)可由两条核酸序列退火形成。
86.在一些实施方式中,修饰的双链模板转换寡聚物(tso)所指的修饰包括但不限于只含有或同时含有5’磷酸化、3’磷酸化及锁核酸等。
87.在一些实施方式中,tso修饰需要得到解除;完成模板转换环节后使用可移除某个或某些特殊修饰的相关酶移除掉某个或某些特殊修饰,使得双链tso暴露出可以进行后续反应的化学基团。在一些实施方式中,所述修饰可能不影响本文的后续反应环节,所以并不一定需要进行特殊修饰的解除。
88.在一些实施方式中,本文所述的特殊修饰可能会采用3’磷酸化进行修饰,此时,可能需要或不需要使用包括但不限于t4多核苷酸激酶(polynucleotide kinase)(neb m0201s或neb m0201l)进行反应去除特殊修饰。
89.在转录组信息的获取程序中,为后续编码的整合在模板转换寡聚物上提供了把手位点,从而保证了编码整合在mrna序列的5’端,其他单细胞双组学测序技术均未采用此种编码整合的方法。同时引入了42℃的孵育环节增加了产物产量。
90.在图1中示例的转录组获取的示意图。通过此环节可以使转录组获得部分5’编码及后续编码整合把手。图1中
①
所示为模板转换寡聚物(tso)单链或双链中的单链,包含
③
、
④
及
⑤
三个部分。图1中
②
所示为转录组,包括但不限于mrna及crispr系统中的grna。图1中
③
所示为后续编码整合把手序列,序列长度是一个范围,可根据实验进行调整,在一些实施方式中亦可以去掉此片段。图1中
④
所示为tso的部分结构区,这一部分可包括一种或多种功能区域,功能区域包括但不限于独特分子识别信号(umi),部分编码(功能包括但不限制于用于细胞识别、组织识别等),双链互补序列等。图1中
⑤
所示为图1中
⑧
互补碱基,m数量是一个范围,可根据实验进行调整,m可为相同碱基或不同碱基,具体碱基可依据图1中
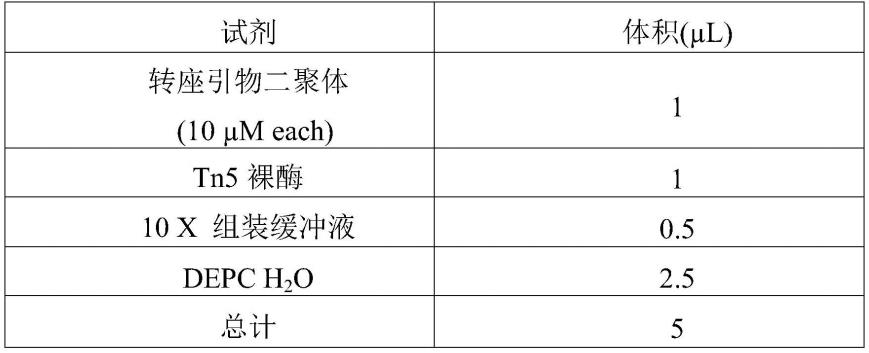
⑧
中的n来确定;m可以是包括但不限于脱氧核糖核酸、核糖核酸及锁核酸。图1中
⑥
所示为与图1中
④
互补的一段序列,可以与模板转换寡聚物形成双链结构以增加
③
区域为单链的可能性;此外图1中
⑥
所示序列可以是经过修饰的,这种修饰包括但不限于3’磷酸化、5’磷酸化等。图1中
⑦
为以图1中
④
部分序列为模板新生成的序列。图1中
⑧
为在逆转录酶的末端转移酶活性下生成的序列,长度是一个范围,具体的序列也与使用的逆转录酶有关。图1中
⑨
所示为在逆转录引物下合成的序列。
91.后续编码
92.可根据需要采用不同的平台或流程对细胞进行单细胞分离或编码整合,这些平台或流程包括但不限于基于微流控技术、基于流式分选技术及基于组合索引的细胞编码技术。图7所示为后续编码的整合环节。通过此环节,后续编码可以整合到转录组文库中相对应rna的5’端,并同时使来自于同一个细胞的转录组文库及染色质开放位点文库带有相同的编码。图7a所示为进行后续编码环节前的文库结构示意图,其中
①
所示为转座后染色质开放位点文库结构示意,其中x为基因组开放序列;
②
所示为模板转换后转录组文库结构示意,其中m和n含义如图1中的
⑤
和
⑧
所述;
③
所示为
①
和
②
的简化版,转录组文库和染色质开放位点文库的编码整合原理相似故下文用
③
的示意图对染色质开放位点文库及转录组文库进行替代;
④
所示为编码整合把手位点;
⑤
所示为另一端突出区域,可根据需要进行设
计,可以与
④
相同或者不同,可用于编码整合或其他用途。图7b所示为基于介质捕获文库示意图(适用于包括但不限于微流控技术、微孔技术等),其中
⑥
为一段结构序列与
⑦
互补,
⑥
与
⑦
形成的复合体可以与编码整合把手位点进行整合,
⑥
与
⑦
中至少一条包含包括但不限于细胞编码信息的序列,
⑥
与
⑦
中至少一条与介质界
⑧
面结合;
⑧
即为介质界面,可包括但不限于平面、曲面、球面等。图7c所示为基于扩增方法将编码整合到
③
文库(适用于包括但不限于流式分选技术等),
④
为编码整合把手位点,在扩增方法中可以不使用此位点,而在
⑨
引物中加入包括但不限于细胞编码信息的序列以将编码整合到
③
文库,
⑩
为另一端的扩增引物,可以根据需要加入或者不加入包括但不限于编码信息的序列;图7d所示为基于非介质及扩增方法介导的编码整合方式(适用于包括但不限于组合索引技术、流式分选技术等),
④
为编码整合把手位点,及组成了第一轮编码序列,可以与
④
位点整合,及组成了第二轮编码序列,可以与的游离端进行整合,此外可根据需要设计第三轮及以上编码,在一些实施方式中可以使用一轮或多轮编码进行整合。
93.所述的“整合”或“结合”包括但不限于依靠序列完全互补整合或结合、依靠序列部分互补整合或结合,或连接整合或结合等。所述的整合或结合结果包括但不限于形成双链结构或形成单链结构等。
94.所述的通过微流控技术将细胞与珠子制备成反应体系,可采用任何合适的细胞或者细胞组成部分(如细胞核等)。
95.所述的“后续反应”包括但不限于连接反应及聚合酶链式扩增(pcr)反应等。
96.在一些实施方式中,细胞可以通过分选技术分选到反应单元中。细胞中染色质开放位点序列的编码整合把手位点及转录组上的编码整合把手位点可以和反应单元中的物质进行结合或反应进行编码整合,从而将新的编码整合至染色质开放位点序列及转录组序列上。
97.所述的分选技术包括但不限于流式分选技术。
98.所述的反应单元包括但不限于孔板的孔中或微孔板的孔中。
99.所述的“结合或反应”包括但不限于依靠序列互补结合、依靠序列部分互补结合或连接结合等。
100.所述的“结合或反应”包括但不限于连接反应及聚合酶链式扩增(pcr)反应等。
101.所述的“结合或反应”结果包括但不限于形成双链结构或形成单链结构等。
102.所述的“编码”包括但不限于源自单链或双链。
103.在一些实施方式中,可以不包含把手位点。
104.在一些实施方式中,细胞可以通过基于组合索引的方式进行多轮编码。细胞混合均匀后随机分散到包含数个反应单元的集合中,染色质开放位点序列的把手位点及tso上的把手位点可以和组合索引的编码反应单元中的物质进行结合或反应进行编码整合;每一轮整合完成后,需要通过使用设计过的阻断物(blocker)对没有整合的编码进行封闭,避免对后续流程的干扰;若有多轮组合索引,每一轮整合完成后会使染色质开放位点序列及转录组序列分别整合到可供后续编码整合的把手位点。通过一轮或多轮的编码整合,可以将新的编码整合至染色质开放位点序列及转录组序列上。
105.所述的反应单元包括但不限于孔板的孔中或微孔板的孔中。
106.所述的“包含数个反应单元的集合”包括但不限于孔板或联管等。
107.所述的“后续反应”包括但不限于连接反应及聚合酶链式扩增(pcr)反应等。
108.在一些实施方式中,需要进行细胞裂解。细胞需要在混合均匀后分配到不同的孔(≥1)中进行裂解,每个孔中的细胞数量≥0。从而,释放染色质开放位点序列以及转录组序列以进行后续流程。
109.所述的裂解反应在一定情况下可能需要进行终止以避免对后续流程的干扰。
110.在一些实施方式中,可使用附着在介质上的核酸序列同时整合到染色质开放位点及转录组上以进行编码,并可以跳过编码整合环节而进入后续流程。
111.本发明可使用模板转换寡聚物上的把手位点,将后续编码整合在模板转换寡聚物上,从而使编码整合在mrna序列的5’端,亦可以将编码设计到扩增引物上在相对mrna的5’端加上编码。相比,本领域已知的其他单细胞双组学技术的编码整合均在mrna序列的3’端。
112.初始文库扩增
113.构建的文库需要进行扩增以增加可供使用的文库片段。
114.在一些实施方式中,可以在终止后的细胞裂解液中加入包含编码的扩增引物及其他进行聚合酶链式反应(pcr)的体系,并进行pcr反应。
115.所述的引物可包含编码结构,可以作为细胞编码组合中的一部分,亦可以根据需要调整为包括但不限于作为样本来源、实验批次、细胞种类等的编码。
116.在一些实施方式中,可以将整合了文库信息的珠子放入文库扩增体系中,进行文库扩增。
117.在本领域中已经报道了在扩增引物上加入编码对细胞进行编码。但是,本发明的技术的编码除了能对细胞进行编码,同时可根据需要对样本来源、试验批次、细胞种类等进行编码标记,在后续整合多样本、多批次及多细胞种类数据时可以进行区分。
118.测序用染色质开放位点文库以及转录组文库的准备
119.在一些实施方式中,可使用链亲和素磁珠对含有生物素标记的染色质开放位点文库或转录组文库进行结合抓取,并将磁珠与文库的混合液置于磁力架上进行分离;不含生物素标记的转录组文库或染色质开放位点文库会存在于上清中,而含有生物素标记的染色质开放位点文库或转录组文库会结合在磁珠上被磁力架吸附。
120.在一些实施方式中,可根据需要再分别对分离后的染色质开放位点文库及转录组文库进行进一步的扩增及纯化。所述的纯化包括但不限于去除杂质及片段长度筛选等。
121.在一些实施方式中,分离获得的染色质开放位点文库可能没有测序用的接头,还无法进行测序,所以需要对文库添加测序通用接头。所述的添加测序通用接头的反应包括但不限于使用pcr反应、连接反应等。
122.在一些实施方式中,添加测序通用接头后还需要进行扩增及纯化。所述的纯化包括但不限于去除杂质及片段长度筛选等。
123.在一些实施方式中,分离获得的全长转录组文库可能没有测序用的接头,还无法进行测序。对纯化后的转录组文库进行处理以加入测序接头。所述的“处理”包括但不限于使用tn5转座、核酸酶切割、pcr反应及连接反应等的单一环节处理或多环节处理。
124.特殊序列文库的制备
125.获得的转录组文库中若含有t细胞受体序列、b细胞受体序列、grna序列等特殊序
列文库,若有需要则可使用相关引物进行富集(包括但不限于使用pcr进行扩增及生物素链亲和素磁珠抓取等),富集后的t细胞受体测序文库、b细胞受体测序文库及grna序列测序文库如果没有测序使用接头,则需要再进行处理,使得特殊序列测序文库获得测序通用接头,包括但不限于使用tn5转座反应、pcr反应、连接反应等。
126.由于编码整合在mrna序列的5’端,可以在富集t细胞受体序列、b细胞受体序列及grna序列等特殊序列文库的同时保留细胞编码组合,所以可以实现兼容相关特殊序列文库的制备及测序。
127.实施例
128.以下的实施例便于更好地理解本发明,但并不限定本发明。
129.如无特殊说明,下述实施例中的实验方法均为常规实验方法,所用试剂均可常规购买得到。
130.下述实施例中的实验试剂、器材及具体实验条件等已被验证可使得本发明得以实现,所述实验试剂、器材及具体实验条件只为便于理解本发明,但并不限定本发明。使用替代试剂、替代器材或替代条件均在本发明的保护范围内。
131.实施例1
132.该实施例中基于k562细胞系及nih3t3细胞系进行混合细胞系文库制备,仅制备染色质开放位点文库及mrna逆转录得到的转录组文库(未包含特殊文库,如tcr文库,bcr文库,crispr grna文库等)。
133.1.制备单细胞悬液
134.k562为悬浮细胞系,无需使用胰蛋白酶处理,nih3t3为贴壁细胞系,需用一定浓度的胰蛋白酶进行处理以使其悬浮。
135.2.细胞固定
136.可以选择对细胞进行固定以完成建库流程,也可以不进行此环节。固定可以选择用66.8μl 1.6%甲醛溶液加入到1ml单细胞悬液(悬于磷酸缓冲盐水(phosphate buffered saline),pbs)中,细胞密度为106个/ml。固定5分钟后用固定终止液(56μl 2.5m甘氨酸,20μl 1m tris-hcl 8.0,13.4μl 7.5%bsa)进行终止5分钟,离心除去。并用pbs-bsa-ri洗液洗涤两次,将细胞重悬于1xtd缓冲液(由4xtd缓冲液稀释)中。
137.表1.pbs-bsa-ri洗液体系
138.试剂体积(微升,μl)1x pbs987.510%bsa10.1m dtt10superasein1rnaseout0.5总计1000
139.表2. 4xtd体系
140.试剂体积(微升,μl)1m tris-acetate,ph7.81325m乙酸钾52.8
1m乙酸镁40二甲基甲酰胺(dmf)640depc h2o135.2总计1000
141.3.染色质开放位点的获取
142.为获取染色质开放位点,先根据表3体系对tn5裸酶进行组装,于室温孵育1小时备用。
143.表3.tn5转座子组装体系
[0144][0145]
表3所示tn5转座子组装体系中10x组装缓冲液为供应商提供组装缓冲液,组装反应根据供应商提供的标准操作流程进行。转座引物二聚体为双链dna结构,退火体系及条件根据供应商提供的标准操作流程进行。用组装好的tn5转座子在表4所示反应体系下于37℃500rpm恒温摇床进行转座30分钟。转座完成后,染色质开放位点序列将带有后续编码整合把手位点。
[0146]
表4.转座反应体系
[0147][0148]
[0149]
4.转录组信息的获取
[0150]
转座完成后加入等体积的nib-bsa-ri(h)洗液(表6),于500xg,4℃离心5min,去掉上清重复洗涤共三次。洗涤完成后用逆转录体系(表7)重悬细胞沉淀,并进行逆转录反应(表8)。
[0151]
表5.nib体系
[0152]
试剂体积(μl)depc h2o98401m tris-hcl 7.51005m nacl201m mgcl2305%毛地黄皂苷10总计10000
[0153]
表6.nib-bsa-ri(h)体系
[0154]
试剂体积(μl)nib10010%bsa0.50.1m dtt1superasein1rnaseout0.5
[0155]
表7.逆转录体系
[0156][0157]
表8.逆转录热循环
[0158][0159][0160]
逆转录反应完成后加入适量体积(如50μl)的nib-bsa-ri(h)洗液,于500xg,4℃离心5min,去掉上清重复洗涤共两次。洗涤完成后用模板转换体系(表9)重悬细胞沉淀,并进行模板转换反应(表10)。若模板转换寡聚物采用双链结构,需要先进行退火。模板转换反应完成后加入适量体积(如50μl)的nib-bsa-ri洗液,于500xg,4℃离心5min,去掉上清重复洗涤共两次。用适量(如1152μl)nib-bsa-ri(l)(表13)重悬细胞。此时,细胞中的转录组已经在rna的5’端提供了后续编码整合把手位点。
[0161]
表9.模板转换体系
[0162][0163]
表10.模板转换反应条件
[0164][0165][0166]
表11.模板转换寡聚物退火体系
[0167]
试剂体积(μl)100μm模板转换寡聚物单链110100μm模板转换寡聚物单链210h2o(退火后加入)30
[0168]
表12.模板转换寡聚物退火条件
[0169][0170]
表13.nib-bsa-ri(l)体系
[0171]
试剂体积(μl)nib10010%bsa0.50.1m dtt1superasein0.25rnaseout0.125
[0172]
5.后续编码整合
[0173]
后续编码整合可采用不同方式,本实施例采用组合索引方式,故下述步骤以组合索引为例。
[0174]
配制后续编码整合反应体系(表14),将细胞混匀分散至含有第一轮编码的96孔板中,并于25℃300rpm恒温摇床孵育30min。反应完成后加入第一轮封闭引物,再于25℃300rpm恒温摇床孵育30min进行封闭。封闭后,将96孔板中所有反应体系合并混匀并加入192μl t4 dna 连接酶(ligase),再混匀分散至每孔含有10μl第二轮编码的96孔板中,于25℃300rpm恒温摇床孵育30min,反应完成后加入第二轮封闭引物,再于25℃300rpm恒温摇床孵育30min进行封闭。封闭后,将96孔板中所有反应体系合并,与500xg,4℃离心5min后去除上清,并用nib-bsa-ri(l)清洗两次。
[0175]
表14.后续编码整合体系(组合索引体系)
[0176][0177]
表15.分子杂交液体系
[0178][0179]
表16.第一轮编码退火体系(针对每一个编码)
[0180][0181]
表17引物退火缓冲体系
[0182]
引物退火缓冲液体积(μl)终浓度1 m tris 8.01010 mm5 m nacl1050 mm0.5 m edta21 mm
h2o(或depc h2o)978 total1000 [0183]
表18.编码退火条件
[0184][0185]
表19.第一轮编码封闭体系
[0186][0187][0188]
表20.第二轮编码退火体系(针对每一个编码)
[0189][0190]
表21.第二轮编码封闭体系
[0191][0192]
6.初始文库扩增
[0193]
此环节可以再引入一轮编码,也可以不加入新的编码。本实施例在此环节引入了一轮编码,以下为引入新一轮编码的流程。
[0194]
细胞洗涤后,用1478μl 10mm tris-hcl ph7.5重悬细胞,并均匀地分散到96孔板中(每孔14μl,会有一定剩余),每孔加入2μl细胞裂解缓冲液及0.2μl 20mg/ml蛋白酶k混合均匀,然后于55℃,500rpm恒温摇床孵育15min.孵育结束后,每孔加入4μl 10%tween-20及0.4μl100 mm pmsf终止。
[0195]
配制初始文库扩增体系,进行10轮线性扩增后加入指数扩增引物再进行5轮指数扩增(所有的扩增轮数可根据情况进行调整)。
[0196]
表22.线性扩增体系
[0197][0198]
表23.线性扩增热循环
[0199][0200]
表24.指数扩增体系
[0201][0202][0203]
表25.指数扩增热循环
[0204][0205]
指数扩增完成后,将所有孔的样品收集混合,然后根据商业试剂盒minelute pcr purification kit(qiagen cat.no./id:28006)的操作流程进行纯化,最后用50μl depc h2o洗脱。洗脱后加入100μl(2x)spriselect beads(beckman coulter)根据操作流程进行片段纯化。纯化产物中,转录组文库已被生物素标记,染色质开放位点文库则不带有生物素。产物中,来自于同一个细胞的染色质开放位点序列及转录组序列将含有相同的细胞编
码组合。
[0206]
纯化后的产物采用(dynabead
tm myone
tm
链霉素亲和素c1)生物素链霉素亲和素磁珠结合,室温孵育1小时,并置于磁力架上进行磁力吸附,以分离染色质开放位点文库及转录组文库,其中磁珠将结合转录组文库,上清将包含染色质开放位点文库。获得的上清用qiagen minelute pcr purification kit进行纯化并洗脱于23μl depc h2o中,用于制备染色质开放位点终文库(可以进行测序)的构建。磁珠在先后用50μl 1x bw-t(表27),50μl 1x bw,50μl depc h2o洗涤后,用21μl depc h2o重悬,配制pcr反应体系并利用pcr反应进行转录组文库释放。
[0207]
表26. 1x bw体系
[0208][0209][0210]
表27. 1x bw-t体系
[0211]
试剂体积(μl)1x bw100010%tween-205
[0212]
表28.转录组文库释放体系
[0213][0214]
表29.转录组文库释放热循环
[0215][0216]
完成转录组文库释放反应后利用磁力架将磁珠与产物体系分离,取得产物体系(上清),并利用pcr反应进行扩增。扩增完成后,用0.6x spriselect beads浓度进行文库纯化,最后用适当体积(如20μl)depc h2o洗脱,备用。后续可用于转录组终文库的构建及特殊序列文库的富集及构建。
[0217]
若转录组文库中存在特殊序列文库,可模拟初始文库扩增的方法用相应的引物进行mrna逆转录文库及特殊序列文库的富集与分离(特殊序列文库可类似于染色质开放位点文库),并构建终文库以进行测序。
[0218]
表30.转录组文库再扩增热循环
[0219][0220]
7-1染色质开放位点终文库的构建
[0221]
在第6节中获得的染色质开放位点文库通过如下体系及反应进行染色质开放位点终文库的构建。构建完成后采用0.5x-0.9x spriselect beads双端筛选的模式纯化染色质开放位点终文库,并洗脱于20μl depc h2o。至此,染色质开放位点文库制备完成,可以进行测序。
[0222]
表31.染色质开放位点终文库构建体系
[0223][0224]
表31中10μm终文库引物1可以根据需要设计包含编码。
[0225]
表32.染色质开放位点终文库构建热循环
[0226][0227]
7-2转录组终文库的构建(若含有特殊序列,在分离出特殊序列文库后可依据此方法进行特殊序列文库终文库的构建)
[0228]
在第6节中获得的转录组文库用配制转录组切割体系,并于55℃恒温环境中静置5min。
[0229]
表33.转录组切割体系
[0230]
试剂体积(μl)转录组文库20ng相应体积(v)4x td缓冲液12.5depc h2o36.5-vtn5(相同转座复合体序列)1总计50
[0231]
切割完成后,在切割体系中加入1μl 10%sds进行终止反应,室温孵育5min。孵育完成后,用0.6x spriselect beads进行产物纯化,并用23μl depc h2o洗脱。洗脱后的产物配制转录组终文库反应体系并进行转录组终文库制备反应。
[0232]
表34.转录组终文库构建体系
[0233]
[0234][0235]
表34中10μm终文库引物1可以根据需要设计包含编码。
[0236]
表35.转录组终文库构建热循环
[0237][0238]
转录组终文库反应完成后,用0.6x spriselect beads进行产物纯化,并用20μl depc h2o洗脱。至此转录组终文库制备完成,文库包含的是转录组5’信息,可以进行测序。
[0239]
实验结果
[0240]
图8为根据本发明实施例所获得的细胞编码效果图。在一次实施中,输入5万细胞可获得7138个细胞的转录组数据,并可获得6665个细胞的染色质开放位点数据。两种组学总共可于6378个细胞同时获得转录组及染色质开放位点数据,同时可接受的双细胞率(~5%)。
[0241]
对获得的转录组数据进行分布比对,结果表明获得的转录组序列主要分布在mrna的5’端,证明本发明的转录组数据可以保留mrna 5’信息(以k562为例)(见图9)。
[0242]
通过相关性分析,发现细胞系的特征基因的染色质开放位点信息及转录组信息可以获得很好的相关性(以k562为例,malat1为常表达管家基因,gata1为k562特征基因)(见图10)。
[0243]
以上所述的具体实施例,对本发明的目的、技术方案和有益效果进行了进一步详细说明,应理解的是,以上所述仅为本发明的具体实施例而已,并不用于限制本发明,凡在本发明的精神和原则之内,所做的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
[0244]
参考文献
[0245]
1cao,j.et al.joint profiling of chromatin accessibility and gene expression in thousands of single cells.science 361,1380-1385,doi:10.1126/
science.aau0730(2018).
[0246]
2liu,l.et al.deconvolution of single-cell multi-omics layers reveals regulatory heterogeneity.nature communications 10,470,doi:10.1038/s41467-018-08205-7(2019).
[0247]
3chen,s.,lake,b.b.&zhang,k.high-throughput sequencing of the transcriptome and chromatin accessibility in the same cell.nature biotechnology 37,1452-1457,doi:10.1038/s41587-019-0290-0(2019).
[0248]
4zhu,c.et al.an ultra high-throughput method for single-cell joint analysis of open chromatin and transcriptome.nature structural&molecular biology 26,1063-1070,doi:10.1038/s41594-019-0323-x(2019).
[0249]
5ma,s.et al.chromatin potential identified by shared single cell profiling of rna and chromatin.biorxiv,2020.2006.2017.156943,doi:10.1101/2020.06.17.156943(2020).
[0250]
6xu,w.et al.issaac-seq enables sensitive and flexible multimodal profiling of chromatin accessibility and gene expression in single cells.biorxiv,2022.2001.2016.476488,doi:10.1101/2022.01.16.476488(2022).
再多了解一些
本文用于创业者技术爱好者查询,仅供学习研究,如用于商业用途,请联系技术所有人。