珠蛋白基因hba1、hba2基因)以及hba基因下游1-2mb范围内紧密连锁的单核苷酸多态性(snp)位点的引物组成(65对),合计86对引物。本发明还提供了检测人类胚胎α-地中海贫血基因突变的方法,使用了上述86对引物组合,并基于高通量测序平台进行检测。
6.α-地中海贫血基因突变检测的多重pcr扩增引物依次为:sea:1~86,具体引物对如下:
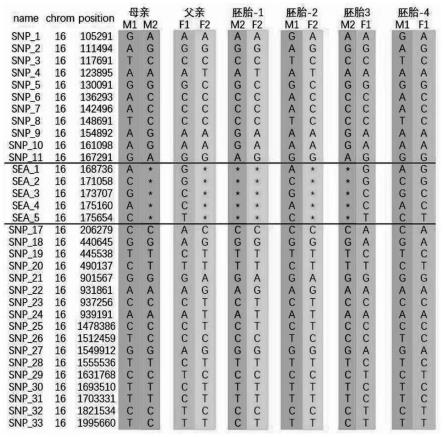
7.snp位点信息如下:
8.引物条件如下:
9.为了实现本发明,我们采用了以下技术和方法:
10.本发明样本的前处理采用了单细胞全基因组扩增技术,可以将微量细胞的基因组进行扩增放大,使其达到能够检测的量。首先将特殊的随机引物跟基因组随机结合、延伸,
这些引物末端带有一段特殊的通用序列,可以使扩增子的尾端互补,从而自身成环,防止dna过度拷贝,扩增产物无法作为新模板,从而很大程度上防止dna复制后的的指数性扩增,增加了全基因组扩增的均一性;预扩增的产物用一对引物进行指数扩增,增加扩增产物产量。建库时,将扩增后的全基因组产物进行末端补平添加a接头,再加上测序通用的接头以及测序引物接头等,即为构建好的测序文库,可用于上机测序。上机测序前,采用乳液pcr技术对文库进行油包水扩增,产生测序用模板文库,再通过对阳性模板进行富集以达到上机测序的总量要求,最后基于半导体测序技术进行测序。
11.靶向捕获方法:多重pcr捕获测序是一种针对临床及确定疾病基因热点的快速检测模式,可以在保证扩增均一性的前提下,对snp、indel等几千甚至上万个位点进行快速靶向线性扩增后,使用测序平台进行大批量样本检测与深度分析的解决方案。发明了一种用于检测α-地中海贫血基因突变的引物组合物,其由特异性扩增中国人群α-地贫基因的sea、4.2kb、3.7kb这三种最常见缺失型突变(20对)以及ws、qs、cs三个最常见点突变的引物(1对),以及特异扩增alpha缺失区域连锁snp位点区域(包括α-珠蛋白基因hba1、hba2基因)以及hba基因下游1-2mb范围内紧密连锁的单核苷酸多态性(snp)位点的引物组成(65对),合计86对引物。引物的筛选标准为:不同缺失类型对应的特异性区域扩增结果,同时对应区域的snp分析可以直接判定胚胎的型别;连锁snp位点筛选标注:genomad,east asain maf(0.2,0.8),gc含量0.3-0.6,距离连锁分析检测位点上下游1-2m区间。
12.高通量测序方法:主要包括使用设计好的多重引物panel,可识别的特异性条形码和dna文库;对构建成功的文库进行上机前质检,将质检通过的文库转均一化后,进行模板制备及模板富集,进行模板的扩增,最终阳性模板在ion torrent测序平台进行测序实验。
13.生物信息分析方法:
14.1、下机数据分析方法:将制作好的测序文库体系在ion torrent proton平台中进行高通量测序,测序结束后,采用torrent_server_5.0_vm软件对生成的原始测序下机数据进行预处理,去除了下机数据中的接头序列。并用tmap比对工具将处理过后的测序数据比对到grch37(hg19)人类参考基因组上。
15.2、地贫基因基因型检测方法:通过torrent suite(v5.4.0)分析套件中的“coverage analysis和“variant caller”等插件对预处理后的测序数据进行基因型分析,分析目标区域chr16:105290-2154894中每个基因位点的单体型多态性位点(snp)的覆盖深度和基因型情况。对于突变阳性的样本,可以通过查看variant caller插件生成的表格,根据表格中的突变频率进一步判断该位点是杂合型突变还是纯合型突变(突变频率在40~60%范围即为杂合型突变,90-100%则是纯合型突变)。利用父母和先验者与α-地贫的致病基因hba1紧密连锁snp在染色体上的位置和基因型构建单体型。通过已经建好的单体型对胚胎是否致病进行判断。
16.3、靶向区域拷贝数分析方法:使用已知的正常样本构建基准数据库,计算每个样本sea缺失区染色体的相对序列值(rc),并利用不同样本的平均值,通过统计分析,确定了参考值的范围。接下来,通过利用参考值对每个样本进行缺失判定。
17.4、缺失区域snp位点判断胚胎致病性方法:在sea缺失区域选择与父母有区别的snp位点,根据孟德尔遗传规律,判断其后代的致病性。
18.本发明的有益技术效果在于:无需对目标区域进行全部扩增,例如缺失型区域内
选取特异性snp位点进行扩增,不同缺失类型对应的特异性区域扩增结果,同时对应区域的snp分析可以直接判定胚胎的缺失类型。同时在先证者缺失的情况下,直接以胚胎的结果作为先证者进行分析(缺失特异性区间的分析可以直接判定胚胎的表型);sea上下游区域会有一定概率的同源重组风险,胚胎的直接判定结果可以作为连锁分析结果的验证。
附图说明
19.图1为snp单体型技术原理图。
20.图2为sea区域覆盖拷贝数,说明该样本为纯合缺失。
21.图3为sea区域覆盖拷贝数。
22.图4为sea区域覆盖拷贝数。
23.图5为sea区域覆盖拷贝数。
24.图6为单体型结果,通过颜色可以区分,m2,f2为缺失链。
具体实施方式
25.下面将结合本发明的附图,对本发明实施例中的技术方案进行清楚、完整地描述。
26.实施例一:4例胚胎α-地贫胚胎移植前检测,基于ion torrent测序平台
27.(一)主要实验步骤:
28.(1)外周血dna提取
29.本实施例中,获取2例α-地贫
‑‑
sea/αα父母的样本,夫妻双方样本来自外周血样本,采用magpure fast blood dna lq kit提取试剂盒,按照试剂盒说明书操作步骤提取。
30.(2)全基因组扩增
31.本实施例中,获取此家系的4枚胚胎活检样本,胚胎细胞样本按照fapon single cell geneme-ampli kit标准操作流程对4个胚胎样本进行全基因组扩增。
32.(3)对步骤(1)(2)获得的样本dna进行qubit 4.0浓度测定。
33.本实施例中,dna样本采用dsdna hs assay kit试剂盒进行浓度测定,按照试剂盒说明书操作步骤进行检测。
34.(4)多重pcr扩增:定量后的产物按照多重扩增体系依照以下体系加入试剂进行多重pcr扩增:试剂(reagent)volume per reaction(μl)1μlwga模板或30ngdn模板1μl/30ng5xhyper buffer2multiprimer dna polymerase0.25多重引物mix0.34nuclease-free water6.41体系总体积10
35.反应条件:
36.多重扩增产物质检:取1.5μl的扩增产物进行电泳质检,琼脂糖凝胶浓度(2%),确认目标片段扩增成功。
37.纯化扩增产物:磁珠纯化扩增产物,本步骤采用dna clean beads kit,样本:磁珠=1:1.2;
38.纯化好的pcr产物,用qubit4.0测定浓度
39.(5)文库构建:本步骤采用的文库构建试剂盒为fapon two-step library prep kit for ion torrent
40.a末端补平
41.末端补平反应体系为50μl体系,反应体系的组成成分具体为:试剂(reagent)volume per reaction(μl)dna/》50ngxend repair buffer10end repair enzyme mix5nuclease-free-water35-xtotal50
42.b接头连接
43.本步骤中使用的p1接头为:ccactacgcctccgctttcctctctatgggcagtcggtgatatcaccgactgcccatagagaggaaagcggaggcgtagtgg-s-t-s-t
44.本步骤中使用的adapter接头(为一段已知的短核苷酸序列,用于链接未知的目标测序片段)是由如下正反两个序列组成的96个barcode x,barcode x代表的具体序列如下:
45.接头连接反应体系为100μl体系,反应体系的组成成分具体为:试剂(reagent)volume per reaction(μl)
末端补平产物50ligation buffer34ligation enzyme mix6p11nuclease-free-water8adapter1total100
46.接头连接pcr反应条件如下:20℃15min;4℃hold
47.c接头连接产物纯化
48.本步骤采用dna clean beads kit,样本:磁珠=1:0.9;获得25μl接头连接的文库片段。
49.d文库片段pcr扩增
50.此中文库扩增引物序列如下:primer-1 ccactacgcctccgctttcctctctatgprimer-2 ccatctcatccctgcgtgtc
51.扩增反应体系为50μl体系,反应体系的组成成分具体为:试剂(reagent)volume per reaction(μl)纯化后连接产物502
×
hifi seq pcr mix34pcr primer mix6total90
52.pcr扩增反应条件如下:72℃10min;98℃45s,(98℃20s,65℃30s,72℃30s)6个循环;72℃5min;4℃hold
53.e扩增产物纯化
54.本步骤采用dna clean beads kit,样本:磁珠=1:1;获得25μl接头连接的文库片段。
55.本步骤获得的dna文库采用1
×
dsdna hs assay kit试剂盒进行浓度测定,按照试剂盒说明书操作步骤进行检测。
56.(6)模板制备:将文库产物均一化,混合至100pm
57.1)将ng/ul浓度换算成nm浓度,如下:(concentration in ng/μl)
×
106÷
(660g/mol
×
average library size)=concentration in nm
58.2)根据以下公式进行取样
59.volume of samples=v(f)*c(f)/#*c(i)
60.注:其中,
61.v(f):混合后的样本终体积;
62.c(f):混合后的所有样本的终浓度;
63.#:混合mix的所有样本数目;
64.c(i):每一个样本的初始浓度(nm);
65.最终用eb补足终体积。
66.按照ion p1 hi-q ot2 200试剂盒的标准操作
67.说明进行模板制备和模板富集。
68.(7)上机测序:按照ion p1 hi-q测序200试剂盒标准操作说明进行上机测序。
69.(二)数据分析:使用已知的正常样本构建基准数据库,计算每个样本sea缺失区染色体的相对序列值(rc),并利用不同样本的平均值,通过统计分析,确定了参考值的范围。接下来,通过利用参考值对每个样本进行缺失判定。
70.(三)单体型分析
71.(1)样本检测结果显示,见表1,2
72.(2)样本拷贝数分析结果见图2,3,4,5
73.(3)样本单体型结果见图6
74.表1样本检测结果显示
75.表2:缺失区snp位点结果显示
76.以上所述实施例仅表达了本发明的部分实施方式,其描述较为具体和详细,但并不能因此而理解为对本发明保护范围的限制。应当指出的是,对于本领域的普通技术人员来说,在不脱离本专利构思的前提下,还可以做出若干变形和改进,这些都属于本发明的保护范围。因此,本发明的保护范围应以权利要求书为准。
再多了解一些
本文用于创业者技术爱好者查询,仅供学习研究,如用于商业用途,请联系技术所有人。