用于分析复杂的基因组区域的方法和系统
交叉引用
1.本技术要求于2019年10月7日提交的美国临时申请第62/911,846号的权益,该申请的全部内容通过引用并入本文。
背景技术:
2.由于遗传变异会影响对药物的反应,因此药物遗传学(pgx)代表了能够对药物反应进行个体化确定的精准医学的一个组成部分。pgx的益处包括降低药物不良反应(sadr)的成本和风险,以及提高药物疗效。虽然目前测试了大量的pgx基因,但细胞色素p450 2d6(cyp2d6)具有巨大的诊断价值,因为高达25%的药物被cyp2d6激活或代谢。这些药物包括抗癌药物、阿片受体激动剂以及几种抗抑郁药和抗焦虑药。cyp2d6酶由cyp2d6基因编码,遗传变异可导致酶功能降低或完全丧失。cyp2d6主要在肝脏中表达,是肝脏药物代谢和清除的主要贡献者。正确诊断cyp2d6遗传变异的问题会直接影响sadr发展的风险。nih临床药物遗传学实施联盟(clinical pharmacogenetics implementation consortium,cpic)目前列出了58种与支持cyp2d6临床测试的证据相关的药物,从而使其成为顶级基因之一。仅在美国,在2019年cyp2d6测试的市场规模评估为5.22亿美元,每年增长率为6-8%。
3.目前,在cyp2d6中有超过100种描述的药物遗传学相关改变(也称为*星等位基因单体型),包括频繁的拷贝数变异。此外,与相邻的、高度同源的(高达94%相同)假基因(cyp2d7和cyp2d8)的基因融合和杂交使变体识别复杂化。在美国,~13%的人携带cyp2d6结构变体,这些变体占与该基因相关的所有变体的7%。这些特征使当前测试平台的遗传分析变得复杂,并且无法准确分析许多稀有或更复杂的单体型。许多小组的工作表明,目前使用的商业基因分型平台容易错误地表征cyp2d6。这会导致不正确的分配,从而导致不正确的剂量推荐。当基于短读段(ngs)或模板长度(sanger测序)时,基因测序同样受到阻碍。虽然已经开发了许多结合被靶向的扩增、拷贝数分析和长程pcr来更精确地确定完整结构的方法,但由于复杂的工作流程、时间要求和总体成本,这些方法不适合常规的临床测试。
技术实现要素:
4.对用于准确和经济有效地分析复杂的基因组区域的改进方法和系统的需求尚未满足。本公开满足了这种未满足的需求。
5.在一个方面,提供了一种分析感兴趣的基因组区域的方法,该方法包括:a)使包含所述感兴趣的基因组区域的基因组dna与规律间隔性成簇短回文重复序列(crispr)相关核酸内切酶和两种或更多种grna接触,从而产生切除的感兴趣的基因组区域;b)分离包含感兴趣的基因组区域的基因组dna;以及c)分析切除的感兴趣的基因组区域,其中该方法不涉及dna扩增。在一些情况下,分析包括对切除的感兴趣的基因组区域进行测序。在一些情况下,分析包括对切除的感兴趣的基因组区域进行基因分型。在一些情况下,分析包括对切除的感兴趣的区域进行结构分析。在一些情况下,在a)的接触之前进行b)的分离。在一些情况下,在a)的接触之后进行b)的分离。在一些情况下,两种或更多种grna各自包含与基因组
dna中存在的不同核苷酸序列基本上互补的核苷酸序列。在一些情况下,不同核苷酸序列位于感兴趣的基因组区域的侧翼。在一些情况下,crispr相关核酸内切酶在位于感兴趣的基因组区域侧翼的基因组位点处切割感兴趣的基因组区域。在一些情况下,crispr相关核酸内切酶是1类或2类crispr相关核酸内切酶。在一些情况下,1类crispr相关核酸内切酶选自:cas3、cas5、cas8a、cas8b、cas8c、cas10d、cse1、cse2、csy1、csy2、csy3、gsu0054、cas10、csm2、cmr5、csx11、csx10和csf1。在一些情况下,2类crispr相关核酸内切酶选自:cas9、cas12a、csn2、cas4、cas12b、cas12c、cas13a、cas13b、cas13c和cas13d。在一些情况下,crispr相关核酸内切酶包含与野生型crispr相关核酸内切酶具有至少80%的序列同一性的氨基酸序列。在一些情况下,crispr相关核酸内切酶是cas9或其变体。在一些情况下,cas9是酿脓链球菌(streptococcus pyogenes)cas9(spcas9)。在一些情况下,cas9变体相对于野生型酿脓链球菌cas9(spcas9)包含选自r780a、k810a、k848a、k855a、h982a、k1003a、r1060a、d1135e、n497a、r661a、q695a、q926a、l169a、y450a、m495a、m694a和m698a的一个或多个点突变。在一些情况下,在a)之前基因组dna没有被片段化、消化或剪切。在一些情况下,在a)之前基因组dna没有经限制酶消化。在一些情况下,感兴趣的基因组区域是复杂的基因组区域。在一些情况下,复杂的基因组区域包括基因及其一种或多种假基因。在一些情况下,一种或多种假基因包含与基因具有至少75%的序列同一性的核苷酸序列。在一些情况下,复杂的基因组区域包含一个或多个重复区域、一个或多个重复段、一个或多个插入、一个或多个倒位、一个或多个串联重复序列、一个或多个逆转录转座子或其任意组合。在一些情况下,感兴趣的基因组区域是高度多态的遗传基因座。在一些情况下,切除的感兴趣的基因组区域的长度为至少1万个碱基。在一些情况下,切除的感兴趣的基因组区域的长度长达25万个碱基。在一些情况下,分离包括分离高分子量dna。在一些情况下,高分子量dna的长度为至少5万个碱基。在一些情况下,测序包括长读段测序。在一些情况下,长读段测序包括单分子实时测序或纳米孔测序。在一些情况下,该方法还包括将一个或多个测序接头连接到切除的感兴趣的基因组区域的一端或两端。在一些情况下,该方法还包括,在a)之前,使基因组dna去磷酸化。在一些情况下,去磷酸化包括用磷酸酶处理基因组dna。在一些情况下,磷酸酶是虾碱性磷酸酶。在一些情况下,该方法还包括在去磷酸化之后,用末端转移酶(tdt)处理基因组dna。在一些情况下,该方法还包括对切除的感兴趣的基因组区域进行末端加尾。在一些情况下,末端加尾包括将一个或多个腺苷核苷酸添加到切除的感兴趣的基因组区域的游离3'末端。在一些情况下,该方法不涉及聚合酶链式反应(pcr)或等温扩增中的任何一种。在一些情况下,该方法不涉及多重置换扩增(mda)、链置换扩增(sda)、基于核酸序列的扩增(nasba)、环介导的等温扩增、滚环扩增(rca)、连接酶链式反应(lcr)、解旋酶依赖性扩增或网状分枝扩增法中的任何一种。在一些情况下,在生物样品中提供基因组dna。在一些情况下,生物样品包括体液(例如,血液(例如,全血、血浆、血清)、尿液、唾液、骨髓、脊髓液、痰、腹水、淋巴液、胸膜液、羊水、精液、阴道分泌物、汗液、粪便、腺体分泌物、眼液、母乳)或固体组织样品。在一些情况下,生物样品是诊断样品。
6.在另一个方面,提供了一种分析长度为至少1万个碱基的感兴趣的复杂的基因组区域的方法,该方法包括:a)提供包含感兴趣的复杂的基因组区域的基因组dna;b)分离包含感兴趣的复杂的基因组区域的高分子量dna;c)使基因组dna与规律间隔性成簇短回文重复序列(crispr)相关核酸内切酶和两种或更多种grna接触以切除感兴趣的复杂的基因组
区域,其中两种或更多种grna各自包含与基因组dna中存在的不同核苷酸序列基本上互补的核苷酸序列,并且其中不同核苷酸序列位于感兴趣的复杂的基因组区域的侧翼;以及d)分析感兴趣的复杂的基因组区域,其中该方法不涉及dna扩增。在一些情况下,分析包括对感兴趣的复杂的基因组区域进行测序。在一些情况下,测序包括长读段测序。在一些情况下,长读段测序包括单分子实时测序或纳米孔测序。在一些情况下,分析包括对感兴趣的复杂的基因组区域进行基因分型。在一些情况下,分析包括对感兴趣的基因组区域进行结构分析。在一些情况下,在(c)的接触之前进行(b)的分离。在一些情况下,在(c)的接触之后进行(b)的分离。在一些情况下,高分子量dna的长度为至少1万个碱基。在一些情况下,感兴趣的复杂的基因组区域包含靶基因及其一种或多种假基因。在一些情况下,一种或多种假基因与靶基因具有至少75%的序列同一性。在一些情况下,感兴趣的复杂的基因组区域包含cyp2d6、cyp2d7和cyp2d8。在一些情况下,感兴趣的复杂的基因组区域包含cyp2c8、cyp2c9、cyp2c18和cyp2c19。在一些情况下,感兴趣的复杂的基因组区域包含一个或多个重复区域、一个或多个重复段、一个或多个插入、一个或多个倒位、一个或多个串联重复序列、一个或多个逆转录转座子或其任意组合。在一些情况下,感兴趣的复杂的基因组区域是高度多态的遗传基因座。在一些情况下,crispr相关核酸内切酶是1类或2类crispr相关核酸内切酶。在一些情况下,1类crispr相关核酸内切酶选自:cas3、cas5、cas8a、cas8b、cas8c、cas10d、cse1、cse2、csy1、csy2、csy3、gsu0054、cas10、csm2、cmr5、csx11、csx10和csf1。在一些情况下,2类crispr相关核酸内切酶选自:cas9、cas12a、csn2、cas4、cas12b、cas12c、cas13a、cas13b、cas13c和cas13d。在一些情况下,crispr相关核酸内切酶包含与野生型crispr相关核酸内切酶具有至少80%的序列同一性的氨基酸序列。在一些情况下,crispr相关核酸内切酶是cas9或其变体。在一些情况下,cas9是酿脓链球菌cas9(spcas9)。在一些情况下,cas9变体相对于野生型酿脓链球菌cas9(spcas9)包含选自r780a、k810a、k848a、k855a、h982a、k1003a、r1060a、d1135e、n497a、r661a、q695a、q926a、l169a、y450a、m495a、m694a和m698a的一个或多个点突变。在一些情况下,在a)之前基因组dna没有被片段化或消化。在一些情况下,在a)之前基因组dna没有经限制酶消化。在一些情况下,感兴趣的复杂的基因组区域的长度长达25万个碱基。在一些情况下,该方法还包括将一个或多个测序接头连接到切除的感兴趣的基因组区域的一端或两端。在一些情况下,该方法不涉及聚合酶链式反应(pcr)或等温扩增中的任何一种。在一些情况下,该方法不涉及多重置换扩增(mda)、链置换扩增(sda)、基于核酸序列的扩增(nasba)、环介导的等温扩增、滚环扩增(rca)、连接酶链式反应(lcr)、解旋酶依赖性扩增或网状分枝扩增法中的任何一种。在一些情况下,在生物样品中提供基因组dna。在一些情况下,生物样品是体液(例如,血液(例如,全血、血浆、血清)、尿液、唾液、骨髓、脊髓液、痰、腹水、淋巴液、胸膜液、羊水、精液、阴道分泌物、汗液、粪便、腺体分泌物、眼液、母乳)或固体组织样品。在一些情况下,生物样品是诊断样品。
7.在另一个方面,提供了一种分析包含cyp2d6、cyp2d7和cyp2d8的遗传基因座的方法,该方法包括:a)提供包含遗传基因座的基因组dna;b)使基因组dna与规律间隔性成簇短回文重复序列(crispr)相关核酸内切酶和两种或更多种grna接触以从基因组dna切除遗传基因座,其中两种或更多种grna各自包含与基因组dna中存在的不同核苷酸序列基本上互补的核苷酸序列,并且其中不同核苷酸序列位于包含cyp2d6、cyp2d7和cyp2d8的遗传基因座的侧翼;以及c)分析遗传基因座。在一些情况下,分析包括对遗传基因座进行测序。在一
些情况下,测序包括长读段测序。在一些情况下,长读段测序包括单分子实时测序或纳米孔测序。在一些情况下,分析包括对遗传基因座进行基因分型。在一些情况下,分析包括对遗传基因座进行结构分析。在一些情况下,该方法还包括,在c)之前,分离包含遗传基因座的高分子量dna。在一些情况下,高分子量dna的长度为至少1万个碱基。在一些情况下,两种或更多种grna包含选自seq id no:1-26的核苷酸序列。在一些情况下,遗传基因座的长度为至少4万个碱基。在一些情况下,crispr相关核酸内切酶是1类或2类crispr相关核酸内切酶。在一些情况下,1类crispr相关核酸内切酶选自:cas3、cas5、cas8a、cas8b、cas8c、cas10d、cse1、cse2、csy1、csy2、csy3、gsu0054、cas10、csm2、cmr5、csx11、csx10和csf1。在一些情况下,2类crispr相关核酸内切酶选自:cas9、cas12a、csn2、cas4、cas12b、cas12c、cas13a、cas13b、cas13c和cas13d。在一些情况下,crispr相关核酸内切酶包含与野生型crispr相关核酸内切酶具有至少80%的序列同一性的氨基酸序列。在一些情况下,crispr相关核酸内切酶是cas9或其变体。在一些情况下,cas9是酿脓链球菌cas9(spcas9)。在一些情况下,cas9变体相对于野生型酿脓链球菌cas9(spcas9)包含选自r780a、k810a、k848a、k855a、h982a、k1003a、r1060a、d1135e、n497a、r661a、q695a、q926a、l169a、y450a、m495a、m694a和m698a的一个或多个点突变。在一些情况下,在a)之前基因组dna没有被片段化、消化或剪切。在一些情况下,在a)之前基因组dna没有经限制酶消化。在一些情况下,该方法还包括将一个或多个测序接头连接到切除的遗传基因座的一端或两端。在一些情况下,该方法不涉及dna扩增。在一些情况下,该方法不涉及聚合酶链式反应(pcr)或等温扩增中的任何一种。在一些情况下,该方法不涉及多重置换扩增(mda)、链置换扩增(sda)、基于核酸序列的扩增(nasba)、环介导的等温扩增、滚环扩增(rca)、连接酶链式反应(lcr)、解旋酶依赖性扩增或网状分枝扩增法中的任何一种。在一些情况下,在生物样品中提供基因组dna。在一些情况下,生物样品是体液(例如,血液(例如,全血、血浆、血清)、尿液、唾液、骨髓、脊髓液、痰、腹水、淋巴液、胸膜液、羊水、精液、阴道分泌物、汗液、粪便、腺体分泌物、眼液、母乳)或固体组织样品。在一些情况下,生物样品是诊断样品。
8.在又一个方面,提供了一种鉴定对象中cyp2d6遗传变异的方法,该方法包括:a)提供包含从对象中获得的基因组dna的生物样品;b)使基因组dna与规律间隔性成簇短回文重复序列(crispr)相关核酸内切酶和两种或更多种grna接触以切除包含cyp2d6、cyp2d7和cyp2d8的遗传基因座;c)对遗传基因座进行长读段测序;以及d)鉴定对象的cyp2d6中的一种或多种遗传变异。在一些情况下,该方法还包括,基于遗传变异将对象鉴定为具有cyp2d6功能的降低、丧失或增加。在一些情况下,该方法还包括基于鉴定向对象推荐治疗或替代治疗。在一些情况下,当对象被鉴定为具有cyp2d6功能的降低、丧失或增加时,该方法还包括向对象推荐替代治疗。在一些情况下,该方法还包括基于鉴定向对象推荐治疗剂的剂量。在一些情况下,当对象被鉴定为具有cyp2d6功能的降低、丧失或增加时,该方法还包括改变治疗剂的剂量。在一些情况下,该方法还包括,在c)之前,分离包含遗传基因座的高分子量dna。在一些情况下,高分子量dna的长度为至少4万个碱基。在一些情况下,两种或更多种grna各自包含与基因组dna中存在的不同核苷酸序列基本上互补的核苷酸序列,并且其中不同核苷酸序列位于包含cyp2d6、cyp2d7和cyp2d8的遗传基因座的侧翼。在一些情况下,两种或更多种grna包含选自seq id no:1-26的核苷酸序列。在一些情况下,遗传基因座的长度为至少4万个碱基。在一些情况下,长读段测序包括单分子实时测序或纳米孔测序。在一
些情况下,crispr相关核酸内切酶是1类或2类crispr相关核酸内切酶。在一些情况下,1类crispr相关核酸内切酶选自:cas3、cas5、cas8a、cas8b、cas8c、cas10d、cse1、cse2、csy1、csy2、csy3、gsu0054、cas10、csm2、cmr5、csx11、csx10和csf1。在一些情况下,2类crispr相关核酸内切酶选自:cas9、cas12a、csn2、cas4、cas12b、cas12c、cas13a、cas13b、cas13c和cas13d。在一些情况下,crispr相关核酸内切酶包含与野生型crispr相关核酸内切酶具有至少80%的序列同一性的氨基酸序列。在一些情况下,crispr相关核酸内切酶是cas9或其变体。在一些情况下,cas9是酿脓链球菌cas9(spcas9)。在一些情况下,cas9变体相对于野生型酿脓链球菌cas9(spcas9)包含选自r780a、k810a、k848a、k855a、h982a、k1003a、r1060a、d1135e、n497a、r661a、q695a、q926a、l169a、y450a、m495a、m694a和m698a的一个或多个点突变。在一些情况下,在a)之前基因组dna没有被片段化、消化或剪切。在一些情况下,在a)之前基因组dna没有经限制酶消化。在一些情况下,该方法还包括将一个或多个测序接头连接到切除的感兴趣的基因组区域的一端或两端。在一些情况下,该方法不涉及dna扩增。在一些情况下,该方法不涉及聚合酶链式反应(pcr)或等温扩增中的任何一种。在一些情况下,该方法不涉及多重置换扩增(mda)、链置换扩增(sda)、基于核酸序列的扩增(nasba)、环介导的等温扩增、滚环扩增(rca)、连接酶链式反应(lcr)、解旋酶依赖性扩增或网状分枝扩增法中的任何一种。在一些情况下,生物样品是体液(例如,血液(例如,全血、血浆、血清)、尿液、唾液、骨髓、脊髓液、痰、腹水、淋巴液、胸膜液、羊水、精液、阴道分泌物、汗液、粪便、腺体分泌物、眼液、母乳)或固体组织样品。
9.在又一方面,提供了一种组合物,其包含:a)规律间隔性成簇短回文重复序列(crispr)相关核酸内切酶;b)第一指导rna(grna),其包含与基因组dna中存在的核苷酸序列基本上互补的核苷酸序列,该基因组dna位于包含cyp2d6、cyp2d7和cyp2d8的遗传基因座上游;c)第二指导rna(grna),其包含与基因组dna中存在的核苷酸序列基本上互补的核苷酸序列,该基因组dna位于包含cyp2d6、cyp2d7和cyp2d8的遗传基因座下游。在一些情况下,第一指导rna包含选自以下的核苷酸序列:seq id no:1、2或13-16。在一些情况下,第二指导rna包含选自以下的核苷酸序列:seq id no:3-12或17-26。在一些情况下,crispr相关核酸内切酶是1类或2类crispr相关核酸内切酶。在一些情况下,1类crispr相关核酸内切酶选自:cas3、cas5、cas8a、cas8b、cas8c、cas10d、cse1、cse2、csy1、csy2、csy3、gsu0054、cas10、csm2、cmr5、csx11、csx10和csf1。在一些情况下,2类crispr相关核酸内切酶选自:cas9、cas12a、csn2、cas4、cas12b、cas12c、cas13a、cas13b、cas13c和cas13d。在一些情况下,crispr相关核酸内切酶包含与野生型crispr相关核酸内切酶具有至少80%的序列同一性的氨基酸序列。在一些情况下,crispr相关核酸内切酶是cas9或其变体。在一些情况下,cas9是酿脓链球菌cas9(spcas9)。在一些情况下,cas9变体相对于野生型酿脓链球菌cas9(spcas9)包含选自r780a、k810a、k848a、k855a、h982a、k1003a、r1060a、d1135e、n497a、r661a、q695a、q926a、l169a、y450a、m495a、m694a和m698a的一个或多个点突变。
10.在又一方面,提供了一种用于对cyp2d6进行基因分型的试剂盒,其包含:a)规律间隔性成簇短回文重复序列(crispr)相关核酸内切酶;b)第一指导rna(grna),其包含与基因组dna中存在的核苷酸序列基本上互补的核苷酸序列,该基因组dna位于包含cyp2d6、cyp2d7和cyp2d8的遗传基因座上游;c)第二指导rna(grna),其包含与基因组dna中存在的核苷酸序列基本上互补的核苷酸序列,该基因组dna位于包含cyp2d6、cyp2d7和cyp2d8的遗
传基因座下游。在一些情况下,第一指导rna包含选自以下的核苷酸序列:seq id no:1、2或13-16。在一些情况下,第二指导rna包含选自以下的核苷酸序列:seq id no:3-12或17-26。在一些情况下,crispr相关核酸内切酶是1类或2类crispr相关核酸内切酶。在一些情况下,1类crispr相关核酸内切酶选自:cas3、cas5、cas8a、cas8b、cas8c、cas10d、cse1、cse2、csy1、csy2、csy3、gsu0054、cas10、csm2、cmr5、csx11、csx10和csf1。在一些情况下,2类crispr相关核酸内切酶选自:cas9、cas12a、csn2、cas4、cas12b、cas12c、cas13a、cas13b、cas13c和cas13d。在一些情况下,crispr相关核酸内切酶包含与野生型crispr相关核酸内切酶具有至少80%的序列同一性的氨基酸序列。在一些情况下,crispr相关核酸内切酶是cas9或其变体。在一些情况下,cas9是酿脓链球菌cas9(spcas9)。在一些情况下,cas9变体相对于野生型酿脓链球菌cas9(spcas9)包含选自r780a、k810a、k848a、k855a、h982a、k1003a、r1060a、d1135e、n497a、r661a、q695a、q926a、l169a、y450a、m495a、m694a和m698a的一个或多个点突变。
11.在又一方面,提供了一种用于分析感兴趣的复杂的基因组区域的系统,该系统包括:(a)至少一个存储单元,其被配置为接收数据输入,该数据输入包括由包括以下步骤的方法生成的数据:(i)从包含感兴趣的复杂的基因组区域的基因组dna中分离高分子量dna;(ii)使基因组dna与规律间隔性成簇短回文重复序列(crispr)相关核酸内切酶和两种或更多种grna接触以切除感兴趣的复杂的基因组区域,其中两种或更多种grna各自包含与基因组dna中存在的不同核苷酸序列基本上互补的核苷酸序列,并且其中不同核苷酸序列位于感兴趣的复杂的基因组区域的侧翼;以及(iii)分析感兴趣的复杂的基因组区域以产生数据,其中方法不涉及dna扩增;和(b)可操作地耦合到至少一个存储单元的计算机处理器,其中计算机处理器被编程为基于数据产生输出。在一些情况下,输出是报告。在一些情况下,输出是感兴趣的复杂的基因组区域的基因型。在一些情况下,输出是感兴趣的复杂的基因组区域的基因序列。在一些情况下,输出是感兴趣的复杂的基因组区域的结构分析。在一些情况下,分析包括对感兴趣的复杂的基因组区域进行基因分型。在一些情况下,分析包括对感兴趣的复杂的基因组区域进行结构分析。在一些情况下,分析包括对感兴趣的复杂的基因组区域进行测序。在一些情况下,测序包括长读段测序。在一些情况下,长读段测序包括单分子实时测序或纳米孔测序。在一些情况下,在(ii)的接触之前进行(i)的分离。在一些情况下,在(ii)的接触之后进行(i)的分离。在一些情况下,高分子量dna的长度为至少1万个碱基。在一些情况下,感兴趣的复杂的基因组区域包含靶基因及其一种或多种假基因。在一些情况下,一种或多种假基因与靶基因具有至少75%的序列同一性。在一些情况下,感兴趣的复杂的基因组区域包含cyp2d6、cyp2d7和cyp2d8。在一些情况下,感兴趣的复杂的基因组区域包含cyp2c8、cyp2c9、cyp2c18和cyp2c19。在一些情况下,感兴趣的复杂的基因组区域包含一个或多个重复区域、一个或多个重复段、一个或多个插入、一个或多个倒位、一个或多个串联重复序列、一个或多个逆转录转座子或其任意组合。在一些情况下,感兴趣的复杂的基因组区域是高度多态的遗传基因座。在一些情况下,crispr相关核酸内切酶是1类或2类crispr相关核酸内切酶。在一些情况下,1类crispr相关核酸内切酶选自:cas3、cas5、cas8a、cas8b、cas8c、cas10d、cse1、cse2、csy1、csy2、csy3、gsu0054、cas10、csm2、cmr5、csx11、csx10和csf1。在一些情况下,2类crispr相关核酸内切酶选自:cas9、cas12a、csn2、cas4、cas12b、cas12c、cas13a、cas13b、cas13c和cas13d。在一些情况下,crispr相关核酸内
切酶包含与野生型crispr相关核酸内切酶具有至少80%的序列同一性的氨基酸序列。在一些情况下,crispr相关核酸内切酶是cas9或其变体。在一些情况下,cas9是酿脓链球菌cas9(spcas9)。在一些情况下,cas9变体相对于野生型酿脓链球菌cas9(spcas9)包含选自r780a、k810a、k848a、k855a、h982a、k1003a、r1060a、d1135e、n497a、r661a、q695a、q926a、l169a、y450a、m495a、m694a和m698a的一个或多个点突变。在一些情况下,在a)之前基因组dna没有被片段化、消化或剪切。在一些情况下,在a)之前基因组dna没有经限制酶消化。在一些情况下,感兴趣的复杂的基因组区域的长度长达25万个碱基。在一些情况下,该系统还包括将一个或多个测序接头连接到切除的感兴趣的基因组区域的一端或两端。在一些情况下,该方法不涉及聚合酶链式反应(pcr)或等温扩增中的任何一种。在一些情况下,该方法不涉及多重置换扩增(mda)、链置换扩增(sda)、基于核酸序列的扩增(nasba)、环介导的等温扩增、滚环扩增(rca)、连接酶链式反应(lcr)、解旋酶依赖性扩增或网状分枝扩增法中的任何一种。在一些情况下,在生物样品中提供基因组dna。在一些情况下,生物样品包括体液(例如,血液(例如,全血、血浆、血清)、尿液、唾液、骨髓、脊髓液、痰、腹水、淋巴液、胸膜液、羊水、精液、阴道分泌物、汗液、粪便、腺体分泌物、眼液、母乳)或固体组织样品。在一些情况下,生物样品是诊断样品。
12.在又一方面,提供了一种用于鉴定对象的cyp2d6的遗传变异的系统,该系统包括:(a)至少一个存储单元,其被配置为接收数据输入,该数据输入包括由包括以下步骤的方法生成的测序数据:(ii)使获自对象的基因组dna与规律间隔性成簇短回文重复序列(crispr)相关核酸内切酶和两种或更多种grna接触,以切除包含cyp2d6、cyp2d7和cyp2d8的遗传基因座;以及(iii)对遗传基因座进行长读段测序以产生测序数据;以及(b)可操作地耦合到至少一个存储单元的计算机处理器,其中计算机处理器被编程为基于测序数据产生输出。在一些情况下,输出是报告。在一些情况下,输出鉴定cyp2d6中的遗传变异。在一些情况下,输出鉴定cyp2d6功能的降低、丧失或增加。在一些情况下,报告基于遗传变异向对象推荐治疗。在一些情况下,报告基于遗传变异向对象推荐治疗剂的剂量。在一些情况下,报告基于遗传变异推荐改变治疗剂的剂量。在一些情况下,治疗剂是由cyp2d6激活或代谢的治疗剂。在一些情况下,该方法还包括,在(ii)之前,分离包含遗传基因座的高分子量dna。在一些情况下,高分子量dna的长度为至少4万个碱基。在一些情况下,两种或更多种grna各自包含与基因组dna中存在的不同核苷酸序列基本上互补的核苷酸序列,并且其中不同核苷酸序列位于包含cyp2d6、cyp2d7和cyp2d8的遗传基因座的侧翼。在一些情况下,两种或更多种grna包含选自seq id no:1-26的核苷酸序列。在一些情况下,遗传基因座的长度为至少4万个碱基。在一些情况下,长读段测序包括单分子实时测序或纳米孔测序。在一些情况下,crispr相关核酸内切酶是1类或2类crispr相关核酸内切酶。在一些情况下,1类crispr相关核酸内切酶选自:cas3、cas5、cas8a、cas8b、cas8c、cas10d、cse1、cse2、csy1、csy2、csy3、gsu0054、cas10、csm2、cmr5、csx11、csx10和csf1。在一些情况下,2类crispr相关核酸内切酶选自:cas9、cas12a、csn2、cas4、cas12b、cas12c、cas13a、cas13b、cas13c和cas13d。在一些情况下,crispr相关核酸内切酶包含与野生型crispr相关核酸内切酶具有至少80%的序列同一性的氨基酸序列。在一些情况下,crispr相关核酸内切酶是cas9或其变体。在一些情况下,cas9是酿脓链球菌cas9(spcas9)。在一些情况下,cas9变体相对于野生型酿脓链球菌cas9(spcas9)包含选自r780a、k810a、k848a、k855a、h982a、k1003a、
r1060a、d1135e、n497a、r661a、q695a、q926a、l169a、y450a、m495a、m694a和m698a的一个或多个点突变。在一些情况下,在a)之前基因组dna没有被片段化、消化或剪切。在一些情况下,在a)之前基因组dna没有经限制酶消化。在一些情况下,该方法还包括将一个或多个测序接头连接到切除的感兴趣的基因组区域的一端或两端。在一些情况下,该方法不涉及dna扩增。在一些情况下,该方法不涉及聚合酶链式反应(pcr)或等温扩增中的任何一种。在一些情况下,该方法不涉及多重置换扩增(mda)、链置换扩增(sda)、基于核酸序列的扩增(nasba)、环介导的等温扩增、滚环扩增(rca)、连接酶链式反应(lcr)、解旋酶依赖性扩增或网状分枝扩增法中的任何一种。在一些情况下,生物样品是体液(例如,血液(例如,全血、血浆、血清)、尿液、唾液、骨髓、脊髓液、痰、腹水、淋巴液、胸膜液、羊水、精液、阴道分泌物、汗液、粪便、腺体分泌物、眼液、母乳)或固体组织样品。援引并入
13.本说明书中提及的所有出版物、专利和专利申请均以引用的方式并入本文,其程度如同每个单独的出版物、专利或专利申请被具体地且单独地指示以引用的方式并入。
附图说明
14.本公开的新颖特征在所附权利要求中特别阐述。通过参考以下阐述了其中利用了本公开的原理的说明性的实施方案的详细说明以及附图,将获得对本公开的特征和优点的更好理解,在附图中:
15.图1描绘了根据本文提供的实施方案的cyp2d6基因座。图a描述了包含cyp2d6基因的单个拷贝的参考遗传基因座相对于cyp2d7和cyp2d8的方向。结构变异的代表性实例说明cyp2d6基因拷贝数变异的复杂性,包括完全cyp2d6缺失(图b)、重复段(图c)和5'(图d)或3'(图e)cypd6/cypd7杂合等位基因的存在。这种排列中的重复基因通常具有包括1.6kb长的间隔序列的类似cyp2d7的下游区域。示出相对于参考序列(ng_008376.3)的5'-3'方向。
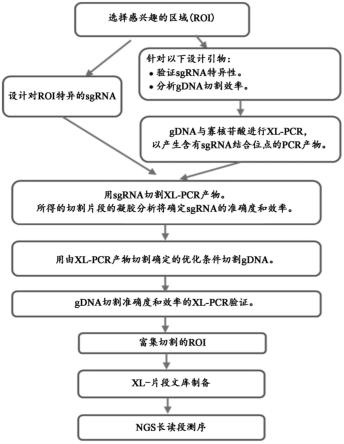
16.图2描绘了流程图的非限制性示例,该流程图描绘了根据本文提供的实施方案对cyp2d6基因座进行分离和测序的方法。
17.图3描绘了根据本文提供的实施方案比较基因组dna提取的非限制性示例。泳道a是使用改进的高分子量方案(》50kb)从淋巴母细胞系(lcl)细胞中提取的50ng gdna,泳道b是使用maxwell快速样品浓缩器(rapid sample concentrator,rsc)(~10-48kb)提取的50ng gdna),泳道c是50ng gdna对照(coriell;~10kb-50kb),泳道d是λ噬菌体dna(~50kda;neb)以及泳道e是hindiiiλ噬菌体消化物。
18.图4a和图4b描绘了根据本文提供的实施方案的靶向cyp2d6基因座的sgrna的设计和验证的非限制性示例。图4a描绘了捕获等位基因cyp2d6和杂合等位基因所需的crispr切割位点的示意图。图4b描绘了靶位点的crispr cut xl-pcr扩增子。样品a接收了不含sgrna的cas9,样品b接收了具有sgrna_1的cas9,而样品c接收了具有sgrna_2的cas9。
19.图5a和图5b描绘了根据本公开的实施方案的靶向基因组dna上的cyp2d6基因座的sgrna的效率的非限制性示例。图5a描绘了包含cyp2d6上游和下游区域的sgrna结合位点的xl-pcr产物的凝胶图像。泳道c是对照。图5b描绘了标准化为阴性对照的未切割gdna的百分比。*=p值《0.010。
20.图6描绘了根据本公开的实施方案的xl-pcr和基于ngs的分析方法的ngs比对的非
限制性示例。
21.图7a-图7c描绘了根据本公开的实施方案的关于cyp2d6基因座的备选crispr/cas9设计方法的问题的非限制性示例。切割位点用剪刀标出。x代表等位基因,其中a等位基因上所示的设计将在b-e等位基因排列上产生不需要的切割。
22.图8描绘了cyp2d6基因座的综合靶设计的非限制性示例。切割位点用剪刀标出。钩形符号代表等位基因,其中a等位基因上所示的设计将仅在b-e等位基因排列上产生中靶切割。
23.图9a-图9c描绘了靶向cyp2d6基因座的sgrna的设计和验证的非限制性示例。图9a描绘了靶向捕获等位基因cyp2d6和杂合等位基因的必要切割位点的示意图。图9b和图9c描绘了靶位点的crispr cut xl-pcr扩增子。样品a接收了不含sgrna的cas9,样品b接收了具有sgrna_1的cas9,而样品c接收了具有sgrna_2的cas9。
24.图10描绘了根据本公开的实施方案分离的高分子量dna的非限制性示例。从lcl细胞沉淀中提取的100ng高分子量的基因组dna的2%dna琼脂糖凝胶与来自coriell研究所的λ对照和预提取的dna进行比较。
25.图11a和图11b描绘了根据本文公开的实施方案的序列运行覆盖率的非限制性示例。
26.图12a和图12b描绘了根据本文公开的实施方案的非限制性示例序列比对大小。
27.图13描绘了根据本文公开的实施方案的比对图的非限制性示例。实现了被靶向的捕获区域的121x覆盖。方框框出了cyp2d6和cyp2d7。
28.图14描绘了根据本文公开的实施方案的显示sgrna特异性的sashimi图的非限制性示例。该图显示了两次测序运行的比对区域。红色比对显示了使用旨在捕获感兴趣的区域(roi)(chr22:42,122,115-41,161,320)的sgrna运行的序列数据。蓝色比对显示使用靶向相反链的sgrna对同一dna样本进行富集。
29.图15描绘了根据本文提供的实施方案的计算机系统的非限制性示例。
具体实施方式
30.本文公开了用于分析基因组感兴趣的区域(roi)(例如,来自基因组dna)的方法。感兴趣的区域可以是例如复杂的(例如高度复杂的)基因组区域。复杂的基因组区域可以包括例如高度多态区域、包含靶基因和与该靶基因具有高度序列同源性的一个或多个假基因的区域、包含一个或多个重复元件、一个或多个倒位、一个或多个插入、一个或多个重复段、一个或多个串联重复序列、一个或多个的逆转录转座子的区域等。本文提供的方法通常涉及使用规律间隔性成簇短回文重复序列(crispr)相关核酸内切酶和两种或更多种指导rna(grna)以从基因组dna中切除感兴趣的区域。本文提供的方法还涉及分析切除的感兴趣的区域(例如测序,例如通过长读段测序方法、基因分型、进行结构分析)。本文进一步提供了分析cyp2d6基因座(例如,包括靶基因cyp2d6和假基因cyp2d7和cyp2d8)的方法。有利地,在一些实施方案中,该方法不涉及使用dna扩增(例如,无扩增)。该方法可以提高对复杂的(例如,高度复杂)基因组区域进行测序的准确性(例如,降低测序错误率)(例如,与传统方法相比),和/或可以减少对复杂的(例如,高度复杂)基因组区域进行测序的时间(例如,与传统方法相比),和/或可以降低对复杂的(例如,高度复杂)基因组区域进行测序的成本(例如,
与传统方法相比)。本文另外提供了用于执行本文提供的方法的系统以及包含crispr相关核酸内切酶和靶向cyp2d6基因座的两种或更多种grna(例如,从基因组dna切除cyp2d6基因座)的组合物和试剂盒。
31.除非上下文另有明确规定,如本文和所附权利要求中使用的,单数形式“a(一个/种)”、“an(一个/种)”和“the(所述)”包括复数指示物。还应注意,可以撰写权利要求以排除任何任选元素。因此,该说明旨在用作使用与权利要求要素的叙述有关的“唯一”、“仅”等排他性术语或使用“否定”限制时的前提依据。
32.在本文中某些范围或数字以术语“约”为开头的数值呈现。在本文中术语“约”用于表示该术语所指的数字的
±
1%、2%、3%、4%或5%。如本文所用,术语“对象”和“个体”可互换使用并且可以是任何动物,包括哺乳动物(例如,人类或非人类动物)。
33.如本文所用,术语“cyp2d6”可以指cyp2d6基因或其任何结构变体或单基因拷贝变体。cyp2d6的结构变体可以包括基因融合、与邻近的高度同源的假基因(例如,cyp2d7和cyp2d8)的杂交、拷贝数变异(cnv)、基因重复和倍增、串联重复和重排。cyp2d6结构变体的一个实例是在cyp2d6外显子9中存在cyp2d7衍生序列(称为“外显子9转换”)。单基因拷贝变体可以包括单核苷酸多态性(snp)或核苷酸的插入或缺失(indel)。cyp2d6的等位基因可以是选自以下的结构变体或单基因拷贝变体:*1、*xn、*2、*2xn、*2a、*2axn、*35、*35xn、*9、*9xn、*10、*10xn、*17、*17xn、*29、*29xn、*36-*10、*36-*10xn、*36xn-*10、*36xn-*10xn、*41、*41xn、*3、*3xn、*4、*4xn、*4n、*5、*6、*6xn、*36和*36xn。在一些情况下,cyp2d6的每个等位基因都是不同的结构变体或单基因拷贝变体。在一些情况下,cyp2d6的每个等位基因都是相同的。
34.如本文所用,术语“cyp2d6基因座”是指包含cyp2d6基因和高度同源的假基因cyp2d7和cyp2d8的基因组区域。在人类中,cyp2d6基因座位于22号染色体上。在一些实施方案中,本文提供的方法涉及分析(例如,测序、基因分型、进行结构分析)部分或整个cyp2d6基因座(例如,包括cyp2d6基因和高度同源的假基因cyp2d7和cyp2d8)。在一些实施方案中,本文提供的方法涉及从基因组dna中切除部分或整个cyp2d6基因座(例如,包括cyp2d6基因和高度同源的假基因cyp2d7和cyp2d8)(例如,通过使用crispr相关核酸内切酶和两种或更多种靶向cyp2d6基因座侧翼的基因组序列的grna)。
35.如本文所用,术语“crispr/cas核酸酶系统”是指包含指导rna(grna)和crispr相关核酸内切酶(cas蛋白)的复合物。术语“crispr”可以指规律间隔性成簇短回文重复序列及其相关系统。crispr/cas核酸酶系统可以是1类或2类crispr/cas核酸酶系统。crispr/cas核酸酶系统可以是i型、ii型、iii型、iv型、v型或vi型crispr/cas核酸酶系统。grna可以与cas蛋白相互作用以将cas蛋白的核酸酶活性引导至靶序列。靶序列可以包含“前间隔序列”和“前间隔序列邻近基序”(pam),并且两个结构域都可能是cas介导的活性(例如,切割)所必需的。grna可以与前间隔序列相反链上的结合位点配对(或杂交),以将cas引导至靶序列。pam位点可以指被cas蛋白识别的短序列,并且在一些情况下,可能是cas蛋白活性所必需的。
36.如本文所用,术语“cas”或“cas蛋白”是指或来源于具有核酸内切酶活性的crispr/cas系统的蛋白。在一些情况下,如本文所用的crispr相关核酸内切酶作为cas蛋白。cas蛋白可以是天然存在的cas蛋白、非天然存在的cas蛋白或其片段。在一些情况下,
cas蛋白是天然存在的cas蛋白的变体(例如,相对于天然存在的cas蛋白具有一个或多个氨基酸置换、插入、缺失等)。在一些情况下,cas蛋白是i类cas蛋白,非限制性实例包括cas3、cas8a、cas5、cas8b、cas8c、cas10d、cse1、cse2、csy1、csy2、csy3、gsu0054、cas10、csm2、cmr5、cas10、csx11、csx10和csf1。在一些情况下,cas蛋白是ii类cas蛋白,非限制性实例包括cas9、csn2、cas4、cas12a(cpf1)、cas12b(c2c1)、cas12c(c2c3)、cas13a(c2c2)、cas13b、cas13c和cas13d。在一些情况下,cas蛋白是cas9。在一些情况下,cas蛋白是cas12a。
37.在本文中术语“指导rna”或“grna”可互换使用,通常是指可与cas蛋白结合并有助于将cas蛋白靶向到靶多核苷酸(例如,dna)内的特定位置的rna分子(或统称为rna分子的组)。指导rna可以包含crispr rna(crrna)片段,以及任选地反式激活crrna(tracrrna)片段。如本文所用的术语“crrna”可指rna分子或其部分,其包括靶向多核苷酸的指导序列、茎序列和任选的5'-悬突序列。crrna可以与结合位点结合。如本文所用,术语“tracrrna”可以指包括蛋白质结合区段(例如,蛋白质结合区段能够与crispr相关蛋白例如cas9相互作用)的rna分子或其部分。术语“指导rna”可以指单个指导rna(sgrna),其中crrna区段和任选的tracrrna区段位于同一rna分子中。术语“指导rna”也可以统称为两种或更多种rna分子的组,其中crrna和tracrrna位于不同的rna分子中。
38.如本文所用,术语“长读段测序”(也称为“第三代测序”)通常是指能够产生比第二代测序显著更长的测序读段(》10,000bp)的任何测序方法。在一些实施方案中,本文提供的方法涉及使用长读段测序(例如,对感兴趣的复杂的基因组区域进行基因分型)。长读段测序系统的非限制性实例包括由pacific biosciences、oxford nanopore technology、quantapore、stratos和helicos开发的那些。在一些情况下,长读段测序方法是单分子实时测序(smrt)(例如,由pacific biosciences开发)。在一些情况下,长读段测序方法是纳米孔测序(例如,由oxford nanopore technology开发的minion、gridion和promethion)。在一些情况下,长读段测序包括目前正在开发或将来开发的任何长读段测序方法或系统(例如,第三代测序方法或系统)。
39.如本文所用,术语“核酸扩增”通常是指从单个核酸分子产生靶核酸(例如,dna)的多个拷贝的任何方法。靶核酸可以是dna(例如dna扩增)或rna(例如rna扩增)。核酸扩增包括聚合酶链式反应(pcr)及其任何和所有变体或修饰,以及可替代类型的核酸扩增方法,例如但不限于环介导的等温扩增(lamp)、基于核酸序列的扩增(nasba)、链置换扩增(sda)、多重置换扩增(mda)、滚环扩增(rca)、连接酶链式反应(lcr)、解旋酶依赖性扩增和网状分枝扩增法(ram)。在本公开的各个方面,本文提供的方法不涉及使用核酸(例如,dna)扩增(例如,无扩增)。
40.本公开的方法
41.在本公开的一个方面,提供了一种分析感兴趣的基因组区域的方法,该方法包括:(a)使包含感兴趣的基因组区域的基因组dna与规律间隔性成簇短回文重复序列(crispr)相关核酸内切酶和两种或更多种grna接触,从而产生切除的感兴趣的基因组区域;(b)分离包含感兴趣的基因组区域的基因组dna;以及(c)分析切除的感兴趣的基因组区域,其中所述方法不涉及dna扩增。
42.在各个方面,该方法包括分离包含感兴趣的基因组区域的基因组dna。在一些实施方案中,该方法涉及分离高分子量的基因组dna。在一些实施方案中,该方法涉及富集高分
子量的基因组dna。在一些实施方案中,高分子量的基因组dna的长度为至少约1万个碱基。例如,高分子量的基因组dna长度为至少约1万个碱基、长度为至少约1.5万个碱基、长度为至少约2万个碱基、长度为至少约3万个碱基、长度为至少约3.5万个碱基、长度为至少约4万个碱基、长度为至少约4.5万个碱基、长度为至少约5万个碱基、长度为至少约5.5万个碱基、长度为至少约6万个碱基、长度为至少约6.5万个碱基、长度为至少约7万个碱基、长度为至少约7.5万个碱基、长度为至少约8万个碱基、长度为至少约8.5万个碱基、长度为至少约9万个碱基、长度为至少约9.5万个碱基、或更大。在一些实施方案中,分离高分子量的基因组dna确保在样品中包含整个、完整的感兴趣的基因组区域。
43.在各个方面,该方法涉及用于分离高分子量的基因组dna的任何方法。用于分离高分子量的基因组dna的方法的非限制性实例包括基因组dna和rna纯化系统(由takara bio制造)和nanobind cbb big dna试剂盒(由circulomics制造)。
44.在一些方面,可以在使基因组dna与crispr相关核酸内切酶和指导rna接触之前进行分离包含感兴趣的基因组区域的基因组dna。在其他方面,可以在使基因组dna与crispr相关核酸内切酶和指导rna接触之后(例如,在从基因组dna中切除感兴趣的基因组区域之后)进行分离包含感兴趣的基因组区域的基因组dna。
45.在各个方面,感兴趣的基因组区域是复杂的基因组区域或高度复杂的基因组区域。在一些情况下,感兴趣的基因组区域是高度多态基因组区域。在一些情况下,感兴趣的基因组区域包含多个重复元件或区域。在一些情况下,感兴趣的基因组区域包含一种或多种靶基因和与靶基因具有高度序列同一性(例如,与靶基因具有至少约75%、至少约80%、至少约85%、至少约90%、至少约91%、至少约92%、至少约93%、至少约94%、至少约95%、至少约96%、至少约97%、至少约98%、至少约99%或更高的序列同一性)的一种或多种另外的基因。在一些情况下,感兴趣的基因组区域包含一种或多种靶基因和与靶基因具有高度序列同一性(例如,与靶基因具有至少约75%、至少约80%、至少约85%、至少约90%、至少约91%、至少约92%、至少约93%、至少约94%、至少约95%、至少约96%、至少约97%、至少约98%、至少约99%或更高的序列同一性)的一种或多种假基因。在一些情况下,感兴趣的基因组区域包含一个或多个重复区域、一个或多个重复段、一个或多个插入、一个或多个倒位、一个或多个串联重复序列、一个或多个逆转录转座子或其任意组合。在一些情况下,感兴趣的基因组区域是通常通过传统方法(例如,通过短读段测序方法)难以准确分析的或通过传统方法(例如,通过短读段测序方法)准确分析基因组区域具有挑战的基因组区域。
46.在一些情况下,感兴趣的基因组区域的长度为至少约1万个碱基。例如,感兴趣的基因组区域的长度可以是至少约1万个碱基、长度为至少约1.5万个碱基、长度为至少约2万个碱基、长度为至少约2.5万个碱基、长度为至少约3万个碱基、长度为至少约3.5万个碱基、长度为至少约4万个碱基、长度为至少约4.5万个碱基、长度为至少约5万个碱基、长度为至少约5.5万个碱基、长度为至少约6万个碱基、长度为至少约6.5万个碱基、长度为至少约7万个碱基、长度为至少约7.5万个碱基、长度为至少约8万个碱基、长度为至少约8.5万个碱基、长度为至少约9万个碱基、长度为至少约9.5万个碱基、长度为至少约10万个碱基、长度为至少约11万个碱基、长度为至少约12万个碱基、长度为至少约13万个碱基、长度为至少约14万个碱基、长度为至少约15万个碱基、长度为至少约16万个碱基、长度为至少约17万个碱基、长度为至少约18万个碱基、长度为至少约19万个碱基、长度为至少约20万个碱基、长度为至
少约21万个碱基、长度为至少约22万个碱基、长度为至少约23万个碱基、长度为至少约24万个碱基、长度为至少约25万个碱基。在一些方面,感兴趣的基因组区域的长度大于约1万个碱基。在一些方面,感兴趣的基因组区域的长度小于约25万个碱基。
47.在各个方面,该方法涉及使包含感兴趣的基因组区域(例如,复杂的基因组区域)的基因组dna与crispr相关核酸内切酶和两种或更多种grna接触。在一些情况下,接触导致从基因组dna中切除整个感兴趣的基因组区域。在一些情况下,接触导致切除部分感兴趣的基因组区域。crispr相关核酸内切酶可以是本文所述的任何crispr相关核酸内切酶。在一些情况下,crispr相关核酸内切酶是i类或ii类crispr相关核酸内切酶。cas i crispr相关核酸内切酶的非限制性实例包括cas3、cas5、cas8a、cas8b、cas8c、cas10d、cse1、cse2、csy1、csy2、csy3、gsu0054、cas10、csm2、cmr5、csx11、csx10和csf1。ii类crispr相关核酸内切酶的非限制性实例包括cas9、cas12a、csn2、cas4、cas12b、cas12c、cas13a、cas13b、cas13c和cas13d。在一些情况下,crispr相关核酸内切酶是cas蛋白或多肽。在一些实施方案中,crispr相关核酸内切酶是cas12a蛋白或多肽。
48.在一些实施方案中,crispr相关核酸内切酶是cas9蛋白或多肽。在一些情况下,cas9蛋白或多肽来源于细菌物种酿脓链球菌。在一些情况下,cas9蛋白或多肽具有与野生型cas9氨基酸序列相同的氨基酸序列。在其他情况下,cas9蛋白或多肽具有相对于野生型cas9氨基酸序列被修饰的氨基酸序列。在一些情况下,cas9蛋白或多肽具有一个或多个突变(例如,相对于野生型cas9蛋白或多肽)。在一些情况下,一个或多个突变是置换、缺失或插入。cas9蛋白或多肽可以具有相对于野生型cas9蛋白或多肽至少约50%序列同一性的氨基酸序列。例如,cas9蛋白或多肽可具有相对于野生型cas9蛋白或多肽至少约50%、至少约55%、至少约60%、至少约65%、至少约70%、至少约75%、至少约80%、至少约85%、至少约90%、至少约95%、至少约96%、至少约97%、至少约98%或至少约99%的序列同一性。在一些情况下,cas9变体可以包含相对于野生型酿脓链球菌cas9的一个或多个点突变。例如,cas9变体可以包含相对于野生型酿脓链球菌cas9选自r780a、k810a、k848a、k855a、h982a、k1003a、r1060a、d1135e、n497a、r661a、q695a、q926a、l169a、y450a、m495a、m694a和m698a的点突变。
49.在各个方面,该方法包括使基因组dna与两种或更多种grna接触。grna可以是crispr rna(crrna)或单指导rna(sgrna)。在一些实施方案中,两种或更多种grna各自包含与基因组dna上的靶核苷酸序列互补或基本上互补的核苷酸序列,使得两种或更多种grna能够与靶核苷酸序列结合,并将crispr复合物引导到所期望的切割位点。在一些实施方案中,两种或更多种grna中的每一种与基因组dna上的不同靶序列结合。在一些实施方案中,两种或更多种grna中的至少一种与感兴趣的基因组区域上游的区域互补或基本上互补,并且两种或更多种grna中的至少一种与感兴趣的基因组下游的区域互补或基本上互补。在一些实施方案中,两种或更多种grna与位于感兴趣的基因组区域侧翼的靶序列结合。通常,grna被设计成使得它们各自靶向感兴趣的基因组区域之外的基因组序列,使得接触(例如,与crispr相关核酸内切酶和两种或更多种grna)从基因组dna切除整个感兴趣的基因组区域。
50.在各个方面,该方法还涉及分析切除的感兴趣的基因组区域。在一些情况下,分析包括对切除的基因组区域进行基因分型。基因分型可以包括通过使用一种或多种测定来检
查感兴趣的基因组区域的序列,并且在一些情况下,将该序列与另一序列(例如,参考序列)进行比较,从而鉴定感兴趣的基因组区域的遗传构成差异的过程。可以通过任何已知方法,包括但不限于dna测序、限制性片段长度多态性鉴定(rflpi)、随机扩增多态性检测(rapd)、扩增片段长度多态性检测(aflpd)、聚合酶链式反应(pcr)、等位基因特异性寡核苷酸(aso)探针以及与dna微阵列或珠子的杂交进行基因分型。在一些情况下,分析包括对感兴趣的基因组区域进行结构分析。
51.在一些情况下,分析包括对感兴趣的基因组区域进行测序。在一些情况下,测序是长读段测序方法(例如,第三代测序方法)。长读段测序方法可以是能够产生比短读段测序方法(例如,第二代测序方法)长得多的测序读段的任何测序方法。在一些情况下,长读段测序方法是能够产生至少10,00万个碱基的测序读段的测序方法。在一些情况下,长读段测序方法是单分子实时测序(例如,smrt测序,pacific biosciences)。在一些情况下,长读段测序方法是纳米孔测序(例如,由oxford nanopore technologies开发的minion、gridion和promethion)。在一些方面,在测序之前,该方法还涉及将接头(例如,测序接头)连接到切除的感兴趣的基因组区域的末端。在一些情况下,该方法可以涉及适用于测序应用的任何其他处理方法,包括末端加尾步骤、去磷酸化步骤等。
52.在各个方面,本文提供的方法是无扩增的(例如,不涉及核酸扩增(例如,dna扩增)步骤)。在一些情况下,本文提供的方法不涉及聚合酶链式反应(pcr)。在一些情况下,本文提供的方法不涉及等温扩增。在一些情况下,本文提供的方法不涉及环介导的等温扩增(lamp)、基于核酸序列的扩增(nasba)、链置换扩增(sda)、多重置换扩增(mda)、滚环扩增(rca)、连接酶链式反应(lcr)、解旋酶依赖性扩增和网状分枝扩增法(ram)中的任何一种。核酸扩增技术经常引入错误。有利地,本文提供的方法避免使用可能将错误引入测序模板的核酸扩增方法。
53.在各个方面,该方法不涉及片段化、剪切或消化基因组dna。在一些情况下,该方法不涉及使用例如限制酶消化基因组dna。换言之,该方法是在没有被剪切、消化或片段化的基因组dna上直接进行的。
54.在本公开的另一个方面,提供了对包含长度为至少1万个碱基的感兴趣的复杂的基因组区域的基因座进行测序的方法,该方法包括:(a)提供包含感兴趣的复杂的基因组区域的基因组dna;(b)分离包含感兴趣的复杂的基因组区域的高分子量dna;(c)使基因组dna与规律间隔性成簇短回文重复序列(crispr)相关核酸内切酶和两种或更多种grna接触,以切除感兴趣的复杂的基因组区域,其中两种或更多种grna各自包含与基因组dna中存在的不同核苷酸序列基本上互补的核苷酸序列,并且其中不同核苷酸序列位于复杂的基因组区域的侧翼;以及c)分析复杂的基因组区域。在一些情况下,该方法不涉及dna扩增(例如,无扩增)。
55.在各个方面,复杂的基因组区域包含靶基因,以及与靶基因具有高度序列同一性的一种或多种假基因。在一些情况下,一种或多种假基因可以与靶基因具有至少约75%(例如,至少约75%、至少约80%、至少约85%、至少约90%、至少约91%、至少约92%、至少约93%、至少约94%、至少约95%、至少约96%、至少约97%、至少约98%、至少约99%)的序列同一性。在一个特定方面,遗传基因座包含靶基因cyp2d6和假基因cyp2d7和cyp2d8。
56.在各个方面,复杂的基因组区域包含靶基因和与靶基因具有高度序列同一性的一
种或多种另外的基因。在一些情况下,一种或多种另外的基因可以与靶基因具有至少约75%(例如,至少约75%、至少约80%、至少约85%、至少约90%、至少约91%、至少约92%、至少约93%、至少约94%、至少约95%、至少约96%、至少约97%、至少约98%、至少约99%)序列同一性。在一个特定方面,遗传基因座包含基因cyp2c8、cyp2c9、cyp2c18和cyp2c19。在一些情况下,通常通过传统方法(例如,通过短读段测序方法)很难对遗传基因座进行准确测序或对遗传基因座进行准确测序具有挑战性。
57.在各个方面,复杂的基因组区域是高度多态的遗传基因座。在各个方面,复杂的基因组区域包含一个或多个重复区域、一个或多个重复段、一个或多个插入、一个或多个倒位、一个或多个串联重复序列、一个或多个逆转录转座子或其任意组合。
58.在一些情况下,感兴趣的复杂的基因组区域的长度为至少约1万个碱基。例如,感兴趣的基因组区域的长度可以是至少约1万个碱基、长度为至少约1.5万个碱基、长度为至少约2万个碱基、长度为至少约2.5万个碱基、长度为至少约3万个碱基、长度为至少约3.5万个碱基、长度为长度为至少约4万个碱基、长度为至少约4.5万个碱基、长度为至少约5万个碱基、至少约5.5万个碱基、长度为至少约6万个碱基、长度为至少约6.5万个碱基、长度为至长度为至少约7万个碱基、长度为至少约7.5万个碱基、长度为至少约8万个碱基、长度为至少约8.5万个碱基、长度为至少约9万个碱基、长度为至少约9.5万个碱基、长度为至少约10万个碱基、长度为至少约11万个碱基、长度为至少约12万个碱基、长度为至少约13万个碱基、长度为至少约14万个碱基、长度为至少约15万个碱基、长度为至少约16万个碱基、少约17万个碱基、长度为至少约18万个碱基、长度为至少约19万个碱基、长度为至少约20万个碱基、长度为至少约21万个碱基、长度为至少约22万个碱基、长度为至少约23万个碱基、长度为至少约24万个碱基或长度为至少约25万个碱基。在一些方面,感兴趣的基因组区域的长度大于约1万个碱基。在一些方面,感兴趣的基因组区域的长度小于约25万个碱基。
59.在各个方面,所述方法涉及使包含遗传基因座的基因组dna与crispr相关核酸内切酶和两种或更多种grna接触。在一些情况下,接触导致从基因组dna中切除整个遗传基因座。在一些情况下,接触导致切除部分感兴趣的遗传基因座。crispr相关核酸内切酶可以是本文所述的任何crispr相关核酸内切酶。在一些情况下,crispr相关核酸内切酶是i类或ii类crispr相关核酸内切酶。i类crispr相关核酸内切酶的非限制性实例包括cas3、cas5、cas8a、cas8b、cas8c、cas10d、cse1、cse2、csy1、csy2、csy3、gsu0054、cas10、csm2、cmr5、csx11、csx10、and csf1。ii类crispr相关核酸内切酶的非限制性实例包括cas9、cas12a、csn2、cas4、cas12b、cas12c、cas13a、cas13b、cas13c和cas13d。在一些情况下,crispr相关核酸内切酶是cas蛋白或多肽。在一些实施方案中,crispr相关核酸内切酶是cas12a蛋白或多肽。
60.在一些实施方案中,crispr相关核酸内切酶是cas9蛋白或多肽。在一些情况下,cas9蛋白或多肽来源于细菌物种酿脓链球菌。在一些情况下,cas9蛋白或多肽具有与野生型cas9氨基酸序列相同的氨基酸序列。在其他情况下,cas9蛋白或多肽具有相对于野生型cas9氨基酸序列被修饰的氨基酸序列。在一些情况下,cas9蛋白或多肽具有一个或多个突变(例如,相对于野生型cas9蛋白或多肽)。在一些情况下,一个或多个突变是置换、缺失或插入。cas9蛋白或多肽可以具有相对于野生型cas9蛋白或多肽至少约50%序列同一性的氨基酸序列。例如,cas9蛋白或多肽可以具有相对于野生型cas9蛋白或多肽至少约50%、至少
约55%、至少约60%、至少约65%、至少约70%、至少约75%、至少约80%、至少约85%、至少约90%、至少约95%、至少约96%、至少约97%、至少约98%或至少约99%的序列同一性。在一些情况下,cas9变体可以包含相对于野生型酿脓链球菌cas9的一个或多个点突变。例如,cas9变体可以包含相对于野生型酿脓链球菌cas9选自r780a、k810a、k848a、k855a、h982a、k1003a、r1060a、d1135e、n497a、r661a、q695a、q926a、l169a、y450a、m495a、m694a和m698a的点突变。
61.在各个方面,该方法包括使基因组dna与两种或更多种grna接触。在一些实施方案中,两种或更多种grna各自包含与基因组dna上的靶核苷酸序列互补或基本上互补的核苷酸序列,使得两种或更多种grna能够与靶核苷酸序列结合,并将crispr复合物引导至所期望的切割位点。在一些实施方案中,两种或更多种grna中的每一种与基因组dna上的不同靶序列结合。在一些实施方案中,两种或更多种grna中的至少一种与感兴趣的复杂的基因组区域上游的区域互补或基本上互补,并且两种或更多种grna中的至少一种与感兴趣的复杂的基因组区域下游的区域互补或基本上互补。在一些实施方案中,两种或更多种grna与位于感兴趣的复杂的基因组区域侧翼的靶序列结合。通常,grna被设计成使得它们各自靶向感兴趣的基因组区域之外的基因组序列,使得接触(例如,与crispr相关核酸内切酶和两种或更多种grna)从基因组dna切除整个感兴趣的基因组区域。
62.在各个方面,该方法进一步涉及对复杂的基因组区域进行分析。分析可以包括本文提供的任何方法,包括基因分型、进行结构分析和/或对切除的感兴趣的基因组区域进行测序。在一些情况下,测序是长读段测序方法(例如,第三代测序方法)。长读段测序方法可以是能够产生比短读段测序方法(例如,第二代测序方法)长得多的测序读段的任何测序方法。在一些情况下,长读段测序方法是能够产生至少10,00万碱基的测序读段的测序方法。在一些情况下,长读段测序方法是单分子实时测序(例如,smrt测序,pacific biosciences)。在一些情况下,长读段测序方法是纳米孔测序(例如,由oxford nanopore technologies开发的minion、gridion和promethion)。在一些方面,在测序之前,该方法还涉及将接头(例如,测序接头)连接到切除的感兴趣的基因组区域的末端。可以使用适合制备用于测序的dna样品的任何其他方法(例如,末端加尾、去磷酸化步骤等)。
63.在各个方面,该方法涉及从包含基因组dna的样品中分离高分子量的基因组dna。在一些实施方案中,该方法涉及富集高分子量的基因组dna。在一些实施方案中,高分子量的基因组dna的长度为至少约1万个碱基。例如,高分子量的基因组dna的长度为至少约1万个碱基、长度为至少约1.5万个碱基、长度为至少约2万个碱基、长度为至少约2.5万个碱基、长度为至少约3万个碱基、长度为至少约3.5万个碱基、长度为至少约4万个碱基、长度为至少约4.5万个碱基、长度为至少约5万个碱基、长度为至少约5.5万个碱基、长度为至少约6万个碱基、长度为至少约6.5万个碱基、长度为至少约7万个碱基、长度为至少约7.5万个碱基、长度为至少约8万个碱基、长度为至少约8.5万个碱基、长度为至少约9万个碱基、长度为至少约9.5万个碱基或更大。在一些实施方案中,分离高分子量的基因组dna确保样品中包含整个、完整的遗传基因座。
64.在各个方面,该方法涉及用于分离高分子量的基因组dna的任何方法。用于分离高分子量的基因组dna的方法的非限制性实例包括基因组dna和rna纯化系统(由takara bio制造)和nanobind cbb big dna试剂盒(由circulomics制造)。
65.在一些方面,可以在使基因组dna与crispr相关核酸内切酶和指导rna接触之前进行分离高分子量的基因组dna。在其他方面,可以在使基因组dna与crispr相关核酸内切酶和指导rna接触之后(例如,在从基因组dna中切除感兴趣的基因组区域之后)进行分离高分子量的基因组dna。
66.在各个方面,本文提供的方法是无扩增的(例如,不涉及核酸扩增(例如,dna扩增)步骤)。在一些情况下,本文提供的方法不涉及聚合酶链式反应(pcr)。在一些情况下,本文提供的方法不涉及等温扩增。在一些情况下,本文提供的方法不涉及环介导的等温扩增(lamp)、基于核酸序列的扩增(nasba)、链置换扩增(sda)、多重置换扩增(mda)、滚环扩增(rca)、连接酶链式反应(lcr)、解旋酶依赖性扩增和网状分枝扩增法(ram)中的任何一种。核酸扩增技术经常引入错误。有利地,本文提供的方法避免使用可能将错误引入测序模板的核酸扩增方法。
67.在各个方面,该方法不涉及片段化、剪切或消化基因组dna。在一些情况下,该方法不涉及使用例如限制酶消化基因组dna。换言之,该方法是在没有被剪切、消化或片段化的基因组dna上直接进行的。
68.在又一方面,提供了一种分析包含cyp2d6、cyp2d7和cyp2d8的遗传基因座的方法,该方法包括:a)提供包含该基因座的基因组dna;b)使基因组dna与规律间隔性成簇短回文重复序列(crispr)相关核酸内切酶和两种或更多种grna接触以从该基因组dna切除遗传基因座,其中两种或更多种grna各自包含与基因组dna中存在的不同核苷酸序列基本上互补的核苷酸序列,并且其中不同核苷酸序列位于包含cyp2d6、cyp2d7和cyp2d8的遗传基因座的侧翼;以及c)分析遗传基因座。在一些情况下,该方法还包括在b)之前分离高分子量dna。
69.在一些情况下,分析包括对遗传基因座(例如,如本文所述的)进行基因分型。在一些情况下,分析包括对遗传基因座进行结构分析(例如,如本文所述)。在一些情况下,分析包括对遗传基因座(例如,如本文所述的)进行测序(例如,长读段测序)。
70.在另一方面,提供了鉴定对象中cyp2d6遗传变异的方法,该方法包括:a)提供包含从对象中获得的基因组dna的生物样品;b)使基因组dna与规律间隔性成簇短回文重复序列(crispr)相关核酸内切酶和两种或更多种grna接触以切除包含cyp2d6、cyp2d7和cyp2d8的遗传基因座;c)对遗传基因座进行长读段测序;以及d)鉴定对象的cyp2d6中的一种或多种遗传变异。
71.在一些情况下,遗传基因座的长度为至少约4万个碱基。例如,遗传基因座的长度可以是至少约4万个碱基、长度为至少约4.5万个碱基、长度为至少约5万个碱基、长度为至少约5.5万个碱基、长度为至少约6万个碱基、长度为至少约6.5万个碱基、长度为至少约7万个碱基、长度为至少约7.5万个碱基、长度为至少约8万个碱基、长度为至少约8.5万个碱基、长度为至少约9万个碱基、长度为至少约9.5万个碱基或长度为至少约10万个碱基。
72.在各个方面,该方法涉及使包含遗传基因座的基因组dna与crispr相关核酸内切酶和两种或更多种grna接触。在一些情况下,接触导致从基因组dna中切除整个遗传基因座(例如,含有cyp2d6、cyp2d7和cyp2d8)。在一些情况下,接触导致切除部分遗传基因座(例如,含有cyp2d6、cyp2d7和cyp2d8)。crispr相关核酸内切酶可以是本文所述的任何crispr相关核酸内切酶。在一些情况下,crispr相关核酸内切酶是i类或ii类crispr相关核酸内切酶。i类crispr相关核酸内切酶的非限制性实例包括cas3、cas5、cas8a、cas8b、cas8c、
cas10d、cse1、cse2、csy1、csy2、csy3、gsu0054、cas10、csm2、cmr5、csx11、csx10和csf1。ii类crispr相关核酸内切酶的非限制性实例包括cas9、cas12a、csn2、cas4、cas12b、cas12c、cas13a、cas13b、cas13c和cas13d。在一些情况下,crispr相关核酸内切酶是cas蛋白或多肽。在一些实施方案中,crispr相关核酸内切酶是cas12a蛋白或多肽。
73.在一些实施方案中,crispr相关核酸内切酶是cas9蛋白或多肽。在一些情况下,cas9蛋白或多肽来源于细菌物种酿脓链球菌。在一些情况下,cas9蛋白或多肽具有与野生型cas9氨基酸序列相同的氨基酸序列。在其他情况下,cas9蛋白或多肽具有相对于野生型cas9氨基酸序列被修饰的氨基酸序列。在一些情况下,cas9蛋白或多肽具有一个或多个突变(例如,相对于野生型cas9蛋白或多肽)。在一些情况下,一个或多个突变是置换、缺失或插入。cas9蛋白或多肽可以具有相对于野生型cas9蛋白或多肽至少约50%序列同一性的氨基酸序列。例如,cas9蛋白或多肽可以具有相对于野生型cas9蛋白或多肽至少约50%、至少约55%、至少约60%、至少约65%、至少约70%、至少约75%、至少约80%、至少约85%、至少约90%、至少约95%、至少约96%、至少约97%、至少约98%或至少约99%的序列同一性。在一些情况下,cas9变体可以包含相对于野生型酿脓链球菌cas9的一个或多个点突变。例如,cas9变体可以包含相对于野生型酿脓链球菌cas9选自:r780a、k810a、k848a、k855a、h982a、k1003a、r1060a、d1135e、n497a、r661a、q695a、q926a、l169a、y450a、m495a、m694a和m698a的点突变。
74.在各个方面,该方法包括使基因组dna与两种或更多种grna接触。在一些实施方案中,两种或更多种grna各自包含与基因组dna上的靶核苷酸序列互补或基本上互补的核苷酸序列,使得两种或更多种grna能够与靶核苷酸序列结合,并将crispr复合物引导至所期望的切割位点。在一些实施方案中,两种或更多种grna中的每一种与基因组dna上的不同靶序列结合。在一些实施方案中,两种或更多种grna中的至少一种与遗传基因座(例如,含有cyp2d6、cyp2d7和cyp2d8)上游的区域互补或基本上互补,并且两种或更多种grna中的至少一种与遗传基因座(例如,含有cyp2d6、cyp2d7和cyp2d8)下游的区域互补或基本上互补。在一些实施方案中,两种或更多种grna与位于遗传基因座(例如,含有cyp2d6、cyp2d7和cyp2d8)侧翼的靶序列结合。通常,设计grna使得它们各自靶向遗传基因座(例如,含有cyp2d6、cyp2d7和cyp2d8)之外的基因组序列,使得接触(例如,与crispr相关核酸内切酶和两种或更多种grna)从基因组dna中切除整个遗传基因座(例如,含有cyp2d6、cyp2d7和cyp2d8)。
75.在一些情况下,至少一种grna包含根据下表1中提供的任何核苷酸序列(例如,seq id no:1-26)的核苷酸序列。在一些情况下,至少一种grna包含与下表1中提供的任何核苷酸序列(例如,seq id no:1-26)具有至少约90%(例如,至少约91%、至少约92%、至少约93%、至少约94%、至少约95%、至少约96%、至少约97%、至少约98%、至少约99%)的序列同一性的核苷酸序列。在一些情况下,第一grna包含seq id no:1、2或13-16中任一个的核苷酸序列或与seq id no:1、2或13-16中的任一个具有至少90%(例如,至少约91%、至少约92%、至少约93%、至少约94%、至少约95%、至少约96%、至少约97%、至少约98%、至少约99%)序列同一性的核苷酸序列。在一些情况下,第二grna包含seq id no:3-12或17-26中任一个的核苷酸序列或与seq id no:3-12或17-26中任一个具有至少90%(例如,至少约91%、至少约92%、至少约93%、至少约94%、至少约95%、至少约96%、至少约97%、至少约
98%、至少约99%)的序列同一性的核苷酸序列。在一些情况下,至少一种grna是crrna。在一些情况下,至少一种grna是sgrna。表1.指导rna序列
76.在各个方面,该方法还涉及分析(例如,基因分型、测序、进行结构分析)切除的感兴趣的基因组区域。在一些情况下,测序是长读段测序方法(例如,第三代测序方法)。长读段测序方法可以是能够产生比短读段测序方法(例如,第二代测序方法)长得多的测序读段的任何测序方法。在一些情况下,长读段测序方法是能够产生至少10,00万个碱基的测序读段的测序方法。在一些情况下,长读段测序方法是单分子实时测序(例如,smrt测序,pacific biosciences)。在一些情况下,长读段测序方法是纳米孔测序(例如,由oxford nanopore technologies开发的minion、gridion和promethion)。在一些方面,在测序之前,该方法还涉及将接头(例如,测序接头)连接到切除的感兴趣的基因组区域的末端。
77.在各个方面,该方法涉及从包含基因组dna的样品中分离高分子量的基因组dna。在一些实施方案中,该方法涉及富集高分子量的基因组dna。在一些实施方案中,高分子量的基因组dna的长度为至少约4万个碱基。例如,高分子量的基因组dna的长度为至少约4万个碱基、长度为至少约4.5万个碱基、长度为至少约5万个碱基、长度为至少约5.5万个碱基、长度为至少约6万个碱基、长度为至少约6.5万个碱基、长度为至少约7万个碱基、长度为至少约7.5万个碱基、长度为至少约8万个碱基、长度为至少约8.5万个碱基、长度为至少约9万个碱基、长度为至少约9.5万个碱基或更大。在一些实施方案中,分离高分子量的基因组dna
确保样品中包含整个、完整的遗传基因座。
78.在各个方面,该方法涉及用于分离高分子量的基因组dna的任何方法。用于分离高分子量的基因组dna的方法的非限制性实例包括基因组dna和rna纯化系统(由takara bio制造)和nanobind cbb big dna试剂盒(由circulomics制造)。
79.在一些方面,可以在使基因组dna与crispr相关核酸内切酶和指导rna接触之前进行分离高分子量的基因组dna。在其他方面,可以在使基因组dna与crispr相关核酸内切酶和指导rna接触之后(例如,在从基因组dna中切除感兴趣的基因组区域之后)进行分离高分子量的基因组dna。
80.在各个方面,本文提供的方法是无扩增的(例如,不涉及核酸扩增(例如,dna扩增)步骤)。在一些情况下,本文提供的方法不涉及聚合酶链式反应(pcr)。在一些情况下,本文提供的方法不涉及等温扩增。在一些情况下,本文提供的方法不涉及环介导的等温扩增(lamp)、基于核酸序列的扩增(nasba)、链置换扩增(sda)、多重置换扩增(mda)、滚环扩增(rca)、连接酶链式反应(lcr)、解旋酶依赖性扩增和网状分枝扩增法(ram)中的任何一种。核酸扩增技术经常引入错误。有利地,本文提供的方法避免使用可能将错误引入测序模板的核酸扩增方法。
81.在各个方面,该方法不涉及片段化、剪切或消化基因组dna。在一些情况下,该方法不涉及使用例如限制酶消化基因组dna。换言之,该方法是在没有被剪切、消化或片段化的基因组dna上直接进行的。
82.在各个方面,遗传变异是cyp2d6中的药物遗传学相关变异(例如,星等位基因单体型)。在一些情况下,遗传变异是cyp2d6的结构变异。在一些情况下,基于遗传变异,鉴定对象为具有降低或丧失的cyp2d6功能。在一些情况下,对象被鉴定为具有增加或获得的cyp2d6功能。
83.在各个方面,该方法还包括基于鉴定向对象推荐治疗。在各个方面,该方法还包括基于鉴定治疗对象。在各个方面,该方法涉及基于鉴定推荐替代治疗。在各个方面,该方法涉及基于鉴定推荐药物剂量。在各个方面,该方法涉及改变施用于对象的药物(例如,由cyp2d6激活或代谢的)的剂量(或推荐改变剂量)。在一些情况下,药物(或治疗剂)是由cyp2d6激活或代谢的药物。
84.组合物和试剂盒
85.在一个方面,提供了一种组合物,其包含:a)规律间隔性成簇短回文重复序列(crispr)相关核酸内切酶;b)第一指导rna(grna),其包含与基因组dna中存在的核苷酸序列基本上互补的核苷酸序列,该基因组dna位于包含cyp2d6、cyp2d7和cyp2d8的遗传基因座的上游;c)第二指导rna(grna),其包含与基因组dna中存在的核苷酸序列基本上互补的核苷酸序列,该基因组dna位于包含cyp2d6、cyp2d7和cyp2d8的遗传基因座的下游。
86.crispr相关核酸内切酶可以是本文所述的任何crispr相关核酸内切酶。在一些情况下,crispr相关核酸内切酶是i类或ii类crispr相关核酸内切酶。cas i crispr相关核酸内切酶的非限制性实例包括,ii类crispr相关核酸内切酶的非限制性实例包括:cas3、cas5、cas8a、cas8b、cas8c、cas10d、cse1、cse2、csy1、csy2、csy3、gsu0054、cas10、csm2、cmr5、csx11、csx10和csf1。cas9、cas12a、csn2、cas4、cas12b、cas12c、cas13a、cas13b、cas13c和cas13d。在一些情况下,crispr相关核酸内切酶是cas蛋白或多肽。在一些实施方
案中,crispr相关核酸内切酶是cas12a蛋白或多肽。
87.在一些实施方案中,crispr相关核酸内切酶是cas9蛋白或多肽。在一些情况下,cas9蛋白或多肽来源于细菌物种酿脓链球菌。在一些情况下,cas9蛋白或多肽具有与野生型cas9氨基酸序列相同的氨基酸序列。在其他情况下,cas9蛋白或多肽具有相对于野生型cas9氨基酸序列被修饰的氨基酸序列。在一些情况下,cas9蛋白或多肽具有一个或多个突变(例如,相对于野生型cas9蛋白或多肽)。在一些情况下,一个或多个突变是置换、缺失或插入。cas9蛋白或多肽可具有相对于野生型cas9蛋白或多肽至少约50%序列同一性的氨基酸序列。例如,cas9蛋白或多肽可具有相对于野生型cas9蛋白或多肽至少约50%、至少约55%、至少约60%、至少约65%、至少约70%、至少约75%、至少约80%、至少约85%、至少约90%、至少约95%、至少约96%、至少约97%、至少约98%或至少约99%的序列同一性。在一些情况下,cas9变体可以包含相对于野生型酿脓链球菌cas9的一个或多个点突变。例如,cas9变体可以包含相对于野生型酿脓链球菌cas9选自:r780a、k810a、k848a、k855a、h982a、k1003a、r1060a、d1135e、n497a、r661a、q695a、q926a、l169a、y450a、m495a、m694a和m698a的点突变。
88.在一些实施方案中,两种或更多种grna各自包含与基因组dna上的靶核苷酸序列互补或基本上互补的核苷酸序列,使得两种或更多种grna能够与靶核苷酸序列结合,并将crispr复合物引导至所期望的切割位点。在一些实施方案中,两种或更多种grna中的每一种与基因组dna上的不同靶序列结合。在一些在实施方案中,两种或更多种grna中的至少一种与遗传基因座(例如,含有cyp2d6、cyp2d7和cyp2d8)的上游区域互补或基本上互补,并且两种或更多种grna中的至少一种与遗传基因座(例如,含有cyp2d6、cyp2d7和cyp2d8)的下游区域互补或基本上互补。在一些实施方案中,两种或更多种grna与位于遗传基因座(例如,含有cyp2d6、cyp2d7和cyp2d8)侧翼的靶序列结合。通常,设计grna使得它们各自靶向遗传基因座(例如,含有cyp2d6、cyp2d7和cyp2d8)之外的基因组序列,使得接触(例如,与crispr相关核酸内切酶和两种或更多种grna)从基因组dna中切除整个遗传基因座(例如,含有cyp2d6、cyp2d7和cyp2d8)。
89.在一些情况下,至少一种grna包含根据表1中提供的任何核苷酸序列(例如,seq id no:1-26)的核苷酸序列。在一些情况下,至少一种grna包含与表1中提供的任何核苷酸序列(例如,seq id no:1-26)具有至少约90%(例如,至少约91%、至少约92%、至少约93%、至少约94%、至少约95%、至少约96%、至少约97%、至少约98%、至少约99%)的序列同一性的核苷酸序列。在一些情况下,至少一种grna是crrna。在一些情况下,至少一种grna是sgrna。
90.本文进一步提供了用于对cyp2d6进行基因分型的试剂盒,其包括:a)规律间隔性成簇短回文重复序列(crispr)相关核酸内切酶;b)第一指导rna(grna),其包含与基因组dna中存在的核苷酸序列基本上互补的核苷酸序列,该基因组dna位于包含cyp2d6、cyp2d7和cyp2d8的遗传基因座的上游;c)第二指导rna(grna),其包含与基因组dna中存在的核苷酸序列基本上互补的核苷酸序列,该基因组dna位于包含cyp2d6、cyp2d7和cyp2d8的遗传基因座的下游。在一些情况下,该试剂盒还包括说明书(例如,用于使用该试剂盒对cyp2d6进行基因分型)。
91.crispr相关核酸内切酶可以是本文所述的任何crispr相关核酸内切酶。在一些情
况下,crispr相关核酸内切酶是i类或ii类crispr相关核酸内切酶。i类crispr相关核酸内切酶的非限制性实例包括cas3、cas5、cas8a、cas8b、cas8c、cas10d、cse1、cse2、csy1、csy2、csy3、gsu0054、cas10、csm2、cmr5、csx11、csx10和csf1。ii类crispr相关核酸内切酶的非限制性实例包括cas9、cas12a、csn2、cas4、cas12b、cas12c、cas13a、cas13b、cas13c和cas13d。在一些情况下,crispr相关核酸内切酶是cas蛋白或多肽。在一些实施方案中,crispr相关核酸内切酶是cas12a蛋白或多肽。
92.在一些实施方案中,crispr相关核酸内切酶是cas9蛋白或多肽。在一些情况下,cas9蛋白或多肽来源于细菌物种酿脓链球菌。在一些情况下,cas9蛋白或多肽具有与野生型cas9氨基酸序列相同的氨基酸序列。在其他情况下,cas9蛋白或多肽具有相对于野生型cas9氨基酸序列被修饰的氨基酸序列。在一些情况下,cas9蛋白或多肽具有一个或多个突变(例如,相对于野生型cas9蛋白或多肽)。在一些情况下,一个或多个突变是置换、缺失或插入。cas9蛋白或多肽可具有相对于野生型cas9蛋白或多肽至少约50%序列同一性的氨基酸序列。例如,cas9蛋白或多肽可具有相对于野生型cas9蛋白或多肽至少约50%、至少约55%、至少约60%、至少约65%、至少约70%、至少约75%、至少约80%、至少约85%、至少约90%、至少约95%、至少约96%、至少约97%、至少约98%或至少约99%的序列同一性。在一些情况下,cas9变体可以包含相对于野生型酿脓链球菌cas9的一个或多个点突变。例如,cas9变体可以包含相对于野生型酿脓链球菌cas9选自r780a、k810a、k848a、k855a、h982a、k1003a、r1060a、d1135e、n497a、r661a、q695a、q926a、l169a、y450a、m495a、m694a和m698a的点突变。
93.在一些实施方案中,两种或更多种grna各自包含与基因组dna上的靶核苷酸序列互补或基本上互补的核苷酸序列,使得两种或更多种grna能够与靶核苷酸序列结合,并将crispr复合物引导至所期望的切割位点。在一些实施方案中,两种或更多种grna中的每一种与基因组dna上的不同靶序列结合。在一些实施方案中,两种或更多种grna中的至少一种与遗传基因座(例如,含有cyp2d6、cyp2d7和cyp2d8)的上游区域互补或基本上互补,并且两种或更多种grna中的至少一种与遗传基因座(例如,含有cyp2d6、cyp2d7和cyp2d8)的下游区域互补或基本上互补。在一些实施方案中,两种或更多种grna与位于遗传基因座(例如,含有cyp2d6、cyp2d7和cyp2d8)侧翼的靶序列结合。通常,设计grna使得它们各自靶向遗传基因座(例如,包含cyp2d6、cyp2d7和cyp2d8)之外的基因组序列,使得接触(例如,与crispr相关核酸内切酶和两种或更多种grna)从基因组dna中切除整个遗传基因座(例如,含有cyp2d6、cyp2d7和cyp2d8)。
94.在一些情况下,至少一种grna包含根据表1中提供的任何核苷酸序列(例如,seq id no:1-26)的核苷酸序列。在一些情况下,至少一种grna包含与表1中提供的任何核苷酸序列(例如,seq id no:1-26)具有至少约90%(例如,至少约91%、至少约92%、至少约93%、至少约94%、至少约95%、至少约96%、至少约97%、至少约98%、至少约99%)的序列同一性的核苷酸序列。在一些情况下,至少一种grna是crrna。在一些情况下,至少一种grna是sgrna。
95.对象和生物样品
96.对象可以提供用于遗传分析的生物样品。生物样品可以是由对象产生的任何物质。通常,生物样品是取自对象的任何组织或由对象产生的任何物质。生物可以是体液,例
如血液(例如全血、血浆、血清)、尿液、唾液、骨髓、脊髓液、痰、腹水、淋巴液、胸膜液、羊水、精液、阴道分泌物、汗液、粪便、腺体分泌物、眼液、母乳等。生物样品可以是细胞和/或固体组织(例如,脸颊组织(例如,来自脸颊拭子)、粪便、皮肤、毛发、器官组织等)。在一些情况下,生物样品是实体瘤或实体瘤的活检。在一些情况下,生物样品是福尔马林固定的石蜡包埋的(ffpe)的组织样品。生物样品可以是包含基因组dna的任何生物样品。
97.生物样品可以来自对象。对象可以是哺乳动物、爬行动物、两栖动物、鸟类或鱼类。哺乳动物可以是人、猿、猩猩、猴子、黑猩猩、牛、猪、马、啮齿类动物、鸟、爬行动物、狗、猫或其他动物。爬行动物可能是蜥蜴、蛇、短嘴鳄、乌龟、鳄鱼和乌龟。两栖动物可以是蟾蜍、青蛙、蝾螈和火蜥蜴。鸟类的实例包括但不限于鸭、鹅、企鹅、鸵鸟和猫头鹰。鱼类的实例包括但不限于鲶鱼、鳗鱼、鲨鱼和箭鱼。优选地,对象是人。对象可能患有疾病或病况。可以为对象开具治疗剂。治疗剂可以是由cyp2d6激活和/或代谢的治疗剂。
98.本公开的系统
99.本文进一步提供了用于执行本文提供的方法的系统。在一个方面,提供了一种系统,其包括用于分析感兴趣的复杂的基因组区域的系统,所述系统包括:(a)至少一个存储单元,其被配置为接收数据输入,该数据输入包括由包括以下步骤的方法产生的数据:(i)从包含感兴趣的复杂的基因组区域的基因组dna中分离高分子量dna;(ii)使基因组dna与规律间隔性成簇短回文重复序列(crispr)相关核酸内切酶和两种或更多种grna以切除感兴趣的复杂的基因组区域,其中两种或更多种grna各自包含与基因组中存在的不同核苷酸序列基本上互补的核苷酸序列,并且其中不同核苷酸序列位于感兴趣的复杂的基因组区域的侧翼;以及(iii)分析感兴趣的复杂的基因组区域以产生数据,其中该方法不涉及dna扩增;和(b)可操作地耦合到至少一个存储单元的计算机处理器,其中该计算机处理器被编程为基于所述数据产生输出。
100.在各个方面,输出是报告。在各个方面,输出是感兴趣的复杂的基因组区域的基因型。在各个方面,输出是感兴趣的复杂的基因组区域的基因序列。在各个方面,输出是感兴趣的复杂的基因组区域的结构分析。在各个方面,分析包括对感兴趣的复杂的基因组区域进行基因分型。在各个方面,分析包括对感兴趣的复杂的基因组区域进行结构分析。在各个方面,分析包括对感兴趣的复杂的基因组区域进行测序。
101.在另一个方面,提供了一种系统,其包括用于鉴定对象的cyp2d6的遗传变异,所述系统包括:(a)至少一个存储单元,其被配置为接收数据输入,该数据输入包括由包括以下步骤的方法生成的测序数据:(ii)将获自对象的基因组dna与规律间隔性成簇短回文重复序列(crispr)相关核酸内切酶和两种或更多种grna接触,以切除包含cyp2d6、cyp2d7和cyp2d8的遗传基因座;以及(iii)对遗传基因座进行长读段测序以产生测序数据;以及(b)可操作地耦合到至少一个存储单元的计算机处理器,其中该计算机处理器被编程为基于测序数据产生输出。
102.在各个方面,输出是报告。在各个方面,输出鉴定cyp2d6中的遗传变异。在各个方面,输出鉴定cyp2d6功能的降低、丧失或增加。在各个方面,报告基于遗传变异向对象推荐治疗。在各个方面,该报告基于遗传变异向对象推荐治疗剂的剂量。在各个方面,报告基于遗传变异推荐改变治疗剂的剂量。在一些情况下,治疗剂是由cyp2d6激活或代谢的治疗剂。
103.本公开进一步提供了用于执行本文所述的方法的基于计算机的系统。在一些方
面,该系统可用于分析由本文提供的方法产生的数据。该系统可以包括一个或多个客户端组件。一个或多个客户端组件可以包括用户界面。该系统可以包括一项或多项服务器组件。服务器组件可以包括一个或多个存储单元。一个或多个存储单元可以被配置为接收数据输入。数据输入可以包括测序数据。测序数据可以从来自对象的核酸样品(例如,基因组dna)产生。已经描述了适合与本公开的系统一起使用的测序数据的非限制性实例。该系统还可以包括一个或多个计算机处理器。一个或多个计算机处理器可操作地耦合到一个或多个存储单元。可以对一个或多个计算机处理器进行编程以生成用于在屏幕上显示的输出。输出可以包括一份或多份报告。
104.本文所述的系统可以包括一个或多个客户端组件。一个或多个客户端组件可以包括一个或多个软件组件、一个或多个硬件组件或其组合。一个或多个客户端组件可以通过一项或多项服务器组件访问一项或多项服务。一个或多个客户端组件可以通过网络访问一项或多项服务。网络可以是互联网、因特网和/或外联网或与互联网通信的内联网和/或外联网。在一些情况下,网络是电信网络和/或数据网络。网络可以包括一个或多个计算机服务器,其可以实现分布式计算,例如云计算。在一些情况下,借助计算机系统,网络可以实现点对点网络,可以使耦合到计算机系统的装置能够充当客户端或服务器。
105.系统可以包括一个或多个存储单元(例如,随机存取存储器、只读存储器、闪存)、电子存储单元(例如,硬盘)、用于与一个或多个其他系统通信的通信接口(例如,网络适配器)和外围装置,例如高速缓存存储器、其他存储器、数据存储和/或电子显示适配器。内存、存储器、接口和外围装置通过诸如主板的通信总线与cpu通信。存储器可以是用于存储数据的数据存储器(或数据存储库)。在一个实例中,一个或多个存储单元可以存储接收的测序数据。
106.系统可以包括一个或多个计算机处理器。一个或多个计算机处理器可操作地耦合到一个或多个存储单元,以例如访问所存储的数据。一个或多个计算机处理器可以执行机器可执行代码,以进行本文所述的方法。
107.可以以软件的形式提供机器可执行或机器可读代码。在使用过程中,可以由处理器执行代码。在一些情况下,可以从存储单元中检索代码并将其存储在存储器中以供处理器随时访问。在一些情况下,可以排除电子存储单元,并将机器可执行指令存储在存储器中。
108.代码可以被预编译并配置用于与具有适于执行该代码的处理器的机器一起使用,可以在运行时编译或者可以在运行时解释。可以选择编程语言来提供代码,以使能够以预编译、编译后或解释的方式执行代码。
109.可以在编程中体现本文提供的系统和方法的方面,例如计算机系统。技术的各个方面可以被认为是通常机器(或处理器)可执行代码和/或在某种类型的机器可读介质上承载或体现的相关数据的形式的“产物”或“制品”。机器可执行代码可以存储在电子存储器上,例如内存(例如,只读内存、随机存取内存、闪存)或硬盘上。“存储”型介质可以包括计算机、处理器等的任何或所有有形存储器或其相关模块,诸如可以在任何时刻为软件编程提供非暂时性存储的各种半导体存储器、磁带驱动器、磁盘驱动器等。整个软件或部分软件可以有时通过因特网或各种其他电信网络进行通信。这样的通信例如能够将软件从一台计算机或处理器加载到另一台计算机或处理器,例如,从管理服务器或主机加载到应用服务器
的计算机平台。因此,可以承载软件元素的另一类型的介质包括光波、电波和电磁波,诸如跨本地设备之间的物理接口、通过有线和光陆线网络以及通过各种空中链路而使用的光波、电波和电磁波。携载这样的波的物理元件,诸如有线或无线链路、光学链路等,也可被认为是承载软件的介质。如本文所使用,除非限制于非暂时性、有形“存储”介质,否则诸如计算机或机器“可读介质”的术语是指参与向处理器提供指令以供执行的任何介质。
110.因此,诸如计算机可执行代码等机器可读介质可以采取许多形式,包括但不限于有形存储介质、载波介质或物理传输介质。非易失性存储介质例如包括光盘或磁盘,诸如任何计算机等中的任何存储装置,例如可以用于实现附图中所示的文库等。易失性存储介质包括动态存储器,诸如这样的计算机平台的主存储器。有形传输介质包括同轴线缆;铜线和光纤,包括在计算机系统内包含总线的电线。载波传输介质可以采取电信号或电磁信号的形式或者采取声波或光波的形式,诸如在射频(rf)和红外(ir)数据通信期间生成的那些。因此,计算机可读介质的常见形式例如包括:软盘、柔性盘、硬盘、磁带、任何其他磁介质、cd-rom、dvd或dvd-rom、任何其他光介质、穿孔卡片纸带、任何其他具有孔洞图案的物理存储介质、ram、rom、prom和eprom、flash-eprom、任何其他存储器芯片或存储器盒、传输数据或指令的载波、传输这样的载波的线缆或链路或者任何其他计算机可从中读取编程代码和/或数据的介质。这些形式的计算机可读介质中的许多介质可以参与将一个或多个指令的一个或多个序列加载到处理器以供执行。
111.本文公开的系统可以包括一个或多个电子显示器或与一个或多个电子显示器通信。电子显示器可以是计算机系统的一部分或者直接或通过网络耦合到计算机系统。计算机系统可以包括用于提供本文公开的各种特征和功能的用户界面(u1)。ui的实例包括但不限于图形用户界面(gui)和基于网络的用户界面。ui可以提供交互工具,用户可以通过该工具利用本文描述的方法和系统。举例来说,如本文所设想的ui可以是基于网络的工具,医疗保健从业者可以通过该工具订购基因测试、定制要测试的遗传变体列表以及接收和查看报告。
112.本文公开的方法可以包括基于来自一个或多个数据库、一个或多个检测、一个或多个数据或结果、基于或源于一个或多个检测的一个或多个输出、基于或源于一个或多个数据或结果的一个或多个输出或其组合的数据和/或信息的生物医学数据库、基因组数据库、生物医学报告、疾病报告、病例对照分析和罕见变体发现分析。
113.如本文所述,一个或多个计算机处理器可以执行机器可执行代码,以进行本公开的方法。机器可执行代码可以包括任意数量的开源或闭源软件。可以执行机器可执行代码来分析数据输入。数据输入可以是从一个或多个测序反应产生的测序数据。该计算机进程可以可操作地耦合到至少一个存储单元。计算机处理器可以访问来自至少一个存储单元的数据(例如,测序数据)。在一些情况下,计算机处理器可以执行机器可执行代码以将测序数据映射到参考序列。在一些情况下,计算机处理器可以执行机器可执行代码以从测序数据确定遗传变体的存在或不存在。在一些情况下,计算机处理器可以执行机器可执行代码以生成在屏幕上显示的输出(例如,报告)。
114.机器可执行代码可以包括一种或多种算法。一种或多种算法可用于实现本公开的方法。
115.本公开的系统可以包括一个或多个计算机系统。图15示出了计算机系统1501(在
本文中也称为“系统”),该计算机系统1501被编程或以其他方式配置为实施本公开的方法,例如接收数据并基于所述数据产生输出。系统1501包括中央处理单元(cpu,本文也称为“处理器”和“计算机处理器”)1505,其可以是单核或多核处理器,也可以是用于并行处理的多个处理器。系统1501还包括存储器1510(例如,随机存取存储器、只读存储器、闪存)、电子存储器1515(例如,硬盘)、用于与一个或多个其他系统通信的通信接口1520(例如,网络适配器)和外围装置1525,例如高速缓存存储器、其他存储器、数据存储和/或电子显示适配器。内存1510、存储器1515、接口1520和外围装置1525通过诸如主板的通信总线(实线)与cpu1505通信。存储器1515可以是用于存储数据的数据存储器(或数据存储库)。系统1501借助通信接口1520可操作地耦合到计算机网络(“网络”)1530。网络1530可以是互联网、因特网和/或外联网或者与互联网通信的内联网和/或外联网。在一些情况下,网络1530是电信网络和/或数据网络。网络1530可以包括可以实现分布式计算,例如云计算的一个或多个计算机服务器。在一些情况下,借助系统1501,网络1530可以实现点对点网络,这可以使耦合到系统1501的装置能够充当客户端或服务器。
116.系统1501与处理系统1540通信。处理系统1540可以被配置为实施本文公开的方法,例如将测序数据映射到参考序列或将分类分配给遗传变体。处理系统1540可以通过网络1530或通过直接(例如,有线、无线)连接与系统1501通信。处理系统1540可以被配置用于分析,例如核酸序列分析。
117.如本文所述的方法和系统可以通过存储在系统1501的电子存储单元上(例如在内存1510或电子存储器1515上)的机器(或计算机处理器)可执行代码(或软件)来实现。在使用过程中,代码可以由处理器1505执行。在一些实例中,可以从存储器1515检索代码并将其存储在内存1510上以供处理器1505随时访问。在一些情况下,可以排除电子存储器1515,并且将机器可执行指令存储在内存1510上。
118.代码可以被预编译并配置为与具有适于执行该代码的处理器的机器一起使用,可以在运行时被编译或者可以在运行时被解释。可以选择编程语言来提供代码,以使代码能够以预编译、编译后或解释的方式执行。
119.本文提供的系统和方法的方面可以体现在编程中。技术的各个方面可以被认为是通常机器(或处理器)可执行代码和/或在某种类型的机器可读介质上承载或体现的相关数据的形式的“产物”或“制品”。机器可执行代码可以存储在电子存储器上,例如内存(例如,只读内存、随机存取内存、闪存)或硬盘上。“存储”型介质可以包括计算机、处理器等的任何或所有有形存储器或其相关模块,诸如可以在任何时刻为软件编程提供非暂时性存储的各种半导体存储器、磁带驱动器、磁盘驱动器等。整个软件或部分软件可以有时通过因特网或各种其他电信网络进行通信。这样的通信例如能够将软件从一台计算机或处理器加载到另一台计算机或处理器,例如,从管理服务器或主机加载到应用服务器的计算机平台。因此,可以承载软件元素的另一类型的介质包括光波、电波和电磁波,诸如跨本地设备之间的物理接口、通过有线和光陆线网络以及通过各种空中链路而使用的光波、电波和电磁波。携载这样的波的物理元件,诸如有线或无线链路、光学链路等,也可被认为是承载软件的介质。如本文所使用,除非限制于非暂时性、有形“存储”介质,否则诸如计算机或机器“可读介质”的术语是指参与向处理器提供指令以供执行的任何介质。
120.因此,诸如计算机可执行代码等机器可读介质可以采取许多形式,包括但不限于
有形存储介质、载波介质或物理传输介质。非易失性存储介质例如包括光盘或磁盘,诸如任何计算机等中的任何存储装置,例如其可以用于实现文库等。易失性存储介质包括动态存储器,诸如这样的计算机平台的主存储器。有形传输介质包括同轴线缆;铜线和光纤,包括在计算机系统内包含总线的电线。载波传输介质可以采取电信号或电磁信号的形式或者采取声波或光波的形式,诸如在射频(rf)和红外(ir)数据通信期间生成的那些。因此,计算机可读介质的常见形式例如包括:软盘、柔性盘、硬盘、磁带、任何其他磁介质、cd-rom、dvd或dvd-rom、任何其他光介质、穿孔卡片纸带、任何其他具有孔洞图案的物理存储介质、ram、rom、prom和eprom、flash-eprom、任何其他存储器芯片或存储器盒、传输数据或指令的载波、传输这样的载波的线缆或链路或者任何其他计算机可从中读取编程代码和/或数据的介质。这些形式的计算机可读介质中的许多介质可以参与将一个或多个指令的一个或多个序列加载到处理器以供执行。
121.计算机系统1501可以包括电子显示器或与之通信,该电子显示器包括用户界面(ui)。ui的实例包括但不限于图形用户界面(gui)和基于网络的用户界面。
122.在一些实施方案中,系统1501包括向用户提供视觉信息的显示器。在一些实施方案中,显示器是阴极射线管(crt)。在一些实施方案中,显示器是液晶显示器(lcd)。在进一步的实施方案中,显示器是薄膜晶体管液晶显示器(tft-lcd)。在一些实施方案中,显示器是有机发光二极管(oled)显示器。在各种进一步的实施方案中,oled显示器上是无源矩阵oled(pmoled)或有源矩阵oled(amoled)显示器。在一些实施方案中,显示器是等离子显示器。在其他实施方案中,显示器是视频投影仪。在更进一步的实施方案中,显示器是诸如本文所公开的那些装置的组合。显示器可以向终端用户提供一个或多个生物医学报告,如通过本文所述的方法生成的生物医学报告。
123.在一些实施方案中,系统1501包括用于从用户接收信息的输入装置。在一些实施方案中,输入装置是键盘。在一些实施方案中,输入装置是定点装置,作为非限制性实例,包括鼠标、轨迹球、轨迹板、操纵杆、游戏控制器或触控笔。在一些实施方案中,输入装置是触摸屏或多点触摸屏。在其他实施方案中,输入装置是用于捕捉语音或其他声音输入的麦克风。在其他实施方案中,输入装置是用于捕捉运动或视觉输入的摄像机。在更进一步的实施方案中,输入装置是诸如本文所公开的装置的组合。
124.系统1501可以包括或可操作地耦合到一个或多个数据库。数据库可以包括基因组、蛋白质组、药物基因组、生物医学和科学数据库。数据库可以是公开可用的数据库。备选地或附加地,数据库可以包括专有数据库。数据库可以是商业可用的数据库。数据库包括但不限于mendeldb、pharmgkb、varimed、regulome、curated breakseq junctions、在线人类孟德尔遗传数据库(omim)、人类基因组突变数据库(hgmd)、ncbi dbsnp、ncbi refseq、gencode、go(基因本体论)和京都基因和基因组百科全书(kegg)。
125.可以在包含与数据用户相同的国家/地区的地理位置产生和/或传输数据。例如,可以从一个国家的地理位置产生和/或传输数据,并且数据用户可以存在于不同的国家。在一些情况下,本公开的系统访问的数据可以从多个地理位置之一传输到用户。数据可以在多个地理位置之间例如通过网络、安全网络、不安全网络、互联网或内联网来回传输。实施例
126.给出以下实施例的目的是为了说明本公开的各种实施方案,并不意味着以任何方
式限制本公开。本实施例连同本文描述的方法目前代表优选的实施方案,是示例性的,并不旨在限制本公开的实施方案的范围。本领域技术人员将想到包含在由权利要求的范围限定的本公开的精神内的变化和其他用途。
127.实施例1.
128.cyp2d6和临床测试
129.cyp2d6遗传结构:cyp2d6是一个小基因(4382bp),有九个外显子。然而,由于该基因座内存在高度相似的非功能性cyp2d7和cyp2d8假基因,因此该高度多态的基因座的遗传分析是困难的,如图1所示。cyp2d6和cyp2d7之间的相似性以及大重复区域的存在不仅产生了基因缺失和基因重复,而且还产生了包含3'cyp2d7与5'cyp2d6或3'cyp2d6和5'cyp2d7的复杂基因杂合体。目前,需要多种测试方法来检测这些结构变体的存在。
130.当前的测试平台:一种分析cyp2d6的常用方法是对长程、等位基因特异性pcr产物进行序列分析。简而言之,等位基因特异性引物用于扩增被靶向的区域。在pcr产物上发现的单核苷酸变体(snv)代表该等位基因的单体型。等位基因特异性扩增子也可以从重复的基因拷贝和cyp2d6-2d7和cyp2d7-2d6杂合基因中产生。最近,诸如单分子实时(smrt)测序或纳米孔测序的长读段测序技术也已用于更准确地表征cyp2d6单体型;然而,长读段cyp2d6测序的文库生成仍然存在局限性。目前用于生成用于测序的cyp2d6模板的xl-pcr反应受到可生成产物大小的限制,具有引物特异性,并不能捕获复杂的杂合体或许多已知的cnv,除非先前已对变体进行了表征并且已知存在于感兴趣的样品中。
131.综上所述,cyp2d6是一种高度多态的基因,直接参与了所有处方药中~25%的代谢。基因的遗传变异,包括拷贝数的变化,可直接影响患者的药物代谢状态。包括拷贝数的准确基因型至关重要,目前的方法不能完全测定基因区域的复杂性。
132.本文提出了一种利用crispr/cas9技术和位点特异性接头连接结合长读段测序来开发用于cyp2d6分析的诊断质量方法的方法。该方法利用单个样本不可知的crispr切割步骤来分离整个cyp2d6基因座以进行长读段测序。该方法能够准确检测单核苷酸多态性(snp)和cnv,并尽可能地分配最准确、定相的cyp2d6基因型和代谢状态。
133.crispr技术可用于在体外和体内靶向和切除基因组感兴趣的区域(roi)。简而言之,crispr-c相关蛋白9(cas9)在与合成生成的靶特异性指导rna(sgrna)复合时,会在与指导rna的靶特异性序列互补的序列处产生双链切割。通过设计sgrna靶向roi两端的序列,crispr-cas9可用于切除长度可达兆碱基的dna。
134.长读段测序:虽然短读段新一代测序(ngs)的发展使人类遗传学发生了革命性的变化,但其局限性也是公认的。分离的hmw dna片段的长读段测序最近引起了人们的兴趣,因为它可以让人们获得定相信息,鉴定小的结构变异并更好地组装包括串联重复的基因组的高复杂性区域。使用crispr技术以靶向特异性的方式分离dna片段,为靶向基因组的相关区域提供了创新且巧妙的方法,以进行长读段测序。
135.get-rm同期群:作为系统地表征cyp2d6基因结构的主要工作的部分,已提供cyp2d6基因分型数据,以建立用于测定开发、验证、质量控制和熟练测试的一套最先进的、表征良好的参考材料。这项工作是与基于疾病控制和预防的遗传测试参考材料协调计划中心、科里尔医学研究所以及其他pgx社区成员的遗传测试参考材料协调计划(get-rm)合作进行的。作为这项研究的一部分,提供了在几个包含复杂结构排列和/或罕见cyp2d6基因型
的样品上的pharmacoscantm基cyp2d6的基因分型。该数据与基于xl-pcr的ngs分析相结合,用于用当前的分析方法确定这些样品的最准确的基因型。所有细胞系和共有基因分型和注释数据的信息为验证提议的新测序和分析方法奠定了基础。
136.研究设计和方法
137.目标1(方法开发):(a)优化特定crispr/cas9方法,以生产包含cyp2d6-d7遗传基因座的高分子量dna区段,用于后续人类基因组dna(例如,血液样品)中的大小分析(例如,凝胶)。(b)分离/富集被靶向的区域和生成用于测序的xl文库。(c)建立用于cyp2d6-d7遗传基因座(例如,pacbio、minion)中的基因组变体的长模板测序的ngs方法。在图2中描绘了所提出的工作流程的概要。
138.hmw dna的分离:roi(cyp2d6和cyp2d7)的正常长度为28-35kb。为确保整个roi对于下游分析是完整的,使用基因组dna和rna纯化系统开发了分离高分子量gdna(高达70kb)的方案。与使用其他方法观察到的10kb-50kb范围相比,修改后的方案能够提取分子量》50kb的gdna(图3)。
139.高度特异性sgrna的设计和验证:由于cyp2d6基因座的复杂性和高度多态性,传统的基于pcr和阵列的技术需要多种测试来进行cnv和snp分析。仅针对cyp2d6基因的crispr cas9方法无法捕获包含结构变异的等位基因,例如d6/d7杂交等位基因或cyp2d6重复事件。为了克服这一限制,确定了位于包含cyp2d6和cyp2d7的区域侧翼的独特序列。通过设计靶向这些独特区域的sgrna,进行了一个crispr/cas9切割反应以分离整个cyp2d6/cyp2d7区域(图4a)。
140.为了确认sgrna的特异性和功效,从gdna生成包含被靶向的sgrna结合位点的xl-pcr产物。xl-pcr产物与cas9且无sgrna(图4b,样品a)或cas9且不同的sgrna(图4b,样品b和c)一起孵育。所有与cas9和sgrna一起孵育的pcr产物都被切割以产生预期大小的dna片段,但不同的sgrna显示出不同程度的切割效率。
141.切割基因组dna中的cyp2d6-cyp2d7位点:sgrna必须高效且特异性地与可能包含脱靶识别位点的gdna结合。为了询问crispr切割效率和特异性,基因组dna与cas9且无sgrna(阴性对照)或cas9且切割cyp2d6的5'和cyp2d7的3'的两个sgrna池一起孵育。用位于每个预测的切割位点侧翼的引物进行pcr反应。如果sgrna与正确的结合位点结合并发生切割,则会预期pcr产物减少。实际上,这是所观察到的(图5a,图5b)。还使用sgrna结合位点内部的引物对cyp2d6基因座进行pcr,以确定cas9介导的脱靶切割是否发生在cyp2d6基因内。没有观察到cyp2d6内的脱靶切割的证据(图5a,图5b)。
142.总之,通过xl-pcr和基因组dna质询证明,cas9-sgrna复合物高效且在基因座内没有显著脱靶活性地切割被靶向的cyp2d6-cyp2d7基因座的两侧。切割产生预测的28kb片段,可用于富集后的下游长读段ngs。
143.实施例2.crispr/cas9方法的进一步优化
144.开发和测试了其他sgrna和cas酶。使用标准软件鉴定并设计如上所述测试的sgrna。目标是获得在roi处以高效率和特异性切割的sgrna。优先考虑较短的dna片段,这些片段仍包含完整的roi。较短的片段可能具有降低的测序和处理成本的好处。还尝试用crispr cas12a酶切割相同区域。cas12a核酸内切酶的功能与cas9相似,但具有不同的pam序列要求(tttv),并在切割后产生5'交错悬突。相反,cas9产生平末端。这对后续步骤很重
要。
145.实施例3.基因组dna中cyp2d6-cyp2d7位点的富集
146.作为概念验证,如上所述,使用cas9-sgrna靶向切割位点cyp2d6的5'和cyp2d7的3'切割5μg的gdna。使用0.75%琼脂糖凝胶盒在bluepippen(sage science)仪器上运行切割的dna,这能够在1-50kb范围内进行大小选择。使用pcr确认洗脱的样品含有期望的cyp2d6-cyp2d7基因座。虽然这种基于凝胶的方法可以分离hmw样品,但也存在一些缺点,包括时间(每次blue pippen运行~10-12小时)、有限的样品数量(每次运行4-5个样品)、显著的材料损失/不良的回收率低且每个样品的高成本(~$50.00)。
147.为了克服这些限制,测试了几种靶富集方法。这能够确定各种方法的优缺点,并最终确定针对进一步临床试验开发的最适合方法。这是临床诊断测试开发的典型方法。下面对长读段测序的讨论是指牛津纳米孔(ont)测序;然而,任何提案都可以经过少量修改来适应pacbio测序要求。
148.方法1:靶的无扩增富集
149.dna制备:这种无扩增文库制备方法涉及dna样品的去磷酸化和3'末端加帽,然后是crispr处理和位点特异性ont接头连接。第一步,用虾碱性磷酸酶处理gdna,去除dna片段5'末端的磷酸基团,以及向3'末端添加单个胸苷双脱氧核苷酸的末端转移酶。此步骤确保gdna末端无法连接。然后用crispr cas9:grna复合物处理dna,产生平端~28-35kb cyp2d6/cyp2d7片段(详见前面的段落)。随后是“a加尾”步骤,在该步骤中,使用dna聚合酶将腺苷核苷酸添加到dna的游离3'末端(例如,未用ddttp加帽的末端)。最后,将带有胸苷悬突的ont接头添加到dna中。只有由crispr-cas9切割产生的dna末端与接头连接,因为它们是唯一具有互补3'-悬突和5'-磷酸基团的末端。
150.测序:生成的文库直接在ont仪器上进行测序。如果通过这种方法生成的dna文库的数量证明对ont测序具有挑战性,则这可以通过在测序前对样本进行多路复用和/或通过增加输入gdna数量来克服。此外,可以通过用核酸外切酶(ont接头对核酸外切酶iii和λ核酸外切酶具有抗性)处理样品来降低背景,这会导致所有背景dna的降解。
151.方法2:使用体外转录进行富集
152.基本原理:如果前述方法未能产生足够的dna或有过量的背景dna过多,则评估另一种方法,即通过体外转录(ivt)来进行被靶向的扩增。ivt与pcr相比有一些优势。(1)转录不太可能传播错误。(2)转录可以产生比大多数长程pcr产物的大小长的长度长达20-30kb的rna分子。
153.dna制备:在crispr切割后,用核酸外切酶处理dna以产生交错末端,并将含有t7启动子和与cyp26-cyp2d7基因座的交错末端互补的悬突的双链dna片段连接到靶片段。dna聚合酶和dna连接酶用于填充间隙并密封任何切口。噬菌体t7 rna聚合酶能够产生长达~20kb的转录本。由于启动子连接到~28kb基因座的两端,因此由t7 rna聚合酶从基因座末端的启动子产生的最长转录本可能足够长以覆盖整个区域。但是,大部分t7产物的长度通常小于4kb。最近发现的syn5噬藻体rna聚合酶能够产生长达30kb的转录本。一起测试syn5启动子与t7启动子。
154.体外转录:使用t7和syn5 rna聚合酶进行ivt。前一种酶是市售的,而后一种酶已在我们实验室中被表达和纯化。有几种商业t7rna聚合酶ivt试剂盒经过优化以产生长rna
转录本。先前的工作表明,随机插入人类基因组中的t7启动子序列在ivt期间产生大于5kb的rna转录本的很大部分。总rna产量、大转录本(》15kb)的比例和错误率是确定哪种聚合酶和ivt方法是更好选择的关键因素。因为可能会产生大范围的rna转录本长度,所以spri珠子可用于选择最大的转录本。在ont仪器上对rna进行直接测序。
155.方法3:用于体外转录的启动子多位点引入
156.基本原理:如果上述方法不够,则将t7或syn5启动子插入被靶向的区域的多个位点。这种方法的一个潜在问题是基因座的片段化使得将变体明确分配给cyp2d7或cyp2d6(因为基因和假基因共享~94%的序列同一性)并获得定相信息具有挑战性。为了克服这一限制,使用多个交错的插入位点来生成重叠片段。
157.启动子的引入:crispr切割发生在roi侧翼位点和基因座内规则间隔(~10kb)的位点。切割是在两个独立的反应中进行的,每个反应都有一组不同的靶位点,使得所产生的重叠片段可用于在测序后将读段缝合在一起。上文描述了核酸外切酶处理、含有启动子的接头的连接、ivt和cdna合成。包含启动子的接头在紧靠启动子下游包含短的固定序列。在进行cdna合成时,使用与该固定序列互补的引物进行逆转录(rt)。如果ivt产生的rna跨越两个插入位点之间的长度,则对该序列具有特异性的rt引物选择跨越相同区域的cdna分子。
158.潜在的替代方案:如有必要,可以使用每个ivt产物开头的固定序列使用几次长程pcr循环,以选择性地扩增跨越插入位点的cdna分子。
159.潜在的替代方案:通过ont进行的rna测序需要大量的rna。如有必要,使用与转录开始相距远(15-20kb)的位点退火的引物进行cdna合成,以选择长转录本。如果很大比例的测序读段没有映射到靶基因座,则将尝试防止接头连接到非靶位点。在crispr处理之前的gdna去磷酸化以及用所谓的“哑铃”接头对gdna的末端进行加帽是两种可能的选择。
160.实施例4.建立用于变体的长模板测序的ngs方法
161.方法:目前有两个主要的适合开发潜在的诊断测试的商业平台。pacbio一直是第一个也是最突出的长读段测序技术,但相关成本很高。最近,纳米孔测序技术已成为一种具有成本效益且潜在可行的平台。牛津纳米孔(oxford nanopore,ont)作为平台在吞吐量、成本和准确性方面不断成熟。考虑到这些优势,本文关注于ont。然而,所提出的方法学和方法在很大程度上与平台无关,并且可以修改以适应两个当前或未来的长读段平台中的任何一个。在牛津纳米孔minion上进行测序运行。
162.目标2(验证):(a)使用当前用于长读段序列比对的软件和平台进行序列分析,以进行变体识别、cnv分析和定相。(b)将cyp2d6-d7长读段序列分析结果与序列/拷贝数变异进行比较,并对共有基因分型和注释结果与来自get-rm项目的结果进行表征,以评估性能特征并指导进一步的诊断测试开发。相对于时间效益和成本效益、所需步骤的最小化和结果的质量对每种方法的可行性进行测试和比较。总体目标是选择最合适的方法来对整个cyp2d6基因进行分离、富集和测序。
163.选择用于验证的样品:一旦开发了样品制备方法,将分析具有已知基因型和单体型的额外样品的扩展组。具有复杂结构(例如重复段、杂交、选定的缺失和复杂的重排)的样品被包括在内,以便在扩展的数据集上评估平台。样品选自get-rm项目(见上文,“get-rm同期群”)。这些细胞系和数据提供了独特的资源,因为它们能够针对当前的黄金标准评估新
的长读段序列数据。对于该提议,已获得这些细胞系的子集——lcl细胞系。从细胞系储库和通过现有合作获得了用于表征其他相关变体和单体型的额外样品。为了使用额外样品进一步验证该方法,使用来自nist coriell同期群的额外细胞系,该同期群被广泛地表征,包括全基因组测序。此外,还获得了代表典型诊断样本的其他样品类型,包括全血和唾液。在这个目标中,总共选择了48个细胞系进行测序,代表重复段、缺失、杂交和串联排列。对总共96个测序样品进行一式两份地分析。
164.变体识别、cnv识别和定相:使用专门为长读段ont数据开发的软件包。clair是clairvoyante的最新版本,是用于预测变体类型、接合性、替代等位基因和插入/缺失长度的多任务五层卷积神经网络模型。最近开发的另一个软件包是megalodon。megalodon的功能集中在将高信息神经网络碱基识别锚定到参考序列上。bowden等人最近使用标准参考样品针对全基因组测序评估了纳米孔技术的性能特征。82倍覆盖率的共识准确率为99.9%,尽管该数据也显示了该平台当前的一些局限性。由于该提案仅对小的被靶向的区域进行测序,并且鉴于能够在超高深度对该区域进行测序,因此预期当前的分析平台可以产生被靶向的序列的足够准确的数据。还监控未来的软件开发,并在新方法可用时使用它们。
165.与共识数据的比较:将数据与get-rm共识结果(基于所有平台的结果以及专家小组对变体的审查)进行比较。确定单体型识别snp和cnv的一致性,评估鉴定杂交单体型的序列特征的能力,并测量确定代谢状态的一致性。接下来,将其他变体与来自get-rm项目的基因分型数据进行比较。结合定相信息(例如,确定的单体型)分析数据以确定定相基因分型数据是否与结果一致,因为这提供了非估算的定相信息。最后,鉴定了仅通过测序鉴定的任何其他变体。还进行了cyp2d6与其假基因之间序列相似性的探索性序列比较。
166.预期问题:一个问题与测序平台的整体准确性有关。最初的方法是在超高深度进行测序。这种方法应该能够确定非系统测序错误,但更难以确定由于平台的技术限制而导致的固有错误。与cyp2d6参考样品的共识数据的比较能够评估这种影响。此外,预期对ont平台的进一步基准研究和改进的序列分析方法增加长读段数据的序列注释。
167.未来方向:在药物遗传学中,cyp2d6是最广泛测试的基因之一,同时对使用当前的测试技术进行分析在技术上具有挑战性。最终目标是开发可以替代当前不完整且容易出错的平台的统一的临床测试方法。该应用程序作为概念验证证明,基于crispr的序列靶向、创新的片段富集和长读段测序是可行的方法。
168.实施例5.
169.用于分析的特定遗传基因座的靶向
170.与如pcr或寡核苷酸杂交的传统方法相比,这种方法使用crispr/cas9系统和基因座特异性指导rna,只对感兴趣的区域(roi)进行被靶向的切割。富集区域选择和sgrna设计的新方法能够捕获包括高度相似的假基因和重复区域的整个基因座,这种区域的一个实例显示在图1中。
171.当前问题
172.常见的dna提取方法和对包含重复区域(例如,rep6等)并与相邻假基因具有高度序列相似性的诸如cyp2d6的高度多态性基因的测序方法有许多弱点。这些问题包括pcr引入的错误、用pcr可捕获的大小的限制、脱靶阵列杂交、需要多种测定(例如,使用qpcr进行测序 cnv分析)、脱靶比对、缺乏变体定相和高金钱时间成本。图6突出显示了6个ngs测序的
传统制备文库实施例的igv比对。这些文库(a-f)由cyp2d6长程pcr(xl-pcr)扩增子生成。在ngs分析之前,扩增子经历了片段化(100-300bp)、接头连接和pcr扩增。这种方法有几个限制。首先,如cyp2d6所示,为了扩增每个样品中的cyp2d6基因,必须在xl-pcr之前了解cyp2d6的拷贝数状态以及是否存在杂合等位基因。每个都必须使用正常、重复段、缺失和杂交等位基因的特异性引物。这需要在ngs之前进行额外的拷贝数测定。此外,xl-pcr扩增时间通常为0.5至1小时/kb长度的靶扩增子。
173.短读段序列数据的分析也受到定相能力降低的阻碍,并且易于与高度相似的假基因或同源区域脱靶比对,例如图1中所示的cyp2d6和94%相似的cyp2d7假基因。此外,同一基因的不同单体型可能与假基因具有不同程度的相似性,并且变体可能无法正确比对。
174.与传统的基于pcr的方法相比,无pcr文库具有显著优势。无pcr文库消除了引入pcr衍生序列错误的可能性,并克服了当前最大pcr产物大小的限制。消除了xl-pcr反应时间,代表了显著时间缩短,并且该方法允许杂合变体定相和检测拷贝数变异(cnv)。
175.sgrna的设计
176.如上所示,由于cyp2d6基因座的复杂性和高度多态性,传统的基于pcr和阵列的技术需要多种测定来执行cnv和snp分析。由于提取和样品处理过程中的dna剪切,为了将用于富集的完整靶区域的数量最大化,将直观地选择尽可能小的crispr/cas9靶区域来捕获感兴趣的基因。然而,仅靶向cyp2d6基因的crispr/cas9方法无法捕获包含结构变异的等位基因,例如d6/d7杂交等位基因或cyp2d6重复事件,这些等位基因至少占检测到的等位基因的20%。对适当的指导rna设计的高度复杂要求的实例显示在图7a-图7c中。
177.第一个设计限制是,将cas9复合物靶向roi的rna不能设计在cyp2d6基因本身附近。这是因为两个主要区域的。第一个原因是存在与cyp2d7不同的cyp2d6侧翼的独特序列的有限位点。那些包含不能很好地工作或能够捕获重要启动子区域变异的重复区域。第二个原因是,如果存在cyp2d6 cnv或d6/d7或d7/d6杂交等位基因,则存在额外的切割和准确的cnv分析和序列比对的能力的丧失(图7a)。图7b和图7c分别显示了在cyp2d7和cyp2d8附近切割的方法的类似限制。
178.为了克服这些限制,已经鉴定了位于包含cyp2d6、cyp2d7和cyp2d8的区域的侧翼的独特序列,并且仍然产生用于长程序列分析的适当大小的切割片段。通过设计靶向这些独特区域的sgrna,进行一次crispr/cas9切割反应,以分离整个cyp2d6/cyp2d7/cyp2d8区域(图8)。此外,根据下游应用,设计必须靶向正确的链( 或-),这取决于sgrna是靶向roi的5'末端还是3'末端。测试的sgrna序列的非限制性实例出现在下表2中。cyp2d6在-链上编码,而指导rna位置(上游或下游)是相对于 链而言的。具有较低染色体位置的序列被认为是更上游的,而具有较高染色体位置的序列被认为是下游的。表2.指导rna序列
179.sgrna性能分析和验证
180.为了确认sgrna的特异性和功效,从gdna生成了含有被靶向的sgrna结合位点的xl-pcr产物。xl-pcr产物与cas9 无sgrna(或脱靶sgrna)或cas9 感兴趣的sgrna一起孵育。图9a显示了代表性琼脂糖凝胶,其显示了在多个反应时间点两种不同的sgrna(t_1和t_2)的切割效率。与cas9和sgrna一起孵育的所有pcr产物都被切割以产生预期大小的dna片段,但不同的sgrna显示出不同程度的切割效率。
181.在确定xl-pcr扩增子的切割效率后,分析对基因组dna的切割效率。这是通过用特定sgrna进行cas介导的切割,然后对切割的dna进行定量pcr反应来完成的。引物设计在预测的sgrna靶切割位点的任一侧。在来自cas9反应或未切割对照的100ng总基因组dna上运
行pcr反应。如果dna在适当的位点被切割,与未切割的对照样品(例如,使用sgrna用于脱靶区域的cas9反应)中产生的pcr产物的量相比,将观察到pcr产物的减少。使用这种方法,确定了sgrna是否能够靶向基因组dna中所期望的roi,并确定了切割效率,如图9b和图9c所示。整个cyp2d6基因的xl-pcr显示切割和未切割对照之间没有差异。这表明跨反应在切割位点中观察到的pcr产物量减少不是由于dna的随机切割,而是这些特定区域的被靶向的cas9介导的切割。
182.高分子量(hmw)dna的分离
183.分离长区段(》50kb)中的高分子量基因组(hmw)dna允许在没有pcr扩增的情况下生成测序文库。如图10所示,使用nanobind ccb dig dna试剂盒(circulomics,madisonwi)从淋巴母细胞(18959和19213)内部提取hmw dna。提取的dna在2%琼脂糖凝胶上运行,并与λhindiii梯(上带23.l kb)、λdna(48.5kb)和先前从cornel研究所获得的提取的基因组dna(通过替代方法提取)进行大小比较。内部提取的dna的大小明显大于通过其他方法(例如coriell gdna 18996)提取的dna,大部分运行高于48.5kbλdna。使用short read eliminator kit(circulomics,madison wi)进一步富集高分子量dna。
184.crispr/cas9富集和文库制备
185.使用纳米孔cas介导协议(vnr_9084_v109_revk_04dec2018)的修改版本,使用上述sgrna进行crispr/cas9富集。对过程中使用的sgrna的体积和浓度进行了修改以实现最佳结果(具体而言,每个sgrna 33.3μl sgrna(3μm))。使用扩增子通过连接协议(sqk-lsk109)连接接头,并在minion测序平台(oxford nanopore,uk)上运行制备好的测序文库并进行数据分析。
186.概念证明
187.利用富集整个cyp2d6-cyp2d7-cyp2d8区域(chr22:42,122,115-42,161,317)的sgrna进行测序证实了3个关键点:(1)sgrna设计成功捕获了整个靶区域,(2)该策略允许显著富集脱靶读段的整个roi和(3)该方法能够成功地对整个roi(~~40kb)进行长读段测序。
188.如图11a所示,仅对含有被靶向的roi的染色体22(chr22)观察到全基因组显著序列富集。所有其他基因组区域显示最小的覆盖率。对chr22的进一步分析发现,仅含有roi的区域被富集并具有》10倍的覆盖率(图11b)。总的来说,映射到chr22的176个读段中有121个是与roi比对的全长读段(68.75%)。所有染色体22读段的每个读段的平均准确度和同一性显示在图11b中。
189.运行校准和时间
190.中位比对读段长度为~39.35kb(图12a),表明靶设计大小的成功测序和比对。值得注意的是,所有比对的读段都是在minion上测序的前2.5小时内捕获的(图12b)。这表明使用本文描述的方法的测序时间可以大大减少标准的长读段测序运行时间。这在结果出报告时间和仪器吞吐量方面都非常有价值。
191.igv分析
192.对序列数据比对的进一步igv分析表明,序列读段与正确的基因组位置(chr22:42,122,115-42,161,317)比对,并且在整个roi中具有均匀的深度和覆盖率。图13显示了与靶cyp2d6区域比对的121个38.5kb读段的igv比对。为了进一步审查该方法的特异性,进行
靶区域中的sgrna富集,但对相反的dna链( 或-)进行sgrna富集,并将序列数据比对与原始链设计上的sgrna富集进行了比较。如图14所示,取决于sgrna链靶,在roi中的cyp2d6-cyp2d7-cyp2d8区域(chr22:42,122,115-42,161,317-在图中以红色显示)或侧翼区域(以蓝色显示)产生100%的序列富集。根据设计的不同,没有观察到与脱靶区域侧翼的重叠。这表明了该方法的两个关键方面:(1)在我们的设计roi内没有产生明显的脱靶切割,(2)富集方法不会导致roi的明显剪切。
193.尽管本文中已经显示和描述了本公开的优选实施方案,但是对于本领域技术人员来说明显的是,这些实施方案仅作为示例提供。在不背离本公开的情况下,本领域技术人员现在将想到许多变化、改变和替换。应当理解,在实施本公开的实施方案时可以采用对本文描述的本公开的实施方案的各种替代。以下权利要求旨在限定本公开的范围,并且由此覆盖这些权利要求范围内的方法和结构及其等价物。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。