聚合酶
1.本技术是基于申请日为2015年11月13日,优先权日为2014年11月14日,申请号为201580066341.1,发明名称为:“聚合酶”的专利申请的分案申请。
2.发明背景
3.热稳定酶可用于以比最初存在的量大的量来扩增存在的核酸序列。引物、模板、三磷酸核苷酸,适当的缓冲液和反应条件以及聚合酶用于pcr过程中,所述pcr过程牵涉靶dna变性,引物杂交和互补链合成。每个引物的延伸产物成为产生所需核酸序列的模板。如果所使用的聚合酶是热稳定酶,由于热不会破坏聚合酶活性,则在每个变性步骤后不需要加入聚合酶。
4.先前的工作已经证明了从水生栖热菌(thermus aquaticus)中分离出热稳定性dna聚合酶,以及该聚合酶在pcr中的用途。尽管水生栖热菌dna聚合酶特别优选用于pcr和其他重组dna技术,但仍然需要其它热稳定聚合酶。
5.因此,本领域期望产生纯化的,热稳定dna聚合酶,其可用于改善上述pcr过程,和当在其他重组技术中(例如dna测序、切口平移,以及甚至逆转录中)使用热稳定dna聚合酶时,改善所获得的结果。
6.序列表
7.本技术与电子形式的序列表一并提交。序列表作为创建于2015年11月13日,名为ip1289.txt的文件提供,其大小为466kb。电子格式的序列表中的信息通过引用整体并入本文。
8.发明概述
9.本文呈现的是热稳定性dna聚合酶的方法和组合物,其可以用于改善pcr过程,和当将热稳定性dna聚合酶用于其他重组技术中(如dna测序、切口平移以及甚至逆转录中)时,其可用于改善所获得的结果。
10.因此,本文呈现的一个实施方案是包含seq id nos:1-66中任一个的氨基酸序列的重组dna聚合酶。
11.还在本文呈现的是重组dna聚合酶,其包含与seq id nos:1-66的任一个至少60%、70%、80%、85%、90%、95%、99%相同的氨基酸序列,并且其中所述重组dna聚合酶展示聚合酶活性。
12.还在本文呈现的是融合蛋白,其包含与另外的多肽融合的如上实施方案中任一个中定义的重组dna聚合酶。在一些实施方案中,融合到聚合酶的多肽结构域可以包含纯化标签、表达标签、溶解度标签或其组合。在一些实施方案中,融合到聚合酶的多肽结构域可以包含例如麦芽糖结合蛋白(mbp)。在一些实施方案中,融合到聚合酶的多肽结构域可以包含例如具有seq id no:72的氨基酸序列的前导肽。
13.还在本文呈现的是核酸分子,其编码如上述实施方案的任一个中定义的重组聚合酶。还呈现的是包含编码上述核酸分子的表达载体。还呈现的是包含上述载体的宿主细胞。
14.还在本文呈现的是将核苷酸掺入dna的方法,其包含允许以下组分相互作用:(i)根据上述实施方案中任一项的聚合酶,(ii)dna模板;和(iii)核苷酸溶液。
15.一个或多个实施方案的细节在附图和下面的描述中阐述。其他特征,目的和优点将从说明书和附图以及权利要求书中变得显而易见。
16.本发明包括以下内容:
17.实施方案1.重组dna聚合酶,其包含seq id nos:1-66的任一个氨基酸序列。
18.实施方案2.重组dna聚合酶,其包含与seq id nos:1-66的任一个氨基酸序列至少90%、95%、99%相同的氨基酸序列,并且其中所述重组dna聚合酶展示聚合酶活性。
19.实施方案3.根据实施方案1-2任一项的重组dna聚合酶,其中所述聚合酶还包含n末端前导肽。
20.实施方案4.根据实施方案3的重组dna聚合酶,其中所述前导肽包含his标签。
21.实施方案5.根据实施方案4的重组dna聚合酶,其中所述前导肽包含seq id no:72的氨基酸序列。
22.实施方案6.核酸分子,其编码如实施方案1-5任一项中定义的改变的聚合酶。
23.实施方案7.表达载体,其包含实施方案6的核酸分子。
24.实施方案8.宿主细胞,其包含实施方案7的载体。
25.实施方案9.将核苷酸掺入dna的方法,其包含允许以下组分相互作用:(i)根据实施方案1-5中任一项的聚合酶,(ii)dna模板;和(iii)核苷酸溶液。
26.实施方案10.根据实施方案1-5中任一项的重组dna聚合酶,其中所述聚合酶相较于vent dna聚合酶展示出增加的保真度。
27.实施方案11.根据实施方案1-5中任一项的重组dna聚合酶,其中所述聚合酶相较于vent dna聚合酶展示出增加的活性。
28.实施方案12.根据实施方案1-5中任一项的重组dna聚合酶,其中所述聚合酶相较于vent dna聚合酶展示出增加的活性和增加的保真度。
29.实施方案13.根据实施方案1-5中任一项的重组dna聚合酶,其中所述聚合酶相较于vent dna聚合酶展示出增加的活性和较低的保真度。
30.实施方案14.根据实施方案1-5中任一项的重组dna聚合酶,其中所述聚合酶相较于vent dna聚合酶展示出增加的保真度和较低的活性。
31.实施方案15.重组dna聚合酶,其包含以下氨基酸序列:seq id no:1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55或56。
32.实施方案16.重组dna聚合酶,其包含以下氨基酸序列的任一个:seq id nos:2、6、9、13、14、16、20、21、24、28、29、33、37、42、43、47、48、49、51、53、54、55、56、57、58、59、60、61、62、63、64、65和66。
33.实施方案17.重组dna聚合酶,其包含以下氨基酸序列的任一个:seq id nos:2、6、9、13、14、16、20、21、24、29、37、43、47、48、49、51、53、54、55、56、58、59、60、61、62、65和66。
34.实施方案18.根据实施方案15-17中任一项的重组dna聚合酶,其中所述聚合酶还包含n末端前导肽。
35.实施方案19.根据实施方案18的重组dna聚合酶,其中所述前导肽包含his标签。
36.实施方案20.根据实施方案19的重组dna聚合酶,其中所述前导肽包含seq id no:72的氨基酸序列。
37.附图简述
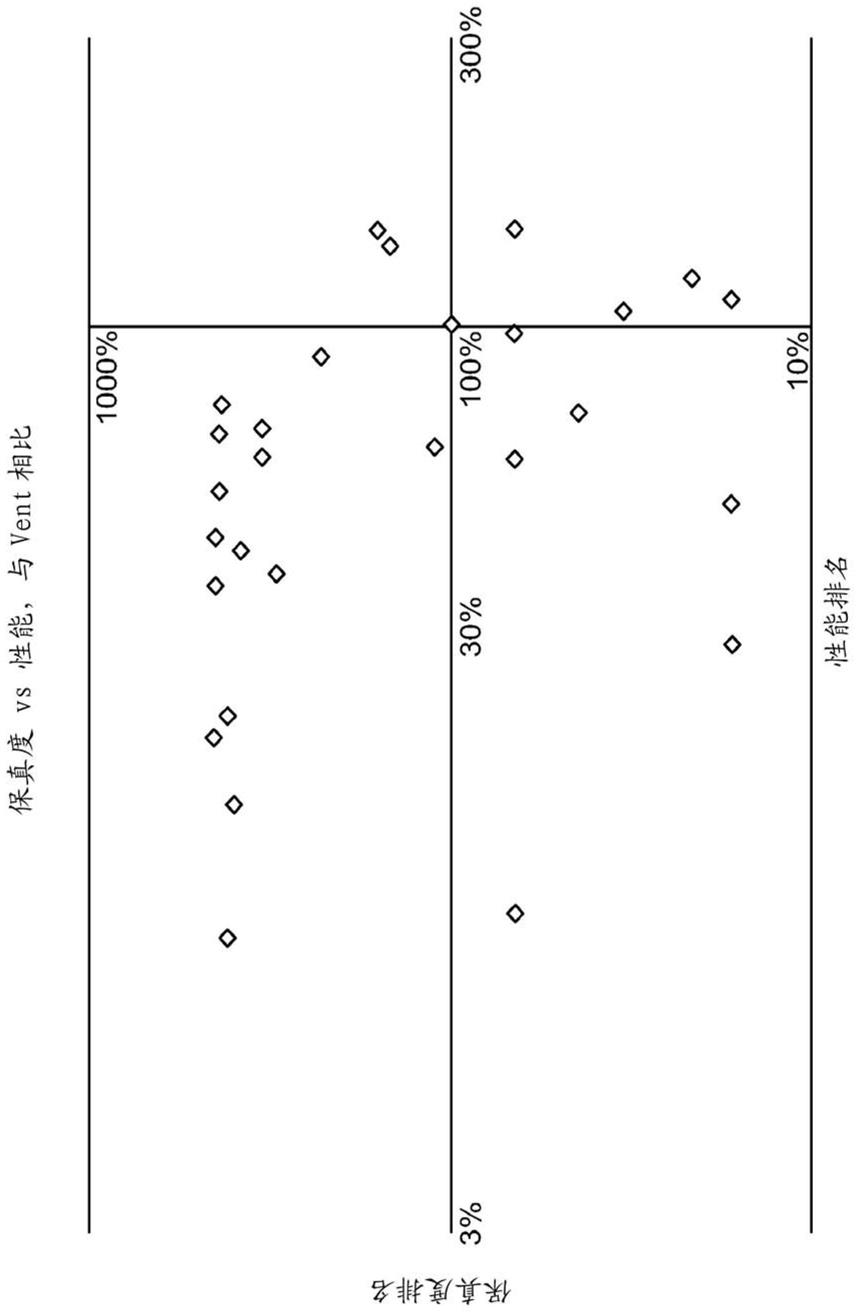
38.图1是显示与vent聚合酶相比,聚合酶保真度和性能的图。
39.发明详述
40.本文呈现的是热稳定性dna聚合酶的方法和组合物,所述热稳定性dna聚合酶可用于改善pcr过程,和当将热稳定性dna聚合酶用于其他重组技术中(如dna测序、切口平移以及甚至逆转录中)时,其可用于改善所获得的结果。本发明人令人惊讶地鉴定了展示改善性能的某些热稳定聚合酶。每种耐热聚合酶的氨基酸序列如seq id nos:1-66所示。
41.因此,本文呈现的一个实施方案是包含seq id nos:1-66中任一个的氨基酸序列的重组dna聚合酶。
42.本文中还呈现的是重组dna聚合酶,其包含与seq id nos:1-66的任一个的氨基酸序列至少60%、70%、80%、85%、90%、95%、99%相同的氨基酸序列,并且所述重组dna聚合酶展示聚合酶活性。本领域技术人员将理解,聚合酶中的一个或多个残基可以通过取代、缺失或添加进行修饰,同时保留聚合酶活性。因此,可以通过添加、缺失或取代一个或多个残基来修饰如seq id nos:1-66所示的聚合酶序列之一,并且仍保留聚合酶活性。可以根据本领域已知的多种方法中的任何一种来测量热稳定聚合酶的聚合酶活性,如以下公开中所例示的:美国专利号5,614,365;美国专利号7,598,032;美国专利号7,312,059;美国专利号6,395,524;美国专利号5,455,170;美国专利号5,405,774;美国专利号5,795,762;其每一个以其整体通过引用并入本文。
43.在一些实施方案中,一种或多种聚合酶(例如用于pcr的那些)的聚合酶活性在扩增条件下展示高性能。如本文所用,性能可以指对引发模板(primed template)的高亲和力和/或高延伸率,也称为持续合成能力(processivity)。如本文所用,“高持续合成能力”指持续合成能力高于20nts(例如,高于40nts、60nts、80nts、100nts、120nts、140nts、160nts、180nts、200nts、220nts、240nts、260nts、280nts、300nts、320nts、340nts、360nts、380nts、400nts或更高)与模板的每结合/离解(association/disassociation)。例如可以根据wo 01/92501a1中的方法测量持续合成能力,其通过引用以其整体并入本文。
44.pcr条件下的性能可以根据描述于elshawadfy et al.(2014)front.microbio.5:224的方法进行测定,或描述于arezi et al.(2014)front.microbio.5:408的那些进行测定,其中的每个以其整体通过引用并入本文。简言之,如elshawadfy等人所述,可以使用rotor-gene 6000热循环仪(corbett research,qiagen)进行qpcr。来自酿酒酵母(s.cerevesiae)的基因组dna可用作模板,其中靶向dna pol 2基因用于扩增。可以使用通用正向引物和一个或多个反向引物来扩增从145个到1040个范围的碱基对长度。在25μl中进行反应,其含有30ng酿酒酵母基因组dna(novagen),1μm每种引物,400μm每种dntp,和2.5μl的sybr green(10,000
×
储液,溶于二甲基亚砜,invitrogen;用水初始稀释1000倍)。通过在相同缓冲液中加入聚合酶(终浓度20nm)来引发反应用于引物-模板延伸。pcr由以下组成:1
×
95℃ 2分钟,然后40循环的:95℃(10s)/58℃(20s)/72℃(用于72℃延伸步骤的时间是可变的)。完成pcr后进行熔解曲线分析,其由以下组成:67℃下90s预熔解步骤,然后每秒增加0.2℃将温度升高至95℃。在1%琼脂糖凝胶(用溴化乙锭检测)上跑20μl的实时pcr混合物以验证扩增是否产生了正确大小的产物。
45.在一些实施方案中,可以使用pcr在高盐浓度和低盐浓度下测量性能。例如,盐中
的性能可以通过公开的示例进行测试,其通过引用以其整体并入本文。
46.在一些实施方案中,一种或多种聚合酶的聚合酶活性展示高保真度。如美国专利号8,481,685所述,并如本文所用,术语“保真度”指通过模板依赖性dna聚合酶的dna聚合的准确性。通常通过错误率(掺入不准确核苷酸的频率,即不以模板依赖的方式掺入的核苷酸)测量dna聚合酶的保真度。通过dna聚合酶的聚合酶活性和3'-5'核酸外切酶活性两者来维持dna聚合的准确性或保真度。术语“高保真度”指错误率低于4.45
×
10-6
(例如低于4.0
×
10-6
、3.5
×
10-6
、3.0
×
10-6
、2.5
×
10-6
、2.0
×
10-6
、1.5
×
10-6
、1.0
×
10-6
、0.5
×
10-6
)突变/nt/倍增(doubling)。可以使用本领域已知的测定法来测量dna聚合酶的保真度或错误率。例如,可以使用cline,j.et al.(96)nar 24:3546-3551中描述的laci pcr保真度测定法来测试dna聚合酶的错误率。简言之,在适当的pcr缓冲液中使用2.5u dna聚合酶(即在72℃,30min掺入25nmol总dntp所必需的酶量)从ppriaz质粒dna扩增编码laciolacza靶基因的1.9kb片段。然后将含有laci的pcr产物克隆到lambda gt10臂中,并且在彩色筛选测定中测定laci突变体的百分比(mf,突变频率),如(lundberg,k.s.,shoemaker,d.d.,adams,m.w.w.,short,j.m.,sorge,j.a.,and mathur,e.j.(1991)gene 180:1-8)所述。错误率表示为突变频率每复制(mf/bp/d),其中bp是lad基因序列中可检测位点的数量(349)并且d是有效目标倍增数。与上述类似,含有laciolacza靶基因的任何质粒可用作pcr的模板。pcr产物可以克隆入允许进行蓝/白色筛选的不同于lambda gt(例如,质粒)的载体中。
47.多年来在文献中已经描述了多种聚合酶保真度测定法,也许thomas kunkel(kunkel,t.a.and tindall,k.r.(1988)的最著名。kunkel方法使用m13噬菌体中的laczα基因的一部分将宿主细菌菌落颜色变化与dna合成中的错误相关联。wayne barnes建立在该测定的基础上,并利用pcr复制整个lacz基因和两个耐药性基因的部分,随后进行连接、克隆、转化和蓝/白色菌落颜色测定(barnes,w.m.(1992)gene,112,29
–
35)。在两种测定中,掺入lacz基因的错误引起β-半乳糖苷酶活性的破坏,导致白色菌落表型。使用这些基于lacz的实验方法,白色菌落的百分比必须转化为每个掺入碱基的错误数。作为更直接的保真度读出,单个克隆pcr产物的sanger测序也可以对dna聚合酶保真度评分,并提供将检测所有突变的优点。使用该方法,可以确定聚合酶的整个突变谱,并且不需要校正非表型变化。测量保真度的其他方法可以直接监控碱基的错误掺入。例如,使用高通量测序系统,还可以通过阅读扩增子的序列并与模板序列相比较,通过测量扩增期间积累的错误来测量保真度。
48.本文提供的一种或多种聚合酶的性质可以与本领域已知的一种或多种其它酶相比较。例如,在一些实施方案中,聚合酶的保真度可以与已知聚合酶如热稳定聚合酶的保真度相比较。在一些实施方案中,可以将聚合酶的持续合成能力与已知聚合酶如热稳定聚合酶的持续合成能力相比较。在一些实施方案中,聚合酶展示出相较于已知聚合酶增加的保真度。在一些实施方案中,聚合酶展示出相较于已知聚合酶增加的持续合成能力。在一些实施方案中,聚合酶展示出相较于已知聚合酶增加的保真度和持续合成能力。在一些实施方案中,聚合酶展示出相较于已知聚合酶增加的保真度但较低的持续合成能力。在一些实施方案中,聚合酶展示出相较于已知聚合酶增加的持续合成能力但较低的保真度。已知的dna聚合酶包括(例如)激烈火球菌(pyrococcus furiosus)(pfu)dna聚合酶(lundberg et al.,1991,gene,108:1),大肠杆菌dna聚合酶i(lecomte and doubleday,1983,nucleic acids res.11:7505),t7 dna聚合酶(nordstrom et al.,1981,j.biol.chem.256:3112),
嗜热栖热菌(thermus thermophilus)(tth)dna聚合酶(myers and gelfand 1991,biochemistry 30:7661),嗜热脂肪芽孢杆菌(bacillus stearothermophilus)dna聚合酶(stenesh and mcgowan,1977,biochim biophys acta 475:32),嗜热高温球菌(thermococcus litoralis)(tli)dna聚合酶(还称为vent dna聚合酶,cariello et al.,1991,nucleic acids res,19:4193),9
°
nm dna聚合酶(来自new england biolabs的停产产品)、海栖热袍菌(thermotoga maritima)(tma)dna聚合酶(diaz and sabino,1998braz j.med.res,31:1239),水生栖热菌(taq)dna聚合酶(chien et al.,1976,j.bacteoriol,127:1550),thermococcus kodakaraensis kod dna聚合酶(takagi et al.,1997,appl.environ.microbiol.63:4504),jdf-3dna聚合酶(专利申请wo 0132887),以及火球菌(pyrococcus)gb-d(pgb-d)dna聚合酶(juncosa-ginesta et al.,1994,biotechniques,16:820)。
49.在一些实施方案中,聚合酶展示出相较于嗜热高温球菌(tli)dna聚合酶(还称为vent dna聚合酶,cariello et al.,1991,nucleic acids res,19:4193)增加的保真度和/或持续合成能力。vent dna聚合酶可商购获得,并可用于本文所述的活性测定中作为聚合酶活性(例如保真度和/或持续合成能力)的基线测量。在一些实施方案中,聚合酶展示出相较于vent dna聚合酶的增加的持续合成能力。在一些实施方案中,聚合酶展示出相较于vent dna聚合酶的增加的保真度和持续合成能力。在一些实施方案中,聚合酶展示出相较于vent dna聚合酶的增加的保真度但较低的持续合成能力。在一些实施方案中,聚合酶展示出相较于vent dna聚合酶的增加的持续合成能力但较低的保真度。
50.制备和分离重组聚合酶
51.通常,编码本文所呈现的聚合酶的核酸可以通过克隆、重组、体外合成、体外扩增和/或其它可用的方法来制备。多种重组方法可用于表达编码如本文所呈现的聚合酶的表达载体。制备重组核酸,表达和分离表达产物的方法是本领域熟知和描述的。本文描述了许多示例性突变和突变的组合,以及设计所需突变的策略。制备和选择聚合酶活性位点突变的方法,包括修饰活性位点内或附近的空间特征以允许改进核苷酸类似物接近的方法可以在上文中以及例如wo 2007/076057和pct/us2007/022459中找到,其以它们的整体通过引用并入本文。
52.突变,重组和体外核酸操作方法(包括克隆,表达,pcr等)的其它有用参考文献包括berger和kimmel,guide to molecular cloning techniques,methods in enzymology volume 152academic press,inc.,san diego,calif.(berger);kaufman et al.(2003)handbook of molecular and cellular methods in biology and medicine second edition ceske(ed)crc press(kaufman);和the nucleic acid protocols handbook ralph rapley(ed)(2000)cold spring harbor,humana press inc(rapley);chen et al.(ed)pcr cloning protocols,second edition(methods in molecular biology,volume 192)humana press;以及viljoen et al.(2005)molecular diagnostic pcr handbook springer,isbn1402034032。
53.此外,许多试剂盒可商购获得用于从细胞中纯化质粒或其他相关核酸,(参见例如easyprep.tm.,flexiprep.tm.均来自pharmacia biotech;strataclean.tm.,来自stratagene;以及,qiaprep.tm.来自qiagen)。可以进一步操作任何分离和/或纯化的核酸
以产生其他核酸,其用于转染细胞,掺入相关载体以感染生物体用于表达和/或诸如此类。典型的克隆载体含有转录和翻译终止子,转录和翻译起始序列,以及可用于调节特定靶核酸表达的启动子。载体任选地包含通用表达盒,其含有至少一个独立终止子序列,在真核细胞或原核细胞或两者中允许盒复制的序列(例如穿梭载体),以及用于原核和真核系统两者的选择标记。载体适用于原核细胞,真核细胞或两者中的复制和整合。
54.其他有用的参考文献,例如用于细胞分离和培养(例如,用于随后的核酸分离)包括freshney(1994)culture of animal cells,a manual of basic technique,third edition,wiley-liss,new york和其中引用的参考文献;payne et al.(1992)plant cell and tissue culture inliquid systems john wiley&sons,inc.new york,n.y.;gamborg and phillips(eds)(1995)plant cell,tissue and organ culture;fundamental methods springer lab manual,springer-verlag(berlin heidelberg new york)和atlas and parks(eds)the handbook of microbiological media(1993)crc press,boca raton,fla。
55.编码本文公开的重组聚合酶的核酸也是本文呈现的实施方案的特征。特定的氨基酸可以由多种密码子编码,并且某些翻译系统(例如,原核或真核细胞)通常显示密码子偏好,如不同的生物体往往优选编码相同氨基酸的几种同义密码子之一。因此,本文提供的核酸是任选地“密码子优化的”,这意味着合成核酸以包括采用的表达聚合酶的特定翻译系统优选的密码子。例如,当期望在细菌细胞(或甚至特定的细菌菌株)中表达聚合酶时,可以合成核酸以包括在该细菌细胞的基因组中最常见的密码子,用于有效表达聚合酶。当希望在真核细胞中表达聚合酶时,可以使用类似的策略,例如核酸可以包括该真核细胞优选的密码子。
56.在一些实施方案中,一个或多个多肽结构域可以与聚合酶融合。在一些实施方案中,所述结构域与聚合酶的c末端融合。在一些实施方案中,所述结构域与聚合酶的n末端融合。在一些实施方案中,所述结构域在聚合酶序列的内部。在一些实施方案中,多肽结构域包含纯化标签。如本文所用,术语“纯化标签”指任何适合纯化或鉴定多肽的肽序列。纯化标签特异性结合对纯化标签具有亲和性的另一部分(moieties)。特异性结合纯化标签的此类部分通常连接到基质或树脂上,如琼脂糖珠。特异性结合纯化标签的部分包括抗体,其他蛋白质(例如蛋白a或链霉亲和素),镍或钴离子或树脂,生物素,直链淀粉,麦芽糖和环糊精。示例性的纯化标签包括组氨酸(his)标签(如六组氨酸肽),其将结合金属离子如镍或钴离子。其他示例性的纯化标签为myc标签(eqkliseedl),strep标签(wshpqfek),flag标签(dykddddk),以及v5标签(gkpipnpllgldst)。术语“纯化标签”还包括“表位标签”,即被抗体特异性识别的肽序列。示例性表位标签包括由单克隆抗flag抗体特异性识别的flag标签。由抗flag抗体识别的肽序列由序列dykddddk或其基本相同的变体组成。在一些实施方案中,与聚合酶融合的多肽结构域包含两个或更多个标签,例如sumo标签和strep标签。术语“纯化标签”还包括纯化标签的基本相同的变体。如本文所用,术语“基本相同的变体”指纯化标签的衍生物或片段,其相较于原始纯化标签是修饰的(例如通过氨基酸取代,缺失或插入),但其保留与特异性识别纯化标签的部分特异性结合的纯化标签的性质。
57.在一些实施方案中,与聚合酶融合的多肽结构域包含表达标签。如本文所用,术语“表达标签”指任何可以连接到第二多肽上的肽或多肽,并且被认为支持感兴趣的重组多肽
的溶解度,稳定性和/或表达。示例性表达标签包括fc标签和sumo标签。原则上,任何肽,多肽或蛋白质都可以用作表达标签。
58.在一些实施方案中,融合到聚合酶的多肽结构域包含seq id no:72所示的前导肽。
59.多种蛋白质分离和检测方法是已知的,并可用于例如从表达本文呈现的重组聚合酶的细胞的重组培养物中分离聚合酶。各种蛋白质分离和检测方法是本领域公知的,包括,例如列于以下中的那些:r.scopes,protein purification,springer-verlag,n.y.(1982);deutscher,methods inenzymology vol.182:guide to protein purification,academic press,inc.n.y.(1990);sandana(1997)bioseparation of proteins,academic press,inc.;bollag et al.(1996)protein methods,2.sup.nd edition wiley-liss,ny;walker(1996)the protein protocols handbook humana press,nj,harris and angal(1990)protein purification applications:a practical approach irl press at oxford,oxford,england;harris and angal protein purification methods:a practical approach irl press at oxford,oxford,england;scopes(1993)protein purification:principles and practice 3.sup.rd edition springer verlag,ny;janson and ryden(1998)proteinpurification:principles,high resolution methods and applications,second edition wiley-vch,ny;以及walker(1998)protein protocols on cd-rom humana press,nj;以及其中引用的参考文献。关于蛋白质纯化和检测方法的更多细节可在satinder ahuja ed.,handbook of bioseparations,academic press(2000)中找到。
60.序列比较,同一性和同源性
61.在两个或多个核酸或多肽序列的语境下,术语“相同”或者“百分比同一性”指当如使用下面描述的序列比较算法(或技术人员可用的其他算法)之一或通过肉眼检查来测量比较和比对最大对应(correspondence)时,两个或更多个序列或子序列相同或具有特定百分比的相同氨基酸残基或核苷酸。
62.在两个核酸或多肽(例如编码聚合酶的dna,或聚合酶的氨基酸序列)的语境下,术语“基本相同”指当如使用序列比较算法或肉眼检查来测量比较和比对最大对应时,具有至少约60%、约80%、约90-95%、约98%、约99%或更多核苷酸或氨基酸残基同一性的两个或多个序列或子序列。此类“基本相同”的序列通常被认为是“同源的”,而不参考实际祖先。优选地,“基本相同”存在于长度为至少约50个残基的序列区域上,更优选在至少约100个残基的区域上,最优选地,序列在要比较的两个序列的至少约150个残基或全长上基本相同。
63.当蛋白质和/或蛋白质序列从共同的祖先蛋白质或蛋白质序列天然或人工衍生时,它们是“同源的”。类似地,当核酸和/或核酸序列从共同的祖先核酸或核酸序列天然或人工衍生时,它们是“同源的”。同源性通常由两个或多个核酸或蛋白质(或其序列)之间的序列相似性推断。可用于建立同源性的序列之间的相似性的精确百分比随核酸和蛋白质的不同而异,但通常使用在50、100、150或更多残基上少至25%的序列相似性来建立同源性。也可以使用更高水平的序列相似性如30%,40%、50%、60%、70%、80%、90%、95%,99%或更高来建立同源性。用于确定序列相似性百分比的方法(例如使用默认参数的blastp和blastn)描述于本文并通常是可用的。
64.对于序列比较和同源性测定,通常一个序列作为比较测试序列的参考序列。当使用序列比较算法时,将测试和参考序列输入到计算机中,指定子序列坐标(如果需要),并且指定序列算法程序参数。然后基于指定的程序参数,序列比较算法计算测试序列相对于参考序列的百分比序列同一性。
65.例如可以通过以下来进行用于比较的序列的最佳比对:smith和waterman,adv.appl.math.2:482(1981)的局部同源性算法,通过needleman&wunsch,j.mol.biol.48:443(1970)的同源比对算法,通过pearson&lipman,proc.nat'l.acad.sci.usa 85:2444(1988)的搜索相似性方法,通过这些算法的计算机化实施(威斯康星遗传学软件包中的gap,bestfit,fasta,和tfasta,genetics computer group,575science dr.,madison,wis.),或通过肉眼观察进行(一般性参见current protocols in molecular biology,ausubel et al.,eds.,current protocols,a joint venture between greene publishing associates,inc.and john wiley&sons,inc.,通过2004补充)。
66.适用于确定百分比序列同一性和序列相似性的算法的一个例子是blast算法,其描述于altschul et al.,j.mol.biol.215:403-410(1990)。用于进行blast分析的软件可通过国家生物技术信息中心(national center for biotechnology information)公开获得。该算法涉及通过识别查询序列中长度为w的短词首先鉴定高分数序列配对(hsp),所述长度为w的短词当与数据库序列中具有相同长度的词对齐时匹配或满足一些正值阈值分数t。t被称为邻域词分数阈值(altschul et al.,同上)。这些初始邻域词命中(hit)作为启动搜索以找到包含它们的较长hsp的种子。然后,词命中沿着每个序列的两个方向延伸,只要可以增加累积对齐分数即可。对于核苷酸序列,使用参数m(一对匹配残基的奖励分数;总是》0)和n(不匹配残基的罚分,总是《0)来计算累积分数。对于氨基酸序列,使用得分矩阵来计算累积分数。当以下情况时中止在每个方向上的词命中的延伸:累积对齐分数从其最大达到值下降量x;由于一个或多个负评分残基比对的积累而累积得分至零或以下;或者到达任一序列的末端。blast算法参数w、t和x确定比对的灵敏度和速度。blastn程序(用于核苷酸序列)使用以下缺省值:11的词长度(w),10的预期值(e),100的截止值,m=5,n=-4,和两条链的比较。对于氨基酸序列,blastp程序使用以下缺省值:3的词长度(w),10的预期值(e),以及blosum62得分矩阵(参见henikoff&henikoff(1989)proc.natl.acad.sci.usa 89:10915)。
67.除了计算百分比序列同一性,blast算法还对两个序列之间的相似性进行统计分析(参见,例如,karlin&altschul,proc.nat'l.acad.sci.usa90:5873-5787(1993))。blast算法提供的一种相似性度量是最小和概率(p(n)),其提供了会偶然发生的两个核苷酸或氨基酸序列之间的匹配的概率的指示。例如,如果在测试核酸与参考核酸的比较中的最小和概率小于约0.1,更优选小于约0.01,以及最优选小于约0.001,则核酸与参考序列相似。
68.编码改变的聚合酶的核酸
69.本文还呈现的是编码本文呈现的改变的聚合酶的核酸分子。对于任何给定的改变的聚合酶(其是聚合酶的突变体版本),其氨基酸序列和优选地编码聚合酶的野生型核苷酸序列也是已知的,可以根据分子生物学的基本原理获得编码突变体的核苷酸序列。例如,鉴于编码聚合酶的野生型核苷酸序列是已知的,可以使用标准遗传密码推断编码具有一个或多个氨基酸取代的聚合酶的任何给定突变体版本的核苷酸序列。类似地,对于其它聚合酶
例如vent
tm
、pfu、t.sp.jdf-3、taq等的突变体版本,可以容易地导出核苷酸序列。然后可以使用本领域已知的标准分子生物学技术构建具有所需核苷酸序列的核酸分子。
70.根据本文呈现的实施例,由于保守氨基酸取代中的简并密码子,定义的核酸不仅包括相同的核酸,而且还包括任何次要的碱基变异,特别是包括导致同义密码子(指定相同氨基酸残基的不同密码子)情况下的取代。术语“核酸序列”还包括与碱基变异相关的任何单链序列的互补序列。
71.本文所述的核酸分子也可以有利地包括于合适的表达载体中以在合适的宿主中表达从其编码的聚合酶蛋白。将克隆的dna掺入合适的表达载体中用于随后转化所述细胞并随后选择转化的细胞是本领域技术人员熟知的,如sambrook et al.(1989),molecular cloning:a laboratory manual,cold spring harbor laboratory中提供的,其通过引用以其整体并入本文。
72.此类表达载体包括具有根据本文呈现的实施方案的核酸的载体,所述核酸可操作地连接于能够影响所述dna片段表达的调节序列,如启动子区。术语“可操作地连接”指并排放置(juxtapositon),其中所述组件在允许它们以它们预期的方式起作用的关系中。可以将此类载体转化入合适的宿主细胞中以提供根据本文呈现的实施方案中的蛋白的表达。
73.核酸分子可以编码成熟蛋白质或具有原序列(prosequence)的蛋白质,其包括编码在前蛋白(preprotein)上的前导序列,其然后被宿主细胞切割以形成成熟蛋白质。所述载体例如可以是提供了复制起点,和任选地用于表达所述核苷酸的表达的启动子以及任选地所述启动子的调节子的质粒、病毒或噬菌体载体。所述载体可以含有一个或多个可选择标记,如例如抗生素抗性基因。
74.表达所需的调节元件包括启动子序列以结合rna聚合酶和以引导合适水平的转录起始,以及还包括用于核糖体结合的翻译起始序列。例如,细菌表达载体可以包括启动子(如lac启动子)以及用于翻译起始的shine-dalgarno序列以及起始密码子aug。类似地真核表达载体可以包括rna聚合酶ii的异源性或同源性启动子,下游聚腺苷酸化信号,起始密码子aug,和用于核糖体脱离的终止密码子。此类载体可以商业获得或由本领域众所周知的方法从序列组装。
75.可以通过在载体中包含增强子序列来优化由高等真核生物编码聚合酶的dna的转录。增强子是作用于启动子以提高转录水平的dna顺式作用元件。除了可选择的标记之外,载体还通常还包括复制起点。
实施例
76.实施例1
77.表达和纯化
78.从dna2.0(menlo park,ca)获得市售的表达载体(pd871),用于编码seq id nos:67-71的氨基酸序列的5个合成亲本序列。为了在其他载体中表达基因,将基因再克隆到pd861表达载体(dna2.0,menlo park,ca)中。然后通过以下产生66个不同的表达载体:以各种组合来组合亲本载体部分以产生编码seq id nos:1-66.的聚合酶氨基酸序列的表达载体。编码的酶还含有包含以下序列的用于纯化的前导肽:mgsshghhhhhhhgvggwshpq fekggtenlyfqggh(seq id no:72)。
79.将每个表达质粒转化入感受态表达菌株(edge biosystems,gaithersburg,md)中,并且过夜培养转化体然后稀释至更大体积。达到诱导密度后,通过添加鼠李糖诱导表达,并允许继续孵育另外的时间段。然后通过离心收获诱导的细胞,然后在-20℃下冷冻。将细胞在100℃下孵育直至裂解细胞。
80.然后将细胞裂解物通过含有ni-nta和肝素亲和树脂的旋转柱以纯化表达的聚合酶。
81.实施例2
82.根据实施例1中描述的方法制备33个编码seq id nos:1-66的聚合酶氨基酸序列的表达载体。编码的酶还含有包含以下序列的用于纯化的前导肽:mgsshghhhhhhhgvggwshpq fekggtenlyfqggh(seq id no:72)。如上文实施例1所述表达和纯化酶,然后如以下实施例所述测试聚合酶活性和保真度。
83.实施例3
84.聚合酶保真度活性
85.测试在实施例2中表达的28个聚合酶的保真度。将所有保真度水平与用作基线的vent dna聚合酶的保真度水平相比较。如图1中所示,除了10个测试的酶之外,所有的酶都具有达到或超过vent的保真度水平。
86.实施例4
87.聚合酶qpcr活性
88.使用qpcr测定来测试实施例2中表达的28个聚合酶的稳健性(robustness)和速度。将所有活性水平与用作基线的vent dna聚合酶活性水平相比较。如图1中所示,至少8个酶具有接近或超过vent的活性水平。进行实验以在各种条件和延伸时间下测试聚合酶。
89.对于qpcr数据分析,聚合酶的排名是从以下指标的组合得出的。
90.1)扩增最大(amplification max)
91.2)至50%扩增最大(amp max)的循环#
92.3)熔融峰最高温度
93.4)熔融峰最大强度
94.将聚合酶排名与vent聚合酶排名相比较,如图1所示。
95.在本技术中,已经引用了各种出版物,专利和/或专利申请。这些出版物的公开通过引用以其整体并入本技术中。
96.术语“包括”旨在是开放式的,不仅包括所述元件,而且还包括任何其它元件。
97.已经描述了许多实施例。然而,应当理解,可以进行各种修改。因此,其它实施方式在以下权利要求的范围内。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。