1.本发明涉及生物技术领域,具体的,本发明涉及炎症性肠病标志基因及其应用,更具体的,本发明涉及一种试剂盒、试剂在制备试剂盒中的用途、用于预防或者治疗炎症性肠病的药物组合物或者食品组合物、确定个体是否患有炎症性肠病方法、确定个体是否患有炎症性肠病的装置、一种装置、一种筛选药物的方法。
背景技术:
2.炎症性肠病(ibd)是一组主要影响胃肠道的慢性炎症性自身免疫性疾病。炎症性肠病的主要症状包括腹痛、腹泻、血便和粘液便。严重者可出现营养不良和肠穿孔。炎症性肠病主要包括溃疡性结肠炎(uc) 和克罗恩病(cd)两种不同的分类,它们影响着全球约500万人,目前没有可用于炎症性肠病的治愈性治疗方法。
3.我国炎症性肠病的患病率正在逐渐升高,这种患病率增加可能与不断变化的环境、饮食和生活方式的西方化以及由此产生的肠道微生物群的变化有关。炎症性肠病的病因是遗传、环境、饮食、感染、心理和其他因素的复杂相互作用,其致病影响被认为与肠脑对话障碍、心理社会因素以及参与该过程的肠道微生物群,包括体育活动和肥胖、压力、睡眠和吸烟在内的各种生活方式因素可能会改变患炎症性肠病的风险。
4.2018年发表在science杂志的研究表明,c1orf106基因的多态性与炎症性肠病的风险增加相关。 cytohesin-1是一种控制arf6活化的鸟嘌呤核苷酸交换因子,c1orf106通过调控cytohesin-1的泛素化降解来调节上皮细胞黏附连接的稳定性。通过限制cytohesin-1依赖性的arf6活化,c1orf106可稳定上皮细胞黏附连接。c1orf106基因变异加速cytohesin-1的降解并缩短其半衰期,引发肠道上皮细胞屏障功能障碍,从而使炎症性肠病风险增加。
5.炎症性肠病治疗的主要药物是氨基水杨酸盐、糖皮质激素、免疫抑制剂和生物制剂。近几年来,氨基水杨酸盐已经被医师和患者广泛接受,免疫抑制剂的应用也趋于规范,应用生物制剂的经验也越来越多。
6.我国目前对炎症性肠病的诊断方法主要有血清标志物、影像学诊断以及内镜诊断。由于早期对于炎症性肠病的认识不足和我国结核病高发,炎症性肠病误诊率和漏诊率较高。近年欧美提出的一些血清标志物在中国似乎并没有获得满意的结果,如在一些国内入组病例数较多的病例对照研究中,抗中性粒细胞胞质抗体(anti-neutrophil cytoplasmic antibodies,anca)对溃疡性结肠炎(uc)诊断敏感度在37.9%~ 56.7%,抗酿酒酵母细胞抗体(anti-sacc/iaroto;yces cerevisiae,asca)对克罗恩病(cd)诊断敏感度为45.2%~65.5%,其敏感性均较欧美研究(anca为60%~80%,asca为55%~65%)偏低,提示东西方ibd发病机制之间可能存在不同,这也给我国的ibd基础科学研究提供了机遇,有机会找到有利于我国疾病人群诊断和鉴别诊断的指标。
技术实现要素:
7.本发明旨在至少解决上述问题至少之一或者提供至少一种可选择的商业手段。
8.为此,在本发明的第一方面,本发明提出了一种试剂盒。根据本发明的实施例,根据本发明的实施例,包括适于检测第一基因集中的至少一种基因的试剂,所述第一基因集由以下基因组成: s100_gi_0002354、s100_gi_0002360、s100_gi_0002362、s100_gi_0002364、s100_gi_0002366、 s100_gi_0002373、s100_gi_0002377、s100_gi_0002402、s100_gi_0002408、s100_gi_0002410、 s100_gi_0008128、s100_gi_0030720、s10_gi_0005425、s186_gi_0019691、s24_gi_0044455和 s254_gi_0026483,其中,所述第一基因集中的基因与seq id no:1-16所示的核酸序列一一对应,所述第一基因集中的基因与其对应的seq id no:1-16中的序列具有不小于90%的同一性。根据本发明具体实施例的试剂盒,可以准确的检测生物样品中是否含有第一基因集中的至少一种基因,更进一步地,也可以准确区分或诊断炎症性肠病患者和健康个体。
9.在本发明的第二方面,本发明提出了试剂在制备试剂盒中的用途,所述试剂适于检测第一基因集中的至少一种基因。根据本发明的实施例,所述试剂盒用于诊断炎症性肠病或检测炎症性肠病的治疗效果,所述第一基因集由以下基因组成:s100_gi_0002354、s100_gi_0002360、s100_gi_0002362、 s100_gi_0002364、s100_gi_0002366、s100_gi_0002373、s100_gi_0002377、s100_gi_0002402、 s100_gi_0002408、s100_gi_0002410、s100_gi_0008128、s100_gi_0030720、s10_gi_0005425、 s186_gi_0019691、s24_gi_0044455和s254_gi_0026483,其中,所述第一基因集中的基因与seq id no: 1-16所示的核酸序列一一对应,所述第一基因集中的基因与其对应的seq id no:1-16中的序列具有不小于90%的同一性。根据本发明具体实施例的试剂制备的试剂盒,可以准确的检测生物样品中是否包含所述第一基因集中的至少一种基因,更进一步地,也可以极准确的区分炎症性肠病患者和健康个体,由此,可以有效的在早期进行炎症性肠病诊断,或用于检测治疗过程中炎症性肠病的变化。
10.在本发明的第三方面,本发明提出了一种用于预防或者治疗炎症性肠病的药物组合物或者食品组合物。根据本发明的实施例,含有提高所述第一基因集中的至少一种基因丰度的物质,所述第一基因集由以下基因组成:s100_gi_0002354、s100_gi_0002360、s100_gi_0002362、s100_gi_0002364、 s100_gi_0002366、s100_gi_0002373、s100_gi_0002377、s100_gi_0002402、s100_gi_0002408、 s100_gi_0002410、s100_gi_0008128、s100_gi_0030720、s10_gi_0005425、s186_gi_0019691、s24_gi_0044455和s254_gi_0026483,其中,所述第一基因集中的基因与seq id no:1-16所示的核酸序列一一对应,所述第一基因集中的基因与其对应的seq id no:1-16中的序列具有不小于90%的同一性。根据本发明实施例的炎症性肠病标志基因中第一基因集的基因可以非侵入性的在早期发现或辅助检测炎症性肠病,确定个体患有炎症性肠病的概率高低或者个体处于健康状态的概率高低;同时,提高炎症性肠病高风险人群或已炎症性肠病患者肠道内的所述第一基因集中的各种基因的丰度,可以降低患炎症性肠病的概率或减缓、治愈炎症性肠病,因此,所述包含提高所述第一基因集中的至少一种基因丰度的药物或者食品组合物能够用于平衡肠道菌群,有效预防或治疗炎症性肠病。
11.在本发明的第四方面,本发明提出了一种确定个体是否患有炎症性肠病的方法。
根据本发明的实施例,包括:(1)确定所述个体的粪便样本中标志基因的丰度,所述标志基因包括第一基因集和第二基因集中的至少一种基因;(2)将步骤(1)中得到的所述丰度与预定的阈值进行比较,以便确定所述个体是否患有炎症性肠病;其中,所述第一基因集由以下基因组成:s100_gi_0002354、s100_gi_0002360、 s100_gi_0002362、s100_gi_0002364、s100_gi_0002366、s100_gi_0002373、s100_gi_0002377、 s100_gi_0002402、s100_gi_0002408、s100_gi_0002410、s100_gi_0008128、s100_gi_0030720、 s10_gi_0005425、s186_gi_0019691、s24_gi_0044455和s254_gi_0026483,其中,所述第一基因集中的基因与seq id no:1-16所示的核酸序列一一对应,所述第一基因集中的基因与其对应的seq id no: 1-16中的序列具有不小于90%的同一性,所述第二基因集由以下基因组成:s100_gi_0015747、 s101_gi_0002517、s152_gi_0024776、s15_gi_0022087、s258_gi_0009120、s47_gi_0011838、 s81_gi_0011061、s103_gi_0016687、s118_gi_0008214、s257_gi_0026750和s57_gi_0025049,其中,所述第二基因集中的基因与seq id no:17-27所示的核酸序列一一对应,所述第二基因集中的基因与其对应的seq id no:17-27中的核酸序列具有不小于90%的同一性。根据本发明实施例的方法可以依据个体的粪便样本中的所述标志基因的丰度确定个体是否患有炎症性肠病,所述标志基因是发明人对大量已知状态的粪便样本进行验证,通过差异比较分析各种肠道基因在炎症性肠病组和健康组粪便样本中的丰度,而确定下来的。
12.在本发明的第五方面,本发明提出了一种确定个体是否患有炎症性肠病的装置。根据本发明的实施例,包括:丰度确定单元,用于确定所述个体的粪便样本中标志基因的丰度,所述标志基因包括第一基因集和第二基因集中的至少一种基因;比较单元,用于将所得到的所述丰度与预定的阈值进行比较,以便确定所述个体是否患有炎症性肠病;其中,所述第一基因集由以下基因组成:s100_gi_0002354、 s100_gi_0002360、s100_gi_0002362、s100_gi_0002364、s100_gi_0002366、s100_gi_0002373、 s100_gi_0002377、s100_gi_0002402、s100_gi_0002408、s100_gi_0002410、s100_gi_0008128、 s100_gi_0030720、s10_gi_0005425、s186_gi_0019691、s24_gi_0044455和s254_gi_0026483,其中,所述第一基因集中的基因与seq id no:1-16所示的核酸序列一一对应,所述第一基因集中的基因与其对应的seq id no:1-16中的序列具有不小于90%的同一性,所述第二基因集由以下基因组成:
13.s100_gi_0015747、s101_gi_0002517、s152_gi_0024776、s15_gi_0022087、s258_gi_0009120、 s47_gi_0011838、s81_gi_0011061、s103_gi_0016687、s118_gi_0008214、s257_gi_0026750和 s57_gi_0025049,其中,所述第二基因集中的基因与seq id no:17-27所示的核酸序列一一对应,所述第二基因集中的基因与其对应的seq id no:17-27中的核酸序列具有不小于90%的同一性。所述标志基因是发明人通过差异比较分析各种肠道基因在炎症性肠病患者和健康人群的粪便样本中的丰度并经过分析和大量已知状态的粪便样本的验证而确定下来的,根据本发明实施例的装置可以准确确定个体是否为炎症性肠病的高风险人群或炎症性肠病患者。
14.在本发明的第六方面,本发明提出了一种装置。根据本发明的实施例,包括:计算机可读存储介质,其上存储有计算机程序,所述程序用于执行第四方面所述的方法;以及一个或者多个处理器,用于执行所述计算机可读存储介质中的程序。根据本发明实施例的装
置可以准确确定个体是否为炎症性肠病的高风险人群或炎症性肠病患者。
15.在本发明的第七方面,本发明提出了一种筛选药物的方法。根据本发明的实施例,所述药物用于治疗或者预防炎症性肠病,所述方法包括:将候选药物施用于受试者,检测施用前后,所述受试者粪便中标志基因的丰度,所述标志基因包括第一基因集和第二基因集中的至少一种基因,其中,满足下列条件至少之一的候选药物适于用于治疗或者预防炎症性肠病:(1)进行所述施用后,所述第一基因集中的至少一种基因的所述丰度升高;和(2)进行所述施用后,所述第二基因集中的至少一种基因的所述丰度降低;其中,所述第一基因集由以下基因组成:s100_gi_0002354、s100_gi_0002360、 s100_gi_0002362、s100_gi_0002364、s100_gi_0002366、s100_gi_0002373、s100_gi_0002377、 s100_gi_0002402、s100_gi_0002408、s100_gi_0002410、s100_gi_0008128、s100_gi_0030720、 s10_gi_0005425、s186_gi_0019691、s24_gi_0044455和s254_gi_0026483,其中,所述第一基因集中的基因与seq id no:1-16所示的核酸序列一一对应,所述第一基因集中的基因与其对应的seq id no: 1-16中的序列具有不小于90%的同一性,所述第二基因集由以下基因组成:s100_gi_0015747、 s101_gi_0002517、s152_gi_0024776、s15_gi_0022087、s258_gi_0009120、s47_gi_0011838、 s81_gi_0011061、s103_gi_0016687、s118_gi_0008214、s257_gi_0026750和s57_gi_0025049,其中,所述第二基因集中的基因与seq id no:17-27所示的核酸序列一一对应,所述第二基因集中的基因与其对应的seq id no:17-27中的核酸序列具有不小于90%的同一性。根据本发明实施例的方法可以生产或筛选出升高所述标志基因中第一基因集中各种基因丰度,和/或降低肠道标志基因中第二基因集中的各种基因丰度的药物,对于辅助减轻炎症性肠病的临床症状具有重要意义。
16.本发明的附加方面和优点将在下面的描述中部分给出,部分将从下面的描述中变得明显,或通过本发明的实践了解到。
附图说明
17.本发明的上述和/或附加的方面和优点从结合下面附图对实施例的描述中将变得明显和容易理解,其中:
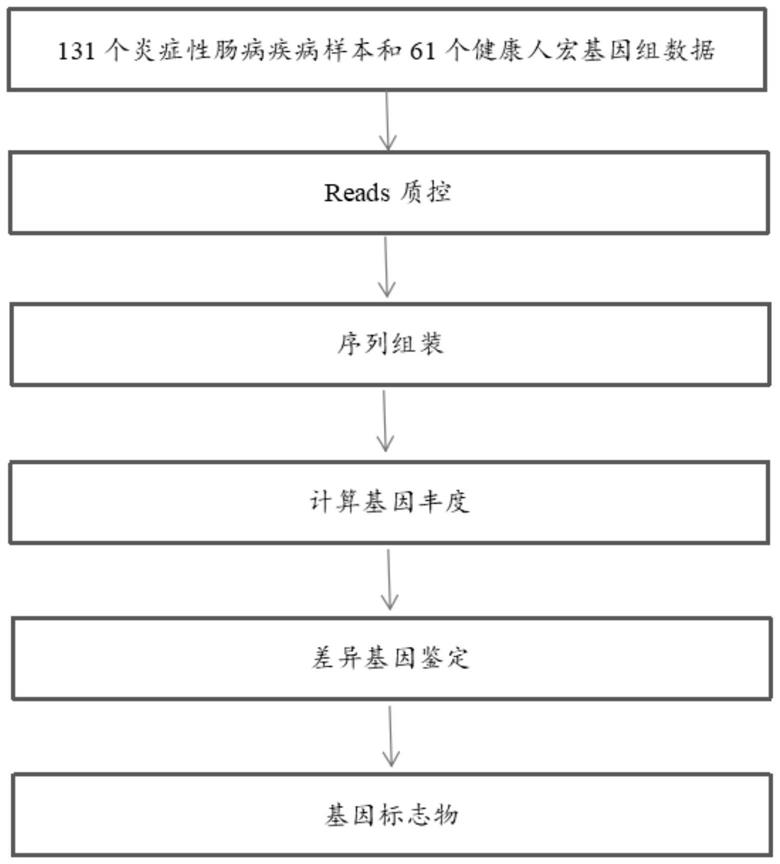
18.图1是根据本发明实施例的筛选炎症性肠病标志基因的实验分析流程示意图;以及
19.图2是根据本发明实施例的标志基因综合指标auc评价结果示意图,其中,specificity表示特异度,即预测为阳性且实际为阳性,真阳性,纵坐标sensitivity表示敏感度,即真阴性:
20.2-a为第一期192个样品数据roc曲线下auc值和置信区间结果图;
21.2-b为第二期102个样品数据roc曲线下auc值和置信区间结果图。
具体实施方式
22.下面详细描述本发明的实施例,所述实施例的示例在附图中示出。下面通过参考附图描述的实施例是示例性的,旨在用于解释本发明,而不能理解为对本发明的限制。
23.此外,术语“第一”、“第二”仅用于描述目的,而不能理解为指示或暗示相对重要性或者隐含指明所指示的技术特征的数量。由此,限定有“第一”、“第二”的特征可以明示或者
隐含地包括至少一个该特征。在本发明的描述中,“多个”的含义是至少两个,例如两个,三个等,除非另有明确具体的限定。
24.术语“任选地”仅用于描述目的,而不能理解为指示或暗示相对重要性。由此,限定有“任选地”的特征可以明示或者隐含地包括或不包括该特征。
25.生物学标志物是从生物学介质中可以检测到的细胞/生物化学或分子改变。生物学介质包括各种体液、组织、细胞、粪便、头发、呼气等。
26.所称的丰度指在某一微生物或者核酸序列群体中该种微生物或者序列的丰富程度。例如在肠道基因群体中该种微生物的丰富程度,可表示为该种微生物在该群体中的含量;又例如在一组核酸序列中某种核酸序的丰富程度,可表示为该种核酸序列的数目占该组序列的总数的比例。
27.同一性,本发明,为了比较两个或更多个核苷酸序列,可以通过将[第一序列中与相应位置的核苷酸相同的核苷酸的数目相除]来计算第一序列和第二序列之间的“序列同一性”的百分比。第二个序列中的核苷酸]减去[第一个序列中核苷酸的总数],然后乘以[100%],其中第二个核苷酸序列中每个核苷酸的缺失,插入,取代或添加-相对于第一核苷酸序列-被认为是单个核苷酸(位置)上的差异。
[0028]
或者,可以使用标准设置,使用用于序列比对的已知计算机算法,例如ncbi blast v2.0,计算两个或多个核苷酸序列之间的序列同一性程度。
[0029]
用于确定序列同一性程度的一些其他技术,计算机算法和设置例如在wo 04/037999,ep 0 967 284, ep 1 085 089,wo 00/55318,wo 00/78972,wo 98/49185和gb 2357768-a。
[0030]
需要说明的是,本技术中所述的“标志基因”的长度不受特别限制,可以为完整基因,也可以为基因的编码区或非编码区,更进一步地,根据不同的实验目的,可以选择相关的任意的核酸片段作为标志基因。
[0031]
根据本发明的一个实施方式提供的一种试剂盒,包括适于检测第一基因集中的至少一个基因的试剂,所述第一基因集由以下基因组成:s100_gi_0002354、s100_gi_0002360、s100_gi_0002362、 s100_gi_0002364、s100_gi_0002366、s100_gi_0002373、s100_gi_0002377、s100_gi_0002402、 s100_gi_0002408、s100_gi_0002410、s100_gi_0008128、s100_gi_0030720、s10_gi_0005425、 s186_gi_0019691、s24_gi_0044455和s254_gi_0026483,其中,所述第一基因集中的基因与seq id no: 1-16所示的核酸序列一一对应,所述第一基因集中的基因与其对应的seq id no:1-16中的序列具有不小于90%的同一性。
[0032]
根据本发明的一个具体的实施方案,所述试剂盒进一步包括适于检测第二基因集中的至少一个基因的试剂,所述第二基因集由以下基因组成:s100_gi_0015747、s101_gi_0002517、s152_gi_0024776、 s15_gi_0022087、s258_gi_0009120、s47_gi_0011838、s81_gi_0011061、s103_gi_0016687、 s118_gi_0008214、s257_gi_0026750和s57_gi_0025049,其中,所述第二基因集中的基因与seq id no: 17-27所示的核酸序列一一对应,所述第二基因集中的基因与其对应的seq id no:17-27中的核酸序列具有不小于90%的同一性。
[0033]
根据本发明的具体实施例,所述试剂盒包括适于检测所述第一基因集中全部所述基因的试剂。
[0034]
根据本发明的具体实施例,所述试剂盒包括适于检测所述第二基因集中全部所述基因的试剂。
[0035]
根据本发明的具体实施例,所述标志基因是发明人通过对大量患炎症性肠病个体和大量健康对照个体的粪便样本中的微生物的基因丰度的差异比较分析、以及验证,而确定下来的,明确了肠道中炎症性肠病相关的标志基因。利用包含检测所述标志基因的试剂的试剂盒能够准确检测生物样品中是否包含所述第一、第二基因集中的至少一种基因,更进一步地,也可以准确确定个体处于患有炎症性肠病状态的概率高低或者处于健康状态的概率高低,能够用于非侵入性的早期发现或辅助检测炎症性肠病。
[0036]
根据本发明的具体实施例,所述适于检测所述第一基因集或第二基因集的试剂不受特别限制,任何可以直接或间接检测所述标志基因的丰度的试剂均包含在本发明的范围内,如:利用pcr技术、高通量测序等方法直接进行检测时使用的试剂,或通过western blot技术检测所述基因对应的蛋白的含量间接获得所述基因丰度时所使用的试剂等。
[0037]
根据本发明提供的试剂在制备试剂盒中的用途,所述试剂适于检测第一基因集中的至少一种基因,所述试剂盒用于诊断炎症性肠病或者检测炎症性肠病的治疗效果,所述第一基因集由以下基因组成: s100_gi_0002354、s100_gi_0002360、s100_gi_0002362、s100_gi_0002364、s100_gi_0002366、 s100_gi_0002373、s100_gi_0002377、s100_gi_0002402、s100_gi_0002408、s100_gi_0002410、 s100_gi_0008128、s100_gi_0030720、s10_gi_0005425、s186_gi_0019691、s24_gi_0044455和 s254_gi_0026483,其中,所述第一基因集中的基因与seq id no:1-16所示的核酸序列一一对应,所述第一基因集中的基因与其对应的seq id no:1-16中的序列具有不小于90%的同一性。
[0038]
根据本发明的具体实施例,所述标志基因是发明人通过对大量患炎症性肠病个体和大量健康对照个体的粪便样本中的微生物基因进行差异比较分析、以及验证,而确定下来的,明确了肠道中炎症性肠病相关的微生物的基因标志物。利用检测所述标志基因的试剂能够确定个体患有炎症性肠病的概率高低或者处于健康状态的概率高低,能够用于非侵入性的早期发现或辅助检测炎症性肠病。
[0039]
根据本发明一些具体的实施例,所述试剂进一步适于检测第二基因集中的至少一种基因,所述第二基因集由以下基因组成:s100_gi_0015747、s101_gi_0002517、s152_gi_0024776、s15_gi_0022087、 s258_gi_0009120、s47_gi_0011838、s81_gi_0011061、s103_gi_0016687、s118_gi_0008214、 s257_gi_0026750和s57_gi_0025049,其中,所述第二基因集中的基因与seq id no:17-27所示的核酸序列一一对应,所述第二基因集中的基因与其对应的seq id no:17-27中的核酸序列具有不小于90%的同一性。
[0040]
根据本发明的具体实施例,所述适于检测所述第一基因集或第二基因集的试剂不受特别限制,任何可以直接或间接检测所述标志基因的丰度的试剂均包含在本发明的范围内,如:利用pcr技术、高通量测序等方法直接进行检测时使用的试剂,或通过western blot技术检测所述基因对应的蛋白的含量间接获得所述基因丰度时所使用的试剂等。
[0041]
根据本发明提供的一种用于预防或者治疗炎症性肠病的药物组合物或者食品组合物,含有提高第一基因集中的至少一种基因丰度的物质,所述第一基因集由以下基因组成:s100_gi_0002354、 s100_gi_0002360、s100_gi_0002362、s100_gi_0002364、s100_gi_0002366、s100_gi_0002373、 s100_gi_0002377、s100_gi_0002402、s100_gi_0002408、
s100_gi_0002410、s100_gi_0008128、 s100_gi_0030720、s10_gi_0005425、s186_gi_0019691、s24_gi_0044455和s254_gi_0026483,其中,所述第一基因集中的基因与seq id no:1-16所示的核酸序列一一对应,所述第一基因集中的基因与其对应的seq id no:1-16中的序列具有不小于90%的同一性。
[0042]
上述标志基因是发明人通过差异比较分析各种肠道微生物的基因在炎症性肠病疾病组和健康组的粪便样本中的丰度,以及经过大量已知状态的粪便样本的验证,而确定下来的。所述标志基因中的第一基因集中的基因相较于炎症性肠病患者群体,在健康群体组中显著富集,所述显著富集是指与在炎症性肠病患者组中的丰度相比,上述基因在健康组中的丰度均具有统计意义地高于或者明显地、实质性地高于在炎症性肠病患者组中的丰度;能够使该部分基因丰度提高的物质能够用于治疗炎症性肠病或者益于炎症性肠病患者服用,所述物质不受特别限制,能够使其丰度提高的物质包括但不限于治疗炎症性肠病的药物和有益肠道菌群平衡的功能性食品。因此,该实施例提供的能够使所述第一基因集中的基因丰度提高的物质能够用于制备治疗炎症性肠病的药物和/或用于制备益于平衡肠道菌群的功能性食品、保健药等,所述药物或食品可有效治疗或缓解炎症性肠病。
[0043]
根据本发明提供的一种确定个体是否患有炎症性肠病的方法,包括步骤(1)和(2)。
[0044]
(1)确定所述个体的粪便样本中的标志基因的丰度。
[0045]
所述标志基因包括第一基因集和第二基因集中的至少一种基因。其中,所述第一基因集由以下基因组成:s100_gi_0002354、s100_gi_0002360、s100_gi_0002362、s100_gi_0002364、s100_gi_0002366、 s100_gi_0002373、s100_gi_0002377、s100_gi_0002402、s100_gi_0002408、s100_gi_0002410、 s100_gi_0008128、s100_gi_0030720、s10_gi_0005425、s186_gi_0019691、s24_gi_0044455和 s254_gi_0026483,其中,所述第一基因集中的基因与seq id no:1-16所示的核酸序列一一对应,所述第一基因集中的基因与其对应的seq id no:1-16中的序列具有不小于90%的同一性,所述第二基因集由以下基因组成:s100_gi_0015747、s101_gi_0002517、s152_gi_0024776、s15_gi_0022087、 s258_gi_0009120、s47_gi_0011838、s81_gi_0011061、s103_gi_0016687、s118_gi_0008214、 s257_gi_0026750和s57_gi_0025049,所述第二基因集中的基因与seq id no:17-27所示的核酸序列一一对应,所述第二基因集中的基因与其对应的seq id no:17-27中的核酸序列具有不小于90%的同一性。
[0046]
根据本发明的一些具体的实施例,步骤(1)进一步包括:获得所述个体的粪便样本中的核酸测序数据;将所述测序数据与参考基因组进行比对;基于所述比对的结果,确定所述标志基因的丰度。
[0047]
根据本发明的具体实施例,在步骤(1)中,按照下列公式确定所述标志基因的丰度: ab(g)=ab(ug) ab(mg),其中,g表示基因的编号,ab(g)表示基因g的丰度,ab(ug)表示所述测序数据与所述基因g的参考序列唯一比对的读段的丰度,ab(mg)表示所述测序数据与所述基因g 的参考序列非唯一比对读段的丰度;ab(ug)=ug/lg,其中,ug表示所述测序数据中与所述基因g 的参考基因唯一比对的读段数目,lg表示所述基因g的参考基因长度;其中,mg为所述测序数据中与所述基因g的参考基因非唯一比对的
读段的数目,i表示所述非唯一比对读段的编号,coi为所述第i读段对应的丰度系数;其中,co
i,g
表示针对所述标志基因g,所述非唯一比对的读段i的丰度系数,n为所述非唯一比对的读段i能够比对的基因的总数,j 表示所述非唯一比对的读段i能够比对的基因的编号。上述丰度确定公式,基于比对结果中的唯一和非唯一比对上组装序列的读段对该组装序列的丰度的贡献情况,充分利用测序数据的同时确定的丰度十分准确。
[0048]
根据本发明的具体实施方案,进行以下以完成该步骤:获得所述个体的粪便样本中的核酸序列的测序数据,所述测序数据包括多个读段;将所述读段比对至所述标志物中的各种基因的基因组,获得比对结果;依据所述比对结果,确定所述标志物中的各种基因的丰度。
[0049]
所述的测序数据通过对样本中的核酸序列进行测序得来,测序依据所选的测序平台的不同,可选择但不限于半导体测序技术平台比如pgm、ion proton、bgiseq-100平台,合成边测序的技术平台,比如illumina公司的hiseq、miseq序列平台以及单分子实时测序平台,比如pacbio序列平台。测序方式可以选择单端测序,也可以选择双末端测序,获得的下机数据是测读出来的片段,称为读段(reads)。
[0050]
比对可以利用已知比对软件进行,例如soap、bwa和teramap等,在比对过程中,一般对比对参数进行设置,设置一个或者一对读段(reads)最多允许有s个碱基错配(mismatch),例如设置s≤2,若reads中有超过s个碱基发生错配,则视为该reads无法比对到(比对上)该组装片段上。所述的获得的比对结果包含各条读段与各参考基因的比对情况,包括读段是否能够比对上某个或某些基因的参考序列、只唯一比对到一种基因还是比对到多种基因的参考序列、比对到基因组的位置、比对到基因组的唯一位置还是多个位置等信息。
[0051]
reads与参考基因组比对,比对上的可以被分为两部分:a)unique reads(u):唯一比对上一个基因的序列;称这些reads为unique reads。即,如果reads比对上的序列均来自同一基因,定义这些reads 为unique reads;b)multiple reads(m):比对上一个以上基因的序列,定义为multiple reads。即,如果 reads比对上的序列来自至少两种基因,定义这些reads为multiple reads。
[0052]
所述参考基因组指预先确定的该基因的序列,可以是预先获得的待测样本所属或者所包含的生物类别的任意参考模板,例如,目标是待测样本中的基因,参考序列可选择ncbi数据库中的各种基因的参考序列,进一步地,也可以预先配置包含更多参考序列的资源库,例如依据待测样本来源的个体的状态、地域等因素选择或是测定组装出更接近的序列作为参考序列。根据本发明的一个实施例,各种基因的参考基因是由发明人进行构建的。
[0053]
(2)丰度比较,以确定个体是否患有炎症性肠病。
[0054]
根据本发明的一个实施例,将步骤(1)中得到的所述丰度与预定的阈值进行比较,以便确定所述个体是否患有炎症性肠病。
[0055]
根据本发明的一些具体实施例,所述阈值为预先设定的。将标志物中的各种基因在对健康个体以及患病个体中的丰度预先测定保存,用以作为设定阈值的依据。所述阈值
可以为一数值或者数值范围,基于已知患病或健康状态个体中的标志基因的丰度均值,该基因对应的阈值可以设为该基因丰度均值的 95%的置信区间(confidence interval)。
[0056]
所述的置信区间是指由样本统计量所构造的总体参数的估计区间。在统计学中,一个概率样本的置信区间是对这个样本的某个总体参数的区间估计。置信区间展现的是这个参数的真实值有一定概率落在测量结果的周围的程度。置信区间给出的是被测量参数的测量值的可信程度,即前面所要求的“一定概率”,这个概率被称为置信水平。
[0057]
根据本发明的一些具体实施例,当步骤(1)中确定的标志基因的丰度达到所述患炎症性肠病丰度阈值,未达到所述不患炎症性肠病丰度阈值时,确定所述个体患有炎症性肠病,当(1)中确定的标志基因的丰度达到不患炎症性肠病丰度阈值,未达到患炎症性肠病丰度阈值时,确定所述个体不患炎症性肠病。
[0058]
需要说明的是,根据目的或要求不同,可能对确定个体状态结果的可信程度有不同的要求,本领域技术人员可以选择不同的显著性水平或阈值。
[0059]
该方法基于检测个体的粪便样本中的标志基因中的各种基因的丰度,分别将检测确定的各种基因的丰度与其阈值进行比较,依据获得的比较结果能够确定个体为炎症性肠病个体或者为健康个体的概率。为早期发现炎症性肠病提供一种非侵入性的辅助检测或者辅助干预治疗的方法。
[0060]
以上任一实施例中的利用标志基因确定个体是否患有炎症性肠病的方法的全部或部分步骤,可以利用包含可拆分的相应单元功能模块的装置/系统来施行,或者将方法程序化、存储于机器可读介质,利用机器运行该可读介质来实现。
[0061]
根据本发明提供的一种确定个体是否患有炎症性肠病的装置,该装置包括:丰度确定单元,用于确定所述个体的粪便样本中标志基因的丰度,所述标志基因包括第一基因集和第二基因集;比较单元,用于将所得到的所述丰度与预定的阈值进行比较,以便确定所述个体是否患有炎症性肠病;其中,所述第一基因集由以下基因组成:s100_gi_0002354、s100_gi_0002360、s100_gi_0002362、s100_gi_0002364、 s100_gi_0002366、s100_gi_0002373、s100_gi_0002377、s100_gi_0002402、s100_gi_0002408、 s100_gi_0002410、s100_gi_0008128、s100_gi_0030720、s10_gi_0005425、s186_gi_0019691、 s24_gi_0044455和s254_gi_0026483,其中,所述第一基因集中的基因与seq id no:1-16所示的核酸序列一一对应,所述第一基因集中的基因与其对应的seq id no:1-16中的序列具有不小于90%的同一性,所述第二基因集由以下基因组成:s100_gi_0015747、s101_gi_0002517、s152_gi_0024776、 s15_gi_0022087、s258_gi_0009120、s47_gi_0011838、s81_gi_0011061、s103_gi_0016687、 s118_gi_0008214、s257_gi_0026750和s57_gi_0025049,所述第二基因集中的基因与seq id no:17-27 所示的核酸序列一一对应,所述第二基因集中的基因与其对应的seq id no:17-27中的核酸序列具有不小于90%的同一性。上述对本发明任一实施例的利用标志基因确定个体是否患有炎症性肠病的方法的技术特征和优点的描述,同样适用本发明这一方面的装置,在此不再赘述。
[0062]
根据本发明的实施例,所述丰度确定单元适于通过下列步骤确定所述丰度:获得所述个体的粪便样本中的核酸测序数据;将所述测序数据与参考基因组进行比对;基于所述比对的结果,确定所述标志基因的丰度。
[0063]
所述的测序数据通过对样本中的核酸序列进行测序得来,测序依据所选的测序平
台的不同,可选择但不限于半导体测序技术平台比如pgm、ion proton、bgiseq-100平台,合成边测序的技术平台,比如illumina公司的hiseq、miseq序列平台以及单分子实时测序平台,比如pacbio序列平台。测序方式可以选择单端测序,也可以选择双末端测序,获得的下机数据是测读出来的片段,称为读段(reads)。
[0064]
比对可以利用已知比对软件进行,例如soap、bwa和teramap等,在比对过程中,一般对比对参数进行设置,设置一个或者一对读段(reads)最多允许有s个碱基错配(mismatch),例如设置s≤2,若reads中有超过s个碱基发生错配,则视为该reads无法比对到(比对上)该组装片段上。所述的获得的比对结果包含各条读段与各参考基因的比对情况,包括读段是否能够比对上某个或某些基因的参考序列、只唯一比对到一种基因还是比对到多种基因的参考序列、比对到基因组的位置、比对到基因组的唯一位置还是多个位置等信息。
[0065]
所述参考基因组指预先确定的该基因的序列,可以是预先获得的待测样本所属或者所包含的生物类别的任意参考模板,例如,目标是待测样本中的基因,参考序列可选择ncbi数据库中的各种基因的参考序列,进一步地,也可以预先配置包含更多参考序列的资源库,例如依据待测样本来源的个体的状态、地域等因素选择或是测定组装出更接近的序列作为参考序列。根据本发明的一个实施例,各种基因的参考基因是由发明人进行构建的。
[0066]
reads与参考基因组比对,比对上的可以被分为两部分:a)unique reads(u):唯一比对上一个基因的序列;称这些reads为unique reads。即,如果reads比对上的序列均来自同一基因,定义这些reads 为unique reads;b)multiple reads(m):比对上一个以上基因的序列,定义为multiple reads。即,如果 reads比对上的序列来自至少两种基因,定义这些reads为multiple reads。
[0067]
所述参考基因组指预先确定的该基因的序列,可以是预先获得的待测样本所属或者所包含的生物类别的任意参考模板,例如,目标是待测样本中的基因,参考序列可选择ncbi数据库中的各种基因的参考序列,进一步地,也可以预先配置包含更多参考序列的资源库,例如依据待测样本来源的个体的状态、地域等因素选择或是测定组装出更接近的序列作为参考序列。根据本发明的一个实施例,各种基因的参考基因是由发明人进行构建的。
[0068]
根据本发明的一个实施例,按照下列公式确定所述标志基因的丰度:ab(g)=ab(ug) ab(mg),其中,g表示基因的编号,ab(g)表示基因g的丰度,ab(ug)表示所述测序数据与所述基因g的参考序列唯一比对的读段的丰度,ab(mg)表示所述测序数据与所述基因g的参考序列非唯一比对读段的丰度;ab(ug)=ug/lg,其中,ug表示所述测序数据中与所述基因g的参考基因唯一比对的读段数目, lg表示所述基因g的参考基因长度;其中,mg为所述测序数据中与所述基因g的参考基因非唯一比对的读段的数目,i表示所述非唯一比对读段的编号,coi为所述第i读段对应的丰度系数;其中,co
i,g
表示针对所述标志基因g,所述非唯一比对的读段i 的丰度系数,n为所述非唯一比对的读段i能够比对的基因的总数,j表示所述非唯一比对的读段i能够比对的基因的编号。上述丰度确定公式,基于比对结果中的唯一和非唯一比对上组装
序列的读段对该组装序列的丰度的贡献情况,充分利用测序数据的同时确定的丰度十分准确。上述对本发明任一实施例的利用标志基因确定个体是否患有炎症性肠病的方法的技术特征和优点的描述,同样适用本发明这一方面的装置,在此不再赘述。
[0069]
根据本发明的又一个实施例提供的一种装置,包括:计算机可读存储介质,其上存储有计算机程序,所述程序用于执行前面所述的一种确定个体是否患有炎症性肠病方法;以及一个或者多个处理器,用于执行所述计算机可读存储介质中的程序。
[0070]
根据本发明的又一个实施例提供的一种筛选药物的方法,所述药物用于治疗或者预防炎症性肠病,所述方法包括:将候选药物施用于受试者,检测施用前后,所述受试者粪便中标志基因的丰度,所述标志基因包括第一基因集和第二基因集中的至少一种基因,其中,满足下列条件至少之一的候选药物适于用于治疗或者预防炎症性肠病:(1)进行所述施用后,所述第一基因集中的至少一种基因的所述丰度升高;和(2)进行所述施用后,所述第二基因集中的至少一种基因的所述丰度降低;其中,所述第一基因集由以下基因组成:s100_gi_0002354、s100_gi_0002360、s100_gi_0002362、s100_gi_0002364、 s100_gi_0002366、s100_gi_0002373、s100_gi_0002377、s100_gi_0002402、s100_gi_0002408、 s100_gi_0002410、s100_gi_0008128、s100_gi_0030720、s10_gi_0005425、s186_gi_0019691、 s24_gi_0044455和s254_gi_0026483,其中,所述第一基因集中的基因与seq id no:1-16所示的核酸序列一一对应,所述第一基因集中的基因与其对应的seq id no:1-16中的序列具有不小于90%的同一性,所述第二基因集由以下基因组成:s100_gi_0015747、s101_gi_0002517、s152_gi_0024776、 s15_gi_0022087、s258_gi_0009120、s47_gi_0011838、s81_gi_0011061、s103_gi_0016687、 s118_gi_0008214、s257_gi_0026750和s57_gi_0025049,所述第二基因集中的基因与seq id no:17-27 所示的核酸序列一一对应,所述第二基因集中的基因与其对应的seq id no:17-27中的核酸序列具有不小于90%的同一性。
[0071]
利用本发明这一方面的生产或筛选治疗炎症性肠病的药物的方法,通过合理有效地应用确定的炎症性肠病标志基因进行筛选,能够获得提高肠道有益基因的丰度和/或降低肠道潜在致病基因的丰度的药物。
[0072]
下面将对实施例作具体介绍。以下实施例中涉及的未特别交待的试剂、序列、软件及仪器,都是常规市售产品。
[0073]
实施例1生物标志物的鉴定
[0074]
本实施例中,发明人通过对202个炎症性肠病患者和92个健康对照的粪便样品进行研究,从而获得肠道菌群基因群落及功能成分特征。总的来说,发明人下载约150.09gb高质量的测序数据(健康人) 以及实验测序得到的470gb高质量测序数据构建了炎症性肠炎参照基因集,并且和下载的lc基因集、igc基因集构建一个更加完整的基因集。宏基因组分析显示,27个基因与炎症性肠炎疾病密切相关,其中16个基因在健康人的肠道基因中富集,11个基因在炎症性肠炎病人的肠道基因中富集。
[0075]
1、测序数据的获取
[0076]
炎症性肠炎疾病和正常对照样本取自已发表的文章公开数据集:prjna398089。
[0077]
参照图1所示的实验流程,鉴定炎症性肠病的相关基因标志物,其中省略的步骤或者细节为本领域技术人员所熟知,几个重要步骤介绍如下面所述。
[0078]
2、基因丰度分析
[0079]
2.1序列优化统计
[0080]
1)首先进行第一期测序,本期测序采集131个样品的数据,获得第一期的131个样品的测序数据以后,对其进行过滤,质控按以下标准进行:a)移除大于5个n碱基的reads;b)去除低质量碱基(q20) 大于50%的reads;c)移除尾部低质量(q20)和n碱基。丢失成对的reads序列被认为是单条reads用于组装,并进行基因预测。
[0081]
2)采用1)所述方法同样处理下载的健康人数据。
[0082]
3)lc基因集获自 qin,n.et al.alterations of the human gut microbiome in liver cirrhosis.nature 513,59-64(2014).,从 ftp://climb.genomics.cn/pub/10.5524/100001_101000/100064/1.genecatalogs/igc.fa.gz下载得到igc基因集。
[0083]
2.2基因丰度分析
[0084]
soapalign 2.21用于匹配针对冗余基因组的paired-end clean reads,这里,所称的冗余基因组来自来自于利用样本数据及igc基因集构建的非冗余基因序列集,比对参数为
–
r 2
–
m 200
–
x 1000。reads与冗余基因组的比对结果,可分为两部分:a)unique reads(u):reads只比对上一个基因的序列;这些reads 被定义为unique reads。即,如果这些序列来自同一基因,发明人将这些reads定义为unique reads。b) multiple reads(m):如果reads比对上两个及两个以上基因的序列,定义为multiple reads。即,如果比对上的序列来自不同基因,发明人定义这些reads为multiple reads。
[0085]
对于给定的基因g,其丰度为ab(s),与u reads和m reads相关,丰度的计算方式如下:
[0086]
ab(g)=ab(ug) ab(mg),
[0087]
其中,g表示基因的编号,ab(g)表示基因g的丰度,ab(ug)表示所述测序数据与所述基因g的参考序列唯一比对的读段的丰度,ab(mg)表示所述测序数据与所述基因g的参考序列非唯一比对读段的丰度;
[0088]
ab(ug)=ug/lg,
[0089]
其中,ug表示所述测序数据中与所述基因g的参考基因唯一比对的读段数目,lg表示所述基因g 的参考基因长度;
[0090][0091]
其中,mg为所述测序数据中与所述基因g的参考基因非唯一比对的读段的数目,i表示所述非唯一比对读段的编号,coi为所述第i读段对应的丰度系数;
[0092][0093]
其中,co
i,g
表示针对所述标志基因g,所述非唯一比对的读段i的丰度系数,n为所述非唯一比对的读段i能够比对的基因的总数,j表示所述非唯一比对的读段i能够比对的基因的编号。
[0094]
对于这些reads,发明人以加和的n个基因的unique reads的丰度作为标准,即对于multiple reads,发明人把其所比对上的n个基因的unique reads丰度之和作为分母。
[0095]
2.3筛选基因基因标记物
[0096]
为了获得与炎症性肠病疾病密切相关的基因标记物,发明人利用炎症性肠病患者组(131例) 与健康人组(61例)两组粪便基因丰度数据,在基因层面做了一个与疾病相关性的研究。
[0097]
基于得到的基因丰度表,发明人设置标准如下:(1)炎症性肠病患者组或健康人组基因丰度的中位数必须大于0.00001;(2)通过结合benjamini hochberg的多重检验的wilcoxon秩和检验进行检验,得到每个基因和炎症性肠病疾病的相关性p值;(3)使用一个相对严格的阈值(p值《 1e-6,fdr值《0.0005)。利用上述参数进行筛选后,发明人得到27个与炎症性肠病疾病密切相关的肠道微生物基因标志物,其中,在炎症性肠病(ibd)患者中富集的基因有11个,在健康人 (ccontrol)中富集的有16个基因,这27个基因标记物如表1所示。
[0098]
表1:
[0099]
基因p值fdr值富集人群s100_gi_00023541.56e-070.000143398controls100_gi_00023603.14e-070.000143398controls100_gi_00023624.76e-070.000143398controls100_gi_00023643.64e-070.000143398controls100_gi_00023666.60e-070.000163836controls100_gi_00023737.99e-080.000111871controls100_gi_00023771.92e-070.000143398controls100_gi_00024029.09e-070.000194334controls100_gi_00024083.74e-070.000143398controls100_gi_00024106.45e-070.000163836controls100_gi_00081287.75e-070.000180729controls100_gi_00157476.73e-070.000163836ibds100_gi_00307201.41e-084.62e-05controls101_gi_00025173.53e-070.000143398ibds10_gi_00054252.47e-084.62e-05controls152_gi_00247764.87e-070.000143398ibds15_gi_00220874.43e-070.000143398ibds186_gi_00196914.38e-070.000143398controls24_gi_00444555.29e-070.000148072controls254_gi_00264832.32e-084.62e-05controls258_gi_00091202.90e-070.000143398ibds47_gi_00118384.59e-070.000143398ibds81_gi_00110619.51e-070.000194334ibds103_gi_00166871.11e-070.000124394ibds118_gi_00082144.04e-070.000143398ibds257_gi_00267504.42e-070.000143398ibd
s57_gi_00250498.49e-070.000190113ibd
[0100]
实施例2基因标记物的验证
[0101]
为了验证实施例1中的发现,发明人进一步分析验证群体中的31个健康人及71个炎症性肠病患者的粪便样本中的所述27个基因的丰度,并根据验证情况对所述27个基因做出删选,验证群体的dna提取、测序以及基因丰度的分析参照实施例1进行。
[0102]
验证结果如下:对于上述富集在健康人群中的16个基因均在验证集中得到高质量的验证(p 值《0.05),验证群体中在健康人富集的基因标志物的p值如表2所示。
[0103]
表2:
[0104]
基因名称p值fdr值富集人群s100_gi_00023541.56e-070.000143398controls100_gi_00023603.14e-070.000143398controls100_gi_00023624.76e-070.000143398controls100_gi_00023643.64e-070.000143398controls100_gi_00023666.60e-070.000163836controls100_gi_00023737.99e-080.000111871controls100_gi_00023771.92e-070.000143398controls100_gi_00024029.09e-070.000194334controls100_gi_00024083.74e-070.000143398controls100_gi_00024106.45e-070.000163836controls100_gi_00081287.75e-070.000180729controls100_gi_00307201.41e-084.62e-05controls10_gi_00054252.47e-084.62e-05controls186_gi_00196914.38e-070.000143398controls24_gi_00444555.29e-070.000148072controls254_gi_00264832.32e-084.62e-05control
[0105]
对于上述富集在炎症性肠病患者中11个基因均在验证集中得到高质量的验证(p值《0.05),炎症性肠病患者富集的肠道微生物基因标志物验证的p值和q值如表3所示。
[0106]
表3:
[0107]
基因名称p值fdr值富集人群s100_gi_00157476.73e-070.000163836ibds101_gi_00025173.53e-070.000143398ibds152_gi_00247764.87e-070.000143398ibds15_gi_00220874.43e-070.000143398ibds258_gi_00091202.90e-070.000143398ibds47_gi_00118384.59e-070.000143398ibds81_gi_00110619.51e-070.000194334ibds103_gi_00166871.11e-070.000124394ibds118_gi_00082144.04e-070.000143398ibds257_gi_00267504.42e-070.000143398ibd
s57_gi_00250498.49e-070.000190113ibd
[0108]
发明人认为,可以将从健康人富集的16个基因标记物,作为炎症性肠病疾病患病的反向指标,或作为治疗炎症性肠病进行研发的基因制剂药物菌群成分,或作为检测炎症性肠病、监测炎症性肠病治疗进程的指标;将炎症性肠病患者富集的11个基因标记物,作为炎症性肠病疾病患病的正向指标,特别用于炎症性肠病疾病简单、非创伤式的检测和诊断。
[0109]
发明人利用这27个基因标记物,构建一个综合指标,估计roc(receiver-operatingcharacteristic)曲线下面积auc,auc越大,表示诊断能力越高,评价综合得分对应其对炎症性肠病的诊断能力。通过对一期(第一阶段)的192个样品和二期(第二阶段)的102个样品进行评测,具体情况如图2所示,都表现出了很好的诊断能力,在一期得到auc=81.7%,如图2-a所示,置信区间为72.55%-90.88%;二期得到auc=90.2%,如图2-b所示,置信区间为85.71%-94.69%。
[0110]
上述基因集中基因的具体信息如下:
[0111]
s100_gi_0002354
[0112]
atgacaagcggagaaaaggctctttatcattccattatagagcagttaaaagataaggaa ctttccgatagccaaaaagacaattttgtgaaaaagctggatgaactgttcaaaagtatg aatgaccaatcaaaagaatttcaattaaactcaattttatga(seq id no:1)。
[0113]
s100_gi_0002360
[0114]
atgaatggttacaattccggaactgagccttgctggatgccattgatggaactcatcgaa acatgcctgttcgacatcattcacacagaagatacaaccggacgattcattcgtctgtat ggagcaggtgactattggcacgcattcgaagaatccgcctaccaattgagtcagctgttc gtgacacacgatgtaactgtactcaggcataaagtttacccatttcccgttttaatggtc agcatctcggatgacgaactccaggcttacggcaaaaatcacatcttcagaaagaaagta tcagggtacagggaacttgttgggataggaatctcgatgaagcggtacaaggaatggcac aaaaaagaggtgatgaaattctcttctcttccatga(seq id no:2)。
[0115]
s100_gi_0002362
[0116]
ttgcagggtaactcgtatgtcacggtgccggaggttgccgactcctgcgggctttcctat accggactggaacagcacctgttgttctaccataaggatctggtgaagcggcgcatcagg atacgcaagaaggctctccgacggcagcggaaaggggaaatcaccggtcggggtaccgtg cacgccccctctccggagttggtggagaagtatgccgaagcggtgcatctgtatgccacg acacccatgtcggccgcacggatagccgggaagacgggagtgtcgaagaagggattctac gagcacctgcaaaggtggcatctggacctggtatgcaggcggaagaatatcccctacgag gaagggcggctagtggactggtctaaagtccggaaatacaaccccgcgacaaaggccaaa tatgccgaagccatccgccggctgaaagaaagcgggctgccgacggcacaggtcgccgcc gagttcgggctgcaacccgaagcgttccgcagctacctgaaggaacacgagccggaactg tatgcccggaagggaatggtcaggacggatacgggcggcgcggtgtcccgccgcagcatg gagaaatacagcgaggccatgcacctgtacggcacgacaaccgaaagcgtgaagtcgttg gccaggcggttcgggttcaacgactgctcgttcgggcagttcatcagaaggaatttcccc gaactggtcgaaaagcacaacgaaatcgtccagaaaaaaggaaaacaaaataaataa(seq idno:3)。
[0117]
s100_gi_0002364
[0118]
atgaagatgctgaatcatacttttttcgcctgtcttttgggcatgagtctcgtttcctgt tcacatgcccaacagcagaaaaccaaggaattccgtctgcccgacgtgcctgtcacgctg acggcgcctgaagacagggcggcgtacttgtccctgcattactgggaccacttcgatttt gccgatacgtcgctcatctcccgtcctgaaatcaccg
agcaggcctttgtcgatttcctc ggcatcctgccctataccgaccgggcacaggctgccatcgatactcttttccgccgcgcc gcaaccggacaggagatgctgtatcacttcatcgggctgagtgacaagtacctctacgag cccaattcgcccatgcatgacgaggaactgcatatcctcgtattgcgggcactgctggac aacccctgcctgtccgatacggacaagatacgtccccgctacctgttggaaatagccatg aagaaccgtccgggagacgtggcggcggatttcaccgtcacttgccgggacggcaggcgc aggcagctctccggcataaaggccgactacgtgctggtttacttcaacgaccccgattgc gaggactgccgccgggtgaaggaattgctcaccctgtccccggttgtgaacgacctgttg gagtccggccgtctgaaactcctctccgtgtgtgtggagggcaagacccccgcgtgggaa aaggcggaatttcccgccggttggatagatggttatgatgccgatcagcggcttacccgc gaacaggtgtacgacctcaaggcgatgcccaccctttacctgctcgatgcggaaaaacgg gtgatactcaaggacgcttcgttcgagcaggtggaagaacggttgtcctga(seq id no:4)。
[0119]
s100_gi_0002366
[0120]
atgatgatttgttgtgcggcggcatggcttcttgccgcttgcagcaaagatgacgatctc cccggcgggcagacaggaggtaacggcaacagcattatcctcgacatctcgtcgggagcg ttgccggtgagtcgtgctaccgtaccggccgaaggggcggaggtcgcagtgagccatatc gacgtgctgattttcaacgataccgacgagaaaacgaaggtgtggagcgaccgggtgaac gcgtcagccaatgaaacgggacggattacactgcccgtaaaacggtcgtcttttacggaa aatgagaggtattgggtgtatctgatcgccaacagcacccacccggaaaaggattttgaa aacctcgcgaacctgaacgccttgaaggcgatgacgcaggaggacgagaacattcacctg acagggaaagacattgaaggtgtcccgggaacattcctcatggacggcatagcctatccc gaagggaacgatgaaccggcggttgctgctccggtcgtcctctacgacggctcgcaaggg aatgacacgcggctggcggtcacgctgcgcagggcagcggcaaaagtcgtggtgaccatc aacaaaggccaagacgtgacttttgattccaaaggcataggttactacctgcgcaatatg ccctataccacatcggtagtggcgggtgcggacggtgcggcaaagctgaggaataccgcc cagacctccggcggctatcttgagtggacggcgaacaaaattaccgtaacggcttacatg tatgcccatgcttgggacaatggaagttccatggagcaggaagtgcgcctggtgatgaac attcccctgacttaccagaaggataccgacccgcaactgcgtcccgataactactaccag attcctgtctgtaacggcaaggccctgaaccgcaacacctgctacagggtaacggcaacg gtcaatgcccccggagcggaaaatccctccacaccggtgcagttgacggatttgaaatat accgtggagaattggatagacgagacggtaaatgtcggaggcggggaccgcccgactttc cttgtgctcaaccggtacgacctggaaatgcacaacaaggaggacgaccgcaccagcctg atcttcacctcctcgtcggaagtgacggcacagattacccgggtttactatatagacaag ttcggaagggaacagaacttggagaaaagataccccgataatccaaacgataacgaatgg ggagtgagtaccggatacagatggcgcagccatggcaacatcctcattactcccgacaag gggctcaacggcaagctggatgtacacagcgacctccccaggaacaatacgatacgttat atcgaagtccgggtaacaaatgaggacggcaacgaacgtaccgtaaccattgagcagtat ccgttggaatatatcacgaatatccaggggtggtactcgtaccgcgatgatttcaagaat tcagatcctaacccgactacctatgaatatgtaggtgataaaattaccggaatcgccctt gccaccagaaatatctccagttggaacggagaatatagatacctaacggggacagatggt ttatttggaggtcctccggacggattcttcgtttcaaaagtagcaaaattgaggaatgat ggaagcggacagtctgatttatatgaatactattacacatcaaactcaaacagacttcgg gaatcccgctatagcgaaattaacggcagaatgtacaccattgtattgacagggacatcc agtgaatatactcttggccgtccacgtatggtccaggatacataccatccggaacttatg gttacggatccgggacaggacaatgcccaattggtctcgccgtcattcatgataggttcc tgtgtaggcgaattccaagttggaggaggaaacctgaagctggacgggagtgacaacagt gtccgcgttgcacgcgagcactgcgcgcattatgtagaaacgtataaagacccgaaaacc gataggacggttaccttggatgactggcgcctgcccacagaagccg
aacttaaaatcatt tttaaattccagggctcgaaagaccaggatgcagacgccatagactaccgcttgaacgga cagtactattacagcgcaagcggaagattttataatgagaaatacaccgttagcggtacc ggagtggcctgtgtacgcgatgcctacggtaaaaaggaataa(seq id no:5)。
[0121]
s100_gi_0002373
[0122]
atgaataggctggacaaaacacttctgataaccgtgctgctgctcggcggcatcctctgg ttgcagcaccggtggacaatacgcctcacggaagaacgcgaccgtttccgcatgaacagc acagccctgctttccgatgtgaagcggatgcggaccgattcagcgacgatggccctcgat gccaaggcactgcggctgaccatagacgagtatgaggagttccgtgccgaggatgccgaa accatcaggcggctcggcgtgaaaatcaggaatctcgaagcggcggcccggcatgaggtc gaggtgaaagcaccgatagacgccgccgtccgcgacacgctcattgtccgtgataccgtc ccgctactgcggcagaaagtggagatggtcacgccgcatatacattttagcggactgatt gaagagaaccgtctcaaaggtgacataaagatacccgtgacgcttaaccaggctgtatgg gtggaatacaagggatggtggttctggaagagggtcaaagctatccgtcagaccatatcc agcaacaacccgtatgtggaaatcaggtattcggaatatataaagatagacaaaaaatga(seq idno:6)。
[0123]
s100_gi_0002377
[0124]
atgtcagacaagatagcgaacacgcaggataggccggaagagaaaccgctcacggagcag gagctgcaattctgcaacctctatgtgaacggcgggctggagtatgccggacggccgaaa aaatgctttgtggaggtattcggtgaggatacggtgaagaatccgtacgcttcggccaat tacctgatgaacaaaccgcatgtgctggcgcatatcaaggcgctgctctcctcggaacgg ttcgagatggagacgatggccgtgaagctgcaggtggccgagaccctcaaggccgtcatg gacgagacggccacttcggactataccgaccgtttcggagtgccgctttcccctgcgccg ctcagggccgtgtcggtcaatgccgccaaggcgctgatggatatcttccccatcaggcac aaggaggagaaccgcctgcgcatcgagggcagtgacggcaatgtcatcttcaatgtgatc gtaccccaaaaccctatgacagatggtgaaaaggaaacataa(seq id no:7)。
[0125]
s100_gi_0002402
[0126]
atgaacatcaccaacgtaacggtaaccaaggttgccgaagagagcacggagaatgccgac taccagttggaatattccatcgtcaatgacgcgctgacccgcgtccacgcctccatccgc aagaaggatacggatggctcggggaacgctccgcagataggtatcatctacatggaacaa ggcgttatttcatgcaacattcctatgggagagccgcttgcacccctgtttcatgatttc gataccatgatcgacgaaataaaaaagagtaacgtacaaaatgcataa(seq id no:8)。
[0127]
s100_gi_0002408
[0128]
atgggcaggatacgacacctataccatctcatcttcgttaccaaaggacgcaagatgacg attccgacaacgggaaaaggattgatgctcgagcggatgacacggatttttacagggaaa ggttgccgcgtacatgccgccaatgcctttctcaaccacgtgcatgtacttgtcgagatt ccgaattccggggatttcagcaagatcgtgaacaaggtgaaaggcgccacgggaacggta taccagaaaaccccggaatatgccgacttctccggttgggctgccggattcgactcgttc agcgtgtcgttcagcgaccttcaccggctcagacggcacatcgaggagcagcatacaatc catcaggagatctcgttcgaggaagaatacggacaactgcttgaagagaatgggttcgac ccgcatcaaacgtccgtgccggcatcctccacggtacatttttccgcgaatacccaccct aaaaacggaatcaaatga(seq id no:9)。
[0129]
s100_gi_0002410
[0130]
atgaaagtattaaagaatttgagcctgatggtgctgcttgcgctggcatttgcaggctgt cgtggcaagagttcccaacttgccgatttcaacaaacagctttacgctcccgaatatgcc tcgggattcaagattgaaagag
cagacggcagacagagcgtgctggtttccgtcatgaat ccgtggcagggcgcggatagcgtcaccacccggttgttcatctcccgtaacggggaaccc gttcccgagggtttcgacgggcaggtcctcgaaggggatgcgcagcgcatcgtggcgatg tcctcgacccatatcgccatgctcgacgccatcggcgagacggcacgtgtggtcggcgtt tcgggactcgactatatctccaatcccgacattcaggcacgccgcgacagcatcggcgac gtgggttacgagggaaatatcaattatgaacttctgttgtcgctcgaccctgaccttgtg ctgcttttcggcgtgaacggggccagtgcgatggagagcaaactcaaggagctcggcatc ccgttcatgtatgtcggcgattatctcgaagaatctccgctgggcaaagccgaatggctg gtggcgctttcggaggtcgtcgggaaacgcgcgaagggcgaaaaggtattcgcggatatt ccgatccgatacaatgcgctgaaacagaaagtgtccggtgccgttctcgatgccccgtcg gtcatgctcaatacgccctacggcgattcgtggttcatgccctcgacggaaaactatgcc gtgcggctgattaccgacgccggaggcgactacatctacaagaagaacacgggcaactct tcgacgcctatcgacttggaggaggcatacctgctcgcttcggaggccgatatgtggctg aacgtcggcatggcgaacacactcgacgaactgaaagccgcctgcccgaagttcgccgat acccgctgcgtccgaaacggctatgtctataacaacaacgcccgcaccaatgccgccgga ggcaacgactactacgaatcggccgtcgtgaatcccgaccttcttctgcgcgacctcgtg aagatattccaccccgagttggtcgaagaagatttcgtatactacaaacaactgaaatag(seq idno:10)。
[0131]
s100_gi_0008128
[0132]
gtgtggcatttcctgccacgcgcccaaaaatttgccgtgcatttcaccctgcacggcaaa atttttcgtaactttgtcgcacttaaaacgtaccccatagcaggctatccaaatgagcgc attttcggcatatcgtgtatcggcttactcacgcatcactcgtgtcgtggctctgcgctg ctttactccaatttcggcaagacgcttctctggcacgtcccggatctttcccaatccggc agctaa(seq id no:11)。
[0133]
s100_gi_0030720
[0134]
atgattaggtatattgaaaaaggcgacatattccgtattgatggagtgagcagttatgcg catggttgtaattgtgccggtgcaatgggtaaaggcattgccgtacagttcaaaagcaaa tatccagatatgtatcttgaatataagcagttgtgcaaagaaaataagttttgccccggt gatgtttttgattatgattatggcaatggacatatatataaccttggtactcaagcaacg tggcgaacacgagctaaaatagagtatatcgaaaaggcactgatacagatgcttgagtta gccagtggtgataatgtaacggcaatagccttacctgcaatcggtgctggattaggagga ttaaaatgggatgatgtaaaagcaattatagagacagtttcaaatgattatcctaatgtt gatttgtatgtagttgaaacttatcaaagcgaataa(seq id no:12)。
[0135]
s10_gi_0005425
[0136]
atgtcattattgcacggaacggaagaggtgcgcgcactgcatgcctctacctgttatgga atggaaagatatagaaatggtgcgcttgatgaaacggaaagccggtcccgccaatcttca tttaatatccggacatggccggataagtcatcggtaatgcagaattgttgcaatgtttcc tgttcggcgcaggctgccggaaacaaccgtatagctgtacagatggcaggatttaccggc acaatgggtctggcggatgtcaaatctgtggattggatggttttccaaactactgttctg gaccaggcccactcgtgcccgttataa(seq id no:13)。
[0137]
s186_gi_0019691
[0138]
cctttaatgaggattatccaccgaacattagttgcggtcaaaaacgaaataaaaatgaat gtaatatcagaaaaagaatattcgttttcaaatgcgcttttttggaacgtaatgttacat catcatattcgagcctttgatgaagaaagggatgcgaattttgatgaagtatgggatgaa gaacttataccaacattgttggatgaaaagaagtacaagaaatattggtgttggctatct caaattgatttgaaaacatccgaaaatcaaggtgaaatagaaaacccaagaactctaaca cttcctatcggcagcgatattgtattgacaatagaatttcatccttgctgtacatattat tttttg
aatgacactttaatcggggaagttagtggcaactttcacttgaagtatctcaca tattcggaattaatgaggattacaaaagagaaatatggagatgtactgttccatttactc ttacctttgtctgcaatcagggaacaagaaaaagacatagcacttaatgaaataatccaa cgattgcaacaaattcccttattcaaggaacattccgcatatataggtaaatgtatcttg aatggattattaatatccaactctgacatacaagaaattcccaagataggaacgatatgt acaagcaataattcatatagaaatgctttggtatatgatgatgaaaggcaggaaataaag gagctaaatatccttttatcgagattataa(seq id no:14)。
[0139]
s24_gi_0044455
[0140]
aaaaagcgtgaatgccaaactgaccaacgggcaggcggaagagatacgggtgagatattc ttccggccaggcttcccagaacagcctggcaaaacagtacggcgtcagccgccagacggt aagcgcgataatacgatacaagaaatatatacgataatggacgaggaagaaatttggaaa gatattcatcattacgagggaatttatcaagtaagctcctgcggtagaattagaagtgtc ggaagatatgtccgtaatgccacatcccaatattggaggaacggacaaatcatcaaaccg tgggtcgggggtacatccccgtattacaatgtttctttggcctctgacggaaaaatatgt aaaaaattaatccatagacttgtcgcagaacattttcttgatgattggaatgcggcaaag gaggttaatcataaggatggcaataagcacaataatcgaagtgacaacctggaaatgtgt accaggcaagaaaacgtcatgcattctatggtgcataaactcagggatgattatggagag aacagtgtgaacgccaaactgacaaatgaacaagcggatcatatcaggcggttacaccgg tcgggagtcatgcaaaatgaactggctgaacaattcaaggtttgcaaacagacgatatgc gatatcgtccactataaaaagtataaaaaatga(seq id no:15)。
[0141]
s254_gi_0026483
[0142]
gatctcgtgcaggtcattctgcaccgtgttgagctgcatcatggtcgcctcgaacacgga cttgtccagcttcatggcgttgatgcggtcatactggttcttgatttccacttccaaacc gatacatttggtctccagttctgcaatctgggccgtgttgttcacatgctgtatgtacag tgtcagcgagaacgaaagtaccacgacgataatcttgaagtacttcactacaaattcctt gaattgttccatatcattattccattaaaagtgagaatgcctccctga(seq id no:16)。
[0143]
s100_gi_0015747
[0144]
atggaatcatcaatcaaggacaaatacatcatcttgggctttgtcggcttcgccatcgtc ctaatatcttccattgccacgctggtaatagcggacagcttcaaccaagacaactttgtc aggtggatagtattcgtatgctgtaacctgttgggatggttgctctatctctcctttcag acacttatctttgatacatacgaaatctacaaaatcaagttcggcaagaaagaaacgatt gccgaagccatagaggtgcaggaagaactgtcacaaaatacacttgaagaagccacatct gtgcctggacctacatcagtccctgagcctgtacccgaatcatccccgacaaaagaagag acacttatccaaacacaaccgatagagcttactatcgccccggatcttcacgaaaagaac cgtgccaattacgcaagcagagagcaacgggaaaaggaagagcgcatccgcatggtcatg gagtattgccattattacctgcctcgcattgccgaccaagaaaccgtgaaccacatctgt actgaggtggacaaatggatgaatcttaacacttataccccgaagcccatacaaagaccg tttaccaaagacatcaacaacattccactccgtcacttcgtatggaatatctctgagcgt ttcctgtacaagagatactacaatggggataaccgtgccaagttcatcaaagcccttttc ccgaaatcgtttgctgatacagacttatcaaccatcaagaatttcaaggtagagccgtta aagacggaaattcccattgatgaacccgaaaacggcaaacttgatttccactatcccgag gattatgtgcggaattag(seq id no:17)。
[0145]
s101_gi_0002517
[0146]
atgaaagcaaccagaaaatgcagtttttgcggcaagtcctttgtaacccgaagcggtatg caaagatattgcagtgaggcttgtcaggcagaagccaaacgagccagagtgatgcagaag aacaacctcttcaaagtcgccc
aacccttgatggagatacagcatcaggagtatctcacc ttttccaaagcagccatcctcatgggctgttcccgacagtacatttacaaacttgtagcc atcggcaagctgaaagcctcacgcatcagcaaccgcatggcattcatccgcagagccgac atcgagcagatgttggagggcaatccctatcaccgcatcctgcccggcaacacctccaca ccaaggaaatcatcttcatcttccttacctgccaaaagagaaaaaagggaaaaggaaagc gaagaagtgttggacttctattcgggcgaggaggtgatgtccctttttaaggtaaagcag tcatggctttacacttccgccaagcgtaaccatatccccatctgccgtatcgcaggaaag aactattacagcaagaagcatattgacgagtttttcggtgtggcagttgatattagcgaa attaccgactggctactgaccgaggaggtggaggaactgttcggcatgaagccgaccgca ctccgtgcctacacctatcgccataagatacccactaaaagagagtacgggcgtacctat tactccaaatcacatttgaacgaactccgcagaactgaccttgtgaacgatgaacgctac tataccgttgagcaggtgcagcaaatctatggtctttcgtcagccaacatctgccatatc gtcaaggtgaagcacatcgaaaagataaaggtgggtgtgaaaaacctgcttttgcgctca gatgtggagcgtgtcatggctgaaaggaacaaataa(seq id no:18)。
[0147]
s152_gi_0024776
[0148]
agatattctcgtcaaacaaatataaataatataaacatggaaaaaaacagaaaaaaacaa atcgtagttttgagtatagctttagtttgcattttcatcttggtattttcattgttccat aaatcagcgacaaaagatagcgcaaatcctcctttaacaaatgttttgactgatagcatt tctcaaattgtctcagcttgtcctggcgaaattggtgtggcggttattgttaataacaga gatacggttaaggtcaataataagagtgtttatcctatgatgagtgtgtttaaggttcat caggcattagctctttgtaatgactttgacaataaaggaatttcacttgataccttagta aatataaatagggataaacttgacccaaagacttggagtcctatgctgaaagattattca gggccagtcatatcattgacagtgagagatttgctgcgttatactcttactcagagtgac aacaatgcaagcaaccttatgtttaaggatatggttaatgtcgctcaaacagatagtttt atagccacactcattcctcgttcaagttttcagatagcttatacggaagaggaaatgtcg gctgaccataacaaggcttactctaactatacatctcctcttggtgctgcaatgttgatg aatcgtttgtttactgaaggtcttatcgatgatgagaaacaaagtttcattaagaatacg ttaaaagaatgcaaaacaggtgtagataggatagcagctccacttcttgataaagaaggg gttgttatagcgcataagacaggttcaggttatgttaatgaaaatggtgttcttgcagct cacaatgatgttgcctatatatgtctgcctaataatatcagttataccttagcggtattt gttaaggatttcaagggaaatgaatcacaagcgtcacaatatgttgcgcatatatcagct gtagtatattctttattaatgcaaacttcagtaaaatcttaa(seq id no:19)。
[0149]
s15_gi_0022087
[0150]
cgtgtcgtggagatattcacggatatatcatttcagtcctttgcgccaagtcttacaaaa gagacgagtacgcgaatggaaaaatcaattcttaccttcaacgacctccccgaggttgtc gctcagcttcgagacgaagtgatgagcctgaaaagcctgctcgccgagcagcgcagtgtg aacaatgccaaaacggtggacacccacgtgcccatgtctgtggacgaggcagcagagtat ttaggtatccctaagggtacgctctacatgaaactgtcagaagggacaatccctgccacc aagcccggcaaacgctattgcctttaccgtgacgaactggacaagtggctggaaaccgcc cgaaagaatcccataccgttgtcagacgaggaactgaacaagtccttatcctcttcccac cgtcgcaagcccaacccacgtaactggtga(seq id no:20)。
[0151]
s258_gi_0009120
[0152]
atgggcaattattcattacagaagtataaaggaacggcaacacggcatacctgccccaaa tgcggagacaggcattctttcgtctattacgtggacgaaaataatgtgccgttgcatcca tcggtcggcagatgtaaccacgaaagcggttgtgggtatcactacactccgaaagagtat tttcaagagcatcctgaacacagaactaccaatgatttctcttttgacaggcaaagagca gagcagaagaaagtgaagcagcaaagtaagccgacagccatcggctatattc
cccctcac tatgtggagaagtcgcaaagcgagcgtagcaatttcttccgtttcctcttcacactcctt acttcctactatggcgacaaggcgaaagaggtgttgaagcggttgttggaggaataccgt ttgggggctacccgtgacggctctgttatcttttggcagatagacaggacgggcaaggta cgcacgggaaaggtacgcacgggaaaggtgatgcagtacaatcccgaagacggacaccgt atcaagggaggacagacatcggcagtgaactggatacacagcatattgaaaaagcagcgt gtgttggcagaggattggcaactatcccaatgccttttcggggaacacttgttgaaaacg catcccgacaaggtggtggtcttggtggaatccgagaagagtgccgttatcggttctgct atcttccccgattatgtatggctggctacgggtggtaagagtcagatgagagaagagaaa ctccgtgtactgtcagggcgaaccgtgcttctctttcccgatgccgatgcttatgccgag tggaaacagcgagccgagagcatgtacttttgtaaggtggtggtttcggacatcatcgaa aggaatgccaccccgaaacaaaaagaagcccatatcgacatagccgattggattatcttt cagatacgggagggcaaggtgatgagtacagccaaccacttggtcgaggctgagagaatc ctccagcggatgatagagaagaatcccgtcctgcaaaaactgatagacgatttagacctt gtgctggtcggtgcatctccaatcggcaacgatgatgaaaaacctccctga(seq id no:21)。
[0153]
s47_gi_0011838
[0154]
aatgatatggaagaggataagaactatatcaacctgatacgtggcgacctcacaaaagca tcccaagcgcataacggtatgcccgacagtgtaggcatgatgaatatcaagacggcaaac caaaccattcttgaagcatcgttattgcctacgccccgtgcgctgtgggacagcttttgg tacgagggggaactctcctgcttgtttgccgattccaacgtgggcaagtccatccttgcc gtgcagatagccgaccgcatcgcccgaaccgacaatgtgctgtatctggactttgaactg tccgaaaagcagttccagctccgctataccaacgagcatggagagctctacacctttccc gacaaaatctatcgggtgtctattgactgcaaccagcttttggatgccaactttgaggaa gctatcataggcggcattgaacagatggctgtgcagaccgactgcaagattttcatcatt gacaatcttacctacctgtgttgcgccatggagaaaggcgatgccgcaggacggctgatg attcagctgaacaatctcaaaaagagatatgcgctctctatccttgtcctagcacatacg cccaaacgctctttggattgtcccatcacatccaacgaccttgccggaagcaaacggctc tacaatttctttgacagcgtgttcaccattggaaaaagtgcccaagacggagggcttcgc tatgtgaagcagcttaaagtgcgctatggcacgttctctcatgatgcggataatgtaatc gtttacgagattgacaaggtggatgctttcttgcagttcgtgttcaggggctattccacg gaaaaggaacacttgaaaaaattgggcgacaatgaatcaagccaaagggattgccaaatt ctgcaactctcccaatcgggcaagtccgtcagggagatagcctcacaggtgaattgtggc aagtccaccgtaaaccgtatcatccagcgcagcaaagagagtaaaaacgcaggtgtccca agtgtcccactgtcccaacccttagagtgtgggacaatgggacaggatgggacagccgac aatcaaccatcaaaaacggactaa(seq id no:22)。
[0155]
s81_gi_0011061
[0156]
gagacaggacccgtacttcgtaattgctgcgcccatcattcaagactgtcaaatcgggtc tttcctgtcaagccaatgatgcctatgcgtatgccaaacacatggattacggatttctcg ttccgtgaacaaacactttatccgcaactctgctatgtggtgtattggcttaactccatt tctatgggcaacacttttgttgcagatttcaagcagcttttatcgaaatacccatcagta agaactcgtttattaggctttcctcataattgggaacaagagcctttgtggagataa(seq idno:23)。
[0157]
s103_gi_0016687
[0158]
atgacccactcagggcgtttctcccctgagaacccagagcaaagagtgaccctctctttg caatctccgcttatgggttgcacccctaagaaccccatgcgtttacggacagcggaaaag caaacaataaagtacaaacccaaaaaacaagtatctatggcaacaaaatcaagcatacat atcaagccctgcaatatcgcatcgagtgaggctcaca
acaggaggactgccgaatacatg cgccacatcggagagtccagaacctatgtggttccagaactctccaccgataatgagcag tggataaatcccgacttcggcagtccggatttgcggatgcattatgacaatatcagacag atggtaaaggaaaagaccggacgtgccatgcaggaaaaggagcgtgaacgcaaaggcaag aacggtaaaatagtcaagattgcgggatgctcccccatacgtgaaggagtgctgcttgtc aggtcggacaccacactggcagacgtgcgtaaattcggtgaggagtgtcaaagacgctgg ggaatcacaccgctgcaaatcttcctgcacaaggatgaagggcattggctgaacggtcag ccggaagcggaagacagggaaagcttcaaagtcggggacagatggttcaagccgaactat catgcccatatcgttttcgactggatgaaccacgaaacaggaaagagccgaaagctcaat gacgatgacatgatgcagatgcagacccttgcatccgacatcctgctgatggaacgcggg cagtcaaaggctgtcactggtaaggagcatctggaacggaacgactttatcattgagaag cagaaagctgaactgcaacgcatggatgcagccaaacggcacaaagaagaacagataaat cttgccgagcaggaactgaaacaggtgaaatcagaaatacgcactgacaagttaaagaag acagccaccacggcagcgacagccataactagtggagttgcttctcttttcgggagtgga aaactgaaagaactggaacgtgccaacgaaaaactgcaagacgaggtttcaaaacggaac accaatattgaaaaattgcagagccaagtacagcagatgcagaaacagcatgatacgcaa atccacaatctcagagaaatgcacaggcaggaacttgacatgaaagaaaaagaactgtca cggctcgccagaatcatagacaaggctttcaggtggtttccgatgttcagggaaatgctg cgcatggaaaagttttgtgccatgctgggattctctaaagaaatgactgaaagtcttata gtcaaaaaagaagccctgaaatgtagcggtaaaatctattccgagcaacacaggcggaac tttgatataaaggatgatattttaagggtggaaaatgaccctgacgatgaaagcaggctg aacctgacaataaacaggaagccgattgccgactggttcagggagcaatggcacaggctt agatatggagcaagagtgccgcaacaggaagaaagaaaaagtagaggattcaaattataa(seqid no:24)。
[0159]
s118_gi_0008214
[0160]
atgaaagacagaaagaattcaagtctgttacgtcgtttggagaagttccagcggcttttc ttcgttgctttgttatccgtattggctgtaggggcctttgcacagagcaaaaccgtttcg ggtactgtccttgacaaaacaggtgagtctgtaatcggtgccagtgtggtggtgaaaggt actaccaatggtacaattaccgactttgatggtaagttcacacttcagaatgttcctgat aatggaacgatccaagtaagttttgtggggtacaagaccgtagatatccaagtgaaagga caatctaccgttaaaataatcttggaggaggataccgaaacactggatgaagtggtagtg gttggttatggtgttcagaaaaaaagtgatgtgactggtgctatggcacgtgttacgagt aaggttatagaggatcgtcctgttcaaaatgctttgcaggctatgcaaggacgtgtgaca ggtgtggatattacgtctaataatcgtcctggtgagttgggcgatattcgtattcgtggt aatcgttctttaactgcggataataatcctttatatgttattgacggaatacctatgtca gcaggttcgattgctgatgttaatccgagtgatattgaatctatggaaattttgaaagat gcttctgctaccgctatttatggctctcgtggtgctaatggtgttatcttggtgacaacc aaaaaaggtaaaacgggtagaactactattaattatgatggctcatttagttttggtttc ttggattcaacaactgattatatgaattctggtcaattgttggattataatcgtcagtca gctattacgggtggtacttataatggtagatatggtgacaaccctgatcctgagcgtgat aaagctttatggctgaatcccaacaatatgtcatatatggataaacgattggaaaaagta tatcagcaaaatgctgatggaacttattcttatgatccttcacaattagaaacaactgat tggggcgaattggtgacacgtacagcatttactcataatcatcagatttctctttcagca ggaacggaaacttccaaattgtatgtatcattgggatatttggatcagaaagtacctatg aaagaccaagatttcaaacgttatacagccaatataaacggtgaaattattccgactaaa tggttgaaagtgggaatgggattgaatgctactcattctattaaaaattatggtattgta agtaatacaagtaatacaggagcaaaagactcttatggtttggctatggatatgatgcca tgggtaccggcttataatgaagatggttctatactggaggttgcaagtgccgatgatcag gcttatcataatcctttgagaaatatagatgat
gcttggaatgaaacacgttattatggc gtgaacttctcttcttatgcagaattggattttggacaattttgggaacctttgaaaggt gtaaaatggcgtacgaatttaggcgctcaatatcgtaactcccgtgtgggtagttattat ggtgagggatatactaatccttttaaaaatcctgcaatggctcctaatgtagccaataat gagcacagtcagaagttgtcatggacattggaaaacttgctgtatatcaataaaacaata aaagatattcattcggtaaatattactttgttgcaatcagctgaacgttatcgtactgaa gggttgaacatgcgtggatatgaaatcacatttccttcttctttgtggtataatattggt gcatcgaataatgcaaaatatgctccaggctccaatttttctacttggtcaagagcttct tatatggcacgtgtgaattatggattgatggataaatatttgttgactgttaccggacga tttgatggcgcttctatgttggctgctggaaacaaatgggatttctttccttctgctgca ttggcatggcgtatggaacaagagaaatggatacagcaaattaattggattaaccaatta aaacttcgtgtaggatatggtgttacaggtaattctgctgttaatccttatcaaacagca ggttctgttacttctacctatgctagcatcccatttggagtgggcaatgtttcgtcgaat actattggtacaaagactaatattatgcctaactattctttaggttgggaaaaaacttca tctttaaatgttggtgttgattttggagtcttggagaatcgtattagtggtagtgttgaa tattatattgctaagacttctgatttgctgatgaatcagtctattccggtaattaccggt tatgcacaaattttgaataatgtaggtaagacagaaaatagaggttttgaagtttcgttg tcaactgtaaatgttaaaacaaaagacttcacatggcaaacggattggactttctctttg aataatgaaaaaattaaagaattggccggtggtgctacgtatgattcaacaggtccttgg atggtgggtgagcctttgaatagtttttatgatttccagtatgaccgcatttggcaaaat actcaggaggataatcatttgatggatgtttatcgttctttgggattgactttccttcct ggacaatataagattgtggatcaaccattagttgaagttccacaaggtacagaaggatca aagactgtagaaataaaagatgctgcaggtaataaaacaggtgaagttgtttcctatatg gataatggatttggtaaatttgatgataatgataagaaaatttataataaatctcctaaa tggacaggcggtatcaataattcatttacctataagaactggaatttgagcttctttact tattttcgatttggtaatacttattatggtctgacccagactattggtcgtcgaattgaa aaagatacatggtcaccaactaatacagatgcaaagtttgctcagcctactactgcaact cgtgaaactaaatatgactatgtaagaaattatacaaaaggaaatatggttcttataaag aatatagctttgtcttatacagttcctcaaagttggttgaagaagtgtggtgcaagcagt gcacaggtatatgctcaggtattgaatccgttcctttttggtggagaattggttaaagct ggtattaatccggatgatttgactggttgggatgcaggtaatcatattggtggacaaact aataatacttgtattactcgcagtcttgttttgggagtaagattgggcttctaa(seq id no:25)。
[0161]
s257_gi_0026750
[0162]
ttgcacattctaaaaggtgaagatttaacgcttttattgattattaactcaaaaaggaca tcaacttgggtcgtttcttctcttttccttgctattcgtttccgatattccacttttgtt ttggtctatctcatatgtactgcaaaggtattgatttttgtttga(seq id no:26)。
[0163]
s57_gi_0025049
[0164]
ggctttcagcttgccgatggctacaagtttgtaaatgtactgtcgggaacagcccatgag gatggctgctttggaaaaggtgagatactcctgatgctgtatctccatcaagggttgggc gactttgaagaggttgttcttctgcatcactctggctcgtttggcttctgcctgacaagc ctcactgcaatatctttgcataccgcttcgggttacaaaggacttgccgcaaaaactgca ttttctggttgctttcataccgttttatcgtttaatggttcatttcttcaatcctatatc tctgaaatggtaaacggatgtgaacggaagtcaaccattgtcgcttttctacacagtgtg actgtttccgcatatcggggcggttattgtcgcttgtcgtaa(seq id no:27)。
[0165]
实施例3个体状态的检测
[0166]
本实施例中,发明人利用89个粪便样本进行样本来源的个体状态的检测。
[0167]
参照实施例2的方法确定各粪便样本中表1所示的s100_gi_0002354、s100_gi_0002360、 s100_gi_0002362、s100_gi_0002364、s100_gi_0002366、s100_gi_0002373、s100_gi_0002377、 s100_gi_0002402、s100_gi_0002408、s100_gi_0002410、s100_gi_0008128、s100_gi_0015747、 s100_gi_0030720、s101_gi_0002517、s10_gi_0005425、s152_gi_0024776、s15_gi_0022087、 s186_gi_0019691、s24_gi_0044455、s254_gi_0026483、s258_gi_0009120、s47_gi_0011838、 s81_gi_0011061、s103_gi_0016687、s118_gi_0008214、s257_gi_0026750和s57_gi_0025049 的丰度,判断各样本中的这27种基因的丰度是否落入各自在疾病对照组或者健康对照组的丰度的 95%的置信区间,判定这27种基因的丰度均落入疾病组的对应区间的样本所对应的个体的状态为炎症性肠病患者,判定27种基因的丰度均落入健康组的对应区间的样本所对应的个体的状态为非炎症性肠病患者。
[0168]
结果显示,利用本实施例所述方法能够对89样本进行个体状态判断,而且这89个粪便样本中的85个粪便样本的个体状态判断结果与记录的该样本来源个体的状态一致。
[0169]
另外,发明人发现对表2和表3中的基因联合检测,例如检测表3中的基因标志物被富集,同时表2中基因标记物不被富集,能够更准确的判断发现炎症性肠病患者或易感人群。
[0170]
在利用标志物治疗炎症性肠病的方案中,发明人发现使3中的基因标志物生长得到抑制或者清除,同时使表2中基因标记物被富集,治疗效果极佳。
[0171]
在本说明书的描述中,参考术语“一个实施例”、“一些实施例”、“示例”、“具体示例”、或“一些示例”等的描述意指结合该实施例或示例描述的具体特征、结构、材料或者特点包含于本发明的至少一个实施例或示例中。在本说明书中,对上述术语的示意性表述不必须针对的是相同的实施例或示例。而且,描述的具体特征、结构、材料或者特点可以在任一个或多个实施例或示例中以合适的方式结合。此外,在不相互矛盾的情况下,本领域的技术人员可以将本说明书中描述的不同实施例或示例以及不同实施例或示例的特征进行结合和组合。
[0172]
尽管上面已经示出和描述了本发明的实施例,可以理解的是,上述实施例是示例性的,不能理解为对本发明的限制,本领域的普通技术人员在本发明的范围内可以对上述实施例进行变化、修改、替换和变型。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。
