梅花鹿全基因组snp分子标记组合、snp芯片及应用
技术领域
1.本发明涉及分子标记开发技术领域,具体而言,涉及梅花鹿全基因组snp分子标记组合、snp芯片及应用。
背景技术:
2.单核苷酸多态性(snp)是指:在基因组水平上由单个核苷酸变异引起的dna序列多态性。snp标记具有准确性高、变异丰富、操作简单等优点,被广泛应用于动物个体鉴别和种源鉴定。目前,cn112210611a专利公开了4个用于鉴别梅花鹿北海道亚种的分子标记,cn107447020b专利公开了24个梅花鹿个体识别的分子标签,现有的这些专利虽然能够一定程度上用于鉴别梅花鹿个体,然而仅仅28个位点难以满足梅花鹿群体的分型和种源鉴定需求。
3.鉴于此,特提出本发明。
技术实现要素:
4.本发明的目的在于提供系统完整的梅花鹿的snp标记,以期后续用于梅花鹿群体的分型和种源鉴定。snp位点的开发对于梅花鹿全基因组关联分析、遗传图谱构建、梅花鹿群体分型和鉴定以及梅花鹿资源的保护具有重要的价值。
5.本发明提供的snp标记具有准确性高、变异丰富、操作简单等优点,且开发出的分子标记不受环境因素影响,能直接反应动物基因水平的差异,因而可以被广泛应用于梅花鹿个体鉴别和种源鉴定。
6.本发明是这样实现的:
7.本发明提供了一种梅花鹿全基因组snp分子标记组合,其包括43316个snp分子标记,43316个snp分子标记的位点信息具体如下:
8.9.10.11.12.13.14.15.16.17.18.19.20.21.22.23.24.25.26.27.28.29.30.31.32.33.34.35.36.37.38.39.40.41.42.43.44.45.46.47.48.49.50.51.52.53.54.55.56.57.58.59.60.61.62.63.64.65.66.67.68.69.70.71.72.73.74.75.76.77.78.79.80.81.82.83.84.85.86.87.88.89.90.91.92.93.94.95.96.97.98.99.100.101.102.103.104.105.106.107.108.109.110.111.112.113.114.115.116.117.118.119.[0120][0121][0122]
其中,位点编号中左侧1~33表示位点所在的染色体,中间数值表示位点在染色体上的位置,位点编号中右侧碱基类型表示该位点在参考基因组中的snp碱基,参考基因组的全基因组序列的版本号为:mhl_v1.0。
[0123]
经过样本测试验证,发明人发现,上述snp分子标记组合对于梅花鹿样本数据量在1.5gb时位点检出率在95%以上,表明本发明提供的snp分子标记组合有用信息量多,具有较大的利用价值。
[0124]
本发明提供的snp分子标记组合在梅花鹿全基因组33条染色体上均匀分布,可以提高相关研究应用的准确性。
[0125]
本领域的技术人员可以根据上述snp分子标记组合开发相应的检测探针、检测引物。
[0126]
本发明还提供了一种梅花鹿全基因组snp分子标记组合,梅花鹿全基因组snp分子标记组合还包括139个gwas分子标记,139个gwas分子标记的位点信息具体如下:
[0127]
1_21328625_c3_112183465_g5_88305985_a7_66091155_a10_3298969_g20_16138247_g1_55465409_c3_37243723_t5_88353618_t7_89934875_c10_19285550_c20_34170371_t1_21328625_c3_45633540_c5_12436359_t7_57408335_a11_19056847_c20_51189148_t1_55465409_c4_28354294_a5_20738265_c7_56997980_c11_1160873_a20_49326097_a1_79155872_a4_49495894_c5_21848587_a7_56980012_a11_954791_a21_40393065_t1_54013883_g4_109314507_g5_24025004_g7_56206726_c11_943935_t21_51005710_t2_62606395_a4_7633884_c5_24127218_g7_56045457_a11_563730_a21_49117095_t2_62531769_g4_4405726_c5_24593086_c7_12896661_g12_56981873_g21_49040655_c2_62508884_t4_4296015_g6_16841362_a7_46448361_c12_54457152_a21_24942219_c2_62377776_t4_4274291_t6_25736030_g7_46326061_t12_53570448_g21_24391167_c2_93165765_g4_69819988_t6_30709714_a7_46236692_g13_55632186_c21_22781159_c2_93038630_g9_23581832_c6_56637998_c7_44568678_c13_55168715_c21_21451926_c2_48957417_a9_21990515_c6_69198357_c7_25886356_a13_55104019_c21_21289006_g2_17520848_c9_39293019_c6_70982537_a8_60716503_a13_55060780_t22_11340107_t2_17894479_a9_70380010_t6_71320845_g8_60438594_a13_55042407_t22_19387129_t2_82496873_a9_70197423_g30_17964474_g8_60396623_g13_55026992_a23_30142419_t15_6154759_c9_65739489_g30_16929045_c8_59823615_c13_54963147_t23_42089261_a15_6304577_c14_61733092_c30_15046844_c8_59787533_g13_54949621_c23_41827741_g15_12646176_g14_15611535_t30_7287629_a8_59723471_g13_54732126_c23_41675429_g17_47279338_t14_15584339_a30_24530425_c8_56079914_g13_54712074_a23_25136409_a18_54959284_g14_41242090_c30_24390252_c8_53467269_t13_54692225_g23_24796660_g19_36698660_a24_23200546_c27_43002013_g28_31577055_t13_54617776_g23_7733789_g19_38266843_g25_39946779_a27_46067259_t28_24845720_c31_34022466_t31_20728636_t19_8512596_g27_13749787_a27_46081075_c
ꢀꢀꢀ
[0128]
其中,位点编号中左侧1~33表示位点所在的染色体,中间数值表示位点在染色体上的位置,位点编号中右侧碱基类型表示该位点在参考基因组中的snp碱基,参考基因组的全基因组序列的版本号为:mhl_v1.0。
[0129]
本发明还提供了一种梅花鹿全基因组snp芯片,snp芯片包含上述43316个snp分子标记组合和/或139个梅花鹿全基因组snp分子标记组合。
[0130]
由上述的梅花鹿全基因组snp分子标记组合单独或组合使用、或梅花鹿全基因组snp芯片在梅花鹿基因分型中的应用。
[0131]
由上述的梅花鹿全基因组snp分子标记组合单独或组合使用、或梅花鹿全基因组snp芯片在梅花鹿全基因组关联分析中的应用。
[0132]
由上述的梅花鹿全基因组snp分子标记组合单独或组合使用、或梅花鹿全基因组snp芯片在梅花鹿聚类分析及亲缘关系鉴定中的应用。
[0133]
由上述的梅花鹿全基因组snp分子标记组合单独或组合使用、或梅花鹿全基因组snp芯片在梅花鹿遗传多样性分析中的应用。
[0134]
由上述的梅花鹿全基因组snp分子标记组合单独或组合使用、或梅花鹿全基因组snp芯片在梅花鹿辅助育种中的应用。
[0135]
由上述的梅花鹿全基因组snp分子标记组合在制备梅花鹿全基因组snp芯片中的应用。
[0136]
通过制备snp芯片,结合液相探针杂交的靶向基因型分型技术,容易实现标准化自动化检测和分析。
[0137]
由上述的梅花鹿全基因组snp分子标记组合在制备梅花鹿全基因组snp位点检测试剂盒中的应用。
[0138]
上述snp位点检测试剂盒包含检测上述梅花鹿全基因组snp分子标记组合的探针和/或引物。
[0139]
本发明具有以下有益效果:
[0140]
本发明提供的snp标记具有准确性高、变异丰富、操作简单等优点,且开发出的分子标记不受环境因素影响,能直接反应动物基因水平的差异,因而可以被广泛应用于梅花鹿全基因组关联分析、遗传图谱构建、梅花鹿群体分型和鉴定以及梅花鹿资源的保护。经过样本测试验证,发明人发现,上述snp分子标记组合对于梅花鹿样本数据量在1.5gb时位点检出率在95%以上,表明本发明提供的snp分子标记组合有用信息量多,具有较大的利用价值。此外,本发明开发的snp分子标记组合在梅花鹿全基因组33条染色体上均匀分布,可以提高相关研究应用的准确性。
附图说明
[0141]
为了更清楚地说明本发明实施例的技术方案,下面将对实施例中所需要使用的附图作简单地介绍,应当理解,以下附图仅示出了本发明的某些实施例,因此不应被看作是对范围的限定,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他相关的附图。
[0142]
图1为设计的探针及覆盖度统计结果图;
[0143]
图2为位点测试结果图;
[0144]
图3为snp标记在不同染色体的分布情况图;
[0145]
图4为snp标记在不同染色体的均匀分布图;
[0146]
图5为gaps分布情况结果图;
[0147]
图6为maf分布统计结果图;
[0148]
图7为snp标记在基因结构上的分布情况。
具体实施方式
[0149]
为使本发明实施例的目的、技术方案和优点更加清楚,下面将对本发明实施例中的技术方案进行清楚、完整地描述。实施例中未注明具体条件者,按照常规条件或制造商建
议的条件进行。所用试剂或仪器未注明生产厂商者,均为可以通过市售购买获得的常规产品。
[0150]
以下结合实施例对本发明的特征和性能作进一步的详细描述。
[0151]
名词解释:
[0152]
snp(single nucleotide polymorphism):单核苷酸多态,指在基因组水平上由单个核苷酸变异引起的dna序列多态性。
[0153]
目标区段(target segment):目标区段的大小一般为100bp左右,区段内可能只含有1个snp位点,可能含有多个snp位点。每个区段内以最终测试挑选的snp位点作为核心位点。
[0154]
maf(minor allele frequency):最小等位基因频率(次等位基因频率),通常是指某位点在特定群体中的不常见的等位基因发生频率,例如tt,tc,cc三个基因型,在群体中c的频率=0.36,t的频率=0.64,则等位基因c就为最小等位基因频率,maf=0.36。当snp存在三个等位变异时,将第二常见的等位基因频率定义为maf(https://www.ncbi.nlm.nih.gov/projects/snp/docs/rs_attributes.html#gmaf),例如snp位点有三个allele:a,c,g,a的频率=0.5,c的频率=0.4,g的频率=0.1,则maf=0.4。一般群体分析中,maf要求>0.05,因较小的maf得到的可用信息量较少,会使统计效能降低,易出现假阳性结果。
[0155]
snp位点检出率:指某个snp位点被成功检测到的样本占所有样本的比值,一般要求≥90%,具体情况会依据目标数据及目标位点数进行调整。分析snp基因型时一般最低深度≥5x,即目标位点至少被5条reads覆盖。
[0156]
实施例1
[0157]
本实施例提供了梅花鹿40ksnp分子标记组合的开发和筛选方法。
[0158]
(1)对178个gwas位点、50个梅花鹿样本重测序数据和9个种公鹿样本位点数据进行数据挑选。
[0159]
保留178个gwas位点;对50个梅花鹿样本重测序数据按照na《10%、杂合率《15%、maf≥0.1筛选出155280个位点;取155280个位点与9个种公鹿样本位点的交集,且在交集中筛选在种公鹿中maf最高,且最多只允许一个样本为na的背景位点共60k。
[0160]
(2)进行位点评估。
[0161]
通过上述的挑选过程,我们最终得到178个gwas位点和60k的背景位点共60178个位点进行后续的探针设计评估。
[0162]
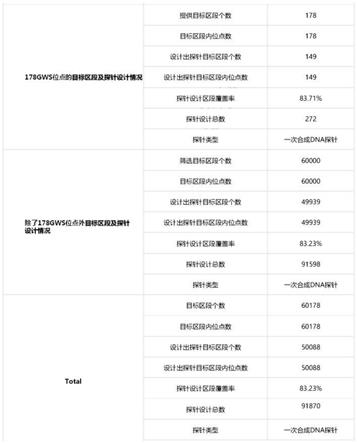
设计的探针及覆盖度参照图1所示,探针设计区段覆盖率=设计出探针目标区段个数/目标区段个数。
[0163]
通过上述的挑选过程,我们得到178个gwas位点和60k的背景位点共60178个位点进行后续的探针设计评估。其中通过评估的位点一共有50088个,但受限于一张芯片的探针数目,从49939个背景位点中删掉部分密集区域位点只挑选45181个位点,即最后用139个gwas位点和43316个背景位点共计43465个位点,对应83071根探针用于后续的测试分析。
[0164]
本实施例提供的梅花鹿40ksnp分子标记组合可以用于制备基因芯片,这些snp分子标记的应用可以极大程度上提高梅花鹿基因型检测准确度、提高检测水平,降低检测成本。
[0165]
实验例1
[0166]
对12个东北鹿样本进行检测,具体方法为:构建文库、杂交捕获、文库质检与测序分析。
[0167]
1.构建文库。
[0168]
按照genobaits文库构建试剂盒的方法进行dna文库的构建。
[0169]
2.文库杂交捕获
[0170]
利用液相基因芯片测定目标样品在40k snp位点的基因型。
[0171]
(1)dna杂交:
[0172]
取500ng已完成构建的样品基因组dna测序文库,加入5μlgenobaits block i和2μlgenobaits block ii,置于eppendorf concentrator plus(eppendorf公司)真空浓缩仪上,在≤70℃的温度下蒸干至干粉。向干粉管中加入8.5μlgenobaits 2
×
hyb buffer、2.7μlgenobaitshyb buffer enhancer、2.8μlnuclease-free water,用移液器吸打混匀后放置于abi 9700pcr仪上95℃温育10分钟,然后取出pcr管加入3μl已经合成的探针(探针的浓度为60ng/μl),旋涡震荡混匀后放置于abi 9700pcr仪上65℃温育2小时,完成探针杂交反应。
[0173]
(2)dna捕获
[0174]
向上一步杂交完成的反应体系中加入100μlgenobaits dna probe beads,上下吸打10次,放入abi 9700pcr仪上65℃温育45分钟,使磁珠与探针结合。用100μlgenobaitswash buffer i、150μlgenobaits wash bufferii分别对结合探针后的磁珠进行65℃热洗,然后再用100μlgenobaits wash buffer i、150μlgenobaits wash buffer ii(和150μlgenobaits wash buffer iii分别对磁珠进行常温洗涤。洗涤完成的磁珠用20μlnuclease-free water进行重悬。
[0175]
取13μl重悬后的dna(带磁珠)加入到新的0.2mlpcr管中,然后加入15μlgenobaits pcr master mix、2μlgenobaits primer mix配置post-pcr体系,用abi9700pcr仪进行文库扩增,扩增程序为:95℃预变性5min,95℃变性30s,60℃退火30s,72℃延伸30s;重复2-4步,共15个循环;72℃延伸5min。
[0176]
向post-pcr产物中加入45μlbeckmenampure xp beads(beackman公司)并用移液器上下吸打均匀,然后将0.2ml pcr管置于磁力架上至溶液澄清,弃去上清并用75vol%乙醇洗涤磁珠两次,用ph为8.0的tris-hcl将文库dna洗脱下来。完成探针的杂交捕获工作。
[0177]
(3)dna杂交捕获文库质检
[0178]
用qubitfluorometric quantitation(thermo fisher)对文库的dna浓度进行测定,然后用琼脂糖凝胶电泳检测文库dna的片段大小是否在300-400bp之间。
[0179]
(4)dna杂交捕获文库测序
[0180]
将构建好的dna文库由石家庄博瑞迪生物技术有限公司进行测序。
[0181]
(5)基因型数据分析
[0182]
测序数据经过:
[0183]
fastqc(www.bioinformatics.babraham.ac.uk/project)质控后,用bwa(bio-bwa.sourceforge.net)的默认参数将测序数据比对到参考基因组上,用gatk(software.broadinstitute.org/gatk)软件对测序数据进行snp鉴定,位点测试结果参照图2所示。图2显示,在其他梅花鹿群体中,本发明提供的snp分子标记的位点检出率超出
97%。表明本发明提供的snp分子标记组合有用信息量多,具有较大的利用价值。
[0184]
图3为snp标记在不同染色体的分布情况图,目标位点在不同染色体上的个数统计结果:横坐标为染色体id;左示例count为位点数/区段数;右示例length为染色体长度(单位bp)。结果显示,snp位点在11条染色体上分布较为均匀。
[0185]
图4为snp标记在不同染色体的均匀分布图,由于原参考非染色体水平,此处snp标记均匀分布图只将挑选位点所在的592个序列拼接在一起展示,图中绘图单位长度为100000bp;且因原参考由10031个序列片段组成,挑选时不考虑均匀分布,所以图中位点分布允许存在部分空洞。
[0186]
图5为gaps分布情况结果图,若两个区段间的距离大于基因组大小/目标区段数*10,则认为是一个gap。采用全部区段进行分析,分析原则:后一个区段的start位置-前一个区段的end位置,得到两个区段间的距离。
[0187]
图6为maf分布统计结果图,图6显示本发明提供的snp组合可用信息量多。区段内核心位点的maf数值统计,目标位点maf值越大说明位点的多态性越好。横坐标maf为maf值的不同范围,纵坐标count为该maf范围内的核心位点数。
[0188]
图7为snp标记在基因结构上的分布情况,显示基因间序列中的snp最多,其次为内含子,外显子中的snp居于第五水平。横坐标为基因结构类型、纵坐标为该类型下的目标位点数。
[0189]
本发明筛选出符合评估要求的43465个位点(包括139个gwas位点和43316个背景位点)和其对应83071根探针进行后续的测试分析,测试使用的12个样本数据量在1.5gb时位点检出率在97.6%-98.0%之间。
[0190]
以上所述仅为本发明的优选实施例而已,并不用于限制本发明,对于本领域的技术人员来说,本发明可以有各种更改和变化。凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。