1.本发明涉及分子生物学技术领域,具体涉及快速鉴定钩吻及其易混淆物的方法以及鉴定待测样本中是否掺入钩吻的方法。
背景技术:
2.钩吻(gelsemium elegans)为马钱科钩吻属常绿木质藤本植物,又名断肠草、烂肠草、大茶药等,主要分布于福建、广东、海南、广西、贵州、云南等地。钩吻主要作为地方性药材资源,具有祛风攻毒,消肿止痛的功效,用于治疗疥癞,湿疹,跌打损伤,风湿痹痛,神经痛等症。但该植物全株有毒,根、茎、枝、叶中均含有剧毒生物碱类成分,少量服用即可引起人体的中毒反应,甚至导致死亡,故又名“断肠草”,属于有毒蜜源之一,因食用含钩吻的蜂蜜中毒事件在我国南部了地区屡见不鲜。因此其药用时只作外用,不可内服。钩吻常见混淆品为金银花、粗叶榕、狼毒和白屈菜,金银花为忍冬科植物忍冬(lonicera japonica)的干燥花蕾,为大宗类药食兼用品;与钩吻花形态、颜色相似,并且二者花期均在夏、秋两季,极易发生误采误食事件;粗叶榕(ficus hirta),又名五指毛桃,其根具有益气健脾、祛痰化湿之效,是华南地区常用的药食同源类中药材,五指毛桃根与钩吻根常缠绕伴随共生,并且形态相近,故在采集时易发生误挖或相互混入的情况。近年来,已报道多起因将钩吻误作为金银花或五指毛桃服用而导致的中毒事件;狼毒(stellera chamaejasme)及白屈菜(chelidonium majus)为多年生草本植物,皆可做药用,但二者均有毒,并且皆被习称为“断肠草”,其相互之间的误采、误用也会对人体的生命健康安全造成威胁。
3.现有研究中,有利用dna条形码技术通过特征核苷酸序列对钩吻及其易混淆品进行鉴别(王虹等,“钩吻属植物及其易混淆品五指毛桃的dna条形码比较分析”,中国现代中药,2020年第12期)。钩吻毒性强,杨川梅等基于近红外光谱结合水光谱组学法对含有钩吻的毒蜂蜜进行鉴定,尚未见到利用高分辨率熔解曲线(high resolution melting curve,hrm)快速鉴定钩吻及其易混淆品的相关报道。
4.因此,有必要建立一种钩吻及其易混淆品的快速鉴别方法,以保障人体的生命健康安全。
技术实现要素:
5.本发明的一个目的在于提供快速鉴定钩吻及其易混淆物的方法;本发明的另一目的在于提供鉴定待测样本中是否掺入钩吻的方法。
6.特别地,一方面,本发明涉及一种用于快速鉴别钩吻及其易混淆品的方法,其包括以下步骤:
7.1)提取钩吻及其易混淆品的植物或药材的样本中的dna,获取样本中的全基因组dna;
8.2)以全基因组dna为模板,通过hrm
‑
pcr扩增,获得包含完整内部转录间隔区2(internal transcribed spacer 2,its2)序列区域的扩增产物;
9.3)对扩增过程中的hrm曲线进行分析,从而实现钩吻及其易混淆品的鉴别。
10.根据本发明的一个优选实施方案,钩吻为马钱科钩吻原植物及其药材。
11.根据本发明的一个优选实施方案,易混淆品为忍冬、粗叶榕、狼毒及白屈菜的原植物及其药材。
12.根据本发明的一个优选实施方案,hrm
‑
pcr扩增使用seq id no.1:5'
‑
atgcgatacttggtgtgaat
‑
3'的正向引物和seq id no.2:5'
‑
gacgcttctccagactacaat
‑
3'的反向引物。
13.根据本发明的一个优选实施方案,hrm
‑
pcr扩增的体系包含12.5ul 2
×
hrm pcr master mix,正向引物和反向引物(2.5um)各1ul,全基因组dna模板2ul,并且向该体系中加灭菌双蒸水至25ul。
14.根据本发明的一个优选实施方案,hrm
‑
pcr扩增的程序为:94℃,5min;94℃30s,56℃30s,72℃45s,40个循环;hrm,2s,75℃
‑
95℃。
15.另一方面,本发明涉及一种用于鉴定待测样本中是否掺入钩吻的方法,其包括以下步骤:
16.1)从待测样本中分离提取总dna并且获取待测样本中的全基因组dna,同时提取已知的钩吻样本的总dna作为阳性对照组;
17.2)以全基因组dna为模板,通过hrm
‑
pcr扩增,获得包含完整内部转录间隔区2(its2)序列区域的扩增产物;
18.3)对扩增过程中的hrm曲线进行分析,将待测样本的hrm曲线与阳性对照组相比较,从而鉴定待测样本中是否掺入钩吻。
19.根据本发明的一个优选实施方案,钩吻为马钱科钩吻原植物及其药材,待测样本包含忍冬、粗叶榕、狼毒及白屈菜的原植物及其药材。
20.根据本发明的一个优选实施方案,hrm
‑
pcr扩增使用seq id no.1:5'
‑
atgcgatacttggtgtgaat
‑
3'的正向引物和seq id no.2:5'
‑
gacgcttctccagactacaat
‑
3'的反向引物。
21.根据本发明的一个优选实施方案,hrm
‑
pcr扩增的体系包含12.5ul 2
×
hrm pcr master mix,正向引物和反向引物(2.5um)各1ul,全基因组dna模板2ul,向该体系中加灭菌双蒸水至25ul;并且hrm
‑
pcr扩增的程序为:94℃,5min;94℃30s,56℃30s,72℃45s,40个循环;hrm,2s,75℃
‑
95℃。
22.与现有技术相比,本发明的上述这些方法具有免测序、快速、灵敏度高等优点。
附图说明
23.下面通过实施方案并且结合附图来进一步说明本发明。但是,本发明并不限于此。
24.其中:
25.图1显示根据本发明的一个实施方案的基于its2序列构建的钩吻及其易混淆品的系统发育树。
26.图2显示根据本发明的另一实施方案中所得的钩吻及其易混淆品的归一化熔解曲线图。
27.图3显示根据本发明的再一实施方案中含有不同的钩吻dna浓度与混合样本总dna
浓度之比的混合样本的归一化熔解峰图。
具体实施方式
28.下面通过特定实施方案来详细说明本发明。但应理解,提供以下实施方案仅是为了说明本发明,并不旨在对本发明的范围进行限制。
29.申请人经过研究发现,最近公开一种鉴定药材布渣叶或其基原植物或植物海南破布叶的方法,其包括以下步骤:s1、根据提供的药材样本或植物样本,提取样本中的dna,获得样本中的全基因组dna;s2、以步骤s1获得的样本中的全基因组dna为模板,经hrm
‑
pcr扩增,获得包含完整的its2基因间隔区的扩增产物;s3、对步骤s2所得包含完整的its2基因间隔区的扩增产物进行hrm曲线比对分析(参见专利公开cn 113025742 a)。但是,在现有技术中,根本没有教导或暗示本发明的用于快速鉴别钩吻及其易混淆品的方法和用于鉴定待测样本中是否掺入钩吻的方法,所述方法如上所述。
30.申请人认为,这主要是因为如下原因,从而导致上述现有技术对本发明根本不存在任何技术启示:
31.首先,现有技术中使用的布渣叶与本发明使用的钩吻在组成和/或结构上相差甚大。布渣叶的主要活性成分为黄酮类化合物,而钩吻的主要活性成分为生物碱,二者的组成和/或结构差异显著,本领域技术人员不可能将鉴定活性成分为黄酮的布渣叶的方法应用到鉴定活性成分为生物碱的钩吻的方法。
32.其次,现有技术的鉴定布渣叶的方法中,在hrm
‑
pcr扩增时采用的引物为its2,其上游引物序列如seq id no.1(5'
‑
ygactctcggcaacggata
‑
3')所示,下游引物序列如seq id no.2(5'
‑
rgtttcttttcctccgctta
‑
3')所示。然而,本发明中,hrm
‑
pcr扩增使用如seq id no.1(5'
‑
atgcgatacttggtgtgaat
‑
3')所示的正向引物和如seq id no.2(5'
‑
gacgcttctccagactacaat
‑
3')所示的反向引物。显然,现有技术中的上游引物序列和下游引物序列分别与本发明的正向引物序列和反向引物序列本质上不对应。因此,本领域技术人员不可能将现有技术中使用的上游引物和下游引物替换为本发明的正向引物和反向引物。
33.再有,现有技术的鉴定体系与本发明的鉴定体系也完全不相同。具体来说,现有技术的鉴定体系涉及药材布渣叶与混淆品海南破布叶,而本发明的鉴定体系涉及马钱科钩吻原植物及其药材与忍冬、粗叶榕、狼毒及白屈菜的原植物及其药材。
34.如本文所述的术语“高分辨率熔解曲线”或“hrm”,是一种基于单核苷酸熔解温度不同而形成不同形态熔解曲线的基因分析新技术。hrm的基本原理如下:双链核苷酸(double strand dna,dsdna)的热稳定性受其长度和碱基组成的影响,序列变化会导致升温过程中dsdna解链行为的改变。因为所用的荧光染料只能嵌入并且结合到dsdna上,因此利用实时pcr技术,通过实时检测dsdna熔解过程中荧光信号值的变化,就能以生成不同形状熔解曲线的方式将pcr产物中存在的差异直观地展示出来。同时,借助于专业性的分析软件就可对测试群体实现基于不同形状熔解曲线的基因分型或归类。hrm具有极高的敏感性,可检测出单个碱基的差异,并且成本低、通量高、速度快、结果准确、不受检测位点的局限。
35.如本文所述的术语“pcr扩增”是指聚合酶链式反应,该聚合酶链式反应(pcr)是一种用于放大扩增特定的dna片段的分子生物学技术,它可看作是生物体外的特殊dna复制,pcr的最大特点是能将微量的dna大幅增加。
36.如本文所述的术语“正向引物(forward primer)”是指处于dna双链上游的引物;如用于测序,则从5
′
向3
′
方向读出dna正链的序列。相应地,如本文所述的术语“反向引物(reverse primer)”是指处于dna双链下游的引物;如用于测序,则从3
′
向5
′
方向读出dna反链的序列。
37.本发明中未明确指明样本材料为药材还是基原植物时,所述“钩吻”包括钩吻(gelsemium elegans)基原植物及钩吻药材两层含义;所述“钩吻易混淆品”或“其易混淆品”包括白屈菜(chelidonium majus)、粗叶榕(ficus hirta)、忍冬(lonicera japonica)和狼毒(stellera chamaejasme)的基原植物及药材两层含义。
38.基于上述原因的分析,本发明人通过付出繁重的创造性劳动,特别提供一种钩吻及其易混淆品的快速鉴别方法,其包括如下步骤:
39.1)提取钩吻及其易混淆品植物或药材样本dna,获取样本中的全基因组dna;
40.2)以步骤1得到的全基因组dna为模板,通过hrm
‑
pcr扩增,获得包含完整内部转录间隔区2(its2)序列区域的扩增产物;
41.3)对步骤2中的扩增过程中的hrm曲线进行分析,即可实现钩吻及其易混淆品的鉴别。
42.另外,本发明人特别提供一种鉴定待测样本中是否掺入钩吻的方法,其包括如下步骤:
43.1)从待测样本中分离提取总dna并且获取待测样本中的全基因组dna,同时提取已知的钩吻样本的总dna作为阳性对照组;
44.2)以步骤1得到的全基因组dna为模板,通过hrm
‑
pcr扩增,获得包含完整内部转录间隔区2(its2)序列区域的扩增产物;
45.3)对步骤2中的扩增过程中的hrm曲线进行分析,将待测样本的hrm曲线与阳性对照组相比较,即可鉴定待测样本中是否掺入钩吻。
46.根据本发明的一个优选实施方案,其中钩吻为马钱科钩吻(gelsemium elegans)原植物及其药材;易混淆品为忍冬(lonicera japonica)、粗叶榕(ficus hirta)、狼毒(stellera chamaejasme)及白屈菜(chelidonium majus)的原植物及其药材。优选地,钩吻的dna浓度与待测样本的总dna浓度之比为0%、10%、25%、50%、75%、90%、100%。
47.根据本发明的另一优选实施方案,hrm
‑
pcr扩增所用正向引物为seq id no.1所示的核苷酸序列及反向引物为seq id no.2所示的核苷酸序列。
48.5'
‑
atgcgatacttggtgtgaat
‑
3'(seq id no.1);
49.5'
‑
gacgcttctccagactacaat
‑
3'(seq id no.2)。
50.根据本发明的再一优选实施方案,hrm
‑
pcr扩增的体系包含12.5ul 2
×
hrm pcr master mix,正向和反向引物各1ul(2.5um),dna模板2ul,加灭菌双蒸水至25ul。扩增程序为:94℃,5min;94℃30s,56℃30s,72℃45s,40个循环;hrm,2s,75℃
‑
95℃。
51.与现有技术相比,本发明提供的方法可以通过免测序的手段实现钩吻与其易混淆品忍冬、粗叶榕、白屈菜及狼毒的快速鉴别。而且,该方法适用性广,可检测所有能获取its2
‑
hrm曲线的钩吻及其易混淆品样本,包括原植物、药材、粉末等,实现钩吻及其易混淆品的准确、快速鉴定;同时本发明适用于检测待测未知样本中是否掺杂剧毒的钩吻成分,以保障人体的生命健康安全。
52.实施例
53.下面通过实施例具体描述本发明。但是,这些实施例并不构成本发明的任何限制。
54.下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。
55.对照例:基于its2序列的钩吻及其易混淆品的鉴别
56.1)样本采集
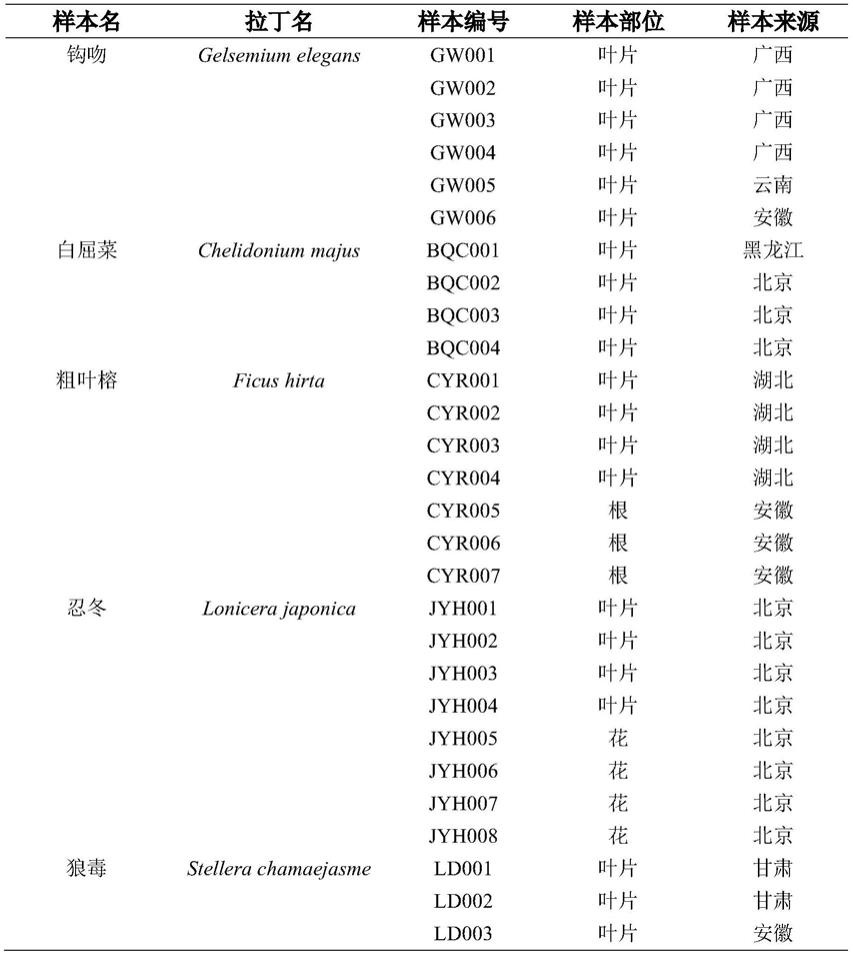
57.采集不同产地钩吻及其易混淆品的基原植物及药材样本共28份,包括钩吻样本6份,白屈菜样本4份,粗叶榕样本7份,忍冬样本8份及狼毒样本3份(按植株来源计,例如,“钩吻样本6份”是指下表1中来源于广西的四个不同植株的样本gw001、gw002、gw003、gw004,以及来源于云南的一个植株的样本gw005和来源于安徽的一个植株的样本gw006的总和),样本信息如表1所示。
58.表1.钩吻及其易混淆品样本信息
[0059][0060]
2)分别取样约30mg,在球磨仪中充分研磨后,利用天根生化科技(北京)有限公司的植物基因组dna提取试剂盒提取样本总dna。
[0061]
3)pcr扩增
[0062]
引物序列:扩增目的片段为its2完整序列,其正向引物序列如seq id no.1所示,反向引物序列如seq id no.2所示。引物序列由北京诺赛基因组研究中心有限公司合成,用无菌去离子水溶解并稀释至2.5μmol/l。
[0063]
pcr反应体系:2
×
pcr master mix 12.5μl,正向引物和反向引物各1.0μl(2.5μmol/l),模板dna 2.0μl(40~60ng),加灭菌双蒸水至25μl,进行pcr扩增。
[0064]
pcr反应程序:94℃,5min;94℃30s,56℃30s,72℃45s,40个循环;72℃10min。
[0065]
4)测序
[0066]
pcr产物直接送中国农业科学院重大工程实验室测序。为确保dna条形码序列的可靠性,需要进行正反向测序或重复测序,然后将正反向测序结果进行拼接获得完整its2条形码序列。
[0067]
5)序列拼接
[0068]
本实施例采用应用软件codoncode aligner 5.2.0(codoncode co.,usa)进行序列拼接及校对。首先,进行测序质量评估及预处理,即去除测序结果两端的低质量部分,并对剩余部分进行质量评估,如果满足质量要求,方可用于序列拼接,具体方法是:以20bp的窗口分别从序列5’端和3’端进行滑动,如果窗口内有多于2个碱基的q值小于20,则删除一个碱基,窗口继续滑动,如果窗口内碱基q值小于20的数目小于或等于2个,窗口停止滑动。
[0069]
6)序列比对
[0070]
用mega
‑
x软件对测序并拼接后得到的各个样本序列进行比对,并通过构建系统发育树对钩吻及其易混淆品进行区分,如图1所示。结果表明,尽管利用its2序列可有效鉴别钩吻及其易混淆品,但由于涉及到测序、序列拼接和序列比对的繁琐步骤,导致该鉴别方法耗时、不快速简便。
[0071]
实施例1:基于hrm分析鉴别钩吻及其易混淆品
[0072]
1)实验材料
[0073]
对照例1的步骤2)中所得钩吻及其易混淆品样本的总dna。
[0074]
2)hrm
‑
pcr扩增
[0075]
引物序列:其正向引物序列如seq id no.1所示,反向引物序列如seq id no.2所示。引物序列由北京诺赛基因组研究中心有限公司合成,用无菌去离子水溶解并稀释至2.5μmol/l。
[0076]
pcr反应体系:2
×
hrm pcr master mix 12.5μl,正向引物和反向引物各1.0μl(2.5μmol/l),模板dna 2.0μl,加灭菌双蒸水至25μl。最终,使得反应体系中钩吻dna含量分别为0ng(单一白屈菜dna组)、5ng(10%钩吻dna组)、12.5ng(25%钩吻dna组)、25ng(50%钩吻dna组)、37.5ng(75%钩吻dna组)、45ng(90%钩吻dna组)、50ng(单一钩吻dna组)。hrm
‑
pcr反应程序:94℃,5min;94℃30s,56℃30s,72℃45s,40个循环;hrm:2s 75℃
‑
95℃,0.15℃的增量。
[0077]
3)数据分析
[0078]
利用96(version 1.1)软件对步骤2)所得高分辨率熔解曲线(hrm)进行分析。结果显示,标准化的随温度变化的熔解曲线能够展示出不同样本dna序列片段之间的差异,如图2所示。这表明该方法可有效用于钩吻及其易混淆品之间的鉴别;而且,该方法
避免测序的繁琐耗时的步骤,因而该方法快速简便。
[0079]
实施例2:基于hrm分析鉴别其它易混淆品中是否掺杂钩吻
[0080]
1)实验材料
[0081]
将对照例1的步骤2)中所得白屈菜及钩吻总dna进行不同比例的混合,使得钩吻的dna浓度与混合样本的总dna浓度之比分别为0%、10%、25%、50%、75%、90%、100%;并分别设置单一钩吻及白屈菜dna作为对照组。
[0082]
2)hrm
‑
pcr扩增
[0083]
引物序列:其正向引物序列如seq id no.1所示,反向引物序列如seq id no.2所示。引物序列由北京诺赛基因组研究中心有限公司合成,用无菌去离子水溶解并稀释至2.5μmol/l。
[0084]
pcr反应体系:2
×
hrm pcr master mix 12.5μl,正向引物和反向引物各1.0μl(2.5μmol/l),模板dna 2.0μl(40~60ng),加灭菌双蒸水至25μl,进行pcr扩增。
[0085]
hrm
‑
pcr反应程序:94℃,5min;94℃30s,56℃30s,72℃45s,40个循环;hrm:2s 75℃
‑
95℃,0.15℃的增量。
[0086]
3)数据分析
[0087]
利用96(version 1.1)软件对步骤2)所得高分辨率熔解曲线(hrm)进行分析。结果显示,单一物种扩增后的熔解曲线为单峰,而掺杂有其它材料的样本会出现不同熔解温度的双峰;掺杂品比例为10%时,hrm曲线仍可反应出其差异,如图3所示。以上结果表明,利用高分辨率熔解曲线可实现钩吻中掺杂情况的分析鉴定,并且该方法具有高灵敏度;而且,该方法避免测序的繁琐耗时的步骤,因而该方法快速简便。
[0088]
尽管上文中已经通过特定实施方案和具体实施例对本发明进行详细描述,但在不偏离本发明精神和宗旨的基础上,可对这些实施方案进行修改、替换和改进,这对本领域技术人员而言是明显的。经过这些修改、替换和改进的实施方案,也均落入本发明要求保护的范围内。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。