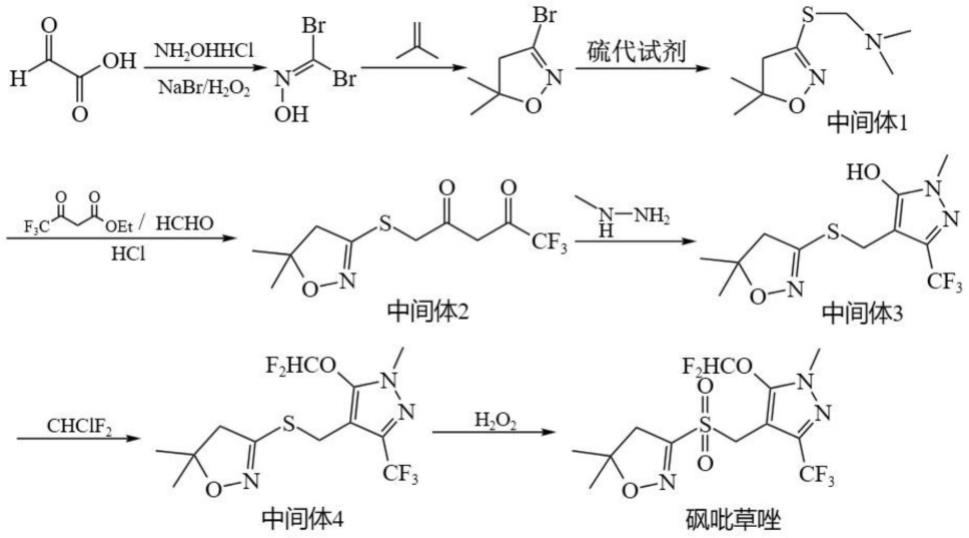
多年冻土微生物dna样本获取及dna的提取和检测方法
技术领域
1.本发明涉及冻土微生物核酸提取技术领域,具体地说是一种多年冻土微生物dna样本获取及dna的提取和检测方法。
背景技术:
2.多年冻土长期处于0℃以下低温环境,被认为是一个储存古老活性细胞的巨大“仓库”,是微生物生存的独特生境,也是探究微生物在零下温度中存活并生长的理想场所。近年来,从多年冻土环境中分离出具有再生能力的巨型dna病毒及细菌的报道,表明微生物(细菌、古菌、真菌和病毒等)能够以某种方式(如停止营养生长的休眠状态)长期存活于多年冻土环境中。尽管它们的代谢速率非常低,但绝非是以休眠状态存在的“幸存者”。事实上,多年冻土微生物在碳、氮、硫等地球化学过程中也发挥重要作用。但是,随着气候的变化,多年冻土融化趋势越来越严重,使得原来储存在冻土中的大量微生物分解。然而,对多年冻土融化释放碳的准确评估仍然是个很大的挑战,其中一部分原因是我们对微生物群落响应及多年冻土层内的微生物的代谢机制缺乏清晰的认识,尤其是其中大量未知的微生物种群。
3.据统计,中国多年冻土微生物细胞数量最高达10
10 cells/g,且不同研究区域的差异较大。相关研究所得可培养微生物数量仅占冻土微生物细胞总数的很小一部分。例如,青藏高原腹地北麓河流域多年冻土可培养微生物数量为102~106cfu/g,而细胞总数则介于107~109cells/g。造成该差异的原因有微生物自身不可培养的特性(如“侏儒”细胞)、不适当的样品预处理(如反复冻融),以及特殊的微生物类群需要特别的培养状况等。同时,多年冻土微生物数量受到各种非生物因素的影响。例如,随着多年冻土深度或年代的增加,从多年冻土中恢复活细胞的容易程度变小,微生物数量呈现下降的趋势。显然,对于多年冻土微生物基因的研究已经刻不容缓。
4.但是,近些年来,多年冻土微生物多样性研究主要集中在南北极,尤其是西伯利亚和加拿大北部多年冻土区的研究最为丰富。多年冻土蕴藏丰富多样的微生物类群,包括细菌、古菌、真菌、蓝细菌、病毒等,其中包括大量未知的稀有或特有微生物。特别是病毒,人们对其物种多样性的认知至今依然十分有限。多年冻土中生存的原核微生物为病毒提供了宿主。病毒不仅影响着原核微生物群落的组成,而且通过裂解宿主细胞和降低原核生物群落的生长率,进一步影响多年冻土中的物质循环。同时,值得注意的是,多年冻土病原微生物有可能通过复苏感染、基因传播等方式导致生物安全风险。人类面对史前病毒并没有很好的防御手段,而群居的生活习性也会导致病毒的扩散速度极快,对人类和动植物造成影响,威胁生物及生态安全,因此对多年冻土中病毒dna的研究具有重要意义。
5.青藏高原是全球中低纬度、高海拔地区最大的多年冻土分布区,其多年冻土发育特点明显,表现为活动层较厚、浅层冻融现象频繁,多年冻土层埋藏深、温度高、含冰量低、稳定性差等的特点。迄今为止,高原多年冻土微生物的研究极为缺乏,尤其是病毒相关研究基本为“盲盒”。而核酸作为遗传信息的携带者,是基因表达的物质基础。在生物的生长、发
育和繁殖等生命活动中发挥非常重要的作用。专家预测,未来青藏高原小于10米厚度的多年冻土可能会消失,而多年冻土中的古病毒即会释放,但是目前对于多年冻土中的病毒dna的研究几乎为空白。同时,以核酸测序为基础的宏基因组测序技术成为微生物生态学研究最主要的方法学之一,是当前揭示微生物(细菌、真菌、古菌和病毒等)群落结构组成、多样性和功能特性等最佳的技术手段。核酸的提取及核酸样品的质量将直接关系到深入分析的成败与研究结果的可靠性。“large
‐
scale evidence for microbial response and associated carbon release after permafrost thaw”,primary research articles,2020年12月,yongliang chen等给出了对青藏高原24个样点的多年冻土的活动层(采样深度为1.5~3.5m)进行大规模取样并运用宏基因组技术(功能基因芯片和illuminamiseq测序)来探索多年冻土融化(对多年冻土样品在5℃下培养11天)对微生物分类和功能群落的影响,具体地采用高通量测序来检测细菌和真菌的分类多样性,旨在获取相同时期、活性高的微生物作为研究对象构建微生物分解过程中冻土碳和气候反馈的方向和强度之间的关系。然而,多年冻土层却难以通过高通量测序获取dna序列,主要原因在于:一方面覆盖在活动层以下的更深的多年冻土层样本难以获取,另一方面微生物生物量和核酸量低、难以提取并满足当前高通量测序的基本要求。
6.为此,本发明提出一种青藏高原多年冻土低生物量样本获取和核酸提取技术,以多年冻土层中的微生物作为研究对象,为多年冻土层的微生物生态学的研究提供助力。
技术实现要素:
7.有鉴于此,本发明的目的在于提供一种多年冻土微生物dna样本获取及dna的提取和检测方法,选取多年冻土不同深度的微生物作为研究对象,采用改良的ctab法进行多年冻土层内的微生物进行dna样本的获取,并通过mda扩增的方法对总dna进行提取,采用荧光法进行定量分析,得到足量的dna量并已经满足后续的宏基因组文库构建和测序。
8.为了达到上述目的,本发明提供如下技术方案。
9.本发明提供了一种多年冻土层土壤微生物样本获取及核酸提取方法,其特征在于,包括以下步骤:s1 采样:采用定点、不同深度剖面多层位采样法对青藏高原多年冻土进行无菌采样,采样时保持多年冻土样品始终处于冻结状态和无菌状态;并于-80℃条件下保存;其中,多年冻土包含活动层和多年冻土层的冻土,所述多年冻土样品包括活动层样品和多年冻土层样品;s2微生物样本获取:该步骤选取所述活动层样品和/或所述多年冻土层样品,采用阶段升温法进行解冻获取微生物样本;s3微生物样本前处理:将所述微生物样本分别进行研磨后,加入pbs缓冲液经振荡-液氮冻融处理后得到冻融悬浮液;s4 采用改良的ctab法提取dna样本:所述冻融悬浮液依次经过菌体裂解-抽提-沉淀-洗涤和溶解得到dna样本;s5 电泳检测:对所述pcr产物进行检测。
10.进一步地,s1中,选取9月底~10上旬青藏高原多年冻土活动层处于最大融深期,采用定点、不同深度剖面多层位采样法进行无菌采样,采样时保持多年冻土样品始终处于冻
结状态和无菌状态;将获取的不同深度的所述多年冻土样品分别存储于独立、无菌容器内,并于-80℃条件下保存;进一步地,s1中,所述不同深度剖面多点采样法是依次选取0~20cm、20~350cm、350~500cm、500~600cm、600~1000cm和1000~1500cm不同深度的活动层和多年冻土层进行采样,其中,0~350cm的样品为活动层样品,其它深度的样品为多年冻土层样品,并分别存储于独立、无菌容器内。
11.进一步地,s2中,所述阶段升温法包括将所述多年冻土依次从-80℃取出后,依次经过-50℃放置30min
‑‑
20℃放置30min-4℃放置1h-常温条件下放置1h。
12.进一步地,s4中,改良的ctab法包括以下步骤:(1)菌体裂解:将s3中所述冻融悬浮液进行高速离心后,弃上清液,加入te缓冲液悬浮沉淀,再加入裂解液混匀,再加预热后的ctab/nacl溶液再次混匀,保温得到裂解样品;所述裂解液包括sds、蛋白酶k。
13.(2)抽提:在所述裂解样品中加入等体积的tris饱和酚:氯仿:异戊醇混合液混匀,在离心得到的上清液中加入等体积的氯仿和异戊醇混合液进行抽提;(3)沉淀:在步骤(2)抽提后的上清液中加入异丙醇离心处理;(4)洗涤和溶解:将步骤(3)的离心后的沉淀物用乙醇洗涤,晾干后溶于ddh2o或ph 8.0的te缓冲液中,得到dna样本。
14.进一步地,所述te悬浮沉淀,加入sds、蛋白酶k后混匀并37℃保温1小时;再加入nacl溶液和 65℃预热的ctab/nacl溶液混匀后,65℃保温30分钟;所述tris饱和酚:氯仿:异戊醇混合液的体积比为25:24:1;所述氯仿和异戊醇混合液的体积比为24:1。
15.为实现本发明的另一个目的,本发明还提供了一种多年冻土微生物dna提取方法,将上述技术方案获取的dna样本或采用传统的ctab获取的dna样本进行 mda扩增,可以获得大量的微生物总dna,尤其是多年冻土层的总dna,从而用于后续的宏基因组文库构建和测序。
16.其中,所述mda扩增包括将所述dna样本经纯化后进行样本变性
‑ꢀ
mda扩增-pcr扩增,获得pcr产物。
17.进一步地,所述dna样本使用nucleospin胶/pcr产物纯化试剂盒进行纯化;所述样本变性采用pcr扩增仪进行变性得到变性产物。
18.进一步地,所述mda扩增体系包括dntp、dtt、phi29酶、bsa和超纯水,将所述pcr产物进行mda扩增。所述mda扩增体系中各组分与所述变性产物的体积百分比分别为dntp:dtt:phi29酶:bsa:变性产物=4:0.8:0.8~2:0~0.2:5~10。
19.进一步地,所述16srdna pcr扩增中,正向引物(341-f):5'-cctacgggaggcagcag-3';反向引物(926-r):5'-ccgtcaattcctttragttt-3'。
20.将上述方案得到的dna样本采用荧光法进行定量分析,结果显示采用改良后的ctab可以直接获取到足量的dna样本,或采用传统的ctab法联合mda扩增的方法也能够提取到足量的多年冻土中的dna。在实现了多年冻土的dna的提取后,能够获取到应用于后续检测用的足量的dna样本,并可以利用本领域已知的方法对其进行检测,包括对其进行测序等。
21.本发明所获得的有益技术效果:1.通过采用本发明的技术方案,提取到了青藏高原冻土微生物的dna,尤其是覆盖在活动层以下多年冻土层的低生物量多年冻土微生物的总dna,不仅为青藏高原的微生物细菌、古菌、真菌、蓝细菌、病毒等微生物的基因序列研究提供了技术支持,而且对青藏高原多年冻土内的种群的研究具有积极的指导作用。
22.2.通过采用本发明的技术方案,为揭示青藏高原特有的微生物类群基因组构建了一条可行的方案,为后续的宏基因组文库构建和测序提供了技术支撑,尤其是在多年冻土层逐渐融化之前,对青藏高原冻土层中的古细菌尤其是古病毒的dna具有重要意义。
23.3.针对多年冻土层中未知的特异性微生物提供了获取和提取dna的方法,该方法具有极强的针对性,且方法简单、便于操作,减少冻土微生物运输和保藏过程中带来的污染。
附图说明
24.图1本发明实施例1中dna样本电泳检测的电泳胶图。
25.图2本发明实施例2中dna样本电泳检测的电泳胶图。
26.图3 本发明实施例2中mda扩增后dna电泳检测的电泳胶图。
具体实施方式
27.为使本发明实施例的目的、技术方案和优点更加清楚,下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例是本发明一部分实施例,而不是全部的实施例。
28.青藏高原多年冻土包括活动层和多年冻土层,其中活动层厚度一般为0~350cm,350cm以下为多年冻土层。本发明研究的对象为多年冻土层的土壤微生物,多年冻土层样本为常年处于冰冻状态的样本。
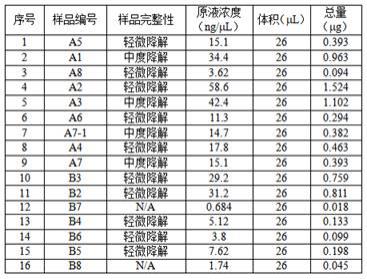
29.采样:选取9月底~10上旬青藏高原多年冻土的活动层处于最大融深期,因此本发明选取该时间段对青藏高原多年冻土的活动层和多年冻土层进行采样。
30.采样方法:采用定点、不同深度剖面多层位采样法进行无菌采样,采样时保持多年冻土样品始终处于冻结状态和无菌状态;将获取的不同深度的所述多年冻土样品分别存储于独立、无菌容器内,-20℃条件下运输保存,而后于-80℃条件下保存。
31.进一步地,定点、不同深度剖面多层位采样法是指对选定某一草甸覆盖下的单点进行同一剖面、不同深度的多点采样,采样深度包括活动层表层(0~20cm)和活动层下层(20~350cm)以及多年冻土层表层(350~500cm)、中层(500~600cm)和下层(600~1500cm)进行采样,获得冻土样品,并进行编号。取样时需遵循样品始终处于冻结和无菌状态,取样工具也要绝对保持无菌状态的基本原则。每个采样深度层,用灭菌后的刀具削去土壤外表面1 cm的部分,将这些样品立即放置于无菌样品袋或铝盒等容器中并密封、编号,迅速保存于-20℃冰柜中并运回保藏试验点,保存于-80℃超低温的冰箱中,以备后续分析。
32.取样时,0-20cm的活动层表层样品直接使用无菌铲或者无菌刀取出并置于无菌样品袋、无菌铝盒等容器中。20-350cm活动层下层样品使用人工探坑法或便携式土壤钻机(钻杆内直径约4.5cm)钻取。350cm以下的多年冻土层样品使用专业机械动力钻机(钻杆内直径
10-20cm)获取。
33.以下实施例和对照例的试验对象包括活动层样品和/或多年冻土层样品。
34.实施例1本实施例选取活动层样品和多年冻土层样品进行dna提取,具体步骤包括:1.样本前处理将活动层样品和多年冻土层样品从-80℃冰箱取出并采用阶段升温法进行解冻,活动层样品和不同剖面深度的多年冻土层样品进行后续处理,首先对所有样本进行研磨,尽可能将块状样本碾碎,并随机选取一份活动层样品和多年冻土层样品进行混合后,将混合后的活动层样品和多年冻土层样本用10xpbs缓冲液进行充分冲洗,涡旋振荡10分钟后,去掉缓冲液里沉积的沙子,保留缓冲液中的土壤悬液并加入pbs缓冲液,经振荡-液氮冻融处理后得到冻融悬浮液,进行后续dna提取。
35.其中,阶段升温法解冻包括将活动层样品和/或多年冻土样品从-80℃取出后,依次经过-50℃放置30min
‑‑
20℃放置30min-4℃放置1h-常温条件下放置1h的分阶段解冻的方法。
36.2.dna提取采用改良的ctab提取法提取基因组dna,具体包括以下步骤:(1)菌体裂解:将获得的冻融悬浮液进行15000r/min高速离心,弃上清,加9.0ml te悬浮液得到沉淀悬浮液,并加1.0ml 10% sds,100μl 20mg/ml(或1mg干粉)蛋白酶k,混匀并37℃保温1小时。然后,加1.5ml 5mol/l nacl并混匀;接着,再加1.5ml 65℃预热过的ctab/nacl溶液再混匀,65℃保温30分钟。该步骤中,sds裂解细菌的细胞壁,蛋白酶k去除蛋白成分,ctab去除多糖成分。
37.(2)抽提:加入等体积的tris饱和酚∶氯仿∶异戊醇(25∶24∶1)并混匀,室温放置5~10分钟;5000rpm离心10分钟,将上清液移至干净离心管。再使用等体积氯仿∶异戊醇(24∶1)抽提,取上清液移至干净管中。
38.(3)沉淀:加等体积的异丙醇并混匀,室温放置10分钟后5000rpm离心10分钟。
39.(4)洗涤:沉淀用75%的乙醇洗涤。
40.(5)溶解:晾干后,溶于ddh2o或ph 8.0 te缓冲液中。
41.3.dna检测采用琼脂糖凝胶对获得的多年冻土微生物dna进行核酸检测,采用dl2000作为marker。
42.具体地,配制1.5%的琼脂糖凝胶,冷却25min,凝固后拔掉胶梳,将凝胶连同胶托一起取出放置在电泳仪中,取扩增结束的pcr产物3μl进行电泳检测,pcr产物混入loading buffer进行点样,同时点样3μl的dl1000 dna marker,200v电泳17min,电泳结束后进行eb(溴化乙锭)染色15 min,然后进行凝胶成像,查看在500片段区域的条带情况。电泳胶图见图1。
43.采用qubit荧光计(赛默飞)对获得的dna进行定量,结果见表1。其中,编号1-5为活动层样品,5以上为多年冻土层样品。
44.表1 样品的dna提取定量分析结果
通过图1和表1所示结果,大部分的样本都能在20k左右的片段区域有明显的条带,表面绝大部分的活动层土壤微生物样本能够提取出一定量的dna。除样品b7外,总dna的量均在0.1mg以上,最高可高达1.5mg,显然,采用本实施例技术方案是能够获取到足量的dna,能用于后续的宏基因组文库构建和测序。总dna获取量的数据也进一步显示了多年冻土中微生物分布的不均一性。由于本实施例的选样为定点采样,可以预测,采用多点采样后混合样品进行dna提取能够获得更高水平的总dna量。
45.同时,通过试验的结果可以推测出,活动层的微生物含量和活性均高于多年冻土层。
46.实施例2本实施例选取多年冻土层样品进行dna提取。具体步骤包括:1.样本前处理将多年冻土层样品(样品编号见表2)从-80℃冰箱取出并采用阶段升温法进行解冻,首先对多年冻土层样本进行研磨,尽可能将块状样本碾碎,不同深度的多年冻土层样本混合均匀后称取20g进行后续处理,然后用10xpbs缓冲液对样本进行充分冲洗,涡旋振荡10分钟后,去掉缓冲液里沉积的沙子,保留缓冲液中的土壤悬液进行后续dna提取。
47.本实施例中的阶段升温法的程序与实施例1相同。
48.2.dna提取采用传统的ctab提取法提取基因组dna,具体包括以下步骤:对获得的土壤悬液进行离心(15000r/min),去除上清,加9.0ml te悬浮液于沉淀中,并加ripa裂解液,包括1ml 1% triton x-100,0.5ml 1% deoxycholate和10ml 0.1% sds,混匀,37℃保温1小时;加1.5ml 5mol/l nacl溶液,混匀;再加1.5ml65℃预热过的 ctab/nacl溶液,混匀,65℃保温20分钟。加入等体积的tris饱和酚∶氯仿∶异戊醇(25∶24∶1),混匀,室温放置5~10分钟,5000rpm离心10分钟,将上清液移至干净离心管。再使用等体积的氯仿∶异戊醇(24∶1)抽提,取上清液移至干净管中。加入等体积的异丙醇,混匀,室温放置10分钟。5000rpm离心10分钟。沉淀用75%的乙醇洗涤。晾干后,溶于ddh2o或ph 8.0 te中。
49.3.dna检测采用琼脂糖凝胶对获得的土壤微生物dna进行核酸检测,采用dl2000作为marker。
具体方法与实施例1相同。获取的电泳胶图参见图2。
50.采用qubit荧光计(赛默飞)对获得的dna进行定量,结果如表2。
51.表2 多年冻土层样品的dna提取定量分析结果如图2所示,图中并没有明显的dna条带,直接进行传统的ctab法对多年冻土层样本中的微生物进行dna提取并不能获得足够量的dna。
52.表2结果显示,基于本方法提取出来的dna量极少,基本低于0.03μg以下。
53.4.mda扩增提取法将步骤2采用传统的ctab提取法提取的dna使用nucleospin 胶/pcr产物纯化试剂盒对提取的dna样本进行纯化。采用mda试剂盒(塞默飞)对纯化后的dna进行扩增,反应体现如下:(1)样本变性:通过pcr扩增仪使样本变性。
54.其中,变性体系(10μl)参阅表3。
55.表3 变性体系的组分和添加比例(体积)pcr程序包括:第一步骤:95℃
ꢀ‑
5 min;第二步骤:95℃
ꢀ‑ꢀ
30 s;第三步骤:75℃
‑ꢀ
30 s;第四步骤:50℃
ꢀ‑ꢀ
30 s;第五步骤:75℃
ꢀ‑ꢀ
20min;第六步骤:4℃
‑ꢀ
∞min。其中,第二步骤-第五步骤执行20次循环操作。
56.(2)mda扩增mda的扩增体系(45μl)参见表4。
57.表4 实施例2中mda扩增体系的组成(45μl)
pcr程序为:30℃24h(3)qubit荧光计对rca后得到的pcr产物进行定量分析(参见表5)。
58.表5qubit荧光计对多年冻土层微生物的mda产物定量分析结果(4)16srdnapcr扩增:取rca后得到的pcr产物1μl稀释10倍作为16srdna扩增的模板。其中,正向引物(341-f):5'-cctacgggaggcagcag-3';反向引物(926-r):5'-ccgtcaattcctttragttt-3'。
59.pcr的扩增体系(25μl)参见表6。
60.表616srdna扩增体系的组成(25μl)pcr扩增程序包括:第一步骤:94℃-4min;第二步骤:94℃-30s;第三步骤:65~56℃-40s;第四步骤:72℃-1min30s;第五步骤:94℃-30s;第六步骤:57℃-40s;第七步骤:72℃-1min30s;第八步骤:72℃-10min;第九步骤:4℃-∞min。其中,第二步骤-第四步骤执行20次循环操作;第五步骤-第七步骤执行10次循环操作。
61.pcr扩增24h后分别采用琼脂糖凝胶电泳(参见图3)。通过图3和表5结果可见,经过一轮mda扩增后,可以获得大量的微生物总dna,能用于后续的宏基因组文库构建和测序。
62.通过上述试验结果可见,基于传统的ctab提取得到的dna样本条件进行mda得到的扩增产物已经能够满足需要,当采用实施例1的改良的ctab的提取后再进行mda扩增也能够
获得更加足量的dna。
63.实施例3本实施例与实施例2的区别在于mda扩增体系的区别。本实施例采用的扩增体系参见表7。本实施例中增加了phi29酶的添加量,经过一轮mda扩增后,可以获得大量的微生物总dna,整体上略优于实施例2,均在1μg以上(参见表8),能用于后续的宏基因组文库构建和测序。
64.表7 实施例3中mda扩增体系的组成(45μl)表8 qubit荧光计对多年冻土层微生物的mda产物定量分析结果实施例4本实施例与实施例2的区别在于mda扩增体系的区别。本实施例采用的扩增体系参见表9。本实施例中增加了phi29酶的添加量,经过一轮mda扩增后,可以获得大量的微生物总dna,经过 qubit荧光计法检测结果显示,整体上略优于实施例2,均在1μg以上(参见表10),能用于后续的宏基因组文库构建和测序。
65.表9 实施例中mda扩增体系的组成(45μl)
表10 qubit荧光计对多年冻土层微生物的mda产物定量分析结果实施例 5本实施例与实施例2相比,增加了phi 29酶的投量,减少了变性产物作为mda扩增模板的投量,可以充分发挥酶的效能,获得更多的mda产物,从而提高了获取的总dna量。
66.表11 实施例5中mda扩增体系的组成(45μl)表12 qubit荧光计对多年冻土层微生物的mda产物定量分析结果
实施例 6本实施例与实施例2的区别在于mda扩增体系的区别。本实施例采用的扩增体系参见表13。本实施例中并没有添加bsa,经过一轮mda扩增后,可以获得一定量的总dna,经过qubit荧光计法检测结果显示,整体上均低于实施例2(参见表14),基本能用于后续的宏基因组文库构建和测序。
67.表13 实施例 6中mda扩增体系的组成(45μl)表14 qubit荧光计对多年冻土层微生物的mda产物定量分析结果
实施例7本实施例与实施例1的区别在于本对照例中对多年冻土样品采取直接解冻的方法,将多年冻土样品从-80℃取出后,于-20℃放置30min后直接常温条件下放置1h进行解冻。
68.采用本实施例的技术方案,获取dna量基本在0.5μg以上,效果低于实施例1采取的升温方案,但是也能够获取到足量的dna。显然,样品解冻的条件对多年冻土层内的微生物从休眠状态激活具有一定的影响。
69.对照例1本对照例与实施例1的区别在于本对照例中对多年冻土样品采取直接解冻的方法,将多年冻土样品从-80℃取出后,常温条件下放置1h进行解冻。
70.采用本对照例的技术方案,提取的dna量远远低于实施例1,基本上低于0.5μg。显然,直接对样品解冻的方法对青藏高原多年冻土内的微生物的提取并不是有利的条件。
71.对照例2本对照例与实施例2的区别在于本对照例中,mda的扩增体系的不同,具体参见表15。
72.表15 对照例3中mda扩增体系的组成(45μl)采用本对照例的技术方案,采用mda扩增后进行dna提取并不能获得足够量的dna,
基本均低于0.5μg。
73.经过上述实施例1和实施例2的结果可见,直接采用传统的ctab方法无法提取到青藏高原多年冻土内的微生物dna,通过改良的ctab方法可以直接获取一定量的dna量。
74.对多年冻土的前处理对dna的提取量具有一定程度的影响,有别于普通土壤的处理方法,本发明的实施例1、实施例7与对照例1的结果显示,通过阶段升温法的升温方案明显优于直接解冻的升温方法。
75.实进一步地,本发明通过mda扩增的方法,对传统的ctab方法进行扩增后能够获取到大量的总dna,通过试验对比并优化试验条件,证明该方案可行,针对多年冻土层的样品进行总dna提取后用于后续的宏基因组文库构建和测序。
76.实施例3-6和对照例2分别给出了mda扩增体系中不同的酶含量对总dna提取量的影响,通过结果显示,当phi29酶添加量低于0.5μl/45μl时,已经无法获取足量的dna。添加量在0.8~2μl/45μl,均能得到足量的dna。可以推测当phi29酶添加量大于2μl也能够达到提取条件,但是综合节约成本角度考虑,添加0.8~2μl/45μl的phi29酶时提取到的dna已经满足后续的宏基因组文库构建和测序。
77.需要说明的是,相比普通土壤,青藏高原的多年冻土中的微生物量和活性都是比较低的,因此,环境污染对多年冻土微生物的干扰是非常显著的。为增加目标dna提取率,同时避免低生物量样本受环境微生物核酸污染,样本前处理和核酸提取应当在设置有生物安全柜的核酸提取室或超净室中进行。且,在进行实施例和对照例的试验时,取样应当在生物安全柜或超净室同时进行空白样本的取样和前处理并作为阴性对照(无模板对照),阴性对照操作属于本领域的常规操作,可根据现有技术中进行操作即可,故不作赘述。同时,多年冻土中不仅微生物量低且具有非均一性,因此应当将解冻后的多年冻土样本充分混匀后,活动层最低称取10g,多年冻土层至少称取20g,相较于普通土壤,多年冻土中的微生物种群具有极大的不同,甚至更多的是未知菌种/群,而且其微生物的活性和微生物量均非常低,常年处于休眠状态,为了实现本发明的技术目的,采用处理普通土壤的常规手段并不能获得高的dna量,因此需要增加样本量、优化样本处理和提取条件,从而获得合适量的基因组dna。
78.采用本发明的技术方案,对500 cm以下的深层的多年冻土层的微生物进行dna提取,不仅为青藏高原的微生物细菌、古菌、真菌、蓝细菌、病毒等微生物的基因序列研究提供了技术支持,而且,对青藏高原多年冻土内的种群的研究具有积极的指导作用,对揭秘多年冻土层的生物多样性的进一步提供技术支持,在极为缺乏对高原多年冻土微生物的了解的情况下,尤其是对古病毒的相关研究具有极大的推动作用。
79.以上仅为本发明的优选实施例而已,其并非因此限制本发明的保护范围,对于本领域的技术人员来说,本发明可以有各种更改和变化。凡在本发明的精神和原则之内,通过常规的替代或者能够实现相同的功能在不脱离本发明的原理和精神的情况下对这些实施例进行变化、修改、替换、整合和参数变更均落入本发明的保护范围内。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。