1.本发明的实施方式涉及利用电子信号测序或鉴定生物聚合物的系统、方法、装置、和组合物。更具体地,本公开包括教导构建系统以基于酶活(包括复制)以电子方式检测生物聚合物的实施方式。本发明中的生物聚合物包括但不限于天然或合成的dna、rna、dna寡核苷酸、蛋白质、肽、多糖等。酶包括但不限于天然的、突变的或合成的dna聚合酶、rna聚合酶、dna解旋酶、dna连接酶、dna外切核酸酶、逆转录酶、rna引物酶、核糖体、蔗糖酶、乳糖酶等。在下文中,主要讨论并使用dna和dna聚合酶以说明本发明概念。
2.发明背景
3.通过酶促合成进行dna测序可以追溯到桑格的链终止法,该方法在靶序列的体外复制期间通过dna聚合酶把双脱氧核苷酸选择性地纳入dna中。
1,2
这种酶促方法已经以高通量或实时方式扩展到下一代测序(ngs)。
3,4
尽管ngs已经将人类基因组测序的成本降低到$1000的范围,但最近的数据显示成本的降低可能已经达到了最低水平(https://www.genome.gov/27565109/the-cost-of-sequencing-a-human-genome)。限制因素之一是ngs依赖于光学信号检测,这需要笨重且昂贵的精密仪器。
4.无标记检测刺激了聚合酶对dna合成的电子读取5,该检测已经被开发为用于基因组测序的平台。6最近的进展表明电子方法可以是手持设备,例如minion测序仪(www.nanoporetech.com),其可测量通过蛋白质纳米孔的离子电流变化进行dna测序,其中dna解旋酶用于控制dna通过纳米孔的易位。7然而,蛋白质纳米孔只能实现低测序准确度(单次读取85%8)。gundlach及其同事已经证明,由耻垢分枝杆菌孔蛋白a(称为mspa)构成的蛋白质纳米孔中的离子电流阻塞是四个核苷酸(四聚体)的集合事件,因此有44(即256)种可能的四聚体产生大量的冗余电流水平。
9,10
由于离子电流受到纳米孔内核苷酸之外的核苷酸的影响,
11
用于测序的原子级薄纳米孔的概念可能无法实现单核苷酸分辨率。
5.collins及其同事报道了一种单壁碳纳米管(swcnt)场效应晶体管(fet)装置,其上系有dna聚合酶i的klenow片段以监测其dna合成。
12,13
在该方法中,当核苷酸被纳入dna链时,记录了低于平均基线电流的δi(t)的短暂偏移。酶纳入不同的核苷酸会导致δi的差异。该方法有可能用于dna测序。碳纳米管是一种仅由锁定在六边形网格中的单层碳原子制成的材料。由于刚性的化学结构,它的感应可能大部分依赖于靠近蛋白质附着位点的带电侧链的静电门控运动。然而,该装置中使用的碳纳米管长度为0.5-1.0μm,
14
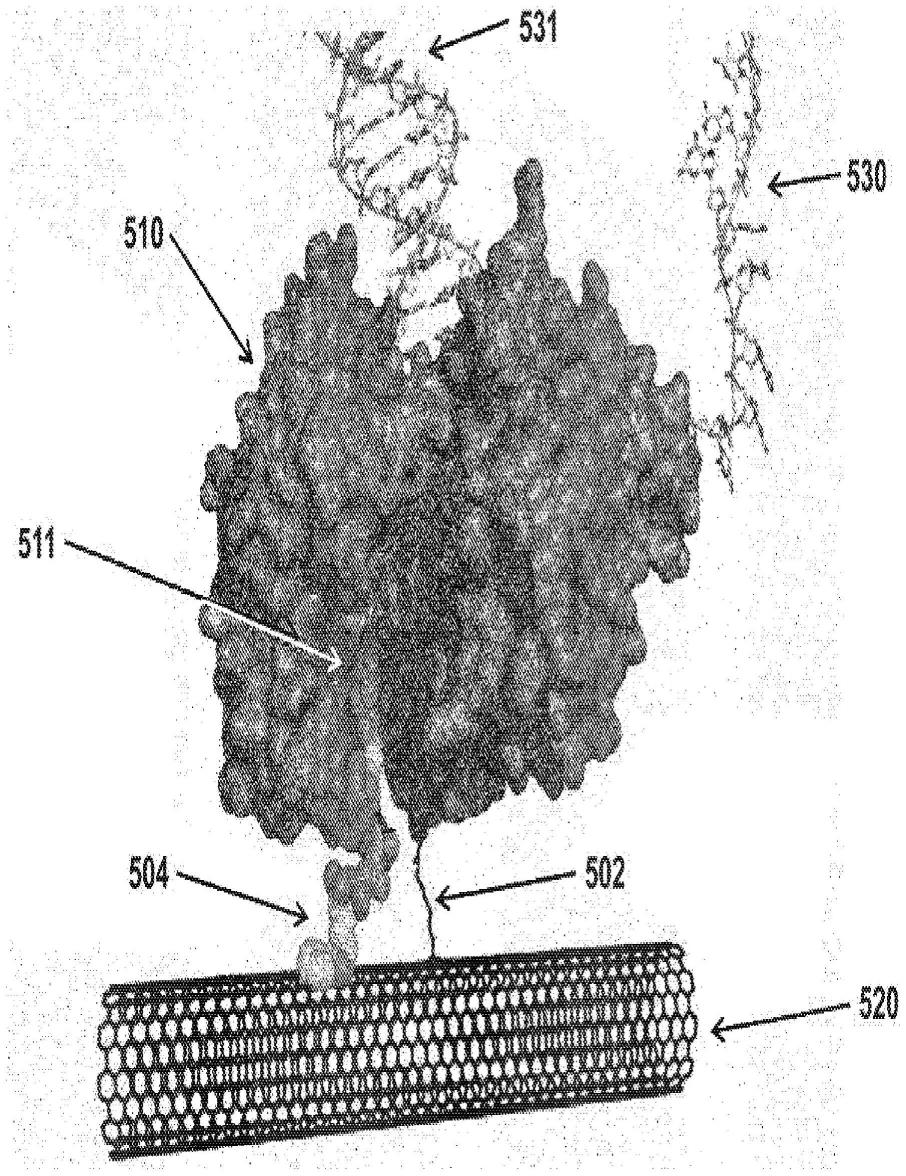
这对在其上可重复地安装单个蛋白质分子提出了挑战。现有技术(wo 2017/024049)揭示了用于dna测序的纳米级场效应晶体管(nanofet),其中dna聚合酶被固定,其核苷酸出口区域朝向碳纳米管栅极,具有一组多磷酸酯标记核苷酸,该标记用于鉴定纳入的核苷酸(图1)。
6.另一项发明(us 2017/0044605)已经揭示了一种使用固定在生物聚合物上的聚合酶测序dna和rna的电子传感器装置,该聚合酶桥接两个分离的电极(图2)。另外,单个酶直接连接正极和负极完成通路,使得所有电流必定流经该分子(us 2018/0305727,wo 2018/208505)。但是,酶可以与电极有两个以上的接触点。
7.dna在分子电子学中引起了极大的关注,因为其独特的碱基堆叠结构使dna成为电
荷转移(ct)的细分子线。此外,dna的序列和长度是可编程的,能够形成无错自组装纳米结构(例如dna折纸),无需昂贵的微细加工技术,这使其成为纳米级集成电路的理想候选。在过去的几十年中,核酸(dna和rna)的程序化自组装已经被开发出来用于纳米结构的构建。
15,16
通常,复杂的dna纳米结构是从分子基序开始组装的,如霍利迪结(holliday junction)、
17,18
多臂结、
19
双交叉(dx)和三交叉(tx)块(tile)、
20,21
平行交叉(paranemic crossover)(px)、
22
张力完整性三角(tensegrity triangle)、
23
六螺旋束、
24
和单链环状dna或dna折纸(图3)。
25
用这些dna基序可以容易地构建各种大小和形状可调的纳米结构。dna纳米结构的刚性比碳纳米管弱,但比dna双链体或分子线更具刚性。它也可以以类似于dna双链体的方式被官能化。它为电子生物传感器的构造提供了独特的试验模型(breadboard),设计结构以具有所需的电导率、电流波动或其他电学性质。据测量,在90%相对湿度下,10
×
60nm
2 tx块纳米结构在45-55nm纳米间隙中具有~70ps的电导。
26
因此,由dna纳米结构桥接的纳米间隙可用来构建用于分子电子检测或鉴定的纳米生物装置。可以设想,dna纳米结构的电导率可以通过其序列、构型和结构动力学进行调整。类似地,rna纳米结构是使用rna基序(图4)通过自组装构建而成。
27,28
rna在结构和功能上比dna更多样化,并且rna双链体比dna的对应物在热力学上更稳定。因此,rna纳米结构可以是对应dna纳米结构的替代。已经证明rna也可以介导电子转移。
29
8.为了编程经典核碱基之外的dna,最近,steven benner和他的同事创建了8核苷酸dna/rna基因系统,称为hachimoji系统。
56
除了四种天然存在的dna核苷酸,a、c、g和t,他们创造了四种另外的非天然dna核苷酸,p、b、s、z,其中p与z配对(p:z),b与s配对(b:s),类似于g:c和a:t配对,其中p和b是嘌呤类似物,z和s是嘧啶类似物,p是2-氨基-8-(1
’‑
β-d-2
’‑
脱氧呋喃核糖基)-咪唑并
‑‑
1,3,5-三嗪-[8h]-4-酮,b是6-氨基-9[(1
’‑
β-d-2
’‑
脱氧呋喃核糖基)-4-羟基-5-(羟基甲基)-氧戊环-2-基]-1h-嘌呤-2-酮,s是3-甲基-6-氨基-5-(1
’‑
β-d-2
’‑
脱氧呋喃核糖基)-嘧啶-2-酮,以及z是6-氨基-3-(1
’‑
β-d-2
’‑
脱氧呋喃核糖基)-5-硝基-1h-吡啶-2-酮(参见图5和参考文献#56)。这些新的核苷酸可以形成与四种天然核苷酸类似的双螺旋,它们还可以与gactzpsb八种碱基混合形成能够被dna聚合酶复制的dna双螺旋。在天然dna中,g:c对有三个氢键,但a:t对只有两个氢键。相比之下,p:z和b:s对都有三个氢键(图5),因此可以预测,由这些非天然碱基形成的dna双链体将会比由天然dna形成的更稳定且导电性更强。对于由非天然核苷酸制成的rna也是如此,其中四种dna碱基pbsz变成rna碱基,p:2-氨基-8-(1
’‑
β-d-呋喃核糖基)-咪唑并
‑‑
1,3,5-三嗪-[8h]-4-酮,b:6-氨基-9[(1
’‑
β-d-呋喃核糖基)-4-羟基-5-(羟基甲基)-氧戊环-2-基]-1h-嘌呤-2-酮,s:2-氨基-1-(1
’‑
β-d-呋喃核糖基)-4(1h)-嘧啶酮,和z:6-氨基-3-(1
’‑
β-d-呋喃核糖基)-5-硝基-1h-吡啶-2-酮,s类似于天然rna中的u(参见参考文献#56)。
[0009]
最近有研究报道了dna聚合酶i在溶液中与px基序结合,其kd约为220nm,与dx基序结合kd约为13μm。
30
尽管px基序不能作为聚合酶延伸的底物起作用。对于dna测序,φ29 dna聚合酶是用于多种平台的酶。
9,31,32
基于氨基酸序列的相似性和其对特定抑制剂的敏感性,φ29 dna聚合酶属于真核细胞型dna依赖性dna聚合酶家族b。
33
如同其他dna聚合酶,它在延长的dna链的3
′‑
oh基团上循序完成模板导向的dnmp单元的添加,对于不匹配的dnmp插入显示出104至106倍的辨别力。
34
此外,φ29 dna聚合酶催化3
′‑5′
核酸外切(exonucleolysis),即从dna链的3
′
末端释放dnmp单元,优先降解错配的引物-末端,这进一步增强了复制的保
真度。
35-37
φ29 dna聚合酶的校正活性、链置换和合成能力可能归因于其独特的结构(图6)。
38-40
[0010]
附图简要说明
[0011]
图1:现有技术纳米级场效应晶体管(nanofet)和用于dna测序的示例性的一组携带可区分的带电的导电标记的核苷酸类似物。
[0012]
图2:使用生物聚合物将dna聚合酶与电极连接的现有技术。
[0013]
图3:用于构建dna纳米结构的示例性dna基序。
[0014]
图4:用于构建rna纳米结构的示例性rna基序。
[0015]
图5:hachimoji氢键合碱基对的dft模型和计算homo和lumo能。
[0016]
图6:φ29 dna聚合酶的结构域组织的带状示意图。
[0017]
图7:用于构建基于核酸的分子线的人工核碱基结构。
[0018]
图8:单分子dna测序装置的示意图。
[0019]
图9:随着dna聚合酶构象变化,核苷酸结合和纳入的动力学机制。
[0020]
图10:制造在纳米间隙区域中的具有暴露氧化硅表面、钝化基材和钝化纳米线的纳米间隙的过程图示。
[0021]
图11:用在dna纳米结构末端用于附接金属电极的5
’‑
巯基-核苷的化学结构。
[0022]
图12:碱基硫族化(chalcogenated)核苷的化学结构。
[0023]
图13:(a)含有羧基官能团的三脚架,作为将dna纳米结构附接到金属电极的锚;(b)在各自核碱基上含有氨基官能团的核苷的化学结构。
[0024]
图14:核碱基硫族化核苷的化学结构。
[0025]
图15:核碱基硫族化核苷的化学结构。
[0026]
图16:使用n-杂环碳烯对纳米间隙的电极(阴极)进行电化学官能化。
[0027]
图17:将dna块固定在纳米间隙中的链霉亲和素上以使其附接电极的示意图。
[0028]
图18:(a)含有两个生物素和两个毒鼠硅(silatrane)官能团的四臂接头的化学结构;(b)分子力学计算得出的其3d结构。
[0029]
图19:生物素化核苷的化学结构。
[0030]
图20:在277和479位置含有对-叠氮苯丙氨酸且两个末端有两个标签的φ29 dna聚合酶的突变体,以及在277和479位置含有对-叠氮苯丙氨酸的突变体。原始结构来自蛋白质数据库(protein data bank)(pdb id:1xhx)。
38
[0031]
图21:将肽附接到φ29 dna聚合酶末端的过程。
[0032]
图22:φ29 dna聚合酶与引物-模板dna和进入的核苷酸底物复合的晶体结构(pdb id:2pyl)。
[0033]
图23:含有乙炔的核苷的化学结构。
[0034]
图24:带dna嵌入剂标签的核苷六磷酸酯的化学结构。
[0035]
图25:用于直接rna测序的单分子装置的示意图。
[0036]
发明概述
[0037]
本发明提供了用于生物聚合物测序或鉴定的纳米结构装置和方法。本公开在各种实施方式的通篇描述中使用单个dna分子的测序说明本发明。本发明还提供了不同实施方式中各种代表性装置、设备和方法的具体技术细节,仅用于说明目的,不意在限制物理尺寸
和排列、化学成分和结构,加工程序和参数,或任何其他适用条件,不以任何方式限制适用范围。
[0038]
在一个实施方式中,如图8所示,通过半导体纳米制造(nanofabrication)技术在两个电极之间制造了10-20nm的纳米间隙,其周围用惰性化学品钝化以防止非特异性吸附,纳米间隙的内部区域暴露用于化学反应。dna块结构被锚定到电极上以桥接纳米间隙,dna聚合酶(如φ29 dna聚合酶)固定于其上。为了测序,目标dna(模板)在该装置中复制。在复制过程中,核苷酸被dna聚合酶纳入正在延长的dna链上。在机制上,核苷酸纳入伴随着聚合酶构象改变(图9)。
41
由于聚合酶是直接附接dna块,构象变化会破坏块的结构,导致电流的波动,电流的波动被用作鉴定不同核苷酸纳入的标志。
[0039]
在一些实施方式中,本发明提供了在两个电极之间制造纳米间隙的方法,间隙大小范围从3nm到1000nm,优选从5nm到100nm,更优选从10nm到30nm。首先,本发明使用电子束光刻(ebl)生成金属纳米线,例如半导体基材上方的au(金)、pd(钯)和pt(铂)纳米线。例如,如图10所示,在氧化硅(sio2)基材(1)或用氮化硅(si3n4)层涂覆的硅基材上,通过ebl制造尺寸为1000x10x10nm(长x宽x高)的金纳米线(3)并通过标准光刻技术连接到大金属接触垫(2)上。纳米线的长度为100nm至100μm,优选1μm至10μm;宽度为5nm至100nm,优选10nm至30nm;并且高度(厚度)为3nm至100nm,优选为5nm至20nm。纳米线阵列也可以通过纳米压印
42
或其他纳米制造技术制造。随后,通过与11-巯基十一烷-六甘醇(cr-1)
43
反应形成单层钝化金属表面,氧化硅表面首先用氨基丙基三乙氧基硅烷(cr-2)处理,然后与n-羟基琥珀酰亚胺2-(ω-o-甲氧基-六甘醇)乙酸酯(cr-3)反应。最后,通过ebl或通过氦气聚焦离子束研磨(he-fib)
44
切割钝化的纳米线以产生10-20nm的纳米间隙,并在切割区域暴露出氧化硅和电极的侧壁。或者,一个或多个纳米线可以被薄绝缘层覆盖而不是钝化,或先被薄绝缘层覆盖然后钝化。
[0040]
在一些实施方式中,dna纳米结构被用以桥接纳米间隙。如图8所示,10nm的纳米间隙由四链dna块桥接。
45
有许多方法可以在溶液中通过自组装形成不同形状和大小的dna纳米结构。
46-48
[0041]
在一个实施方式中,非天然dna碱基(pbsz)用于构建桥接纳米间隙的纳米结构。众所周知,有g/c碱基的双螺旋dna是比仅含有a和t核苷酸的双螺旋dna更好的导体。鸟嘌呤碱基的易氧化性使得产生电荷载流(空穴)成为可能。据信通过dna的电荷传输主要是通过碱基最高占据分子轨道(homo)的空穴传输,因为这些轨道比碱基最低未占分子轨道(lumo)更接近电极费米能级。
57
如图5所示,非自然碱基对z:p的homo能量高于a:t碱基对之一,而碱基对s:b的homo能量高于g:c碱基对之一。因此,由这些非天然碱基对构成的dna分子比由天然碱基对构成的dna分子具有更高的电导率。
[0042]
在一个实施方式中,非天然dna碱基(pbsz)用于构建桥接纳米间隙的导电线性分子线。线性分子线是由简单双螺旋dna双链体(双链dna)构成。线性分子线可包含用于附接或连接聚合酶或其他酶的一种或多种修饰的核苷酸。使用非天然dna碱基构建分子线的一个益处是其潜在的较高电导率。
[0043]
在一个实施方式中,非天然dna碱基(pbsz)用于构建更复杂的导电分子纳米结构,二维的或三维的、不可分离的单一结构或可分离的多结构复合体。
[0044]
在另一个实施方式中,非天然dna碱基(pbsz)与天然碱基(acgt)混合以构建桥接
纳米间隙的简单线性导线分子线或更复杂的导电分子纳米结构,二维的或三维的、不可分离的单一结构或可分离的多结构复合体。例如,可以使用天然c:g对加非天然s:b对以构建dna纳米结构,以利用它们的高homo能量特性;此外,可以包括天然a:t对以形成六核苷酸dna纳米结构,以利用其较弱的碱基配对能量状态来诱导dna纳米结构中有利的结构变化。另一个实例是形成八个核苷酸的dna纳米结构,其更复杂,意味着更可调节或更高的概率实现高精度测序。
[0045]
在一些实施方式中,本发明提供了用于形成基于核酸的分子线(不一定呈螺旋形式)的非天然尺寸扩大的核碱基
58
(图7)。与天然存在的核碱基相比,这些尺寸扩大的碱基具有更大的π共轭,提供更好的核碱基堆叠,从而更高效地传输电荷。
[0046]
在一些实施方式中,本发明提供非氢键合核碱基作为基于核酸的分子线的部分(图7)。这些核碱基对其周围的改变更敏感,这使得分子线对生物传感和化学传感更敏感。
[0047]
在一个实施方式中,本发明使用了芘作为通用碱基(py,图7),其可以不加选择地与那些核碱基中的任一个碱基配对。由于其大的π共轭,它可以被插入到分子线中,以取代氢键合核碱基用以增加电导率。
[0048]
在一个实施方式中,未配对的或非配对的一种或多种核酸碱基可以插入到dna纳米结构中以有意地引起结构不连续,从而实现对生物聚合物的测序或鉴定有利的结构变化。
[0049]
本发明还提供了将所述dna纳米结构附接到电极上的方法。在一个实施方式中,dna纳米结构在其5’末端具有5
’‑
巯基核苷,在其3’末端具有3
’‑
巯基核苷,如图11所示。核苷是脱氧核糖核苷(r=h)和核糖核苷(r=oh)。此外,硫原子可被硒取代,硒可能是用于电子传输的更好的锚。
49
[0050]
在另一个实施方式中,本发明提供了在末端用rxh和rxxr官能化dna纳米结构的方法,其中r是脂肪族或芳香族基团;x是硫族元素优选s和se。
[0051]
在一些实施方式中,本发明提供碱基硫族化核苷,其可被纳入dna纳米结构用于附接电极(图12)。已经证明通过核碱基连接电极dna与电极比通过糖部分提供了更高效的电接触。
[0052]
在一个实施方式中,本发明提供了负载四苯基甲烷的三脚架锚,以硫(s)或硒(se)作为附接到金属电极的锚定原子,以羧基用于附接dna纳米结构(图13,a)。同时,用氨基官能化核苷在末端修饰dna纳米结构(图13,b),用于附接三脚架。
[0053]
本发明还提供了另一种用叠氮化物官能化的三脚架(图14,a),其允许通过叠氮化物-炔烃点击反应将dna纳米结构附接到金属电极上。因此,本发明提供了用环辛炔官能化的核苷(图14,b)用于修饰dna纳米结构末端。
[0054]
本发明还提供了用硼酸官能化的三脚架(图15,a)和用二醇官能化的核苷(图15,b)用于修饰dna纳米结构末端。因此,如先前公开(美国临时专利62/772,837)所公开的通过硼酸和二醇的反应将dna纳米结构附接到金属电极。
[0055]
在一个实施方式中,本发明提供了在纳米间隙中用n-杂环碳烯(nhc)选择性地官能化两个电极之一的方法。如图16所示,5-羧基-1,3-二异丙基-1h-苯并[d]咪唑-2-碳烯通过其金络合物在溶液中的电化学还原沉积到金电极上。
51
nhc的羧基通过将其转化为活化的酯用作锚定点。因此,dna纳米结构(具有分别由胺和硫醇官能化的末端)通过其胺官能化末
端与nhc电极反应,其硫醇官能化末端与裸露的金电极直接反应,桥接纳米间隙。
[0056]
本发明提供了防止纳米结构接触纳米间隙底部的方法。如图17所示,单个链霉亲和素分子通过生物素化的四臂接头固定在纳米间隙中,使得生物素化dna块可以连接到链霉亲和素上,然后通过上述方法之一附接到电极上。本发明还提供了用于链霉亲和素固定的四臂接头,其中两臂由生物素官能化,其他两臂由毒鼠硅官能化(图18,a)。通过分子力学计算,四臂接头似乎是四面体几何形状(图18,b)。两个生物素部分与链霉亲和素相互作用形成二价复合物。对于链霉亲和素的固定,毒鼠硅部分首先与氧化硅反应,允许四臂接头固定在表面上,然后将链霉亲和素添加到表面。
[0057]
在另一个实施方式中,本发明提供了生物素化核苷,其可以通过亚磷酰胺化学纳入dna以构建dna纳米结构(图19)。
[0058]
在一些实施方式中,本发明提供将dna聚合酶附接到dna纳米结构的方法。本发明使用多重定点诱变方法
52
和遗传密码扩展技术
53
,以在多重特定位点用非天然氨基酸(uaa)取代dna聚合酶的标准氨基酸。如图20所示,φ29 dna聚合酶突变体表达为对-叠氮苯丙氨酸取代w277(10)和k479(11)。uaa对-叠氮苯丙氨酸用于通过点击化学作为固定聚合酶的锚定点,已经进化出促进其纳入的aars。
53,54
φ29 dna聚合酶突变体进一步表达为在n末端具有肽序列mlvprg(12)和c末端具有lpxtg-his6(13)。通过这种方式,可以在酶的两个末端用肽修饰。图21显示了用分选酶a将肽附接到酶上的过程。
55
通过观察φ29 dna聚合酶与引物-模板dna以及进入的核苷酸底物复合的结构,技术人员可以看到蛋白质的c端(14)离dna非常近(图22),表明蛋白质中dna的任何运动都可能对dna纳米结构造成多米诺效应,导致电流的波动,电流的波动可被用作dna核苷酸纳入事件的标志。因此,微调dna纳米结构可以实现单碱基分辨率。
[0059]
在一个实施方式中,本发明提供包含乙炔的核苷,其可以被纳入dna中以构建dna纳米结构,用于在铜催化剂存在的情况下通过点击反应附接dna聚合酶(图23)。
[0060]
在一个实施方式中,本发明提供了用与dna纳米结构相互作用的带不同dna嵌入剂标签的修饰核苷酸(dn6p)(图24)。这些修饰核苷酸被用作dna聚合酶的底物以将dna核苷酸纳入dna。首先,dna聚合酶与目标dna模板和核苷多磷酸酯形成复合物,这也稳定了嵌入剂标签与dna纳米结构的相互作用。当核苷酸被纳入目标dna时,它释放带嵌入剂标签的五磷酸酯。因为静电排斥使嵌入剂与dna的相互作用去稳定,导致带标签的五磷酸酯释放到溶液中。这种过程会改变dna纳米结构的电导。由于每个dn6p携带不同的嵌入剂,不同核苷酸的纳入会导致不同的电流波动,这可用于鉴定被纳入dna的核苷酸。
[0061]
以上公开的实施方式中的大多数方法应用于rna测序。在一个实施方式中,重新工程化的莫洛尼鼠白血病病毒逆转录酶(m-mlv rt)被固定在dna块上用于rna逆转录,如图25所示。当带聚(dt)引物的靶标rna被引入装置时,dna核苷酸被纳入聚(dt)引物。在此过程中,每次纳入造成聚合酶的构象改变,导致电流的波动。随着纳入的持续,记录一串电信号,用分析程序从中推断出rna序列。
[0062]
一般性说明:
[0063]
本文中述及的所有出版物、专利申请、专利和其他文件都通过引用全文纳入本文。
[0064]
除非另外定义,否则,本文中所使用的所有技术和科学术语都具有本发明所属领域普通技术人员通常所理解的同样含义。虽然通过本发明实施方式介绍本发明,并且相当
with a mutant mspa nanopore and phi29 dna polymerase.)nat.biotechnol.2012,30,349-353.
[0076]
10.laszlo,a.h.;derrington,i.m.;ross,b.c.;brinkerhoff,h.;adey,a.;nova,i.c.;craig,j.m.;langford,k.w.;samson,j.m.;daza,r.;doering,k.;shendure,j.;gundlach,j.h.,对天然dna的长纳米孔测序读取进行解码(decoding long nanopore sequencing reads of natural dna.)nat.biotechnol.2014,32,829-833.
[0077]
11.lindsay,s.,固态测序的前景和挑战(the promises and challenges of solid-state sequencing.)nat nanotechnol 2016,11(2),109-11.
[0078]
12.olsen,t.j.;choi,y.;sims,p.c.;gul,o.t.;corso,b.l.;dong,c.;btown,w.a.;collins,p.g.;weiss,g.a.,dna聚合酶i(klenow片段)的单分子加工的电子测量(electronic measurements of single-molecule processing by dna polymerase i(klenow fragment).)j.am.chem.soc.2013,135,7855-60.
[0079]
13.pugliese,k.m.;gui,o.t.;choi,y.;olsen,t.j.;sims,p.c.;collins,p.g.;weiss,g.a.,单分子dna聚合酶i(klenow片段)纳米电路对三磷酸脱氧核苷类似物的渐进式纳入(processive incorporation of deoxynucleoside triphosphate analogs by single-molecule dna polymerase i(klenow fragment)nanocircuits.)j.am.chem.soc.2015,137,9587-94.
[0080]
14.choi,y.;olsen,t.j.;sims,p.c.;moody,i.s.;corso,b.l.;dang,m.n.;weiss,g.a.;collins,p.g.,用蛋白质工程剖析碳纳米管电路中的单分子信号转导(dissecting single-molecule signal transduction in carbon nanotube circuits with protein engineering.)nano lett.2013,13,625-631.
[0081]
15.xavier,p.l.;chandrasekaran,a.r.,基于dna的纳米级构建:新兴趋势和应用(dna-based construction at the nanoscale:emerging trends and applications.)nanotechnology 2018,29,062001.
[0082]
16.ge,z.;gu,h.;li,q.;fan,c.,框架核酸的概念和发展(concept and development of framework nucleic acids.)j.am.chem.soc.2018,140,17808-17819.
[0083]
17.sha,r.;liu,f.;seeman,n.c.,原子力显微镜测量对称霍利迪结的域间角(atomic force microscopic measurement of the interdomain angle in symmetric holliday junctions.)biochemistry 2002,41,5950-5955.
[0084]
18.mao,c.;sun,w.;seeman,n.c.,用原子力显微镜观察到的设计的二维dna霍利迪结阵列(designed two-dimensional dna holliday junction arraysvisualizedby atomic force microscopy.)j.am.chem.soc.1999,121,5437-5443.
[0085]
19.wang,x.;seeman,n.c.,8臂和12臂dna支链结的组装和表征(assembly and characterization of 8-arm and 12-arm dna branched junctions.)j.am.chem.soc.2007,129,8169-8176.
[0086]
20.fu,t.-j.;seeman,n.c.,dna双交叉分子(dna double-crossover molecules.)biochemistry&analytical biochemistry 1993,32,3211-3220.
[0087]
21.labean,t.h.;yan,h.;kopatsch,j.;liu,f.;winfree,e.;reif,j.h.;seeman,n.c.,dna三交叉复合物的构建、分析、连接和自组装(construction,analysis,ligation,
and self-assembly of dnatriple crossover complexes.)j.am.chem.soc.2000,122,1848-1860.
[0088]
22.wang,x.;chandrasekaran,a.r.;shen,z.;ohayon,y.p.;wang,t.;kizer,m.e.;sha,r.;mao,c.;yan,h.;zhang,x.;liao,s.;ding,b.;chakraborty,b.;jonoska,n.;niu,d.;gu,h.;chao,j.;gao,x.;li,y.;ciengshin,t.;seeman,n.c.,平行交叉的dna:在那里,又回来了。(paranemic crossover dna:there and back again.)chem.rev.2018,119,6273-6289.
[0089]
23.liu,d.;wang,m.;deng,z.;walulu,r.;mao,c.,张力完整性:构建具有柔性四臂dna结的刚性dna三角(tensegrity:construction ofrigid dna triangles with flexible four-arm dna junctions.)j.am.chem.soc.2004,126,2324-2325.
[0090]
24.mathieu,f.;liao,s.;kopatsch,j.;wang,t.;mao,c.;seeman,n.c.,从dna中设计的六螺旋束(six-helix bundles designedfrom dna.)nanolett.2005,5,661-665.
[0091]
25.rothemund,p.w.,折叠dna以创建纳米级的形状和图案(folding dna to create nanoscale shapes and pattems.)nature 2006,440,297-302.
[0092]
26.linko,v.;leppiniemi,j.;paasonen,s.t.;hytonen,v.p.;toppari,j.j.,用于分子电子学的确定大小的dna三交叉构建体:修饰、定位和电导特性(defined-size dna triple crossover construct for molecular electronics:modification,positioning and conductance properties.)nanotechnology 2011,22,275610.
[0093]
27.grabow,w.w.;jaeger,l.,rna自组装和rna纳米技术(rna self-assembly and rna nanotechnology.)acc.chem.res.2014,47,1871-80.
[0094]
28.li,m.;zheng,m.;wu,s.;tian,c.;liu,d.;weizmann,y.;jiang,w.;wang,g.;mao,c.,通过单链rna的程序化折叠在体内生产rna纳米结构(in vivo production of rna nanostructures via programmed folding of single-stranded rnas.)nat.commun.2018,9,2196.
[0095]
29.maie,k.;miyagi,k.;takada,t.;nakamura,m.;yamana,k.,rna介导的电子转移:双指数距离的依赖性(rna-mediated electron transfer:double exponential distance dependence.)j.am.chem.soc..2009,131,13188-13189.
[0096]
30.gao,x.;gethers,m-;han,s.p;goddard,w.a.;sha,r.;cunningham,r.;seeman,n.c.,dna的px基序与大肠杆菌dna聚合酶i的特异性结合(the px-motif of dna binds specifically to e.coli dna polymerase i.)biochemistry 2019,58,575-581
[0097]
31.korlach,j.;marks,p.j.;cicero,r.l.;gray,j.j.;murphy,d.l.;roitman,d.b.;pham,t.t.;otto,g.a.;foquet,m.;turner,s.w.,选择性的铝钝化,在零模式波导纳米结构中定向固定单个dna聚合酶分子(selective aluminum passivationfor targeted immobilization of single dna polymerase molecules in zero-mode waveguide nanostructures.)proc.nati.acad.sci.usa 2008,105,1176-81.
[0098]
32.stranges,p.b.;palla,m.;kalachikov,s.;nivala,j.;dorwart,m.;trans,a.;kumar,s.;porel,m.;chien,m.;tao,c.;morozova,i.;li,z.;shi,s.;aberra,a.;arnold,c.;yang,a.;aguirre,a.;harada,e.t.;korenblum,d.;pollard,j.;bhat,a.;gremyachinskiy,d.;bibillo,a.;chen,r.;davis,r.;russo,j.j.;fuller,c.w.;roever,
s.;ju,j.;church,g.m.,用于在电极阵列上合成单分子dna测序的纳米孔耦合聚合酶的设计和表征(design and characterization of a nanopore-coupled polymerase for single-molecule dna sequencing by synthesis on an electrode array.)proc.nati.acad.sci.usa 2016,113,e6749-e6756.
[0099]
33.bemad,a.;zabalios,a.;salas,m.;blanco,l.,原核生物和真核生物dna聚合酶的结构和功能关系(stmctural and functional relationships between prokaryotic and eukaryotic dna polymerases.)the embo journal 1987,6,4219-4225.
[0100]
34.esteban,j.a.;salas,m.;blanco,l.,phi29 dna聚合酶的保真度(fidelity of phi29 dna polymerase.)j.bio.chem.1993,268,2713-2726.
[0101]
35.blanco,l.;salas,m.,噬菌体phi29编码的dna聚合酶中3
′‑5′
外切酶活性的表征(characterization of a 3
′‑5′
exonuclease activityin the phage phi29-encoded dna polymerase.)nucleic acids research 1985,13,1239-1249.
[0102]
36.garmendia,c.;bernad,a.;estebane,j.a.;blanco,l.;salad,m.,噬菌体phi29 dna聚合酶,一种校对酶(the bacteriophage phi29 dna polymerase,a proofreading enzyme.)j.bio.chem.1992,267,2594-2599.
[0103]
37.esteban,j.a.;soengas,m.s.;salasn,m.;blanco,l.,429dna聚合酶的3
′
至5
′
外切酶活性位点(3’to 5’exonuclease active site of429 dna polymerase.)j.bio.chem.1994,269,31946-31954.
[0104]
38.kamtekar,s.;berman,a.j.;wang,j.;lazaro,j.m.;de vega,m.;blanco,l.;salas,m.;steitz,t.a.,从噬菌体phi29的蛋白质-引物dna聚合酶的晶体结构中洞察到链的置换和合成(insights into strand displacement and processivity from the crystal structure of the protein-primed dna polymerase ofbacteriophage phi29.)molecular cell 2004,16,609-18.
[0105]
39.berman,a.j.;kamtekar,s.;goodman,j.l.;lazaro,j.m.;vega,m.d.;blanco,l.;salas,m.;steitz,t.a.,phi29 dna聚合酶与底物复合的结构:b族聚合酶的转位机制(structures of phi29 dna polymerase complexed with substrate:the mechanism of translocation in b-family polymerases.)theembo journal 2007,26,3494-3505|.
[0106]
40.rodr1
′
guez,i.;zaro,j.m.l.;blanco,l.;kamtekar,s.;berman,a.j.;wang,j.;steitz,t.a.;salas,m.;vega,m.d.,φ29 dna聚合酶中的一个特定亚结构域同时赋予了持续合成能力和链置换能力(a specific subdomain inφ29 dna polymerase confers both processivityand strand-displacement capacity.)proc.nati.acad.sci.usa 2005,102,6407-6412.
[0107]
41.raper,a.t.;reed,a.j.;suo,z.,dna聚合酶的动力学机制:构型动力学和第三种二价金属离子的贡献(kinetic mechanism ofdna polymerases:contributions of conformational dynamics and a third divalent metal ion.)chem.rev.2018,118,6000-6025.
[0108]
42.pi,s.;lin,p.;xia,q.,小于10纳米金属纳米线阵列的制造与小于1纳米的关键尺寸控制(fabrication of sub-10 nm metal nanowire arrays with sub-1 nm critical dimension control.)nanotechnology 2016,27,464004.
[0109]
43.senapati,s.;biswas,s.;manna,s.;ros,r.;lindsay,s.;zhang,p.,利用afm和spr探测蛋白质受体-配体之间二价相互作用的y形三臂结构(ay-shaped three-arm structure for probing bivalent interactions between protein receptor-ligand using afm and spr.)langmuir 2018,34,6930-6940.
[0110]
44.scholder,o.;jefimovs,k.;shorubalko,i.;hafner,c.;sennhauser,u.;bona,g.l.,氦气聚焦离子束制造的具有小于5纳米间隙的等离子体天线(helium focused ion beam fabricated plasmonic antennas with sub-5nm gaps.)nanotechnology 2013,24,395301.
[0111]
45.evans,c.g.;winfree,e.,dna块自组装的物理原理(physical principles for dna tile self-assembly.)chem.soc.rev.2017,46,3808-3829.
[0112]
46.endo,m.;sugiyama,h.,dna纳米技术的化学方法(chemical approaches to dna nanotechnology.)chembiochem 2009,10,2420-43.
[0113]
47.hong,f.;jiang,s.;lan,x.;narayanan,r.p.;sulc,p.;zhang,f.;liu,y.;yan,h.,用于2d和3d dna晶体工程的具有精确可调角度的分层交叉块(layered-crossover tiles with precisely tunable angles for 2d and 3ddna crystal engineering.)j.am.chem.soc.2018,140,14670-14676.
[0114]
48.wang,w.;lin,t.;zhang,s.;bai,t.;mi,y.;wei,b.,从双交叉块中自组装出完全可寻址的dna纳米结构(self-assembly of fully addressable dna nanostructures from double crossover tiles.)nucleic acids res2016,44,7989-96.
[0115]
49.kaull,r.p.;sawhne,r.s.;kaul,r.;engles#,d.,基于硒醇终止的锚的单分子结-有前景的电子传输候选者(single-molecule junctions based on selenol terminated anchor-a promising candidate for electron nansport.)proceedings of the international conference on advanced nanomaterials&emerging engineeringtechnologies 2013,icanmeet-20j3,431-435.
[0116]
50.xiang,l.;palma,j.l.;bruot,c.;mujica,v.;ratner,m.a.;tao,n.,dna电荷传输中的中间隧穿-跳跃方案(intermediate tunnelling-hopping regime in dna charge transport.)nat.chem.2015,7,221-6.
[0117]
51.doud,e.a.;inkpen,m.s.;lovat,g.;montes,e.;paley,d.w.;steigerwald,m.l.;vazquez,h.;venkataraman,l.;roy,x.,n-杂环碳烯结合单分子结的原位形成(in situformation of n-heterocyclic carbene-bound single-molecule junctions.)j.am.chem.soc.2018,140,8944-8949.
[0118]
52.liang,x.;peng,l.;li,k.;peterson,t.;katzen,f.,一种基于同源重组的多位点定向诱变的方法(a method for multi-site-directed mutagenesis based on homologous recombination.)anal.biochem.2012,427,99-101.
[0119]
53.chatterjee,a.;sun,s.b.;furman,j.l.;xiao,h.;schultz,p.g.,一个在大肠杆菌中进行单一和多重非天然氨基酸诱变的多功能平台(aversatile platform for single-and multiple-unnatural amino acid mutagenesis in escherichia coli.)biochemistry 2013,52,1828-37.
[0120]
54.chin,j.w.;santoro,s.w.;martin,a.b.;king,d.s.;wang,l.;schultz,p.g.,
在大肠杆菌的遗传密码中加入对-叠氮-l-苯丙氨酸(addition of p-azido-l-phenylalanine to the genetic code of escherichia coli.)j.am.chem.soc.2002,124,9026-9027.
[0121]
55.antos,j.m.;chew,g.-l.;guimaraes,c.p.;yoder,n.c.;grotenbreg,g.m.;popp,m.w.-l.;ploegh,h.l.,使用不同特异性的分选酶对单一多肽进行位点特异性n-和c-末端标记(site-specific n-and c-terminal labeling of a single polypeptide using sortases of different specificity.)j.am.chem.soc.2009,131,10800-10801.
[0122]
55.ishiguro,t.,yamaji,k.和saito,g.,《有机超导体》(organic superconductors),斯普林格固态科学系列,第二版,issn 0171-1873,1998.
[0123]
56.hoshika,s.;leal,n.a.;kim,m.-j.;kim,m.-s.;karalkar,n.b.;kim,h.-j.;bates,a.m.;jr.,n.e.w.;santalucia,h.a.;meyer,a.j.;dasgupta,s.;piccirilli,j.a.;ellington,a.d.;jr.,j.s.;georgiadis,m.m.;benner,s.a.,hachimoji dna和rna:一个有八个构件的遗传系统(hachimoji dna and rna:a genetic system with eight building blocks.)science 2019,363,884-887
[0124]
57.xiang,l.;palma,j.l.;bruot,c.;mujica,v.;ramer,m.a.;tao,n.,dna电荷传输中的中间隧道-跳跃方案(intermediatetunnelling-hopping regime in dna charge transport.)nat chem 2015,7,221-6.
[0125]
58.winnacker,m.;kool,e.t.,由尺寸扩大的碱基对组成的人工遗传组(artificial genetic sets composed of size-expanded base pairs.)angew chem int ed engl 2013,52,12498-508.
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。
