1.本公开总体涉及用于药物再利用的系统和方法,更具体地,一些实施例涉及大规模并行图数据库,其处理以知识图形式表示的多模态数据并且加速领域特定函数以进行蛋白质相似性分析,以生成药物假设。
附图说明
2.根据一个或多个各个实施例,参考以下附图对本公开进行详细描述。提供这些附图仅用于说明目的并且仅描绘典型实施例或示例实施例。
3.图1图示了根据各个实施例的示例图引擎。
4.图2图示了根据各个实施例的图引擎查询执行的示例。
5.图3图示了根据各个实施例的用于执行蛋白质序列分析的图引擎(诸如cge)的示例实施方式。
6.图4图示了可以用于执行蛋白质序列分析的样本查询的示例。
7.图5图示了根据各个实施例的可以用于实现蛋白质序列分析的示例计算部件500。
8.图6是可以用于实现本公开中所描述的实施例的各个特征的示例计算部件。
9.附图并非详尽的,并且不将本公开限制为所公开的精确形式。
具体实施方式
10.在新型冠状病毒所引起的流行中,药物再利用(调查用于新治疗目的的现有药物)成为发现医疗护理的第一缕希望。然而,药物再利用不仅限于冠状病毒,而且还可以用于标识针对多种不同病症中的任一病症的治疗剂/药物。药物再利用管道牵涉到了解致病生物的蛋白质结构,解释生物的蛋白质结构与人体的相互作用,挖掘潜在药物分子的特性,连接精选文献中的点以解释动作机制,在测定数据中寻找证据,并且使用先前试验的数据分析潜在安全性和有效性等。传统上讲,这个过程手动完成,并且花费几个月。
11.该问题的繁琐性质归因于生命科学研究人员进行以下各项所需的时间:(a)通过将蛋白质序列与先前已知或研究的致病生物(超过4百万个序列)匹配和比较来理解致病生物;(b)处置并处理多模态大数据(蛋白质序列、蛋白质组学相互作用、生化途径、过去临床试验的结构化数据等);(c)整合并搜索跨多个多模态多太字节数据集连接的模式;(d)安装、配置并运行大量工具(遗传学、蛋白质组学、分子动力学、数据科学等)以生成见解;以及最后(e)验证并证实药理学解释的科学严谨性。本文中所公开的系统和方法的实施例超越了仅仅使传统过程自动化并且提供用于实现再利用管道的新技术和技巧。实施例可以使用大规模并行处理图数据库技术来提供更快速的响应以在流行中加速药物再利用管道。这表示当前技术的巨大改进,该当前技术用于通过蛋白质序列分析来标识用于重新购买/重新定位已知和新型疾病的药物的候选药物,这些疾病包括例如新型流感毒株、冠状病毒、遗传性罕见病等。
12.实施例涉及大规模并行处理图数据库用于快速响应药物再利用的应用。各实现方
式可以使用可扩展图数据库,该可扩展图数据库被配置为托管从多个知识源集成的医学相关事实的知识图,并且还充当能够进行数据库内蛋白质序列分析的计算引擎。实施例可以被配置为基于受试者病毒或其他病症的处理序列来使用图数据库进行多模态药物再利用,标识具有相似或匹配序列的其他已知病毒/病症,并且查询与那些已知病毒/病症相互作用的化合物和治疗剂的特性。
13.实施例可以提供大规模并行图数据库,该大规模并行图数据库(a)存储、处置、托管和处理以知识图形式表示的多模态数据;(b)为数据驱动发现提供交互式查询和语义遍历能力;(c)加速领域特定函数,诸如进行蛋白质相似性分析的史密斯-沃特曼(smith-waterman)算法、用于图论连通性和相关性分析的以顶点为中心的全图算法,诸如页面等级(pagerank);以及(d)运行/执行跨多个数据集来以秒为量级而非几个月为量级来生成药物假设的查询工作流程。实施例实现了多个多模态生命科学数据库的综合知识图,并行进行蛋白质序列匹配,并且提供了一种新型快速药物再利用方法,该新型快速药物再利用方法能够跨4百万多个蛋白质、155十多亿个事实查询,同时处置大约30tb的数据。
14.一些应用对covid-19大流行病之外的其他生物医学发现问题实现可推广大数据平台,该可推广大数据平台允许:(a)可扩展图数据库,该可扩展图数据库提供知识遍历和发现所需的数量级计算加速和交互性;(b)集成生命科学知识图,该综合生命科学知识图捕获了可用生物医学事实的开放科学领域;(c)对正在进行的大流行病的潜在候选药物的假设;(d)可复制代码和结果,用于对生物医学事实领域(对病毒、蛋白质、药物、生化途径)的未来研究,而非局限于疾病特定知识图的实践状态。
15.实施例可以使用cray图引擎或其他类似引擎来实现。cray图引擎(cge)为存储器中语义图数据库,该存储器中语义图数据库被设计为扩展到cray xc超级计算机上的数百个节点和数万个进程,以支持对大型数据集(约100tb)的交互式查询。cge基于标准化资源描述框架(rdf)格式,以摄取n-三元组/n-四元组的数据集,并且使用sparql查询语言实现查询。rdf数据被表达为带有“四元组”标签的有向图,其中“四元组”由四个字段组成:主语、谓语、宾语和图。三元组只是被存储在“默认图”中的四元组。例如,以下是来自可以被加载到cge中的uniprot covid-19数据的示例rdf三元组的简化版本:
[0016][0017]
作为数据结构的图可以包括可能连接的网络。顶点或节点通常是指实体(数据、人员、企业等),并且实体之间的连接为边缘。图数据库可以被用于标识被连接到其他实体的实体。通常,本地处理可以被用于处理节点周围的少量数据。然而,其他任务可能涉及在更全面的基础上(例如,在全图分析中)评估边缘/连接。语义图可以包括这样的三元组的集合,其中主语和宾语表示顶点,而谓语表示顶点之间的边缘。语义图数据库与关系数据库的不同之处在于底层数据结构是图,而非表的结构化集合。图结构使语义数据库成为分析松散连接或无架构式的多模态非结构化数据和结构化数据的理想选择,如同社交网络相互作用或活体生物中蛋白质和基因之间的相互作用一样。
[0018]
在各个实施例中,cge可以包括两个主要部件:字典和查询引擎。字典负责构建数据库,这是从高性能lustre文件系统中提取原始n-三元组/n-四元组文件并且将它们转换为cge所使用的内部表示的过程。字典存储来自n-三元组/n-四元组的唯一rdf串,并且提供
唯一串与查询引擎内部用于四元组的整数标识符之间的映射。大部分字典构建时间可能由lustre i/o时间主导。
[0019]
cge查询引擎处理sparql查询和sparul更新请求,提供可以应用于查询数据的若干个内置图算法(诸如例如,中心性测量、页面等级、连通性分析)以及向用户返回结果。查询引擎所执行的核心工作可以包括:匹配sparql查询中的基本图图案以及支持对查询结果的操作(诸如filter和order),这些操作允许用户分别移除和排序解决方案。
[0020]
可以实现与cge接口以使用高性能互连提高具有通用处理器的超级计算机产品的性能和可扩展性的实施例。在传统cge之上和之外添加了多种特征,以专门支持快速响应药物再利用。
[0021]
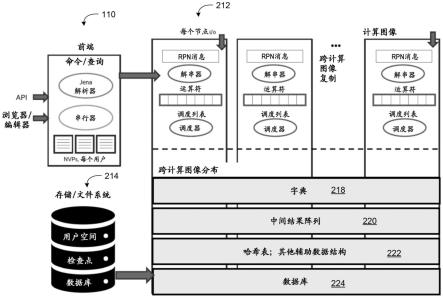
图1图示了根据各个实施例的示例图引擎。该示例包括前端210和跨多个计算机图像212复制的多个资源。这些资源可以包括例如如计算图像212中所示的解串器、运算符和调度器。示例图引擎还可以包括字典218、待提升中间结果220、哈希表、以及其他令人振奋的数据结构222和数据库224。存储文件系统214可以被用于容纳数据库224以及用户空间、检查点和其他数据。
[0022]
前端210提供接口,通过该接口,用户可以诸如例如通过提交查询并且从查询接收回结果来与图引擎交互。在背内侧上,图引擎在硬件上运行,它们可以构建在经过分区的全局地址空间的顶部上,这可以允许系统将独立进程和图像视为其自身实体,但您可以细分数据并且使用通信库跨图像共享数据。图引擎可以被配置为以协调方式运行数万个图或数十万个图,其中所有图都可以在其自身子集上独立运行,然后,当需要结果时使用库进行同步。
[0023]
图2图示了根据各个实施例的图引擎查询执行的示例。现在,参考图2,用户可以提交示例查询320。通信接口324可以提供前端(例如,前端210)计算节点与后端计算节点之间的通信和控制。在该示例中,通信接口可以包括诸如sparql协议和rdf查询语言(sparql)转换器、与网络浏览器的ip接口、显示或转发sparql和命令结果的服务、生成低级别查询(例如,rpn)的服务、以及传递非sparql命令的服务之类的元素。
[0024]
可以提供多个计算节点328来执行查询操作。在该示例中,图像被接收(一个图像被示为图像0 334)。计算节点可以接收、验证并且向所有图像发送rpn,并且当获得结果时,将这些结果连同指针一起发送到输出文件。计算节点可以包括对图像执行操作的多个运算符336。该示例中包括的运算符336为scan、join、merge、optional、union、filter和bind,尽管还可以使用其他操作。这些操作可以被用于以不同方式遍历各个数据库338中的数据以完成查询。在图2图示的示例中,示例查询320用于标识在商店处销售dvd的人员。该查询可以被转换(例如,sparql)并且被提交到计算节点,该计算节点执行适合该查询的各种操作(例如,运算符336)以标识销售dvd的人员。
[0025]
在各个实施例中,也可以改进计算节点328以执行蛋白质序列分析。这可以作为进入数据库中的领域特定能力实现,以为各种应用(包括例如药物标识)提供序列分析并且再利用它们。图3图示了根据各个实施例的用于执行蛋白质序列分析的图引擎诸如cge的示例实施方式。该示例还示明了示例查询420,它们可以经由接口424被提交给cge中的计算节点428。与图3的示例一样,在该示例中,所包括的运算符436包括scan、join、merge、optional、union、filter和bind等。然而,与图3的示例不同,在该示例中,运算符436还包括执行蛋白
质序列分析的运算符。在一个示例中,cge被修改以定义函数的接口,该接口可以被用作运算符(诸如order或filter)中的评估表达式的一部分。该函数被称为用户定义函数,因为cge的用户可以编写该函数以将领域特定知识应用于查询结果。cge提供了通用函数,用户可以通过它们对其自身函数进行改写,cge将在程序启动时将其加载到存储器中以在执行期间使用。cge定义了与函数的接口,以便使得能够向用户函数传递参数以及出于评估运算符的表达式的目的,允许用户向cge返回信息。用户定义函数返回给cge的信息使得领域特定函数能够轻松地对结果进行排名或过滤。在各个实施例中,系统可以被配置为使得用户还能够添加用户定义函数来执行自定义搜索/查询。
[0026]
图4图示了可以用于执行蛋白质序列分析的样本查询的示例。这可以是上文在图3中引入的查询420的示例。如在该示例中看到的,在442处,示例查询可以包括给定病症的蛋白质的标识,针对该蛋白质,用户想要将其标识为可行治疗剂。在该示例中,用户标识了sars2刺突蛋白,并且查询包括助记符,该助记符在该示例中为
‘
spike_sars2’。查询的这一部分还标识了感兴趣病症的蛋白质序列,该感兴趣病症在这种情况下为病毒。核心序列可以包括病毒的蛋白质序列的全部或一个或多个片段。
[0027]
还如在该示例中所见,在454处,查询可以指定查找具有相同序列信息的所有其他蛋白质的请求。这可以包括请求,以查找整个序列或该序列的一个或多个部分以查看是否存在具有匹配序列或匹配序列片段的任何其他蛋白质。在455处,查询请求将感兴趣病症(sars2刺突)与数据库中的其他蛋白质进行比较,并且基于它们的距离对序列匹配进行评分以提供相似性得分。在457处,查询请求返回结果并且这些结果以基于相似性得分的降序次序来进行排序列举。
[0028]
图5图示了根据所公开的技术的一个实施例的可以被用于实现蛋白质序列分析的示例计算部件500。计算部件500可以为例如服务器计算机、控制器、或能够处理数据的任何其他类似计算部件。在图5的示例实施方式中,计算部件500包括硬件处理器502和机器可读存储介质504。硬件处理器502可以为一个或多个中央处理单元(cpu)、基于半导体的微处理器、和/或适用于检索并执行机器可读存储介质504中存储的指令的其他硬件设备。硬件处理器502可以获取、解码和执行指令(诸如指令506至514),以控制用于合并局部参数的过程或操作,以实现用于快速响应药物再利用的蛋白质序列分析。作为检索和执行指令的备选或补充,硬件处理器502可以包括一个或多个电子电路,该一个或多个电子电路包括用于执行一个或多个指令的函数的电子部件,诸如现场可编程门阵列(fpga)、专用集成电路(asic)、或其他电子电路。
[0029]
机器可读存储介质(诸如机器可读存储介质504)可以是包含或存储可执行指令的任何电子、磁、光或其他物理存储设备。因此,机器可读存储介质504可以为例如随机存取存储器(ram)、非易失性ram(nvram)、电可擦除可编程只读存储器(eeprom)、存储设备、光盘等。在一些实施例中,机器可读存储介质504可以为非暂态存储介质,其中术语“非暂态”不涵盖暂态传播信号。如下文所详细描述的,机器可读存储介质504可以被编码有可执行指令,例如指令506至514。
[0030]
硬件处理器502可以执行指令506以接收包括给定病毒或其他病症的序列的查询。例如,蛋白质或基因测序装置可以被用于确定感兴趣病症的序列。如上文关于图4中的示例查询所描述的,可以构建查询,该查询包括给定病症的蛋白质序列的标识,针对该蛋白质序
列,用户想要将其标识为可行治疗剂。所构造的查询还可以包括感兴趣病症的助记符以及蛋白质序列或该蛋白质序列的片段。
[0031]
硬件处理器502可以响应于所接收的查询而执行指令508,搜索序列数据库以标识具有与感兴趣病毒(或蛋白质)的序列相似的序列的其他病毒或蛋白质。如上文参考图4所指出的,系统可以搜索数据库以标识与整个序列或该序列的一个或多个所标识的片段的匹配或相似性。关于该操作,硬件处理器502可以执行指令510,可以将序列数据库中的序列与查询序列(整个序列或该序列的一个或多个所标识的片段)进行比较以确定是否可以标识任何匹配。
[0032]
硬件处理器502可以执行指令512以基于比较来确定相似性得分。相似性得分可以包括例如数值(例如,作为定义范围内的数字或作为百分比等)或者指示给定序列与查询序列的相似程度的其他指示符。例如,如果表达为百分比,则100%可以表示完全匹配。该系统可以被配置为返回相似性得分大于给定阈值的蛋白质的结果。例如,系统可以标识与大于70%(或某个其他阈值)的相似性得分匹配的蛋白质。在各个实施例中,阈值水平也可以由查询来设置。
[0033]
硬件处理器502可以执行指令514以查询图数据库,以标识对蛋白质序列具有大于所标识的阈值的相似性得分的蛋白质具有抑制效果的治疗剂(药物或分子)。例如,针对具有大于阈值的相似性得分的每个蛋白质,系统可以在图数据库标识对那些蛋白质具有效果的治疗剂。系统可以返回标识具有期望效果的治疗剂的结果,并且可以根据相似性得分对结果进行评分。换言之,数据库中已知的对序列数据库中的蛋白质具有期望效果的治疗剂可以被视为对感兴趣病症具有期望效果的可能性更高。
[0034]
各个实施例中可以包括两个核心cge改进以支持药物再利用:(a)支持用于数据库中蛋白质序列分析的用户定义函数(udf);(b)能够使用上文所描述的sparql前端用户接口并行执行这种领域特定udf以加速和横向扩展。
[0035]
例如,因为cge利用jena的sparql查询解析器接口,所以cge中用户定义函数的语法可以遵循apache jena准则。作为查询的一部分,sparql接口允许在查询表达式内使用自定义函数来对数据实现领域特定操作。这是允许用户定义、表达和执行领域特定数学运算以对sparql不支持的查询结果进行评估和排名的特征。这样的图操作可以被实现为表达式中的由uri定义的自定义函数。该能力可以被配置为允许用户定义他们自己的函数。对这些用户定义函数的调用可以采用以下形式:
[0036][0037]
用于药物再利用的两个自定义用户定义函数(udf)(新uri,arq:user_func),可以被包括在cge中,以调用与cge分开存在的udf的调出。单个c接口可以被定义名为cge_user_eval的函数,cge可以将该函数作为表达式的一部分来执行。cge_user_eval函数接受四个参量,这四个参量提供参量总数、参量列表、返回值和返回类型。这可以允许用户将数据从cge传递到udf、评价参量、以及返回可以用于评价sparql表达式的原始值(例如,布尔、整数或双精度)。
[0038]
因为cge以大规模并行方式执行,其中可能同时运行数万个图像,所以作为查询的一部分,调取的任何udf也可以在查询解决方案集合上并行执行。udf的并行执行使得其能够扩展到数据集,否则这些数据集可能会因跨并行图像划分数据而变得太大。udf还可以以令人尴尬的并行方式被应用到整个解决方案集合。此外,经由udf的并行执行可以通过将复杂处理任务分解为需要处理的相当小的输入集合的图像来实现计算密集型算法的分布式执行。
[0039]
实施例可以包括集中于改进数据库操作(诸如filter或group)的执行的cge的增强。这些操作可以使得用户能够比较作为查询匹配的一部分所找到的术语,以应用某种次序或排名。这些术语的原始串可以存储在cge字典中,该cge字典可以实现为将串分布在所有进程中的分布式哈希表。当需要将术语作为操作的一部分时(诸如filter或group),这些术语的这种分布会产生将术语从本地提取到进程的大量工作。
[0040]
为了提高这些操作的整体性能和可扩展性,图像所使用的串可以以协调方式作为大块来被获取。每个图像可以通过结果并且从每个其他图像中创建它需要的串列表。然后,所有图像可以从彼此获取作为单个块的所需串,而非单独地发出每个串的远程获取。这会增加每个消息的大小,但会显著减少所需消息的总数。这种通信模式与cge中为核心图操作(诸如join和merge)所做的工作相匹配,并且在先前研究中已经证明可以通过一次减少未完成消息的数目来显著提高性能[8]。这种cge改进对于查询蛋白质序列与数百万开放科学序列的平行成对比较以及对结果集进行排名排序至关重要。
[0041]
实施例使用生物医学或其他生命科学数据资源整合生命科学知识图。例如,实施方式可以将通常用于生命科学和系统生物学研究的可用生物医学数据资源用于知识图。研究人员的通常工作流程是在数据库中的一个数据库中执行搜索,然后构造另一数据库的查询,并且进行迭代。在各个数据源的本体之间手动映射并且将来自多个查询端点的结果拼凑在一起(或使用又一数据库来执行该转换)是个繁琐过程。在各个实施例中,cge的可扩展性使得能够在一个环境中加载所有相关数据库,从而实现无缝跨数据库查询。在各个实施例中,联合查询还可以被用于跨多个数据库进行查询,然而,由于诸如来自防火墙系统的网络访问、查询速率限制或复杂联合查询的简单性能问题之类的挑战,该方法可能不太适合复杂查询。cge的可扩展加载时间还支持频繁重新加载数据集,包括在工作流程期间将内部数据集成到公共数据库的背景的顶部上。此外,用于数据库构建过程的cge的性能和可扩展性使得能够在不到一个小时的时间内快速拉入更新过的数据并且完全重建数据库。
[0042]
为研究covid-19的潜在药物再利用候选物而组装的集成生命科学知识图由公众可用的数据库集合生成。现在对集合中较大数据库的描述以及本文档中所明确提及的描述进行描述。
[0043]
uniprot:uniprot数据库为关于蛋白质的功能信息集合,并且包括注释、相互关系、以及在某些情况下,蛋白质本身的氨基酸序列。蛋白质为用于研究药物蛋白质结构和相互作用的基石。蛋白质之间的相互作用复杂并且有广泛联系,因此图表示特别有用。uniprot专注于人类蛋白质,尽管其他广泛研究的生物也有很好的代表性。
[0044]
uniprot联盟是欧洲生物信息学研究所(ebi)、瑞士生物信息学研究所(sib)和蛋白质信息资源(pir)之间的合作品。它一直是语义网技术的先驱,自2008年以来,uniprot一直以rdf格式分发。uniprot随着更多科学数据的加入而不断增长。新uniprot版本每四个星
期分发一次。
[0045]
对于本研究,大部分uniprot rdf数据来自2020年3月19日的发布。存在新uniprot门户,用于提供covid-19冠状病毒蛋白入口和受体的最新信息,该最新信息独立于一般uniprot发布周期而更新。对于covid-19研究,这使我们能够更快地拉取更新过的covid数据。知识图的covid-19uniprot数据已于2020年5月22日更新。uniprot数据库包含大约876亿个三元组。以n-三元组(.nt)文件的形式,它在磁盘上粗略为12.7tb。为了简化跨多个数据库的查询,我们将所有命名图合并为单个默认图。
[0046]
pubchem:pubchem是由美国国立卫生研究院(nih)维护的开放化学数据库。pubchemrdf项目为pubchem化合物、物质和生物测定数据库提供rdf格式化信息。本研究的知识图使用了于2020年3月30日从ftp://ftp.ncbi.nlm.nih.gov/pubchem/rdf下载的pubchemrdf的v1.6.3测试版本。pubchemrdf数据库包含大约800亿个rdf三元组。以n-三元组的形式,这在磁盘上相当于约13tb。
[0047]
chembl:chembl是具有类似药物特性的生物活性分子的人工精选数据库。它将化学、生物活性和基因组数据汇集在一起,以帮助将基因组信息转化为有效新药。数据被定期更新,大约每3个月至4个月发布一次。chembl-rdf版本27.0(2020年5月18日)已经被集成到本研究的知识图中。chembl数据库包含大约539m个三元组。以n-三元组的形式,这在磁盘上相当于大约81gb。
[0048]
bio2rdf数据集:bio2rdf是个开源项目,该开源项目使用语义网技术来将来自多个数据提供者的不同的数据集拉取到一起。除了提供用于跨异构数据集集合进行查询的基于virtuoso的在线sparql端点之外,bio2rdf还提供了门户来下载用于bio2rdf数据库所包含的数据集的经过转换的rdf数据文件。下载知识图中所包含的bio2rdf数据集。
[0049]
完全bio2rdf集合由跨35个数据集的大约110亿个三元组组成,并且包括drugbank、pubmed和mesh数据集。
[0050]
orthodb:orthodb(https://www.orthodb.org)提供直向同源物(即,现存物种从它们最后的共同祖先继承的基因)的进化和功能注释。由于直向同源物是最有可能保留其祖先基因的功能的候选物,所以orthodb旨在缩小关于基因功能的假设并且进行比较进化研究。
[0051]
orthodb数据库包含大约22亿个rdf三元组,这些rdf三元组描述了来自1.5万个生物的4000万个基因的进化和功能特性。以n-三元组的形式,这在磁盘上相当于约275gb。
[0052]
表1知识图数据集特点。重复删除之前的原始大小。
[0053]
数据集大小(盘上)大小(三元组)源uniprot(2020年3月)12.7tb876亿[7]pubchemrdf(v1.6.3beta)13.0tb800亿[12]chembl-rdf(27.0)81gb5.39亿[13]bio2rdf(版本4)2.4tb115亿[14]orthodb(v10)275gb22亿[17]biomodels(r31)5.2gb0.28亿[20]biosamplpe(20191125)112.8gb11亿[21]ols(2018年3月)10.2gb0.788亿[22]
reactome(r71)3.2gb0.19亿[23]
[0054]
biomodels:biomodels数据库是表示生物系统的数学模型的储存库。它目前拥有一系列描述过程的模型,如发信号通知、一个或多个蛋白质-药物相互作用、代谢途径、流行病模型等。biomodels所托管的模型通常在同行评审的科学文献中进行描述,并且在一些情况下,它们从通路资源(path2models)自动生成。这些模型由人工精选,并且通过对外部数据资源(诸如出版物、化合物及途径的数据库、本体等)的交叉引用在语义上进行丰富。
[0055]
实施例提供cge支持udf的能力,可以被实现为使得可以编写查询,这些查询组合来自知识图的信息并且将领域特定udf应用于数据以便更好地细化结果。对于药物再利用,实施例可以实现执行蛋白质序列相似性以推断蛋白质之间的连接的udf。这可以被配置为使得能够推断出鲜为人知的蛋白质(诸如covid-19)与开放数据集中详细记录的蛋白质(诸如uniprot和chembl)之间的连接。
[0056]
实施例可以利用史密斯-沃特曼(sw)蛋白质相似性算法来比对序列对并且计算比对的相似性得分。对于长度为m和n的两个序列,sw算法返回最优局部比对和相似性得分,其中计算时间复杂度为0(mn)。局部比对用于提供描述序列内最相似区域的比对,而非由全局比对返回的序列的端到端比对。由于sw返回最佳局部比对,所以它是许多比对器的重要部件。然而,计算复杂度限制了该算法被用于比较大序列集的程度。
[0057]
因为偏好使用最佳局部比对来对相似性进行评分并且高度优化的开源实施方式作为可以由cge加载的独立c/c 库的可用性[25],所以sw算法在各个实施例中可以是合乎需要的。鉴于cge的高度并行实施方式,用户可以在几秒钟内查询知识图,并且执行数百万次蛋白质相似性计算,使得能够通过相似性得分对解决方案轻松进行过滤和排名。
[0058]
为了对得分进行归一化,每个序列可以与其自身进行比较。这些得分的平方根的乘积被用作分母[26],如清单1中所概述的。
[0059][0060]
清单1.史密斯-沃特曼得分的归一化方法
[0061]
本文中所描述的系统和方法的实施例可以允许研究人员了解新型冠状病毒与其他已知病毒的相似程度或不同程度。如果构成新型病毒的蛋白质序列的部分与其他已知病毒具有序列和功能重叠,则集成知识图中的信息帮助我们推断搜索,以标识抑制已知病毒上的病毒致病活性的潜在候选的已知药物。现在,提供在以下段落中实现基于相似性的推断的简单示例查询。
[0062]
covid-19相似性:covid-19蛋白序列由数个非结构蛋白、包膜蛋白、刺突蛋白等组成。为了假设与covid-19病毒蛋白质的不同部分结合或相互作用的潜在药物,实施例可以首先标识具有与新型covid-19突变相似的结构的开放科学蛋白质。清单2中的查询是查找与covid-19刺突白质序列最相似的蛋白质的示例。
[0063][0064]
清单2.对与参考蛋白质相似的蛋白质进行排名的sparql查询
[0065]
清单2中的示例相似性查询首先使用uniprot助记符查找spike_sars2的蛋白质序列。接下来,检索具有序列和名称的所有蛋白质的序列。最后,这些序列中的每个序列与spike_sars2的序列进行比较,并且相似性得分被保存在变量sim中。bind子句将所有sim值保存在临时表中,以使它们可以被用于其他操作以及被返回给用户。在该查询中,删除相似性得分小于0.1的任何结果,并且将这些结果通过相似性得分以降序次序返回。
[0066]
所返回的相似性得分最高的蛋白质为a0a2d1px97,其为“蝙蝠sars样冠状病毒”,其中相似性得分为0.817。排名靠前的结果中的数个结果为蝙蝠冠状病毒或其他物种的冠状病毒,如表ii中列出的前10个结果所示。相似性得分从0.79迅速下降到0.37,这是其中蛋白质a0a2i6pix8(即,“中东呼吸综合征相关冠状病毒”)的相似性得分为0.368的中东呼吸综合征(mers)首次出现在结果中的点。mers的数个蛋白质之后是其他物种中的若干个冠状病毒,这些物种包括牛、人、兔和鼠,并且数个非冠状病毒蛋白质开始出现,诸如相似性得分为0.322的“传染性支气管炎病毒”的a0a1b2rx89。这些得分与表明covid-19可能起源于蝙蝠并且与mers非常相似的研究相吻合[27]。
[0067]
表ii
[0068]
与covid-19刺突最为相似的前10个蛋白质序列
[0069]
蛋白质学名得分a0a2dipx9t"ba sarsike coonavins"0.817a0a0u2wm2"sars-ike corona inis wtv16"0.817a0a 2dipxa9"bat sars-ike coronavins"0.816u5wlk5"bat sars-ke coronavins rshc01470.814a0a2d1px29"bat sars-ke coronavins"0.814
u5whz7"bat sars-ire comonavins rs33677"0.813u5w105"bat sa rss-ike coronavius wivt"0.813a0a2d1pxc0"bat sarshke corouavins"0.813a0a2d1pxds"bat sar-ie coronav ins5"0.812a0a4y6gl47"coronavinus btrs-betacov/yn2018b"0.812
[0070]
covid-19药物再利用:在获得相似性分析结果之后,可以实现实施例以利用知识图来基于相似性得分排名,以寻找可以被再利用用于covid-19的潜在药物。为此,使用被配置为反向工作而非查找给定化合物的所有已知目标的sparql查询,查询从未知蛋白质开始并且搜索可能以它为目标的潜在化合物。在这种情况下,该示例集中于对蛋白质具有抑制效果的化合物。清单3中的示例查询可以被用于进行该搜索。
[0071]
[0072][0073]
清单3.找出可以再利用的潜在药物的sparql查询
[0074]
该示例查询存在三个主要部件。首先,顶部的内部查询在chembl中搜索关于已经通过某个开发阶段的具有抑制活性化合物的信息。因为意图是再利用现有药物,所以这些化合物可能仅限于处于3期或更高期的临床试验开发阶段的化合物。在第二内部查询中,作为给定化合物的已知靶标的所有蛋白质与covid-19刺突蛋白质进行比较。将结果通过与spike_sars2蛋白质的相似性放入降序次序,并且仅返回前150个同种型的蛋白质。给定蛋白质通常存在多个序列,在这种情况下,前150个同种型与大约前50个最相似的蛋白质相关联。查询的最后部分再次将选定蛋白质与靶向它们的化合物以及来自第一内部查询的活性信息进行匹配。最终结果通过相似性得分以降序次序返回,以基于与covid-19刺突的相似性而突出显示潜在地可能被再利用的化合物。
[0075]
反向查询返回靶向蛋白质的化合物,其中相似性得分范围从0.2降到0.183。这些化合物中的数个化合物用于已投入临床试验的药物,因为它们有可能被再利用来对抗covid-19[29]。表iii中还示出了在临床试验中的通过反向查询发现的对抗covid-19刺突的得分最高的蛋白质序列中的一些蛋白质序列。
[0076]
表iii
[0077]
当前反向查询结果中出现的covid-19的临床试验中的示例药物
[0078]
蛋白质化合物名称得分p52333baricitinib0.194p17948ribavirin0.189p17948ritonavir0.189
p17948dexamethasone0.189p17948azithromycin0.189p08183lopinavir0.187
[0079]
我们用来验证反向查询所返回的结果的一种方法是比较所返回的化合物与当前用于covid-19的临床试验的化合物之间的重叠。基于目前在6月初进行临床试验中的一部分的药物,我们创建了包含196种独特药物的清单,以与我们的结果进行比较。对于考虑与spike_sars2蛋白质最相似的前150个同种型序列的上述查询,知识图所返回的结果包括196个化合物中的91个化合物(46%)。反向查询所发现的化合物与临床试验列表之间的显著重叠也有助于定义可以被认为是感兴趣的得分范围。由于反向查询所发现的蛋白质的相似性得分都介于0.183与0.20之间,所以靶向得分在相同范围内的其他蛋白质的化合物也可能对covid-19产生有益影响似乎是合理的。
[0080]
新假设(破伤风):使用spike_sars2的反向查询所返回的一个潜在感兴趣结果为破伤风毒素,它具有uniprot标识符p04958和助记符tetx_clote。针对刺突的反向查询返回tetx_clote作为最高匹配项,其中相似性得分为0.20。鉴于无症状covid-19阳性病例的比例很高,疾病控制和预防中心(cdc)目前估计该比例为40%[30],tetx_clote结果引起了一个意外但感兴趣假设,即,破伤风疫苗可能通过使得免疫系统能够对病毒生成合理响应来有助于无症状率,并且降低症状的严重程度。根据cdc,在2017年,美国19岁及以上成年人中约有63.4%在过去10年内按照建议接种了某种形式的破伤风疫苗,其中65岁以上的个体显著下降。虽然破伤风由细菌引起并且covid-19是一种病毒,但是细菌与病毒之间存在多种异源免疫示例。这种异源免疫至少最初归因于不同病原体抗原的t和b细胞表位的氨基酸序列相似性。
[0081]
数据库性能
[0082]
为了促进covid-19研究,生命科学知识图托管在少数较大cray xc-40开发系统上。这些系统主要包含intel broadwell、skylake和cascade lake处理器的组合。文件包含用于构建数据库的n-三元组以及所构建的数据库,该文件被存储在所附带的lustre文件系统上,并且进行条带化以匹配文件系统中的可用数目的对象服务器目标(ost)。
[0083]
本章节中的性能结果是在频率范围为2.1ghz至2.4ghz的具有双插槽48核skylake节点以及48核skylake节点和56核skylake节点的混合的内部370节点(336个计算节点、34个服务节点)xc-40开发系统上运行的。这些节点中的大多数节点具有192gb ddr-2666存储器,但63个cascade lake节点具有更大的384gb ddr4-2933存储器。所附带的lustre文件系统为提供655tb的存储的具有8个ost的sonexion cs-l300n系统。数据库构建和加载时间由进出lustre文件系统的i/o性能决定,因此i/o系统性能为重要的考虑因素。所报告的查询执行时间是严格的查询时间,并且不包括将结果写入lustre文件系统所需的时间,这是常见做法。
[0084]
数据库构建:如先前在cge背景章节中所提及的,cge所完成的第一步是从输入n-三元组/n-四元组文件集合构建数据库,以在查询引擎所使用的表示中产生编译数据库。用于生命科学数据库的原始n-三元组输入文件在lustre上为28.29tb。构建过程由cge的字典部件处置,并且该构建过程由数个步骤组成,表v中概述了这些步骤以及每个步骤的时间(以秒为单位)。
[0085]
表v
[0086]
生命科学数据库的构建步骤的时间
[0087][0088]
如数字所示,数据库的构建时间主要取决于从lustre读取原始n-三元组文件的时间,这是预期的。其余构建步骤(即,摄取、同步和更新)的时间从128个节点(2048个图像)扩展到256个节点(4096个图像)很好地扩展。所构建的数据库的检查点写入lustre,所以后续使用相同数据库重新启动cge可以加载经过编译的数据库,而不必再次摄取原始n-三元组。所构建的数据库在磁盘上只有约5.4tb,而原始n-三元组在磁盘上约为28.29tb,并且cge可以在大约568秒内在256个节点(其中每个节点有16幅图像)上使用所构建的数据库来重新启动。
[0089]
刺突相似性查询:用于使用生命科学知识图测试cge性能的第一查询是清单2中的相似性查询。该查询从uniprot中搜索满足特定条件(诸如具有序列值和推荐名称)的已知蛋白质,并且将它们与spike_sars2的序列值进行比较。相似性查询找到49,299,877个蛋白质序列以与spike_sars2序列进行比较。表vi示出了用于计算4930万个蛋白质序列的sw计算的时间以及当每个节点使用16幅图像或32幅图像在128个节点和256个节点上执行时的总查询时间。
[0090]
查看sw计算的时间,我们观察到计算相似性的时间通过图像计数(即,核)和节点计数两者很好地扩展。这归因于计算与其他计算无关的事实,所以所有图像都可以并行计算蛋白质序列的子集的计算。扩展还突出显示了在cge的大规模并行上下文内利用sw计算的优点。如果在单个进程上执行的知识图执行相同sw计算,则可能花费约21,709秒(即,10.6
×
2048),从而基本使得在串行上下文中无法进行查询。严格的查询时间确实示出了从128个节点到256个节点的合理扩展,每个节点至少16幅图像,但查询的扩展受到group运算符的性能限制。由于相似性得分四舍五入到小数点后三位,所以当将值作为新变量(即,?sin查询变量)存储在cge内时需要删除大量重复值。由于cge跨图像分配这些新变量的方式,所以大量重复可能导致数个图像等待少量图像完成它们将存储的值的处理。此外,查询扩展还与最近性能改进有关,这些改进使得诸如group和filter之类的操作能够将所需串作为块而非单独来获取。蛋白质序列可能很长,范围从数百个到数千个氨基酸不等,所以从远程进程获取这些长串作为块对于防止通信开销主导查询性能至关重要。
[0091]
刺突反向查询:用于测试cge在生命科学知识图上的性能的下一查询是来自列表3的反向查询。该查询从spike_sars2的蛋白质序列开始并且搜索作为具有期望效果(即,抑
制性)的可接受化合物的靶标的相似蛋白质。因为必须对大型中间结果进行若干个连结才能找到唯一期望蛋白质或化合物,所以反向查询比相似性查询复杂得多。虽然复杂连结会影响整体查询时间,但连结所施加的额外病症显著减少了必须比较的蛋白质序列的数目。cge已经被优化以通过重用来自查询的先前部分的信息,以在扫描和连结阶段期间在查询的早期滤出解决方案。该优化可以被用于减少必须连结的中间结果的大小,这不仅对性能有用,而且还对像反向查询这样复杂的查询的存储器要求也很有用。对于spike_sars2反向查询的情况,所比较的蛋白质的数目仅为1,165,914,其远小于相似性查询中所比较的4930万个蛋白质。
[0092]
如在表vii中示出的数字,由于cge能够利用知识图内的信息显著减少所考虑的蛋白质的数目,所以sw时间是查询时间的非常小的部分。反向查询的严格查询时间是相似性查询时间的两倍之多,这是预料之中的,因为反向查询中的复杂连结的数目较多。大多数严格查询时间由连结主导。例如,在每个节点具有16幅图像的256个节点上,严格查询时间为49.0秒,其中34.52秒用于进行连结。即使使用复杂连结,当每个节点使用16幅图像(1.82倍加速)时,从128个节点到256个节点的性能扩展得相当好,但是虽然每个节点32幅图像的查询速度更快,但扩展效率并不高。每个节点具有更多核的有限扩展与由于图像访问同一节点上但在不同插槽上的存储器而导致的存储器访问瓶颈有关。
[0093]
因为没有其他已知大型语义图引擎能够加载这种幅度的现实世界生命科学数据集,所以将cge的性能与其他数据库引擎进行比较是不容易的。然而,先前基准已经通过标准lubm万亿三元组数据集清楚地表明,cge比任何竞争对手快至少一个数量级,尤其是当执行复杂查询时[8]。对于lubm的情况,通常基准查询为数字9,它进行多个复杂连结来搜索数据集以查找实体之中的某个三角关系[9]。
[0094]
表vi
[0095]
刺突相似性查询的扩展结果
[0096][0097]
表vii
[0098]
反向查询的扩展结果
[0099]
[0100][0101]
可扩展性优点:cge的性能和可扩展性已在先前研究[8]、[10]以及上文所讨论的结果中得到证实。当尝试以交互方式搜索如此大的数据集时,这种大规模性能至关重要。虽然可以利用较小图引擎来分析相同数据,但是执行简单查询所需的时间几乎肯定会太长而无法使系统发挥作用。即使当跨图执行最复杂的查询时,cge能够扩展到数百个节点和数万幅图像的能力也使得能够在几秒钟内快速摄取并且搜索非常大的数据集。这种可扩展性对于udf也非常有利,因为它使得领域特定函数(其中一些可能在计算上很复杂)能够轻松应用于改进查询结果的细化。cge[8]的这些无可比拟的能力使研究人员能够搜索非常大的真实世界数据集,以便找到可以实时有效地再利用的化合物。
[0102]
关于药物再利用问题的见解
[0103]
虽然本研究主要关注知识图和sw序列比较对covid-19的应用,但这些变化和查询可以应用于任何数目的感兴趣疾病。例如,如果将清单2中相似性查询中的spike_sars2助记符改变为牛痘病毒的uniprot助记符的u5tgx1_cowpx,则相似性查询可以用于查找与给定牛痘最相似的蛋白质。在这种情况下,运行该相似性查询会返回多个牛痘病毒作为前三个结果,其次是taterapox,再次是三个骆驼痘病毒。更感兴趣的结果开始出现在相似性列表上的第7位,即,用于“痘苗病毒wau86/88-1”的蛋白质v5qzd2的uniprot助记符v5qzd2_9poxv,其中相似性得分为0.892。由于痘苗病毒与天花病毒(天花的病原体)的相似性,所以它比任何其他疫苗都更多地用于人类免疫接种。
[0104]
反向查询还可以用于其他疾病以搜索可以再利用的潜在药物。例如,将spike_sars2助记符替换为用于“人类疱疹病毒1”(hhv1)的uniprot助记符kith_hhv11,返回溴夫定(brivudine)作为抑制蛋白质p06479的顶级化合物,其中相似性得分为0.984。已知溴夫定对水痘带状疱疹病毒和1型单纯疱疹病毒具有很强的抗病毒活性[44]。前10个查询结果实际上都是针对各种hhv1蛋白质与诸如喷昔洛韦(penciclovir)和阿昔洛韦(acyclovir)之类的化合物,这些化合物为靶向1型单纯型疱疹病毒的已知抗病毒药物[44]、[45]。
[0105]
这些结果清楚地演示了cge能够使得具有sw序列相似性udf的知识图能够快速有效地找到可以再用于靶向不同疾病的潜在药物。cge的这些能力可以让研究人员以交互方式搜索潜在候选药物,并且应用主题专业知识来进一步细化查询结果,从而可能提高所重新靶向的药物的有效性。
[0106]
结论和未来工作
[0107]
本文中所描述的示例演示了cge、大规模并行语义图引擎和其他能够轻松摄取和搜索这种规模的图数据库的类似引擎,并且使得研究人员能够在几秒钟内跨数据集执行复杂查询。此外,cge的性能使研究人员能够以交互方式查询包含1550亿个三元组的给定数据库,以搜索图的节点之间可能无法找到的潜在隐藏连接。
[0108]
本论文还讨论了最近对cge所做的改变,以启用用户定义函数,这些用户定义函数允许用户将领域特定专业知识应用于诸如filter、group和order之类的操作。我们的工作重点是利用史密斯-沃特曼序列相似性算法的开源实施方式作为查询内的udf,以基于与给
定参考序列的相似性来对已知化合物所靶向的蛋白质应用排名。使用spike_sars2蛋白作为参考,我们展示了如何编写查询,该查询使得cge能够在几秒钟内找到可以再用于covid-19的潜在药物。附加地,我们演示了cge的这些能力不是特定针对covid-19的,并且它能够很容易地用于寻找潜在药物,以再用于其他已知感兴趣疾病或新感兴趣疾病。
[0109]
使用我们两个主要感兴趣查询,我们演示了查询内史密斯-沃特曼函数的强大扩展,并且示出了了查询本身的良好整体扩展。扩展测试示出了未来需要关注的一些领域以改进扩展。首先,group运算符存在由于生成过多重复而导致的性能瓶颈,并且随着核计数的增加,查询的扩展性不如预期。然而,即使有这些限制,针对复杂查询,我们也能够显示出良好的扩展性,从而进一步演示了cge的独特能力。
[0110]
最后,我们已经展示了包括大规模并行性、复杂查询性能、以及可以摄取的数据规模在内的cge的独特能力与蛋白质序列相似性结合可以使得研究人员能够快速有效地再利用现有药物以靶向新疾病。
[0111]
图xyz描绘了示例计算机系统xyz00的框图,其中可以实现本文中所描述的各个实施例。计算机系统xyz00包括用于通信信息的总线xyz02或其他通信机制、与总线xyz02耦合以处理信息的一个或多个硬件处理器xyz04。硬件处理器(多个)xyz04可以为例如一个或多个通用微处理器。
[0112]
计算机系统xyz00还包括主存储器xyz06,诸如随机存取存储器(ram)、高速缓存和/或其他动态存储设备,该主存储器xyz06被耦合到总线xyz02,用于存储信息以及将由处理器xyz04执行的指令。主存储器xyz06还可以被用于在执行将由处理器xyz04执行的指令期间,存储临时变量或其他中间信息。这些指令当被存储在处理器xyz04可访问的存储介质中时,将计算机系统xyz00呈现为被定制为执行指令中指定的操作的专用机器。
[0113]
计算机系统xyz00还包括只读存储器(rom)xyz08或其他静态存储设备,该其他静态存储设备被耦合到总线xyz02,用于存储处理器xyz04的静态信息和指令。提供存储设备xyz10,诸如磁盘、光盘或usb拇指驱动器(闪存)等,并且将其耦合到总线xyz02,用于存储信息和指令。
[0114]
计算机系统xyz00可以经由总线xyz02被耦合到显示器xyz12,诸如液晶显示器(lcd)(或触摸屏),用于向计算机用户显示信息。包括字母数字键和其他键的输入设备xyz14被耦合到总线xyz02,用于将信息和命令选择通信到处理器xyz04。另一类型的用户输入设备为光标控制xyz16(诸如鼠标、轨迹球或光标方向键),用于将方向信息和命令选择通信到处理器xyz04以及用于控制光标在显示器xyz12上的移动。在一些实施例中,可以经由在没有光标的触摸屏上接收触摸来实现与光标控制相同的方向信息和命令选择。
[0115]
计算系统xyz00可以包括用于实现gui的用户接口模块,该gui可以作为由计算设备(多个)执行的可执行软件代码存储在大容量存储设备中。通过示例,该模块和其他模块可以包括部件,诸如软件部件、面向对象的软件部件、类部件和任务部件、过程、函数、属性、过程、子例程、程序代码段、驱动程序、固件、微码、电路、数据、数据库、数据结构、表、数组、以及变量。
[0116]
一般而言,如本文中所使用的,单词“部件”、“引擎”、“系统”、“数据库”、“数据存储”等可以是指以硬件或固件体现的逻辑,或是指以编程语言(诸如java、c或c )编写的可能具有入口点和出口点的软件指令集合。软件部件可以被编译并且链接成可执行程序、被
安装在动态链接库中、或可以以解释性编程语言(诸如例如,basic、perl或python)编写。应当理解,软件部件可以从其他部件或从它们自身调用,和/或可以响应于检测到的事件或中断而被调用。被配置为用于在计算设备上执行的软件部件可以在计算机可读介质上(诸如光盘、数字视频盘、闪存驱动器、磁盘或任何其他有形介质)提供或作为数字下载(并且最初可能以需要在执行前进行安装、解压缩或解密的压缩或可安装格式被存储)提供。这种软件代码可以部分或全部被存储在执行计算设备的存储器设备上,以供计算设备执行。软件指令可以被嵌入在诸如eprom之类的固件中。还应当理解,硬件部件可以由所连接的逻辑单元(诸如门和触发器)组成,和/或可以由可编程单元(诸如可编程门阵列或处理器)组成。
[0117]
计算机系统xyz00可以使用定制硬连线逻辑、一个或多个asic或fpga、固件和/或程序逻辑来实现本文中所描述的技术,该程序逻辑与计算机系统相结合使得计算机系统xyz00成为专用机器或将其编程为专用机器。根据一个实施例,本文中的技术由计算机系统xyz00响应于处理器(多个)xyz04执行被包含在主存储器xyz06中的一个或多个指令的一个或多个序列而被执行。这样的指令可以从另一存储介质(诸如存储设备xyz10)读入主存储器xyz06。执行被包含在主存储器xyz06中的指令序列使处理器(多个)xyz04执行本文中所描述的处理步骤。在备选实施例中,硬连线电路系统可以代替软件指令或与软件指令组合使用。
[0118]
本文中所使用的术语“非暂态介质”和类似术语是指存储使机器以特定方式操作的数据和/或指令的任何介质。这种非暂态介质可以包括非易失性介质和/或易失性介质。非易失性介质包括例如光盘或磁盘,诸如存储设备xyz10。易失性介质包括动态存储器,诸如例如主存储器xyz06。非暂态介质的常见形式包括例如软盘、柔性盘、硬盘、固态驱动器、磁带或任何其他磁性数据存储介质、cd-rom、任何其他光学数据存储介质、带有孔图案的任何物理介质、ram、prom和eprom、flash-eprom、nvram、任何其他存储器芯片或盒带、以及其联网版本。
[0119]
非暂态介质与传输介质不同但可以与传输介质结合使用。传输介质参与非暂态介质之间的信息传输。例如,传输介质包括同轴电缆、铜线、以及光纤,该光纤包括电线,该电线包括总线xyz02。传输介质还可以采用声波或光波的形式,诸如在无线电波和红外数据通信期间生成的声波或光波。
[0120]
计算机系统xyz00还包括被耦合到总线xyz02的通信接口xyz18。通信接口xyz18提供双向数据通信,该双向数据被通信耦合到一个或多个网络链路,该一个或多个网络链路被连接到一个或多个本地网络。例如,通信接口xyz18可以是集成服务数字网络(isdn)卡、电缆调制解调器、卫星调制解调器、或调制解调器,以提供与对应类型的电话线的数据通信连接。作为另一示例,通信接口xyz18可以为局域网(lan)卡以提供与兼容lan(或与wan通信的wan部件)的数据通信连接。还可以实现无线链接。在任何这样的实施方式中,通信接口xyz18发送以及接收携带表示各种类型信息的数字数据流的电学信号、电磁信号或光学信号。
[0121]
网络链路通常通过一个或多个网络向其他数据设备提供数据通信。例如,网络链路可以通过本地网络提供与主机计算机或由互联网服务提供商(isp)所操作的数据设备的连接。isp进而通过现在通常称为“互联网”的全球分组数据通信网络提供数据通信服务。本地网络和互联网两者都使用携带数字数据流的电学信号、电磁信号或光学信号。携带进出
计算机系统xyz00的数字数据的通过各种网络的信号和网络链路上的信号以及通过通信接口xyz18的信号为传输介质的示例形式。
[0122]
计算机系统xyz00可以通过一个或多个网络、网络链路和通信接口xyz18发送并接收包括程序代码的数据。在互联网示例中,服务器可以通过互联网、isp、本地网络和通信接口xyz18传输应用程序的请求代码。
[0123]
所接收的代码可以在其被接收到时由处理器xyz04执行,和/或被存储在存储设备xyz10或其他非易失性存储器中以供稍后执行。
[0124]
前述章节中描述的过程、方法和算法中的每个过程、方法和算法都可以体现在由一个或多个计算机系统或包括计算机硬件的计算机处理器执行的代码部件中,并且由这些代码部件完全或部分自动化。一个或多个计算机系统或计算机处理器还可以操作以支持“云计算”环境中的相关操作的执行或作为“软件即服务”(saas)操作。过程和算法可以部分或全部在专用电路系统中实现。上文所描述的各种特征和过程可以彼此独立使用,或可以以各种方式组合。不同组合和子组合旨在落入本公开的范围内,并且在一些实施方式中可以省略某些方法或过程块。本文中所描述的方法和过程也不限于任何特定顺序,并且与其相关的块或状态可以以其他适当顺序执行,或可以并行执行,或以某种其他方式执行。块或状态可以被添加到所公开的示例实施例或从所公开的示例实施例中移除。操作或过程中的某些操作或过程的执行可能分布在计算机系统或计算机处理器之间,不仅驻留在单个机器内,而且还跨多个机器部署。
[0125]
如本文中所使用的,可以利用任何形式的硬件、软件或其组合来实现电路。例如,一个或多个处理器、控制器、asic、pla、pal、cpld、fpga、逻辑部件、软件例程或其他机制可以被实现为组成电路。在实施方式中,本文中所描述的各种电路可以被实现为分立电路,或所描述的功能和特征可以在一个或多个电路之间部分或全部共享。尽管各种功能特征或功能元件可以作为单独电路进行个别描述或要求保护,但这些特征和功能可以在一个或多个公共电路之间共享,并且这种描述不应要求或暗示需要单独电路来实现这些特征或功能。在电路全部或部分使用软件实现的情况下,这种软件可以实现为与能够执行关于其所描述的功能的计算或处理系统(诸如计算机系统xyz00)一起操作。
[0126]
如本文中所使用的,术语“或”可以被解释为包括意义或排他意义。此外,单数形式的资源、操作或结构的描述不应被理解为排除复数形式。除非另有特别说明或在所使用的上下文中以其他方式理解,否则诸如“可以(can)”、“可能(could)”、“可能(might)”或“可能(may)”之类的条件语言通常旨在传达某些实施例包括,而其他实施例不包括,某些特征、元件和/或步骤。
[0127]
除非另有明确说明,本文档中所使用的术语和短语及其变型应当被解释为开放式而非限制性的。诸如“传统(conventional)”、“传统(traditional)”、“正常(normal)”、“标准(standard)”、“已知(known)”等之类的形容词以及具有类似含义的术语不应被解释为将所描述的项目局限于给定时间段或仅局限于自给定时间起可用的项目,相反,应理解为涵盖可以在现在或将来任何时间可用或已知的传统、传统、正常或标准技术。在一些实例中,诸如“一个或多个”、“至少”、“但不限于”或其他类似短语之类的拓宽单词和短语的存在不应被理解为意味着在可能没有这样的拓宽短语的实例中,意图或需要更窄的情况。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。
