1.本发明整体涉及表示由测序机器产生的基因组测序数据的方法,并且更具体地涉及用于压缩这样的基因组测序数据的计算机实现的方法。本公开提供了一种基于参考的压缩方法,其允许快速压缩和解压缩,同时不引起信息丢失,并且具有高压缩比。
背景技术:
2.下一代测序机器现在以可承受的价格产生海量的测序数据。最新的系统在36小时的单次运行中产生超过60亿个150个核苷酸长的序列,足以对20个完整的人类基因组进行测序。这为遗传疾病的诊断和个性化医疗的发展开辟了许多新的视角,旨在适应基于人基因组特异性的治疗。
3.然而,这也带来了新的挑战,特别是与存储海量数据相关的成本。用于原始(未比对)序列数据的最常用的文件格式为fastq格式,其保存序列数据(a、c、t、g核苷酸的字符串,也称为读段)、质量值(测序平台对每个核苷酸造成测序错误的概率)和序列名称。这是普通ascii文本文件,通常用通用文本压缩方案lz(lempel-ziv方案,在gzip软件中实现)来压缩。然而,使用此类压缩方法带来了几个问题:
4.–
由于数据的冗余未被完全利用而导致的低压缩比
5.–
压缩和解压缩缓慢
6.还存在专用于fastq编码的压缩方法,分为基于参考的方法或基于非参考的方法。然而,它们中没有一个完全令人满意,原因是:a)基于参考的方法具有良好的压缩比,但速度缓慢,b)基于非参考的方法速度较快,但压缩比较低。这种基于非参考的方法的一个示例由软件spring提供,该软件是用于fastq文件的无参考压缩器(万维网地址:github.com/shubhamchandak94/spring)。然而,由软件spring提供的压缩方法的压缩比低。
7.在基于参考的压缩方法中,已经提出了一些使用序列比对并且旨在具有更快的速度且具有良好压缩比的方法。然而,此类方法遭遇了几个问题,值得注意的一个主要问题是,它们并非完全无损。这种已知的基于参考的压缩方法例如描述于专利文献wo 2018/068829 a1中。在该所描述的方法中,在与一个或多个参考序列比对之后,根据匹配准确度将核苷酸序列分类(从而创建比对读段的类别),然后使用不同的源模型和熵编码器针对其中数据被分区的每一层将这些核苷酸序列编码为多层语法元素。因此,数据的类别被单独编码,并且被构造在语法元素的不同层中,每个层均包含描述符,这些描述符单义地表示所述层的经分类和比对的读段。该方法旨在获得具有简化的信息熵的不同信息源,从而允许提高压缩性能以及对特定类别压缩数据的选择性访问。然而,这种压缩方法以与在读段比对步骤结束时获得的顺序不同的顺序对读段进行重新排序(即,根据其类别对读段进行重新排序)。于是在压缩过程(尤其是初始序列排序)中丢失了一些信息。因此,一些分析结果的再现性可能受到影响,因为一些下游分析软件可能依赖于读段的顺序。此外,以与读段的初始顺序不同的顺序解压缩数据使得检查未压缩文件是否与初始文件相同更加困难。此外,这种压缩方法速度相对缓慢,尤其是在与最先进的基于非参考的压缩方法相比时。
技术实现要素:
8.本公开通过提供用于压缩基因组序列数据的系统、方法、计算机程序和硬件电路系统来解决现有技术解决方案的问题。在一个方面,公开了用于压缩由测序机器产生的基因组序列数据的计算机实现的方法,所述基因组序列数据包括已与参考序列比对的核苷酸或碱基的序列的读段,从而产生比对读段,所述比对读段作为读段列表存储在初始文件中,所述方法包括:
[0009]-针对每个比对读段,确定所述读段与所述参考序列是完全映射的还是不完全映射的,或者所述读段是否与所述参考序列是未映射的,
[0010]-根据所述确定对所述读段进行编码,其中根据第一编码过程对被确定为完全映射的所述读段进行编码,并且根据第二编码过程对被确定为未映射的所述读段进行编码,
[0011]-其中所述确定步骤包括针对每个不完全映射的读段,将所述读段与所述参考序列之间的失配的数目与阈值进行比较,
[0012]-其中,在所述编码步骤中,根据所述第二编码过程或第三编码过程对被确定为不完全映射的所述读段进行编码,当所述失配数目大于所述阈值时,根据所述第二编码过程对所述不完全映射的读段进行编码,当所述失配数目低于所述阈值时,根据所述第三编码过程对所述不完全映射的读段进行编码,
[0013]-其中,在所述第二编码过程中,所述读段的每个核苷酸或碱基被单独编码,
[0014]-其中所述第一编码过程和所述第三编码过程包括不同的描述符集合,每个描述符集合单义地表示与对应的编码过程相关联的读段,所述第一编码过程和所述第三编码过程中的每一者是简化的信息源熵编码过程。
[0015]
本公开克服了现有压缩方法的缺点,方式为:允许快速压缩和解压缩,同时不引起信息丢失,并且提供高压缩比。更具体地,本公开的重点是以最紧凑的方式对最频繁出现的情况进行编码,即使这意味着对于罕见的最不频繁出现的情况采用降级的编码模式。这导致压缩性能的巨大提高。此外,由于本公开所使用的基因组信息表示格式,由本文所述的方法执行的压缩更快。最后但并非最不重要的是,本公开照此保持读段的初始顺序,并且不根据读段的类别对读段进行重新排序。因此,在该过程期间没有信息丢失,这使得能够更容易地进行下游分析,以及在解压缩步骤之后进行有效的一致性检查。
[0016]
根据附图和以下具体实施方式,本公开的这些和其他特征和优点将变得更加明显。此外,尽管阈值在本文中可以被称为被超过或未被超过,但是应当理解,可以概念性地采用此类阈值,使得确定此类阈值是否被满足、符合或以其他方式被检测到,而不管用于实现那些阈值评估的数目或值是使用正值还是负值来描述。
[0017]
根据本公开的一个创新方面,公开了一种用于压缩基因组序列数据的方法。在一个方面,所述方法可以包括经由通过一个或多个计算机执行软件指令来进行一个或多个操作,其中所述操作包括:由所述一个或多个计算机获得读段记录;由所述一个或多个计算机确定所述读段记录是对应于被完全映射到参考序列的读段还是被不完全映射到所述参考序列的读段;基于由所述一个或多个计算机确定所述读段记录对应于被不完全映射到所述参考序列的读段,由所述一个或多个计算机确定所述不完全映射的读段的失配数目是否满足预先确定的失配阈值数目,以及基于确定所述失配数目满足所述预先确定的失配阈值数目,由所述一个或多个计算机将所述不完全映射的读段的每个失配编码为具有1个字节大
小的记录。
[0018]
其他方面包括执行如本文所公开的方法的动作的对应系统、装置和计算机程序,如由在计算机可读存储设备上编码的指令所定义的。
[0019]
这些和其他版本可任选地包括以下特征中的一个或多个特征。例如,在一些具体实施中,由所述一个或多个计算机确定所述不完全映射的读段的失配数目是否满足预先确定的失配阈值数目可以包括由所述一个或多个计算机确定所述不完全映射的读段的所述失配数目是否大于所述预先确定的失配阈值数目。
[0020]
在一些具体实施中,每个读段记录可以包括:指示比对读段相对于所述参考序列的绝对起始位置的数据;指示所述读段的长度的数据;指示所述读段是完全映射的还是不完全映射的数据;指示在所述读段中识别的失配的数目的数据;以及指示所述读段中的所述可能失配中的每一者的相对位置的数据。
[0021]
在一些具体实施中,将所述不完全映射的读段的每个失配编码为具有1个字节大小的记录包括:针对每个特定失配,由所述一个或多个计算机编码所述字节的前两位以包括表示存在于所述读段中的替代核苷酸或碱基而不是所述参考序列中的相应参考核苷酸或碱基的数据;以及由所述一个或多个计算机编码所述字节的剩余六位以包括表示所述参考序列中的所述失配的位置的数据,所述位置被计算为相对于所述读段的前一个失配的偏移。
[0022]
在一些具体实施中,所述方法可以还包括:由一个或多个计算机确定所述偏移是否大于最大可编码值;以及基于确定所述偏移大于所述最大编码值,由一个或多个计算机在所述特定失配与所述前一个失配之间插入至少一个假失配。
[0023]
在一些具体实施中,所述方法可以还包括:基于确定所述失配数目不满足所述预先确定的失配阈值数目,由一个或多个计算机使用简化的信息熵编码过程将对应于所述失配中的每一者的位置的所述参考序列的位置列表编码到所述参考序列中。
[0024]
在一些具体实施中,所述方法可以还包括:基于确定所述读段记录对应于被完全映射到所述参考序列的读段,由一个或多个计算机使用简化的信息熵编码来编码所述读段记录的至少一部分。
[0025]
在一些具体实施中,所述一个或多个计算机可以包括一个或多个硬件处理器。
[0026]
在一些实施方案中,所述一个或多个硬件处理器可以包括一个或多个现场可编程门阵列(fpga)。
[0027]
在一些具体实施中,所述用于压缩基因组序列数据的方法可以由一个或多个硬件处理器执行。在此类具体实施中,所述硬件处理器可以包括被配置为执行一个或多个操作的硬件处理电路系统。在一个方面,所述操作可以包括:由所述硬件处理电路系统获得读段记录;由所述硬件处理电路系统确定所述读段记录是对应于被完全映射到参考序列的读段还是被不完全映射到所述参考序列的读段;基于由所述硬件处理电路系统确定所述读段记录对应于被不完全映射到所述参考序列的读段,由所述一个或多个计算机确定所述不完全映射的读段的失配数目是否满足预先确定的失配阈值数目,以及基于确定所述失配数目满足所述预先确定的失配阈值数目,由所述硬件处理电路系统将所述不完全映射的读段的每个失配编码为具有1个字节大小的记录。
[0028]
在一些具体实施中,每个读段记录可以包括:指示所述比对读段相对于所述参考
序列的绝对起始位置的数据;指示所述读段的长度的数据;指示所述读段是完全映射的还是不完全映射的数据;指示在所述读段中识别的失配的数目的数据;以及指示所述读段中的所述可能失配的相对位置的数据。
[0029]
在一些具体实施中,由所述硬件处理电路系统确定所述不完全映射的读段的失配数目是否满足预先确定的失配阈值数目可以包括由所述硬件处理电路系统确定所述不完全映射的读段的所述失配数目是否大于所述预先确定的失配阈值数目。
[0030]
在一些具体实施中,将所述不完全映射的读段的每个失配编码为具有1个字节大小的记录可以包括:针对每个特定失配,由所述硬件处理电路系统编码所述字节的前两位以包括表示存在于所述读段中的替代核苷酸或碱基而不是所述参考序列中的相应参考核苷酸或碱基的数据;以及由所述硬件处理电路系统编码所述字节的剩余六位以包括表示所述参考序列中的所述失配的位置的数据,所述位置被计算为相对于所述读段的前一个失配的偏移。
[0031]
在一些具体实施中,所述硬件处理电路系统被进一步配置为执行以下操作,所述操作包括:由所述硬件处理电路系统确定所述偏移是否大于最大可编码值;以及基于确定所述偏移大于所述最大编码值,由所述硬件处理电路系统在所述特定失配与所述前一个失配之间插入至少一个假失配。
[0032]
在一些具体实施中,所述硬件处理电路系统被进一步配置为执行以下操作,所述操作包括:基于确定所述失配数目不满足所述预先确定的失配阈值数目,由所述硬件处理电路系统使用简化的信息熵编码过程将对应于所述失配中的每一者的位置的所述参考序列的位置列表编码到所述参考序列中。
[0033]
在一些具体实施中,所述硬件处理电路系统被进一步配置为执行以下操作,所述操作包括:基于确定所述读段记录对应于被完全映射到所述参考序列的读段,由所述硬件处理电路系统使用简化的信息熵编码来编码所述读段记录的至少一部分。
[0034]
在一些具体实施中,该硬件处理电路系统包括一个或多个现场可编程门阵列(fpga)。
[0035]
根据本公开的另一个创新方面,公开了一种用于压缩基因组序列数据的方法。在一个方面,所述方法可以包括以下操作:由所述一个或多个处理器访问存储装置,该存储装置以保留由映射和比对模块产生的多个读段记录的序列排序的方式存储所述多个读段记录,针对所述多个读段记录中的每个特定读段记录:由所述一个或多个处理器获得所述特定读段记录;由所述一个或多个处理器确定所述特定读段记录是对应于被完全映射到参考序列的读段还是被不完全映射到所述参考序列的读段;基于由所述一个或多个处理器确定所述特定读段记录对应于被不完全映射到所述参考序列的读段,由所述一个或多个处理器确定所述不完全映射的读段的失配数目是否满足预先确定的失配阈值数目;基于确定所述失配数目满足所述预先确定的失配阈值数目,由所述一个或多个处理器将所述不完全映射的读段的每个失配编码为具有预先确定的压缩记录大小的压缩记录;以及由所述一个或多个处理器将所述压缩记录存储在所述存储装置中,同时维持所述读段记录的所述序列排序。
[0036]
其他方面包括执行如本文所公开的方法的动作的对应系统、装置和计算机程序,如由在计算机可读存储设备上编码的指令所定义的。
[0037]
这些和其他版本可任选地包括以下特征中的一个或多个特征。例如,在一些具体实施中,所述多个读段记录中的每个读段记录可以包括:指示所述比对读段相对于所述参考序列的绝对起始位置的数据;指示所述读段的长度的数据;指示所述读段是完全映射的还是不完全映射的数据;指示在所述读段中识别的失配的数目的数据;指示所述读段是否包括至少一个未确定的碱基n的数据;指示所述读段中的未确定的碱基n的数目的数据;指示所述读段是映射的还是未映射的数据;指示所述读段记录在由所述映射和比对模块输出的读段记录序列中的位置的数据;以及指示所述读段中的所述可能失配的相对位置的数据。
[0038]
在一些实施方案中,预先确定的压缩记录大小是一个字节。
[0039]
在一些具体实施中,将所述不完全映射的读段的每个失配编码为具有一个字节大小的压缩记录可以包括:针对每个特定失配,由一个或多个处理器编码所述字节的前两位以包括表示存在于所述读段中的替代核苷酸或碱基而不是所述参考序列中的相应参考核苷酸或碱基的数据;以及由一个或多个处理器编码所述字节的剩余六位以包括表示所述参考序列中的所述失配的位置的数据,所述位置被计算为相对于所述读段的前一个失配的偏移。
[0040]
在一些具体实施中,所述方法可以还包括:由一个或多个处理器确定所述偏移是否大于最大可编码值;以及基于确定所述偏移大于所述最大编码值,由一个或多个处理器在所述特定失配与所述前一个失配之间插入至少一个假失配。
[0041]
在一些具体实施中,所述方法可以还包括:基于确定所述失配数目不满足所述预先确定的失配阈值数目,由一个或多个处理器使用简化的信息熵编码过程将对应于所述失配中的每一者的位置的所述参考序列的位置列表编码到所述参考序列中。
[0042]
在一些具体实施中,所述方法可以还包括:基于确定所述读段记录对应于被完全映射到所述参考序列的读段,由所述一个或多个处理器使用简化的信息熵编码来编码所述读段记录的至少一部分。
[0043]
在一些具体实施中,由所述一个或多个计算机确定所述不完全映射的读段的失配数目是否满足预先确定的失配阈值数目可以包括由所述一个或多个处理器确定所述不完全映射的读段的所述失配数目是否大于所述参考阈值。
[0044]
根据本公开的另一个创新方面,公开了一种硬件处理器。在一个方面,该硬件处理器可以包括被配置为执行一个或多个操作的硬件处理电路系统。在一个方面,所述硬件处理电路系统被配置为执行的操作包括:由所述硬件处理电路系统访问存储装置,该存储装置以保留由映射和比对模块产生的多个读段记录的序列排序的方式存储所述多个读段记录,针对所述多个读段记录中的每个特定读段记录:由所述硬件处理电路系统获得所述特定读段记录;由所述硬件处理电路系统确定所述特定读段记录是对应于被完全映射到参考序列的读段还是被不完全映射到所述参考序列的读段;基于由所述硬件处理电路系统确定所述特定读段记录对应于被不完全映射到所述参考序列的读段,由所述硬件处理电路系统确定所述不完全映射的读段的失配数目是否满足预先确定的失配阈值数目;基于确定所述失配数目满足所述预先确定的失配阈值数目,由所述硬件处理电路系统将所述不完全映射的读段的每个失配编码为具有预先确定的压缩记录大小的压缩记录;以及由所述硬件处理电路系统将所述压缩记录存储在所述存储装置中,同时维持所述读段记录的所述序列排
序。
[0045]
这些和其他版本可任选地包括以下特征中的一个或多个特征。例如,在一些具体实施中,可以由所述硬件处理电路系统访问的所述多个读段记录中的每个读段记录可以包括:指示所述比对读段相对于所述参考序列的绝对起始位置的数据;指示所述读段的长度的数据;指示所述读段是完全映射的还是不完全映射的数据;指示在所述读段中识别的失配的数目的数据;指示所述读段是否包括至少一个未确定的碱基n的数据;指示所述读段中的未确定的碱基n的数目的数据;指示所述读段是映射的还是未映射的数据;指示所述读段记录在由所述映射和比对模块输出的读段记录序列中的位置的数据;以及指示所述读段中的所述可能失配的相对位置的数据。
[0046]
在一些具体实施中,由所述硬件处理电路系统生成的预先确定的压缩记录大小可以是一个字节。
[0047]
在一些具体实施中,将所述不完全映射的读段的每个失配编码为具有一个字节大小的压缩记录可以包括:针对每个特定失配,由所述硬件处理电路系统编码所述字节的前两位以包括表示存在于所述读段中的替代核苷酸或碱基而不是所述参考序列中的相应参考核苷酸或碱基的数据;以及由所述硬件处理电路系统编码所述字节的剩余六位以包括表示所述参考序列中的所述失配的位置的数据,所述位置被计算为相对于所述读段的前一个失配的偏移。
[0048]
在一些具体实施中,硬件处理器可以被进一步配置为包括硬件处理电路系统,该硬件处理电路系统被配置为执行以下操作,所述操作包括:由所述硬件处理电路系统确定所述偏移是否大于最大可编码值;以及基于确定所述偏移大于所述最大编码值,由所述硬件处理电路系统在所述特定失配与所述前一个失配之间插入至少一个假失配。
[0049]
在一些具体实施中,硬件处理器可以被进一步配置为包括硬件处理电路系统,该硬件处理电路系统被配置为执行以下操作,所述操作包括:基于确定所述失配数目不满足所述预先确定的失配阈值数目,由所述硬件处理电路系统使用简化的信息熵编码过程将对应于所述失配中的每一者的位置的所述参考序列的位置列表编码到所述参考序列中。
[0050]
在一些具体实施中,硬件处理器可以被进一步配置为包括硬件处理电路系统,该硬件处理电路系统被配置为执行以下操作,所述操作包括:基于确定所述读段记录对应于被完全映射到所述参考序列的读段,由所述硬件处理电路系统使用简化的信息熵编码来编码所述读段记录的至少一部分。
[0051]
在一些具体实施中,由所述硬件处理电路系统确定所述不完全映射的读段的失配数目是否满足预先确定的失配阈值数目包括:
[0052]
由所述硬件处理电路系统确定所述不完全映射的读段的所述失配数目是否大于所述预先确定的失配阈值数目。
[0053]
根据本公开的另一个创新方面,公开了一种用于压缩由测序机器产生的基因组序列数据的计算机实现的方法,所述基因组序列数据包括已与参考序列比对的核苷酸或碱基的序列的读段,从而产生比对读段,所述比对读段作为读段列表存储在初始文件中。在一个方面,所述方法可以包括针对每个比对读段的以下动作:确定所述读段与所述参考序列是完全映射的还是不完全映射的,或者所述读段是否与所述参考序列是未映射的;根据所述确定对所述读段进行编码,其中根据第一编码过程对被确定为完全映射的所述读段进行编
码,并且根据第二编码过程对被确定为未映射的所述读段进行编码,其中所述确定步骤包括针对每个不完全映射的读段,将所述读段与所述参考序列之间的失配的数目与阈值进行比较,其中,在所述编码步骤中,根据所述第二编码过程或第三编码过程对被确定为不完全映射的所述读段进行编码,当所述失配数目大于所述阈值时,根据所述第二编码过程对所述不完全映射的读段进行编码,并且当所述失配数目小于所述阈值时,根据所述第三编码过程对所述不完全映射的读段进行编码,其中,在所述第二编码过程中,所述读段的每个核苷酸或碱基被单独编码,其中所述第一编码过程和所述第三编码过程包括不同的描述符集合,每个描述符集合单义地表示与对应的编码过程相关联的所述读段,所述第一编码过程和所述第三编码过程中的每一者是简化的信息源熵编码过程。
[0054]
其他方面包括执行如本文所公开的方法的动作的对应系统、装置和计算机程序,如由在计算机可读存储设备上编码的指令所定义的。
[0055]
这些和其他版本可任选地包括以下特征中的一个或多个特征。例如,在一些具体实施中,所述确定步骤可以包括当确定读段与所述参考序列不完全映射并且具有低于所述阈值的失配数目时的进一步确定,所述进一步确定有关于所述读段与所述参考序列是全局映射的还是局部映射的,并且其中所述第三编码过程包括第一编码子过程和第二编码子过程,根据所述第一编码子过程对被确定为全局映射的所述读段进行编码,根据所述第二编码子过程对被确定为局部映射的所述读段进行编码,所述第一编码子过程和所述第二编码子过程包括不同的描述符集合,每个描述符集合单义地表示与对应的编码子过程相关联的所述读段。
[0056]
在一些具体实施中,所述第一编码子过程的所述描述符可以包括所述参考序列中的比对起始位置、读段长度和由符号替换表示的失配列表,并且其中所述第二编码子过程的所述描述符包括所述参考序列中的局部比对起始位置、读段长度、由符号替换表示的失配列表,以及不是所述比对的一部分的所述读段的剪切部分的长度。
[0057]
在一些具体实施中,在所述编码步骤中,将要根据所述第二编码子过程编码的读段的所述剪切部分串联,所述剪切部分的每个核苷酸或碱基被单独编码。
[0058]
在一些具体实施中,在所述编码步骤中,在1个字节上编码不完全映射的读段的每个失配。
[0059]
在一些具体实施中,在所述编码步骤中,不完全映射的读段的每个失配如下编码:所述字节的前两位用于编码所述读段中存在的替代核苷酸或碱基而不是所述参考序列中的相应参考核苷酸或碱基,并且所述字节的后六位用于编码所述参考序列中的所述失配的位置,所述位置被计算为相对于所述读段的前一个失配的偏移。
[0060]
在一些具体实施中,在所述编码步骤中,如果在给定失配与所述前一个失配之间计算出的所述偏移大于最大可编码值,则在所述两个失配之间插入至少一个假失配,直到所述失配中的每一者与所述至少一个假失配之间的每个偏移都低于所述最大可编码值,假失配被定义为这样的失配:对于所述失配,所述字节的位用于编码所述失配,或者用于编码与所述参考序列中的相应参考核苷酸或碱基相等的核苷酸或碱基。
[0061]
在一些具体实施中,初始步骤是将所述读段列表划分为读段块,其中每个块开始于包含对所述块进行解码所需的信息的标头,其中所述压缩方法逐块进行。
[0062]
在一些具体实施中,读段块具有相同的块大小。
[0063]
在一些实施方案中,最终步骤是提供包含编码读段列表的压缩文件,所述编码读段以与存储在初始文件中的读段的顺序相同的顺序存储在所述压缩文件中。
[0064]
在一些具体实施中,所述阈值等于31。
[0065]
在一些具体实施中,针对每个比对读段,提供了确定所述读段是否包含至少一个对应于测序机器不能检出任何碱基或核苷酸的情况的失配的步骤。
[0066]
在一些具体实施中,针对包含每个至少一个对应于测序机器不能检出任何碱基或核苷酸的情况的失配的读段,提供了确定此类失配的数目的步骤,以及将所述数目与参考阈值进行比较的步骤。
[0067]
在一些具体实施中,在所述编码步骤中,如果此类失配的数目大于所述参考阈值,则将要根据所述第二编码过程编码的读段的每个核苷酸或碱基以4位单独编码,并且如果此类失配的数目小于所述参考阈值,则将要根据所述第二编码过程编码的读段的每个核苷酸或碱基以2位单独编码,并且所述编码步骤还包括编码沿着所述参考序列的位置的列表,所述位置对应于所述参考序列中的此类失配的位置。
附图说明
[0068]
图1是示出本文所述压缩方法的一个示例的流程图。
[0069]
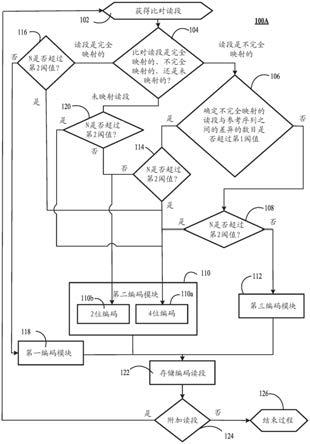
图1a是示出图1的压缩方法的更详细示例的流程图。
[0070]
图2是示出用于实施本文所述的一个或多个压缩方法的系统的一个示例的图示。
[0071]
图2a是示出用于实施本文所述的压缩方法的系统的另一个示例的图示。
[0072]
图2b是示出用于实施本文所述的压缩方法的系统的另一个示例的图示。
[0073]
图3是示出与参考序列全局映射的读段的第一示例的示意图。
[0074]
图4是示出在必须插入假失配的情况下与参考序列全局映射的读段的第二示例的示意图。
[0075]
图5是可以用于实施执行图1和图1a的压缩方法的系统的计算部件的一个示例的图示。
[0076]
图6描绘了示出本公开的实验结果的多个柱形图。
[0077]
图7描绘了示出本公开的附加实验结果的多个柱形图。
[0078]
图8描绘了示出本公开的附加实验结果的多个柱形图。
具体实施方式
[0079]
本公开所提及的基因组序列包括例如但不限于核苷酸序列、脱氧核糖核酸(dna)序列、核糖核酸(rna)序列和氨基酸序列。尽管本文关于核苷酸序列形式的基因组信息相当详细地描述了本公开,但是应当理解,如本领域技术人员所理解的,根据本发明的压缩方法也可以被实施用于其他基因组序列,尽管有一些变化。
[0080]
基因组测序信息通过测序机器以由来自限定词汇表的字母串表示的核苷酸(或更一般地,碱基)序列的形式产生。最小词汇表由五个符号表示:{a、c、g、t、n},表示存在于dna中的4种类型的核苷酸,即腺嘌呤、胞嘧啶、鸟嘌呤和胸腺嘧啶。在rna中,胸腺嘧啶被尿嘧啶(u)替代。n指示测序机器不能检出任何碱基,因此该位置的真实性质是不确定的。因此,出于本公开的目的,符号“n”是指未确定的碱基,并且读段中的“n”的数目是指该读段中的未
确定的碱基的数目。
[0081]
由测序机器产生的核苷酸序列可以被称为“读段”。序列读段的长度可以介于几十个核苷酸至几千个核苷酸之间。一些技术成对产生序列读段,其中所述对的第一读段来自一条dna链,并且所述对的第二读段来自另一条dna链。贯穿本公开,“参考序列”是由测序机器产生的核苷酸或碱基序列组成的读段能够与之比对/映射的任何序列。这种参考序列的一个示例实际上可以是参考基因组,即由科学家组装的作为物种的基因集合的代表性示例的序列。然而,参考序列还可以由合成序列组成,考虑到它们要被进一步处理,该合成序列被设想为仅仅改善读段的可压缩性。
[0082]
在一些情况下,测序机器可能在序列读段中引入错误,并且值得注意的是,可能使用错误符号(即,表示不同的核酸)来表示实际存在于经测序的样品中的核酸或碱基。这种类型的替换错误可能最终被映射和比对模块识别为“失配”。这是因为当读段与参考序列比对时,读段中的替换错误可能与参考序列的相应位置不匹配。然而,“失配”的含义并不限于此类情况。相反,“失配”可以是由测序装置检出的读段的以下任何碱基或核苷酸:当读段以阈值水平的准确度与参考序列比对时,其与参考序列的相应位置不匹配。此类失配可以包括候选变体、变体,或者比对读段与参考序列位置之间的其他差异。
[0083]
本公开涉及基于参考的压缩方法,其接收核苷酸或碱基序列的读段作为输入,此类读段先前已通过映射和比对模块与参考序列进行比对,从而产生比对读段。在一些具体实施中,先前比对的读段可以包括已使用软件映射和比对模块进行比对的读段,该模块执行所接收的读段到参考序列的映射和比对。例如,在一些具体实施中,软件映射器可以通过使用一个或多个处理器(诸如一个或多个中央处理单元(cpu)、一个或多个图形处理单元(gpu),或它们的任何组合)执行软件指令来进行所接收的读段的基于散列表的映射和比对。在其他具体实施中,先前比对的读段可以包括已使用硬件映射和比对模块进行比对的读段,该模块执行所接收的读段到参考序列的映射和比对。例如,在一些具体实施中,硬件映射和比对模块可以通过使用一个或多个硬件处理器(诸如一个或多个现场可编程门阵列(fpga))来进行基于散列表的映射和比对,所述硬件处理器具有硬连线数字逻辑电路,该数字逻辑电路被配置为进行所接收的读段的基于散列表的映射和比对。
[0084]
然后将比对读段存储为初始文件中的读段列表。比对读段以及一旦在初始文件中比对就将这些读段存储的方式对于本发明不是至关重要的,并且不是本公开的目的。然后将每个读段编码为参考序列上的位置以及与所述参考序列的差异的列表。然后可以通过如本文通过本公开所述配置的适当解压缩软件,从比对编码信息和参考序列重建每个读段。
[0085]
在一些具体实施中,本公开的压缩模块可以经由由一个或多个cpu或gpu执行软件指令、执行一个或多个硬件处理器的硬连线数字逻辑电路或两者的组合来实施,以处理并压缩比对读段。在压缩读段之前,可以将读段与参考序列比对,而不考虑在序列读段中引入的某些类型的错误,诸如插入错误或缺失错误。插入错误在于,在一个序列读段中插入一个或多个不涉及任何实际存在的核酸的额外符号。缺失错误在于,从一个序列读段中缺失一个或多个代表实际存在于测序样品中的核酸的符号。更准确地讲,在给定序列读段中存在插入错误或缺失错误的情况下,比对软件随后将所得到的错误核酸视为替换错误,也称为“失配”。比对软件配置的这种优先选择允许更快的后续编码,特别是在速度与压缩比之间提供更好的折衷。
[0086]
针对每个比对读段,该映射和比对模块可以生成并且提供读段记录。在一些具体实施中,每个读段记录可以直接作为输入从映射和比对模块提供给压缩模块。在其他具体实施中,由映射和比对模块生成的每个读段记录可以输出并且存储在存储器或其他存储装置中。在此类具体实施中,压缩模块随后可以访问所存储的读段记录并且压缩所存储的读段记录。
[0087]
由映射和比对模块生成、提供或存储的每个读段记录包括由映射和比对模块生成的描述由读段记录表示的读段的数据。此类读段记录可以至少包括以下信息:所述比对读段相对于所述参考序列的绝对起始位置、所述读段的长度、所述读段的比对类型(诸如所述读段是映射读段还是未映射读段)、在所述读段中识别的失配的数目、有关所述读段是完全映射的读段还是不完全映射的读段的指示、所述读段中的所述可能失配的相对位置,等等。
[0088]
尽管此处描述的示例指示读段记录中的数据和其中所包含的数据由映射和比对模块生成,但是本公开不限于此。相反,映射和比对模块与压缩模块之间的其他中间模块可以用于生成读段记录、其中所包含的数据。
[0089]
在一些具体实施中,由映射和比对模块提供或存储的读段记录能够以保留由映射和比对模块生成的读段记录的顺序排序的方式来提供或存储。在一些具体实施中,例如,每个读段记录还可以包含以读段记录的顺序排序指示读段记录放置的数据。指示读段记录放置的这种数据可以包括例如序列_id。在一些具体实施中,该序列_id可以例如是对于由映射和比对模块产生的第一读段记录以“1”开始的数字,该数字然后对于由映射和比对模块生成的每个后续读段记录递增。本公开的压缩模块然后可以访问这些读段记录并且以其当前的顺序压缩这些读段记录,而不需要将读段记录重新排序为读段记录的簇以便压缩。以保留由映射和比对模块生成的读段记录初始排序的方式压缩这些读段记录通过实现读段记录的无损压缩而提供了优于常规方法的优点,因为甚至保留了读段记录的顺序排序。此外,在压缩期间保留读段记录的次序还使得对读段记录压缩的验证更为容易。
[0090]
现在将参考图1描述本公开的压缩方法。在一些具体实施中,例如,该方法可以由图2中所示的设备20执行。设备20可以包括至少一个处理器22和至少一个可操作地耦接到至少一个处理器22以形成计算装置的存储器24。存储器24可以存储计算机程序代码或软件26,所述计算机程序代码或软件包含计算机可执行指令,这些计算机可执行指令在由处理器22执行时,使得处理器22执行压缩模块的操作,包括执行本文所述的一种或多种压缩方法的多个阶段。然而,本公开不必限于由设备20实施。
[0091]
例如,在一些具体实施中,本公开的压缩方法可以由图2a所示的设备20a实施。设备20a类似于设备20,原因是设备20a也包括处理器22和至少一个可操作地耦接到至少一个处理器22以形成计算装置的存储器24。设备20a的存储器24还存储计算机程序代码或软件26,所述计算机程序代码或软件包含计算机可执行指令,这些计算机可执行指令在由处理器22执行时,使得处理器22执行包括本文所述的一种或多种压缩方法的多个阶段的操作。然而,此外,设备20a还包括计算机程序代码或软件28,所述计算机程序代码或软件包含计算机可执行指令,这些计算机可执行指令在由处理器22执行时,使得处理器22执行操作以实现映射和比对模块的功能。其功能经由执行计算机软件指令来实现的映射和比对模块可以生成一个或多个比对读段29并且将所述比对读段29存储在存储器24中。然后,处理器22可以执行压缩模块的软件指令26以访问比对读段29中的一个或多个比对读段,并且使用本
文所述的一种或多种压缩方法的多个阶段来压缩所述一个或多个比对读段29。在一些具体实施中,设备20a可以是核酸测序装置。
[0092]
作为另一个示例,在一些具体实施中,本公开的压缩方法可以由图2b所示的设备20b实施。设备20b不同于设备20,原因是设备20b包括一个或多个硬件处理器22b,诸如一个或多个现场可编程门阵列(fpga)。在该示例中,一个或多个硬件处理器可以实现本文所述的一个或多个压缩方法的多个阶段的功能,以及一个或多个硬件处理器22b的硬件电路系统中的映射和比对模块的多个阶段的功能。例如,硬件处理器22b可以包括硬连线数字逻辑电路26b,其被配置为压缩模块以执行本文所述的一种或多种压缩方法的多个阶段。同样,硬件处理器22b可以包括硬连线数字逻辑电路28b,其被配置为执行映射和比对模块的操作,该映射和比对模块被配置为生成比对读段29b并且将比对读段29b存储在存储器24中。被配置为压缩模块以实现本文所述的一种或多种压缩方法的多个阶段的功能的硬连线数字逻辑电路26b可以从存储器24访问比对读段29b,并且使用本文所述的压缩方法来压缩比对读段29b。在一些具体实施中,设备20b可以是核酸测序装置。其中存储了比对读段记录作为读段列表的初始文件例如存储在设备20的存储器中。在一些具体实施中,该读段列表可以包括以保留由映射和比对模块产生的多个比对读段记录的序列排序的方式存储在该设备的存储器中的所述多个比对读段记录。所述比对读段记录的这种序列排序可以是与在映射和比对阶段结束时获得的顺序相同的顺序。
[0093]
在一些具体实施中,可以将比对读段的初始列表划分为读段块。例如,在一些具体实施中,可以将比对读段的列表划分为50000个读段的块。然而,50000个读段的块的该特定值不应被理解为限制本公开的范围,因为本公开的具体实施可以使用其他值以相同的方式来实现。
[0094]
在一些具体实施中,读段块可以具有相同的块大小。然而,在其他具体实施中,读段块可以具有不同的块大小。在任何情况下,每个读段块均能够开始于包含对该块进行解码所需的信息的标头,诸如该块的内容的以字节为单位的大小,以及/或者该块或其内容的标识符,以及/或者包含在该块中的读段的数目。这允许支持压缩文件的串联,以及流式传输能力(每个读段块均包含对该块的读段进行解码所需的所有信息)。此外,由于压缩方法然后可以逐块进行,所以这也允许对读段块进行多线程处理,从而允许并行化和处理时间的一些所得增益。如果给定块的所有读段具有相同的长度,则读段长度也被存储在标头中,否则在压缩方法期间明确地存储每个读段长度的列表。
[0095]
返回图1,该方法优选地包括初始阶段2,其中所述设备从设备20、20a或20b的存储器获得比对读段记录。在一些具体实施中,这可以包括:由所述设备访问存储器或其他存储装置,所述存储器或其他存储装置以保留由映射和比对模块产生的多个读段记录的序列排序的方式存储所述多个读段记录。例如,所述设备可以基于前一个读段记录的序列_id和存储在存储器中的一个或多个其他读段记录的序列_id来确定用于压缩的下一个读段记录。在一些具体实施中,序列_id可以是数值,该数值针对映射和比对模块所产生的每个后续读段记录而递增,并且压缩模块可以维护计数器,该计数器在图1的压缩过程的每次迭代时递增并且提供关于应当在阶段2处访问的下一个读段记录的指示。
[0096]
每个读段记录包含关于读段比对的类型的信息。关于读段的比对类型的信息可以包括描述读段相对于参考基因组的映射和比对水平的任何信息。在一些具体实施中,比对
类型可以包括完全比对、不完全比对,或者“未映射”读段比对。“完全比对”或“完全映射的读段”可以包括其中读段的每个核苷酸映射至参考基因组的一部分并且与之比对的读段。在一些具体实施中,“完全比对”或“完全映射的读段”可以具有零个失配和零个未确定的碱基“n”。在其他具体实施中,“完全比对”或“完全映射”读段可以具有零个失配,但是可能具有一个或多个未确定的碱基“n”。一般而言,“不完全比对”或“不完全映射的读段”的定义取决于在本文所述的压缩方法的特定具体实施中实现的“完全映射的读段”的含义。例如,如果使用其中完全映射的读段可以包含零个失配和零个未确定的碱基n的具体实施,则“不完全比对”或“不完全映射的读段”意指与参考序列的至少一部分匹配并且包括至少一个失配或至少一个n的任何读段。然而,如果例如使用其中完全映射的读段可以包含零个由一个或多个n引起的失配的具体实施,则“不完全比对”或“不完全映射的读段”意指具有至少一个除未公开的碱基n之外的失配的任何读段,而所述读段的至少一部分与参考序列的一部分匹配(根据不完全映射的读段的这种另外的定义,不完全映射的读段可以包含一个或多个n,条件是其还包含一个或多个其他失配)。因此,任何特定的系统或具体实施如何被配置为识别完全映射的读段将确定该具体实施的不完全映射的读段的含义。“未映射读段”可以包括尚未映射至参考基因组也尚未与之比对的读段。
[0097]
在一些具体实施中,每个读段记录包括描述该读段的属性的多个位标记。在一些具体实施中,所述多个位标记可以使用在读段记录开始处的一个或多个字段来存储。然而,在其他具体实施中,读段记录的其他字段可以用于存储所述多个位标记。所述多个位标记中的每个位标记可以使用多个值中的一个值来指示其对应的读段属性的值。在一些具体实施中,以下位标记可以用于指示读段记录的读段属性的值:
[0098]-第一位标记,其指示相对于参考序列的正向或反向取向,
[0099]-第二位标记,其指示完全比对或未完全比对,
[0100]-第三位标记,其指示读段是否包含至少一个n,
[0101]-第四位标记,其指示位置信息是以16位还是32位编码,
[0102]-第五位标记,其指示读段是映射的还是未映射的。
[0103]
针对多个读段中的每个读段执行以下阶段4至12。如果读段被分组成块,则针对每个读段块的每个读段执行阶段4至12。
[0104]
本公开的压缩方法可以包括下一个阶段4,其通过设备20、20a或20b针对每个比对读段确定所述读段与参考序列是完全映射的还是不完全映射的,或者所述读段是否与参考序列是未映射的。在一些具体实施中,设备20、20a、20b可以基于从映射和比对模块接收的信息来确定读段是完全映射的读段、不完全映射的读段,还是未映射的读段。该信息可以包括以下信息:诸如,由所获得的读段记录表示的读段是映射的还是未映射的;由读段记录表示的读段是完全映射的还是不完全映射的;总失配(诸如变体或测序错误、未确定的碱基)的数目的指示,或它们的任何组合。在一些具体实施中,该信息可以包含在所获得的读段记录本身之内。
[0105]
在一些具体实施中,设备20、20a或20b可以首先确定比对读段是映射的还是未映射的。如果设备20、20a或20b确定比对读段是未映射的,则所述设备可以在阶段6处继续执行图1的过程。替代性地,如果设备20、20a或20b确定读段是映射的,则设备20、20a或20b可以确定该读段是不完全映射的还是完全映射的。
[0106]
在一些具体实施中,设备20、20a或20b可以通过评估读段中的失配总数的数目来确定读段是不完全映射的还是完全映射的。在一些具体实施中,该失配总数可以由映射和比对模块提供并且从所获得的读段记录获得。在此类具体实施中,如果设备20、20a或20b确定失配总数等于零,则设备20、20a或20b可以在阶段4处确定所获得的比对读段是完全映射的读段,并且可以在阶段6处继续执行图1的过程。替代性地,如果在阶段4处,设备20、20a或20b确定失配总数大于零,则设备20、20a或20b可以在阶段4处确定与读段记录对应的读段是不完全映射的读段,并且设备20、20a或20b可以在阶段6处继续执行图1的过程。
[0107]
然而,需注意,上述具体实施仅仅是有关设备20、20a或20b可以如何确定比对读段记录是完全映射的、不完全映射的或未映射的示例。例如,在一些具体实施中,这种确定可以基于所获得的读段记录中所包含的信息并且在不将失配数目与零阈值进行比较的情况下进行。以举例的方式,读段记录可以在该读段记录的标头或其他部分中保持位标记,其指示该读段是映射的、未映射的、完全映射的还是不完全映射的,等等。在此类具体实施中,设备20、20a或20b可以基于所获得的读段记录的位标记并且在不将失配数目与零阈值进行比较的情况下,在阶段4处确定比对读段记录是映射的、未映射的、完全映射的还是不完全映射的。其他具体实施也落入本公开的范围之内。例如,可以想到,可以采用以下具体实施:其中可以访问存储在与所获得的读段记录不同的数据结构中的信息,并且将其视为读段位标记或其他数据,以指示特定读段记录是映射的、未映射的、完全映射的还是不完全映射的。
[0108]
在一些具体实施中,该确定步骤4可以还包括针对每个不完全映射的读段,将所述读段与所述参考序列之间的失配的数目与阈值进行比较4a。这可以包括失配总数,其中失配总数包括比对读段与参考测序之间的任何差异(包括变体、测序错误和未确定的碱基n)的总和。在一些具体实施中,失配数目可以由映射和比对模块提供并且从读段记录获得。
[0109]
在一些具体实施中,所述阈值可以是31。可以选择该特定值,以便提供用于以足够紧凑的方式存储失配数目的最佳可能折衷,如稍后关于阶段12将更好理解的。实际上,已经在统计学上观察到,在绝大多数情况下,不完全映射的读段具有少于31个失配。该选择背后的原理在于以最紧凑的方式对最频繁出现的情况进行编码,留下一些非常少的降级情况。然而,尽管在一些具体实施诸如短读段具体实施(其中读段的长度为大约150个核苷酸或碱基可能是有利的)中使用31个失配的阈值,但是本公开并不仅限于其中阈值等于31的那些具体实施。相反,对于其他具体实施,可能期望使用比31更高的阈值。例如,尽管各方面(例如,31个失配的阈值)可以旨在用于压缩表示由短读段定序器生成的读段的读段记录,但是预期本发明的基因组数据压缩方法可以用于其他具体实施中,诸如用于压缩由长读段定序器生成的读段记录。因此,在此类具体实施中,在读段由长度显著长于150个核苷酸或碱基的读段记录表示的情况下,阈值可以被设定为比31更高的值,以实现用于长读段系统的本公开的压缩方法的功能。
[0110]
如果读段被确定为以低于阈值的失配数目发生不完全映射,则确定阶段4还可以包括关于读段与参考序列是全局映射的还是局部映射的附加确定。“全局映射的读段”是不完全映射的读段,其整个序列(包括该读段的起点和终点)与参考序列是不完全映射的。“局部映射的读段”是含有与参考序列是不完全映射的核苷酸或碱基的区段的不完全映射的读段。因此,核苷酸或碱基的所述区段对应于初始读段的一部分。
[0111]
在一些具体实施中,所述压缩方法可以还包括阶段6,其针对每个比对读段,确定
所述读段是否包含至少一个未确定的碱基“n”,即,所述读段是否包含至少一个对应于测序机器不能检出任何碱基或核苷酸的情况的失配。对于包含至少一个“n”的每个读段,该方法然后包括阶段8,其确定此类未确定的碱基“n”的数目;和阶段10,其将所述未确定的碱基“n”的数目与参考阈值进行比较。在一些具体实施中,参考阈值可以等于31。然而,在其他具体实施中,可以将参考阈值设定为其他值。
[0112]
无论确定阶段4的结果如何,该方法均包括下一个阶段12,其根据所述确定对读段进行编码。更准确地讲,根据第一编码过程对被确定为与参考序列完全映射的读段进行编码,无论所述读段是不包含未确定的碱基“n”,还是具有数目低于参考阈值的未确定的碱基“n”。根据第二编码过程对被确定为未映射的读段或者被确定为完全映射但具有数目大于参考阈值的未确定的碱基“n”的读段进行编码,其中每个核苷酸或碱基被单独编码,而不管所述核苷酸或碱基是比对的还是未比对的。根据第二编码过程或第三编码过程对被确定为不完全映射的读段进行编码。更准确地讲,根据第二编码过程对被确定为以大于阈值的失配数目发生不完全映射的读段进行编码。如果读段被确定为以低于阈值的失配数目发生不完全映射,则如果所述读段不包含n或具有低于参考阈值的n数目,则根据第三编码过程对所述读段进行编码。如果不是这样,即,如果读段具有大于参考阈值的n数目,则根据第二编码过程对所述读段进行编码。
[0113]
不管给定读段是已被确定为完全映射的、不完全映射的还是未映射的,如果所述读段包含至少一个n但具有低于参考阈值的n数目,则编码阶段12包括编码沿着参考序列的位置的列表,所述位置对应于参考序列中的n的位置。然后将该位置列表存储在计算装置的存储器中,所述装置实施所述压缩方法。如果读段包含至少一个n但具有低于参考阈值的n数目,则将根据第二编码过程进行编码,并且该读段的每个核苷酸或碱基以2位单独编码。
[0114]
如果读段包含至少一个n但具有大于参考阈值的n数目,则在任何情况下都将根据第二编码过程对所述读段进行编码,并且该读段的每个核苷酸或碱基以4位单独编码。在这种情况下,编码阶段12不包括对参考序列中的n位置的列表进行编码和存储。实际上,然后根据第二编码过程以与读段的其他核苷酸或碱基极其相同的方式对每个n失配直接进行编码。
[0115]
第一编码过程和第三编码过程包括不同的描述符集合。每个描述符集合单义地表示与对应的编码过程相关联的读段,第一编码过程和第三编码过程中的每一者是简化的信息熵编码过程。更准确地讲,第三编码过程包括第一编码子过程和第二编码子过程。根据第一编码子过程对在阶段4期间被确定为全局映射的不完全映射的读段进行编码。根据第二编码子过程对在阶段4期间被确定为局部映射的不完全映射的读段进行编码。第一编码子过程和第二编码子过程包括不同的描述符集合,每个描述符集合单义地表示与对应的编码子过程相关联的读段。
[0116]
于是,针对每个读段编码并且使得能够在数据解压缩期间重建整个读段序列的比对信息取决于用于所述读段的对应的编码过程或子过程。
[0117]
例如,在一些具体实施中,用于第一编码过程的第一描述符集合可以包括:
[0118]
o完全映射的读段相对于参考序列的绝对起始位置(以16位或32位编码),以及
[0119]
o读段的长度(采用差分编码法相对于前一个读段的长度进行编码,其中可变长度码在2位至34位的范围内)。
[0120]
作为另一个示例,在一些具体实施中,用于第一编码子过程的第二描述符集合可以包括:
[0121]
o不完全映射的读段相对于参考序列的绝对起始位置(以16位或32位编码),
[0122]
o读段的长度(采用差分编码法相对于前一个读段的长度进行编码,其中可变长度码在2位至34位的范围内),以及
[0123]
o读段的失配列表。
[0124]
作为另一个示例,在一些具体实施中,用于第二编码子过程的第三描述符集合可以包括:
[0125]
o读段的不完全映射的部分相对于参考序列的绝对起始位置
–
也称为局部比对起始位置(以16位或32位编码),
[0126]
o读段的长度(采用差分编码法相对于前一个读段的长度进行编码,其中可变长度码在2位至34位的范围内),
[0127]
o读段的失配列表,以及
[0128]
o读段的不是比对的一部分的剪切部分的长度(对于每个剪切部分以8位编码)。
[0129]
优选地,在第一子过程和第二子过程中编码的失配列表可以包括标头。例如,在一些具体实施中,该标头可以使用位标记编码并且在一个字节上编码。在此类具体实施中,一字节标头的前五位可以用于编码读段中所包含的失配的数目。在其中阈值等于31的具体实施中,失配数目可以在介于0与31之间的范围内。一字节标头的一位可以用于编码不完全映射的读段是全局映射还是局部映射。一字节标头的另一位可以用于编码2位模式是否被激活用于第二编码过程。一字节标头的最后一位可以用于编码4位模式是否被激活用于第二编码过程。在一些具体实施中,针对在编码阶段12期间根据第二编码子过程编码的每个读段,将所述读段的剪切部分(即,不是局部比对的一部分的那些部分)串联,并且所述剪切部分的每个核苷酸或碱基被单独编码。在一些具体实施中,所述读段的这种剪切部分的每个核苷酸或碱基以2位单独编码。
[0130]
在一些具体实施中,在不完全映射的读段的失配列表中编码(即,根据第一编码子过程或第二编码子过程编码)的每个失配可以在1个字节上编码。更准确地讲,要根据第一编码子过程或第二编码子过程编码的不完全映射的读段的每个失配可以如下编码:
[0131]
o字节的前两位用于编码读段中存在的替代核苷酸或碱基,而不是参考序列中的相应参考核苷酸或碱基,
[0132]
o后六位用于编码参考序列中的失配的位置,所述位置被计算为相对于读段的前一个失配的偏移。该计算出的位置可以是失配的相对位置,绝对位置被编码的读段的第一失配除外。因此,该偏移的范围(以6位编码)可以为[0-63]。
[0133]
由完成图1的过程产生的编码记录或压缩记录可以存储在所述设备的存储器或其他存储装置中。在一些具体实施中,这种编码记录或压缩记录可以以维持所述读段记录的序列排序的方式存储在所述设备的存储器或其他存储装置中。这有助于确保比对读段记录的压缩是无损的,因为甚至保留了比对读段记录的初始序列排序。
[0134]
在阶段102处获得比对读段记录
[0135]
参考图1a的压缩方法100a更详细地描述图1的压缩方法。由设备20、20a或20b执行压缩方法100a能够开始于初始阶段102,该初始阶段包括获得比对读段记录(以下也称为“所获得的读段记录”或者“未映射读段”/“映射读段”/“完全映射的读段”/“不完全映射的读段”,基于在方法100a的执行期间所获得的读段记录的后续分类)。在一些具体实施中,该比对读段记录能够从多个比对读段记录获得,所述多个比对读段记录以使得由测序装置提供的它们的初始顺序被保留的方式存储。因此,映射和比对模块以及压缩模块的整个操作能够将读段记录以由测序装置提供的它们的初始顺序保持。在一些具体实施中,可以通过使用序列_id来存储比对读取记录以保留它们的初始顺序,该序列_id与每个比对读段记录一起存储并且随着由映射和比对模块产生的每个比对读段记录而递增。
[0136]
在阶段104处确定对应于比对读段记录的读段是完全映射的、不完全映射的还是未映射的
[0137]
本公开的压缩方法可以包括下一个阶段104,其通过设备20、20a或20b确定所获得的读段记录是对应于与于参考序列完全映射的读段、与参考序列不完全映射的读段,还是与参考序列未映射的读段。在一些具体实施中,设备20、20a、20b可以基于从映射和比对模块接收的信息来确定读段是完全映射的读段、不完全映射的读段,还是未映射的读段。该信息可以包括以下信息:诸如,由所获得的读段记录表示的读段是映射的还是未映射的;由读段记录表示的读段是完全映射的还是不完全映射的;总失配(诸如变体或测序错误、未确定的碱基)的数目的指示,或它们的任何组合。在一些具体实施中,该信息可以包含在读段记录本身之内。
[0138]
在一些具体实施中,设备20、20a或20b可以首先在阶段104处确定比对读段是映射的还是未映射的。如果设备20、20a或20b确定比对读段是未映射的,则所述设备可以在阶段120处继续执行图1a的过程100a。替代性地,如果设备20、20a或20b确定读段是映射的,则设备20、20a或20b可以在阶段104期间进一步确定该读段是不完全映射的还是完全映射的。
[0139]
在一些具体实施中,设备20、20a或20b可以在阶段104期间通过评估读段中的失配数目来确定读段是不完全映射的还是完全映射的。在一些具体实施中,失配的数目可以由映射和比对模块提供并且从读段记录获得。对于不同的具体实施,可以以不同的方式来记录失配数目。在一些具体实施中,阶段104处的失配数目可以不包括未确定的碱基n的数目。在其他具体实施中,在阶段104处确定的失配数目可以包括失配数目和未确定的碱基n的数目的总和。
[0140]
在图1a的该示例中,假设未确定的碱基n不是失配。因此,完全映射的读段可以包括0个失配和一个或多个未确定的碱基n。因此,在该具体实施中,不完全映射的读段将需要具有至少一个失配,并且可以具有或可以不具有任何未确定的碱基n。然而,在其他具体实施中,图1a的过程可以通过假设读段中存在n可能是失配来修改。在此类具体实施中,只有当读段被确定为具有0个失配和0个未确定的碱基n时,该读段才可以被确定为完全映射的读段,其中具有0个失配和一个或多个未确定的碱基n的读段被分类为不完全映射的读段。
[0141]
在第一具体实施中,在阶段104处,如果设备20、20a或20b确定失配总数等于零并且未确定的碱基n的总数为零或更多,则设备20、20a或20b可以在阶段4处确定所获得的比对读段是完全映射的读段并且可以在阶段116处继续执行图1a的过程100a。替代性地,在该第一具体实施中,如果在阶段104期间,设备20、20a或20b确定失配总数大于零并且未确定的碱基n的总数为零或更多,则设备20、20a或20b可以在阶段104期间确定与所获得的读段记录对应的读段是不完全映射的读段,并且设备20、20a或20b可以在阶段106处继续执行图
1a的过程100a。
[0142]
在第二具体实施和替代性具体实施中,在阶段104处,设备20、20a或20b将仅在失配总数等于零并且未确定的碱基n的总数为零的情况下才确定读段是完全映射的读段,并且在这样的情形下,设备20、20a或20b可以在阶段116处继续执行图1a的过程100a。替代性地,在该第二具体实施中,如果在阶段104期间,设备20、20a或20b确定失配总数大于零或者未确定的碱基n的总数大于零,则设备20、20a或20b可以在阶段104期间确定与所获得的读段记录对应的读段是不完全映射的读段,并且设备20、20a或20b可以在阶段106处继续执行图1a的过程100a。
[0143]
然而,需注意,上述具体实施仅仅是有关设备20、20a或20b可以如何在阶段104处确定对应于所获得的读段记录的读段是完全映射的、不完全映射的或未映射的示例。例如,在一些具体实施中,这种确定可以替代地基于所获得的读段记录中所包含的信息并且在不将失配数目与阈值进行比较、不将未确定的碱基n的数目与阈值进行比较,或不进行这两种比较的情况下进行。以举例的方式,读段记录可以在该读段记录的标头或其他部分中保持位标记,其指示该读段是映射的、未映射的、完全映射的还是不完全映射的,等等。在此类具体实施中,设备20、20a或20b可以基于读段记录的位标记并且在不将失配或未公开的碱基n的数目与阈值进行比较的情况下,在阶段4处确定比对读段记录是映射的、未映射的、完全映射的还是不完全映射的。其他具体实施也落入本公开的范围之内。例如,可以想到,可以采用以下具体实施:其中可以访问存储在与读段记录不同的数据结构中的信息,并且将其视为读段位标记或其他数据,以指示与特定读段记录对应的读段是映射的、未映射的、完全映射的还是不完全映射的。
[0144]
阶段104的“读段不完全映射”分支
[0145]
如果设备20、20a或20b在阶段104处确定与所获得的读段记录对应的读段是不完全映射的读段,则设备20、20a或20b可以在阶段106处确定所述不完全映射的读段与参考序列之间的差异的数目是否超过第一阈值。这可以包括失配总数,其中失配总数包括比对读段与参考序列之间的任何差异(包括变体、测序错误和未确定的碱基n)的总和。在其他具体实施中,阶段106处的差异数目可以仅包括失配数目而不将未确定的碱基n的数目考虑在内。在一些具体实施中,失配数目可以由映射和比对模块提供并且从读段记录获得。
[0146]
在一些具体实施中,第一阈值可以是31。可以选择该特定值,以便提供用于以足够紧凑的方式存储失配数目的最佳可能折衷,如稍后关于后续阶段将更好理解的。实际上,已经在统计学上观察到,在绝大多数情况下,不完全映射的读段具有少于31个失配。该选择背后的原理在于以最紧凑的方式对最频繁出现的情况进行编码,留下一些非常少的降级情况。然而,虽然使用第一阈值31可以实现特定的优点,但是本公开不仅限于其中第一阈值等于31的那些具体实施。相反,对于其他具体实施,可能期望使用比31更高的阈值。例如,尽管各方面(例如,31个失配的阈值)可以旨在用于压缩表示由短读段定序器生成的读段的读段记录,但是预期本发明的基因组数据压缩方法可以用于其他具体实施中,诸如用于压缩由长读段定序器生成的读段记录。因此,在此类具体实施中,在读段由长度显著长于150个核苷酸或碱基的读段记录表示的情况下,阈值可以被设定为比31更高的值,以实现用于长读段系统的本公开的压缩方法的功能。
[0147]
阶段106的“是”分支
[0148]
如果设备20、20a或20b在阶段106处确定不完全映射的读段与参考序列之间的差异的数目超过第一阈值,则该设备可以在阶段114处继续执行过程100a。在阶段114处,设备20、20a或20b可以确定不完全映射的读段中的未确定的碱基“n”的数目是否超过第二阈值。在一些具体实施中,第二阈值也可以等于31。然而,类似于第一阈值,本公开的第二阈值也不限于值31。相反,基于在该具体实施中公布的读段的长度,可以将任何数值(包括高于31的值)用于第二阈值。此外,不需要第一阈值和第二阈值使用相同的阈值。
[0149]
阶段114的“是”分支
[0150]
如果由设备20、20a或20b确定在不完全映射的读段中未公开的碱基“n”的数目超过第二阈值,则设备20、20a或20b可以确定将使用第二编码模块110对该不完全映射的读段进行编码,以使用第二编码过程来编码该不完全映射的读段。第二编码过程与上文关于图1所述的第二编码过程相同,其中每个核苷酸或碱基被单独编码,而不管所述核苷酸或碱基是比对的还是未比对的。在一些具体实施中,由于设备20、20a或20b在阶段114处确定未确定的碱基“n”的数目超过第二阈值,所以设备20、20a或20b可以使用第二编码模块来使用第二编码过程将读段编码为4位110a。一旦使用第二编码过程110使用4位编码110a对读段进行编码,设备20、20a或20b就可以在阶段122处将编码读段存储在存储器或其他存储装置中。设备20、20a或20b可以在阶段124处确定是否存在要压缩的另一个顺序排序的比对读段。并且,如果存在要压缩的另一个顺序排序的比对读段,则设备20、20a或20b可以执行阶段102的操作,以便获得下一个顺序排序的比对读段记录并且再次执行过程100a。然后,设备20、20a或20b继续迭代地执行过程100a,直到在阶段124处不再识别出顺序排序的比对读段记录。在这样的确定之后,过程100a可以终止于阶段126处。
[0151]
阶段114的“否”分支
[0152]
如果设备20、20a或20b在阶段114期间确定,在不完全映射的读段中未公开的碱基“n”的数目不超过第二阈值,则设备20、20a或20b可以确定将使用第二编码模块110对该不完全映射的读段进行编码,以使用第二编码过程来编码该不完全映射的读段。第二编码过程与上文关于图1所述的第二编码过程相同,其中每个核苷酸或碱基被单独编码,而不管所述核苷酸或碱基是比对的还是未比对的。在一些具体实施中,由于设备20、20a或20b在阶段114处确定未确定的碱基“n”的数目不超过第二阈值,所以设备20、20a或20b可以使用第二编码模块来使用第二编码过程将读段编码为2位110b。一旦使用第二编码过程110使用2位编码110b对读段进行编码,设备20、20a或20b就可以在阶段122处将编码读段存储在存储器或其他存储装置中。设备20、20a或20b可以在阶段124处确定是否存在要压缩的另一个顺序排序的比对读段。并且,如果存在要压缩的另一个顺序排序的比对读段,则设备20、20a或20b可以执行阶段102的操作,以便获得下一个顺序排序的比对读段记录并且再次执行过程100a。然后,设备20、20a或20b继续迭代地执行过程100a,直到在阶段124处不再识别出顺序排序的比对读段记录。在这样的确定之后,过程100a可以终止于阶段126处。
[0153]
阶段106的“否”分支
[0154]
如果设备20、20a或20b在阶段106处确定不完全映射的读段与参考序列之间的差异的数目不超过第一阈值,则设备20、20a或20b可以在阶段108处继续执行过程100a。在阶段108处,设备20、20a或20b可以确定不完全映射的读段是否包括多于第二阈值数目的未确定的碱基“n”。
[0155]
阶段108的“是”分支
[0156]
如果设备20、20a或20b在阶段108处确定,在不完全映射的读段中未公开的碱基“n”的数目超过第二阈值,则设备20、20a或20b可以在阶段108处确定将使用第二编码模块110对该不完全映射的读段进行编码,以使用第二编码过程来编码该不完全映射的读段。第二编码过程与上文关于图1所述的第二编码过程相同,其中每个核苷酸或碱基被单独编码,而不管所述核苷酸或碱基是比对的还是未比对的。在一些具体实施中,由于设备20、20a或20b在阶段108处确定未确定的碱基“n”的数目超过第二阈值,所以设备20、20a或20b可以使用第二编码模块来使用第二编码过程将读段编码为4位110a。一旦使用第二编码过程110使用4位编码110a对读段进行编码,设备20、20a或20b就可以在阶段122处将编码读段存储在存储器或其他存储装置中。设备20、20a或20b可以在阶段124处确定是否存在要压缩的另一个顺序排序的比对读段。并且,如果存在要压缩的另一个顺序排序的比对读段,则设备20、20a或20b可以执行阶段102的操作,以便获得下一个顺序排序的比对读段记录并且再次执行过程100a。然后,设备20、20a或20b继续迭代地执行过程100a,直到在阶段124处不再识别出顺序排序的比对读段记录。在这样的确定之后,过程100a可以终止于阶段126处。
[0157]
阶段108的“否”分支
[0158]
如果设备20、20a或20b在阶段108期间确定不完全映射的读段包括数目不满足第二阈值的未确定的碱基“n”,则设备20、20a或20b可以使用第三编码模块112来使用第三编码过程对该不完全映射的读段进行编码。图1a中的第三编码过程与上文参考图1的过程所述的第三编码过程相同,并且使用与上述第三编码过程相同的描述符。一旦使用第三编码模块112的第三编码过程对读段进行编码,设备20、20a或20b就可以在阶段122处将编码读段存储在存储器或其他存储装置中。设备20、20a或20b可以在阶段124处确定是否存在要压缩的另一个顺序排序的比对读段。并且,如果存在要压缩的另一个顺序排序的比对读段,则设备20、20a或20b可以执行阶段102的操作,以便获得下一个顺序排序的比对读段记录并且再次执行过程100a。然后,设备20、20a或20b继续迭代地执行过程100a,直到在阶段124处不再识别出顺序排序的比对读段记录。在这样的确定之后,过程100a可以终止于阶段126处。
[0159]
阶段104的“读段完全映射”分支
[0160]
替代性地,如果在阶段104处确定与所获得的读段记录对应的读段是完全映射的读段,则设备20、20a或20b可以在阶段116处确定完全映射的读段是否包括数目超过第二阈值的未确定的碱基“n”。在一些具体实施中,第二阈值也可以等于31。然而,类似于第一阈值,本公开不限于第二阈值31。相反,基于在该具体实施中公布的读段的长度,可以将任何数值(包括高于31的值)用于第二阈值。此外,不需要第一阈值和第二阈值使用相同的阈值。
[0161]
阶段116的“否”分支
[0162]
如果设备20、20a或20b在阶段116处确定完全映射的读段不包括多于第二阈值数目的未确定的碱基“n”,则设备20、20a或20b可以确定使用第一编码模块122使用第一编码过程对该读段进行编码。如果完全映射的读段不包括任何未确定的碱基“n”,则第一编码模块122执行与上文参考图1所述的第一编码过程相同的第一编码过程,并且使用与上文所述的第一编码过程相同的描述符。替代性地,如果完全映射的读段包括一个或多个“n”,则第一编码模块122使用上文参考图1所述的第一编码过程并且使用用于上述第一编码过程的相同描述符来编码该完全映射的读段。此外,在其中完全映射的读段包括一个或多个n(但
小于n的第二阈值数目)的特定具体实施中,第一编码模块118还可以在读段上存储未确定的碱基n的位置列表。
[0163]
一旦使用第一编码模块118对读段进行编码,设备20、20a或20b就可以在阶段122处将编码读段存储在存储器或其他存储装置中。设备20、20a或20b可以在阶段124处确定是否存在要压缩的另一个顺序排序的比对读段。并且,如果存在要压缩的另一个顺序排序的比对读段,则设备20、20a或20b可以执行阶段102的操作,以便获得下一个顺序排序的比对读段记录并且再次执行过程100a。然后,设备20、20a或20b继续迭代地执行过程100a,直到在阶段124处不再识别出顺序排序的比对读段记录。在这样的确定之后,过程100a可以终止于阶段126处。
[0164]
阶段116的“是”分支
[0165]
然而,如果设备在阶段116处确定读段确实包括多于第二阈值数目的未确定的碱基“n”,则设备20、20a或20b可以使用第二编码模块110来使用第二编码过程将读段编码为4位110a。图1a的第二编码过程与上文关于图1所述的第二编码过程相同,其中每个核苷酸或碱基被单独编码,而不管所述核苷酸或碱基是比对的还是未比对的。一旦使用第二编码过程110使用4位编码110a对读段进行编码,设备20、20a或20b就可以在阶段122处将编码读段存储在存储器或其他存储装置中。设备20、20a或20b可以在阶段124处确定是否存在要压缩的另一个顺序排序的比对读段。并且,如果存在要压缩的另一个顺序排序的比对读段,则设备20、20a或20b可以执行阶段102的操作,以便获得下一个顺序排序的比对读段记录并且再次执行过程100a。然后,设备20、20a或20b继续迭代地执行过程100a,直到在阶段124处不再识别出顺序排序的比对读段记录。在这样的确定之后,过程100a可以终止于阶段126处。
[0166]
阶段104的“未映射读段”分支
[0167]
替代性地,如果在阶段104处确定与所获得的读段记录对应的读段是未映射的读段,则设备20、20a或20b可以在阶段120处确定该未映射的读段是否包括数目超过第二阈值的未确定的碱基“n”。在一些具体实施中,第二阈值也可以等于31。然而,类似于第一阈值,本公开不限于第二阈值31。相反,基于在该具体实施中公布的读段的长度,可以将任何数值(包括高于31的值)用于第二阈值。此外,不需要第一阈值和第二阈值使用相同的阈值。
[0168]
阶段120的“否”分支
[0169]
如果设备20、20a或20b在阶段120处确定未映射的读段不包括多于第二阈值数目的未确定的碱基“n”,则设备20、20a或20b可以确定使用第二编码模块110使用第二编码过程对该读段进行编码。第二编码过程与上文关于图1所述的第二编码过程相同,其中每个核苷酸或碱基被单独编码,而不管所述核苷酸或碱基是比对的还是未比对的。在一些具体实施中,由于设备20、20a或20b在阶段120处确定未确定的碱基“n”的数目不超过第二阈值,所以设备20、20a或20b可以使用第二编码模块来使用第二编码过程将读段编码为2位110b。一旦使用第二编码过程110使用2位编码110b对读段进行编码,设备20、20a或20b就可以在阶段122处将编码读段存储在存储器或其他存储装置中。设备20、20a或20b可以在阶段124处确定是否存在要压缩的另一个顺序排序的比对读段。并且,如果存在要压缩的另一个顺序排序的比对读段,则设备20、20a或20b可以执行阶段102的操作,以便获得下一个顺序排序的比对读段记录并且再次执行过程100a。然后,设备20、20a或20b继续迭代地执行过程100a,直到在阶段124处不再识别出顺序排序的比对读段记录。在这样的确定之后,过程
100a可以终止于阶段126处。
[0170]
阶段120的“是”分支
[0171]
然而,如果设备在阶段120处确定未映射的读段确实包括多于第二阈值数目的未确定的碱基“n”,则设备20、20a或20b可以使用第二编码模块110来使用第二编码过程将读段编码为4位110a。图1a的第二编码过程与上文关于图1所述的第二编码过程相同,其中每个核苷酸或碱基被单独编码,而不管所述核苷酸或碱基是比对的还是未比对的。一旦使用第二编码过程110使用4位编码110a对读段进行编码,设备20、20a或20b就可以在阶段122处将编码读段存储在存储器或其他存储装置中。设备20、20a或20b可以在阶段124处确定是否存在要压缩的另一个顺序排序的比对读段。并且,如果存在要压缩的另一个顺序排序的比对读段,则设备20、20a或20b可以执行阶段102的操作,以便获得下一个顺序排序的比对读段记录并且再次执行过程100a。然后,设备20、20a或20b继续迭代地执行过程100a,直到在阶段124处不再识别出顺序排序的比对读段记录。在这样的确定之后,过程100a可以终止于阶段126处。
[0172]
图3提供了根据第一编码子过程对读段的失配进行编码的一个示例。该读段是不完全映射的读段,其与参考序列是全局映射的。该读段具有两个失配:
[0173]
o第一失配,其位于该读段中的第12位,其在于在该读段中用t核苷酸替换参考序列中的a核苷酸,以及
[0174]
o第二失配,其位于该读段中的第21位,其在于在该读段中用g核苷酸替换参考序列中的c核苷酸。
[0175]
该读段的失配列表然后被编码为:
[0176]
o《12,t》,值“12”对应于该读段中的第一失配的绝对位置,以及
[0177]
o《9,g》,值“9”对应于该读段中的第二失配的相对位置,即第二失配与第一失配之间的偏移。
[0178]
例如,《12,t》可以被转换为值“51”(在1个字节上编码),并且《9,g》可以被转换为值“38”(在1个字节上编码)。这种字节编码用以下方式获得:
[0179]
偏移位置x4 核苷酸值(其中a=0,c=1,g=2,t=3)
[0180]
优选地,对于要根据第一编码子过程或第二编码子过程进行编码的每个不完全映射的读段,如果在该读段的给定失配与前一个失配之间计算出的偏移大于最大可编码值,则在所述两个失配之间插入至少一个“假”失配,直到所述失配中的每一者与所述至少一个“假”失配之间的每个偏移都低于所述最大可编码值。“假”失配被定义为这样的失配:对于该失配,字节的位用于编码该失配,或者用于编码与参考序列中的相应参考核苷酸或碱基相等的核苷酸或碱基。在一些具体实施中,最大可编码值等于63,对应于能够以6位编码的最大值。然而,本公开并不限于具有最大可编码值63的具体实施。对于具有大于63的最大可编码值的具体实施,可以使用附加的位来编码该值。在此类具体实施中,这可能需要例如调整用于该读段的标头中的其他位长度、标头大小的超过一个字节的增加,或这两者的组合。因此,本公开的算法的特征对于特定使用案例是灵活的,但是可能由设计改变引起的具体实施可能导致性能的相应折衷,这在任何特定具体实施的某些情况下可能是可接受的并且甚至是有益的。
[0181]
图4提供了在必须插入“假”失配的情况下,根据第一编码子过程对读段的失配进
行编码的一个示例。该读段是不完全映射的读段,其与参考序列是全局映射的。该读段具有两个失配:
[0182]
o第一失配,其位于该读段中的第22位,其在于在该读段中用t核苷酸替换参考序列中的a核苷酸,以及
[0183]
o第二失配,其位于该读段中的第134位,其在于在该读段中用g核苷酸替换参考序列中的c核苷酸。
[0184]
第二失配与第一失配之间的位置偏移为112,其大于最大可编码值63。因此,“假”失配必须插入所述两个失配之间,使得所述失配中的每一者与该“假”失配之间的每个偏移都低于所述最大可编码值。具有t核苷酸的“假”失配(对应于参考序列中的“真”t核苷酸)例如插入该读段中的第85位。该“假”失配与第一失配之间计算出的位置偏移为63,其对应于最大可编码值。第二失配与该“假”失配之间计算出的位置偏移为49,其低于63。
[0185]
该读段的失配列表然后被编码为:
[0186]
o《22,t》,值“22”对应于该读段中的第一失配的绝对位置,
[0187]
o《63,t》,值“63”对应于该读段中的“假”失配的相对位置,即“假”失配与第一失配之间的偏移,以及
[0188]
o《49,g》,值“49”对应于该读段中的第二失配的相对位置,即第二失配与“假”失配之间的偏移。
[0189]
例如,《22,t》可以被转换为值“91”(在1个字节上编码),《63,t》可以被转换为值“255”(在1个字节上编码),并且《49,g》可以被转换为值“198”(在1个字节上编码)。这种字节编码用以下方式获得:
[0190]
偏移位置x4 核苷酸值(其中a=0,c=1,g=2,t=3)
[0191]
所述方法包括提供包含编码读段列表的压缩文件的最终步骤14。所述编码读段以与存储在初始未压缩文件中的读段的顺序相同的顺序存储在所述压缩文件中。然后可以通过根据本发明配置的适当解压缩软件和/或方法,从比对编码信息和参考序列重建每个读段。
[0192]
尽管参考计算装置20的示例性架构(出于说明的目的在图2中示出)进行了描述,但是此处公开的本发明的技术可以在硬件、软件、固件或它们的任何组合中实现。当在软件中实现时,计算机程序代码可以存储在计算机介质上,并且由包括一个或多个处理器的硬件处理单元执行,这与使用图2的装置20时的情况相同。应当理解,如本文所用的术语“处理器”旨在包括一个或多个处理装置,包括信号处理器、微处理器、微控制器、专用集成电路(asic)、现场可编程门阵列(fpga)或其他类型的处理电路系统,以及此类电路系统元件的部分或组合。另外,如本文所用的术语“存储器”旨在包括与处理器相关联的电子存储器,诸如以任何组合使用的随机存取存储器(ram)、只读存储器(rom)或其他类型的存储器。
[0193]
因此,用于执行本文所述的方法和协议的软件指令或代码可以存储在相关联的存储器装置(例如,rom、固定存储器或可移动存储器)中的一者或多者中,并且当准备使用时,被加载到ram中并由处理器执行。
[0194]
本公开的技术可以在各种各样的装置或设备中实施,包括例如移动电话、计算机、服务器、平板电脑和类似装置。
[0195]
尽管本文已参考附图描述了本发明的说明性实施方案,但是应当理解,本发明并
不限于那些确切的实施方案,并且在不脱离本发明的范围或实质的情况下,本领域的技术人员可以作出各种其他的改变和修改。
[0196]
图5是可以用于实施执行图1和图1a的压缩方法的系统的计算部件的一个示例的图示。
[0197]
计算装置500旨在表示各种形式的数字计算机,诸如膝上型电脑、台式电脑、工作站、个人数字助理、服务器、刀片式服务器、大型机和其他适当的计算机。计算装置550旨在表示各种形式的移动设备,诸如个人数字助理、移动电话、智能电话和其他类似的计算装置。此外,计算装置500或550可以包括通用串行总线(usb)闪存驱动器。usb闪存驱动器可存储操作系统和其他应用程序。usb闪存驱动器可包括输入/输出部件,诸如可插入到另一个计算装置的usb端口中的无线发射器或usb连接器。此处所示的部件、它们的连接和关系以及它们的功能仅意在作为示例,并不意在限制本文档中描述和/或要求保护的本发明的具体实施。
[0198]
计算装置500包括处理器502、存储器504、存储装置508、连接到存储器504和高速扩展端口510的高速接口508,以及连接到低速总线514和存储装置508的低速接口512。部件502、504、508、508、510和512中的每一者使用各种总线互连,并且可以安装在公共母板上或视情况以其他方式安装。处理器502可以处理用于在计算装置500内执行的指令,包括存储在存储器504中或存储装置508上的指令,以在外部输入/输出装置(诸如耦接到高速接口508的显示器516)上显示gui的图形信息。在其他具体实施中,可视情况将多个处理器和/或多个总线与多个存储器和多种类型的存储器一起使用。另外,可以连接多个计算装置500,每个装置提供必要操作的一些部分,例如,作为服务器库、一组刀片式服务器或多处理器系统。
[0199]
存储器504将信息存储在计算装置500内。在一个具体实施中,存储器504是一个或多个易失性存储器单元。在另一具体实施中,存储器504是一个或多个非易失性存储器单元。存储器504还可以是另一种形式的计算机可读介质,诸如磁盘或光盘。
[0200]
存储装置508能够为计算装置500提供海量存储。在一个具体实施中,存储装置508可以是或包含计算机可读介质,诸如软盘装置、硬盘装置、光盘装置或磁带装置、闪存存储器或其他类似的固态存储器装置,或装置阵列,包括存储区域网络中的装置或其他配置。计算机程序产品可在信息载体中有形地体现。计算机程序产品还可包含指令,该指令在被执行时,执行一种或多种方法,诸如上文所述的那些方法。信息载体是计算机可读介质或机器可读介质,诸如存储器504、存储装置508,或者处理器502上的存储器。
[0201]
高速控制器508管理计算装置500的带宽密集型操作,而低速控制器512管理带宽较不密集型操作。这种功能分配仅为示例。在一个具体实施中,高速控制器508例如通过图形处理器或加速器耦接到存储器504、显示器516,并且耦接到高速扩展端口510,该端口可以接受各种扩展卡(未示出)。在该具体实施中,低速控制器512耦接到存储装置508和低速扩展端口514。低速扩展端口(该端口可包括各种通信端口,例如usb、蓝牙、以太网、无线以太网)可例如通过网络适配器耦接到一个或多个输入/输出设备,诸如键盘、指向设备、麦克风/扬声器对、扫描仪或联网设备诸如交换机或路由器。计算装置500可以多种不同的形式实施,如图所示。例如,该计算装置可被实施为标准服务器520,或者在一组此类服务器中被实施多次。它还可被实施为机架式服务器系统524的一部分。此外,该计算装置可在个人计
算机诸如膝上型计算机522中实施。替代性地,来自计算装置500的部件可以与移动装置(未示出)诸如装置550中的其他部件组合。此类装置中的每一者可以包含计算装置500、550中的一个或多个装置,并且整个系统可以由彼此通信的多个计算装置500、550组成。
[0202]
计算装置500可以多种不同的形式实施,如图所示。例如,该计算装置可被实施为标准服务器520,或者在一组此类服务器中被实施多次。它还可被实施为机架式服务器系统524的一部分。此外,该计算装置可在个人计算机诸如膝上型计算机522中实施。替代性地,来自计算装置500的部件可以与移动装置(未示出)诸如装置550中的其他部件组合。此类装置中的每一者可以包含计算装置500、550中的一个或多个装置,并且整个系统可以由彼此通信的多个计算装置500、550组成。
[0203]
计算装置550包括处理器552、存储器564和输入/输出装置诸如显示器554、通信接口566和收发器568,以及其他部件。设备550还可设置有存储设备,诸如微驱动器或其他设备,以提供额外的存储。部件550、552、564、554、566和568中的每一者使用各种总线互连,并且这些部件中的若干部件可以安装在公共母板上或视情况以其他方式安装。
[0204]
处理器552可以执行计算装置550内的指令,包括存储在存储器564中的指令。处理器可被实现为包括独立的多个模拟处理器和数字处理器的芯片的芯片组。另外,处理器可使用多种架构中的任一种架构来实现。例如,处理器510可以是cisc(复杂指令集计算机)处理器、risc(精简指令集计算机)处理器或misc(最小指令集计算机)处理器。处理器可以提供例如装置550的其他部件的协调,诸如对用户接口的控制、由装置550运行的应用程序,以及由装置550进行的无线通信。
[0205]
处理器552可以通过耦接到显示器554的控制接口558和显示接口556与用户通信。显示器554可以是例如tft(薄膜晶体管液晶显示器)显示器或oled(有机发光二极管)显示器,或其他适当的显示技术。显示接口556可以包括用于驱动显示器554以向用户呈现图形和其他信息的适当电路系统。控制接口558可以接收来自用户的命令并且转换这些命令以提交给处理器552。此外,可以提供与处理器552通信的外部接口562,以便实现装置550与其他装置的近区通信。外部接口562可以例如在一些具体实施中提供有线通信,或者在其他具体实施中提供无线通信,并且还可以使用多个接口。
[0206]
存储器564将信息存储在计算装置550内。存储器564可被实施为一个或多个计算机可读介质、一个或多个易失性存储器单元或一个或多个非易失性存储器单元中的一者或多者。还可以提供扩展存储器574并且通过扩展接口572将其连接到装置550,该扩展接口可以包括例如simm(单列直插式存储器模块)卡接口。这种扩展存储器574可以为装置550提供额外的存储空间,或者还可以为装置550存储应用程序或其他信息。具体地讲,扩展存储器574可以包括用于执行或补充上述过程的指令,并且还可以包括安全信息。因此,例如,扩展存储器574可被提供为用于设备550的安全模块,并且可被编程为具有允许设备550安全使用的指令。此外,安全应用程序可经由simm卡连同附加信息一起提供,诸如将识别信息以不可破解的方式放置在simm卡上。
[0207]
存储器可包括例如闪存存储器和/或nvram存储器,如下所述。在一个具体实施中,计算机程序产品在信息载体中有形地体现。计算机程序产品包含指令,该指令在被执行时,执行一种或多种方法,诸如上文所述的那些方法。信息载体是计算机可读介质或机器可读介质,诸如存储器564、扩展存储器574或处理器552上的可通过例如收发器568或外部接口
562接收的存储器。
[0208]
装置550可以通过通信接口566进行无线通信,该通信接口在必要时可以包括数字信号处理电路系统。通信接口566可以提供在各种模式或协议下的通信,诸如gsm语音呼叫、sms、ems或mms信息收发、cdma、tdma、pdc、wcdma、cdma2000或gprs等。此类通信可通过例如射频收发器568发生。此外,可发生近程通信,诸如使用蓝牙、wi-fi或其他此类收发器(未示出)。此外,gps(全球定位系统)接收器模块570可向设备550提供附加的导航相关和位置相关的无线数据,该无线数据可由在设备550上运行的应用程序视情况使用。
[0209]
装置550还可以使用音频编解码器560可听地通信,该音频编解码器可以从用户接收口头信息并且将其转换为可用的数字信息。音频编解码器560同样可诸如通过扬声器(例如,在设备550的手持终端中)为用户生成可听声。这种声音可以包括来自语音电话呼叫的声音,可以包括录制的声音(例如,语音消息、音乐文件等),并且还可以包括由在装置550上操作的应用程序生成的声音。
[0210]
计算装置550能够以多种不同的形式实施,如图所示。例如,该计算装置可被实施为移动电话580。该计算装置还可被实施为智能电话582、个人数字助理或其他类似的移动设备的一部分。
[0211]
本文所述的系统和方法的各种具体实施可在数字电子电路、集成电路、特别设计的asic(专用集成电路)、计算机硬件、固件、软件和/或此类具体实施的组合中实现。这些各种具体实施可包括在一个或多个计算机程序中的具体实施,该一个或多个计算机程序能够在包括至少一个可编程处理器的可编程系统上执行和/或解释,该至少一个可编程处理器可以是专用或通用处理器,被耦接以从存储系统、至少一个输入设备和至少一个输出设备接收数据和指令以及将数据和指令发送到存储系统、至少一个输入设备和至少一个输出设备。
[0212]
这些计算机程序(也称为程序、软件、软件应用程序或代码)包括用于可编程处理器的机器指令,并且可以高级程序化和/或面向对象的编程语言和/或以汇编语言/机器语言来实现。如本文所用,术语“机器可读介质”、“计算机可读介质”是指用于向可编程处理器提供机器指令和/或数据的任何计算机程序产品、装置和/或设备,例如磁盘、光盘、存储器、可编程逻辑设备(pld),包括接收机器指令作为机器可读信号的机器可读介质。术语“机器可读信号”是指用于向可编程处理器提供机器指令和/或数据的任何信号。
[0213]
为了提供与用户的交互,本文所述的系统和技术可在计算机上实现,该计算机具有用于向用户显示信息的显示设备(例如crt(阴极射线管)或lcd(液晶显示器)监视器),以及用户可用来向该计算机提供输入的键盘和指向设备(例如鼠标或轨迹球)。也可使用其他类型的设备来提供与用户的交互;例如,提供给用户的反馈可以是任何形式的感官反馈,例如视觉反馈、听觉反馈或触觉反馈;并且可以任何形式接收来自用户的输入,包括声音、语音或触觉输入。
[0214]
本文所述的系统和技术可在计算系统中实现,该计算系统包括后端部件(例如,作为数据服务器)或包括中间件部件(例如,应用程序服务器)或包括前端部件(例如,具有图形用户界面或web浏览器的客户端计算机),用户可通过该计算系统与本文所述的系统和技术的具体实施进行交互,或者与此类后端部件、中间件部件或前端部件的任何组合进行交互。该系统的部件可通过数字数据通信的任何形式或介质(例如,通信网络)互连。通信网络
的示例包括局域网(“lan”)、广域网(“wan”)和互联网。
[0215]
该计算系统可包括客户端和服务器。客户端和服务器通常彼此远离,并且通常通过通信网络进行交互。客户端和服务器的关系借助于在相应计算机上运行并彼此具有客户端-服务器关系的计算机程序而产生。
[0216]
其他实施方案
[0217]
已经描述了多个实施方案。然而,应当理解,在不脱离本发明的实质和范围的情况下,可进行各种修改。此外,附图中所示的逻辑流程不需要所示的特定顺序或有序顺序来实现所需的结果。此外,可在所述流程中提供其他步骤,或者可消除步骤,并且可将其他部件添加到所述系统或从所述系统中移除。因此,其他实施方案也在以下权利要求书的范围内。
[0218]
实验结果
[0219]
根据本发明的压缩方法的统计和数值示例
[0220]
以下比较实施例是在大小为35,770mb的未压缩数据文件上进行的,该文件含有4800万个核苷酸读段或序列。该比较实施例的结果也在图6中用图形描绘。
[0221]
以下结果指示当使用每个相应算法压缩时具有4800万个读段的大小为35,770mb的未压缩文件的压缩版本的大小。这些结果描绘于图表610中。
[0222]
o已用gzip软件压缩的文件的大小:6,649mb(612)
[0223]
o已用基于非参考的spring软件压缩的文件的大小:1,402mb(614)
[0224]
o已用根据本公开的基于参考的压缩方法压缩的文件的大小:1,179mb(6160
[0225]
以下结果指示使用每个相应算法将大小为35,770mb的未压缩文件与4800万个读段进行比较所花费的时间量。这些结果描绘于图表620中。
[0226]
o使用基于非参考的spring软件的压缩时间:1,722s(622)
[0227]
o使用根据本发明的基于参考的压缩方法的压缩时间:181s(624)
[0228]
以下结果指示当使用每个相应算法压缩时具有4800万个读段的大小为35,770mb的未压缩文件的压缩版本的以位/核苷酸定量计的平均大小。这些结果描绘于图表630中。
[0229]
o未压缩数据文件的以位/核苷酸计的平均大小(ascii编码):8位/核苷酸(630)
[0230]
o已用适于4个可能字符a、t、c、g的编码压缩的文件的以位/核苷酸计的平均大小:2位/核苷酸(634)
[0231]
o已用根据本发明的基于参考的压缩方法压缩的文件的以位/核苷酸计的平均大小:0.33位/核苷酸(636)
[0232]
图7描绘了使用不同压缩算法来压缩使用wxs novaseq和参考基因组srr8604734生成的样品读段的附加比较结果。
[0233]
图表710示出了对使用参考基因组srr8604734的wxs novaseq读段进行gzip压缩(712)、对使用参考基因组srr8604734的wxs novaseq读段进行spring压缩(716)以及对使用参考基因组srr8604734的wxs novaseq读段进行本公开的压缩(716)之间的所得压缩大小(mb)的比较。测量结果以兆字节为单位。
[0234]
图表720示出了720中的压缩算法对使用参考基因组srr8604734的wxs novaseq读段进行压缩的压缩速度的比较。对使用参考基因组srr8604734的wxs novaseq读段进行压缩的spring压缩速度在(722)中示出,与之相比的对使用参考基因组srr8604734的wxs novaseq读段进行压缩的本公开的速度在724中示出。测量结果以秒为单位。
[0235]
图表730示出了730中的压缩算法在对使用参考基因组srr8604734的wxs novaseq读段进行压缩期间的存储器使用情况的比较。spring压缩利用13,428mb的存储器来压缩使用参考基因组srr8604734的wxs novaseq读段732,并且本公开使用3,604mb的存储器来压缩使用参考基因组srr8604734的wxs novaseq读段(734)。测量结果以兆字节为单位。
[0236]
图8描绘了使用不同的压缩算法来以不同的压缩比压缩使用不同的定序器和不同的参考基因组生成的样品读段的附加比较结果。
[0237]
图表810示出了使用novaseq生成的读段文件的以千兆字节(gb)为单位的原始大小(812)。使用gzip来压缩大小为100gb的novaseq读段,gzip将原始数据压缩为17.7gb(814)。本公开将100gb novaseq所生成读段的相同原始大小812文件压缩为3.4gb的压缩文件(816)。如810中所示,用于通过gzip和本公开两者进行的压缩的参考基因组为srr6882909,并且压缩比为5.2x。
[0238]
图表820示出了使用hiseq x ten生成的读段文件的以千兆字节(gb)为单位的原始大小(822)。使用gzip来压缩大小为100gb的hiseq x ten读段,gzip将原始数据压缩为24.9gb(824)。本公开将100gb hiseq x ten所生成读段的相同原始大小822文件压缩为8.1gb的压缩文件(826)。如820中所示,用于通过gzip和本公开两者进行的压缩的参考基因组为srr7725247,并且压缩比为3x。
[0239]
图表830示出了使用hiseq 2000生成的读段文件的以千兆字节(gb)为单位的原始大小(832)。使用gzip来压缩大小为100gb的hiseq 2000读段,gzip将原始数据压缩为27.6gb(834)。本公开将100gb hiseq2000所生成读段的相同原始大小832文件压缩为11.3gb的压缩文件(836)。如830中所示,用于通过gzip和本公开两者进行的压缩的参考基因组为err174324,并且压缩比为2.4x。
[0240]
上文指出的数值示例说明本发明允许快速压缩和解压缩,同时提供高压缩比。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。