1.本发明涉及基于单细胞拉曼光谱快速预测食醋发酵进程的方法,属于食醋酿造技术领域。
背景技术:
2.固态的开放式天然发酵是我国酿醋的主要方式。在醋醅这样一个微生物群落环境中,由复杂的生理生化和物理化学反应,包括微生物之间的相互作用,来生成醋酸和其他的主体风味物质。固态发酵食醋是指以大米、糯米或高粱等粮食谷物等为主要原料酿醋,再辅以麸皮、稻壳等填充剂为辅料,以固态方式在开放式环境下经多菌种协同参与醋酸发酵酿造而成的食醋。固态发酵食醋仅存在于中国,如中国东部地区的镇江香醋、中国西部地区的四川麸醋、中国华北的山西老陈醋和天津独流醋以及中国西北地区的凉州熏醋等。酿醋原料的复杂性和环境的开放性造就了酿醋微生物群落的多样性和复杂性,酿醋微生物群落的多样性和复杂性又赋予食醋丰富的口感和协调的香,生产出酸味柔和、醋香浓郁、酯香突出的食醋。
3.固态发酵食醋的酿造主要包括两个过程:酒精发酵和醋酸发酵。酒精发酵阶段包括淀粉质原料的糊化、糖化和酒化,主要是通过霉菌和酵母菌等所产生的糖化酶、淀粉酶以及酒化酶系共同催化完成;而在醋酸发酵阶段主要是通过醋酸菌、乳酸菌等微生物将乙醇氧化为醋酸,这些微生物除了产生乙酸还能产生大量的风味物质,赋予食醋丰富的口感和协调的香。
4.通常,固态发酵食醋产品的质量在很大程度上取决于对发酵进程的准确监控并在适当的时间终止发酵。目前,监控发酵进程的方法主要是对ph值、可滴定酸度、总酸含量等进行检测监控。但,这种方法具有滞后性。因为,正是微生物群落的活动导致了发酵体系的上述各项理化指标的变化。当依赖理化指标去调控发酵条件或终止发酵,往往难以获得符合预期品质的食醋产品。因此,有必要寻找一种能够从源头上对最终产品质量有影响的指标进行实时监测。酿造固态发酵食醋的核心为稳定的微生物群落,主要包括乳酸菌、醋酸菌等。群落中的微生物在发酵过程中多具有独特的功能,通过代谢过程产生风味物质和功能因子,形成食醋的风味骨架,进而影响食醋的最终品质。
5.目前,固态发酵食醋酿醋微生物群落的检测以高通量测序技术为主,但是应用高通量测序技术检测醋酸发酵进程的微生物群落变化存在耗时长等问题,无法及时辅助生产的研判,导致了生产过程中对酿醋微生物群落的了解有一定的滞后性;且环境、季节、工人的操作等因素都会影响固态发酵食醋酿醋微生物群落结构。因此,如何快速的检测固态发酵食醋醋酸发酵阶段的微生物群落变化,预测食醋固态发酵进程是目前亟待解决的问题。
6.拉曼光谱(raman spectra),是一种散射光谱。拉曼光谱分析法是基于拉曼散射效应,对与入射光频率不同的散射光谱进行分析以得到分子振动、转动方面信息,并应用于分子结构研究的一种分析方法。拉曼光谱检测作为一种快速、无损、无标记、非接触的快速检测技术,已被应用于各行各业中。特别是在生命科学领域,由于拉曼光谱分析时样品的用量
很少,不需要对生物样品进行脱水、包埋、切片、染色、标记等繁琐的前处理程序,不仅操作简单,而且不会损伤样品从而能够获得样品最真实的信息。另外,生物大分子多是处在水溶液环境中,水的拉曼散射很弱,干扰小,而且单细胞拉曼光谱能提供细胞内核酸、蛋白质、脂质、碳水化合物含量等大量信息,可在不损伤细胞的条件下实时动态地监测细胞分子结构变化,可以对细胞、病毒等进行原位检测分析,表征微生物细胞的基因型、表型和生理状态。
7.目前,拉曼光谱已被应用于探究微生物单细胞对物质吸收后特征峰的变化、推导物质循环过程、进行微生物分类鉴定和探索基因型与表型的关系。但,食醋固态发酵过程具有微生物群落结构复杂、酿造周期长、无法实时检测等特殊性,应用拉曼光谱还存在无法原位检测、部分微生物的拉曼光谱信噪比较低等问题。
技术实现要素:
8.[技术问题]
[0009]
本发明要解决的技术问题是现有食醋固态发酵进程部分理化指标或高通量测序来判断,存在滞后性、成本高的问题。
[0010]
[技术方案]
[0011]
针对现有技术存在的问题与不足,本发明提供了一种基于单细胞拉曼光谱快速预测食醋固态发酵进程的方法及应用,在对固态发酵食醋不同醋酸发酵阶段的微生物单细胞进行单细胞拉曼光谱的采集后,建立固态发酵食醋酿造过程中基于单细胞拉曼光谱的微生物群落的数据模型,基于此数据模型预测新的醋醅的理论发酵时间。所述理论发酵时间不同于实际的发酵时间,而是根据微生物群落信息预测的发酵时间。实际的发酵时间是从接种种醅开始计时的,理论发酵时间通常比实际的发酵时间短,在某些情况下,也可能比实际的发酵时间长,这主要取决于微生物群落的生长代谢情况。
[0012]
作为本发明的一个方面,提供基于单细胞拉曼光谱的分析食醋固态发酵过程的微生物群落结构的方法,包括以下步骤:
[0013]
(1)醋醅样品的采集:采集不同发酵时间的醋醅样品1~5g;
[0014]
(2)醋醅样品的预处理:取步骤(1)中的醋醅样品,加入4~20ml无菌ddh2o振荡1~3min后静置30s~1min,吸取1~4ml菌悬液用无菌水洗涤1~3次后重悬获得醋醅微生物群落的菌悬液;
[0015]
(3)醋醅样品的单细胞拉曼光谱数据采集:吸取步骤(2)中的菌悬液进行单细胞拉曼光谱的采集,其中光谱采集条件为:使用532nm激光,采集范围为280-3800cm-1
,激光强度为6mw,采集时间为5~20s/次,累积次数1~3次,不同发酵时间的醋醅分别采集100~500个细胞;
[0016]
(4)单细胞拉曼光谱数据的预处理:所有测得的醋醅样品的单细胞拉曼光谱数据均需要进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、并对所有的数据进行归一化处理,设置最大峰为100;
[0017]
(5)醋醅样品的单细胞拉曼光谱数据分型划分:汇总所有发酵时间下的醋醅样品的单细胞拉曼光谱数据进行层次聚类分析,按照距离将光谱数据分型,计算在不同发酵时间下的醋醅样品的微生物群落分型的组成,得到食醋固态发酵全过程的微生物群落结构。
[0018]
作为本发明的另一个方面,提供基于单细胞拉曼光谱的用于快速预测食醋固态发
酵进程的模型的构建方法,包括以下步骤:
[0019]
(1)醋醅样品的采集:
[0020]
采集不同发酵时间的醋醅样品1~5g;
[0021]
(2)醋醅样品的预处理:
[0022]
取步骤(1)中的醋醅样品,加入4~20ml无菌ddh2o振荡1~3min后静置30s~1min,吸取1~4ml菌悬液用无菌水洗涤1~3次后重悬获得醋醅微生物群落的菌悬液;
[0023]
(3)醋醅样品的单细胞拉曼光谱数据采集:
[0024]
吸取步骤(2)中的菌悬液进行单细胞拉曼光谱的采集,其中光谱采集条件为:使用532nm激光,采集范围为280-3800cm-1
,激光强度为6mw,采集时间为5~20s/次,累积次数1~3次,不同发酵时间的醋醅分别采集100~500个细胞;
[0025]
(4)单细胞拉曼光谱数据的预处理:
[0026]
所有测得的醋醅样品的单细胞拉曼光谱数据均需要进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、并对所有的数据进行归一化处理,设置最大峰为100;
[0027]
(5)醋醅样品的单细胞拉曼光谱数据分型划分:
[0028]
汇总所有发酵时间下的醋醅样品的单细胞拉曼光谱数据进行层次聚类分析,按照距离将光谱数据分型,计算在不同发酵时间下的醋醅样品的微生物群落分型的组成,得到食醋固态发酵全过程的微生物群落结构;
[0029]
(6)不同发酵时间的醋醅样品中微生物群落分型的曲线拟合:
[0030]
基于不同发酵时间醋醅样品的微生物群落分型数据建立模型,并使用多元线性回归方法拟合曲线以预测理论发酵时间,式中y代表发酵时间(day),x代表不同的分型(opu),n为总opu的个数,a为曲线中自变量的系数,b为该曲线的常数项系数,拟合方程如下所示;
[0031][0032]
在一种实施方式中,其中步骤(1)所述的醋醅样品取自发酵池,从发酵池的中间以及四个角从上至下取样,混匀后得到样品。
[0033]
在一种实施方式中,其中步骤(1)所述的醋醅样品源于工艺流程固定、发酵稳定的样品池。
[0034]
在一种实施方式中,其中步骤(2)中的醋醅微生物群落的菌悬液还需要进行100~1000倍稀释,稀释倍数视样品情况而定。稀释倍数以显微视野内看到的细胞不重叠、彼此之间存在距离、不会影响不同细胞的拉曼光谱为合适。
[0035]
在一种实施方式中,其中步骤(3)测定单细胞拉曼光谱的醋醅细胞为视野范围内的所有共计100~500个细胞。
[0036]
在一种实施方式中,其中步骤(4)所有醋醅样品的单细胞拉曼光谱数据进行分析时均选取400~1800cm-1
的指纹区图谱。
[0037]
在一种实施方式中,其中步骤(5)中的层次聚类分析使用的距离为欧式距离。对微生物群落进行距离的计算时通常采用欧氏距离。
[0038]
在一种实施方式中,其中步骤(5)中进行层次聚类分析得到不同发酵时间的醋醅样品的分型时,把分型的相对低于5%的分型视为其他分型。
[0039]
在一种实施方式中,其中步骤(6)中拟合曲线的方法为多元线性回归的方法。
[0040]
在一种实施方式中,其中步骤(6)中拟合曲线时使用的软件为spss 26。
[0041]
在一种实施方式中,其中步骤(6)中拟合曲线时的n视为总opu的个数,该个数的取值视欧氏距离自行选取。
[0042]
在一种实施方式中,其中步骤(6)中拟合曲线时最优曲线有时并不是利用所有的分型计算得到的。
[0043]
作为本发明的另一个方面,提供应用所述模型进行食醋固态发酵的方法,是在发酵过程中从食醋发酵池取样,将醋醅样品进行预处理,然后采集单细胞的拉曼光谱数据,将数据进行规范处理后进行分型划分,得到该醋醅样品的微生物群落分型的组成,代入公式ⅰ,计算得到理论发酵时间,如果理论发酵时间早于或晚于实际发酵时间,则采取措施调整发酵过程。可采取的措施包括:添加种醅、表层覆盖糠壳保温,提前或推后翻醅时间等。
[0044]
[有益效果]
[0045]
使用本发明方法分析食醋固态发酵过程中的微生物群落结构,耗时短、对操作者的水平要求不高,避免了使用高通量测序技术手段获取醋醅微生物群落结构耗时长、要求操作者的水平和经验等特点。
[0046]
本发明以工艺流程固定、发酵稳定的样品池为基础,取样,并基于单细胞拉曼光谱快速检测微生物群落结构、建立模型。然后,在其他样品池(例如新启用的发酵池)或不同批次的发酵过程中,取样,并基于单细胞拉曼光谱快速检测微生物群落结构,代入模型后,如果模型计算得到的理论发酵时间与实际发酵时间一致,则说明发酵进程正常,如果模型计算得到的理论发酵时间早于或晚于发酵时间,则说明需要采取措施调控发酵进程,以确保发酵结束时能得到合格的产品。
[0047]
使用本发明方法预测固态发酵食醋醋酸发酵阶段耗时短、对操作者的水平要求不高,避免了使用高通量测序技术手段获取醋醅微生物群落结构耗时长、要求操作者的水平和经验等特点。
[0048]
本发明建模过程中,对分型进行分类的方法为层次聚类分析,该方法的距离和规则的相似度容易定义,限制少,不需要预先制定聚类数,相较于其他分类方法有一定的优势。
[0049]
使用本发明方法利用单细胞拉曼光谱可以从单细胞水平快速检测微生物群落变化,以便于实时监控食醋发酵状态,及时辅助生产的研判,为现代化智能酿醋设备的建立提供参考。
附图说明
[0050]
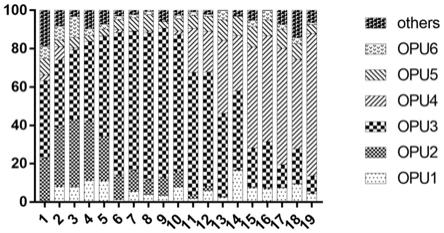
图1为实施例1中固态发酵食醋不同醋酸发酵阶段的醋醅微生物的组成。
[0051]
图2为实施例2中固态发酵食醋不同醋酸发酵阶段的醋醅微生物的组成。
[0052]
图3为实施例3中固态发酵食醋不同醋酸发酵阶段的醋醅微生物的组成。
[0053]
图4为实施例4中获得的固态发酵食醋在不同发酵时间下的醋醅微生物群落分型结构。
[0054]
图5为实施例5中获得的固态发酵食醋在不同发酵时间下的醋醅微生物群落分型结构。
[0055]
图6为实施例6中获得的固态发酵食醋在不同发酵时间下的醋醅微生物群落分型结构。
具体实施方式
[0056]
以下结合具体实施例和附图对本发明作进一步说明。应当理解的是,此处所描述的具体实施方式仅用于说明和解释本发明,并不用于限制本发明。
[0057]
还需要说明的是,术语“包括”、“包含”或者其任何其他变体意在涵盖非排他性的包含,从而使得包括一系列要素的过程、方法、商品或者设备不仅包括那些要素,而且还包括没有明确列出的其他要素,或者是还包括为这种过程、方法、商品或者设备所固有的要素。在没有更多限制的情况下,由语句“包括一个
……”
限定的要素,并不排除在包括所述要素的过程、方法、商品或者设备中还存在另外的相同要素。
[0058]
下述实施例中,作为取样来源的编号为35a的发酵池所采用的发酵工艺是:以麸皮、糠壳为原料,接种发酵第8天的醋醅作为种醅,接种种醅混匀后开始发酵,并将接种种醅的时间作为发酵第1天,发酵过程中采用逐层翻醅的方式,每天翻醅一次,向下翻相同的深度,翻醅至第8天露底,后续每天整体翻醅,持续至醋酸发酵结束,即发酵第19天结束(不同编号的样品池中的发酵工艺均是一致的)。
[0059]
下述实施例所述微生物群落分型,是指利用所有的酿醋微生物的光谱进行聚类分析,欧氏距离数值固定的不同分支,一个分支即为一个分型。
[0060]
下述实施例所述微生物群落分型的组成,是指,利用所有的酿醋微生物的光谱进行聚类分析,得到的所有的在欧氏距离数值固定的情况下的不同分支的数量和比例。
[0061]
下述实施例所述微生物群落结构,是指使用单细胞拉曼光谱检测醋醅中的不同微生物,通过聚类分析得到不同的分型,不同发酵时间下分型的组成及其占比情况。
[0062]
下述实施例所述食醋固态发酵全过程的微生物群落结构,是指使用单细胞拉曼光谱检测醋醅中的不同微生物,通过聚类分析得到不同的分型,发酵全过程中分型的组成及其占比情况。
[0063]
下述实施例所述不同发酵时间的微生物群落分型的相对丰度,是指分型计算完成之后,每天的不同分型在当天所有测得的光谱中的组成及其占比。
[0064]
实施例1构建基于单细胞拉曼光谱的用于快速预测食醋固态发酵进程的模型
[0065]
(1)醋醅样品的采集:在不同的实际发酵时间,在编号为35a的发酵池的中间以及四个角从上至下打洞取样,将五个位置的样品混匀后作为醋醅样品;
[0066]
(2)醋醅样品的预处理:取步骤(1)中的1g醋醅样品,加入4ml无菌ddh2o振荡1min后静置30s,吸取2ml菌悬液用无菌水洗涤3次,获得醋醅微生物群落的菌悬液,菌悬液稀释1000倍;
[0067]
(3)醋醅样品的单细胞拉曼光谱数据采集:吸取步骤(2)中的菌悬液进行单细胞拉曼光谱的采集,其中光谱采集条件为:使用532nm激光,采集范围为280-3800cm-1
,激光强度为6mw,采集时间为10s,累积次数1次,不同发酵时间的醋醅采集视野范围内的所有共计200个细胞;
[0068]
(4)单细胞拉曼光谱数据的预处理:所有测得的醋醅样品的单细胞拉曼光谱数据均需要进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、并对所有的数据进行
归一化处理,设置最大峰为100;所有醋醅样品的单细胞拉曼光谱数据进行分析时均选取400~1800cm-1
的指纹区图谱;(使用德国witec公司的suit 5软件进行光谱预处理)
[0069]
(5)醋醅样品的单细胞拉曼光谱数据分型划分:汇总所有发酵时间下的醋醅样品的单细胞拉曼光谱数据进行层次聚类分析,当欧氏距离为100时,把不同分支上的光谱数据作为不同的分型,共得到七个分型,计算在不同发酵时间下的醋醅样品的微生物群落分型的组成,得到食醋固态发酵全过程的微生物群落结构。结果见图1。
[0070]
(6)不同发酵时间的醋醅样品中微生物群落分型的曲线拟合:基于不同发酵时间醋醅样品的微生物群落分型数据建立模型,并使用多元线性回归方法拟合曲线以预测实际醋酸发酵时间。式中y代表发酵时间(day),x代表不同的分型(opu),拟合方程如下所示:
[0071]
y=13.56 0.32x
opu1-0.45x
opu2
0.09x
opu4-0.61x
opu5
0.40x
opu6
0.01x
others
[0072]
从图1可以看出,2号分型(opu2)随着发酵的进行其相对丰度逐渐降低,而4号分型(opu4)在发酵后期相对丰度逐渐升高,3号分型(opu3)在发酵全过程中均维持有一定程度的数量,1号、6号(opu1、opu6)分型在发酵过程中的相对丰度较低。拟合曲线的r2为0.974。
[0073]
实施例2构建基于单细胞拉曼光谱的用于快速预测食醋固态发酵进程的模型
[0074]
(1)醋醅样品的采集:在不同的实际发酵时间,在编号为26b的发酵池的中间以及四个角从上至下打洞取样,将五个位置的样品混匀后作为醋醅样品;
[0075]
(2)醋醅样品的预处理:取步骤(1)中的3g醋醅样品,加入12ml无菌ddh2o振荡2min后静置1min,吸取2ml菌悬液用无菌水洗涤3次,获得醋醅微生物群落的菌悬液,菌悬液稀释500倍;
[0076]
(3)醋醅样品的单细胞拉曼光谱数据采集:吸取步骤(2)中的菌悬液进行单细胞拉曼光谱的采集,其中光谱采集条件为:使用532nm激光,采集范围为280-3800cm-1
,激光强度为6mw,采集时间为15s,累积次数2次,不同发酵时间的醋醅采集视野范围内的所有共计100个细胞;
[0077]
(4)单细胞拉曼光谱数据的预处理:所有测得的醋醅样品的单细胞拉曼光谱数据均需要进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、并对所有的数据进行归一化处理,设置最大峰为100;所有醋醅样品的单细胞拉曼光谱数据进行分析时均选取400~1800cm-1
的指纹区图谱;
[0078]
(5)醋醅样品的单细胞拉曼光谱数据分型划分:汇总所有发酵时间下的醋醅样品的单细胞拉曼光谱数据进行层次聚类分析,当欧氏距离为80时,把不同分支上的光谱数据作为不同的分型,共得到八个分型,计算在不同发酵时间下的醋醅样品的微生物群落分型的组成,得到食醋固态发酵全过程的微生物群落结构。结果见图2。
[0079]
(6)不同发酵时间的醋醅样品中微生物群落分型的曲线拟合:基于不同发酵时间醋醅样品的微生物群落分型数据建立模型,并使用多元线性回归方法拟合曲线以预测实际醋酸发酵时间。式中y代表发酵时间(day),x代表不同的分型(opu),拟合方程如下所示:
[0080]
y=18.26-0.38x
opu1-0.41x
opu3-0.31x
opu4
0.05x
opu5-0.14x
opu6
0.01x
opu7
0.51x
others
[0081]
从图2可以看出,1号、3号分型(opu1、opu3)在发酵过程中相对丰度一直较低,4号分型(opu4)在发酵前期的相对丰度逐渐下降,6号分型(opu6)为发酵中期的优势微生物分型,2号分型(opu2)在发酵后期的相对丰度较高。拟合曲线的r2为0.963。
[0082]
实施例3构建基于单细胞拉曼光谱的用于快速预测食醋固态发酵进程的模型
[0083]
(1)醋醅样品的采集:在不同的实际发酵时间,在编号为4c的发酵池的中间以及四个角从上至下打洞取样,将五个位置的样品混匀后作为醋醅样品;
[0084]
(2)醋醅样品的预处理:取步骤(1)中的5g醋醅样品,加入20ml无菌ddh2o振荡2min后静置30s,吸取1ml菌悬液用无菌水洗涤5次,获得醋醅微生物群落的菌悬液,菌悬液稀释100倍;
[0085]
(3)醋醅样品的单细胞拉曼光谱数据采集:吸取步骤(2)中的菌悬液进行单细胞拉曼光谱的采集,其中光谱采集条件为:使用532nm激光,采集范围为280-3800cm-1
,激光强度为6mw,采集时间为15s,累积次数3次,不同发酵时间的醋醅采集视野范围内的所有共计100个细胞;
[0086]
(4)单细胞拉曼光谱数据的预处理:所有测得的醋醅样品的单细胞拉曼光谱数据均需要进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、并对所有的数据进行归一化处理,设置最大峰为100;所有醋醅样品的单细胞拉曼光谱数据进行分析时均选取400~1800cm-1
的指纹区图谱;
[0087]
(5)醋醅样品的单细胞拉曼光谱数据分型划分:汇总所有发酵时间下的醋醅样品的单细胞拉曼光谱数据进行层次聚类分析,当欧氏距离为200时,把不同分支上的光谱数据作为不同的分型,共得到四个分型,计算在不同发酵时间下的醋醅样品的微生物群落分型的组成,得到食醋固态发酵全过程的微生物群落结构。结果见图3。
[0088]
(6)不同发酵时间的醋醅样品中微生物群落分型的曲线拟合:基于不同发酵时间醋醅样品的微生物群落分型数据建立模型,并使用多元线性回归方法拟合曲线以预测实际醋酸发酵时间。式中y代表发酵时间(day),x代表不同的分型(opu),拟合方程如下所示:
[0089]
y=8.52-0.30x
opu2
0.09x
opu3
0.18x
others
[0090]
从图3可以看出,1号分型(opu1)在发酵前期相对丰度逐渐增加,在发酵中期丰度达到最高,随着发酵的继续进行其相对丰度逐渐减低。3号分型(opu3)在发酵后期的相对丰度较高。拟合曲线的r2为0.905。
[0091]
在上述3个实施例中,对测定好的总计19天的醋醅样品的单细胞拉曼光谱数据使用层次聚类分析进行分型的选取,把分型的相对丰度<5%的类别视为其他,得到了固态发酵食醋不同发酵时间的微生物群落分型的相对丰度变化情况。3个实施例的结果显示利用不同的单细胞拉曼光谱条件我们可以得到固态发酵食醋酿造过程中基于单细胞拉曼光谱的微生物群落的数据模型,后续可以利用此数据模式预测新的醋醅的理论发酵时间。
[0092]
综上所述,本发明采集不同发酵时间的醋醅样品的单细胞拉曼光谱数据,然后利用层次聚类分析对所有醋醅样品的单细胞拉曼光谱数据进行分型的划分,得到食醋固态发酵过程中的微生物群落不同分型的组成,并以此分型结构数据使用多元线性回归方法进行曲线拟合得到预测醋醅发酵时间的曲线。此后采集新的醋醅样品后,在比较光谱的相似性后得到该天的微生物分型组成,并代入回归曲线了解其具体的发酵天数,实现快速检测醋醅样品的发酵天数的目的。我们从单细胞水平快速检测微生物群落变化,以便于实时监控食醋发酵状态,及时辅助生产的研判,为现代化智能酿醋设备的建立提供参考,具有极高的价值。
[0093]
实施例4应用模型快速预测食醋固态发酵进程
[0094]
(一)建模
[0095]
(1)醋醅样品的采集:选取编号为22c的发酵池的正常发酵的全过程(即每一天)的醋醅样品,取样点是发酵池的中间以及四个角,从上至下打洞取样,混匀后作为醋醅样品;
[0096]
(2)醋醅样品的预处理:取步骤(1)中的3g醋醅样品,加入12ml无菌ddh2o振荡2min后静置1min,吸取2ml菌悬液用无菌水洗涤3次,获得醋醅微生物群落的菌悬液,菌悬液稀释500倍;
[0097]
(3)醋醅样品的单细胞拉曼光谱数据采集:吸取步骤(2)中的菌悬液进行单细胞拉曼光谱的采集,其中光谱采集条件为:使用532nm激光,采集范围为280-3800cm-1
,激光强度为6mw,采集时间为15s,累积次数2次,不同发酵时间的醋醅采集视野范围内的所有共计100个细胞;
[0098]
(4)单细胞拉曼光谱数据的预处理:所有测得的醋醅样品的单细胞拉曼光谱数据均需要进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、并对所有的数据进行归一化处理,设置最大峰为100;所有醋醅样品的单细胞拉曼光谱数据进行分析时均选取400~1800cm-1
的指纹区图谱;
[0099]
(5)醋醅样品的单细胞拉曼光谱数据分型划分:汇总所有发酵时间下的醋醅样品的单细胞拉曼光谱数据进行层次聚类分析,当欧氏距离为100时,把不同分支上的光谱数据作为不同的分型,共得到七个分型,得到不同发酵时间下光谱分型的相对丰度情况;
[0100]
(6)不同发酵时间的醋醅样品中微生物群落分型的曲线拟合:基于步骤(5)不同发酵时间醋醅样品的微生物群落分型数据建立模型,并使用多元线性回归方法拟合曲线以预测实际醋酸发酵时间。
[0101]
拟合得到的曲线是:
[0102]
y=13.56 0.32x
opu1-0.45x
opu2
0.09x
opu4-0.61x
opu5
0.40x
opu6
0.01x
others
[0103]
(二)预测
[0104]
醋醅样品的采集:选取新一批次的编号为22c的正常发酵的发酵池的实际发酵时间5、10、16天的醋醅样品,取样点是发酵池的中间以及四个角,从上至下打洞取样,混匀后作为醋醅样品。
[0105]
参照(一)中步骤(2)~(5),得到5、10、16天的光谱分型的相对丰度情况,将5、10、16天的光谱分型的相对丰度情况,代入(一)中(6)拟合的曲线中,得到理论发酵时间,如表1所示,分别是5.9、10.7、17.0天(与实际发酵时间相差≤1天)。这说明正常发酵情况下,模型计算得到的理论发酵时间与实际发酵时间相差不大,说明本发明方法可以有效预测正常发酵的样品。
[0106]
5、10、16天的光谱分型的相对丰度情况结果见图4,图4结果显示,在发酵第5天时,opu2和opu3占据了较高的相对丰度,而在发酵第十天时,opu3是群落中的优势微生物分型,但在发酵第16天时,opu4的相对丰度较第10天有所上涨,但是opu3的相对丰度降低了。
[0107]
实施例5应用模型快速预测食醋固态发酵进程
[0108]
(一)建模
[0109]
(1)醋醅样品的采集:选取编号为15d的发酵池的正常发酵全过程(即每一天)的醋醅样品,取样点是发酵池的中间以及四个角,从上至下打洞取样,混匀后作为醋醅样品;
[0110]
(2)醋醅样品的预处理:取步骤(1)中的3g醋醅样品,加入20ml无菌ddh2o振荡2min
后静置30s,吸取1ml菌悬液用无菌水洗涤5次,获得醋醅微生物群落的菌悬液,菌悬液稀释100倍;
[0111]
(3)醋醅样品的单细胞拉曼光谱数据采集:吸取步骤(2)中的菌悬液进行单细胞拉曼光谱的采集,其中光谱采集条件为:使用532nm激光,采集范围为280-3800cm-1
,激光强度为6mw,采集时间为15s,累积次数3次,不同发酵时间的醋醅采集视野范围内的所有共计100个细胞;
[0112]
(4)单细胞拉曼光谱数据的预处理:所有测得的醋醅样品的单细胞拉曼光谱数据均需要进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、并对所有的数据进行归一化处理,设置最大峰为100;所有醋醅样品的单细胞拉曼光谱数据进行分析时均选取400~1800cm-1
的指纹区图谱;
[0113]
(5)醋醅样品的单细胞拉曼光谱数据分型划分:汇总所有发酵时间下的醋醅样品的单细胞拉曼光谱数据进行层次聚类分析,当欧氏距离为100时,把不同分支上的光谱数据作为不同的分型,共得到七个分型,得到不同发酵时间下光谱分型的相对丰度情况;(6)不同发酵时间的醋醅样品中微生物群落分型的曲线拟合:基于不同发酵时间醋醅样品的微生物群落分型数据建立模型,并使用多元线性回归方法拟合曲线以预测实际醋酸发酵时间。拟合得到的曲线如下:
[0114]
y=14.12 0.36x
opu1-0.45x
opu2
0.10x
opu4-0.62x
opu5
0.39x
opu6
0.03x
others
[0115]
(二)预测
[0116]
醋醅样品的采集:选取编号为15d的发酵池的正常发酵的发酵池的实际发酵时间3、9、12天的醋醅样品,取样点是发酵池的中间以及四个角,从上至下打洞取样,混匀后作为醋醅样品。
[0117]
参照(一)中步骤(2)~(5),得到3、9、12天天的光谱分型的相对丰度情况,将3、9、12天的光谱分型的相对丰度情况,代入(一)中(6)拟合的曲线中,得到理论发酵时间,如表1所示。
[0118]
3、9、12天的光谱分型的相对丰度情况如图5所示。图5结果显示,在发酵第3天时,opu2和opu3占据了较高的相对丰度,发酵第9天时,opu3是群落中的优势微生物分型,但在发酵第12天时,opu4的相对丰度较第9天有所上涨,但是opu3的相对丰度相较第9天有一定程度的下降。
[0119]
实施例6应用模型快速预测食醋固态发酵进程
[0120]
(一)建模
[0121]
(1)醋醅样品的采集:选取编号为8c的发酵池的正常发酵全过程(即每一天)的醋醅样品,取样点是发酵池的中间以及四个角,从上至下打洞取样,混匀后作为醋醅样品;
[0122]
(2)醋醅样品的预处理:取步骤(1)中的1g醋醅样品,加入4ml无菌ddh2o振荡1min后静置30s,吸取2ml菌悬液用无菌水洗涤3次,获得醋醅微生物群落的菌悬液,菌悬液稀释1000倍;
[0123]
(3)醋醅样品的单细胞拉曼光谱数据采集:吸取步骤(2)中的菌悬液进行单细胞拉曼光谱的采集,其中光谱采集条件为:使用532nm激光,采集范围为280-3800cm-1
,激光强度为6mw,采集时间为10s,累积次数1次,不同发酵时间的醋醅采集视野范围内的所有共计200个细胞;
[0124]
(4)单细胞拉曼光谱数据的预处理:所有测得的醋醅样品的单细胞拉曼光谱数据均需要进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、并对所有的数据进行归一化处理,设置最大峰为100;所有醋醅样品的单细胞拉曼光谱数据进行分析时均选取400~1800cm-1
的指纹区图谱;
[0125]
(5)醋醅样品的单细胞拉曼光谱数据分型划分:汇总所有发酵时间下的醋醅样品的单细胞拉曼光谱数据进行层次聚类分析,当欧氏距离为100时,把不同分支上的光谱数据作为不同的分型,共得到七个分型,得到不同发酵时间下光谱分型的相对丰度情况;
[0126]
(6)不同发酵时间的醋醅样品中微生物群落分型的曲线拟合:基于不同发酵时间醋醅样品的微生物群落分型数据建立模型,并使用多元线性回归方法拟合曲线以预测实际醋酸发酵时间。拟合得到的曲线如下:
[0127]
y=13.81 0.41x
opu1-0.53x
opu2
0.04x
opu4-0.76x
opu5
0.44x
opu6
0.26x
others
。
[0128]
(二)预测
[0129]
醋醅样品的采集:选取编号为8c的发酵池的正常发酵的发酵池的实际发酵时间4、7、11天的醋醅样品,取样点是发酵池的中间以及四个角,从上至下打洞取样,混匀后作为醋醅样品。
[0130]
参照(一)中步骤(2)~(5),得到4、7、11天的光谱分型的相对丰度情况,将4、7、11天的光谱分型的相对丰度情况,代入(一)中(6)拟合的曲线中,得到理论发酵时间,如表1所示。
[0131]
4、7、11天的光谱分型的相对丰度情况如图6所示。图6结果显示在发酵第4天时,opu2和opu3占据了较高的相对丰度,而在发酵第11天时,opu3是群落中的最具优势的微生物分型,opu4也有较高的相对丰度,但在发酵第17天时,opu4的相对丰度较第11天涨幅明显,但是opu3的相对丰度明显降低。
[0132]
表1实施例4、5、6中醋醅的实际发酵时间及预测的理论发酵时间
[0133][0134]
对已知实际发酵时间的样品测定单细胞拉曼光谱,获得该发酵天数下的醋醅微生物群落分型结构,并拟合得到曲线。将采用相同工艺的、不同批次的发酵池的已知实际发酵时间的样品测定单细胞拉曼光谱,获得该发酵天数下的醋醅微生物群落分型结构,然后代入拟合得到的曲线,求得理论发酵时间。结果显示,预测的理论发酵时间与实际发酵时间相差不大,说明本发明方法可以有效预测正常发酵的样品。
[0135]
实施例7应用模型控制优化食醋固态发酵进程
[0136]
(一)建模
[0137]
(1)醋醅样品的采集:选取编号为24a的发酵池的正常发酵全过程(即每一天)的醋醅样品,取样点是发酵池的中间以及四个角,从上至下打洞取样,混匀后作为醋醅样品;
[0138]
(2)醋醅样品的预处理:取步骤(1)中的3g醋醅样品,加入20ml无菌ddh2o振荡2min后静置30s,吸取1ml菌悬液用无菌水洗涤5次,获得醋醅微生物群落的菌悬液,菌悬液稀释100倍;
[0139]
(3)醋醅样品的单细胞拉曼光谱数据采集:吸取步骤(2)中的菌悬液进行单细胞拉曼光谱的采集,其中光谱采集条件为:使用532nm激光,采集范围为280-3800cm-1
,激光强度为6mw,采集时间为15s,累积次数3次,不同发酵时间的醋醅采集视野范围内的所有共计100个细胞;
[0140]
(4)单细胞拉曼光谱数据的预处理:所有测得的醋醅样品的单细胞拉曼光谱数据均需要进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、并对所有的数据进行归一化处理,设置最大峰为100;所有醋醅样品的单细胞拉曼光谱数据进行分析时均选取400~1800cm-1
的指纹区图谱;
[0141]
(5)醋醅样品的单细胞拉曼光谱数据分型划分:汇总所有发酵时间下的醋醅样品的单细胞拉曼光谱数据进行层次聚类分析,当欧氏距离为100时,把不同分支上的光谱数据作为不同的分型,共得到七个分型,得到不同发酵时间下光谱分型的相对丰度情况;
[0142]
(6)不同发酵时间的醋醅样品中微生物群落分型的曲线拟合:基于正常发酵时不同发酵时间醋醅样品的微生物群落分型数据建立模型,并使用多元线性回归方法拟合曲线以预测实际醋酸发酵时间。拟合得到的曲线如下:
[0143]
y=13.88 0.41x
opu1-0.38x
opu2
0.12x
opu4-0.62x
opu5
0.35x
opu6
0.08x
others
[0144]
(二)控制优化食醋固态发酵进程
[0145]
包括以下步骤:
[0146]
(1)醋醅样品的采集:选择发酵偏快(约比正常批次生产的醋醅发酵进程快2天,即微生物大量生长繁殖,醋醅样品的总酸含量大于当前天数正常发酵下的总酸含量,此时需要调整工艺,否则会生产出品质较差的食醋)的样品池的样品,在发酵池中间以及四个角从上至下打洞取样,混匀后作为醋醅样品;
[0147]
(2)醋醅样品的预处理:取步骤(1)中的3g醋醅样品,加入20ml无菌ddh2o振荡2min后静置30s,吸取1ml菌悬液用无菌水洗涤5次,获得醋醅微生物群落的菌悬液,菌悬液稀释100倍;
[0148]
(3)醋醅样品的单细胞拉曼光谱数据采集:吸取步骤(2)中的菌悬液进行单细胞拉曼光谱的采集,其中光谱采集条件为:使用532nm激光,采集范围为280-3800cm-1
,激光强度为6mw,采集时间为15s,累积次数3次,不同发酵时间的醋醅采集视野范围内的所有共计100个细胞;
[0149]
(4)单细胞拉曼光谱数据的预处理:所有测得的醋醅样品的单细胞拉曼光谱数据均需要进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、并对所有的数据进行归一化处理,设置最大峰为100;所有醋醅样品的单细胞拉曼光谱数据进行分析时均选取400~1800cm-1
的指纹区图谱;
[0150]
(5)醋醅样品的单细胞拉曼光谱数据分型划分:将先前测得的不同分型的光谱数据与现有的光谱数据进行对比,按照光谱相似性将新测得的光谱进行分型的分类,得到不同发酵时间下光谱分型的相对丰度情况。
[0151]
(6)将步骤(5)中求得的不同分型数据带入步骤(一)中步骤(6)得到的曲线,计算
理论发酵时间。计算结果表明:实际发酵时间是13天,理论发酵时间是15天,实际发酵时间比理论发酵时间慢了两天。
[0152]
(7)工艺调整:将步骤(1)的发酵过快的醋醅分为两部分,其中一部分继续按照传统发酵工艺进行生产,另一部分醋醅则在在发酵17天时终止发酵。最终在发酵结束时,测定两种工艺下的醋醅的总酸含量。结果见表2。
[0153]
实施例8应用模型控制优化食醋固态发酵进程
[0154]
(一)建模
[0155]
(1)醋醅样品的采集:选取编号为4c的发酵池的正常发酵全过程(即每一天)的醋醅样品,取样点是发酵池的中间以及四个角,从上至下打洞取样,混匀后作为醋醅样品;
[0156]
(2)醋醅样品的预处理:取步骤(1)中的1g醋醅样品,加入4ml无菌ddh2o振荡1min后静置30s,吸取2ml菌悬液用无菌水洗涤3次,获得醋醅微生物群落的菌悬液,菌悬液稀释1000倍;
[0157]
(3)醋醅样品的单细胞拉曼光谱数据采集:吸取步骤(2)中的菌悬液进行单细胞拉曼光谱的采集,其中光谱采集条件为:使用532nm激光,采集范围为280-3800cm-1
,激光强度为6mw,采集时间为10s,累积次数1次,不同发酵时间的醋醅采集视野范围内的所有共计200个细胞;
[0158]
(4)单细胞拉曼光谱数据的预处理:所有测得的醋醅样品的单细胞拉曼光谱数据均需要进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、并对所有的数据进行归一化处理,设置最大峰为100;所有醋醅样品的单细胞拉曼光谱数据进行分析时均选取400~1800cm-1
的指纹区图谱;
[0159]
(5)醋醅样品的单细胞拉曼光谱数据分型划分:汇总所有发酵时间下的醋醅样品的单细胞拉曼光谱数据进行层次聚类分析,当欧氏距离为100时,把不同分支上的光谱数据作为不同的分型,共得到七个分型,得到不同发酵时间下光谱分型的相对丰度情况;
[0160]
(6)不同发酵时间的醋醅样品中微生物群落分型的曲线拟合:基于正常发酵时不同发酵时间醋醅样品的微生物群落分型数据建立模型,并使用多元线性回归方法拟合曲线以预测实际醋酸发酵时间。拟合得到的曲线如下:
[0161]
y=12.90 0.44x
opu1-0.39x
opu2
0.08x
opu4-0.52x
opu5
0.39x
opu6
0.13x
others
[0162]
(二)控制优化食醋固态发酵进程
[0163]
(1)醋醅样品的采集:选择发酵偏慢(约比正常批次生产的醋醅发酵进程慢2天,即醋醅样品的总酸含量低于当前天数正常发酵下的总酸含量)的样品池的样品,在发酵池中间以及四个角从上至下打洞取样,混匀后作为醋醅样品;
[0164]
(2)醋醅样品的预处理:取步骤(1)中的1g醋醅样品,加入4ml无菌ddh2o振荡1min后静置30s,吸取2ml菌悬液用无菌水洗涤3次,获得醋醅微生物群落的菌悬液,菌悬液稀释1000倍;
[0165]
(3)醋醅样品的单细胞拉曼光谱数据采集:吸取步骤(2)中的菌悬液进行单细胞拉曼光谱的采集,其中光谱采集条件为:使用532nm激光,采集范围为280-3800cm-1
,激光强度为6mw,采集时间为10s,累积次数1次,不同发酵时间的醋醅采集视野范围内的所有共计200个细胞;
[0166]
(4)单细胞拉曼光谱数据的预处理:所有测得的醋醅样品的单细胞拉曼光谱数据
均需要进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、并对所有的数据进行归一化处理,设置最大峰为100;所有醋醅样品的单细胞拉曼光谱数据进行分析时均选取400~1800cm-1
的指纹区图谱;
[0167]
(5)醋醅样品的单细胞拉曼光谱数据分型划分:将先前测得的不同分型的光谱数据与现有的光谱数据进行对比,按照光谱相似性将新测得的光谱进行分型的分类,得到不同发酵时间下光谱分型的相对丰度情况。
[0168]
(6)将步骤(5)中求得的不同分型数据带入步骤(一)中步骤(6)得到的曲线,计算理论发酵时间。计算结果表明:实际发酵时间是6天,理论发酵时间是4天,实际发酵时间比理论发酵时间快了两天。
[0169]
(7)工艺调整:将步骤(1)的发酵过慢的醋醅分为两部分,其中一部分继续按照传统发酵工艺进行生产,另一部分醋醅则在当天进行接种种醅拌匀操作,并随后增强翻醅强度。最终在发酵结束时,测定两种工艺下的醋醅的总酸含量。结果见表2。
[0170]
实施例9应用模型控制优化食醋固态发酵进程
[0171]
(一)建模
[0172]
(1)醋醅样品的采集:选取编号为16b的发酵池的正常发酵全过程(即每一天)的醋醅样品,取样点是发酵池的中间以及四个角,从上至下打洞取样,混匀后作为醋醅样品;
[0173]
(2)醋醅样品的预处理:取步骤(1)中的3g醋醅样品,加入12ml无菌ddh2o振荡2min后静置1min,吸取2ml菌悬液用无菌水洗涤3次,获得醋醅微生物群落的菌悬液,菌悬液稀释500倍;
[0174]
(3)醋醅样品的单细胞拉曼光谱数据采集:吸取步骤(2)中的菌悬液进行单细胞拉曼光谱的采集,其中光谱采集条件为:使用532nm激光,采集范围为280-3800cm-1
,激光强度为6mw,采集时间为15s,累积次数2次,不同发酵时间的醋醅采集视野范围内的所有共计100个细胞;
[0175]
(4)单细胞拉曼光谱数据的预处理:所有测得的醋醅样品的单细胞拉曼光谱数据均需要进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、并对所有的数据进行归一化处理,设置最大峰为100;所有醋醅样品的单细胞拉曼光谱数据进行分析时均选取400~1800cm-1
的指纹区图谱;
[0176]
(5)醋醅样品的单细胞拉曼光谱数据分型划分:汇总所有发酵时间下的醋醅样品的单细胞拉曼光谱数据进行层次聚类分析,当欧氏距离为100时,把不同分支上的光谱数据作为不同的分型,共得到七个分型,得到不同发酵时间下光谱分型的相对丰度情况;
[0177]
(6)不同发酵时间的醋醅样品中微生物群落分型的曲线拟合:基于正常发酵时不同发酵时间醋醅样品的微生物群落分型数据建立模型,并使用多元线性回归方法拟合曲线以预测实际醋酸发酵时间。拟合得到的曲线如下:
[0178]
y=14.78 0.33x
opu1-0.68x
opu2
0.12x
opu4-0.59x
opu5
0.38x
opu6
0.06x
others
[0179]
(二)控制优化食醋固态发酵进程
[0180]
(1)醋醅样品的采集:选择发酵异常(即人工在发酵中期添加非酿造微生物的杂菌污染醋醅)的样品池的样品,在发酵池中间以及四个角从上至下打洞取样,混匀后作为醋醅样品;
[0181]
(2)醋醅样品的预处理:取步骤(1)中的3g醋醅样品,加入12ml无菌ddh2o振荡2min
后静置1min,吸取2ml菌悬液用无菌水洗涤3次,获得醋醅微生物群落的菌悬液,菌悬液稀释500倍;
[0182]
(3)醋醅样品的单细胞拉曼光谱数据采集:吸取步骤(2)中的菌悬液进行单细胞拉曼光谱的采集,其中光谱采集条件为:使用532nm激光,采集范围为280-3800cm-1
,激光强度为6mw,采集时间为15s,累积次数2次,不同发酵时间的醋醅采集视野范围内的所有共计100个细胞;
[0183]
(4)单细胞拉曼光谱数据的预处理:所有测得的醋醅样品的单细胞拉曼光谱数据均需要进行宇宙射线的消除、背景噪音的去除、对基线进行校正处理、并对所有的数据进行归一化处理,设置最大峰为100;所有醋醅样品的单细胞拉曼光谱数据进行分析时均选取400~1800cm-1
的指纹区图谱;
[0184]
(5)醋醅样品的单细胞拉曼光谱数据分型划分:将先前测得的不同分型的光谱数据与现有的光谱数据进行对比,按照光谱相似性将新测得的光谱进行分型的分类,得到不同发酵时间下光谱分型的相对丰度情况。
[0185]
(6)将步骤(5)中求得的不同分型数据带入步骤(一)中步骤(6)得到的曲线,计算理论发酵时间。计算结果表明:实际发酵时间是12天,理论发酵时间为28天。同时获得的分型中出现大量的opu
others
,与正常发酵下的分型存在显著差异。
[0186]
(7)工艺调整:将步骤(1)的发酵异常的醋醅分为两部分,其中一部分继续按照传统发酵工艺进行生产,另一部分醋醅则停止发酵,即时中止,避免损失。最终在发酵结束时,测定两种工艺下的醋醅的总酸含量。结果见表2。
[0187]
表2实施例7、8、9中调整工艺前后发酵结束时醋醅样品的总酸含量
[0188][0189]
对发酵过快、发酵过慢、发酵过程中污染杂菌的样品分别进行理论发酵时间的预测,并基于预测结果调整实际工艺,表3中我们可以看出及时进行工艺调整后,最终醋醅样品的总酸含量相差不大,与正常发酵下的醋醅样品的总酸含量保持一致,但是在发酵异常后未进行工艺调整的样品却存在出现异味、酸度较低、发酵失败等问题。同时在检测出污染杂菌之后及时停止发酵也有利于避免造成更大的损失。以上结果表明,我们的方法可以判断发酵过程中出现的各种问题,有助于提升工厂效率,避免经济损失。
[0190]
综上所述,本发明采集不同发酵时间的醋醅样品的单细胞拉曼光谱数据,然后利用层次聚类分析对所有醋醅样品的单细胞拉曼光谱数据进行分型的划分,得到食醋固态发酵过程中的微生物群落不同分型的组成,并以此分型结构数据使用多元线性回归方法进行曲线拟合得到预测醋醅发酵时间的曲线。此后采集新的醋醅样品后,在比较光谱的相似性后得到该天的微生物分型组成,并代入回归曲线了解其具体的发酵天数,实现快速检测醋醅样品的发酵天数的目的。我们从单细胞水平快速检测微生物群落变化,以便于实时监控食醋发酵状态,及时辅助生产的研判,为现代化智能酿醋设备的建立提供参考,具有极高的价值。
[0191]
虽然本发明已以较佳实施例公开如上,但其并非用以限定本发明,任何熟悉此技术的人,在不脱离本发明的精神和范围内,都可做各种的改动与修饰,因此本发明的保护范围应该以权利要求书所界定的为准。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。