1.本发明涉及植物育种和基因,特别是涉及到提高植物对非生物胁迫的耐受性。
2.发明背景
3.植物的胁迫可以是有生物的和非生物的因素引起的。例如,生物引发的胁迫包括病菌的感染、昆虫的取食,另一种植物的寄生如榭寄生。非生物胁迫包括,如过量的或不足的可用水、极端温度、和化学合成品如除草剂。
4.非生物胁迫是造成全球作物损失的主要原因,导致主要作物的平均产量损失超过50%(boyer,j.s.(1982)science 218:443-448;bray,e.a.等(2000)in biochemistry and molecular biology of plants,由buchanan,b.b.等人编辑,amer.soc.plant biol.,第1158-1249页)。
5.因此,需要开发增加植物对非生物胁迫耐受性的组合物和方法。本发明提供了这样的组合物和方法。
6.发明概述
7.以下实施例属于本发明所涵盖的实施例:
8.在一个实施例中,本发明提供了一种抑制dna构建体,所述构建体包含至少一个可操作地与抑制元件连接的异源调控元件。其中所述抑制元件减少了一种内源靶向多核苷酸的表达,所述内源靶向多核苷酸编码一个多肽,其包含的氨基酸序列与seq id no:3、6、9、12、15、18或21有至少90%的序列一致性。在某些实施例中,所述抑制元件包含至少100个连续编码多肽的一种多核苷酸的碱基对,所述多肽的氨基酸序列与seq id no:3、6、9、12、15、18或21有至少90%的序列一致性。在某些实施例中,所述抑制元件包含seq id no:51的多核苷酸。
9.本发明还提供了一种crisp/cas构建体,所述crisp/cas构建体包含至少一个可操作地连接grna的异源调控序列,其中所述grna是靶向包含内源bcs1-2、dnaj7、lntp10、gh17.2、duf6、atap1或pcl1基因和/或其调控元件的基因组区域,以降低内源bcs1-2、dnaj7、lntp10、gh17.2、duf6、atap1或pcl1多肽的表达或活性。在某些实施例中,所述内源基因编码的多肽的氨基酸序列与seq id no:3、6、9、12、15、18或21有至少90%的一致性。在某些实施例中,所述bcs1-2、dnaj7、lntp10、gh17.2、duf6、atap1或pcl1基因包含的多核苷酸的核苷酸序列为seq id no:1、2、4、5、7、8、10、11、13、14、16、17、19或20或其等位基因变异包括1至约10个的核苷酸变化。在某些实施例中,所述内源调控元件包含一个多核苷酸,其核苷酸序列为seq id no:74或75。
10.本发明进一步提供了一种能够减少内源bcs1-2、dnaj7、lntp10、gh17.2、duf6、atap1或pcl1多肽的表达或活性的改良植物或种子。在某些实施例中,所述改良的植物或种子包含的一种抑制dna构建体含有至少一个可操作地连接抑制元件的异源调控元件,其中所述抑制元件减少了所述内源bcs1-2、dnaj7、lntp10、gh17.2、duf6、atap1或pcl1多肽的表达。在某些实施例中,所述多肽包含的氨基酸序列与seq id no:3、6、9、12、15、18或21有至少90%的序列一致性。在某些实施例中,所述抑制元件包含至少100个能够编码氨基酸序列
与seq id no:3、6、9、12、15、18或21有至少90%序列一致性的多核苷酸的连续碱基对。在某些实施例中,所述抑制元件包含的所述多核苷酸为seq id no:51。
11.在某些实施例中,所述改良植物或种子在其基因位点上包含有一个靶向基因修饰,所述靶向基因修饰包含一个能编码bcs1-2、dnaj7、lntp10、gh17.2、duf6、atap1或pcl1多肽的多核苷酸,其中所述基因修饰能减少所述多肽的表达和/或活性。在某些实施例中,所述多核苷酸编码的多肽的氨基酸序列与seq id no:3、6、9、12、15、18或21有至少90%的序列一致性。
12.在某些实施例中,所述改良植物或种子表现出以下至少一种表型:增加的耐旱性、增加的籽粒产量、增加的非生物胁迫耐性、提高的氮素胁迫耐性、或提高的氮素利用效率(nue)。在某些实施例中,所述改良植物或种子降低bcs1-2、dnaj7、lntp10、gh17.2、duf6、atap1或pcl1多肽的表达和/或活性来提高干旱耐性、提高籽粒产量和/或提高非生物胁迫耐性。在某些实施例中,当生长在低氮条件下,所述的改良植物或种子,降低lntp10、duf6或atap1多肽的表达和/或活性,与对照植物相比较,可提高氮素胁迫耐性、或提高氮素利用效率(nue)和/或增加籽粒产量。
13.在某些实施例中,本发明中所述的组合物和方法的植物选自水稻、玉米、大豆、向日葵、高粱、油菜、小麦、苜蓿、棉花、大麦、小米、甘蔗和柳枝稷。
14.本发明还提供了一种提高植物耐旱性的方法,这种方法包括减少植物中至少一种编码bcs1-2、dnaj7、lntp10、gh17.2、duf6、atap1或pcl1多肽的多核苷酸的表达和/或活性。在某些实施例中,所述多肽的氨基酸序列与seq id no:3、6、9、12、15、18或21有至少80%的序列一致性。
15.在某些实施例中,所述提高耐旱性的方法保括:(a)向可再生植物细胞引入一个抑制dna构建体,其中所述抑制dna构建体包含有至少一个可操作地连接抑制元件的异源调控元件;(b)由可再生植物的细胞再生一个改良植物,其中所述植物包含抑制dna构建体。在某些实施例中,所述抑制元件减少氨基酸序列与seq id no:3、6、9、12、15、18或21有至少90%序列一致性的内源靶向多核苷酸的表达。在某些实施例中,所述抑制元件包含至少100个连续的碱基对,其多核苷酸编码的氨基酸序列与seq di no:3、6、9、12、15、18或21有至少90%的序列一致性。在某些实施例中,所述抑制元件包含的多核苷酸为seq id no:51。
16.在某些实施例中,所述提高耐旱性的方法包括:(a)向一个可再生植物细胞的基因组位点引入一个靶向基因修饰,所述靶向基因修饰包含一个编码bcs1-2、dnaj7、lntp10、gh17.2、duf6、atap1或pcl1多肽的多核苷酸;和(b)再生这个植物,其中所述植物在其基因组内含有所述引入的基因修饰,并能减少所述多肽的表达和/或活性。在某些实施例中,所述多肽的氨基酸序列与seq id no:3、6、9、12、15、18或21相比较时有至少80%的序列一致性。在某些实施例中,所述靶向基因修饰使用以下基因组修饰技术被引入:多核苷酸引导的核酸内切酶、crispr-cas核酸内切酶、碱基编辑脱氨酶、锌指核酸酶、转录激活因子样效应核酸酶(talen)、工程位点特异性大范围核酸酶或argonaute。在某些实施例中,所述靶向基因修饰存在于(a)编码区域;(b)非编码区域;(c)调控序列;(d)非翻译区;或(e)基因位点上(a)-(d)任意组合。所述基因位点编码多肽的氨基酸序列和seq id no:3、6、9、12、15、18或21比较时有至少80%的序列一致性。
17.在某些实施例中,所述靶向基因修饰通过crispr/cas构建体被引入,所述crispr/
cas构建体含有至少一个可操作地连接grna的异源调控序列,其中所述grna靶向所述内源bcs1-2、dnaj7、lntp10、gh17.2、duf6、atap1或pcl1基因和/或它的调控元件。
18.还提供了一种提高植物氮素胁迫耐性、氮素利用效率和/或籽粒产量的方法,所述方法包括减少植物中编码lntp10、duf6或atap1多肽的至少一个多核苷酸的表达和/或活性。在某些实施例中,所述多肽的氨基酸序列与seq id no:9、15或18有至少80%的序列一致性。
19.在某些实施例中,提高氮素胁迫耐性、氮素利用效率和/或籽粒产量的方法包括:(a)向一个可再生植物细胞引入一个抑制dna构建体,其中所述抑制dna构建体含有至少一个可操作地连接抑制元件的异源调控元件;(b)由可再生的植物细胞再生一个改良植物,其中所述植物包含所述抑制dna构建体。在某些实施例中,所述抑制元件抑制氨基酸序列与seq id no:9、15或18有至少90%的序列一致性的内源靶多核苷酸的表达。在某些实施例中,所述抑制元件含有至少100个连续的多核苷酸碱基对,其多核苷酸的氨基酸序列与seq id no:9、15或18有至少90%的序列一致性。
20.在某些实施例中,提高氮素胁迫耐性、氮素利用效率和/或籽粒产量的方法包括:(a)向可再生植物细胞的基因位点引入一个靶向基因修饰,其包含一个编码lntp10、duf6或atap1多肽的多核苷酸;和(b)再生一个植物,其中所述植物在其基因组中包含引入的基因修饰,减少所述多肽的表达和/或活性。在某些实施例中,所述多肽的氨基酸序列与seq id no:9、15或18比较时,有至少80%的序列一致性。在某些实施例中,所述靶向基因修饰使用以下基因修饰技术被引入:多核苷酸引导的核酸内切酶,crispr-cas核酸内切酶,碱基编辑脱氨酶,锌指核酸酶,转录激活因子样效应核酸酶(talen),工程位点特异性大范围核酸酶或argonaute。在某些实施例中,所述靶向基因修饰存在于:(a)编码区;(b)非编码区;(c)调控序列;(d)非翻译区;或(e)基因位点上(a)-(d)任意的组合。所述基因位点编码的多肽的氨基酸序列在和seq id no:9、15或18比较时有至少80%的序列一致性。
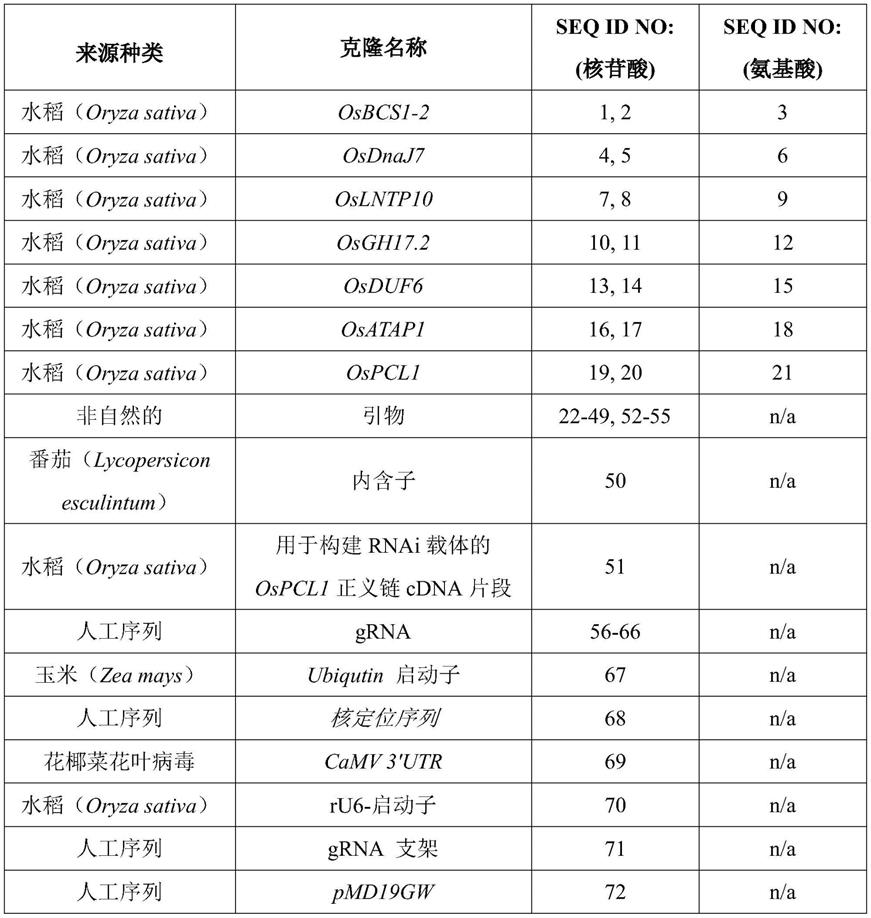
21.附图说明及序列表
22.要全面理解本发明的内容,详见以下详细说明以及构成本技术组成部分的附图和序列列表。本附件中的序列描述和序列列表符合37c.f.r.
§§
1.821和1.825中规定的专利申请中核苷酸和氨基酸序列披露的规则。序列描述包括37c.f.r.
§§
1.821和1.825中定义的氨基酸的三个字母代码,其通过引用并入本文。
23.图1显示了osbcs1-2基因基因组中sgrna分布的示意图。
24.图2显示了osdnaj7基因基因组中sgrna分布的示意图。
25.图3显示了oslntp10基因及其调控元件基因组中sgrna分布的示意图。
26.图4显示了osgh17.2基因及其调控元件基因组中sgrna分布的示意图。
27.图5显示了osduf6基因基因组中sgrna分布的示意图。
28.序列列表详述
29.30.[0031][0032]
详细描述
[0033]
本发明所述的每个参考文献的公开内容都作为引用并入本文。
[0034]
在本文中和所附的权利要求中,“一个”、“一种”、“所述”包括复数引用,除非文中另有明确规定。例如,“植物”是指多个这样的植物;“细胞”包括本领域技术人员已知的一个或多个细胞及其它对应物,以此类推。
[0035]
定义
[0036]
在本文中,植物的“增加的耐旱性”是指在干旱条件下生长时相对于参照或对照植物测量的生理或物理特征(例如产量)的任何可测量的改善。通常,当在其基因组中包含重组dna构建体或dna修饰的植物相对于参照或对照植物表现出增加的耐旱性时,参照或对照植物在其基因组中不包含重组dna构建体或dna修饰。
[0037]
在本文中,植物的“增加的氮胁迫耐受性”是指当在低氮和/或氮限制条件下生长时相对于参照或对照植物测量的生理或物理特征(例如产量)的任何可测量的改善。通常,当在其基因组中包含重组dna构建体或dna修饰的植物相对于参照或对照植物表现出增加的氮胁迫耐受性时,参照或对照植物在其基因组中不包含重组dna构建体或dna修饰。
[0038]
在本文中,“氮利用效率(nue)”是指植物吸收利用的肥料中的氮量与施用的肥料氮量之间的比率。因此,在某些实施方案中,氮利用效率的提高是指任何可检测的植物吸收利用的肥料氮的量和施用的肥料氮的量的增加。本领域普通技术人员可以使用本领域中的常规方法计算氮使用效率。
[0039]“农艺性状”是一个可测量的参数,包括但不限于:绿量、籽粒产量、生长速率、总生物量或积累速率、成熟期鲜重、成熟期干重、果实产量、种子产量、植物总氮含量、果实总氮含量、种子总氮含量、营养组织中的氮含量、植物游离氨基酸总含量、果实游离氨基酸含量、种子游离氨基酸含量、营养组织中游离氨基酸含量、植物总蛋白质含量、果实蛋白质含量、种子蛋白质含量、营养组织中的蛋白质含量、耐旱性、氮吸收、根系倒伏、收获指数、茎秆倒
和“组织优选启动子”指主要但非必须专一地在一种组织或器官中表达,而且也可在一种特定细胞或细胞型中表达的启动子。“发育调控启动子”指其活性由发育事件决定的启动子。
[0051]“可操作地连接”指核酸片段连接成单一片段,使得其中一个核酸片段的功能受到另一个核酸片段的调控。例如,在启动子能够调节核酸片段的转录时,该启动子与该核酸片段进行了可操作地连接。
[0052]“表达”指功能产物的产生。例如,核酸片段的表达可指核酸片段的转录(如转录生成mrna或功能rna)和/或rna翻译成前体或成熟蛋白质。
[0053]
在本文中,“增加的”、“增多的”等是指与对照组(例如,不包含dna修饰的野生型植物)相比,实验组(例如,具有本文所述的dna修饰的植物)中任何可检测到的增加。因此,蛋白质表达的增加包括样品中蛋白质总水平的任何可检测出的增加,可以使用本领域的常规方法如western印迹和elisa来确定。
[0054]
在本文中,“产量”是指每单位土地收获的农作物产品的量,并且可以包括指根据谷物收获时经过水分调整后每英亩或每公斤作物的蒲式耳(例如,通常为玉米15%,大米13.5%)。谷物水分是在收获时的谷物中测量的。调整后的谷物测试重量确定为每蒲式耳磅数或每株植物克数的重量,根据收获时的谷物水分水平进行调整。
[0055]“抑制dna构建体”是重组dna构建体,当其转化或稳定整合到植物的基因组中时能导致植物中靶基因的“沉默”。靶基因可以是植物的内源或转入的基因。
[0056]
本文所用的关于靶基因“沉默”通常是指抑制靶基因表达的mrna或蛋白质/酶的数量,和/或酶活性或功能蛋白质的数量。术语“抑制”,“抑制”和“沉默”在本文可互换使用,还包括降低、减少、下降、减少、抑制、消除或预防。
[0057]
抑制dna构建体在本领域中是众所周知的,并且一旦选择目的靶基因就可以容易地构建,包括但不限于共抑制构建体、反义构建体、病毒抑制构建体、发夹抑制构建体、茎环抑制构建体、产生双链rna的构建体、更一般地、rnai(rna干扰)构建体和小rna构建体、例如sirna(短干扰rna)构建体和mirna(microrna)构建体。
[0058]“反义抑制”是指产生能够抑制靶基因或基因产物表达的反义rna转录物。“共抑制”是指产生能够抑制靶基因或基因产物表达的正义rna转录物。“正义”rna是指包含mrna的rna转录本,可以在细胞内或体外翻译成蛋白质。另一种变体描述了使用植物病毒序列来指导抑制近端mrna编码序列(1998年8月20日发表的pct出版物no.wo 98/36083)。
[0059]
rna干扰(rnai)是指由短干扰rna(sirna)介导的动物中序列特异性转录后基因沉默的过程(fire等,nature 391:806(1998))。植物中的相应过程通常称为转录后基因沉默(ptgs)或rna沉默,也被称为真菌中的平息。转录后基因沉默的过程被认为是一种进化上保守的细胞防御机制,用于阻止外源基因的表达,并且通常由不同的植物群和门共享(fire等,trends genet.15:358(1999))。
[0060]
在本文中,在两个多核苷酸或多肽序列的背景下,“序列同一性”或“同一性”是指在指定的比较窗口内比对以获得最大对应性时相同的两个序列中的残基。当序列同一性百分比用于蛋白质时,人们认识到不完全相同的残基位置往往因保守的氨基酸替代而有所不同,其中氨基酸残基被其他具有相似化学性质(如电荷或疏水性)的氨基酸残基所取代因此不会改变分子的功能特性。当序列在保守取代中不同时,可以向上调整序列同一性百分比以校正取代的保守性质。通过这种保守取代而不同的序列被称为具有“序列相似性”或“相
id no:7)编码,其在tigr中注释为“表达的蛋白质”。“lntp10多肽”在本文中是指oslntp10多肽及其旁系同源基因(例如,由seq id no:96编码的seq id no:97)或来自其他生物的同源基因,例如高粱(由seq id no:98编码的seq id no:99)。
[0069]“osgh17.2”是过表达时能赋予敏旱表型的水稻多肽。osgh17.2多肽(seq id no:12)由水稻基因座loc_os01g58730.1的编码序列(cds)(seq id no:11)编码,或核苷酸序列(seq id no:10)编码,在tigr中注释为“糖基水解酶家族17,推定,表达”。gh17.2多肽在此指osgh17.2多肽及其旁系同源基因(例如,由seq id no:100编码的seq id no:101)或来自其他生物的同源基因,例如玉米(由seq id no:102编码的seq id no:103),高粱(由seq id no:104编码的seq id no:105),拟南芥(由seq id no:106编码的seq id no:107)或大豆(由seq id no:108编码的seq id no:109)。
[0070]“osduf6”是在过表达时能赋予敏旱表型的水稻多肽。osduf6多肽(seq id no:15)由水稻基因座loc_os03g02280.1的编码序列(cds)(seq id no:14)或核苷酸序列(seq id no:13)编码,在tigr中注释为“duf584结构域蛋白,假定,表达”。”本文中,duf6多肽是指osduf6多肽及其旁系同源基因(如seq id no:110编码的seq id no:111)或其他生物的同源基因,如玉米(seq id no:112编码的seq id no:113)、高粱(seq id no:114编码的seq id no:115),拟南芥(由seq id no:116编码的seq id no:117)或大豆(由seq id no:118编码的seq id no:119)。
[0071]“osatap1”是过表达时能赋予干旱敏感表型的水稻多肽。osatap1多肽(seq id no:18)由水稻基因座loc_os03g02330.1的编码序列(cds)(seq id no:17)或核苷酸序列(seq id no:16)编码,在tigr注释为“aaa型atp酶家族蛋白,假定,表达”。“atap1多肽”是指osatap1多肽及其旁系同源基因(如由seq id no:120编码的seq id no:121)或其他生物的同源基因,如玉米(由seq id no:122编码的seq id no:123)、高粱(由seq id no:124编码的seq id no:125),拟南芥(由seq id no:126编码的seq id no:127)或大豆(由seq id no:128编码的seq id no:129)。
[0072]“ospcl1”是当过表达时能赋予敏旱表型的水稻多肽。ospcl1多肽(seq id no:21)由水稻基因座loc_os03g02400.1的编码序列(cds)(seq id no:20)或核苷酸序列(seq id no:19)编码,在tigr中其注释为“含有质体蓝素样结构域的蛋白质,推定,表达”。“pcl1多肽”在本文中指ospcl1多肽及其旁系同源基因(例如,由seq id no:130编码的seq id no:131)或来自其他生物的同源基因,例如玉米(由seq id no:132编码的seq id no:133,),高粱(由seq id no:134编码的seq id no:135),拟南芥(由seq id no:136编码的seq id no:137)或大豆(由seq id no:138编码的seq id no:139)。
[0073]
本领域技术人员应当理解的,本
技术实现要素:
涵盖的不仅仅是特定的示例性序列。引起在给定位点产生化学等价氨基酸的核酸片段的改变,但是不影响编码多肽的功能特性,在领域内是众所周知的。例如,氨基酸丙氨酸(疏水性氨基酸)的密码子可以被编码另一疏水性较低残基(例如甘氨酸)的密码子或更疏水性残基(例如缬氨酸,亮氨酸,或异亮氨酸)替代。类似地,导致一个带负电荷的残基取代另一个(例如谷氨酸取代天冬氨酸),或另一种带正电荷的残基取代另一个(例如精氨酸取代赖氨酸),也可以预期产生功能上等同的产物。导致多肽分子的n-末端和c-末端部分改变的核苷酸变化也不会改变多肽的活性。所提出的每种修饰都完全在本领域的常规技术范围内,确定编码产物的生物活性的保留也是如
此。
[0074]
抑制dna构建体和crisp/cas构建体
[0075]
提供了一个能够减少bcs1-2、dnaj7、lntp10、gh17.2、duf6、atap1或pcl1多肽表达和/或活性的抑制dna构建体。在某些实施例中,所述抑制dna构建体是一种共抑制构建体、反义构建体、病毒抑制构建体、发夹抑制构建体、茎环抑制构建体、双链rna产生构建体、更一般地、rnai(rna干扰)构建体和小rna构建体如sirna(短干扰rna)构建体和mirna(microrna)构建体。
[0076]
在某些实施例中,所述抑制dna构建体含有至少一个可操作地连接抑制元件的异源调控元件,其中所述抑制元件抑制内源靶向多核苷酸的表达,所述内源性靶向多核苷酸的氨基酸序列与seq id no:3、6、9、12、15、18或21有至少90%的序列一致性。在某些实施例中,所述抑制元件含有至少100个连续碱基对,其多核苷酸的氨基酸序列与seq id no:3、6、9、12、15、18或21有至少90%的序列一致性。在某些实施例中,所述抑制元件含有的多核苷酸为seq id no:51。
[0077]
本发明中还提供了一个crispr/cas构建体,所述crispr/cas构建体含有至少一个异源调控序列可操作地连接了grna,其中所述grna的目标是在基因组区域包含有内源bcs1-2、dnaj7、lntp10、gh17.2、duf6、atap1或pcl1基因和/或其调控元件能减少内源bcs1-2、dnaj7、lntp10、gh17.2、duf6、atap1或pcl1多肽的表达或活性。在某些实施例中,所述内源基因编码的多肽的氨基酸序列与seq id no:3、6、9、12、15、18或21有至少90%的一致性。进一步,所述bcs1-2、dnaj7、lntp10、gh17.2、duf6、atap1或pcl1基因含有的多核苷酸的核苷酸序列为seq id no:1、2、4、5、7、8、10、11、13、14、16、17、19或20或包含有1到10左右的核苷酸变化的等位基因。在某些实施例中,所述内源调控元件含有的多核苷酸的核苷酸序列为seq id no:74或75。在某些实施例中,所述grna含有的序列包含一个或多个seq id no:56-66序列。
[0078]
在某些实施例中,至少一个调控元件是异源调控元件。在某些实施例中,至少一个重组dna构建体的调控元件包含一个启动子。在某些实施例中,所述启动子是一个异源启动子。
[0079]
大量启动子可以被用在本发明所述的重组dna构建体中。所述启动子可以依据期望的结果被挑选,且可以包括组成型、组织特异型、或其它用于在宿主生物体中表达的启动子。
[0080]“组成型”启动子是一种启动子,在大多数环境条件下都具有活性。组成型启动子包括例如wo 99/43838和美国专利号6072050中公开的rsyn7启动子的核心启动子和其他组成型启动子、核心camv 35s启动子(odell等(1985)nature313:810-812)、水稻肌动蛋白(mcelroy等(1990)plant cell 2:163-171)、泛素(christensen等(1989)plant mol.biol.12:619-632和christensen等(1992)plant mol.biol.18:675-689)、pemu(last等(1991)theor.appl.genet.81:581-588)、mas(velten等(1984)embo j.3:2723-2730)、als启动子(美国专利号5659026)等。其他组成型启动子包括例如美国专利号5608149、5,608,144、5,604,121、5,569,597、5,466,785、5,399,680、5,268,463、5,608,142和6177611。
[0081]
组织特异性或发育调节的启动子是一种dna序列,能选择性地调节植物细胞/组织中dna序列的表达,例如对穗发育、种子成熟或对两者都至关重要的那些细胞/组织中的dna
序列,并且通常将这种dna序列的表达限制在植物中期望的发育期(例如穗发育或种子成熟)。引起所需时间和空间表达的任何可识别的启动子可以用于本发明的方法中。
[0082]
本领域已知许多叶片优选的启动子(yamamoto等(1997)plant j.12(2):255-265;kwon等(1994)plant physiol.105:357-367;yamamoto等(1994)plant cell physiol.35(5):773-778;gotor等(1993)plant j.3:509-518;orozco等(1993)plant mol.biol.23(6):1129-1138;和matsuoka等(1993)proc.natl.acad.sci.usa90(20):9586-9590)。
[0083]
种子或胚胎特异性且可用于公开的启动子包括大豆kunitz胰蛋白酶抑制剂(kti3,jofuku和goldberg。(1989)植物细胞1:1079-1093)、convicilin、vicilin和豆科植物(豌豆子叶)(rerie,w.g。等人(1991)mol.gen.genet.259:149-157;newbigin,e.j。等人(1990)planta 180:461-470;higgins,t.j.v。等人(1988)plant.mol.biol.11:683-695)、玉米醇溶蛋白(玉米胚乳)(schemthaner,j.p。等人(1988)embo j.7:1249-1255),菜豆蛋白(豆子叶)(segupta-gopalan,c。等人(1985)proc.natl.acad.sci.82:3320-3324)、植物血凝素(豆子叶)(voelker,t。等人(1987)embo j.6:3571-3577)、b-伴大豆球蛋白和大豆球蛋白(大豆子叶)(chen,z-l等(1988)embo j.7:297-302)、谷蛋白(水稻胚乳)、大麦醇溶蛋白(大麦胚乳)(marris,c.,et al。(1988)plant mol.biol.10:359-366)、谷蛋白和麦醇溶蛋白(小麦胚乳)(colot,v。等(1987)embo j.6:3559-3564)。与嵌合基因构建中的异源编码区可操作连接的种子特异性基因的启动子在转基因植物中维持其时间和空间表达模式。这些实例包括拟南芥2s种子贮藏蛋白基因启动子以在拟南芥和甘蓝型油菜种子中表达脑啡肽(vanderkerckhove等人(1989)bio/technology 7:l929-932),豆凝集素和豆β-菜豆蛋白启动子以表达荧光素酶(riggs等(1989)plant sci.63:47-57),和小麦谷蛋白启动子表达氯霉素乙酰转移酶(colot等(1987)embo j 6:3559-3564)。
[0084]
诱导型启动子选择性表达可操作连接的dna序列以响应内源或外源刺激的出现,例如通过化合物(化学诱导剂)或响应于环境、激素、化学和/或发育信号。诱导型或调节型启动子包括例如由光、热、胁迫、洪水或干旱、植物激素、伤口或化学物质如乙醇、茉莉酮酸盐、水杨酸盐或安全剂调节的启动子。
[0085]
还考虑了合成启动子,其包括一种或多种异源调节元件的组合。
[0086]
本发明的抑制dna的启动子构建体可以是本领域已知的任何类型或类别的启动子,使得许多启动子中的任何一个可以用于表达本文公开的各种多核苷酸序列,包括天然启动子的目的多核苷酸序列。用于本发明的抑制dna构建体的启动子可以基于期望的结果进行选择。
[0087]
本发明的抑制性dna构建体还可以包括其他调控元件,包括但不限于翻译前导序列、内含子和聚腺苷酸化识别序列。在某些实施例中,抑制dna构建体还包含增强子或沉默子。
[0088]
可以将内含子序列添加到5'非翻译区、蛋白质编码区或3'非翻译区,以增加在胞质溶胶中积累的成熟信息的量。已经在植物和动物表达构建体中的转录单元中包含可剪接的内含子可使mrna和蛋白质水平的基因表达增加高达1000倍(buchman和berg。(1988)mol.cell biol.8:4395-4405;callis等(1987)genes dev.1:1183-1200)。
[0089]
植物和植物细胞
[0090]
提供了一种植物、植物细胞、植物组织、种子和籽粒,在其基因组中包含本文所述
的任何抑制dna构建体。因而所述植物、植物细胞、植物组织、种子和/或籽粒减少了编码多肽的表达。
[0091]
还提供了一种植物、植物细胞、植物组织、种子和籽粒,在其基因位点含有引入的编码本文所述多肽的基因修饰。在某些实施例中,所述多肽含有的氨基酸与seq id no:3、6、9、12、15、18或21这些氨基酸序列有至少80%的一致性。在某些实施例中,所述基因修饰降低编码多肽的活性。在某些实施例中,所述基因修饰减少编码多肽的水平。在某些实施例中,所述基因修饰同时降低编码多肽的水平和活性。
[0092]
所述植物可以是单子叶或双子叶植物,例如,水稻或玉米或大豆植物,就像是玉米杂交植物或玉米自交植物。所述植物同样可以是向日葵、高粱、油菜、小麦、苜蓿、棉花、大麦、小米、甘蔗或柳枝稷。
[0093]
在某些实施例中,所述植物当和对照植物比较时表现出增加的干旱耐性和/或氮素胁迫耐性。在某些实施例中,所述植物和对照植物相比较时表现出至少一个农艺性状的改变。
[0094]
本领域普通技术人员熟悉模拟干旱条件和评估经受模拟或自然发生的干旱条件的植物的耐旱性的方案。例如,人们可以通过给予植物比通常需要的水少或一段时间内没有水来模拟干旱条件,并且可以通过寻找生理和/或物理条件的差异来评估耐旱性,包括(但不限于)活力、生长、大小或根长,或特别是叶色或叶面积大小。评估耐旱性的其他技术包括测量叶绿素荧光、光合速率和气体交换速率。
[0095]
其它目的性状的堆叠
[0096]
在某些实施例中,本发明公开的创新的多核苷酸被设计成分子堆栈。这样,本发明中多种宿主细胞、植物、植物细胞、植物组织、种子和/或籽粒能进一步包含一种或多种期望性状。在某些实施例中,为了创造出拥有期望性状组合的植物,所述宿主细胞、植物、植物细胞、植物组织、种子和/或籽粒可以被堆栈成任何期望的多核苷酸序列的组合。在本文中,术语“性状堆叠”是指在同一植物或期望的生物体中存在多种性状。例如,“性状堆叠”可以包含序列在物理上彼此相邻的分子堆叠。在本文中,性状是指来源于特定序列或序列组的表型。在一个实例中,所述分子堆叠包含有至少一个能赋予草甘膦耐性的多核苷酸。能赋予草甘膦耐性的多核苷酸在本领域内是已知的。
[0097]
在某些实施例中,所述分子堆叠包含有至少一个能赋予草甘膦耐性的多核苷酸和至少一个能赋予第二除草剂耐性的补充核苷酸。
[0098]
在某些实施例中,具有本发明的多核苷酸序列的植物、植物细胞、种子和/或籽粒可以与一个或多个序列堆叠并赋予耐受性如:als抑制剂、hppd抑制剂、2,4-d、其他苯氧基生长素除草剂、芳氧基苯氧基丙酸除草剂、麦草畏、草铵膦除草剂、靶向protox酶的除草剂(也称为“protox抑制剂”)。
[0099]
包含有本文描述的减少多肽表达和/或活性的植物、植物细胞、植物组织、种子和/或籽粒也可以与至少一个其它性状进行结合,进一步产生包含有多种期望性状组合的植物。例如,植物、植物细胞、植物组织、种子和/或籽粒可以堆叠能编码具有杀虫和/或杀虫活性的多肽的多核苷酸,或具有本发明的多核苷酸序列植物、植物细胞、植物组织、种子和/或籽粒可以与植物抗病基因组合。
[0100]
这些堆叠的组合物的产生可以由以下任一方法,包括但不限于,任何常规育种或
遗传转化培育植物。如果所述序列通过遗传转化堆叠在植物中,期望的多核苷酸序列可以在任何时间以任何目的结合。所述性状可以由转化盒的任何组合提供的期望多核苷酸的共转化协议同时被引入。例如,如果两个序列将被引入,则两个序列可以包含在单独的转化盒(反式)中或包含在相同的转化盒(顺式)中。序列的表达可以由相同的启动子或不同的启动子驱动。在某些情况下,可能需要引入抑制目的多核苷酸表达的转化盒。这可以与其他抑制盒或过表达盒的任何组合物结合以产生植物中所需的性状组合。进一步认识到,可以使用位点特异性重组系统将多核苷酸序列堆叠在所需的基因组位置。参见例如wo99/25821,wo99/25854,wo99/25840,wo99/25855和wo99/25853,所有这些都通过引用并入本文。
[0101]
方法
[0102]
增加植物耐旱性、籽粒产量和/或氮素利用效率的方法
[0103]
为植物提供了一种增加耐旱性、提高籽粒产量和/或提高氮素利用效率的方法,包括减少至少一种多核苷酸编码的bcs1-2、dnaj7、lntp10、gh17.2、duf6、atap1或pcl1多肽的表达和/或活性。在某些实施例中,多核苷酸编码的多肽的氨基酸序列与seq id no:3、6、9、12、15、18或21有至少80%(比如,80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)的序列一致性。
[0104]
在某些实施例中,所述方法包括:(a)在一个可再生植物细胞中表达本文中所述的抑制dna构建体;和(b)再生所述植物,其基因组中含有所述抑制dna构建体。在某些实施例中所述调控元件是一种异源启动子。
[0105]
在某些实施例中,所述方法包括:(a)向一个可再生植物细胞的基因位点上引入一个靶向基因修饰来编码所述多肽;和(b)再生所述植物,植物中所述编码多肽的水平和/或活性是降低的。在某些实施例中,引入所述靶向基因修饰所使用的基因组修饰技术如下:多核苷酸引导的核酸内切酶、crispr-cas核酸内切酶、碱基编辑脱氨酶、锌指核酸酶、转录激活因子样效应核酸酶(talen)、工程位点特异性大范围核酸酶或argonaute。在某些实施例中,所述靶向基因修饰存在于基因组位点的(a)编码区;(b)非编码区;(c)调控序列;(d)非转录区;或(e)任意(a)-(d)的组合,且编码多肽的氨基酸序列与seq id no:3、6、9、12、15、18或21这些序列的一致性至少为80%。
[0106]
使用本发明中方法的植物可以是本文描述的的任何种类的植物。在某些实施例中,所述植物是玉米、大豆或水稻。
[0107]
多种方法可以用于将期望序列引入植物、植物部分、植物细胞、种子和/或籽粒中。“引入”意味着把本发明中的多核苷酸或以这种方式产生的多肽给植物、植物细胞、种子和/或籽粒,这样序列得以进入植物细胞的内部。本发明所述的方法不依靠特别的向植物、植物细胞、种子和/或籽粒中引入序列的方法,仅依靠所述多核苷酸或获得的多肽进入至少一个植物细胞的内部。
[0108]
和向植物引入多肽或多核苷酸序列的协议一样的转化协议,可以取决于植物或植物细胞的种类(即,单子叶植物或双子叶植物)有针对性地转化。合适的引入植物细胞多肽和多核苷酸的方法包括显微注射(crossway等(1986)biotechniques 4:320-334)、电穿孔(riggs等人(1986)proc.natl.acad.sci.usa83:5602-5606,农杆菌介导的转化(美国专利号5563055和美国专利号5981840)、直接基因转移(paszkowski等人(1984)embo j.3:2717-2722)和弹道粒子加速度(参见例如美国专利号4945050、美国专利号5879918、美国专利号
5886244和5932782;tomes等人(1995)在植物细胞、组织和器官培养中:基本方法,编辑gamborg和phillips(springer-verlag,berlin);mccabe等人(1988)biotechnology 6:923 926)和lec1转化(wo 00/28058)参见weissinger等人(1988)ann.rev.genet.22:421 477;sanford等人(1987)particulate science and technology5:27 37(洋葱);christou等人(1988)plant physiol.87:671 674(大豆);mccabe等人(1988)bio/technology 6:923 926(大豆);finer和mcmullen(1991)in vitro cell dev.biol.27p:175-182(大豆);singh等人1998)theor.appl.genet.96:319-324(大豆);datta等人(1990)biotechnology 8:736 740(水稻);klein等人(1988)proc.natl.acad.sci.usa 85:4305 4309(玉米);klein等人(1988)biotechnology6:559 563(玉米);美国专利号5240855;5322783;和5324646;klein等人(1988)plant physiol.91:440 444(玉米);fromm等人(1990)生物技术8:833 839(玉米);hooykaas van slogteren等(1984)nature(london)311:763-764;美国专利号5736369(谷物);bytebier等(1987)proc.natl.acad.sci.usa 84:5345-5349(百合科);de wet等(1985)在胚珠组织的实验操作中,ed.chapman等(longman,new york),第197-209页(花粉);kaeppler等(1990)plant cell reports 9:415-418and kaeppler等(1992)theor.appl.genet.84:560-566(whisker-mediated transformation);d'halluin等(1992)plant cell 4:1495-1505(electroporation);li等(1993)plant cell reports 12:250-255and christou and ford(1995)annals of botany 75:407-413(rice);osjoda等(1996)nature biotechnology 14:745-750(通过根癌土壤杆菌的玉米);所有这些都通过参考并入本文。
[0109]
在其他的实施例中,本发明中创造性的多核苷酸可以通过用病毒或病毒核酸接触植物被引入植株。通常,这些方法涉及将公开的核苷酸构建体合并入dna或rna分子中。应当认识到,所述创造性的多核苷酸序列可以开始作为病毒多蛋白的一部分合成,所述病毒多蛋白之后可以在体内或体外进行蛋白水解处理以产生所需的重组蛋白。应该进一步认识到,本文披露的启动子还包括用于病毒rna聚合酶转录的启动子。向植物引入多核苷酸并在其中进行编码表达蛋白,相关的病毒dna或rna分子在领域内是已知的。参见例如美国专利号5889191、5889190、5866785、5589367、5316931和porta等(1996)molecular biotechnology5:209-221;在此通过引用并入本文中。
[0110]
已经转化的细胞可以按照常规方式生长成植物。参见,例如,mccormick等人(1986)plant cell reports 5:81-84。然后这些植物可以生长,并用相同的转化菌株或不同的菌株授粉,并鉴定所得后代是否具有所需表型特征的组成型表达。可以生长两代或更多代以确保所需表型特征的表达被稳定维持和遗传,然后收获种子以确保已经实现所需表型特征的表达。以这种方式,本发明提供了具有本文公开的多核苷酸的转化种子(也称为“转基因种子”),例如,作为表达盒的一部分,稳定地掺入其基因组中。
[0111]
可以培养通过植物转化技术衍生的转化植物细胞,包括上面讨论的那些,以再生具有转化基因型(即本发明的多核苷酸)并因此具有所需表型的整株植物,例如增加的产量。对于玉米的转化和再生,参见gordon kamm等,the plant cell,2:603-618(1990)。
[0112]
可以使用各种方法在植物、植物部分、植物细胞、种子和/或籽粒的基因组位点处引入遗传修饰,并编码本文公开的多肽。在某些实施例中,靶向dna修饰有以下基因修饰技术:多核苷酸引导的内切核酸酶、crispr-cas内切核酸酶、碱基编辑脱氨酶、锌指核酸酶、转
录激活物样效应核酸酶(talen)、工程位点特异性大范围核酸酶或argonaute。
[0113]
在一些实施例中,可以通过在基因组中靠近所需改变的限定位置诱导双链断裂(dsb)或单链断裂来促进基因组修饰。可以使用任何可用的dsb诱导剂诱导dsb,包括但不限于talen、大范围核酸酶、锌指核酸酶、cas9-grna系统(基于细菌crispr-cas系统)、引导的cpf1内切核酸酶系统等。在一些实施方案中,dsb的引入可以与引入多核苷酸修饰模板组合。
[0114]
多核苷酸修饰模板可以通过本领域已知的任何方法引入细胞,例如但不限于瞬时引入方法、转染、电穿孔、显微注射、颗粒介导的递送、局部应用、晶须介导的递送、通过细胞穿透肽递送、或介孔二氧化硅纳米粒子(msn)介导的直接递送。
[0115]
多核苷酸修饰模板可以作为单链多核苷酸分子、双链多核苷酸分子或作为环状dna(载体dna)的一部分引入细胞中。多核苷酸修饰模板也可以连接到向导rna和/或cas内切核酸酶。
[0116]“修饰的核苷酸”或“编辑的核苷酸”是指与未修饰的核苷酸序列相比包含至少一个目的核苷酸序列的改变。这种“改变”包括,例如:(i)至少一个核苷酸的替换、(ii)至少一个核苷酸的缺失、(iii)至少一个核苷酸的插入、或(iv)任何(i)-(iii)的组合。
[0117]
术语“多核苷酸修饰模板”包括一个多核苷酸,其与未编辑的核苷酸序列相比包含至少一个核苷酸修饰。核苷酸修饰可以是至少一个核苷酸的取代、添加或缺失。任选地,多核苷酸修饰模板可以进一步包含一个同源核苷酸序列,同源基因连接至少一个核苷酸修饰,其中同源基因同源核苷酸序列与待编辑的所需核苷酸序列提供足够的同源基因。
[0118]
编辑结合dsb和修饰模板的基因组序列的过程通常包括:向宿主细胞提供识别染色体中靶序列的dsb诱导剂或编码dsb诱导剂的核酸序列,并能够在基因组序列中诱导dsb。与待编辑的核苷酸序列相比,至少一个多核苷酸修饰模板中包含有至少一个核苷酸的改变。多核苷酸修饰模板可以进一步包含位于至少一个核苷酸改变同源基因的核苷酸序列,其中同源基因序列与dsb同源基因的染色体区域基本同源。
[0119]
核酸内切酶可以通过本领域已知的任何方法提供给细胞,例如但不限于瞬时引入方法、转染、显微注射和/或局部应用或间接通过重组构建体。内切核酸酶可以作为蛋白质或向导多核苷酸复合物,直接提供给细胞或通过重组构建体间接提供。内切核酸酶可以瞬时引入细胞中,或者可以使用本领域已知的任何方法掺入宿主细胞的基因组中。在crispr-cas系统的情况下,如2016年5月12日公布的wo2016073433中所述,可以用细胞穿透肽(cpp)促进内切核酸酶和/或引导的多核苷酸摄入细胞。
[0120]
除了通过双链断裂技术进行修饰之外,使用碱基编辑技术实现对没有这种双链断裂的一个或多个碱基的修饰,参见例如gaudelli等,(2017)可编程碱基编辑基因组dna中的a*t至g*c而没有dna切割。自然551(7681):464-471;komor等,(2016)基因组dna中靶碱基的可编程编辑,无需双链dna切割,nature 533(7603):420-4。
[0121]
这些融合物含有dcas9或cas9切口酶以及合适的脱氨酶,它们可以将例如胞嘧啶转化为尿嘧啶而不诱导靶向dna的双链断裂。然后通过dna复制或修复将尿嘧啶转化为胸腺嘧啶。具有目标灵活性和特异性的改进的基础编辑器用于编辑内源基因位点以产生目标变异并提高谷物产量。同样,腺嘌呤碱基编辑器可使腺嘌呤变为肌苷,然后通过修复或复制将其转化为鸟嘌呤。因此,目标碱基改变,即c
·
g到t
·
a转换和a
·
t到g
·
c转换在使用适当的
站点特定基础编辑器的多个位置进行。
[0122]
在一个实施例中,碱基编辑是一种基因组编辑方法,能够在目标基因组位点将一个碱基对直接转化为另一个碱基对,而不需要双链dna断裂(dsbs)、同源定向修复(hdr)过程、或外部供体dna模板。在一个实施例中,碱基编辑器包括(i)催化受损的crispr-cas9突变体,其突变使得其核酸酶结构域之一不能形成dsb;(ii)单链特异性胞苷/腺嘌呤脱氨酶,其在由cas9产生的单链dna泡中的适当核苷酸窗口内将c转化为u或a转化为g;(iii)阻碍尿嘧啶切除的尿嘧啶糖基化酶抑制剂(ugi)和降低碱基编辑效率和产物纯度的下游过程;(iv)切口酶活性以切割未编辑的dna链,然后进行细胞dna修复过程以替换含g的dna链。
[0123]
如本文所用,“基因组区域”是细胞基因组中染色体的区段,其存在于靶位点的任一侧,或者还包含靶位点的一部分。基因组区域可至少包含5-10、5-15、5-20、5-25、5-25、5-30、5-35、5-40、5-45、5-45、5-50、5-55、5-55、5-60、5-60、5-65、5-70、5-75、5-80、5-85、5-90、5-95、5-100、5-200、5-100、5-200、5-300、5-400、5-500、5-10、5-15、5-15、5-15、5-15、5-15、5-15、5-15、5-15、5-15、5-15、5-15、5-15-20、5-20、5-20、5-25、5-25、5-25、5-25、5
‑‑
1800、5-1900、5-2000、5-2100、5-2200、5-2300、5-2400、5-2500、5-2600,5-2700、5-2800.5-2900、5-3000、5-3100或更多碱基,以使基因组区域具有足够的同源性,以与相应的同源性区域进行同源重组。
[0124]
tal效应核酸酶(talen)是一类序列特异性核酸酶,可用于在植物或其他生物基因组的特定靶序列上产生双链断裂(miller等(2011)nature biotechnology 29:143-148)。
[0125]
内切核酸酶是切割多核苷酸链内的磷酸二酯键的酶。核酸内切酶包括在特定位点切割dna而不破坏碱基的限制性内切酶,以及与限制性内切酶一样在特定识别位点结合和切割的大范围核酸酶(也称为归巢核酸内切酶(hease)),但是大范围核酸酶的识别位点通常更长,约18bp或更多(专利申请pct/us12/30061,于2012年3月22日提交)。大范围核酸酶根据保守序列基序分为四个家族,这些家族是laglidadg,giy-yig,h-n-h和his-cys-box家族,这些基序参与金属离子的配位和磷酸二酯键的水解。酶因其长识别位点和耐受某些序列而著称大范围核酸酶的命名约定与其他限制性核酸内切酶的命名约定相似,大范围核酸酶的特征还在于前缀f-,i-或pi-,分别由独立的orf,内含子和内含子编码。重组过程的一步涉及多核苷酸在识别位点处或附近切割。切割活性可用于产生双链断裂。对于位点特异性重组酶及其识别位点的综述,参见sauer(1994)curr op biotechnol 5:521-7;和sadowski(1993)faseb 7:760-7。在一些实例中,重组酶来自整合酶或分解酶家族。
[0126]
锌指核酸酶(zfn)是由锌指dna结合结构域和双链断裂诱导剂结构域组成的工程化双链断裂诱导剂。识别位点特异性由锌指结构域赋予,其通常包含两个、三个或四个锌指,例如具有c2h2结构,然而其他锌指结构锌指结构域适用于设计特异性结合所选多核苷酸识别序列的多肽.zfn包括与非特异性内切核酸酶结构域连接的工程化dna结合锌指结构域,例如来自类型的核酸酶结构域iis内切核酸酶,如foki.addi功能性功能可以与锌指结合结构域融合,包括转录激活因子结构域、转录抑制因子结构域和甲基化酶。在一些实例中,核酸酶结构域的二聚化是切割活性所必需的。在目标dna每个锌指识别三个连续的碱基对。例如,3-指结构域识别9个具有核酸酶的二聚化要求的、连续核苷酸的序列,使用两组锌指三联体结合18个核苷酸的识别序列。
[0127]
使用dsb诱导剂(例如cas9-grna复合物)进行基因组编辑已被描述,例如2015年3
月19日发布的美国专利申请us 2015-0082478 a1,2015年2月26日发布的wo2015/026886 a1,2016年1月14日发布的wo2016007347和2016年2月18日发布的wo201625131,所有这些都是在此作为参考并合并入本文中。
[0128]
实例
[0129]
以下是本发明某些方面的具体实施例的实例。这些实例仅用于说明目的,并不以任何方式显示本发明的范围。
[0130]
实例1
[0131]
敏旱基因的克隆和载体构建
[0132]
使用含有来自花椰菜花叶病毒35s(camv 35s)启动子的四个多聚增强子元件的双元构建体,并且水稻激活标签群体(atls)由四个粳稻(oryza sativa ssp.japonica)品种(中花11、超优1号、台中65和日本晴)通过农杆菌介导的转化方法转化产生,如lin和zhang((2005)plant cell rep.23:540-547)所述。产生的转基因株系成长并收获转基因种子以形成水稻激活标签群体。
[0133]
敏旱标签株系(atls)在田间重复试验中得到证实,其t-dna插入位点通过连接介导的巢式pcr(osbcs1-2、oslntp10)或质粒拯救法(osgh17.2、osatap1、osdnaj7、osduf6)或反向pcr方法(ospcl1)来测定。克隆t-dna左边界和右边界附近的基因,并通过田间筛选重现功能基因。本文仅显示了概括的功能基因。并基于表2中所示基因的loc id,设计引物用于克隆水稻干旱敏感基因osbcs1-2、osdnaj7、oslntp10、osgh17.2、osduf6、osatap1、ospcl1。
[0134]
表2.水稻基因名称、基因ids(来自于tigr)和构建体ids
[0135][0136][0137]
使用柱试剂盒在琼脂糖凝胶电泳后提取pcr扩增产物,然后与ta克隆载体连接。通过测序确认这些构建体中的序列和方向。将每个基因克隆到植物双元构建体中。
[0138]
实例2
[0139]
转基因水稻株系的转化和基因表达分析
[0140]
如lin和zhang((2005)plant cell rep.23:540-547)所述,通过农杆菌介导的转化,用实施例1中制备的载体或空载体(dp0158)转化中花11号(oryza sativa l.)。将转化实验室产生的转基因幼苗(t0)移植到田间以获得t1种子。筛选t1和随后的t2种子以确认转
化,并将阳性鉴定的转基因种子用于接下来的性状筛选。
[0141]
通过rt-pcr测定转基因水稻植物叶中的基因表达水平。设计引物用于过表达转基因水稻中osbcs1-2、osdnaj7、oslntp10、osgh17.2、osduf6、osatap1和ospcl1基因的rt-pcr。将zh11-tc(组织培养的zh11水稻)中的表达水平设定为1.00,并将转基因植物中的表达水平与zh11-tc进行比较。基于ef-1αmrna水平对基因表达进行标准化,并且基因表达分析的结果在下表3中提供。
[0142]
表3.转基因水稻植株中相对表达水平增幅
[0143]
基因名称构建体id相对表达水平增幅osbcs1-2dp0962从38.25到4456.27osdnaj7dp0396从6.4到4052.74oslntp10dp0866从0.43到237.78osgh17.2dp0334从37794.93到446767osduf6dp0786从1.59到125.82osatap1dp0329从2.01到17.67ospcl1dp0780从1.75到1994.24
[0144]
实例3
[0145]
转基因水稻植株的表型
[0146]
实例2中的转基因水稻和zh11-tc以及dp0158水稻植株需测定以下指标:(a)耐旱性、(b)水充足条件下的籽粒产量、(c)低氮胁迫/氮素利用效率。
[0147]
实例2植物产生的t2种子在32℃温度下经800ppm多菌灵处理8小时并清洗3-5次后,在32℃温度下,在水中浸泡16小时,然后在35-37℃的烘箱中催芽18小时。发芽的种子用于以下测定:
[0148]
耐旱-发芽的种子种植在苗床中。在三叶期,将幼苗移植到测试田中,每个转基因株系重复4次,每次重复种植10株,并将4个重复种植在同一田块中。zh11-tc和dp0158幼苗在同一区块的转基因株系附近,并在统计分析中用作对照。水稻植物通过常规做法使用杀虫剂和肥料进行管理。在穗开始阶段停止浇水,以便根据天气条件(温度和湿度)在开花阶段给予干旱胁迫。使用tdr30(spectrum technologies,inc。)以每块约10个位点每4天测量土壤水含量。在实验期间观察并记录植物表型。表型包括抽穗期、卷叶度、干旱敏感性和耐旱性。中午特别注意卷叶度。在生长季节结束时,从每行的中间收获每个转基因株系的6株代表性植物,并测量每株植物的籽粒产量。使用混合线性模型对谷物产量数据进行统计分析。
[0149]
水分充足条件下的籽粒产量-将发芽的种子种植在苗床中。在三叶期,将幼苗移植到试验田中,每个转基因株系重复4次,每个重复种植40株,并将4个重复种植在同一田块中。zh11-tc、dp0158和阴性幼苗在同一区块的转基因株系附近,并在统计分析中用作对照。水稻植物通过常规做法使用杀虫剂和肥料进行管理。在生长季节结束时,从每行的中间收获每个转基因株系的代表性植物,并测量每株植物的籽粒产量。使用混合线性模型对籽粒产量数据进行统计分析。
[0150]
低氮耐性/氮利用效率-一个氮水平:实验中设定n-0(使用无氮肥料)。将发芽的种子种植在苗床中。在三叶期,将幼苗移植到试验田中,每个转基因株系重复4次,每次重复种
植10株,并将4个重复种植在同一田块中。将zh11-tc、dp0158和阴性植物种植在同一块转基因株系附近,并用作统计分析中的对照。水稻试验采用常规农药管理,但施用磷肥和钾肥进行n-0处理。
[0151]
在季节结束时,从每行的中间收获每个转基因品系的6个代表性植物,并测量每株植物的籽粒产量。通过asreml程序使用混合线性模型统计分析每株植物的籽粒产量数据。基于分析选择阳性转基因株系(p《0.1)。
[0152]
这些研究的结果在表4中提供,其提供了每种构建体的转基因系的组合数据。
[0153]
表4.转基因水稻植株的农艺性状
[0154][0155]
dp0962-转基因水稻植株两年分别在海南和宁夏进行了五次测试。其中四次试验表明,与对照相比,dp0962-转基因水稻在田间干旱条件下单株平均产量下降。在osbcs1-2
高表达株系中观察到卷叶表型,而osbcs1-2低表达株系表现出良好的结实率,且没有卷叶表型。这些结果表明,dp0962-转基因植物的产量和干旱敏感表型与osbcs1-2基因表达水平相关。如表4所示,宁夏田间12个事件中有9个显示单株产量显著下降(p《0.1)。这12个事件的单株平均产量分别比zh11-tc和dp0158对照低60%和40%。产量和表型观察一致表明osbcs1-2是水稻干旱敏感基因。
[0156]
dp0396-转基因水稻两年内在海南、北京和宁夏进行了四次测试。所有实验一致表明,在田间干旱条件下,dp0396-转基因水稻单株平均产量下降,osdnaj7高表达株系也可观察到卷叶表型。在海南田间试验中,6个osdnaj7高表达株系中的3个表现出单株植物的产量明显低于zh11-tc和dp0158对照。这3个事件的平均单株产量分别比zh11-tc和dp0158对照低68%和56%。但另外3个osdnaj7低表达株系的单株产量比dp0158对照高,这3个事件的单株平均产量分别比zh11-tc和dp0158对照高15%和58%(表4)。产量和表型观察一致表明osdnaj7是水稻干旱敏感基因。
[0157]
dp0866-转基因水稻植株两年内在海南和宁夏进行了三次测试。所有实验一致表明,在田间干旱条件下,oslntp10高表达株系单株平均产量下降,而oslntp10低表达株系单株平均产量增加。在海南田间试验中,9个低表达株系中有5个观察到良好的结实率,并且单株植物的产量明显高于对照zh11-tc和dp0158。这5个低表达株系的平均产量分别比zh11-tc和dp0158对照高52%和89%。另外4个高表达株系单株植物的产量比zh11-tc和dp0158对照低,平均产量分别比zh11-tc和dp0158对照低29%和11%。这4个低表达株系的平均单株植物的产量显示在表4中。在田间低氮条件下,dp0866-转基因水稻植株在北京进行了三次测试。所有实验均获得一致的结果。在北京的第一年,9个高表达株系中有4个的单株产量低于zh11-tc和dp0158对照。这4个高表达事件的平均产量分别比zh11-tc和dp0158对照低36%和23%。另外5个低表达株系单株产量相对于对照是增加的。这5个低表达系的单株平均产量分别比zh11-tc和dp0158对照高2%和24%(表4)。产量和表型观察一致表明,oslntp10是水稻干旱敏感和低氮敏感基因。
[0158]
dp0334-转基因水稻植株一年内在海南和宁夏进行了两次测试。所有实验一致表明,在田间干旱条件下,在dp0334-转基因株系中过表达osgh17.2基因降低了单株植物的产量。在宁夏田间,观察到12个株系的卷叶表型,平均单株产量显著低于zh11-tc和dp0158对照。这12个株系的平均单株产量分别比zh11-tc和dp0158对照低77%和66%(表4)。产量和表型观察一致表明osgh17.2是水稻干旱敏感基因。
[0159]
dp0786-转基因水稻植株两年内在海南和宁夏的田间干旱条件下进行了三次测试。所有实验一致表明,在田间干旱条件下,dp0786-转基因株系中osduf6基因的过表达降低了单株植物的产量。在宁夏田间,6个株系中有3个株系的单株产量显著低于zh11-tc和dp0158对照。这3个阳性株系的平均单株产量分别比zh11-tc和dp0158对照低72%和70%(表4)。在田间低氮条件下,dp0786-转基因株系在北京进行了两次测试。两次实验都获得了一致的结果。在北京的第二年,所有12个株系的单株产量均低于zh11-tc和dp0158对照。12个株系中的9个弹株植物的产量显著低于zh11-tc和dp0158对照。这12个株系的平均单株产量分别比zh11-tc和dp0158对照低72%和70%(表4)。这些数据一致表明osduf6是一种水稻干旱敏感和低氮敏感基因。
[0160]
dp0329-转基因水稻植物一年内分别在海南和宁夏进行了两次测试。两次实验一
致表明,osatap1的过表达降低了田间干旱条件下每株植物的平均产量。在海南田间实验中,12个株系中有10个株系的单株产量显著低于zh11-tc和dp0158对照。如表4所示,这10个株系的单株平均产量分别比zh11-tc和dp0158对照低67%和55%。在田间低氮条件下,dp0329-转基因株系在北京进行了一次测试。如表4中的结果,dp0329-转基因植物的单株植物的平均产量明显低于zh11-tc和dp0158对照的平均产量。12个事件中的9个表现出平均单株产量显著低于zh11-tc对照,并且12个株系中的3个表现出平均单株植物产量明显低于dp0158对照。这些数据一致表明osatap1是一种水稻干旱敏感和低氮敏感基因。
[0161]
dp0780-转基因水稻植株两年分别在海南和宁夏进行了三次测试。所有实验一致表明,ospcl1基因的过表达降低了田间干旱条件下单株植物的产量。在海南田间试验中,7个株系中有2个株系的单株产量明显低于zh11-tc和dp0158对照。如表4所示,这7个株系的平均产量分别比zh11-tc和dp0158对照低21%和30%。这些数据一致表明ospcl1是一种水稻干旱敏感基因。
[0162]
总之,这些结果表明osbcs1-2、osdnaj7、oslntp10、osgh17.2、osduf6、osatap1和ospcl1转基因水稻植株在营养生长期受到干旱胁迫后,表现出干旱敏感表型,且单株产量低于对照。转基因水稻植株的产量和干旱敏感表型分别与osbcs1-2、osdnaj7和osgh17.2基因表达水平相关。osbcs1-2、osdnaj7和osgh17.2高表达株系单株平均产量下降,而osbcs1-2、osdnaj7和osgh17.2低表达株系单株平均产量增加。oslntp10、osduf6和osatap1转基因水稻植株对田间低氮胁迫敏感。
[0163]
实例4
[0164]
rnai和crisp/cas9载体构建体和转化
[0165]
rnai构建体的构建:
[0166]
使用模板(seq id no:51)和引物(seq id no:52-55)克隆ospcl1基因的正向cdna片段和反向cdna片段。然后将正向cdna片段、内含子(seq id no:50)和反向cdna片段连接在一起,并与pmd19gw载体(seq id no:72)连接。通过测序确认构建体中的序列和方向后,将rnai结构片段(正向cdna内含子反向cdna)克隆到pcambia1300dsred-35s-gw构建体(seq id no:73)中以获得rnai构建体(dp3022)。
[0167]
crispr/cas9构建体的构建:
[0168]
在crispr-cas9系统中,玉米ubi启动子(seq id no:67)驱动cas9蛋白的优化编码序列(seq id no:68);camv35s 3'-utr(seq id no:69)提高cas9蛋白的表达水平;水稻u6启动子(seq id no:70)驱动grna的表达(grna支架,seq id no:71)。
[0169]
使用可用工具分析靶基因组序列以产生候选sgrna序列。sgrna序列也可以由其他网络工具生成,包括但不限于,网站cbi.hzau.edu.cn/crispr/和crispr-plant,可在线获得。
[0170]
分析osbcs1-2(seq id no:1和seq id no:2)、osdnaj7(seq id no:4和seq id no:5)、oslntp10(seq id no:7和seq id no:8)、osgh17.2(seq id no:10和seq id no:11)和osduf6(seq id no:13和seq id no:14)基因的序列以产生sgrna序列。sgrna序列列于seq id no:56-66中。
[0171]
一个sgrna可用于制备基因组编辑构建体;sgrna可以选自片段的任何区域,例如启动子、外显子、内含子和utr。单个sgrna可以将cas9酶引导至靶区域并在靶向dna序列处
产生双链断裂,触发非同源末端连接(nhej)修复机制和同源定向修复(hdr),并且它经常在目标点诱导随机插入、删除和替换。例如,该编辑可以去除调节元件区域中的表达元件以降低mrna水平,或者可以引起多肽的结构变化,这可能导致蛋白质活性降低。
[0172]
两个sgrna可用于制备基因组编辑构建体。可以从片段的任何区域选择两个或更多个sgrna,例如启动子、外显子、内含子和utr。该构建体可导致片段缺失或点突变(小插入、缺失和取代)。
[0173]
表5显示了osbcs1-2、osdnaj7、oslntp10、osgh17.2和osduf6基因及其调控元件的基因组编辑的靶基因、靶位点和特异性链。dp3039和dp2801分别是用于编辑oslntp10和osgh17.2的一个目标位点的构建体。dp2805、dp3092和dp3093分别是用于编辑osbcs1-2、osdnaj7和osduf6的两个目标位点的构建体。dp3041是用于编辑oslntp10基因的三个靶位点的构建体。为了编辑一个靶位点,首先将靶引物退火以形成短双链片段,然后将该片段插入phsg396gw-urs-uc-mpcas9&ru6-dsred构建体中。在确认sgrna片段的核苷酸序列后,将sgrna片段与pcambia1300dsred-gw-adv.ccdb的表达载体连接。同时,为了编辑两个或多个靶位点,不同的引物应首先退火以形成双链片段,然后堆叠在一起并插入phsg396gw-urs-uc-mpcas9和ru6-dsred构建体中,然后与pcambia1300dsred-gw-adv.ccdb verctor。
[0174]
表5.用于构建crispr/cas9构建体的grnas
[0175][0176][0177]
如实例2所述,将rnai和crispr/cas9构建体转化到水稻植物中。
[0178]
实例5
[0179]
鉴定基因组编辑水稻植株中的切割位点和干旱敏感基因的修饰
[0180]
设计引物以使用转化幼苗的基因组dna作为模板扩增基因组编辑靶位点附近的靶序列。对扩增的靶序列进行测序以确认编辑结果。产生修饰,如插入至少一个核苷酸、缺失至少一个核苷酸、替换至少一个核苷酸,这导致编码序列的提前终止,翻译移位和/或缺失至少一个氨基酸残基。
[0181]
在dp2805水稻植株中,在预期的位点鉴定了10个修饰。9个突变体导致orf提前终止,进一步导致长度为147至387个氨基酸的残基;1个突变体导致27个核苷酸的缺失和7个氨基酸残基的缺失。
[0182]
在dp3092水稻植株中,在预期的位点鉴定了6个修饰。两个突变体导致翻译移位,但翻译并未在原始终止密码站点停止;1个突变体导致翻译移位,但翻译在原始终止密码站点停止;2个突变体导致orf提前终止,进一步导致长度为553个氨基酸的残基;1个突变体导致插入一个核苷酸并缺失169个核苷酸,最后终止于原始的终止密码位点并导致56个氨基酸残基的缺失。
[0183]
在dp3039水稻植株中,在预期的位点鉴定了5个修饰。三个突变体导致翻译移位,但翻译并未在原始终止密码站点停止;1个突变体通过20个核苷酸的替换导致orf的早期终止,并进一步导致长度为74个氨基酸的残基;1个突变体导致orf提前终止,并进一步导致长度为28个氨基酸的残基。
[0184]
在dp3041水稻植株中,在预期的位点鉴定出29个修饰。六个突变体导致翻译移位,但翻译并未在原始终止密码站点停止;6个突变体导致orf提前终止,进一步导致29个氨基酸残基的长度;在启动子和/或utr区域编辑了17个突变体。
[0185]
在dp2801水稻植株中,在预期的位点鉴定出17个修饰。十个突变体导致utr区缺失1至732个核苷酸;3个突变体导致在utr区插入一个核苷酸;4个突变体导致utr区核苷酸片段置换。
[0186]
在dp3093水稻植株中,在预期的位点鉴定了9个修饰。所有突变体均导致移码突变,并且未在原始终止密码位点停止。
[0187]
基因组编辑的纯合水稻植物用于以下功能测试。
[0188]
实例6
[0189]
基因表达抑制或基因组编辑水稻植株的特征
[0190]
在田间筛选t2种子以验证降低基因表达是否可以增强基因表达抑制或基因组编辑的水稻植物的耐旱性。筛选方法在实施例3中有描述。zh11-tc、dp0158或基因组编辑的阴性水稻植物在同一块中的改良株系附近,并在统计分析中用作对照。表6提供了这些研究的结果。
[0191]
表6.基因表达抑制或基因编辑水稻植株的农艺性状
[0192][0193][0194]
osbcs1-2基因编辑植物(dp2805)两年内在宁夏和海南的田间干旱和水分充足条
件下进行了三次测试。所有实验一致表明,在田间干旱和水分充足的条件下,dp2805植物的单株平均产量增加。在海南田间干旱条件下,11个品系中有8个产量显著高于dp0158对照。这8个阳性系的单株平均产量比dp0158对照高53%。在海南田间水分充足的条件下,15个品系中有8个品系的单株产量显著高于dp0158对照。这8个阳性系的单株平均产量比dp0158对照高27%。所有这些来自海南田间试验的结果如表6所示。
[0195]
osdnaj7基因编辑植物(dp3092)分别在宁夏和海南的田间干旱和水分充足条件下,一年内进行了两次测试。两个实验一致表明,在田间干旱和水分充足的条件下,dp3092植物提高了单株植物的平均产量。在宁夏田间干旱条件下,19个品系中有5个品系的单株产量显著高于zh11-tc和dp0158对照。这19个品系的单株平均产量分别比zh11-tc和dp0158对照高10%。在宁夏田间水分充足的条件下,18个品系中有7个品系的单株产量明显高于zh11-tc和dp0158对照。这18个品系的单株平均产量分别比zh11-tc和dp0158对照高3%和21%。宁夏田间试验的所有这些结果如表6所示。
[0196]
oslntp10基因编辑的植物(dp3039)在海南的田间干旱和水分充足的条件下进行了一次测试。实验表明,dp3039植株在田间干旱和水分充足条件下提高了单株平均产量。在田间干旱条件下,8个品系中的6个显示出比zh11-tc和dp0158对照显著增加的单株产量。这8个品系的单株平均产量分别比zh11-tc和dp0158对照高9%和45%。在田间水分充足的条件下,10个品系中的6个显示出比zh11-tc和dp0158对照明显增加的单株产量。这10个品系的单株平均产量分别比zh11-tc和dp0158对照高5%和22%。所有这些来自海南田间试验的结果如表6所示。
[0197]
oslntp10基因编辑的植物(dp3041)在海南的田间干旱和水分充足的条件下进行了一次测试。实验表明,dp3041植物在田间干旱和充分浇水条件下提高了单株平均产量。在田间干旱条件下,11个品系中的7个显示出比zh11-tc和dp0158对照明显增加的单株产量。这11个品系的单株平均产量分别比zh11-tc和dp0158对照高5%和39%。在田间充分浇水的条件下,13个品系中的6个显示出比zh11-tc和dp0158对照明显增加的单株产量。这13个品系的单株平均产量分别比zh11-tc和dp0158对照高5%和21%。所有这些来自海南田间试验的结果如表6所示。
[0198]
osgh17.2基因编辑植物(dp2801)在宁夏田间干旱和水分充足的条件下进行了一次测试。实验表明,dp2801植株提高了单株平均产量。在田间干旱条件下,19个品系中的5个显示出比zh11-tc和dp0158对照显著增加的单株产量。这19个品系的单株平均产量均比zh11-tc和dp0158对照高10%。在田间充分浇水的条件下,18个品系中的7个分别显示出比zh11-tc和dp0158对照明显增加的单株产量。这18个品系的单株平均产量分别比zh11-tc和dp0158对照高20%和7%。宁夏田间试验的所有这些结果如表6所示。
[0199]
osduf6基因编辑植物(dp3093)在宁夏田间干旱条件下进行了一次测试。实验表明,dp3093植株提高了单株平均产量。在田间干旱条件下,10个品系中的4个显示出比zh11-tc和dp0158对照明显增加的单株产量。这10个品系的单株平均产量分别比zh11-tc和dp0158对照高6%和13%。宁夏田间试验的所有这些结果如表6所示。
[0200]
ospcl1基因抑制植物(dp3022)在宁夏和海南的田间干旱条件下进行了两次测试。实验表明,dp3022植株提高了单株平均产量。在宁夏田间,7个品系中有5个品系的单株产量显著高于zh11-tc和dp0158对照。如表6所示,这7个品系的单株平均产量分别比zh11-tc和
dp0158对照高18%和20%。
[0201]
总之,这些结果表明osbcs1-2、osdnaj7、oslntp10、osgh17.2、osduf6、osatap1和ospcl1基因编辑或基因抑制水稻植株在干旱胁迫和/或正常条件下,比对照表现出营养期耐旱性和单株产量的增加。
[0202]
实例7
[0203]
玉米中水稻同源敏旱基因表达减少的转化和评估
[0204]
如本文所述,可以修饰玉米植物(例如抑制dna构建体或靶向遗传修饰)以降低来自玉米同源基因的表达和/或活性。抑制元件在玉米转化载体中的表达可以在组成型启动子例如玉米泛素启动子的控制下(christensen等人(1989)plant mol.biol.12:619-632和christensen等人(1992))plant mol.biol.18:675-689)或在另一个启动子的控制下,例如应激反应性启动子或组织优选的启动子。基本上如国际专利公开wo2009/006276中所述,可以通过粒子轰击将抑制dna构建体引入玉米细胞中。或者,玉米植物可以通过农杆菌介导的转化用抑制性dna构建体转化,基本上如zhao等人在meth.mol.biol.318:315-323(2006)和zhao等人,mol.breed.8:323-333(2001)和1999年11月9日发布的美国专利号5981840中所述。或者,可以使用本领域已知的方法在编码同源蛋白的基因组基因位点处引入靶向遗传修饰。
[0205]
再生植物的后代,例如t1植物,可以经受基于土壤的干旱胁迫。使用图像分析,可以在干旱胁迫之前和期间多次测量植物面积、体积、生长速率和颜色。相对于对照,干旱胁迫期间萎或叶面积减少,黄色积累减少和/或生长速率增加的显着延迟将被认为是该基因在玉米中起作用以增强耐旱性的证据。
[0206]
实例8
[0207]
高粱中水稻同源敏旱基因表达减少的评估
[0208]
如本文所述,高粱可以被修饰(例如抑制dna构建体或靶向遗传修饰)以降低来自高粱的同源基因的表达和/或活性。
[0209]
再生植物的后代,例如t1植物,可以经受基于土壤的干旱胁迫。使用图像分析,可以在干旱胁迫之前和期间多次测量植物面积,体积,生长速率和颜色。相对于对照,干旱胁迫期间叶片萎蔫或叶面积减少、黄色积累减少和/或生长速率增加等的显著延迟被认为是该基因在高粱中起作用并增强耐旱性的证据。
[0210]
实例9
[0211]
大豆中水稻同源敏旱基因表达减少的评估
[0212]
如本文所述,大豆植物可以通过修饰(例如,抑制dna构建体或靶向遗传修饰)以降低来自大豆的同源基因的表达和/或活性。
[0213]
再生植物的后代,例如t1植物,可以经受基于土壤的干旱胁迫。使用图像分析,可以在干旱胁迫之前和期间多次测量植物面积、体积、生长速率和颜色。相对于对照,干旱胁迫期间叶片萎蔫或叶面积减少、黄色积累减少和/或生长速率增加等的性状延迟将被认为是该基因在大豆中起作用以增强耐旱性的证据。
[0214]
实例10
[0215]
拟南芥中水稻敏旱基因的实验室干旱筛选
[0216]
为了解水稻抗旱基因是否能提高双子叶植物的耐旱性或其他性状,本文所述的水
稻载体可以通过农杆菌介导的转化程序使用花浸法转化到拟南芥(哥伦比亚)中,并鉴定转基因植物(clough,s.t.和bent,a.f.(1998)the plant journal 16,735-743;zhang,x.等(2006)nature protocols 1:641-646)。
[0217]
再生植物的后代,如t1植物,可以受到基于土壤的干旱胁迫。使用图像分析,可以在干旱胁迫之前和期间多次测量植物面积、体积、生长速率和颜色。相对于对照,干旱胁迫期间叶片萎蔫或叶面积减少、黄色积累减少和/或生长速率增加等的性状延迟被认为是该基因在双子叶植物中起作用以增强耐旱性的证据。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。
