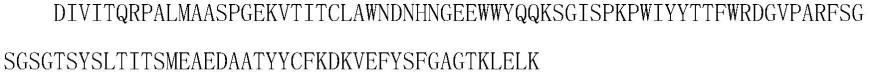hiv-1蛋白酶和逆转录酶区、整合酶区基因型的检测方法
技术领域
1.本发明属于生物技术领域,特别涉及一种hiv-1蛋白酶和逆转录酶区、整合酶区基因型的检测方法。
背景技术:
2.艾滋病(acquired immune deficiency syndrome,aids)是一种由人类免疫缺陷病毒(human immunodeficiency virus,hiv)感染cd4
t淋巴细胞导致机体免疫功能下降而引起的慢性传染性疾病。抗逆转录病毒治疗(antiretroviral therapy,art)作为一种降低hiv感染者死亡、减少hiv传播的有效手段,正在全球范围广泛推行。然而由于hiv具有高度的遗传变异性和极快的病毒复制速率,导致hiv容易出现耐药突变并在药物选择压力下成为优势毒株,进而导致hiv感染者对抗病毒治疗药物耐药。
3.目前hiv基因型耐药检测方法多采用一代测序检测耐药突变位点。该方法通过双脱氧核苷酸末端终止法进行测序,由sanger等人于1977年发明,又称sanger测序法(sanger sequencing,ss)。sanger测序一直是流行病学调查或临床中hiv耐药检测的通用方法,但此方法的检测通量较低,且只能检测准种中比例在20%以上的突变株,很难检测劣势株,因此近些年来,国际上一直在寻求新的、高通量和高灵敏度的替代检测方法。
4.二代测序,也称下一代测序(next generation sequence,ngs),最早于2005年发明。roche公司研发的454测序平台,基于光纤微流体技术和包裹待测dna片段的乳化液滴技术,是第一个商业化的平台,标志着二代测序技术时代的到来。该平台具有检测速度快、灵敏度高、长读取的优势,读长可以达到700bp,但其错误率高达4%,难以精确检测病毒准种间的细微差别。2006年,solexa公司研发了ga测序仪,利用边合成边测序的原理,实现了核酸的大规模平行测序。基于ga平台,illumina研发了一系列测序平台,极大地提高了二代测序的效率,并进一步降低了错误率。其中miseq平台可进行双端测序,可获得长达600bp的读长,且运行成本较低,便于小型微生物,包括病毒和细菌基因组的测序,是目前应用最广泛的二代测序平台之一。
5.尽管ngs应用前景很好,但是现有技术中并没有一种可靠的使用ngs检测hiv特定区域基因型的方法,并未如预测的那样实现检测的快速常规化,以往基于扩增子建库的ngs方法测出的文库浓度低,测序深度低,且基因型的分析需依靠编程等生物信息学专业的知识,导致成本的大幅上升和应用的严重受限。
技术实现要素:
6.为了适用于二代测序,尤其是使用miseq高通量测序仪进行二代测序的要求,本发明提供了一种hiv-1蛋白酶和逆转录酶区、整合酶区基因型的检测方法。
7.本发明提供了一种hiv-1蛋白酶和逆转录酶区、整合酶区基因型的检测方法,所述方法包括:
8.提取样品rna,并以提取的所述rna为模板,逆转录成cdna;
9.以所述cdna为模板,进行pcr扩增,分别获得hiv-1病毒的蛋白酶和逆转录酶区的dna片段、整合酶区的dna片段;
10.构建基因文库;
11.对构建的所述基因文库,采用二代测序技术进行测序,获取hiv-1蛋白酶和逆转录酶区、整合酶区的dna序列组成,然后以hiv-1参考株hxb2为参考序列,从而获得hiv-1蛋白酶和逆转录酶区、整合酶区的基因型。
12.本发明提供的hiv-1蛋白酶和逆转录酶区、整合酶区基因型的检测方法,操作步骤简单、成本低、效率高,非常适合二代测序、尤其是使用miseq高通量测序仪进行二代测序的要求。
附图说明
13.图1为本发明实施例4中,二代测序的深度和覆盖范围的示意图。
14.图2为本发明实施例4中,同一样本不同测序方法序列相似性的比较示意图。
15.图3为本发明实施例4中,二代测序方法精确性和可重复性验证的示意图。
16.图4为本发明实施例4中,基于一代测序与二代测序的配对样本突变位点检出情况示意图;其中图4a示出了一代测序检出突变位点者的二代测序结果(105例),图4b示出了一代测序未检出突变位点者的二代测序结果(227例)。
具体实施方式
17.为使本发明的技术方案、目的和优点更加清楚,下面通过具体的实施例子对本发明做进一步的详细描述。应当理解的是,此处所描述的具体实施方式仅用于说明和解释本发明,并不用于限制本发明。
18.本发明提供了一种hiv-1蛋白酶和逆转录酶区、整合酶区基因型的检测方法,所述方法适用于非诊断/治疗目的的hiv-1蛋白酶和逆转录酶区、整合酶区基因型的筛查。本发明通过对hiv-1蛋白酶和逆转录酶区、整合酶区基因型的筛查,可以为非诊断/治疗目的的hiv-1蛋白酶和逆转录酶区、整合酶区的低频耐药突变位点的筛查提供重要的参考。
19.本发明提供的hiv-1蛋白酶和逆转录酶区、整合酶区基因型的检测方法,包括如下步骤:
20.提取样品rna,并以提取的所述rna为模板,逆转录成cdna;
21.以所述cdna为模板,进行pcr扩增,分别获得hiv-1病毒的蛋白酶和逆转录酶区的dna片段、整合酶区的dna片段;
22.构建基因文库;
23.对构建的所述基因文库,采用二代测序技术进行测序,获取hiv-1蛋白酶和逆转录酶区、整合酶区的dna序列组成,然后以hiv-1参考株hxb2为参考序列,从而获得hiv-1蛋白酶和逆转录酶区、整合酶区的基因型。
24.根据本发明,所述方法包括第一轮pcr反应和第二轮pcr反应;其中,所述第一轮pcr反应以提取的所述rna为模板,逆转录成cdna,并且对逆转录成的cdna进行初步扩增,以获取hiv-1病毒的蛋白酶和逆转录酶区的dna片段、整合酶区的dna片段;所述第二轮pcr反应将所述第一轮pcr反应获得的hiv-1病毒的蛋白酶和逆转录酶区的dna片段、整合酶区的
dna片段进一步扩增。
25.其中,所述第一轮pcr反应所采用的引物如下:
26.当用于扩增蛋白酶和逆转录酶区的dna片段时:
27.正向引物1:5
′‑
tgaargaitgyactgaragrcaggctaat-3
′
,即序列表中的序列1;
28.正向引物2:5
′‑
actgaragrcaggctaattttttag-3
′
,即序列表中的序列2;
29.反向引物:5
′‑
atccctgcataaatctgacttgc-3
′
,即序列表中的序列3;
30.当用于扩增整合酶区的dna片段时:
31.正向引物1:5
′‑
ggratyattcargcacaaccag-3
′
,即序列表中的序列4;
32.正向引物2:5
′‑
gcattaggratyattcargcac-3
′
,即序列表中的序列5;
33.反向引物:5
′‑
tgggatrtgtacttcygarctta-3
′
,即序列表中的序列6;
34.所述第二轮pcr反应所采用的引物如下:
35.当用于扩增蛋白酶和逆转录酶区的dna片段时:
36.正向引物:5
′‑
ctttarcttccctcaratcactct-3
′
,即序列表中的序列7;
37.反向引物:5
′‑
cttctgtatgtcattgacagtcc-3
′
,即序列表中的序列8;
38.当用于扩增整合酶区的dna片段时:
39.正向引物:5
′‑
tctayctgkcatgggtrccagcac-3
′
,即序列表中的序列9;
40.反向引物:5
′‑
catcctgtctacytgccacac-3
′
,即序列表中的序列10。
41.根据本发明,所述构建基因文库的操作包括如下步骤:
42.构建基因文库之前,首先对获得的蛋白酶和逆转录酶区的dna片段、整合酶区的dna片段进行纯化。
43.当采用两轮pcr反应时,对第二轮pcr产物进行纯化。所述纯化可以为采用磁珠纯化的方式。
44.当采用磁珠纯化时,磁珠悬浮液与第二轮pcr产物之间的体积比可以为(0.7-0.9):1,优选为0.8:1。
45.进一步地,还可以包括对纯化回收的片段进行浓度测定,并将每个纯化回收片段的浓度调整为同一浓度的操作,例如,可以将每个纯化回收片段的浓度统一调整为1ng-1ug,优选为600ng。
46.所述构建基因文库的操作包括:
47.1)将获得的蛋白酶和逆转录酶区的dna片段、整合酶区的dna片段进行酶切处理,获得酶切反应产物;
48.所述酶切处理采用的酶可以为无偏好性的片段化酶,即不受特定酶切位点限制的随机内切酶,例如可以为罗氏公司的kapa 片段化酶。通过所述酶切处理,可以将获得的蛋白酶和逆转录酶区的dna片段、整合酶区的dna片段进行随机切割,再结合采用一定的酶切处理条件(酶切时间和温度),即可将蛋白酶和逆转录酶区的dna片段、整合酶区的dna片段切成不同大小的片段,从而应用于后续的测序处理。
49.所述酶切处理的条件可以为:30-37℃酶切处理7-10分钟;优选地,所述酶切处理的条件为:37℃酶切处理7min,或者30℃酶切处理10min。通过酶切处理,即可将蛋白酶和逆转录酶区的dna片段、整合酶区的dna片段切割成大部分都位于400-600bp长度的dna片段。
50.本发明通过采用无偏好性的片段化酶,结合采用合适的酶切处理条件,可以为后
续的处理步骤提供合适的dna片段,从而获得合适的、最终应用于二代测序的需要的dna片段。
51.2)对所述酶切反应产物进行末端修复和加a处理,获得末段修复和加a处理反应产物;
52.由于酶切之后形成的片段化的dna产物具有粘性末端,通过末段修复处理,可以将损坏的或不完全的dna末端修复成5’磷酸化的平头末端dna,以用于平末端dna连接。对修复处理之后的dna片段进行加a处理,其目的是在3’末端加上腺嘌呤(a)尾巴,从而与接头t互补配对,增加接头连接效率。
53.3)对所述末段修复和加a处理反应产物进行接头连接处理,获得接头连接反应产物;
54.所述接头连接处理中的接头为具有特定固定序列的dna片段,例如:与测序芯片(flowcell)结合的p5/p7 oligo,区分样品的标签(index)以及测序需要的引物等。
55.通过接头连接处理,可以使得获得的文库中的dna片段与测序芯片配对发生成簇反应,进而对目的片段进行测序。
56.4)对所述接头连接反应产物进行纯化处理之后,对所述纯化后的接头连接反应产物进行片段筛选处理,获得所述基因文库。
57.其中,所述纯化处理可以为磁珠纯化。所述磁珠纯化的条件包括:控制磁珠悬浮液与接头连接反应产物之间的体积比为(0.7-0.9):1,优选为0.8:1。
58.通过所述纯化处理,可以去除接头连接反应产物中的二聚体、残留的酶等蛋白质,以避免影响测序质量。
59.所述片段筛选可以包括第一次筛选和第二次筛选。所述第一次筛选和第二次筛选均可以采用磁珠筛选。
60.所述磁珠筛选的条件包括:控制磁珠悬浮液与纯化后的接头连接反应产物之间的体积比为(0.5-0.8):1。
61.优选地,当步骤1)中的所述酶切处理的条件为37℃酶切处理7min时,所述磁珠筛选的条件包括:第一次筛选时,控制磁珠悬浮液与纯化后的接头连接反应产物之间的体积比为0.5:1,第二次筛选时,控制磁珠悬浮液与纯化后的接头连接反应产物之间的体积比为0.7:1;
62.当步骤1)中的所述酶切处理的条件为30℃酶切处理10min时,所述磁珠筛选的条件包括:第一次筛选时,控制磁珠悬浮液与纯化后的接头连接反应产物之间的体积比为0.6:1,第二次筛选时,控制磁珠悬浮液与纯化后的接头连接反应产物之间的体积比为0.8:1。
63.第一次磁珠筛选时,弃去与磁珠结合的dna片段,从而将大于1kbp的dna片段丢弃。
64.第二次磁珠筛选时,弃去上清液,从而将小于350bp的dna片段丢弃。
65.经过两次磁珠筛选,可以使得基因文库中保留了350至750bp,且大部分(至少80%)分布在600bp附近(580-620bp)的dna片段,从而满足二代测序的要求。
66.采用磁珠筛选时,磁珠悬浮液与待筛选样本之间的体积比是决定筛选得到的dna片段大小分布的关键因素。本发明中,将酶切的条件和磁珠筛选的条件进行紧密配合,由于37℃酶切处理7min时的产物可能出现过大的片段,采用上述的磁珠悬浮液与纯化后的接头
连接反应产物之间的体积比是最合适的;而30℃酶切处理10min时的产物可能出现过小的片段,因此采用上述的磁珠悬浮液与纯化后的接头连接反应产物之间的体积比是最合适的。采用上述的酶切条件与磁珠筛选条件,可以使得最终获得的基因文库中,dna片段的大小大部分分布在在600bp附近,同时文库中dna片段的浓度可以控制在4nm以上,非常适合二代测序的要求,例如满足illumina miseq平台测序的要求。
67.根据本发明,所述hiv-1蛋白酶和逆转录酶区、整合酶区基因型的检测方法还包括对构建的所述基因文库在测序前进行质控的操作。
68.所述质控可以包括对所述基因文库中的dna片段的浓度进行测量,和/或对所述基因文库中的dna片段的大小进行测量的操作。
69.下述实施例中所使用的各种试剂、材料等,若无特别说明,均为可以从商业渠道获得的产品;下述实施例中所使用的各种测试、检测方法若无特别说明,均为本领域中的常规测试、检测方法,均可以从教科书、工具书或学术期刊中获得。
70.本发明中所使用的磁珠均为购自罗氏公司货号为kk8002的磁珠筛选试剂盒,该试剂盒是一种磁珠悬浮液,磁珠缓冲液中含有peg/nacl,用于驱动dna分子与磁珠的结合。
71.实施例1
72.本实施例用来说明目标片段的获取。
73.首先从样本材料中提取rna,以用于基因文库的构建。其中,样本材料为待检测hiv-1病人的血清或者血浆。rna的提取采用qiagen viral rnamini kit病毒rna提取试剂盒并按照试剂盒的使用说明来进行。
74.1、特异性目的片段的巢式pcr扩增:
75.采用特异性扩增引物,以上述提取的rna为模板,通过rt-pcr(一步法)进行目的片段的扩增,引物具体的位置以hiv-1b亚型hxb2为标准。用来扩增蛋白酶和逆转录酶区的具体引物信息如表1所示,用来扩增整合酶区的具体引物信息如表2所示。
76.表1用来扩增蛋白酶和逆转录酶区的引物
[0077][0078]
表2用来扩增整合酶区的引物
[0079][0080]
上述引物序列中,字母“a”代表“腺嘌呤”,字母“t”代表“胸腺嘧啶”,字母c代表“胞嘧啶”,字母g代表“鸟嘌呤”;字母r\y\k\m\v均代表混合碱基,其中:字母“r”代表“a g”,字母“y”代表“c t”,字母“k”代表“g t”;字母“m”代表“a c”,字母“v”代表“a g c”,字母“w”代表“a t”,字母“i”代表次黄嘌呤。本发明中,如无特殊说明,在核酸序列中所使用的字母均为上述含义。名词“混合碱基”,是指该位置处具有相应的两种或三种碱基,例如,某个位置
是混合碱基“y”,即是指这个序列的混合碱基“y”所处的位置是c和t均有,且摩尔比例一般为1:1,在合成引物时同时加入这两种dntp,两种dntp的掺入几率一般均为50%。
[0081]
第一轮pcr反应体系:
[0082]
第一轮pcr反应采用promega公司货号为a1702 accessquick
tm rt-pcr system的试剂盒来进行。反应体系(总体积25μl)如下表3所示:
[0083]
表3第一轮pcr反应体系
[0084]
组分用量pcr mix12.5μl模板rna5μl反转录酶0.5μl正向引物10.25μl正向引物20.25μl反向引物0.5μlddh2o6μl
[0085]
其中,pcr mix中含有tfl dna聚合酶、dntps、硫酸镁和反应缓冲液。模板rna即为按照上述方法提取得到的rna。各个引物的终浓度均为20μmol/l。
[0086]
当用于扩增蛋白酶和逆转录酶区时,上述反应体系中的正向引物1、正向引物2、反向引物依次为表1中的引物1、引物2、引物3。当用于扩增整合酶区时,上述反应体系中的正向引物1、正向引物2、反向引物依次为表2中的引物6、引物7、引物8。
[0087]
反应程序如下:
[0088]
对于蛋白酶和逆转录酶区的扩增,按照如下程序进行:
[0089]
50℃45分钟;94℃2分钟;94℃15秒,50℃20秒,72℃2分钟,35个循环;72℃10分钟;4℃保存。
[0090]
对于整合酶区的扩增,按照如下程序:
[0091]
50℃45分钟;95℃2分钟;95℃20秒,50℃30秒,72℃2分30秒,3个循环;95℃15秒,50℃20秒,72℃2分钟,35个循环;72℃10分钟;4℃保存。
[0092]
经过第一轮pcr扩增,即可获得1302bp的蛋白酶和逆转录酶区dna片段,和1161bp的整合酶区dna片段。
[0093]
第二轮pcr反应体系:
[0094]
第二轮pcr采用tiangen公司货号为kt205-02 2x taq plus pcr预混试剂(含染料)的试剂盒来进行。反应体系(总体积50μl)如下表4所示:
[0095]
表4第二轮pcr反应体系
[0096]
组分用量pcr mix25μl模板5μltaq酶0.5μl正向引物1μl反向引物1μlddh2o18μl
[0097]
其中,pcr mix预混试剂中含有2倍终浓度taq dna聚合酶、dntps、mgcl2、反应缓冲液、pcr反应的增强剂和优化剂以及稳定剂。各个引物的终浓度均为20μmol/l。
[0098]
当用于扩增蛋白酶和逆转录酶区时,上述反应体系中的模板为第一轮pcr反应获得的蛋白酶和逆转录酶区dna片段,正向引物、反向引物依次为表1中的引物4、引物5。
[0099]
当用于扩增整合酶区时,上述反应体系中的模板为第一轮pcr反应获得的整合酶区dna片段,正向引物、反向引物依次为表2中的引物9、引物10。
[0100]
反应程序如下:
[0101]
对于蛋白酶和逆转录酶区的扩增,按照如下程序进行:
[0102]
95℃4分钟;95℃20秒,55℃30秒,72℃2分30秒,3个循环;95℃15秒,55℃20秒,72℃2分钟,35个循环;72℃10分钟;4℃保存。
[0103]
对于整合酶区的扩增,按照如下程序进行:
[0104]
95℃4分钟;95℃20秒,55℃30秒,72℃2分30秒,3个循环;95℃15秒,55℃20秒,72℃2分钟,35个循环;72℃10分钟;4℃保持。
[0105]
经过第二轮pcr扩增,即可获得1062bp的蛋白酶和逆转录酶区dna片段,和948bp的整合酶区dna片段。
[0106]
pcr反应产物的鉴定
[0107]
使用1%质量浓度的琼脂糖凝胶,对第二轮pcr产物进行鉴定,结果发现:蛋白酶和逆转录酶区的第二轮扩增产物可以在稍大于1000bp的dnamarker处出现荧光条带(实际大小1062bp);整合酶区的第二轮扩增产物可以在稍小于1000bp的dnamarker处出现荧光条带(实际大小948bp),说明经过两轮pcr扩增成功获得了蛋白酶和逆转录酶区的dna片段,以及整合酶区的dna片段。
[0108]
使用磁珠回收第二轮pcr扩增产物中的dna片段
[0109]
采用罗氏公司货号为kk8002的磁珠筛选试剂盒对第二轮pcr扩增产物进行回收。其中,磁珠悬浮液与第二轮pcr扩增产物之间的体积比为0.8:1,使用前将磁珠悬浮液从冰箱取出置于室温平衡大约30分钟。磁珠回收的具体操作流程如下:
[0110]
目标dna和磁珠的结合:涡旋磁珠悬浮液使其充分混合均匀,在96孔板上每孔加入40μl混合均匀的磁珠悬浮液,然后加入50μl的第二轮pcr扩增产物,混匀后于室温静置5分钟。将96孔板于磁力分离器上静置至少5分钟使所有的磁珠都通过磁力吸附到管底上,弃去上清液。
[0111]
使用体积分数为80%的乙醇清洗两次:清洗时将96孔板置于磁力分离器上,每孔加入200μl的80%乙醇,切勿碰触磁珠,于室温静置至少30秒,小心移走并弃掉上清。如上所述,再重复清洗一次,最后要完全吸走上部的乙醇。
[0112]
充分晾干反应板中的乙醇:将96孔板置于磁力分离器上,在室温条件下静置5-15分钟,使所有残留酒精挥发完全。直到孔壁及底部无可见液滴,但同时要避免过干。
[0113]
将目的片段从吸附磁珠上洗脱下来:将96孔板从磁力分离板上取下,置于水平台面上,向每孔中加入10-50μl纯水,重悬磁珠,室温条件下静置2-5分钟。将96孔板于磁力分离板上静置5分钟,使所有的磁珠都吸附到管底上,使得纯化后的dna样本和磁珠充分分离,将含有纯化的dna片段的纯水转移到一个新的96孔板上,将纯化回收好的dna片段置于-20℃冰箱备用。
[0114]
测定磁珠回收后的dna片段的浓度
[0115]
采用微量蛋白和核酸定量仪对纯化回收的片段进行浓度测定。按照仪器的操作流程进行操作,在定量的试剂的选取过程中选取高敏感度的核酸定量试剂盒(invitrogen
tm
的qubit
tm dsdnahs assay kits)进行检测,软件版本选用2.0对纯化回收的dna片段进行定量。
[0116]
准备足够的薄壁干净0.5ml微量离心管,需要注意的是在该部分的测量过程中需要两个预制测量标准品用来确定测量阈值曲线的上限和下限。由于使用的是高灵敏试剂盒,需要对样本进行10倍稀释,即2μl样本加18μl纯水配比。
[0117]
将qubit
tm dsdnahs的测定缓冲液和相应的荧光染料试剂按照1:200的比例配比稀释(即为1μl荧光染料 199μl的测定缓冲液),获得工作液。吸取190μl工作液,分装到两个0.5ml的微量离心管中,再分别吸取10μl的qubit标准品1和标准品2,小心短暂涡旋避免气泡产生,避光保存备用。向0.5ml离心管中加入198μl工作液,吸取2μl稀释后的待测样本到上述管中,小心短暂涡旋避免气泡产生,每个待测样本的测定最终体积为200μl。混匀后室温静置2分钟,避光保存备用。
[0118]
在2.0首页,选择测定dna样品,然后选择dsdnahigh sensitivity试剂盒。进入选择标准品的测定页面,选择yes开始测定标准品。将标准品1的0.5ml微量离心管放入测定槽中,盖上测定槽的盖子,选择read开始测定;依照同样的步骤放入标准品2的0.5ml微量离心管,进行标准品2的测定。待两个标准品均完成测定之后,便显示标准曲线。随后开始进行样本的测定过程,依照顺序将每一个待测样本放入待测槽中,选择read进行测定,然后设置浓度单位为ng/μl,加样量为2μl,将所测得的结果乘以10则为样本的终浓度。该步骤需要在15分钟内测定完成,时间过长容易导致误差。
[0119]
根据测得的浓度将来自同一样本的蛋白酶和逆转录酶区和整合酶区两个片段进行混合。例如:测得同一样本的两个片段浓度均为100ng/μl,而建库前目的片段的体积要为35μl,dna片段初始含量500ng,则每个片段选取2.5μl回收后的样本(共5μl),混合后再加30μl水;则dna片段含量为100ng/μl*2.5μl*2=500ng。
[0120]
实施例2
[0121]
本实施例用来说明基因文库的构建。
[0122]
酶切片段化:
[0123]
采用kapa酶切试剂盒,对实施例1获得的回收片段进行酶切处理。
[0124]
根据实施例1获得的回收片段的浓度以及基因文库的起始量(1ng-1μg间均可)计算出所需的样本体积。在冰盒上配置如下表5所示的酶切反应体系:
[0125]
表5酶切反应体系
[0126]
组分用量回收双链dna片段样本和补足去离子水35μlkapa片段化缓冲液(10x)5μlkapa片段化酶10μl
[0127]
将配好的酶切反应体系在37℃下酶切7min。
[0128]
末端修复和加a处理:
[0129]
对酶切反应产物进行末端修复和加a处理,采用购自罗氏公司货号为kk8515的试剂盒进行,具体操作如下:
[0130]
将酶切反应产物在冰盒上按下述反应体系,进行末端修复和加a反应:
[0131]
表6末端修复和加a反应体系
[0132]
组分用量酶切反应产物50μl末端修复和加a尾的缓冲液7μl末端修复和加a尾的酶混合物3μl
[0133]
反应程序为:65℃30分钟;4℃保持(如果立即进行下一步,则20℃保持)。同时也可以将盖子的加热温度设定在85℃(而不是通常的105℃)。
[0134]
接头连接:
[0135]
将末端修复和加a反应产物进行接头连接。
[0136]
首先按照表7将要连接的接头稀释到合适的浓度。其中,接头为购自罗氏公司货号为kk8722的双端接头试剂盒(适用于illumina平台)。
[0137]
表7 dna起始量为1ng
–
1μg间构建的文库的建议接头浓度
[0138][0139]
按照表8的接头连接反应体系,在20℃下孵育15分钟,4℃保持15分钟,从而在末端修复和加a反应产物中的dna片段上连接好接头。
[0140]
表8接头连接反应体系
[0141]
组分体积/反应末端修复和加a反应之后的产物60μl稀释后的接头5μlpcr级水5μl连接缓冲液30μldna连接酶10μl
[0142]
磁珠纯化:
[0143]
将连接了接头的末端修复和加a反应产物,采用磁珠进行纯化:
[0144]
将磁珠悬浮液置于室温平衡大约30分钟,按磁珠悬浮液与接头连接反应产物的体积比为0.8:1混合,然后在室温下静置5-15分钟,使dna与磁珠结合;然后置于磁力架上捕捉磁珠直到液体清澈,小心取出并丢弃上清液。保持pcr管/板在磁力架上,加入200μl80%乙醇,在室温下孵育30秒后,小心地取出并丢弃乙醇,重复再用200μl 80%乙醇清洗一次。尽量去除所有残留乙醇,室温下干燥3-5分钟,直到所有乙醇蒸发。
[0145]
磁珠洗脱:向每孔中加入55μl纯水,混匀后室温下静置2分钟。然后在磁力架上静置5分钟,直至上清液清澈。将含有纯化的dna片段的上清液转移到一个新的pcr管/板上,将
纯化回收好的dna片段保存备用。
[0146]
在采用ngs测序的基因文库构建时,一般都会对连接了接头的dna文库进行一次特异性pcr扩增,以增加基因文库中连有接头的dna片段的含量,从而满足ngs测序的要求。而在本发明中,将磁珠纯化之后的产物直接用于片段筛选,不再进行pcr反应对文库进行扩增富集,避免了引入pcr偏见及突变。因为再一次的pcr扩增会提高接头二聚体以及占比较高的dna片段的比例,且扩增后需要又一次的纯化会导致本身占比较低的dna片段的损失。本发明中通过控制酶切的条件和对接头连接产物的两次筛选条件的配合,使得无需对连接了接头的dna文库进行pcr扩增,而是直接将纯化之后的产物直接用于片段筛选,即可达到上机测序的浓度要求;因此虽然采用本发明的方法,不对连接了接头的dna文库进行pcr扩增,而是直接将纯化之后的产物直接用于片段筛选,构建用于ngs测序的文库,依然能够很好地满足ngs测序的要求。
[0147]
片段筛选
[0148]
将磁珠悬浮液从冰箱取出置于室温平衡大约30分钟。
[0149]
第一次筛选:控制磁珠悬浮液与纯化后的接头连接反应产物之间的体积比为0.5:1。具体操作为:向50μl接头连接反应产物中添加25μl kapa磁珠悬浮液,混合均匀后在室温下静置5-15分钟,使大于1kb的dna片段与磁珠结合,然后置于磁力架上捕捉磁珠直到液体清澈,小心地将含有小于1kb的dna片段的上清液转移到一个新的板/管中,切记上清液中不能带有磁珠,从而去除大于1kb的dna片段。
[0150]
第二次筛选:控制磁珠悬浮液与纯化后的接头连接反应产物之间的体积比为0.7:1。具体操作为:在经过第一次筛选之后的上清液(75μl)中加入10μl kapa磁珠悬浮液(磁珠悬浮液的总体积为第一次筛选时加入的25μl 第二次筛选时加入的10μl=35μl,因此,磁珠悬浮液与纯化后的接头连接反应产物之间的体积比为35μl:50μl=0.7:1),混合均匀后在室温下静置5-15分钟,使大于350bp的dna片段结合磁珠,然后置于磁力架上捕捉磁珠直到液体清澈,小心取出上清液并丢弃,从而将小于350bp的dna片段去除。保持pcr管/板在磁力架上,加入200μl 80%乙醇,在室温下孵育30秒后,小心地取出并丢弃乙醇;重复再用200μl 80%乙醇清洗一次。尽量去除所有残留乙醇,在室温下干燥3-5分钟,直到所有乙醇蒸发。注意不要过干燥,以免可能导致回收率降低。
[0151]
磁珠洗脱:从磁力架上取下pcr管/板,向每孔中加入30μl纯水,上下吹打10次或充分涡旋震荡,室温条件下静置2分钟,然后在磁力架上静置5分钟,直至上清液清澈。将含有纯化的dna片段的上清液转移到一个新的pcr管/板上,将纯化回收好的dna片段保存备用,一般置于4℃存放1-2周,或置于-20℃冰箱长期保存。
[0152]
实施例3
[0153]
本实施例用来说明建库后基因文库的质控及测序。
[0154]
文库的质控
[0155]
依照购自罗氏公司货号为kk4824的文库定量试剂盒(适用于illumina),按照试剂盒的操作说明,对文库进行实时荧光定量,以对文库进行质控。具体操作如下:
[0156]
使用纯水或dna稀释缓冲液[10mm tris-hcl,ph 8.0(25℃) 0.05%20(可选)]稀释样本。使用前一定要将纯水或缓冲液平衡到室温。确保定量试剂盒的所有试剂完全解冻并彻底混合。如果该试剂盒是第一次使用,将10x引物预混试剂(1毫升)加入2x kapa
混合物(5毫升)瓶中,用涡旋搅拌器彻底混合。
[0157]
用纯水或dna稀释缓冲液对待测定样本进行浓度稀释,以10倍系列稀释至10000倍,在稀释过程中注意充分混合,用移液器多吹打几次。确定好最终的反应总数并按表9配备主混合物:试剂盒中的6个dna标准品(每个标准品重复三次);稀释后的待测定样本;无模板对照(ntcs)。
[0158]
表9荧光定量pcr反应体系
[0159][0160]
将主混合物分配到每个pcr孔中,然后向所有无模板对照孔中添加4μl的pcr级水;将每个dna标准品4μl分配到孔中,从最稀释(标准品6)到最浓缩(标准品1);分配4μl的稀释后的待测样本分配到适当的孔中。盖上密封pcr板或盖,转移到qpcr仪器。根据表10的循环方案执行qpcr,在仪器软件中选择绝对定量(absolute quantification)选项,调整运行参数(如样本分布情况,样本名字,参考染料,标准品浓度等)。
[0161]
表10荧光定量pcr反应程序
[0162][0163]
仪器运行结束,检查标准曲线以确保满足以下标准:标准品间的δcq值在3.1-3.6范围内;计算的反应效率在90-110%的范围内(即pcr产物每周期增加1.8-2.2倍,标准曲线的斜率在-3.1-3.6之间);r2≥0.99。如果标准曲线不满足这些标准,则计算的文库浓度将不可靠,必须重复测定。
[0164]
使用agilent 2100进行文库片段大小的鉴定
[0165]
将高灵敏dna配套试剂盒(购自安捷伦公司货号为5067-4626的商品)从冷藏室内取出,避光放于室温环境下,至少30分钟使试剂恢复到室温。
[0166]
从试剂盒中取出上盖蓝色的高灵敏dna染料浓缩物,涡旋混匀,使其中的dmso充分混匀。稍微离心使粘在管壁和上盖的试剂沉到管底。然后取15μl的上述染料加入到盖子为红色的高灵敏dna凝胶基质瓶中,稍微涡旋混匀。将混合好的液体加入到过滤柱中,再将过滤柱置于离心机中,在室温条件下2240g离心10分钟。将离心柱丢弃,保存好离心后所得试剂,在保存的过程中切记要避光,并在管壁详细记录首次混匀的时间,每次制备好的试剂可以用于测定5次。将制备好试剂的保存于4℃冰箱内,用锡箔纸包裹避光保存,每次使用前需要将先其置于室温30分钟平衡。
[0167]
测定芯片的制备:
[0168]
将先前已经制备好的基质胶和试剂盒内试剂从冰箱中取出,放置在室温下30分钟,使其充分恢复到室温。从配套试剂中取出新的芯片,将芯片固定于注胶板的芯片槽内,盖好上盖,使用配套的1ml微量注射器检查芯片和注胶板的气密性。在测定芯片中注入基底胶,在带有g(黑底)字母的孔中加入9μl已经制备好的预制胶,加样务必加在加样孔底部,不能加在管壁上。设定计时器60s倒计时,将注胶器上的注射器位置放于1ml处,盖上注胶器,将注射器缓慢推下,固定在最低档后开始倒计时60s。倒计时60s完成后,松开注射器上方的卡子,默数5s后,待注射器内的柄缓慢的上提,至1ml处时停止。打开注胶板,将9μl已经制备好的预制胶加在芯片中另外三个注胶孔里。在测定芯片上所有的12个孔(11个样本槽和1个ladder孔)中加入5μl绿色盖子的marker,在标识梯子的ladder孔中加入1μl黄色盖子的ladder。加入待测样本(待测定样本的浓度范围为100pg/μl-10ng/μl),在各个样本孔中加入1μl样本,如果样本不足11个,在没有样本的孔中加入1μl超纯水代替。将制备好的芯片置于agilent 2100专用的震荡器中,待其自动震荡程序完成,震荡时间为1min,震荡完成后在5min内进行测定。
[0169]
在agilent 2100机进行测定:
[0170]
打开agilent 2100检测仪,点击软件打开页面。将芯片放于芯片槽内,固定好后,关上机盖。选择测定程序:选择接头(电脑与2100的连接默认为1,如果脱机运行选择demo),电脑与2100连接后会显示online,选择测试类型(点击analysis,选择high sensitivity dna)。样本详细信息的输入,在芯片位置处进行双击后进行输入。点击start开始运行。大约10min后开始出现ladder,如果15条ladder正常,则运行基本正常。在45min后仪器运行结束。仪器运行结束后进行机器的清洗,将空白的清洗芯片每孔加入9μl超纯水,换下运行结束的芯片,盖上机盖,半小时后取出。
[0171]
各个测定样本的浓度均一化调整及样本混合:
[0172]
使用不含有核酸酶的超纯水调整每个样本的最终浓度到4nmol/l,从而使每个样本的浓度达成一致。
[0173]
在最终的稀释过程中,可简化为每个样本均加入10μl,然后分别计算每孔中需要加入超纯水的体积,按照计算好的超纯水的体积在新的96孔pcr反应板中加入超纯水。从存放纯化好的96孔pcr反应板中吸取10μl,加入到已经加好水的96孔pcr反应板中。最终所有样本都加好后,使用覆板膜仔细盖好反应板,使用震荡器小心涡旋使其充分混匀后,使用2000rpm/min离心2min,使得粘在管壁和膜上的样本离心到管中。
[0174]
取一只1.5ml干净的薄壁离心管,做好标记,并将该管置于预冷的冰盒上,分别吸取10μl已完成均一化的各个样本到离心管中,待所有样本都吸取到该离心管中后,小心盖上离心管盖,避免溅出,使用震荡器小心涡旋使其充分混匀后,使用2000rpm离心2分钟,使得粘在管壁和膜上的样本离心到管中。对混合后的样本再次进行qpcr定量检测混合后的文库浓度。
[0175]
上机前准备及上机测序:
[0176]
接通miseq高通量测序仪器上的电源,打开电源开关,打开仪器,使用默认的登录用户名和密码登录机器,等待仪器中的操作系统完成装载,完成仪器初始化,自动载入miseq控制软件(mcs)。在仪器界面中可看到四个传感器的相应指示,包括试剂冷却器的温
度等。观察仪器的各个状态提示,是否有仪器报错等。若有要及时处理并解决问题。使用新配置的0.5%吐温20仔细清洗机器3遍。miseq v3试剂盒中配套的杂交液(pre-chilled ht1)需在室温条件下放置30min,使得该试剂温度恢复到室温。
[0177]
使用0.2n的naoh溶液将上一步所构建的文库变性并进行稀释:使用1000μl的微量移液器吸取800μl的超纯水和200μl的1n的naoh溶液配制成0.2n的naoh溶液备用。
[0178]
使4nmol的dna构建文库变性并稀释至20pmol:准备好一个0.2ml的微量离心管置于冰盒之上,吸取5μl的浓度为4nmol的dna文库和5μl的0.2n的naoh加入管中,短暂涡旋使得充分混匀,然后280g离心一分钟,在室温条件下放置5min,使得dna的双链变性为单链。取一个新的1.5ml离心管置于冰盒上,分别加入10μl的上一步变性好的dna文库和990μl的pre-chilled ht1杂交液,获得1ml的20pm变性文库。
[0179]
质控品phix文库变性并稀释至20pmol:准备一个0.2ml的微量离心管置于冰盒之上,吸取2μl的浓度为10nmol的phix文库和3μl的超纯水加入管中,短暂涡旋使得充分混匀,使其稀释至浓度为4nmol的phix文库。再准备一个0.2ml的微量离心管置于冰盒之上,吸取5μl的浓度为4nmol的phix文库和5μl的0.2n的naoh加入管中,短暂涡旋使得充分混匀,然后280g离心一分钟,在室温条件下放置5min,使得dna的双链变性为单链。取一个新的1.5ml离心管置于冰盒上,分别加入10μl的上一步变性好的dna文库和990μl的pre-chilled ht1杂交液,结果是1毫升的20pm phix文库。
[0180]
将变性后的样本文库及phix文库均稀释到10pm或12pm的上机浓度:1)10pm:加入300μl上述已经配制好的20pm dna或phix文库和300μl pre-chilled ht1杂交液;2)12pm:加入360μl上述已经配制好的20pm dna和240μl pre-chilled ht1杂交液。在涡旋仪上稍涡旋后,离心机短暂离心,置于冰上备用。
[0181]
将60%的10pm(或12pm)样本文库和40%的10pm(或12pm)phix文库进行混合。将混合后的最终文库加入试剂槽中相对应的样本孔位置,上机测序。上机最终浓度控制在10pm~12pm左右,此时的数据量与测序质量均是最优状态。
[0182]
对比例
[0183]
除了下述参数之外,其他均与实施例1-3相同:
[0184]
将实施例2中片段筛选操作中的两次磁珠筛选条件修改为:
[0185]
第一次筛选时,控制磁珠悬浮液与纯化后的接头连接反应产物之间的体积比为0.7:1,第二次筛选时,控制磁珠悬浮液与纯化后的接头连接反应产物之间的体积比为0.8:1。结果发现:在对基因文库进行质控时,基因文库中dna片段的大小没有落在350至750bp范围内,出现过多的小于350bp的dna片段,影响测序过程中簇的生成质量,且不适用于miseq v3(600cycles)的上机,造成测序数据量的浪费。
[0186]
第一次筛选时,控制磁珠悬浮液与纯化后的接头连接反应产物之间的体积比为0.4:1,第二次筛选时,控制磁珠悬浮液与纯化后的接头连接反应产物之间的体积比为0.5:1。结果发现:在对基因文库进行质控时,基因文库中dna片段的大小没有落在350至750bp的范围内,出现过多且过长的未被酶切完全的dna片段,同样不适用于miseq v3(600cycles)的上机要求,而且会造成测序得到的序列无法覆盖目的dna片段。
[0187]
实施例4
[0188]
本实施例用来说明本发明提供的hiv-1蛋白酶和逆转录酶区、整合酶区基因型的
检测方法的扩增灵敏度,测序深度和覆盖率,核苷酸序列相似性和准确性,可重复性,精确性,敏感度,特异性,阳性预测值(ppv),阴性预测值(npv)。
[0189]
样本数量及信息
[0190]
评估扩增灵敏度,测序深度和覆盖率,核苷酸序列相似性,准确性所采用的20份血浆样本来源于我国部分地区接受art(抗逆转录病毒治疗)后的hiv/aids病人;评估可重复性,精确性所用的样本为art前病人的5份血浆样本,以及3份hiv质粒pnl4-3稀释后的样本。评估该方法的敏感度、特异性、阳性预测值(ppv)及阴性预测值(npv)的样本为332份血浆样本。
[0191]
表11样本数量及信息
[0192][0193]
扩增灵敏度
[0194]
为了评估本发明提供的hiv-1蛋白酶和逆转录酶区、整合酶区基因型的检测方法的扩增敏感度,选取vl((viral load病毒载量)在1000-5000拷贝/ml和vl》10000拷贝/ml的各10个血浆样本,结果显示vl在1000-5000拷贝/ml,及vl≥10000拷贝/ml的20份血浆样本均扩增成功,且通过二代测序方法成功获得序列。
[0195]
测序深度和覆盖率
[0196]
测序深度是指测序得到的总碱基数与待测基因组大小的比值。覆盖率是指测序获得的序列占整个基因组的比例。二代测序的原始数据按质控标准进行过滤后得到高质量完整的测序序列,样本的平均测序深度为109798
×
。所有样本过滤后得到的序列均能覆盖蛋白酶和逆转录酶区具有临床意义的耐药突变位点对应的基因区域,且研究对象每个碱基位置的最低测序深度均在10000
×
以上,如图1所示。
[0197]
核苷酸序列相似性分析
[0198]
核苷酸序列相似性比较用来描述检测序列和目标序列之间相同dna碱基或氨基酸残基序列所占比例,需要注意的是,序列中的混合碱基被界定为不具有相似性。为了评估ngs miseq平台测序的能力,我们通过blast分析同一样本不同ngs突变频率阈值(1%、5%、10%和20%)下所获得的序列分别与ss序列之间的核苷酸相似性。结果显示当突变频率为5%、10%及20%时,相比1%的突变频率,配对样本一代、二代序列间的相似度较高(≥97.0%),突变频率组间差异性分析发现1%突变频率组与其他3组间的差异有统计学意义
(p《0.05)。当突变频率从1%提高到5%时,其配对样本序列间的相似性也随之升高,趋势较明显;而当突变频率从5%提高到10%,再到20%时,发现相似性增加的幅度较平缓,组间变化不明显,如表12和图2所示。
[0199]
表12同一样本ss(sanger测序)与ngs序列相似性的比较(%)
[0200] 1%突变频率5%突变频率10%突变频率20%突变频率最小值89.095.096.096.0最大值98.099.099.0100.0平均数94.797.298.098.6标准偏差2.51.01.11.3中位数95.097.098.099.095%ci下限93.596.897.598.095%ci上限95.897.798.599.3
[0201]
准确性
[0202]
以一代测序为标准,其结果显示7名患者有耐药突变位点,均是对非核苷类反转录酶抑制剂耐药,主要耐药位点是k103n位点(25.0%),其次为p225h(5.0%)、e138a(5.0%)和v179d(5.0%)位点。突变频率是指耐药突变株在准种中所占的比例,不同突变频率下的二代测序结果一般是不同的,此次结果中只展示了1%、5%、10%和20%阈值的ngs耐药结果。通过比较一代测序与各阈值下二代测序所测耐药位点的结果,发现各阈值下ngs均能检出sanger测序所检出的所有位点,当耐药位点突变频率高于10%时,两种测序方法的检测结果完全一致,而突变频率低于10%的耐药位点只被二代测序检测到,如表13所示。
[0203]
表13 ss与ngs耐药位点判别的比较
[0204]
[0205][0206]
可重复性
[0207]
为了评估该方法的可重复性,将野生株pnl4-3质粒稀释成3个不同拷贝数的样本,病毒载量分别为10000拷贝/ml、5000拷贝/ml和1000拷贝/ml,每个样本重复两次试验,测序结果显示无任何耐药突变位点的存在。此外,选取5份不同亚型和病毒载量的血浆样本重复两次试验得到两组位点突变频率结果。wilcoxon秩和检验发现两次测量结果无统计学差异(p=0.478),如表14所示。
[0208]
表14 hiv耐药位点突变频率的可重复性结果
[0209][0210]
精确性
[0211]
为了评估该方法的精确性,选取两份血浆样本在同一批试验中重复4次。friedman检验结果显示配对样本多次测量结果间无统计学差异(p=0.175)。此外,一般来说组内相关系数(intraclass correlation efficient,icc)介于0-1之间,就方法验证而言,若icc值小于0.4,则认为精确性较差,若icc值大于0.75,则认为精确性较好,结果显示icc值为0.998(95%ci:0.996-0.999,p《0.001),如表15所示。
[0212]
对ss和不同阈值的ngs共享序列通过mega软件,以n-j法构建进化树,构建模型为kimura 2-parameter,bootstrap值设为1000,结果显示除样本19gx074(批内第二、四次的序列)1%阈值的序列未在一个簇上外,同一样本的其余序列均成簇,且交联值高(》70),序
列间基因距离短,如表15和图3所示。
[0213]
表15 hiv耐药位点突变频率的精确性结果
[0214][0215]
配对样本的一代和二代测序检测hiv突变位点的结果
[0216]
一致性检验用于评价两种测序方法检测结果的一致性程度,用kappa系数表示。有5个级别:0.0~0.20极低的一致性、0.21~0.40一般的一致性、0.41~0.60中等的一致性、0.61~0.80高度的一致性和0.81~1几乎完全一致。
[0217]
敏感度、特异性、阴性预测值和阳性预测值是用来评估基于二代测序的hiv基因型耐药检测方法是否可靠的重要参数,敏感度也称真阳性率,反应待评估方法检出突变位点的漏检率;特异性也称真阴性率,反应待评估方法检出突变位点的错误率;阴性预测值,反应未检出突变位点的样本总数中,真正不携带突变位点的样本数占其样本总数的百分比;阳性预测值,反应检出突变位点的样本总数中,真正携带突变位点的样本数占其样本总数的百分比。本发明中将当前临床或实验室所通用的检测hiv耐药的sanger测序方法所产生的结果作为“金标准”,将二代测序结果与之进行比较。
[0218]
选取病毒载量≥1000拷贝/ml的332份血浆样本基于一代和二代测序的基因型耐药检测方法平行检测。其中,一代测序结果显示有105份携带突变位点;227份未被检出突变位点。一代测序检出突变位点的样本中,ngs不同突变频率下检出数分别为102、94、94和90。采用配对卡方检验比较两种方法检测hiv基因型突变位点是否有统计学差异,从结果可以看出,当阈值为1%、5%时,两种检测方法是有差异的,差异具有统计学意义;但一致性检验结果显示阈值设为1%时,两方法间的一致性较低(κ=0.0941)。随着阈值从1%升高到20%,二代测序的敏感度随之降低,而特异性从16.7%上升到94.7%。此外,有3.6%(12/332)的病人其突变频率在20%以上却只被ngs检测到,见表16。
[0219]
表16配对样本的一代和二代测序检测hiv突变位点的统计结果(332人)
[0220][0221]
表16(续表)配对样本的一代和二代测序检测hiv突变位点的统计结果(332人)
[0222][0223]
对一代测序检测到耐药位点的105名病人进行两种测序方法的比较,发现当突变频率高于20%时,二代测序检出90例病人有突变位点,pis的突变位点是q58e,nrtis的突变位点以k65r、q151m和m184v为主,而耐nnrtis的患者较多,突变位点多为k103n、y181c、g190a、e138a和v179de;一代测序检出而二代测序未检出的突变位点主要有m46v、k65r、l210w、k103n、v106m和v179e等,手动检查一代及二代测序的序列后,发现由于sanger测序无法判读两个相邻核苷酸混合碱基导致假阳性结果。观察一代测序未检测出突变位点的227名患者的二代测序结果,发现位点突变频率集中在5%以下。此外,二代测序结果显示突变位点k65er、d67gen、f77l、l100vi在1%阈值检测下出现频率较高,几乎存在于每个样本的检测结果中,而在5%以上阈值的二代测序结果中很少检测到这些突变位点,如图4所示。
[0224]
综上可见,采用本发明提供的hiv-1蛋白酶和逆转录酶区、整合酶区基因型的检测方法,在准确性、扩增灵敏度、精确性、可重复性方面均符合who推荐的标准,完全可以适用于我国流行的hiv-1病毒株针对低频耐药位点的检测。
[0225]
以上所述仅为本发明的优选实施例而已,并不用于限制本发明,对于本领域的技术人员来说,本发明可以有各种更改和变化。凡在本发明的精神和原则之内所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
再多了解一些
本文用于创业者技术爱好者查询,仅供学习研究,如用于商业用途,请联系技术所有人。