一种血浆游离dna来源的甲基化检测方法、肺癌诊断标志物以及试剂盒
技术领域
1.本发明涉及一种血浆游离dna来源的甲基化检测方法、肺癌诊断标志物以及试剂盒,属于基因检测技术领域。
背景技术:
2.肺癌是世界上最常见的恶性肿瘤,也是我国死亡率最高的癌种。肺癌可以分为两大类:小细胞肺癌(small cell lung cancer)和非小细胞肺癌(non small cell lung cancer)。非小细胞肺癌(nsclc)包含腺癌、鳞状细胞癌和大细胞癌。非小细胞肺癌约占所有肺癌的80%,腺癌约占非小细胞肺癌的55%。早期肺癌症状不明显,大约75%的患者发现时已处于中晚期,且5年生存率很低。由于肺癌的异质性,往往预后效果不佳。
3.目前,胸部低剂量ct是现阶段肺癌早期筛查的最常用的方法,能检测直径5mm以上的肿瘤。但该方法存在辐射暴露、过度诊断、灵敏度不够等问题。液体活检是近年来备受关注的一种体外诊断技术,通过非侵入性取样的方法,能检测肿瘤或转移到血液循环中的肿瘤细胞和肿瘤dna片段。对于筛选检测早期肿瘤、评价药物疗效具有十分重要的意义。目前最常用的是ctdna 的检测。ctdna是肿瘤细胞破裂后释放到人体循环系统中的小片段dna,可能携带肿瘤突变、插入、缺失、拷贝数变异等遗传信息。因此基于人血液来源的ctdna检测对于癌症的早期诊断和癌症治疗过程中的疗效评估具有十分重要的意义。
4.dna甲基化是表观遗传学(epigenetics)的重要组成部分。在人体中,经常发生在cpg二核苷酸的胞嘧啶上。dna甲基化能引起染色质结构、dna构象、dna稳定性及dna与蛋白质相互作用方式的改变,从而控制基因表达。大量研究表明,在肿瘤的发生和发展中存在dna 甲基化水平和模式的混乱,包括整体基因组甲基化水平过低和局部基因启动子区域甲基化过高。已有研究发现,在肺癌中甲基化的异常不仅在组织中被发现,在血浆、血液等体液中也能够检测到,并且随着肺癌的发生,甲基化异常程度也会增加。
5.基于液体活检技术的ctdna甲基化研究在近年来越来越热。有研究表明,ctdna 甲基化可在肿瘤发生的早期被检测到,而且具有较好的稳定性,因此 ctdna 甲基化成为肿瘤液体活检中一个极具价值的标志物。甲基化检测方法包括重亚硫酸氢盐转换,其代表方法包括wgbs (whole-genome bisulfite sequencing)。状态进行判断,以及基于蛋白质亲和富集的mbd-seq(methylated dna binding domain sequencing)技术,又称蛋白质富集全基因组甲基化测序。mbd-seq方法利用mbd蛋白家族可以特异性的结合甲基化dna的特性,从而富集到基因组上cpg高密度的甲基化dna片段,并结合高通量测序技术,对富集到的dna片段进行测序,从而检测全基因组范围内的甲基化水平。目前常用mbd-seq技术进行甲基化dna富集,其方法均是基于需要较高的dna投入,投入量越高,mbd富集效果越好,因此大部分商品化试剂盒制造商会将mbd实验的进入量规定在1000ng以上,但是肿瘤来源的cfdna提取量非常低,不可能实现大规模甲基化dna富集。
技术实现要素:
6.为了解决以上mbd-seq富集cfdna的问题,以及传统wgbs方法检测成本较高等问题,本发明对mbd-seq技术进行了优化,以用于针对肺癌血浆cfdna来源的,且较低投入量的cfdna样本的富集甲基化检测,开发了一种新的cfdna基因检测技术,辅助基于液体活检的早期肺癌的检测。本发明同时找到了通过上述方法获得的具有肺癌检测较高的甲基化标志物。
7.一种肺癌血浆游离dna来源的甲基化检测方法,包括如下步骤:步骤1,制备链霉亲和素磁珠悬浮液,在链霉亲和素磁珠悬浮液中加入mbd蛋白,室温旋转孵育后洗涤后重悬,得到磁珠-蛋白混合物;步骤2,提取得到cfdna,再加入至步骤1中得到的磁珠-蛋白混合物中,孵育后并洗涤;步骤3,对步骤2中的分离得到的磁珠进行洗脱,采用的洗脱液是nacl溶液,洗脱过程步骤是先进行第一浓度洗脱,再进行第二浓度洗脱,收集全部洗脱液;所述的第一浓度是400-600mm,第二浓度是800-1200mm;步骤4,洗脱液中加入醋酸钠溶液、无水乙醇和糖原,使析出沉淀,经过分离、洗涤、干燥、复溶后,得到富集的dna;步骤5,对步骤4中得到的富集的dna进行测序,并进行甲基化分析。
8.所述的步骤1中,磁珠-蛋白混合物以100μl计,其中含有5-15μl mbd蛋白和5-15μl链霉亲和素磁珠。
9.所述的步骤2中,cfdna与磁珠-蛋白混合物的配比是:10-20ng:4-10μl。
10.所述的步骤3中,第一浓度洗脱次数1-3次,第二浓度洗脱次数2-4次。
11.所述的步骤4中,醋酸钠溶液与洗脱液的体积比、无水乙醇与洗脱液的体积比分别为1:8-12、1:1-3;所述的醋酸钠溶液浓度2-4m;沉淀过程于-70℃以下至少2h。
12.检测血浆游离dna来源的甲基化标志物的试剂在用于制备非小细胞肺癌诊断试剂中的应用;所述的甲基化标志物是由基因组上的20个甲基化区域所组成,所述的甲基化区域在基因组上的位置如下所示:
。
13.一种试剂盒,其中包含有用于对以上甲基化区域进行检测的试剂。
14.一种用于肺癌血浆游离dna来源的甲基化检测装置,包括:提取模块:获取肺癌和健康人血浆样本,进行cfdna提取;mbd富集甲基化dna模块:用于通过mbd蛋白对cfdna进行富集,其中通过具有优化的离子浓度的洗脱液进行洗脱,进行富集高甲基化dna;甲基化文库构建模块:将富集到的高甲基化dna通过接头连接构建预文库,然后通过酶转化、pcr扩增构建甲基化文库;测序模块:用于对甲基化文库进行全基因组甲基化高通量测序;质控模块:用于将测序数据接头、低质量序列去除,获得高质量数据;比对模块:用于将测序数据比对到参考基因组,并获得cpg位点上的支持甲基化的reads数和未甲基化的reads数;甲基化率数值计算模块:用于计算每个甲基化区域上的甲基化率;富集峰鉴定模块:用于比较阴性对照和富集样本鉴定出富集到的高甲基化区域;差异分析模块:用于基于富集峰鉴定出差异甲基化区域;
肺癌标志物筛选模块:利用机器学习,创建健康人和肺癌患者分类器,筛选出肺癌特异的甲基化标志物,并构建肺癌诊断回归模型。
15.所述的每个甲基化区域上的甲基化率是根据在这个区域上的全部的发生了甲基化的cpg位点上的有甲基化的reads数除以甲基化与未甲基化的总reads数计算得到。
16.鉴定富集峰可以使用未经mbd蛋白富集的全基因组甲基化测序样本。
17.一种计算机可读取介质,其记载有可以运行对肺癌甲基化状态进行检测的计算机程序,其特征在于,所述的计算机程序包括执行以下步骤:步骤1,获得血浆样品进行提取后获得血浆cfdna;步骤2,提取的cfdna使用优化的mbd富集方法,富集高甲基化dna;步骤3,对富集的高甲基化dna进行甲基化建库和测序后得到的测序数据;步骤4,将测序数据比对到参考基因组获得cpg位点,并获得在所述的cpg位点上的有甲基化的reads数和未甲基化的reads数;步骤5,计算每个甲基化区域上的甲基化率;步骤6,比较阴性对照和富集样本鉴定出富集到的高甲基化区域,并对富集峰进行基因注释;步骤7,基于富集峰进行差异分析,鉴定出健康人和肺癌患者血浆中具有显著性的甲基化区域;步骤8,利用机器学习,创建健康人和肺癌患者分类器,筛选出肺癌特异的甲基化标志物,建立肺癌诊断回归模型。
18.有益效果:本发明首次提供了一种使用优化的mbd-seq技术对微量肺癌血浆cfdna进行甲基化检测的技术。该技术可以更加灵敏的富集甲基化dna,更好的进行甲基化分析,节省样本材料。本发明获得了血浆游离dna来源的甲基化标志物,其具有较高的肺癌诊断准确性。
附图说明
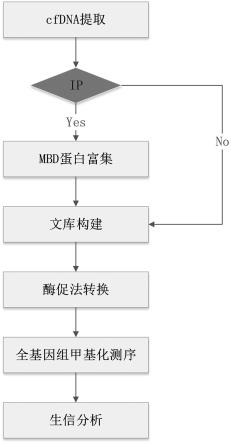
19.图1示出了本专利的流程图;图2示出了mbd富集方法优化前后,cpg位点甲基化率分布图。
20.图3a示出了优化后的mbd方法成功富集到高甲基化区域。
21.图3b示出了mbd富集峰在全基因组范围内的分布图。
22.图4示出了肺癌患者和健康人血浆游离dna的473个dmr热图。
23.图5示出了glm模型下最佳建模dmr组合筛选。
24.图6示出了最佳20个甲基化标志物在肺癌血浆和正常血浆样本中的箱型图。
25.图7a示出了20个甲基化标志物在训练集的受试者工作特征曲线(roc)和相关曲线下面积(auc)。
26.图7b示出了20个甲基化标志物在验证集的受试者工作特征曲线(roc)和相关曲线下面积(auc)。
具体实施方式
27.本发明提供的一种优化的mbd富集方法,该方法可以较好地从低进入量的血浆
cfdna中富集到高甲基化dna,减少了富集过程中dna与mbd蛋白的非特异性结合。这种优化富集方法主要是通过调整cfdna与mbd蛋白、磁珠的质量比,以及选择合适盐离子浓度的洗脱液来实现的。
28.以下的样本富集实验中,2例血浆样本来源于早期非小细胞肺癌患者,当天采血当天分离血浆。mbd富集实验cfdna的投入量为15ng。对照使用mbd说明书推荐进行富集实验,测试组使用优化的mbd富集方法进行实验。
29.提取血浆游离dna,使用qiagen公司的qiaamp circulating nucleic acid 试剂盒。此步骤可以替换成市场上可购买到的其他同类型试剂盒。
30.mbd富集甲基化dna步骤,使用invitrogen公司的methylminer
tm methylated dna enrichment试剂盒。
31.对照例mbd富集实验,富集过程如下:按照试剂盒说明书要求进行实验,步骤:1)针对15ng肺癌cfdna投入量,取10μl链霉亲和素磁珠,上磁力架,吸弃上清液,加入100μl的1x bind/wash buffer进行2次洗涤,最后使用100μl的1x bind/wash buffer悬浮磁珠,制备成磁珠悬浮液;2)取7μl mbd蛋白,加1x bind/wash buffer,使终体积达到100μl。将mbd蛋白加入到步骤1)的100μl的磁珠悬浮液中,室温旋转孵育1h,此时mbd蛋白与磁珠结合在一起,得到磁珠-蛋白混合物;3)将磁珠-蛋白混合物上磁力架,加入1x bind/wash buffer进行2次洗涤,最后使用100μl的1x bind/wash buffer进行悬浮;4)取6μl步骤3)的悬浮液中加入15ng提取的肺癌cfdna,补1x bind/wash buffer至200μl,放入旋转仪中4℃下,过夜孵育;5)将步骤4)的产物上磁力架,收集上清液,并使用1x bind/wash buffer洗涤2次,收集2次的洗涤液,与上清液一起冰上暂存;6)使用250mmnacl浓度的洗脱液对步骤5)的磁珠进行2次洗脱,收集250mm的洗脱液,再用500mm盐离子浓度的洗脱液洗脱2次,收集500mm洗脱液,冰上暂存;7)将步骤5)、6)的回收的上清液、洗涤液、洗脱液分别按照10:1、1:2的比例加入3m的醋酸钠(ph=5.2)和无水乙醇,然后分别加入1μl的糖原(20μg/μl),充分混匀后,-80℃放置至少2h,沉淀富集到的dna。4℃,16000g离心回收沉淀,并使用70%乙醇洗涤沉淀;空气干燥5min,最后加入25μl的无酶水进行沉淀复溶,即得到mbd富集到的高甲基化dna、非/低甲基化dna,-20℃保存。
32.实施例1mbd富集实验,步骤如下:1)取5μl链霉亲和素磁珠,上磁力架,吸弃上清液加入50μl的1x bind/wash buffer进行2次洗涤,并使用50μl的1x binding washing buffer悬浮磁珠;2)将3.5μlmbd蛋白与磁珠混合,补1x bind/wash buffer至100μl,室温进行1h旋转孵育,得到磁珠-蛋白合混合溶液;3)将磁珠-蛋白混合物上磁力架,加入1x bind/wash buffer进行2次洗涤,最终使
用100μl的1x bind/wash buffer进行悬浮;4)取6μl步骤3)磁珠-蛋白合混合溶液,加入15ng提取的肺癌cfdna,补1x bind/wash buffer至200μl,放入旋转仪中4℃下,过夜孵育;5)将步骤4)的产物上磁力架,收集上清液,并使用1x bind/wash buffer洗涤2次,收集2次的洗涤液,与上清液一起冰上暂存;6)优化条件2:使用2组不同盐离子浓度的洗脱液进行洗脱。
33.条件a:先使用250mmnacl浓度的洗脱液对步骤5)的磁珠进行2次洗脱,收集250mm的洗脱液,再用500mmnacl浓度的洗脱液洗脱2次,收集500mm洗脱液,冰上暂存,得到250 500m组合的洗脱液;条件b. 先使用500mm盐离子浓度的洗脱液对步骤5)的磁珠进行2次洗脱,收集500mm洗脱液,再用1000mm盐离子浓度的洗脱液洗脱3次,收集1000mm洗脱液,冰上暂存,得到500 1000mm组合的洗脱液;7)将步骤5)、6)的回收的上清液、洗涤液、洗脱液分别按照10:1、1:2的比例加入3m的醋酸钠(ph=5.2)和无水乙醇,然后分别加入1μl的糖原(20μg/μl),充分混匀后,-80℃放置至少2h,沉淀富集到的dna。4℃,16000g离心回收沉淀,并使用70%乙醇洗涤沉淀;空气干燥5min,最后加入25μl的无酶水进行沉淀复溶,即得到mbd富集到的高甲基化dna、非/低甲基化dna,-20℃保存。
34.8)定量:将对照条件、优化条件下富集的高甲基化dna使用qubit 4.0荧光定量仪及qubit dsdna hs assays试剂盒,按照荧光定量仪说明书操作要求进行浓度定量。
35.文库构建和测序过程:富集到的高甲基化dna、非/低甲基化dna一起进行甲基化建库,使用neb公司的nebnext
® enzymatic methyl-seq试剂盒,并在样本中添加内部对照(试剂盒提供的一段未甲基化的dna片段)。将构建的文库进行甲基化相关的酶促转化,经过pcr扩增,最后获得转化后的甲基化文库。具体操作步骤见试剂盒说明书。
36.对上述构建的血浆游离dna甲基化文库,采用illumina公司novaseq测序仪进行全基因组甲基化测序(whole genome bisulfite sequencing,wgbs)。测序完成下机后,使用bcl2fastq生成fastq文件。用fastqc软件对数据进行质量控制,trimmomatic软件去除接头和低质量序列,得到的cleandata使用bismark进行基因组(hg19)的比对。比对后得到发生了甲基化的cpg位点,再根据获得的位点确定出在每个cpg位点甲基化的reads数和在这个位点区域上未甲基化的reads数,计算每个位点甲基化率,进一步统计不同水平下cpg位点占总cpg数目的占比(图2)。
37.与对照组相比,优化mbd条件在250 500mm盐离子浓度下明显富集到高甲基化dna,且提升盐离子浓度500 1000mm条件下,富集的高甲基化dna占比更高。
38.甲基化样本的检测和标志物的确定:选取22例晚期非小细胞肺癌患者、18例健康人血浆,进行cfdna提取,使用qiagen公司的qiaamp circulating nucleic acid 试剂盒。用实施例1中优化mbd富集方法,使用500 1000mm的盐离子洗脱条件,对15ng的cfdna进行甲基化dna富集。
39.测试组:富集的高甲基化dna全部投入进行甲基化建库,使用neb公司的nebnext
® enzymatic methyl-seq试剂盒,并在样本中添加内部对照(试剂盒提供的一段未甲基化的
dna片段)。对mbd富集的高甲基化dna进行末端修复、末端加碱基a、加接头,经过磁珠纯化后,得到预文库;将构建的预文库进行甲基化相关的酶促转化,获得转化后的预文库;将转化后的预文库进行pcr扩增,获得扩增的甲基化文库产物。文库进行甲基化相关的酶促转化,经过pcr扩增,最后获得转化后的甲基化文库。
40.对照组:使用10ng提取的cfdna直接进行甲基化建库。在样本中添加内部对照,进行末端修复、末端加碱基a、加接头,经过磁珠纯化后,得到预文库;将构建的预文库进行甲基化相关的酶促转化,获得转化后的预文库;将转化后的预文库进行pcr扩增,获得扩增的甲基化文库产物。
41.具体操作步骤见试剂盒说明书。
42.对上述构建的血浆游离dna甲基化文库,采用illumina公司novaseq测序仪进行全基因组甲基化测序(whole genome bisulfite sequencing,wgbs),对照组上机通量30g,测试组上机通量10g。测序完成下机后,使用bcl2fastq生成fastq文件。用fastqc软件对数据进行质量控制,trimmomatic软件去除接头和低质量序列,得到的cleandata使用bismark进行基因组(hg19)的比对。比对后得到发生了甲基化的cpg位点,再根据获得的位点确定出在每个cpg位点甲基化的reads数和在这个位点区域上未甲基化的reads数。然后测试组样本和对照组样本对比,使用软件macs2鉴定富集峰(peaks,图3a和图3b)。图3a深色代表高甲基化,浅色代表低甲基化,对比对照组和测试组可以看到高甲基化区域明显被富集,并且peaks在全基因组范围内均有被富集(图3b)。
43.进一步比较非小细胞肺癌患者和健康人富集峰,鉴定差异甲基化区域(differentially methylated regions, dmrs)。在一个dmr区域内含有一个或多个cpg位点,将在此dmr区域内所有cpg位点甲基化reads数之和除以在此dmr区间内所有cpg位点甲基化与未甲基化总reads数之和,得到dmr的甲基化率。通过以上的测序和数据处理步骤,可以获得每个cfdna样本中的每个dmr的甲基化率。
44.从健康人和非小细胞肺癌患者中分别选取14例和14例作为训练集,剩余4例和8例作为验证集,将训练集样本通过对比健康人和非小细胞肺癌患者富集峰,筛选出有显著性差异dmrs 473个(图4)。健康人与非小细胞肺癌患者可以很好的聚类,且能观察到明显的差异甲基化信号。
45.采用机器学习方法(广义线性模型,glm)创建分类器,对上述筛选得到的473个dmr的预测能力(判断发生肺癌)进行排序,对训练集执行了10次交叉验证计算,然后按10次交叉验证重要性总名次从前到后对候选dmr进行排名(图5)。最终筛选出模型预测准确性最高的,用于非小细胞肺癌诊断的20个最优dmrs(图6)。20个最优dmrs的基因组位置及碱基序列分别如下表所示。
46.采用上述20个dmrs作为诊断非小细胞肺癌cfdna的甲基化标记物,用h2o方法建立诊断回归模型,对验证集和训练集非小细胞肺癌血浆和健康人血浆样本进行诊断评分。如图7a和图7b所示,区分非小细胞肺癌和健康人,训练集结果显示能够稳定达到100%准确,验证集结果显示能够稳定达到100%准确。
再多了解一些
本文用于创业者技术爱好者查询,仅供学习研究,如用于商业用途,请联系技术所有人。