用于治疗神经退行性疾病的组合物和方法
1.关于序列表的声明
2.与本技术相关的序列表以文本格式代替纸质副本提供,并在此通过引用并入本说明书中。含有序列表的文本文件的名称是630264_401wo_sequence_listing_st25.txt。所述文本文件为651kb,创建于2021年2月4日,并通过efs-web以电子方式提交。
背景技术:
3.ataxin-2(atxn2)蛋白是一种细胞质蛋白,是应激颗粒的组成部分。应激颗粒被认为是由蛋白质翻译终止诱导的瞬时亚细胞区室,并且包含许多已知在患有神经退行性疾病的受试者中发生突变的蛋白质(brown和al-chalabi,《新英格兰医学杂志(n engl j med)》(2017)377:162-172)。ataxin-2含有谷氨酰胺残基的序列,所述序列称为多聚谷氨酰胺重复序列,在正常个体中其长度为约22个氨基酸。在患有神经退行性疾病脊髓小脑共济失调-2(sca2)的个体中发现了这种多聚谷氨酰胺重复序列扩展到34或更长的长度。此疾病的特征在于小脑中的浦肯野神经元(purkinje neuron)和其它神经元细胞类型的进行性死亡。患有脊髓小脑共济失调-2的患者出现共济失调、感觉问题和其它临床特征,所述特征会随着时间的推移而恶化。据报道,与正常受试者相比,患有运动神经元疾病肌萎缩性侧索硬化症(als)的个体中ataxin-2多聚谷氨酰胺重复序列的中度扩展的频率显著升高,所述重复序列比在大多数个体中观察到的要长,但比在脊髓小脑共济失调-2受试者中通常观察到的(例如,介于27与33个谷氨酰胺残基之间)要短(elden等人,《自然(nature)》(2010)466:7310)。这表明这些中等长度,即介于正常个体中发现的多聚谷氨酰胺重复序列与脊髓小脑共济失调-2患者中发现的多聚谷氨酰胺重复序列之间的多聚谷氨酰胺重复序列会增加als的风险。目前,sca2和als的治疗选择有限。
技术实现要素:
4.本公开的方面涉及用于调节与脊髓小脑共济失调-2(sca2)、肌萎缩性侧索硬化症(als)和与tdp-43蛋白病相关的病状相关的基因的表达的组合物和方法。具体地,提供了可用于抑制ataxin 2(atxn2)的表达或活性的抑制性核酸。例如,提供了靶向atxn2的一种或多种同种型,例如atxn2同种型的子集或所有atxn2同种型的抑制性核酸。
5.一方面,本公开提供了一种分离的核酸分子,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、
268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、1176-1288、1811-1827、2015、2065、2083、2152、2203和2209。
6.在一些实施方式中,所述抑制性核酸是sirna双链体、shrna、mirna或dsrna。
7.在一些实施方式中,所述抑制性核酸进一步包括过客链序列,任选地其中所述过客链序列选自表1、19、23和24或选自表1、19、23和24并且具有1个到10个插入、缺失、取代、错配、摆动或其任何组合的过客链序列。
8.在一些实施方式中,所述抑制性核酸是人工mirna,其中所述向导链序列包含在mirna骨架序列内。
9.在一些实施方式中,所述人工mirna的所述向导链序列和所述过客链序列包含在mirna骨架序列内。在一些实施方式中,所述mirna骨架序列是mir-155骨架序列、mir-155e骨架序列、mir-155m骨架序列、mir1-1骨架序列、mir-1-1_m骨架序列、mir-100骨架序列、mir-100_m骨架序列、mir-190a骨架序列、mir-190a_m骨架序列、mir-124骨架序列、mir-124_m骨架序列、mir-132骨架序列、mir-9骨架序列、mir-138-2骨架序列、mir-122骨架序列、mir-122_m骨架序列、mir-130a骨架序列、mir-16-2骨架序列、mir-128骨架序列、mir-144骨架序列、mir-451a骨架序列或mir-223骨架序列。
10.在一些实施方式中,所述抑制性核酸是包括以下中的任一个中所示的核酸序列的mirna:seq id no:443-490、1109-1111、1114、1121-1168、1405-1520、1908-2007、2011、2017、2021、2025、2027、2031、2035、2039、2041、2045、2049、2053、2057、2061、2067、2071、2075、2079、2085、2089、2093、2097、2101、2105、2109、2113、2117、2120、2124、2128、2132、2136、2140、2144、2148、2154、2158、2162、2166、2170、2174、2176、2180、2182、2184、2187、2189、2191、2193、2195、2197、2199、2205、2211、2261、2263、2265和2267。
11.在一些实施方式中,编码所述抑制性核酸的所述核酸序列位于所述表达构建体的非翻译区中。在一些实施方式中,所述非翻译区是内含子、5'非翻译区(5'utr)或3'非翻译区(3'utr)。
12.在一些实施方式中,包括编码抑制性核酸的表达构建体的所述分离的核酸进一步包括启动子。在一些实施方式中,所述启动子是rna pol iii启动子(例如,u6、h1等)、鸡-β肌动蛋白(cba)启动子、cag启动子、h1启动子、cd68启动子、人突触蛋白启动子或jet启动子。在一些实施方式中,所述启动子是h1启动子,所述h1启动子包括seq id no:1522的核苷酸113-203、seq id no:1521的核苷酸1798-1888、seq id no:2257的核苷酸113-343或seq id no:2257的核苷酸244-343。
13.在一些实施方式中,所述表达构建体侧接5'腺相关病毒(aav)反向末端重复(itr)序列和3'aav itr序列或其变体。在一些实施方式中,所述itr序列之一缺少功能性末端解链位点。在一些实施方式中,所述itr源自aav血清型,所述aav血清型选自由以下组成的组:aav1、aav2、aav5、aav6、aav6.2、aav7、aav8、aav9、aavrh10、aav11以及其变体。在一些实施方式中,所述5'itr包括seq id no:2257的核苷酸1-106,并且所述3'itr包括seq id no:
2257的核苷酸2192-2358。
14.另一方面,本公开提供了一种载体,所述载体包括如本公开提供的分离的核酸。在一些实施方式中,所述载体是质粒或病毒载体。在一些实施方式中,所述病毒载体是重组腺相关病毒(raav)载体或杆状病毒(baculovirus)载体。在一些实施方式中,所述载体是自互补的raav载体。在一些实施方式中,所述载体(例如,raav载体)进一步包括填充序列。在一些实施方式中,所述填充序列包括seq id no:1522的核苷酸348-2228或seq id no:2257的核苷酸489-2185。在一些实施方式中,所述载体(例如,raav载体)包括seq id no:2257-2260中的任一个的核苷酸序列。
15.另一方面,本公开提供了一种重组腺相关(raav)颗粒,所述颗粒包括如本公开提供的分离的核酸分子或raav载体。在一些实施方式中,所述raav颗粒包括衣壳蛋白。在一些实施方式中,所述衣壳蛋白能够穿过血脑屏障。在一些实施方式中,所述衣壳蛋白是aav9衣壳蛋白或aavrh.10衣壳蛋白。在一些实施方式中,所述raav颗粒转导中枢神经系统(cns)的神经元细胞和/或非神经元细胞。
16.另一方面,本公开提供了一种药物组合物,所述药物组合物包含如本公开提供的分离的核酸、如本公开提供的载体或如本公开提供的raav颗粒,以及任选地药学上可接受的载体。
17.另一方面,本发明提供了一种宿主细胞,所述宿主细胞包括如本公开提供的分离的核酸、如本公开提供的载体或如本公开提供的raav颗粒。
18.另一方面,本公开提供了用于治疗患有或疑似患有神经退行性疾病的受试者的方法,所述方法包括向所述受试者施用如本公开提供的分离的核酸分子、如本公开提供的载体、如本公开提供的raav颗粒或如本公开提供的药物组合物。在一些实施方式中,所述施用包括直接注射到所述受试者的所述cns。在一些实施方式中,所述直接注射是脑内注射、脑实质内注射、鞘内注射、纹状体内注射、软膜下注射或其任何组合。在一些实施方式中,所述直接注射是直接注射到所述受试者的脑脊液(csf)中,任选地其中所述直接注射是脑池内注射、脑室内注射和/或腰椎内注射。在一些实施方式中,所述受试者的特征在于具有atxn2等位基因,所述atxn2等位基因具有至少22个cag三核苷酸重复序列,任选地其中所述atxn2等位基因具有至少24个cag三核苷酸重复序列、至少27个cag三核苷酸重复序列、至少30个cag三核苷酸重复序列或至少33个或更多个cag三核苷酸重复序列。在一些实施方式中,所述神经退行性疾病是脊髓小脑共济失调-2、肌萎缩性侧索硬化症、额颞叶痴呆、原发性侧索硬化症、进行性肌肉萎缩、边缘为主的年龄相关性tdp-43脑病、慢性创伤性脑病、路易体痴呆(dementia with lewy bodies)、皮质基底节变性、进行性核上性麻痹(psp)、关岛型痴呆帕金森氏症als复合征(dementia parkinsonism als complex of guam,g-pdc)、皮克氏病(pick's disease)、海马硬化症、亨廷顿氏病(huntington's disease)、帕金森氏病(parkinson's disease)或阿尔茨海默氏病(alzheimer's disease)。
19.另一方面,本公开提供了一种抑制细胞中atxn2表达的方法,所述方法包括将如本公开提供的分离的核酸分子、如本公开提供的载体、如本公开提供的raav颗粒或如本公开提供的药物组合物递送到所述细胞。在一些实施方式中,所述细胞具有atxn2等位基因,所述atxn2等位基因具有至少22个cag三核苷酸重复序列,任选地其中所述atxn2等位基因具有至少24个cag三核苷酸重复序列、至少27个cag三核苷酸重复序列、至少30个cag三核苷酸
no:443-490、1109-1111、1114、1121-1168、1405-1520、1908-2007、2011、2017、2021、2025、2027、2031、2035、2039、2041、2045、2049、2053、2057、2061、2067、2071、2075、2079、2085、2089、2093、2097、2101、2105、2109、2113、2117、2120、2124、2128、2132、2136、2140、2144、2148、2154、2158、2162、2166、2170、2174、2176、2180、2182、2184、2187、2189、2191、2193、2195、2197、2199、2205、2211、2261、2263、2265和2267。
24.另一方面,本公开提供了一种分离的rna双链体,所述分离的rna双链体包括向导链序列和过客链序列,其中所述向导链序列包括以下中的任一个中所示的核酸序列:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、以及1176-1288、1811-1827、2015、2065、2083、2152、2203和2209,任选地其中所述向导链序列和所述过客链序列通过环区连接以形成包括双链体结构和环区的发夹结构。在一些实施方式中,所述环结构包括6个到25个核苷酸。
25.另一方面,本公开提供了一种试剂盒,所述试剂盒包括容纳本公开所描述的组合物的容器。
附图说明
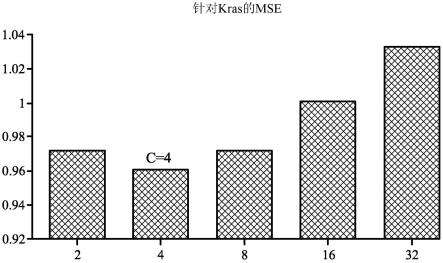
26.图1示出了mir-30数据集的调整均方误差(pelossof等人,《自然生物技术(nature biotechnology)》(2017)35:350-353)。数据来自用于训练支持向量机模型以预测shrna性能的shrna数据集,所述数据示出shrna预测算法对靶向kras的一组shrna的预测性能的均方误差(mse)的数据。均方误差计算为支持向量机(svm)预测器的评分与标签1或-1之间差异的平方,所述标签1或-1与经验确定的产生良好敲低或不良敲低的shrna相对应。对这些平方差在测试的shrna间进行平均。改变超参数c并计算每个值c的均方误差。
27.图2示出了在靶向tile数据集中其它基因的shrna上训练后,应用于靶向trp53基因的留出shrna的svm模型的精度与召回率的图(pelossof等人,《自然生物技术》(2017)35:350-353)。大约0.19处的水平线表示在所有靶向trp53的shrna集中呈阳性,即产生良好敲低的shrna占shrna总数的分数。精度-召回率线表示在给定的svm评分截止点处,数据集中包含的真阳性的分数(“召回率”)与相对于假阳性的真阳性的分数(“精度”)随svm评分的值而变化。因此,在最不严格的svm评分下,所有真阳性都包含在内(召回率=1),但精度很低,因为包含许多阴性shrna。
28.图3示出了针对svm评分绘制的两条曲线。在一条曲线中,示出了随着分类器评分
的增加而预期丢失的阳性shrna的累积分数。这由粗线表示。在另一条曲线中,示出了对低性能shrna排斥的改善百分比。这由较浅色的线表示。从左到右的竖直虚线表示数据集中svm评分的第25个百分位(浅虚线)和第50个百分位(粗体虚线),所述数据集即靶向trp53的shrna。
29.图4示出了svm评分预测的分布随靶向atxn2的shrna序列的向导序列的第一个碱基而变化的抖动图。示出了所有数据点;“小提琴”(“violin”)的水平宽度与每个svm评分处的点数成正比,所述评分绘制在y轴上。在左侧,计算与atxn2序列完全互补的向导序列的评分(位置1处的向导序列碱基是a、u、c或g)。在右侧,如果第一个碱基转化为u,则计算评分(如果位置1处的向导不是天然以u开头,则将位置1处的向导序列碱基编辑为u)。应注意,最初以u开头的向导序列在右图中将具有相同的评分,而以a、g或c开头的序列将具有不同的评分。通常,如果第一个碱基是u,则svm评分会增加。
30.图5示出了跨一组常用细胞系的atxn2 quantigene测定值的图。使用30μl(左条柱)或10μl(右条柱)的裂解物报告信号。
“‑”
表示没有细胞材料的阴性对照。y轴是测定信号。额外的水平线表示最小信号选择标准。
31.图6a至6b示出了来自人脑或来自hepg2.5细胞的ataxin-2转录物的可变剪接的“sashimi”图。图6a:对于脑,示出了来自两个不同个体的代表性图。根据图下方的图表,图中条柱的高度表示与ataxin-2中的位置对齐的读段数。弯曲弧上的数字表示跨外显子-外显子连接对齐的读段计数。在弧线位于顶部和底部两者的连接处,这表明转录物的潜在可变剪接。箭头指出经受大量可变剪接的外显子,使得这些外显子不会出现在人脑中的大量atxn2转录物中。图底部的图表表示转录物enst00000377617.7的结构,其中外显子为实心矩形。转录物从右向左定向,其中外显子1位于右侧。图6b:示出了来自hepg2细胞的类似数据。与转录物的对齐不是按比例的。
32.图7示出了在20nm、1nm和200pm剂量下跨测试的sirna的atxn2 mrna值。x轴示出与对应的sirna互补的atxn2序列(seq id no:2)的位置。atxn2 mrna值表示来自quantigene测定的atxn2与gapdh信号的比率,所述比率相对于模拟对照归一化。x轴上的3'utr示出了atxn2转录物的3'非翻译区的大致位置。
33.图8.atxn2 mrna敲低(相对于模拟转染对照归一化的atxn2与gapdh信号的比率)与svm评分的相关图。观察到预期的相关性,这表明高svm评分预测良好的敲低性能。
34.图9.在指定条件下,从间接免疫荧光获得的来自atxn2 sirna处理的u2os细胞的atxn2信号图。xd-id no表示用对应于表1的不同sirna以指定剂量(20nm(顶部)或1nm(底部))处理。其它处理表示如下:“无_一级_二级”=在抗体染色期间,ataxin-2一级抗体被省略,二级抗体被包含在内;“无_一级_无二级”=在抗体染色期间,ataxin-2一级抗体和针对ataxin-2抗体的二级荧光抗体均被省略;“一级_无二级”=在抗体染色期间,ataxin-2一级抗体被包含在内,但针对ataxin-2抗体的二级荧光抗体被省略;“smp”=从dharmacon获得的具有化学修饰的核苷酸的靶向atxn2的4个sirna的池;“一级_二级”=用一级抗体和二级抗体染色的未经处理的细胞;“ntc”=用“非靶向对照”sirna处理的细胞,所述sirna预期不会靶向任何人转录物,所述sirna具有化学修饰的核苷酸,所述sirna从dharmacon获得;“xd-luccontrol”=一种sirna,其仅由靶向sirna的atxn2中的rna碱基构成,所述sirna预期靶向荧光素酶基因但不靶向atxn2。在图中,每个点表示跨孔中所有细胞的平均信号。在
表6和7中,从跨孔的平均敲低的计算中排除的离群点被示出为浅色点。
35.图10a至10b示出了如图9中所描述的atxn2 sirna处理的u2os细胞的代表性图像。图10a:sirna(20nm)处理的u2os细胞的代表性图像。上图,赫斯特染色(hoechst staining)标定细胞核。下图,atxn2间接免疫荧光。处理/染色程序如下图所示。图10b:如图11a中一样,但所针对的是用1nm的sirna处理的u2os样品。
36.图11示出了归一化的atxn2间接免疫荧光信号随沿atxn2转录物(seq id no:2)的位置而变化的图。x轴限于沿atxn2转录物跨越测试的sirna的结合位点的位置。
37.图12a至12c示出了测试的各种sirna的剂量应答。图12a(上)示出了跨组1中测试的sirna id的log ic50图。条形表示ic50值的95%置信区间的跨度。图12a(下)示出了sirna的代表性剂量应答曲线。y轴表示来自quantigene测定的mrna水平的atxn2与gapdh信号的比率,其来自用指定浓度的sirna给药的hepg2细胞裂解物。拟合表示3参数逻辑回归拟合,其中hill斜率设置为常数1。离群点被自动识别,从曲线拟合和ic50估计中排除。图12b示出了跨组2中测试的sirna id的log ic50图。条形表示ic50值的95%置信区间的跨度。图12c示出了sirna的代表性剂量应答曲线。y轴表示来自quantigene测定的mrna水平的atxn2与gapdh信号的比率,其来自用指定浓度的sirna给药的hepg2细胞裂解物。拟合表示3参数逻辑回归拟合,其中hill斜率设置为常数1。离群点被自动识别,从曲线拟合和ic50估计中排除,并且在图上指示。
38.图13示出了如使用基于网络的服务器mfold创建的嵌入mirna骨架中的向导序列的预测折叠模式。获得了多个折叠预测;示出了代表性折叠。注意向导序列附近每个mirna中若干个位置处的未配对的“凸起”(“bulged”)核苷酸,“密封”(“sealed”)变体除外。
39.图14示出了荧光自动细胞分选数据,其证明通过人工mirna,表达gfp(终止)
–
atxn2报告构建体的u2os细胞系的信号强度降低。用含有插入物的载体转染细胞,所述插入物包含嵌入mirna骨架中的xd-14792(seq id no:112)的向导序列或对照向导序列。y轴绘制每个重复中细胞的中值荧光强度。重复源自含有用载体转染的细胞的96孔板的孔。在facs分析之前用胰蛋白酶解离细胞。
40.图15示出了使用慢病毒包装的atxn2特异性人工mirna在成像实验中区分转导的细胞和未转导的细胞的阈值化程序。慢病毒载体(类似于plvx-ef1a_mcherry-mir-1-1-xd_14890-wpre_cmv(seq id no:546))表达mcherry,并且因此mcherry表达的鉴定可区分转导的细胞和未转导的细胞。左图示出了用于检测mcherry信号的荧光通道中的信号的直方图(包含来自抗mcherry抗体和荧光二级抗体的间接免疫荧光)。右图示出了来自用mcherry编码载体转导的细胞的信号的直方图,其中信号的清晰双峰分布表示未转导的细胞(低信号)和转导的细胞(高信号)。竖直线示出用于将mcherry阳性细胞与mcherry阴性细胞分开的阈值,所述竖直线被放置成使得没有未转导的细胞超过此信号阈值,并且使得转导的细胞中mcherry信号双峰直方图的大部分右峰超过此阈值。
41.图16示出了人工mirna高含量成像测定的atxn2信号归一化程序。每个点表示用于检测atxn2的间接免疫荧光的通道中跨孔中的细胞平均的信号。atxn2敲除细胞用于确定atxn2抗体的间接免疫荧光的背景水平。示出了不同的细胞类型和染色条件,其中y轴归一化,100%设置为来自野生型、未转导的细胞的信号,并且0%设置为来自未转导的atxn2敲除细胞的信号。atxn2抗体染色的atxn2敲除细胞中的信号在一定程度上超过了未用抗体染
色的细胞的信号,从而表明存在与抗体相关的一些背景信号,并且使用atxn2敲除可以帮助修正此背景信号,以提高测量atxn2蛋白信号的准确性。
42.图17a至17b示出了来自用慢病毒载体转导的孔的atxn2信号的图,所述慢病毒载体表达嵌入mirna骨架中的向导序列(在x轴上示出)(mir-155e-图17a;mir1-1-图17b)。表11中列出了向导序列和mirna上下文序列。
43.图18a至18b示出了来自如图17中量化的细胞的赫斯特33342染色(顶部行)、mcherry信号(中间行)和atxn2间接免疫荧光信号(底部行)的代表性图像。图18a示出了嵌入mir-155e骨架中的向导序列的数据;图18b示出了嵌入mir1-1骨架中的向导序列的数据。
44.图19示出了来自嵌入mirna的抗atxn2向导序列的atxn2蛋白信号与来自抗atxn2 sirna处理的atxn2 mrna信号的图。mrna与蛋白质敲低之间存在跨测试的条件的相关性。
45.图20a至20c示出了crispr向导rna在破坏u2os细胞中的ataxin-2基因和敲除ataxin-2蛋白中的验证。图20a示出了与cas9蛋白复合的用靶向atxn2的crispr grna核转染的u2os细胞的蛋白质印迹分析。处理方式包含无核转染对照、靶向cd81或预期非靶向的对照向导rna,以及五种独特的atxn2靶向向导。示出了针对ataxin-2蛋白和α-微管蛋白加载对照的免疫印迹。图20b示出了代表性直方图,并且图20c示出了用如图20a所示的以指定处理方式处理的核转染的细胞的ataxin-2间接免疫荧光信号的孔内的中值荧光强度。
46.图21a至21b示出了为测定校准而生成的u2os atxn2敲除克隆。图21a示出了atxn2 u2os敲除细胞系生成方案。图21b示出了来自在用指定的atxn2靶向grna核转染后生成的克隆系的蛋白质印迹分析。含有来自选择使用的克隆(克隆43)的裂解材料的蛋白质的泳道由箭头指示。
47.图22示出了aav载体化amirna递送后体内ataxin-2蛋白的敲低。通过尾静脉注射将嵌入mir-1-1骨架的编码mirna xd-14792或xd-14887的aav或缺乏mirna的对照构建体静脉内递送到成年野生型小鼠。注射后15天,对动物实施安乐死,并且收集肝脏并速冻。在蓝光照射下,在肝脏中检测到由载体编码的gfp产生的gfp荧光。图22(左):针对ataxin-2、β-肌动蛋白和gfp(未示出),对肝脏裂解物进行免疫印迹。每条泳道都源自不同的动物。图22(右):将ataxin-2信号相对于β-肌动蛋白信号归一化。所有mirna给药的动物的ataxin-2信号都低于用对照aav载体给药的动物。每个点表示来自个体动物的atxn2与β肌动蛋白信号的比率。
48.图23a至23b示出了atxn2靶向mirna的集合库筛选的质量指标(“深度筛选1”)。图23a示出了比较两个重复中高分选样品和低分选样品的比率的散点图,其显示出密切的相关性。图23b示出了所有测试的样品之间的相关矩阵。计算所有样品之间的向导序列计数向量之间的斯皮尔曼相关性(spearman correlation)。
49.图24示出了比率基线减除程序。原始计数比率(对数底数2转换)示出在x轴上,顶部为atxn2靶向mirna,并且底部为乱序mirna。对于随后的计算,减去乱序mirna的比率的中值。
50.图25示出了atxn2信号耗竭与细胞耗竭的图。每个点表示一个库元件,所述库元件含有靶向以下的mirna:atxn2转录物;乱序序列;或靶向必需基因并预期降低细胞增殖和/或活力的序列。x轴是跨源自高atxn2 facs门群和低atxn2 facs门群中细胞的序列计数比率的重复的平均值。y轴是跨初始转导后和16天后源自hela细胞的序列计数比率的重复的
平均值。落向轴底部的点表示相对于初始转导时间点从16天时间点耗竭的元素。
51.图26示出了atxn2信号耗竭与向导序列互补性的atxn2转录物上位置的图。朝底部的点表示具有更大atxn2敲低的向导序列;朝y轴顶部的点表示具有更小atxn2敲低的向导序列。
52.图27示出了与图26中类似的曲线,但在atxn2转录物的3'端上放大。黑色是被认为是所述atxn2转录物的3'utr中“热点”部分的序列。
53.图28示出了跨与在指定位置从所述pri-mirna切除的与向导序列相匹配的乱序向导序列平均的读段百分比。上图示出了示例序列,其中左侧的粗体文本是mir骨架序列,并且常规文本是向导序列。箭头和数字表示切割位置(对于此处描述的平铺筛选,在mir 16-2骨架中,drosha是此切割事件的预期酶)。示出了在预期位置处切割的向导序列的种子序列。如果在与预期位置不同的位置从所述pri-mirna中切除向导位置,则此种子序列的位置将移位。
54.图29示出了用于评估干细胞分化方案中运动神经元产生的代表性图像。左上图像示出了来自抗hb9和抗β3微管蛋白(tuj1)抗体的间接免疫荧光信号的叠加。右上示出了来自抗islet1的信号和tuj1信号的叠加。左下示出了hb9、islet1和tuj1信号的叠加。右下示出了hb9、islet1、tuj1和核dapi染色的叠加。在所述图像中,可以清楚地看到神经元突起被tuj1抗体标记。神经元核由运动神经元标志物hb9和islet1标记,其中25%到35%的神经元被hb9标记,50%到60%的神经元被islet1标记,并且70%到80%的细胞对tuj1信号呈阳性。
55.图30a至30c示出了来自在干细胞源性运动神经元中以慢病毒形式转导靶向atxn2的amirna后测试atxn2 mrna和蛋白质敲低的实验数据。图30a是包装在慢病毒载体中的所述盒的示意图,其中h1启动子驱动人工mirna,随后是pol iii终止信号(6t)。在此mir表达盒之后,cmv pol ii启动子驱动所述荧光报告gfp的表达,然后是wpre元件以稳定所述gfp转录物。图30b示出了来自qpcr的针对atxn2 mrna的数据。每个点表示一个生物学重复,所述重复源自运动神经元的不同组织培养孔。数据表示根据atxn2与gusb或b2m的qpcr阈值(ct)的变化计算的平均信号。条是重复的平均值,并且误差条是跨重复的标准偏差。将atxn2信号相对于从未用载体处理的孔中生长的运动神经元测量的水平归一化。用多克隆位点(mcs)代替amirna的对照慢病毒载体处理的孔中的数据被示出为“mcs”。测试了两个amirna,指示了amirna靶向的atxn2转录物中的指定位置(1784或4402);amir嵌入mir16-2骨架中。靶向atxn2位置1784的向导序列也称为xd-14792。慢病毒载体以两种浓度给药。基于u2os细胞中的滴定(gfp信号的facs分析,计算gfp阳性细胞百分比)计算达到2.5或4.5的感染复数(moi)的病毒剂量。使用这些值和每孔铺板的神经元数量,使用对应剂量的载体以在运动神经元培养物中达到2.5或4.5的moi(基于u2os感染计算)。观察培养物中的gfp荧光证实转导接近完成,如果所述u2os moi与所述运动神经元moi相似,则如预期的那样。图30c示出了来自与图30b中相同处理的培养物的atxn2蛋白评估的评估。上图示出了蛋白质印迹,其明显证明了来自用靶向atxn2的amirna处理的孔中的蛋白质与未处理的孔或用对照mcs载体处理的孔相比,泳道中的信号减少。下图量化了atxn2免疫印迹信号,其中每个点表示一个生物学重复,条表示跨重复的平均值,并且误差条表示标准偏差。
56.图31.数据根据与图30所示类似执行的实验而呈现。在此实验中,moi(根据u2os细
胞中的感染计算)为3.5。示出了用具有靶向指定的atxn2转录物位置的嵌入mir 16-2骨架的amirna的慢病毒载体处理的运动神经元中的敲低。水平虚线表示80%敲低的阈值。在此实验中,很明显,靶向atxn2转录物中3'utr的amirna不会产生与靶向atxn2编码序列的amirna相同水平的敲低。条示出相对于未用慢病毒载体处理的孔归一化的平均敲低;每个点都是一个生物学重复(来自单个孔的神经元),并且误差条是跨重复的标准偏差。如上所述,mcs表示具有代替mir盒的对照多克隆位点的慢病毒载体。
57.图32.2%琼脂糖tae凝胶显示在aav9中包装的嵌入mir16-2骨架的amirna中的截短。将aav基因组dna进行柱纯化并且通过qubit荧光计来进行浓度量化。通过qubit测量,将等量的载体基因组dna加载到凝胶中并进行电泳。应注意,为清楚起见,示出的凝胶图像被拼接在一起。最左侧的泳道是dna大小梯,示出了以千碱基为单位的指示的dna大小。从左到右,样品是(所有dna都源自纯化的aav载体基因组):(1)驱动mir1-1 xd-14792(1784)的h1启动子,后接驱动gfp的cbh启动子;(2)h1启动子,后接非mir多克隆位点,后接填充序列“amely_v1”;(3至11)从左到右,具有靶向atxn2的1784、1479、1755、3330、4402、4405、4406、4409和4502位置的amirna的aav。每个泳道都有靶向atxn2的amirna,其载体基因组形式与泳道2相同,用指定的mir盒替换所述mcs,具有mir16-2骨架。应注意,在来自具有mir16-2骨架mir盒的aav基因组的所有材料中,都存在以预期大小运行的上带,以及更快迁移的下带。
58.图33a至33b.来自深度筛选2的数据示出了重复之间的一致性(图33a)和跨mir骨架的性能(图33b)。在图33a中,每个点表示库元件的相对丰度,其中x轴上的位置表示来自第一个筛选重复的第10个百分位atxn2分选与未分选细胞之间丰度的log2倍数变化,并且y轴表示来自第二个筛选重复的对应的log2倍数变化。图最右侧的点表示分选和未分选细胞的序列计数比率中的分母为0并且因此在对数转换时未定义的数据。对于表现出大量敲低(log2倍数变化《-1)的元素的重复之间存在良好的对应关系,但对于非活性对照(包含靶向amirna的必需基因、911对照和乱序对照),与深度筛选1相比,在此筛选中重复之间的可变性更大。因此,在阴性对照中值中,在筛选重复之间存在一些偏差。由于活性amirna的log2倍数变化值一致,没有进行基线相减。在图33b中,箱形图表示跨嵌入各种mir骨架中的amirna的atxn2敲低性能。在每个箱形图中,中心线是中值,箱的上边缘和下边缘表示第75个和第25个百分位数,并且所述线延伸到箱边缘之外的最大值/最小值或四分位距的1.5倍(第25个与第75个百分位数之间的差值),以更接近中值的为准。重叠点(非常微弱、透明)表示来自单个mirna的atxn2敲低信号。y轴表示在atxn2信号的第10个百分位中检测到的元素的测序读段丰度相对于未分选细胞中向导的丰度之间的平均log2倍数变化。在此筛选中,理论最大倍数变化是第10个百分位分选与未分选细胞之间的10倍。
59.图34.在晚期时间点t1(转导后18天)与早期时间点t0(转导后1天),各种mir骨架中靶向必需基因的amirna的耗竭。y轴表示两个时间点之间丰度的log2倍数变化,并且没有进行基线相减。可以看到如在此图中当表达靶向必需基因的amirna时每个mir骨架在诱导向导随时间耗竭方面的“性能”与如在图33中当表达靶向atxn2的amirna时mir骨架在atxn2敲低方面的性能之间相似的排名。
60.图35.琼脂糖凝胶具有纯化的具有各种mir骨架的aav载体基因组,嵌入的amirna在位置4402(前10个)处靶向atxn2,或靶向位置1784(最后2个;1784向导序列与xd-14792相同)。应注意,为清楚起见,图像被拼接(将包含dna大小梯的泳道放置在紧邻相关泳道的位
置)。一些泳道的带迁移方式与其它泳道不同(mir122、mir1-1-4402、mir-1-1xd14792),这可能是由于加载或染料结合的差异,而不是真正的迁移差异。更重要的是,跨mir骨架,第二强带的相对强度存在差异,比最强烈的上带迁移得更远,所述上带是假定的预期载体基因组。尤其是具有mir100和mir128骨架的aav载体基因组具有比其它更弱的快速迁移带。
61.图36.具有来自顺式质粒池的aav载体基因组的琼脂糖凝胶。每个池包含通过pcr扩增从含有嵌入多个mir骨架中的amir混合物的寡核苷酸池生成的元素,并且使用的pcr引物不区分亲本与“_m”形式的mir骨架。因此,标记为mir-1-1的池将包含在骨架mir-1-1和mir-1-1_m中的amir;标记为mir-100的池将含有mir-100和mir-100_m骨架;标记为mir-190a的池将含有mir-190a和mir-190a_m骨架;池mir-124将含有mir-124和mir-124_m骨架;池mir-138-2将含有mir-138-2和mir-138-2_m骨架。mir-155m和mir-155e虽然彼此不通过“_m”修饰规则相关,但也具有高度序列相似性,因此标记为“mir-155m”的池可能含有mir-155m和mir-155e骨架的混合。每个泳道含有来自aav的纯化载体基因组dna,所述aav用指定的质粒池生成。最后一条泳道源自所述凝胶中显示的5个所述微池以及具有mir骨架mir-124、mir-128、mir-138-2、mir-144和mir-155m的微池的混合物。如图35所示,具有mir-100骨架(虚线框)的aav池与其它aav池相比,具有较弱的快速迁移带。
62.图37.来自深度筛选2的数据,仅包含具有mir-100或mir-100_m骨架的元素。如在图33a中,每个点表示库元件的相对丰度,其中x轴上的位置表示来自第一个筛选重复的第10个百分位atxn2分选与未分选细胞之间丰度的log2倍数变化,并且y轴表示来自第二个筛选重复的对应的log2倍数变化。
63.图38.rt-ddpcr数据表明在用表达靶向atxn2的amirna的scaav-dj载体处理后7天干细胞源性运动神经元中atxn2 mrna的敲低。每个点表示一个生物学重复(以每个细胞指定的载体基因组剂量用aav处理的神经元的孔)。以指定的amirna标记x轴,所述amirna表示为mir骨架-atxn2靶向位置。amirna嵌入自互补的载体基因组中,其中h1启动子驱动amir,并且填充序列从mir盒的psg11、“psg11_v5”(seq id no:2257的核苷酸489-2185)3'修饰到野生型itr。y轴表示rt-ddpcr信号,其中每单位微升每个转录物的拷贝来自对atxn2、gusb或b2m具有特异性的引物/探针集的阳性到阴性液滴的百分比。这些点表示atxn2/gusb和atxn2/b2m比率的平均值。
64.图39.此图示出了除了跨越更广泛的指定剂量之外,与图38相似的rt-ddpcr实验。由于可用细胞数量的限制,并非所有amirna都用所有剂量处理。在此实验中,atxn2 mrna水平通过atxn2/b2m rt-ddpcr比率计算。
65.图40.用如图38和39中的scaav-dj载体处理的干细胞源性运动神经元的图像。用每个细胞1e4载体基因组的剂量处理细胞。dapi染色(标记细胞核)、抗isl1抗体的间接免疫荧光信号(标记运动神经元)和tuj1信号,标记神经元突起的代表性图像。在scaav-dj中用活性靶向atxn2的amirna(1755)和非活性(1755_911)amirna处理的神经元之间的神经元突起没有明显差异。右侧图(顶部)量化了由dapi染色定义的细胞总数,并且(底部)量化了isl1阳性的细胞分数。每个点表示给定孔的跨场的平均量化。星号表示通过单因素方差分析(one-way anova)随后邓尼特多重比较检验(dunnett's multiple comparisons test)计算的与媒剂(pbs 0.001%pf-68)对照的显著(p《.05)差异。载体编码靶向mir100或mir100_m骨架中指定atxn2转录物位置的amirna(图38和39示出了哪些amirna在mir100中,
哪些在mir100_m骨架中)。“pbs”表示用媒剂(pbs 0.001%pf-68)处理的运动神经元的孔;gfp表示包装在scaav-dj中的amirna和gfp表达载体h1-mir1-1.xd-14792-cbh-gfp。
66.图41a至41c.类似于图40,图41a示出了跨用指定的编码特定amirna的scaav-dj载体处理的干细胞源性运动神经元的神经元形态的代表性图像,所述指定amirna嵌入mir100或mir100_m骨架载体中。与媒剂相比,任何处理的神经元形态都没有明显的改变。量化了aav处理的干细胞源性运动神经元中hoechst 核的总数(图41b)和isl1 占总核的百分比(图41c)。
67.图42.示出了rnaseq数据的“火山图”(“volcano plot”),其比较用活性amirna与其非活性“9-11”对照对应物处理的神经元中的基因表达。911对照不降低atxn2水平,但与活性amirna仅相差3个核苷酸(碱基9、10和11)。因此,不涉及碱基9、10和11的amirna的脱靶效应可能与同源非911对照amirna保持一致,并且可以认为所述比较可以富集降低atxn2水平的“中靶”转录影响。到目前为止,在mir100_1755和mir100_2945与其911对照的比较中观察到的最强大的转录效应是atxn2。在图中,每个点表示一个基因(不同转录物的计数按基因折叠);y轴表示标称p值;x轴是条件之间基因表达的log2倍数变化。数据源自n=每次处理5个到6个生物学重复。用1e4载体基因组/细胞的剂量处理神经元,并且7天后收集rna用于rnaseq(quantseq)。
68.图43.一组“火山图”将每个指定的amir aav处理(具有与图42中描述的相同处理条件)与示出的所有其它amirna处理(n=6个重复/条件)进行比较。轴与图42中的一样;水平虚线表示10%的错误发现率阈值。在此,绘制的是每个amir的预测脱靶转录物(在此系统中具有可检测的表达水平),即与所述向导序列的碱基2-18具有互补性且具有2个或更少错配的转录物。对于大多数amirna,没有或只有极少数的预测脱靶相对于一组其它amirna被下调,并且超过10%的错误发现率阈值。
69.图44.atxn2 mrna与来自小鼠的atxn2 amirna表达载体(mir1-1-1784(左)和mir100-3330(右))的生物分布的图,所述小鼠用表达指定amirna aav构建体的载体纹状体内给药。每个点表示从单个纹状体活检中分离的rna和dna的rt-ddpcr mrna和载体分布数据,所述数据取atxn2/gusb和atxn2/tbp液滴比的平均值,相对于媒剂处理的动物归一化。包含了多种不同的载体形式,都带有一个版本的所述h1启动子和各种填充序列。
70.图45a至45b.来自用指定amirna aav构建体给药的动物的纹状体活检的taqman qpcr数据的图(mir1784
–
图45a;mir3330
–
图45b)。对于评估的每个纹状体活检,显示了两个数据点:y轴绘制来自外源性amir和内源性mir、mir124的cdna扩增之间的ct阈值差异;或两个内源性mir的扩增之间的差异。x轴示出如图44中的载体分布数据(的对数底数2转换)。虚线是线性拟合。注意ct与表达之间的关系是一种类似于表达~2^
ct
的形式,与ct差异与log2(载体基因组/二倍体基因组)之间明显的线性关系一致。
71.图46.qpcr数据(图45中所示数据的子集)是针对源自相同纹状体穿孔活检的rna的外源性amir表达/总mir表达的小rnaseq量化绘制的。外源性amir与内源性mir的δct和小rnaseq量化之间的关系分别拟合到每个指定amir的线性模型(线性回归)。两个amir的qpcr与小rnaseq的拟合斜率相似,并且拟合良好,如残差r2所量化。
72.图47.此图示出了对于仅具有通过qpcr测量的amir表达的剩余样品,使用图46中的线性模型来导出预测的绝对amir表达水平随总mir表达而进行的变化。此预测的amir表
达水平绘制在x轴上。每个点表示单独的纹状体穿孔活检。y轴表示所述活检的rt-ddpcr量化的atxn2 mrna水平,与图44中相同。本地散点平滑估计拟合(loess fit)用于单独地将曲线拟合到来自用表达mir1-1.1784的aav(黑色实心圆,虚线);或表达mir100.3330的aav(空心菱形;点线)给药的动物的活检的数据。
73.图48a至48b.在静脉内用表达指示amir的aav给药后2周或3周,从下颌下静脉收集的血液的肝酶数据、丙氨酸转氨酶(alt)(图48a)和天冬氨酸转氨酶(ast)(图48a)。平行监测原初动物。
74.图49.在来自用表达指定amirna的aav给药的动物的纹状体活检中,如图44中的atxn2 mrna敲低和载体分布的图。线表示在r语言中实施的每个系列的loess(本地散点平滑估计)拟合(统计:本地散点平滑估计)。
75.图50a至50b.amirna在用表达指定amirna的aav给药的动物的组织中的表达。分析来自静脉内给药的动物的肝脏组织(图50a);从通过纹状体内注射给药的动物分析纹状体组织(图50b)。amirna表达被绘制为相对于总mirna表达归一化。
76.图51.示出了在纹状体内给药的动物的纹状体组织中加工的mirna的5'端均匀性的图。y轴(log
10
尺度)绘制了在“预期”位置0、预期起始位点的5'(负数)或预期起始位点的3'(正数)处开始的成熟amirna跨所有样品(n=4/aav)的累积测序读段。对于所有这些amir,绝大多数成熟的加工amirna在预期的起始位点处开始。
77.图52a至52d.(上)嵌入mir100骨架中的amirna mir100_1755(图52a)、mir100_2586(图52b)、mir100_2945(图52c)和mir100_3330(图52d)的代表性预测折叠结构(mfold)的图。箭头表示加工的mirna向导链的典型起始位置。(下)观察到的小rnaseq测序读段。左侧是观察到的序列,右侧是跨所有样品的观察次数(n=3-4次肝脏、n=6次纹状体活检)。应注意,序列读段是dna,并且在对应的mirna中,将通过用“u”碱基取代读段中的“t”来生成序列。少数序列是amir与内源性mir之间的融合,但这些序列被认为是小rnaseq程序期间连接反应的人造产物,并且因此被排除在外。通过将观察到的序列与顶部的pri-mirna序列进行比较。应注意的是,在一些情况下会发生3'修饰,如在amirna的3'端处添加“a”或“u”碱基(dna测序读段中的“t”)。
78.图53a至53c示出了aav9载体化mirna递送到脑脊液后体内ataxin-2蛋白的敲低。如图22,嵌入mir-1-1骨架的编码mirna xd-14792或xd-14887的aav或缺乏mirna的对照构建体被给药,在此情况下,在出生后第0天的小鼠中双侧脑室内(icv)注射,每半球3微升。amirna在神经元特异性突触蛋白启动子(如在seq id no:622的核苷酸1128-1575或seq id no:623的核苷酸1128-1575中)或普遍存在的cag启动子的控制下表达。在注射后的指定时间点收获脑组织(皮层)。(图53a)示出了使用的表达盒的图。(图53b)类似于图22的来自蛋白质印迹分析的代表性免疫印迹。针对ataxin-2、β-肌动蛋白和gfp进行免疫印迹。对于每次处理,列出了每个半球施用的剂量,所述剂量通过针对载体基因组中的gfp区域的qpcr滴度计算。在图53c中,示出了相对于总蛋白信号强度(revert 700,licor公司(licor))归一化的atxn2蛋白或gfp蛋白的信号强度的定量。将atxn2信号在指定时间缩放到cag-mcs和syn-mcs对照的平均值,并且将gfp信号在4周时间点缩放到gfp最大值,或者在8周时间点缩放到加载到每个蛋白质印迹上的多个cag-mcs载体iv给药的肝脏样品的平均gfp信号。每个点表示来自单个皮层(来自单个动物)的数据,所述数据跨技术重复取平均值。误差条示出
跨技术重复的标准偏差。对于表达所述xd-14792mir的cag载体在4周和8周时间点,以及对于具有突触蛋白启动子的载体的8周时间点,atxn2水平相对于对照aav载体(mcs)的降低是明显的。
79.图54a至54b示出了来自用aav9对照或如图53中所示表达(mir-1-1骨架中的xd-14792,seq id no:1133)的amirna载体i.c.v.给药的动物的皮层和小脑的组织切片的代表性免疫荧光显微照片。红色对应于抗atxn2抗体的间接免疫荧光信号;绿色对应于抗gfp信号;并且蓝色是核(dapi染色)。在图54a中,可以看到假定的第5层皮质锥体神经元,其中尖端树突在图像中向上突出。来自gfp报告的强度存在于神经元中,所述神经元很可能使用所述aav转导。在左侧,用所述对照amirna转导的动物中表达gfp的神经元也具有强烈的atxn2(红色)信号,并且在gfp和atxn2信号下都可以清楚地看到神经元。相比之下,在对应于用atxn2 amirna(mir-1-1骨架中的xd-14792,seq id no:1133)表达载体给药的动物的组织图像的右侧,具有强gfp强度的神经元也不具有强atxn2强度,并且总体上具有强atxn2信号的神经元的数量似乎减少了。图54b示出了与图54a相似的结果,但捕获小脑中的浦肯野细胞。在右侧,图像示出了注射了atxn2 amirna(mir-1-1骨架中的xd-14792,seq id no:1133)表达载体的动物的小脑组织。gfp标记的aav转导的浦肯野细胞不具有强atxn2信号,而缺乏gfp转导的浦肯野细胞具有强atxn2表达。相比之下,在对应于来自用对照载体给药的动物的图像的左侧,具有gfp信号的细胞也具有atxn2信号。
具体实施方式
80.atxn2多聚谷氨酰胺重复序列扩展到34或更长的长度会导致2型脊髓小脑共济失调(sca2)。此外,atxn2中的中等长度多聚谷氨酰胺扩展会增加als的风险。atxn2水平的降低已被证明在脊髓小脑共济失调-2和als的动物模型中具有治疗益处。使用基于核酸的疗法敲低atxn2蛋白可减轻在表达含有扩展的多聚谷氨酰胺重复序列的人atxn2的变体的动物模型中发生的进行性神经变性。在过表达tdp-43蛋白(als患者中最常见的神经病理学的组分)的als动物模型中,动物通常会出现运动神经元的进行性死亡。然而,用atxn2敲除小鼠繁殖这些动物显著增加了存活时间(elden等人,《自然》(2010)466:7310)。类似地,通过引入反义寡核苷酸核酸来降低atxn2蛋白水平也增加了tdp-43转基因小鼠的存活率。降低atxn2水平可显著延长tdp-43转基因小鼠的寿命并改善运动功能,并减少tdp-43内含物的负担。axtn2可能通过影响tdp-43的聚集倾向来调节毒性。在许多神经退行性疾病中也观察到tdp-43蛋白病,包含als、ftd、原发性侧索硬化症、进行性肌肉萎缩、边缘为主的年龄相关性tdp-43脑病、慢性创伤性脑病、路易体痴呆、皮质基底节变性、进行性核上性麻痹(psp)、关岛型痴呆帕金森氏症als复合征(g-pdc)、皮克氏病、海马硬化症、亨廷顿氏病、帕金森氏病和阿尔茨海默氏病。因此,降低atxn2水平可用于治疗atxn2是致病因子的神经退行性疾病(例如,sca2),以及atxn2不是致病因子但改变tdp-43病理聚集的神经退行性疾病。
81.本发明的方面涉及抑制性核酸(例如,sirna、shrna、mirna,包含人工mirna),所述抑制性核酸在向受试者施用时降低受试者中ataxin-2的表达或活性。因此,本公开中提供的组合物和方法可用于治疗神经退行性疾病,包括2型脊髓小脑共济失调(sca2)、肌萎缩性侧索硬化症(als)、阿尔茨海默氏额颞叶痴呆(ftd)、帕金森氏病和与tdp-43蛋白病相关的病状。
82.在更详细地阐述本公开之前,对本发明的理解可能有助于提供本文所使用的某些术语的定义。贯穿本公开阐述了额外的定义。
83.在本说明书中,任何浓度范围、百分比范围、比率范围或整数范围应当理解为包含所述范围内的任何整数的值,以及在适当时其分数(如整数的十分之一和百分之一),除非另有说明。而且,除非另外指出,否则本文所述的涉及任何物理特征的任何数值范围,如聚合物亚基、尺寸或厚度应当理解为包含所述范围内的任何整数。如本文中所使用的,术语“约”是指所指定范围、值或结构的
±
20%,除非另外指明。应当理解,如本文所用,术语“一个”和“一种”是指所列举的组分中的“一个或多个”。替代方案(例如,“或”)的使用应该理解为意指替代方案中的一个、两个或其任何组合。如本文所使用的,“包含”、“具有”和“包括”同义地使用,所述术语和其变体旨在被解释为非限制性的。
84.如本文所使用的,术语“核酸”或“多核苷酸”是指由共价连接的核苷酸亚基组成的任何核酸聚合物,如聚脱氧核糖核苷酸或多核糖核苷酸。核酸的实例包含rna和dna。
85.如本文所使用的,“rna”是指包括一个或多个核糖核苷酸的分子,并且包含双链rna、单链rna、分离的rna、合成rna、重组rna,以及通过添加、缺失、取代和/或替换一个或多个核苷酸而不同于天然rna的经修饰的rna。rna分子的核苷酸可以包括标准核苷酸或非标准核苷酸,如非天然存在的核苷酸或化学合成的核苷酸。
86.如本文所使用的,“dna”是指包括一个或多个脱氧核糖核苷酸的分子,并且包含双链dna、单链dna、分离的dna、合成dna、重组dna,以及通过添加、缺失、取代和/或替换一个或多个核苷酸而不同于天然dna的经修饰的dna。dna分子的核苷酸可以包括标准核苷酸或非标准核苷酸,如非天然存在的核苷酸或化学合成的核苷酸。
[0087]“分离的”是指从其自然环境中分离的或人工产生的物质。如本文关于细胞所使用的,“分离的”是指已从其自然环境(例如,从受试者、器官、组织或体液)中分离的细胞。如本文关于核酸所使用的,“分离的”是指已从其自然环境(例如,从细胞、细胞器或细胞质)分离的或纯化的、重组产生的、扩增的或合成的核酸。在实施方式中,分离的核酸包含载体内含有的核酸。
[0088]
如本文所使用的,术语“野生型”或“非突变”形式的基因是指编码与正常或非致病性活性相关的蛋白质(例如,没有突变,如导致神经退行性疾病的发展、发作或进展风险更高的重复序列区域扩展的蛋白质)的核酸。
[0089]
如本文所使用的,术语“突变”是指基因结构(例如,基因序列)的产生基因的改变的形式的任何变化,所述基因的改变的形式可以传递给后代(遗传突变)或不传递给后代(体细胞突变)。基因突变包含dna中单个碱基的取代、插入或缺失,或者基因或染色体的多个碱基或更大部分的取代、插入、缺失或重排,包含重复序列扩展。
[0090]
如本文所使用的,术语“ataxin 2”或“atnx2”是指由atxn2基因编码的蛋白质,所述蛋白质含有多聚谷氨酰胺(polyq,cag重复序列)链。atxn2基因或转录物可以指通常具有22个或23个重复序列的atxn2的正常等位基因,或具有中间(~24个至32个重复序列)或更长重复序列扩展(~33个至》100个重复序列)的突变等位基因。在一些实施方式中,atxn2是指哺乳动物atnx2,包含人atxn2。在一些实施方式中,野生型atxn2是指如seq id no:1中所示的q99700.2的蛋白质序列或其天然存在的变体。在一些实施方式中,野生型atxn2核酸是指nm_002973.3(seq id no:2)、enst00000377617.7、enst00000550104.5、
enst00000608853.5或enst00000616825.4或其天然存在的变体的核酸序列。
[0091]
如本文所使用的,术语“抑制性核酸”是指包括向导链序列的核酸,所述向导链序列与靶核酸,例如atxn2 rna、mrna、前mrna或成熟mrna的至少一部分杂交并且抑制其表达或活性。抑制性核酸可以靶向靶核酸的蛋白质编码区(例如,外显子)或非编码区(例如,5'utr、3'utr、内含子等)。在一些实施方式中,抑制性核酸是单链或双链分子。抑制性核酸可以进一步包括在单独链(例如,双链双链体)上或同一链(例如,单链、自退火双链体结构)中的过客链序列。在一些实施方式中,抑制性核酸是rna分子,如sirna、shrna、mirna或dsrna。
[0092]
如本文所使用的,“微小rna”或“mirna”是指能够通过靶mrna的切割、靶mrna的翻译抑制、靶mrna降解或其组合介导靶基因沉默的小非编码rna分子。通常,mirna转录为发夹或茎环(例如,具有自互补的单链骨架)双链体结构,所述结构称为初级mirna(pri-mirna),经过酶加工(例如,通过drosha、dgcr8、pasha等)成mirna前体(pre-mirna)。mirna前体被输出到细胞质中,在那里其由dicer酶加工以产生具有过客链的mirna双链体,然后是单链成熟mirna分子,所述单链成熟mirna分子随后被加载到rna诱导的沉默复合物(risc)中。所提及的mirna可以包含合成或人工mirna。
[0093]
如本文所使用的,“合成mirna”或“人工mirna”或“amirna”是指内源性、经修饰的或合成的pri-mirna或mirna前体(例如,mirna骨架或支架),其中茎序列中的内源性mirna向导序列和过客序列已被mirna向导序列和指导靶基因的高效rna沉默的mirna过客序列替代(参见例如,eamens等人(2014),《分子生物学方法(methods mol.biol.)》1062:211-224)。在一些实施方式中,所述向导序列和过客序列的互补性性质(例如,碱基数量、错配位置、凸起类型等)可以与构建所述合成mirna的内源性mirna骨架中所述向导序列和过客序列的互补性性质相似或不同。
[0094]
如本文所使用的,术语“微小rna骨架”、“mir骨架”、“微小rna支架”或“mir支架”是指茎序列被所关注的mirna替代的pri-mirna或mirna支架前体,并且能够产生指导被所关注的mirna靶向的基因处的rna沉默的功能性成熟mirna。mir骨架包括5'侧接区(也称为5'mir上下文,≥9个核苷酸)、包括mirna双链体(向导链序列和过客链序列)和基底茎(5'和3',每个约4个至13个核苷酸)的茎区、包含末端环(对于末端环,》10个核苷酸)的至少一个环基序区、3'侧接区(也称为3'mir上下文,≥9个核苷酸)以及任选地茎中的一个或多个凸起。mir骨架可以完全或部分源自野生型mirna支架或是完全人工序列。
[0095]
如本文所使用的,抑制性核酸的术语“反义链序列”或“向导链序列”是指与靶向用于沉默的所述基因的所述mrna的约10个到50个核苷酸(例如,约15个到30个、16个到25个、18个到23个或19个到22个核苷酸)区基本上互补(例如,至少45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%或100%互补)的序列。所述反义序列与靶mrna序列充分互补以指导靶特异性沉默,例如,以通过rnai机制或过程触发靶mrna的破坏。在一些实施方式中,所述反义序列或向导链序列是指被dicer切割后剩余的所述成熟序列。
[0096]
如本文所使用的,术语抑制性核酸的“有义序列”或“过客链序列”是指与所述靶mrna同源并且与抑制性核酸的所述反义链序列或向导链序列部分或完全互补的序列。抑制性核酸的所述反义链序列和有义链序列杂交形成双链体结构(例如,形成双链双链体或单链自退火双链体结构)。在一些实施方式中,所述有义序列或过客链序列是指被dicer切割后剩余的所述成熟序列。
[0097]
如本文所使用的,“双链体”在用于提及抑制性核酸时是指杂交在一起以形成双链体结构的两条核酸链(例如,向导链和过客链)。双链体可以由两条单独的核酸链或由具有自互补的区(例如,发夹或茎环)的单个核酸链形成。
[0098]
如本文所使用的,术语“互补”是指多核苷酸彼此形成碱基对的能力。碱基对通常由反平行多核苷酸链或单个自退火多核苷酸链中的核苷酸亚基之间的氢键形成。互补多核苷酸链可以以沃森-克里克方式(watson-crick manner)(例如,a与t、a与u、c与g)或以允许形成双链体的任何其它方式形成碱基对。如本领域技术人员显而易见的,当使用rna而不是dna时,认为与腺苷互补的碱基是尿嘧啶而不是胸腺嘧啶。此外,当在本发明的上下文中表示“u”时,可以理解其取代“t”的能力,除非另外说明。互补性还涵盖未经修饰的核碱基与经修饰的核碱基之间的沃森-克里克碱基配对(例如,5-甲基胞嘧啶替代胞嘧啶)。两条多核苷酸链之间的完全互补、完美互补或100%互补是其中一条多核苷酸链的每个核苷酸可以与第二条多核苷酸链的核苷酸单元形成氢键。互补性百分比是指核酸分子中与对齐的参考序列(例如,靶mrna、过客链)互补的连续核苷酸序列的核苷酸数除以核苷酸总数并乘以100。在此类对齐中,不形成碱基对的核碱基/核苷酸称为错配。在计算连续核苷酸序列的互补性百分比时,不允许插入和缺失。本领域技术人员可以理解,在计算互补性时,只要保留核碱基的沃森-克里克碱基配对能力,就不会考虑对核碱基的化学修饰(例如,为了计算互补性百分比,5-甲基胞嘧啶被认为与胞嘧啶相同)。
[0099]
两个或更多个核酸序列之间的“同一性百分比”是指由参考序列共享的核酸分子中连续核苷酸序列的核苷酸比例(即,同一性百分比=相同核苷酸的数量/对齐的区中核苷酸的总数(例如,连续核苷酸序列)
×
100)。在计算连续核苷酸序列的同一性百分比时,不允许插入和缺失。本领域技术人员可以理解,在计算统一性时,只要保留核碱基的沃森-克里克碱基配对能力,就不会考虑对核碱基的化学修饰(例如,为了计算同一性百分比,5-甲基胞嘧啶被认为与胞嘧啶相同)。
[0100]
如本文所使用的,术语“杂交(hybridizing或hybridizes)”是指两条核酸链在反平行链上的碱基对之间形成氢键,从而形成双链体。两条核酸链之间的杂交强度可以通过解链温度(tm)来描述,所述解链温度定义为在给定的离子强度和ph下,50%的靶序列与互补多核苷酸杂交的温度。
[0101]
如本文所使用的,“表达构建体”是指任何类型的含有核酸的基因构建体(例如,转基因),其中部分或全部核酸编码序列能够被转录。在一些实施方式中,表达包含核酸的转录,例如以从转录的基因产生生物活性多肽产物或抑制性rna(例如,sirna、shrna、mirna)。在一些实施方式中,转基因与表达控制序列可操作地连接。
[0102]
如本文所使用的,术语“转基因”是指已经自然地或通过基因工程手段转移到另一个细胞中并且能够被转录和任选地翻译的外源性核酸。
[0103]
如本文所使用的,术语“基因表达”是指核酸从核酸分子转录并且通常翻译成肽或蛋白质的过程。所述过程可以包含转录、转录后控制、转录后修饰、翻译、翻译后控制、翻译后修饰或其任何组合。提及“基因表达”的测量可以指转录产物(例如,rna或mrna)、翻译产物(例如,肽或蛋白质)的测量。
[0104]
如本文所使用的,术语“抑制基因的表达”是指减少、下调、抑制、阻断、降低或停止所述基因的表达。基因的所述表达产物可以是从所述基因转录的rna分子(例如,mrna)或从
所述基因转录的mrna翻译的多肽。通常,mrna水平的降低导致从其翻译的多肽水平的降低。可以使用用于测量mrna或蛋白质的标准技术来确定表达水平。
[0105]
如本文所使用的,“载体”是指能够在细胞之间转运核酸分子(例如,编码抑制性核酸的转基因)并且当可操作地连接到合适的表达控制序列时影响所述核酸分子表达的基因构建体。表达控制序列可以包含转录起始、终止、启动子和增强子序列;有效的rna加工信号,如剪接和聚腺苷酸化(polya)信号;稳定细胞质mrna的序列;增强翻译效率的序列(即,kozak共有序列);增强蛋白稳定性的序列;以及在需要时,增强所编码产物的分泌的序列。载体可以是质粒、噬菌体颗粒、转座子、粘粒、噬菌粒、染色体、人工染色体、病毒、病毒粒子等。一旦转化到合适的宿主细胞中,所述载体可以独立于宿主基因组进行复制和发挥作用,或者可以在一些情况下整合到所述基因组本身中。
[0106]
如本文所使用的,“宿主细胞”是指含有或能够含有所关注的组合物(例如抑制性核酸)的任何细胞。在实施方式中,宿主细胞是哺乳动物细胞,如啮齿动物细胞(小鼠或大鼠)或灵长类动物细胞(猴、黑猩猩或人)。在实施方式中,宿主细胞可以在体外或体内。在实施方式中,宿主细胞可以来自已建立的细胞系或原代细胞。在实施方式中,宿主细胞是cns的细胞,如神经元、神经胶质细胞、星形胶质细胞和小胶质细胞。
[0107]
如本文所使用的,“神经退行性疾病”或“神经退行性病症”是指表现出神经细胞死亡作为病理状态的疾病或病症。神经退行性疾病可以表现出慢性神经变性,例如,在若干年的时间段内缓慢的进行性神经细胞死亡,或者急性神经变性,例如,突然发作或神经细胞死亡。慢性神经退行性疾病的实例包含阿尔茨海默氏病、帕金森氏病、亨廷顿氏病、2型脊髓小脑共济失调(sca2)、额颞叶痴呆(ftd)和肌萎缩性侧索硬化症(als)。慢性神经退行性疾病包括以tdp-43蛋白病为特征的疾病,其特征在于细胞核到细胞质的错误定位、泛素化和高度磷酸化的tdp-43沉积到包涵体中、蛋白质截短导致毒性c端tdp-43片段的形成,以及蛋白质聚集。tdp-43蛋白病疾病包含als、ftd、原发性侧索硬化症、进行性肌肉萎缩、边缘为主的年龄相关性tdp-43脑病、慢性创伤性脑病、路易体痴呆、皮质基底节变性、进行性核上性麻痹(psp)、关岛型痴呆帕金森氏症als复合征(g-pdc)、皮克氏病、海马硬化症、亨廷顿氏病、帕金森氏病和阿尔茨海默氏病。急性神经变性可能由缺血(例如,中风、外伤性脑损伤)、脱髓鞘引起的轴突横断或外伤(例如,脊髓损伤或多发性硬化)引起。神经退行性疾病可以表现出主要一种类型的神经元或多种类型的神经元的死亡。
[0108]
如本文所使用的,“受试者”、“患者”和“个体”在本文中可互换使用,并且是指选择用于治疗或疗法的活生物体(例如,哺乳动物)。受试者的实例包含人和非人哺乳动物,如灵长类动物(猴、黑猩猩)、牛、马、羊、狗、猫、大鼠、小鼠、豚鼠、猪以及其转基因物种。
[0109]
抑制性核酸
[0110]
一方面,本公开提供了抑制ataxin 2(atxn2)的表达或活性的分离的抑制性核酸。抑制性核酸是与如atxn2 rna、mrna前体、mrna等所述atxn2核酸的至少一部分特异性结合(例如,杂交)并抑制其表达或活性的核酸。在一些实施方式中,所述抑制性核酸与atxn2的蛋白质编码区或非编码区(例如,5'utr、3'utr、内含子等)互补。在一些实施方式中,所述抑制性核酸与野生型atxn2核酸或其天然存在的变体互补。在一些实施方式中,所述atxn2基因编码由ncbi参考序列np_002964.4或np_002964.3确定的多肽。在一些实施方式中,atxn2转录物包括在seq id no:2中所示的序列或编码在seq id no:1中所示的氨基酸的序列。在
一些实施方式中,所述atxn2等位基因含有大约22个cag三核苷酸重复序列。在一些实施方式中,所述atxn2等位基因具有至少22个cag三核苷酸重复序列、至少24个cag三核苷酸重复序列、至少27个cag三核苷酸重复序列、至少30个cag三核苷酸重复序列或至少33个或更多个cag三核苷酸重复序列。在一些实施方式中,抑制性核酸是单链的或双链的。在一些实施方式中,所述抑制性核酸是sirna、shrna、mirna或dsrna。
[0111]
在一些实施方式中,与未与所述抑制性核酸接触的细胞中atxn2的表达水平相比,所述抑制性核酸能够将细胞中atxn2的表达或活性抑制至少10%、至少15%、至少20%、至少25%、至少30%、至少35%、至少40%、至少45%、至少50%、至少60%、至少70%、至少80%、至少90%、至少95%或更多。在一些实施方式中,与未与所述抑制性核酸接触的细胞中atxn2的表达水平相比,所述抑制性核酸能够将atxn2的表达或活性抑制10-20%、10-30%、10-40%、10-50%、10-60%、10-70%、10-80%、10-90%、10-95%、20-30%、20-40%、20-50%、20-60%、20-70%、20-80%、20-90%、20-95%、20-100%、30-40%、30-50%、30-60%、30-70%、30-80%、30-90%、30-95%、30-100%、40-50%、40-60%、40-70%、40-80%、40-90%、40-95%、40-100%、50-60%、50-70%、50-80%、50-90%、50-95%、50-100%、60-70%、60-80%、60-90%、60-95%、60-100%、70-80%、70-90%、70-95%、70-100%、80-90%、80-95%、80-100%、90-95%、90-100%。测量atxn2表达,例如rna、mrna多肽的水平的方法是本领域已知的,包括本文所描述的那些方法。
[0112]
在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括表1、3、9、11、12、13、19、23、24和25中的向导序列中的任一个中所示的核酸序列或由其组成。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、1176-1288、1811-1827、2015、2065、2083、2152、2203和2209。
[0113]
在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括表1、3、9、11、12、13、19、23、24和25中的向导序列中的任一个,例如以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、
180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、1176-1288、1811-1827、2015、2065、2083、2152、2203和2209。
[0114]
在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括与表1、3、9、11、12、13、19、23、24和25中的向导序列中的任一个,例如以下中的任一个具有至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%同一性的核酸序列或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、1176-1288、1811-1827、2015、2065、2083、2152、2203和2209。
[0115]
在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括表1、3、9、11、12、13、19、23、24和25中的向导序列中的任一个,例如以下中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、1176-1288、1811-1827、2015、2065、2083、2152、2203和2209,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。
[0116]
在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括表1、3、9、11、12、13、19、23、24和25的向导序列中的任一个,例如以下中的任一个的序列或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、1176-1288、1811-1827、2015、2065、2083、2152、2203和2209,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0117]
在一些实施方式中,所述抑制性核酸包括表12的向导链序列。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括与seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0118]
在一些实施方式中,所述抑制性核酸包括表13的向导链序列。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:14、40、100、108、112、128、166、198、242、308、336和362。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:
seq id no:14、40、100、108、112、128、166、198、242、308、336和362。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括与seq id no:14、40、100、108、112、128、166、198、242、308、336和362中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括seq id no:14、40、100、108、112、128、166、198、242、308、336和362中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括seq id no:14、40、100、108、112、128、166、198、242、308、336和362中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0119]
在一些实施方式中,所述抑制性核酸包括表19的向导链序列。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:1176-1288、40、108和166。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:1176-1288、40、108和166。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括与seq id no:1176-1288、40、108和166中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括seq id no:1176-1288、40、108和166中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括seq id no:1176-1288、40、108和166中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0120]
在一些实施方式中,所述抑制性核酸包括表23的向导链序列。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:1908-2007。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的与靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:1908-2007。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括与seq id no:1908-2007中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括seq id no:1908-2007中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括seq id no:1908-2007中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再
与所述atxn2靶序列互补。
[0121]
在一些实施方式中,所述抑制性核酸包括表24的向导链序列。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括与seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0122]
在一些实施方式中,所述抑制性核酸包括表25的向导链序列。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括与seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0123]
在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:1185、1816、1213和1811。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:1185、1816、1213和1811。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括与seq id no:1185、1816、1213和1811中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括seq id no:1185、1816、1213和1811中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括seq id no:1185、1816、1213和1811中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0124]
在一些实施方式中,所述抑制性核酸是靶向atxn2 mrna以通过mrna降解或翻译抑制干扰atxn2表达的分离的sirna双链体。sirna双链体是短的双链rna,所述短的双链rna包括与所述靶atxn2 mrna互补的向导链和与所述靶atnx2 mrna同源的过客链。所述向导链和过客链杂交在一起以形成双链体结构,并且所述向导链与所述atxn2mrna序列具有足够的互补性以指导atxn2特异性rna干扰。所述sirna双链体的所述向导链的长度为约18个核苷酸、19个核苷酸、20个核苷酸、21个核苷酸、22个核苷酸、23个核苷酸、24个核苷酸、25个核苷酸、26个核苷酸、27个核苷酸、28个核苷酸、29个核苷酸或30个核苷酸,或者长度为18-30、18-29、18-28、18-27、18-26、18-25、18-24、18-23、18-22、18-20、19-30、19-29、19-28、19-27、19-26、19-25、19-24、19-23、19-22、19-21、20-30、20-29、20-28、20-27、20-26、20-25、20-24、20-23、20-22、21-30、21-29、21-28、21-27、21-26、21-25、21-24、21-23、22-30、22-29、22-28、22-27、22-26、22-24、23-30、23-29、23-28、23-27、23-26、23-25、24-30、24-29、24-28、24-27、24-26、25-30、25-29、25-28、25-27、26-30、26-29、26-28、27-30、27-29、28-30个核苷酸。所述sirna双链体的所述过客链的长度可以是约18个核苷酸、19个核苷酸、20个核苷酸、21个核苷酸、22个核苷酸、23个核苷酸、24个核苷酸、25个核苷酸、26个核苷酸、27个核苷酸、28个核苷酸、29个核苷酸或30个核苷酸,或者长度为18-30、18-29、18-28、18-27、18-26、18-25、18-24、18-23、18-22、18-20、19-30、19-29、19-28、19-27、19-26、19-25、19-24、19-23、19-22、19-21、20-30、20-29、20-28、20-27、20-26、20-25、20-24、20-23、20-22、21-30、21-29、21-28、21-27、21-26、21-25、21-24、21-23、22-30、22-29、22-28、22-27、22-26、22-24、23-30、23-29、23-28、23-27、23-26、23-25、24-30、24-29、24-28、24-27、24-26、25-30、25-29、25-28、25-27、26-30、26-29、26-28、27-30、27-29、28-30个核苷酸。在一些实施方式中,所述sirna双链体在每条链上含有2个或3个核苷酸3'突出端。在一些实施方式中,3'突出端与所述atxn2转录物互补。在一些实施方式中,所述sirna双链体的所述向导链和所述过客链彼此至少60%、65%、70%、75%、80%、85%、90%、95%、97%或100%互补,不包含突出端的任何核苷酸。
[0125]
在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括表1、
3、9、11、12、13、19、23、24和25的向导序列中的任一个,例如以下中的任一个中所示的核酸序列或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、1176-1288、1811-1827、2015、2065、2083、2152、2203和2209。
[0126]
在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括表1、3、9、11、12、13、19、23、24和25的向导序列中的任一个,例如以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、1176-1288、1811-1827、2015、2065、2083、2152、2203和2209。
[0127]
在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括与表1、3、9、11、12、13、19、23、24和25的向导序列中的任一个,例如以下中的任一个具有至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%同一性的核酸序列或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、
330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、1176-1288、1811-1827、2015、2065、2083、2152、2203和2209。
[0128]
在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括表1、3、9、11、12、13、19、23、24和25的向导序列中的任一个,例如以下中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、1176-1288、1811-1827、2015、2065、2083、2152、2203和2209,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。
[0129]
在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括表1、3、9、11、12、13、19、23、24和25的向导序列中的任一个向导序列,例如以下中的任一个的序列或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、1176-1288、1811-1827、2015、2065、2083、2152、2203和2209,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0130]
在一些实施方式中,所述sirna双链体包括表12的向导链序列。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、
242、302、306、308、330、336和362。在一些实施方式中,sirna双链体包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括与seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0131]
在一些实施方式中,所述sirna双链体包括表13的向导链序列。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:14、40、100、108、112、128、166、198、242、308、336和362。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:14、40、100、108、112、128、166、198、242、308、336和362。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括与seq id no:14、40、100、108、112、128、166、198、242、308、336和362中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括seq id no:14、40、100、108、112、128、166、198、242、308、336和362中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括seq id no:14、40、100、108、112、128、166、198、242、308、336和362中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0132]
在一些实施方式中,所述sirna双链体包括表19的向导链序列。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:1176-1288、40、108和166。在一些实施方式中,sirna双链体包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:1176-1288、40、108和166。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括与seq id no:1176-1288、40、108和166中的任一个至少60%、70%、75%、80%、85%、90%、95%、
97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括seq id no:1176-1288、40、108和166中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括seq id no:1176-1288、40、108和166中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0133]
在一些实施方式中,所述sirna双链体包括表23的向导链序列。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:1908-2007。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:1908-2007。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括与seq id no:1908-2007中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括seq id no:1908-2007中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括seq id no:1908-2007中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0134]
在一些实施方式中,所述sirna双链体包括表24的向导链序列。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括与seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,sirna双链体包括向导链序
列,所述向导链序列包括seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0135]
在一些实施方式中,所述sirna双链体包括表25的向导链序列。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括与seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,sirna双链体包括向导链序列,所述向导链序列包括seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0136]
在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:1185、1816、1213和1811。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:1185、1816、1213和1811。在一些实施方式中,sirna双链体包括向导链序列,所述向导链序列包括与seq id no:1185、1816、1213和1811中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括seq id no:1185、1816、1213和1811中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括seq id no:1185、1816、1213和1811中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0137]
在一些实施方式中,所述sirna双链体包括表1、19、23和24提供的sirna双链体中的任一个sirna双链体的向导链序列和过客链序列。在一些实施方式中,所述sirna双链体包括向导链序列和过客链序列,其包括以下中的任一个:seq id no:12和11;seq id no:14和13;seq id no:40和39;seq id no:60和59;seq id no:100和99;seq id no:104和103;
seq id no:108和107;seq id no:112和111;seq id no:124和123;seq id no:126和125;seq id no:128和127;seq id no:166和165;seq id no:198和197;seq id no:220和219;seq id no:242和241;seq id no:302和301;seq id no:306和305;seq id no:308和307;seq id no:330和320;seq id no:336和335;以及seq id no:362和361。在一些实施方式中,所述sirna双链体包括向导链序列和过客链序列,其包括以下中的任一个:seq id no:14和13;seq id no:40和39;seq id no:100和99;seq id no:108和107;seq id no:112和11;seq id no:128和127;seq id no:166和165;seq id no:198和197;seq id no:242和241;seq id no:308和307;seq id no:336和335;以及seq id no:362和361。
[0138]
表1:atxn2特异性sirna双链体序列
[0139]
[0140]
[0141]
[0142]
[0143]
[0144]
[0145]
[0146]
[0147]
[0148][0149]
在一些实施方式中,本公开的所述分离的sirna双链体,特别是当不作为表达构建体或在载体内递送时,包括至少一个经修饰的核苷酸,包含经修饰的碱基、经修饰的糖或经修饰的骨架。具有核苷酸修饰的sirna可能具有增加的稳定性、增加的特异性、降低的免疫原性或其组合。经修饰的核苷酸可以出现在向导链、过客链或向导链和过客链两者上。
[0150]
经修饰的碱基是指已通过替代或添加一个或多个原子或基团而被修饰的核苷酸碱基,例如腺嘌呤、鸟嘌呤、胞嘧啶、胸腺嘧啶、尿嘧啶、黄嘌呤、肌苷和辫苷。核碱基部分修饰的一些实例包括但不限于单独或组合的烷基化、卤化、硫醇化、胺化、酰胺化或乙酰化碱基。更具体的实例包含例如5-丙炔基尿苷、5-丙炔基胞苷、6-甲基腺嘌呤、6-甲基鸟嘌呤、n,n,-二甲基腺嘌呤、2-丙基腺嘌呤、2-丙基鸟嘌呤、2-氨基腺嘌呤、1-甲基肌苷、3-甲基尿苷、5-甲基胞苷、5-甲基尿苷和在5位置处有修饰的其它核苷酸、5-(2-氨基)丙基尿苷、5-卤代胞苷、5-卤代尿苷、4-乙酰胞苷、1-甲基腺苷、2-甲基腺苷、3-甲基胞苷、6-甲基尿苷、2-甲基鸟苷、7-甲基鸟苷、2,2-二甲基鸟苷、5-甲基氨基乙基尿苷、5-甲氧基尿苷、脱氮核苷酸,例如7-脱氮-腺苷、6-氮杂核苷、6-偶氮胞苷、6-偶氮胸苷、5-甲基-2-硫代尿苷、其它硫碱基,如2-硫代尿苷和4-硫代尿苷和2-硫代胞苷、二氢尿苷、假尿苷、辫苷、古嘌苷、萘基和经取代的萘基、任何o-和n-烷基化的嘌呤和嘧啶,如n6-甲基腺苷、5-甲基羰基甲基尿苷、尿苷5-氧乙酸、吡啶-4-酮、吡啶-2-酮、苯基和修饰的苯基,如氨基苯酚或2,4,6-三甲氧基苯,作为g-钳位核苷酸的经修饰的胞嘧啶、8-经取代的腺嘌呤和鸟嘌呤、5-经取代的尿嘧啶和胸腺嘧啶、氮杂嘧啶、羧基羟基烷基核苷酸、羧基烷基氨基烷基核苷酸和烷基羰基烷基化核苷酸。糖修饰的核苷酸包括但不限于2'-氟、2'-氨基和2'-硫修饰的核糖核苷酸,例如,2'-氟修饰的核糖核苷酸。
[0151]
经修饰的核苷酸可以在糖部分上进行修饰,也可以是具有非核糖基糖或其类似物的核苷酸。例如,糖部分可以是或基于甘露糖、阿拉伯糖、吡喃葡萄糖、吡喃半乳糖、4'-硫代核糖和其它糖、杂环或碳环。
[0152]
如本文所使用的,正常“骨架”是指dna或rna分子中重复交替的糖-磷酸序列。脱氧核糖/核糖在3'-羟基和5'-羟基处与酯链中的磷酸基团连接,也称为“磷酸二酯”键或连接。一个或多个或所有磷酸二酯键可以被修饰为硫代磷酸酯键、硼代磷酸酯键、酰胺键、二硫代磷酸酯键或三唑键。
[0153]
在一些实施方式中,所述抑制性核酸是shrna。在一些实施方式中,所述shrna是茎-环双链体分子,所述茎-环双链体分子包括通过间隔子序列(即,环)连接的如本文提供
200、60-100、60-90、60-80、70-500、70-400、70-300、70-200、70-100、70-90、80-500、80-400、80-300、80-200、80-100、90-500、90-400、90-300、90-200、100-500、100-400、100-300、100-200、200-500、200-400、200-300、300-500、300-400或400-500个核苷酸。
[0156]
所述mirna前体通过输出蛋白-5从细胞核转运到细胞质,并由dicer进一步加工以产生包括向导链和过客链的成熟双链mirna双链体。然后将成熟mirna双链体并入由trbp(hiv反式激活应答rna结合蛋白)介导的rna诱导沉默复合物(risc)中。所述过客链通常被释放和切割,而所述向导链保留在risc中并与所述靶mrna结合并介导沉默。在一些实施方式中,成熟mirna是指成熟mirna双链体的向导链。在一些实施方式中,成熟mirna的长度为约19个、20个、21个、22个、23个、24个、25个、26个、27个、28个、29个或30个核苷酸,或者长度范围为约19个到30个核苷酸、19个到29个核苷酸、19个到28个核苷酸、19个到27个核苷酸、19个到26个核苷酸、19个到25个核苷酸、19个到24个核苷酸、19个到23个核苷酸、19个到21个核苷酸、20个到30个核苷酸、20个到29个核苷酸、20个到28个核苷酸、20个到27个核苷酸、20个到26个核苷酸、20个到25个核苷酸、20个到24个核苷酸、20个到23个核苷酸、20个到22个核苷酸、21个到30个核苷酸、21个到29个核苷酸、21个到28个核苷酸、21个到27个核苷酸、21个到26个核苷酸、21个到25个核苷酸、21个到24个核苷酸、21个到23个核苷酸、22个到30个核苷酸、22个到29个核苷酸、22个到28个核苷酸、22个到27个核苷酸、22个到26个核苷酸、22个到25个核苷酸、22个到24个核苷酸、23个到30个核苷酸、23个到29个核苷酸、23个到28个核苷酸、23个到27个核苷酸、23个到26个核苷酸、23个到25个核苷酸、24个到30个核苷酸、24个到29个核苷酸、24个到28个核苷酸、24个到27个核苷酸、24个到26个核苷酸、25个到30个核苷酸、25个到29核苷酸、25个到28个核苷酸、25个到27个核苷酸、26个到30个核苷酸、26个到29个核苷酸、26个到28个核苷酸、27个到30个核苷酸、27个到29个核苷酸或28个到30个核苷酸。
[0157]
人工mirna是指能够产生功能性成熟mirna的内源性、经修饰的或合成pri-mrna或前mrna支架或骨架,其中茎区内mirna双链体的向导链序列和过客链序列已被替代为所关注的向导链序列和过客链序列,所述所关注的向导链序列和过客链序列指导所关注的靶mrna的沉默。以下描述了人工mirna设计:eamens等人(2014)《分子生物学方法》1062:211-24(通过全文引用的方式并入)。合成mirna骨架在美国专利公开2008/0313773(通过全文引用的方式并入)中进行了描述。在一些实施方式中,所述人工mirna的长度为约100个到200个核苷酸、100个到175个核苷酸、100个到150个核苷酸、125个到200个核苷酸、125个到175个核苷酸或125个到150个核苷酸。在一些实施方式中,所述人工mirna的长度为约100个核苷酸、约120个核苷酸、约130个核苷酸、约140个核苷酸、约150个核苷酸、约160个核苷酸、约170个核苷酸、约180个核苷酸、约190个核苷酸或约200个核苷酸。
[0158]
在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括与表1、3、9、11、12、13、19、23、24和25的向导序列中的任一个,例如以下中的任一个中所示的核酸序列或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、
188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、1176-1288、1811-1827、2015、2065、2083、2152、2203和2209。
[0159]
在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括表1、3、9、11、12、13、19、23、24和25的向导序列中的任一个,例如以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、1176-1288、1811-1827、2015、2065、2083、2152、2203和2209。
[0160]
在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括与表1、3、9、11、12、13、19、23、24和25的向导序列中的任一个,例如以下中的任一个具有至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%同一性的序列或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、1176-1288、1811-1827、2015、2065、2083、2152、2203和2209。
[0161]
在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工
mirna或成熟mirna,所述向导链序列包括表1、3、9、11、12、13、19、23、24和25的向导序列中的任一个,例如以下中的任一个的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、1176-1288、1811-1827、2015、2065、2083、2152、2203和2209,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。
[0162]
在一些实施方式中,所述mirna是pri-mirna、mrna前体、人工mirna或成熟mirna,所述mirna包括向导链序列,所述向导链序列包括表1、3、9、11、12、13、19、23、24和25的任一向导序列的序列或由其组成,例如,以下中的任一个:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、1176-1288、1811-1827、2015、2065、2083、2152、2203和2209,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0163]
在一些实施方式中,所述mirna是包括表12的向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、
308、330、336和362。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括与seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0164]
在一些实施方式中,所述mirna是包括表13的向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:14、40、100、108、112、128、166、198、242、308、336和362。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:14、40、100、108、112、128、166、198、242、308、336和362。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括与seq id no:14、40、100、108、112、128、166、198、242、308、336和362中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括seq id no:14、40、100、108、112、128、166、198、242、308、336和362中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括seq id no:14、40、100、108、112、128、166、198、242、308、336和362中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0165]
在一些实施方式中,所述mirna是包括表19的向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:1176-1288、40、108和166。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸
序列或由其组成:seq id no:1176-1288、40、108和166。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括与seq id no:1176-1288、40、108和166中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括seq id no:1176-1288、40、108和166中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括seq id no:1176-1288、40、108和166中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0166]
在一些实施方式中,所述mirna是包括表23的向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:1908-2007。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:1908-2007。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括与seq id no:1908-2007中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括seq id no:1908-2007中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括seq id no:1908-2007中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0167]
在一些实施方式中,所述mirna是包括表24的向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209。在一些实施方式中,所述mirna是包括向导链序列的
pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括与seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0168]
在一些实施方式中,所述mirna是包括表25的向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括与seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0169]
在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:1185、1816、1213和1811。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组
成:seq id no:1185、1816、1213和1811。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括与seq id no:1185、1816、1213和1811中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括seq id no:1185、1816、1213和1811中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述mirna是包括向导链序列的pri-mirna、mrna前体、人工mirna或成熟mirna,所述向导链序列包括seq id no:1185、1816、1213和1811中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0170]
在一些实施方式中,所述人工mirna包括根据本文描述的任何实施方式所述的向导链序列,所述向导链序列包含在mir骨架序列内。在一些实施方式中,所述人工mirna的向导链序列和过客链序列包含在mirna骨架序列内。在一些实施方式中,所述mirna骨架序列是mir-155骨架序列、mir-155e骨架序列、mir-155m骨架序列、mir1-1骨架序列、mir-1-1_m骨架序列、mir-100骨架序列、mir-100_m骨架序列、mir-190a骨架序列、mir-124骨架序列、mir-124_m骨架序列、mir-16-2骨架序列、mir-132骨架序列、mir-9骨架序列、mir-138-2骨架序列、mir-122骨架序列、mir-122_m骨架序列、mir-130a骨架序列、mir-128骨架序列、mir-144骨架序列、mir-451a骨架序列或mir-223骨架序列。
[0171]
在一些实施方式中,所述mirna骨架序列是mir-155e骨架序列、mir-155m骨架序列、mir1-1骨架序列、mir-1-1_m骨架序列、mir-100骨架序列、mir-100_m骨架序列、mir-190a骨架序列、mir-190a_m骨架序列、mir-124骨架序列、mir-124_m骨架序列、mir-132骨架序列、mir-138-2骨架序列、mir-122骨架序列、mir-122_m骨架序列、mir-130a骨架序列、mir-16-2骨架序列、mir-128骨架序列、mir-144骨架序列、mir-451a骨架序列或mir-223骨架序列。
[0172]
在一些实施方式中,所述mirna骨架序列是mir1-1骨架序列、mir-1-1_m骨架序列、mir-100骨架序列、mir-100_m骨架序列、mir-122骨架序列、mir-122_m骨架序列、mir-124骨架序列、mir-130a骨架序列、mir-132骨架序列、mir-138-2骨架序列、mir-144骨架序列、mir-155e骨架序列、mir-155m骨架序列、mir-190a_m骨架序列或mir-190a_m骨架序列。
[0173]
在一些实施方式中,所述mirna骨架序列是mir-100骨架序列或mir-100_m骨架序列。
[0174]
表2提供了表示以下中的区段的dna序列的实例:mir-1-1骨架、mir-100骨架、mir-122骨架、mir-124骨架、mir-128骨架、mir-130a骨架、mir-155e骨架、mir-155-m骨架和mir-138-2骨架。表21提供了表示以下中的区段的dna序列的实例:mir-1-1骨架、mir-1-1_m骨架、mir-100骨架、mir-100_m、mir-122骨架、mir-122_m骨架、mir-124骨架、mir-124_m骨架、mir-128骨架、mir-130a骨架、mir-155e骨架、mir-155m骨架、mir-138-2骨架、mir-144骨架、mir-190a骨架、mir-190a_m骨架、mir-132骨架、mir-451a骨架、mir-223骨架和mir-16-2骨架。应当理解,表2和21中的mir骨架区段的rna序列可以通过将表2和21的序列中的“t”核苷酸转化为“u”核苷酸来获得。人工mirna可以被设计为将本公开的期望的向导序列和过客序
列插入如表2或21中定义的mirna骨架中,并且任选地其中过客序列是根据表8中的规则设计的。例如,可以根据表21设计具有为dna形式的mir-100骨架的人工mirna(例如,用于插入转移质粒),所述人工mirna从5'到3'包括:seq id no:1529的5'mir上下文(侧接)序列;seq id no:1530的5'基底茎序列;期望的向导序列;seq id no:1531的环序列;根据表8中的规则设计的期望的过客序列;seq id no:1532的3'基底茎序列;以及seq id no:1533的3'mir上下文(侧接)序列。
[0175]
表2:mir骨架序列的注释
[0176][0177]
[0178]
在一些实施方式中,所述mir-155骨架序列、mir1-1骨架序列、mir-100骨架序列、mir-190a骨架序列、mir-124骨架序列、mir-16-2骨架序列、mir-132骨架序列、mir-9骨架序列、mir-138-2骨架序列、mir-122骨架序列、mir-130a骨架序列、mir-128骨架序列、mir-144骨架序列、mir-451a骨架序列或mir-223骨架序列的所述末端环、茎、5'侧接区段、3'侧接区段或其任何组合被修饰(例如,具有核苷酸插入、缺失、取代、错配、摆动或其任何组合)。
[0179]
先前已经确定了能够有效加工pri-mirna骨架的序列基序。这些包含所述mirna前体5'端处的ug基序、所述茎中错配的ghg基序和3'cnnc基序。在一些实施方式中,所述mir骨架序列已被修饰以并入这些基序,包含例如mir-155e骨架序列、mir-1-1_m骨架、mir-100_m骨架序列、mir-124_m骨架序列和mir-122_m骨架序列。此类经修饰的mir骨架在本文中用后缀“_m”标记。
[0180]
在一些实施方式中,所述mirna(pri-mirna、mrna前体、人工mirna或成熟mirna)包括表1、19、23和24中所示的双链体序列中的任一个的向导链序列和对应的过客链序列或由其组成。在一些实施方式中,所述mirna的过客链序列包括与向导链序列100%互补或完美互补的序列。例如,向导链序列可以包括以下的序列或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434或436(表1中的向导序列),并且过客链序列可以分别包括以下的序列或由其组成:seq id no:3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、57、59、61、63、65、67、69、71、73、75、77、79、81、83、85、87、89、91、93、95、97、99、101、103、105、107、109、111、113、115、117、119、121、123、125、127、129、131、133、135、137、139、141、143、145、147、149、151、153、155、157、159、161、163、165、167、169、171、173、175、177、179、181、183、185、187、189、191、193、195、197、199、201、203、205、207、209、211、213、215、217、219、221、223、225、227、229、231、233、235、237、239、241、243、245、247、249、251、253、255、257、259、261、263、265、267、269、271、273、275、277、279、281、283、285、287、289、291、293、295、297、299、301、303、305、307、309、311、313、315、317、319、321、323、325、327、329、331、333、335、337、339、341、343、345、347、349、351、353、355、357、359、361、363、365、367、369、371、373、375、377、379、381、383、385、387、389、391、393、395、397、399、401、403、405、407、409、411、413、415、417、419、421、423、425、427、429、431、433或435(表1中的过客序列)。在一些实施方式中,所述mirna的过客链序列不与向导链序列100%互补。例如,向导链序列可以包括seq id no:1176的序列或由其组成,并且对应的过客链序列可以包括seq id no:1289的序列或由其组成(参见表19)。
[0181]
在一些实施方式中,所述mirna(pri-mirna、mrna前体、人工mirna或成熟mirna)包括:向导链序列,所述向导链序列包括以下中的任一个或由其组成:seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362;以及过客链序列,所述过客链序列包括与向导链序列100%互补或完美互补的序列。例如,向导链序列可以包括以下的序列或由其组成:seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336或362,并且过客链序列可以分别包括以下的序列或由其组成:seq id no:11、13、39、59、99、103、107、111、123、125、127、165、197、219、241、301、305、307、329、335或361。
[0182]
在一些实施方式中,所述mirna(pri-mirna、mrna前体、人工mirna或成熟mirna)包括向导链序列,所述向导链序列包括以下中的任一个或由其组成:seq id no:14、40、100、108、112、128、166、198、242、308、336和362,并且所述mirna的过客链序列包括与向导链100%互补或完美互补的序列或由其组成。例如,向导链序列可以包括以下的序列:seq id no:14、40、100、108、112、128、166、198、242、308、336或362,并且过客链序列可以分别包括以下的序列:seq id no:13、39、99、107、111、127、165、197、241、307、335或361。
[0183]
在一些实施方式中,所述mirna(pri-mirna、mrna前体、人工mirna或成熟mirna)包括向导链序列,所述向导链序列包括表1、19、23和24的向导序列中的任一个或由其组成,并且过客链序列包括表1、19、23和24的对应过客序列或由其组成,所述对应过客序列相对于表1、19、23和24的过客链序列具有1个、2个、3个、4个、5个、6个、7个、8个、9个、10个或更多个插入、缺失、取代、错配、摆动或其任何组合。在一些实施方式中,所述mirna(pri-mirna、mrna前体、人工mirna或成熟mirna)包括:向导链序列,所述向导链序列包括以下中的任一个或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436;以及过客链序列,所述过客链序列分别包括以下的序列或由其组成:seq id no:3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、57、59、61、63、65、67、69、71、73、75、77、79、81、83、85、87、89、91、93、95、97、99、101、103、105、107、109、111、113、115、117、119、121、123、125、127、129、131、133、135、137、139、141、143、145、147、149、151、153、155、157、159、161、163、165、167、169、171、173、175、177、179、181、183、185、187、189、191、193、195、197、199、201、203、205、207、209、211、213、215、217、219、221、223、225、227、229、231、233、235、237、239、241、243、245、247、249、251、253、255、257、259、261、263、265、267、269、271、273、275、277、279、281、283、285、287、289、291、293、295、297、299、
301、303、305、307、309、311、313、315、317、319、321、323、325、327、329、331、333、335、337、339、341、343、345、347、349、351、353、355、357、359、361、363、365、367、369、371、373、375、377、379、381、383、385、387、389、391、393、395、397、399、401、403、405、407、409、411、413、415、417、419、421、423、425、427、429、431、433、435,其中分别相对于以下的过客链序列,所述过客链序列具有1个、2个、3个、4个、5个、6个、7个、8个、9个、10个或更多个插入、缺失、取代、错配、摆动或其任何组合:seq id no:3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、57、59、61、63、65、67、69、71、73、75、77、79、81、83、85、87、89、91、93、95、97、99、101、103、105、107、109、111、113、115、117、119、121、123、125、127、129、131、133、135、137、139、141、143、145、147、149、151、153、155、157、159、161、163、165、167、169、171、173、175、177、179、181、183、185、187、189、191、193、195、197、199、201、203、205、207、209、211、213、215、217、219、221、223、225、227、229、231、233、235、237、239、241、243、245、247、249、251、253、255、257、259、261、263、265、267、269、271、273、275、277、279、281、283、285、287、289、291、293、295、297、299、301、303、305、307、309、311、313、315、317、319、321、323、325、327、329、331、333、335、337、339、341、343、345、347、349、351、353、355、357、359、361、363、365、367、369、371、373、375、377、379、381、383、385、387、389、391、393、395、397、399、401、403、405、407、409、411、413、415、417、419、421、423、425、427、429、431、433、435。在一些实施方式中,错配是所述过客链序列中的g
→
c、c
→
g、a
→
t或t
→
a转化。在一些实施方式中,错配(以与向导链产生凸起)是所述过客链序列中的g
→
t、c
→
a、a
→
c或t
→
g转化。在一些实施方式中,摆动是g-u摆动,其中在所述过客链序列中c转化为t。在一些实施方式中,根据表8的规则修饰所述过客链序列。
[0184]
在一些实施方式中,所述mirna(pri-mirna、mrna前体、人工mirna或成熟mirna)包括:向导链序列,所述向导链序列包括以下中的任一个或由其组成:seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362;以及过客链序列,所述过客链序列包括相对于分别包括seq id no:11、13、39、59、99、103、107、11、123、125、127、165、197、219、241、301、305、307、329、335和361的序列或由其组成的过客链序列具有1个、2个、3个、4个、5个、6个、7个、8个、9个、10个或更多个插入、缺失、取代、错配、摆动或其任何组合的序列或由其组成。在一些实施方式中,错配是所述过客链序列中的g
→
c、c
→
g、a
→
t或t
→
a转化。在一些实施方式中,错配(以与向导链产生凸起)是所述过客链序列中的g
→
t、c
→
a、a
→
c或t
→
g转化。在一些实施方式中,摆动是g-u摆动,其中在所述过客链序列中c转化为t。在一些实施方式中,根据表8的规则修饰所述过客链序列。
[0185]
在一些实施方式中,所述mirna(pri-mirna、mrna前体、人工mirna或成熟mirna)包括:向导链序列,所述向导链序列包括以下中的任一个或由其组成:seq id no:14、40、100、108、112、128、166、198、242、308、336和362;以及过客链序列,所述过客链序列包括相对于分别包括seq id no:13、39、99、107、111、127、165、197、241、307、335或361的序列或由其组成的过客链序列具有1个、2个、3个、4个、5个、6个、7个、8个、9个、10个或更多个插入、缺失、取代、错配、摆动或其任何组合的序列或由其组成。在一些实施方式中,错配是所述过客链序列中的g
→
c、c
→
g、a
→
t或t
→
a转化。在一些实施方式中,错配(以与向导链产生凸起)是所述过客链序列中的g
→
t、c
→
a、a
→
c或t
→
g转化。在一些实施方式中,摆动是g-u摆动,其中在所述过客链序列中c转化为t。在一些实施方式中,根据表8的规则修饰所述过客链序
列。
[0186]
在一些实施方式中,所述mirna是人工mirna,所述人工mirna包括根据本文描述的任何实施方式所述的向导链序列,包含在mir-155骨架序列、mir1-1骨架序列、mir-100骨架序列、mir-124骨架序列、mir-138-2骨架序列、mir-122骨架序列、mir-128骨架序列、mir-130a骨架序列或mir-16-2骨架序列内,其中所述人工mirna包括根据表8修饰的过客链序列。在一些实施方式中,过客链序列包括错配,其中错配是所述过客链序列中的g
→
c、c
→
g、a
→
t或t
→
a转化;错配(以与向导链产生凸起)是所述过客链序列中的g
→
t、c
→
a、a
→
c或t
→
g转化;并且摆动是g-u摆动,其中在所述过客链序列中c转化为t。
[0187]
在一些实施方式中,人工mirna包括表3、9、11、19、23、24和25中的任一个表中所示的核酸序列或由其组成。在一些实施方式中,人工mirna包括以下中的任一个的核酸序列或由其组成:seq id no:443-490、1109-1111、1114、1121-1168、1405-1520、1908-2007、2011、2017、2021、2025、2027、2031、2035、2039、2041、2045、2049、2053、2057、2061、2067、2071、2075、2079、2085、2089、2093、2097、2101、2105、2109、2113、2117、2120、2124、2128、2132、2136、2140、2144、2148、2154、2158、2162、2166、2170、2174、2176、2180、2182、2184、2187、2189、2191、2193、2195、2197、2199、2205、2211、2261、2263、2265和2267。
[0188]
表3:atxn2特异性amirna
[0189]
[0190]
[0191]
[0192]
[0193]
[0194]
[0195]
[0196][0197]
在一些实施方式中,人工mirna包括和表3中所示的核酸序列或由其组成。在一些实施方式中,人工mirna包括seq id no:443-490中的任一个的核酸序列或由其组成。
[0198]
在一些实施方式中,人工mirna包括和表9中所示的核酸序列或由其组成。在一些实施方式中,人工mirna包括seq id no:1109-1111和1114中的任一个的核酸序列或由其组
成。
[0199]
在一些实施方式中,人工mirna包括和表11中所示的核酸序列或由其组成。在一些实施方式中,人工mirna包括seq id no:1121-1168中的任一个的核酸序列或由其组成。
[0200]
在一些实施方式中,人工mirna包括和表19中所示的核酸序列或由其组成。在一些实施方式中,人工mirna包括seq id no:1405-1520中的任一个的核酸序列或由其组成。
[0201]
在一些实施方式中,人工mirna包括和表23中所示的核酸序列或由其组成。在一些实施方式中,人工mirna包括seq id no:1908-2007中的任一个的核酸序列或由其组成。
[0202]
在一些实施方式中,人工mirna包括和表24中所示的核酸序列或由其组成。在一些实施方式中,人工mirna包括以下中的任一个的核酸序列或由其组成:seq id no:1908-1934、1936-1977、1979-1982、1984-1994、1997、1998、2000、2001、2005-2007、2011、2017、2021、2025、2027、2031、2035、2039、2041、2045、2049、2053、2057、2061、2067、2071、2075、2079、2085、2089、2093、2097、2101、2105、2109、2113、2117、2120、2124、2128、2132、2136、2140、2144、2148、2154、2158、2162、2166、2170、2174、2176、2180、2182、2184、2187、2189、2191、2193、2195、2197、2199、2205、2211、2261、2263、2265和2267。
[0203]
在一些实施方式中,人工mirna包括和表25中所示的核酸序列或由其组成。在一些实施方式中,人工mirna包括seq id no:1915、1982、1965、1937、1985、1921和2021的任一个的核酸序列或由其组成。
[0204]
表达构建体
[0205]
另一方面,本公开提供了一种分离的核酸,所述分离的核酸包括编码抑制如本文所描述的atxn2的表达或活性的抑制性核酸(例如,sirna、shrna、dsrna、mirna、amirna等)中的任一种的表达构建体或表达盒。
[0206]
在一些实施方式中,所述分离的核酸包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括表1、3、9、11、12、13、19、23、24和25的向导序列中的任一个向导序列,例如以下中所示的核酸序列或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、1176-1288、1811-1827、2015、2065、2083、2152、2203和2209。
[0207]
在一些实施方式中,所述分离的核酸包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括表1、3、9、11、12、13、19、23、24和25的向导序列中的任一个向导序列,例如以下中所示的与所述靶
atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、1176-1288、1811-1827、2015、2065、2083、2152、2203和2209。
[0208]
在一些实施方式中,所述分离的核酸包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括与表1、3、9、11、12、13、19、23、24和25的向导序列中的任一个向导序列,例如以下具有至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%同一性的核酸序列或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、1176-1288、1811-1827、2015、2065、2083、2152、2203和2209。
[0209]
在一些实施方式中,所述分离的核酸包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括表1、3、9、11、12、13、19、23、24和25的向导序列中的任一向导序列,例如以下的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、
264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、1176-1288、1811-1827、2015、2065、2083、2152、2203和2209,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。
[0210]
在一些实施方式中,所述分离的核酸包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括表1、3、9、11、12、13、19、23、24和25的向导序列中的任一个,例如以下的序列或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、1176-1288、1811-1827、2015、2065、2083、2152、2203和2209,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0211]
在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括表12的向导链序列。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362。在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362。在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括与seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括seq id no:12、14、40、60、
100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0212]
在一些实施方式中,所述分离的核酸包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括表13的向导链序列。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:14、40、100、108、112、128、166、198、242、308、336和362。在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的与靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:14、40、100、108、112、128、166、198、242、308、336和362。在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括与seq id no:14、40、100、108、112、128、166、198、242、308、336和362中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括seq id no:14、40、100、108、112、128、166、198、242、308、336和362中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括seq id no:14、40、100、108、112、128、166、198、242、308、336和362中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0213]
在一些实施方式中,所述分离的核酸包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括表19的向导链序列。在一些实施方式中,抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:1176-1288、40、108和166。在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:1176-1288、40、108和166。在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括与seq id no:1176-1288、40、108和166中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、
99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括seq id no:1176-1288、40、108和166中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括seq id no:1176-1288、40、108和166中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0214]
在一些实施方式中,所述分离的核酸包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括表23的向导链序列。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:1908-2007。在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:1908-2007。在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括与seq id no:1908-2007中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括seq id no:1908-2007中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括seq id no:1908-2007中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0215]
在一些实施方式中,所述分离的核酸包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括表24的向导链序列。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209。在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209。在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建
体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括与seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0216]
在一些实施方式中,所述分离的核酸包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括表25的向导链序列。在一些实施方式中,所述抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314。在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314。在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括与seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0217]
在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括以下
中的任一个中所示的核酸序列或由其组成:seq id no:1185、1816、1213和1811。在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:1185、1816、1213和1811。在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括与seq id no:1185、1816、1213和1811中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括seq id no:1185、1816、1213和1811中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述分离的核酸分子包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体,其中所述抑制性核酸包括向导链序列,所述向导链序列包括seq id no:1185、1816、1213和1811中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0218]
在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体靶向atxn2 mrna以通过mrna降解或翻译抑制干扰atxn2表达。在一些实施方式中,所述sirna双链体的所述向导链的长度为约18个核苷酸、19个核苷酸、20个核苷酸、21个核苷酸、22个核苷酸、23个核苷酸、24个核苷酸、25个核苷酸、26个核苷酸、27个核苷酸、28个核苷酸、29个核苷酸或30个核苷酸,或者长度为18-30、18-29、18-28、18-27、18-26、18-25、18-24、18-23、18-22、18-20、19-30、19-29、19-28、19-27、19-26、19-25、19-24、19-23、19-22、19-21、20-30、20-29、20-28、20-27、20-26、20-25、20-24、20-23、20-22、21-30、21-29、21-28、21-27、21-26、21-25、21-24、21-23、22-30、22-29、22-28、22-27、22-26、22-24、23-30、23-29、23-28、23-27、23-26、23-25、24-30、24-29、24-28、24-27、24-26、25-30、25-29、25-28、25-27、26-30、26-29、26-28、27-30、27-29、28-30个核苷酸。在一些实施方式中,所述sirna双链体的所述过客链的长度为约18个核苷酸、19个核苷酸、20个核苷酸、21个核苷酸、22个核苷酸、23个核苷酸、24个核苷酸、25个核苷酸、26个核苷酸、27个核苷酸、28个核苷酸、29个核苷酸或30个核苷酸,或者长度为18-30、18-29、18-28、18-27、18-26、18-25、18-24、18-23、18-22、18-20、19-30、19-29、19-28、19-27、19-26、19-25、19-24、19-23、19-22、19-21、20-30、20-29、20-28、20-27、20-26、20-25、20-24、20-23、20-22、21-30、21-29、21-28、21-27、21-26、21-25、21-24、21-23、22-30、22-29、22-28、22-27、22-26、22-24、23-30、23-29、23-28、23-27、23-26、23-25、24-30、24-29、24-28、24-27、24-26、25-30、25-29、25-28、25-27、26-30、26-29、26-28、27-30、27-29、28-30个核苷酸。在一些实施方式中,所述sirna双链体在每条链上含有2个或3个核苷酸3'突出端。在一些实施方式中,所述3'突出端与atxn2转录物互补。在一些实施方式中,所述sirna双链体的所述向导链和过客链彼此至少60%、65%、70%、75%、80%、85%、90%、95%、97%或100%互补,不包含突出端的任何核苷酸。
[0219]
在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括表1、3、9、11、12、13、19、23和24中的向导序列中的任一个向导序列,例如以下中的任一个中所示的核酸序列或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、1176-1288、1811-1827、2015、2065、2083、2152、2203和2209。
[0220]
在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括表1、3、9、11、12、13、19、23和24的向导序列中的任一个向导序列,例如以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、1176-1288、1811-1827、2015、2065、2083、2152、2203和2209。
[0221]
在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括与表1、3、9、11、12、13、19、23和24的向导序列中的任一向导序列,例如以下中的任一个具有至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%同一性的序列或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、
192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、1176-1288、1811-1827、2015、2065、2083、2152、2203和2209。
[0222]
在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括表1、3、9、11、12、13、19、23和24的向导序列中的任一个向导序列,例如以下中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、1176-1288、1811-1827、2015、2065、2083、2152、2203和2209,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。
[0223]
在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括表1、3、9、11、12、13、19、23和24的向导序列中的任一个向导序列,例如以下中的任一个或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、1176-1288、1811-1827、2015、2065、2083、2152、2203和2209,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体
核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0224]
在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括表12的向导链序列。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362。在一些实施方式中,所述sirna双链体包括向导链序列,所述向导链序列包括与seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0225]
在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括表13的向导链序列。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:14、40、100、108、112、128、166、198、242、308、336和362。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:14、40、100、108、112、128、166、198、242、308、336和362。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括与seq id no:14、40、100、108、112、128、166、198、242、308、336和362中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括seq id no:14、40、100、108、112、128、166、198、242、308、336和362中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,
所述sirna双链体包括向导链序列,所述向导链序列包括seq id no:14、40、100、108、112、128、166、198、242、308、336、362中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0226]
在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括表19的向导链序列。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:1176-1288、40、108和166。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:1176-1288、40、108和166。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括与seq id no:1176-1288、40、108和166中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括seq id no:1176-1288、40、108和166中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括seq id no:1176-1288、40、108和166中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0227]
在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括表23的向导链序列。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:1908-2007。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:1908-2007。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括与seq id no:1908-2007中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括seq id no:1908-2007中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括seq id no:1908-2007中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列
互补。
[0228]
在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括表24的向导链序列。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括与seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括向导链序列,所述向导链序列包括seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0229]
在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括表25的向导链序列。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括与seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链
体包括向导链序列,所述向导链序列包括seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列,所述向导链序列包括seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0230]
在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括表1、19、23和24中提供的sirna双链体中的任一个sirna双链体的向导链序列和过客链序列。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列和过客链序列,其包括以下中的任一个或由其组成:seq id no:12和11;seq id no:14和13;seq id no:40和39;seq id no:60和59;seq id no:100和99;seq id no:104和103;seq id no:108和107;seq id no:112和111;seq id no:124和123;seq id no:126和125;seq id no:128和127;seq id no:166和165;seq id no:198和197;seq id no:220和219;seq id no:242和241;seq id no:302和301;seq id no:306和305;seq id no:308和307;seq id no:330和320;seq id no:336和335;以及seq id no:362和361。在一些实施方式中,所述分离的核酸包括编码sirna双链体的表达构建体,所述sirna双链体包括向导链序列和过客链序列,其包括以下中的任一个或由其组成:seq id no:14和13;seq id no:40和39;seq id no:100和99;seq id no:108和107;seq id no:112和11;seq id no:128和127;seq id no:166和165;seq id no:198和197;seq id no:242和241;seq id no:308和307;seq id no:336和335;以及seq id no:362和361。
[0231]
在一些实施方式中,所述分离的核酸包括编码shrna的表达构建体,所述shrna包括本文提供的sirna双链体的向导链和过客链,所述向导链和过客链通过短间隔子序列,即环连接。在一些实施方式中,环序列的长度为4个、5个、6个、7个、8个、9个或10个核苷酸或长度为4个到10个、4个到9个、4个到8个、4个到7个、4个到6个、5个到10个、5个到9个、5个到8个、5个到7个、6个到9个、6个到8个、7个到10个、7个到9个或8个到10个核苷酸。
[0232]
在一些实施方式中,所述分离的核酸包括编码如pri-mirna、前mrna、人工mirna或成熟mirna等mirna的表达构建体,其中所述mirna包括向导链序列,所述向导链序列包括与表1、3、9、11、12、13、19、23、24和25的向导序列中的任一个向导序列,例如以下中的任一个中所示的核酸序列或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、
id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、1176-1288、1811-1827、2015、2065、2083、2152、2203和2209,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。
[0236]
在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,其中所述mirna包括向导链序列,所述向导链序列包括表1、3、9、11、12、13、19、23、24和25的向导序列中的任一个向导序列,例如以下中的任一个的核酸序列或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436、1176-1288、1811-1827、2015、2065、2083、2152、2203和2209,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0237]
在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,所述mirna包括表12的向导链序列。在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,所述mirna包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362。在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,其中所述mirna包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:12、14、40、60、100、104、
108、112、124、126、128、166、198、220、242、302、306、308、330、336和362。在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,其中所述mirna包括向导链序列,所述向导链序列包括与seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,其中所述mirna包括向导链序列,所述向导链序列包括seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,其中所述mirna包括向导链序列,所述向导链序列包括seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0238]
在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,所述mirna包括表13的向导链序列。在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,所述mirna包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:14、40、100、108、112、128、166、198、242、308、336和362。在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,其中所述mirna包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:14、40、100、108、112、128、166、198、242、308、336和362。在一些实施方式中,所述mirna是pri-mirna、mrna前体、人工mirna或成熟mirna,其中所述mirna包括向导链序列,所述向导链序列包括与seq id no:14、40、100、108、112、128、166、198、242、308、336和362中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,其中所述mirna包括向导链序列,所述向导链序列包括seq id no:14、40、100、108、112、128、166、198、242、308、336和362中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,其中所述mirna包括向导链序列,所述向导链序列包括seq id no:14、40、100、108、112、128、166、198、242、308、336和362中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0239]
在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna
或成熟mirna等mirna的表达构建体,所述mirna包括表19的向导链序列。在一些实施方式中,所述分离的核酸包括编码如pri-mirna、前mrna、人工mirna或成熟mirna等mirna的表达构建体,所述mirna包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:1176-1288、40、108和166。在一些实施方式中,所述分离的核酸包括编码如pri-mirna、前mrna、人工mirna或成熟mirna等mirna的表达构建体,其中所述mirna包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:1176-1288、40、108和166。在一些实施方式中,所述mirna是pri-mirna、mrna前体、人工mirna或成熟mirna,其中所述mirna包括向导链序列,所述向导链序列包括与seq id no:1176-1288、40、108和166中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,其中所述mirna包括向导链序列,所述向导链序列包括seq id no:1176-1288、40、108和166中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,其中所述mirna包括向导链序列,所述向导链序列包括seq id no:1176-1288、40、108和166中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0240]
在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,所述mirna包括表23的向导链序列。在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,所述mirna包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:1908-2007。在一些实施方式中,所述分离的核酸包括编码如pri-mirna、前mrna、人工mirna或成熟mirna等mirna的表达构建体,其中所述mirna包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:1908-2007。在一些实施方式中,所述mirna是pri-mirna、mrna前体、人工mirna或成熟mirna,其中所述mirna包括向导链序列,所述向导链序列包括与seq id no:1908-2007中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,其中所述mirna包括向导链序列,所述向导链序列包括seq id no:1908-2007中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,其中所述mirna包括向导链序列,所述向导链序列包括seq id no:1908-2007中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0241]
在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,所述mirna包括表24的向导链序列。在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,所述mirna包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209。在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,其中所述mirna包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209。在一些实施方式中,mirna是pri-mirna、mrna前体、人工mirna或成熟mirna,其中所述mirna包括向导链序列,所述向导链序列包括与seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,其中所述mirna包括向导链序列,所述向导链序列包括seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,其中所述mirna包括向导链序列,所述向导链序列包括seq id no:100、112、166、202、246、306、308、314、1180、1185、1196、1200、1211、1213、1215、1216、1224、1811-1822、1824-1827、2015、2065、2083、2152、2203和2209中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0242]
在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,所述mirna包括表25的向导链序列。在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,所述mirna包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314。在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,其中所述mirna包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314。在一些实施方式中,mirna是pri-mirna、mrna前体、人工mirna或成熟mirna,其中所述mirna包括向导链序列,所述向导链序列包括与seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314
中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,其中所述mirna包括向导链序列,所述向导链序列包括seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,其中所述mirna包括向导链序列,所述向导链序列包括seq id no:1185、1816、1213、1819、2083、1215、1216、1811和314中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0243]
在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,所述mirna包括向导链序列,所述向导链序列包括以下中的任一个中所示的核酸序列或由其组成:seq id no:1185、1816、1213和1811。在一些实施方式中,所述分离的核酸包括编码如pri-mirna、前mrna、人工mirna或成熟mirna等mirna的表达构建体,其中所述mirna包括向导链序列,所述向导链序列包括以下中的任一个中所示的与所述靶atxn2 mrna序列具有至少1个、2个、3个、4个或5个错配的核酸序列或由其组成:seq id no:1185、1816、1213和1811。在一些实施方式中,所述mirna是pri-mirna、mrna前体、人工mirna或成熟mirna,其中所述mirna包括向导链序列,所述向导链序列包括与seq id no:1185、1816、1213和1811中的任一个至少60%、70%、75%、80%、85%、90%、95%、97%、99%或100%相同的核酸序列或由其组成。在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrna前体、人工mirna或成熟mirna等mirna的表达构建体,其中所述mirna包括向导链序列,所述向导链序列包括seq id no:1185、1816、1213和1811中的任一个的序列的至少15个、16个、17个、18个、19个、20个、21个或22个连续核苷酸或由其组成,优选地其中所述向导链序列保留所选的seq id no的位置2-7(“种子序列”)。在一些实施方式中,所述分离的核酸包括编码如pri-mirna、mrnaq前体、人工mirna或成熟mirna等mirna的表达构建体,其中所述mirna包括向导链序列,所述向导链序列包括seq id no:1185、1816、1213和1811中的任一个的序列或由其组成,其中位置19-22处的1个、2个、3个或4个核苷酸与所选的seq id no不同(变体核苷酸),使得所述向导链序列在所述变体核苷酸处不再与所述atxn2靶序列互补。
[0244]
在一些实施方式中,所述分离的核酸包括编码如人工mirn的表达构建体,所述人工mirna包括根据本文描述的任何实施方式所述的向导链序列,所述向导链序列包含在mir骨架序列内。在一些实施方式中,所述人工mirna的所述向导链序列和过客链序列包含在mirna骨架序列内。在一些实施方式中,所述mirna骨架序列包含在mir-155骨架序列、mir-155e骨架序列、mir-155m骨架序列、mir1-1骨架序列、mir-1-1_m骨架序列、mir-100骨架序列、mir-100_m骨架序列、mir-190a骨架序列、mir-124骨架序列、mir-124_m骨架序列、mir-16-2骨架序列、mir-132骨架序列、mir-9骨架序列、mir-138-2骨架序列、mir-122骨架序列、mir-122_m骨架序列、mir-130a骨架序列、mir-128骨架序列、mir-144骨架序列、mir-451a骨架序列或mir-223骨架序列中。在一些实施方式中,所述mir-155骨架序列、mir1-1骨架序
列、mir-100骨架序列、mir-190a骨架序列、mir-124骨架序列、mir-16-2骨架序列、mir-132骨架序列、mir-9骨架序列、mir-138-2骨架序列、mir-122骨架序列、mir-130a骨架序列、mir-128骨架序列、mir-144骨架序列、mir-451a骨架序列或mir-223骨架序列的所述末端环、茎、5'侧接区段、3'侧接区段或其任何组合被修饰(例如,核苷酸插入、缺失、取代、错配、摆动或其任何组合)。
[0245]
在一些实施方式中,所述分离的核酸包括编码mirna(pri-mirna、mrna前体、人工mirna或成熟mirna)的表达构建体,所述mirna包括向导链序列和对应的过客链序列,所述过客链序列包括表1、19、23和24中所示的双链体序列中的任一双链体序列或由其组成。在一些实施方式中,所述mirna的所述过客链序列包括与所述向导链序列100%互补或完美互补的序列。例如,编码的向导链序列可以包括以下的序列或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434或436(表1中的向导序列),并且编码的过客链序列可以分别包括以下的序列或由其组成:seq id no:3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、57、59、61、63、65、67、69、71、73、75、77、79、81、83、85、87、89、91、93、95、97、99、101、103、105、107、109、111、113、115、117、119、121、123、125、127、129、131、133、135、137、139、141、143、145、147、149、151、153、155、157、159、161、163、165、167、169、171、173、175、177、179、181、183、185、187、189、191、193、195、197、199、201、203、205、207、209、211、213、215、217、219、221、223、225、227、229、231、233、235、237、239、241、243、245、247、249、251、253、255、257、259、261、263、265、267、269、271、273、275、277、279、281、283、285、287、289、291、293、295、297、299、301、303、305、307、309、311、313、315、317、319、321、323、325、327、329、331、333、335、337、339、341、343、345、347、349、351、353、355、357、359、361、363、365、367、369、371、373、375、377、379、381、383、385、387、389、391、393、395、397、399、401、403、405、407、409、411、413、415、417、419、421、423、425、427、429、431、433或435(表1中的过客序列)。在一些实施方式中,所述mirna的所述过客链序列不与所述向导链序列100%互补。例如,向导链序列可以包括seq id no:1176的序列或由其组成,并且对应的过客链序列可以包括seq id no:1289的序列或由其组成(参见表19)。
[0246]
在一些实施方式中,所述分离的核酸包括编码mirna(pri-mirna、mrna前体、人工mirna或成熟mirna)的表达构建体,所述mirna包括:向导链序列,所述向导链序列包括以下中的任一个或由其组成:seq id no:12、14、40、60、100、104、108、112、124、126、128、166、
198、220、242、302、306、308、330、336和362;以及过客链序列,所述过客链序列包括与所述向导链序列100%互补或完美互补的序列。例如,编码的向导链序列可以包括以下的序列或由其组成:seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336或362,并且编码的过客链序列可以分别包括以下的序列或由其组成:seq id no:11、13、39、59、99、103、107、111、123、125、127、165、197、219、241、301、305、307、329、335或361。
[0247]
在一些实施方式中,所述分离的核酸包括编码mirna(pri-mirna、mrna前体、人工mirna或成熟mirna)的表达构建体,其中所述mirna包括:向导链序列,所述向导链序列包括以下中的任一个或由其组成:seq id no:14、40、100、108、112、128、166、198、242、308、336和362;以及过客链序列,所述过客链序列包括与所述向导链100%互补或完美互补的序列。例如,编码的向导链序列可以包括以下的序列或由其组成:seq id no:14、40、100、108、112、128、166、198、242、308、336或362,并且编码的过客链序列可以分别包括以下的序列或由其组成:seq id no:13、39、99、107、111、127、165、197、241、307、335或361。
[0248]
在一些实施方式中,所述分离的核酸包括编码mirna(pri-mirna、mrna前体、人工mirna或成熟mirna)的表达构建体,其中所述mirna括向导链序列,所述向导链序列包括表1、19、23和24的向导序列中的任一个或由其组成,并且过客链序列包括表1、19、23和24的对应过客序列或由其组成,所述对应过客序列相对于表1、19、23和24的过客链序列具有1个、2个、3个、4个、5个、6个、7个、8个、9个、10个或更多个插入、缺失、取代、错配、摆动或其任何组合。在一些实施方式中,所述分离的核酸包括编码mirna(pri-mirna、mrna前体、人工mirna或成熟mirna)的表达构建体,其中所述mirna包括向导链序列,所述向导链序列包括以下中的任一个或由其组成:seq id no:4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92、94、96、98、100、102、104、106、108、110、112、114、116、118、120、124、126、128、130、132、134、136、138、140、142、144、146、148、150、152、154、156、158、160、162、164、166、168、170、172、174、176、178、180、182、184、186、188、190、192、194、196、198、200、202、204、206、208、210、212、214、216、218、220、222、224、226、228、230、232、234、236、238、240、242、244、246、248、250、252、254、256、258、260、262、264、266、268、270、272、274、276、278、280、282、284、286、288、290、292、294、296、298、300、302、304、306、308、310、312、314、316、318、320、324、326、328、330、332、334、336、338、340、342、344、346、348、350、352、354、356、358、360、362、364、366、368、370、372、374、376、378、380、382、384、386、388、390、392、394、396、398、400、402、404、406、408、410、412、414、416、418、420、422、424、426、428、430、432、434、436,以及过客链序列,分别相对于以下对应的过客链序列,所述过客链序列具有1个、2个、3个、4个、5个、6个、7个、8个、9个、10个或更多个插入、缺失、取代、错配、摆动或其任何组合:seq id no:3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、57、59、61、63、65、67、69、71、73、75、77、79、81、83、85、87、89、91、93、95、97、99、101、103、105、107、109、111、113、115、117、119、121、123、125、127、129、131、133、135、137、139、141、143、145、147、149、151、153、155、157、159、161、163、165、167、169、171、173、175、177、179、181、183、185、187、189、191、193、195、197、199、201、203、205、207、209、211、213、215、217、219、221、223、225、227、229、231、233、235、237、239、241、243、245、247、249、
251、253、255、257、259、261、263、265、267、269、271、273、275、277、279、281、283、285、287、289、291、293、295、297、299、301、303、305、307、309、311、313、315、317、319、321、323、325、327、329、331、333、335、337、339、341、343、345、347、349、351、353、355、357、359、361、363、365、367、369、371、373、375、377、379、381、383、385、387、389、391、393、395、397、399、401、403、405、407、409、411、413、415、417、419、421、423、425、427、429、431、433、435。在一些实施方式中,错配是所述编码的过客链序列中的g
→
c、c
→
g、a
→
t或t
→
a转化。在一些实施方式中,错配(以与向导链产生凸起)是所述编码的过客链序列中的g
→
t、c
→
a、a
→
c或t
→
g转化。在一些实施方式中,摆动是g-u摆动,其中在所述编码的过客链序列中c转化为t。在一些实施方式中,根据表8的规则修饰所述过客链序列。
[0249]
在一些实施方式中,所述分离的核酸包括编码mirna(pri-mirna、mrna前体、人工mirna或成熟mirna)的表达构建体,其中所述mirna包括:向导链序列,所述向导链序列包括以下中的任一个或由其组成:seq id no:12、14、40、60、100、104、108、112、124、126、128、166、198、220、242、302、306、308、330、336和362;以及过客链序列,所述过客链序列包括相对于分别包括seq id no:11、13、39、59、99、103、107、11、123、125、127、165、197、219、241、301、305、307、329、335和361或由其组成的过客链序列具有1个、2个、3个、4个、5个、6个、7个、8个、9个、10个或更多个插入、缺失、取代、错配、摆动或其任何组合的序列或由其组成。在一些实施方式中,错配是所述过客链序列中的g
→
c、c
→
g、a
ꢀ→
t或t
→
a转化。在一些实施方式中,错配(以与向导链产生凸起)是所述过客链序列中的g
→
t、c
→
a、a
→
c或t
→
g转化。在一些实施方式中,摆动是g-u摆动,其中在所述过客链序列中c转化为t。在一些实施方式中,根据表8的规则修饰所述过客链序列。
[0250]
在一些实施方式中,所述分离的核酸包括编码mirna(pri-mirna、mrna前体、人工mirna或成熟mirna)的表达构建体,其中所述mirna包括:向导链序列,所述向导链序列包括以下中的任一个或由其组成:seq id no:14、40、100、108、112、128、166、198、242、308、336和362;以及过客链序列,所述过客链序列包括相对于分别包括seq id no:13、39、99、107、111、127、165、197、241、307、335和361或由其组成的过客链序列具有1个、2个、3个、4个、5个、6个、7个、8个、9个、10个或更多个插入、缺失、取代、错配、摆动或其任何组合的序列。在一些实施方式中,错配是所述编码的过客链序列中的g
→
c、c
→
g、a
→
t或t
→
a转化。在一些实施方式中,错配(以与向导链产生凸起)是所述编码的过客链序列中的g
→
t、c
→
a、a
→
c或t
→
g转化。在一些实施方式中,摆动是g-u摆动,其中在所述编码的过客链序列中c转化为t。在一些实施方式中,根据表8的规则修饰所述过客链序列。
[0251]
在一些实施方式中,所述分离的核酸包括编码人工mirna的表达构建体,所述人工mirna包括根据本文描述的任何实施方式所述的向导链序列,包含在mir-155m骨架序列、mir-155e骨架序列、mir1-1骨架序列、mir-100骨架序列、mir-124骨架序列、mir-138-2骨架序列、mir-122骨架序列、mir-128骨架序列、mir-130a骨架序列或mir-16-2骨架序列内,其中所述人工mirna包括根据表8修饰的过客链序列。在一些实施方式中,过客链序列包括错配,其中错配是所述过客链序列中的g
→
c、c
→
g、a
→
t或t
→
a转化;错配(以与向导链产生凸起)是所述过客链序列中的g
→
t、c
→
a、a
→
c或t
→
g转化;并且摆动是g-u摆动,其中所述在过客链序列中c转化为t。
[0252]
在一些实施方式中,所述分离的核酸包括编码人工mirna的表达构建体,所述人工
mirna包括表3、9、11、1923、24和25中的任一个中所示的核酸序列或由其组成。在一些实施方式中,所述分离的核酸包括编码人工mirna的表达构建体,所述人工mirna包括以下中的任一个或由其组成:seq id no:443-490、1109-1111、1114、1121-1168、1405-1520、1908-2007、2011、2017、2021、2025、2027、2031、2035、2039、2041、2045、2049、2053、2057、2061、2067、2071、2075、2079、2085、2089、2093、2097、2101、2105、2109、2113、2117、2120、2124、2128、2132、2136、2140、2144、2148、2154、2158、2162、2166、2170、2174、2176、2180、2182、2184、2187、2189、2191、2193、2195、2197、2199、2205、2211、2261、2263、2265和2267。
[0253]
在一些实施方式中,所述分离的核酸包括编码人工mirna的表达构建体,所述人工mirna包括表3中所示的核酸序列或由其组成。在一些实施方式中,所述分离的核酸包括编码人工mirna的表达构建体,所述人工mirna包括以下中的任一个的核酸序列或由其组成:seq id no:443-490。
[0254]
在一些实施方式中,所述分离的核酸包括编码人工mirna的表达构建体,所述人工mirna包括表9中所示的核酸序列或由其组成。在一些实施方式中,所述分离的核酸包括编码人工mirna的表达构建体,所述人工mirna包括以下中的任一个的核酸序列或由其组成:seq id no:1109-1111和1114。
[0255]
在一些实施方式中,所述分离的核酸包括编码人工mirna的表达构建体,所述人工mirna包括表11中所示的核酸序列或由其组成。在一些实施方式中,所述分离的核酸包括编码人工mirna的表达构建体,所述人工mirna包括以下中的任一个的核酸序列或由其组成:seq id no:1121-1168。
[0256]
在一些实施方式中,所述分离的核酸包括编码人工mirna的表达构建体,所述人工mirna包括表19中所示的核酸序列或由其组成。在一些实施方式中,所述分离的核酸包括编码人工mirna的表达构建体,所述人工mirna包括以下中的任一个的核酸序列或由其组成:seq id no:1405-1520。
[0257]
在一些实施方式中,所述分离的核酸包括编码人工mirna的表达构建体,所述人工mirna包括表23中所示的核酸序列或由其组成。在一些实施方式中,所述分离的核酸包括编码人工mirna的表达构建体,所述人工mirna包括以下中的任一个的核酸序列或由其组成:seq id no:1908-2007。
[0258]
在一些实施方式中,所述分离的核酸包括编码人工mirna的表达构建体,所述人工mirna包括表24中所示的核酸序列或由其组成。在一些实施方式中,所述分离的核酸包括编码人工mirna的表达构建体,所述人工mirna包括以下中的任一个的核酸序列或由其组成:seq id no:1908-1934、1936-1977、1979-1982、1984-1994、1997、1998、2000、2001、2005-2007、2011、2017、2021、2025、2027、2031、2035、2039、2041、2045、2049、2053、2057、2061、2067、2071、2075、2079、2085、2089、2093、2097、2101、2105、2109、2113、2117、2120、2124、2128、2132、2136、2140、2144、2148、2154、2158、2162、2166、2170、2174、2176、2180、2182、2184、2187、2189、2191、2193、2195、2197、2199、2205、2211、2261、2263、2265和2267。
[0259]
在一些实施方式中,所述分离的核酸包括编码人工mirna的表达构建体,所述人工mirna包括表25中所示的核酸序列或由其组成。在一些实施方式中,所述分离的核酸包括编码人工mirna的表达构建体,所述人工mirna包括以下中的任一个的核酸序列或由其组成:seq id no:1915、1982、1965、1937、1985、1921和2021。
[0260]
在一些实施方式中,编码靶向atxn2 mrna的所述抑制性核酸的表达构建体包括以dna形式公开的任何向导链序列或人工mirna序列或由其组成。例如,表9、11、23和24提供了dna形式的amirna序列,所述dna序列可以插入表达构建体中。可替代地,可以通过用“t”核苷酸取代每个“u”核苷酸来将本文提供的amirna序列转化为dna形式。
[0261]
在一些实施方式中,所述表达构建体编码两种或更多种靶向本文描述的atxn2 mrna转录物的抑制性核酸。在一些实施方式中,所述表达构建体编码靶向atxn2转录物的抑制性核酸和靶向除atxn2之外的第二靶转录物的抑制性核酸。在一些实施方式中,所述第二靶转录物是c9orf72。靶向c9orf72的抑制性核酸的实例在美国专利公开us2019/0316126中进行了描述(通过全文引用的方式并入)。在一些实施方式中,所述表达构建体编码靶向atxn2转录物并编码治疗多肽或蛋白质的抑制性核酸。
[0262]
在一些实施方式中,所述表达构建体是单顺反子。在一些实施方式中,所述表达构建体是多顺反子(例如,表达构建体编码两种或更多种肽或多肽)。在一些实施方式中,在表达构建体中编码第一基因产物(例如,靶向atxn2 mrna的抑制性核酸)的核酸序列和编码第二基因产物的核酸序列由内部核糖体进入位点(ires)、弗林蛋白酶切割位点或病毒2a肽分离。在一些实施方式中,病毒2a肽是猪肠病毒-1(porcine teschovirus-1,p2a)、明脉扁刺蛾病毒(thosea asigna virus,t2a)、马鼻炎a病毒(equine rhinitis a virus,erav)、口蹄疫病毒(foot-and-mouth disease virus,f2a)、家蚕胞质多角体病毒(b.mori cytoplasmic polyhedrosis virus,bmcpv 2a)、家蚕软化病病毒(b.mori flacherie virus,bmifv 2a)或其变体。
[0263]
在一些实施方式中,所述表达构建体进一步包括与转基因(例如,编码人工mirna的核酸)可操作地连接的一种或多种表达控制序列(调节序列)。“可操作地连接的”序列包含与转基因邻接或以反式或远离转基因起作用以控制其表达的表达控制序列。表达控制序列的实例包括转录起始序列、终止序列、启动子序列、增强子序列、阻遏子序列、剪接位点序列、聚腺苷酸化(polya)信号序列或其任何组合。
[0264]
在一些实施方式中,启动子是内源性启动子、合成启动子、组成型启动子、诱导型启动子、组织特异性启动子(例如,cns特异性)或细胞特异性启动子(神经元、胶质细胞或星形胶质细胞)。组成型启动子的实例包括劳氏肉瘤病毒(rous sarcoma virus,rsv)ltr启动子(任选地具有rsv增强子)、巨细胞病毒(cmv)启动子(任选地具有cmv增强子)、sv40启动子和二氢叶酸还原酶启动子。诱导型启动子的实例包括锌诱导型绵羊金属硫蛋白(mt)启动子、地塞米松(dexamethasone,dex)诱导型小鼠乳腺肿瘤病毒(mmtv)启动子、t7聚合酶启动子系统、蜕皮激素昆虫启动子、四环素(tetracycline)抑制系统、四环素诱导系统、ru486诱导系统和雷帕霉素(rapamycin)诱导系统。可以使用的启动子的其它实例包括例如鸡β-肌动蛋白启动子(cba启动子)、cag启动子、h1启动子、cd68启动子、jet启动子、突触蛋白启动子、rna pol ii启动子或rna pol iii启动子(例如,u6、h1等)。在一些实施方式中,所述启动子是组织特异性rna pol ii启动子。在一些实施方式中,所述组织特异性rna pol ii启动子源自表现出神经元特异性表达的基因。在一些实施方式中,所述神经元特异性启动子是突触蛋白1启动子或突触蛋白2启动子。
[0265]
在一些实施方式中,所述启动子是h1启动子,所述h1启动子包括seq id no:1522的核苷酸113-203中所示的序列或由其组成。在一些实施方式中,所述启动子是h1启动子,
所述h1启动子包括seq id no:1521的核苷酸1798-1888中所示的序列或由其组成。在一些实施方式中,所述启动子是h1启动子,所述h1启动子包括seq id no:2257-2260中的任一个的核苷酸113-343中所示的序列或由其组成。在一些实施方式中,所述启动子是h1启动子,所述h1启动子包括seq id no:2257-2260中的任一个的核苷酸244-343中所示的序列或由其组成。
[0266]
在一些实施方式中,编码本公开的抑制性核酸的序列位于表达构建体的非翻译区中。
[0267]
在一些实施方式中,编码本公开内容的抑制性核酸的序列位于表达构建体的内含子、5'非翻译区(5'utr)或3'非翻译区(3'utr)中。在一些实施方式中,编码本发明的抑制性核酸的序列位于启动子下游和表达基因上游的内含子中。
[0268]
在一些实施方式中,所述分离的核酸包括编码抑制性核酸的表达构建体,其侧接两个aav反向末端重复序列(itr)(例如,5'itr和3'itr)。在一些实施方式中,每个aav itr是全长itr(例如,长度约为145bp,并且含有功能性rep结合位点(rbs)和末端解链位点(trs))。在一些实施方式中,所述itr之一被截短(例如,缩短或非全长)。在一些实施方式中,截短的itr缺少功能性末端解链位点(trs)并用于产生自互补的aav载体(scaav载体)。在一些实施方式中,截短的itr是被称为aitr的aav2 itr的截短版本(d序列和trs缺失)。在一些实施方式中,所述itr选自以下aav血清型:aav1、aav2、aav3、aav4、aav5、aav6、aav7、aav8、aav9、aav9.47、aav9(hu14)、aav10、aav11、aav 12、aavrh8、aavrh10、aav-dj8、aav-dj、aav-php.a、aav-php.b、aavphp.b2、aavphp.b3、aavphp.n/php.b-dgt、aavphp.b-est、aavphp.b-ggt、aavphp.b-atp、aavphp.b-att-t、aavphp.b-dgt-t、aavphp.b-ggt-t、aavphp.b-sgs、aavphp.b-aqp、aavphp.b-qqp、aavphp.b-snp(3)、aavphp.b-snp、aavphp.b-qgt、aavphp.b-nqt、aavphp.b-egs、aavphp.b-sgn、aavphp.b-egt、aavphp.b-dst、aavphp.b-dst、aavphp.b-stp、aavphp.b-pqp、aavphp.b-sqp、aavphp.b-qlp、aavphp.b-tmp、aavphp.b-ttp、aavphp.s/g2a12、aavg2a 15/g2a3、aavg2b4、aavg2b5以及其变体。
[0269]
在一些实施方式中,包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体的分离的核酸分子包括seq id no:2257-2260中的任一个中所示的核苷酸序列。在一些实施方式中,包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体的分离的核酸分子包括seq id no:2257中所示的核苷酸序列。在一些实施方式中,包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体的分离的核酸分子包括seq id no:2258中所示的核苷酸序列。在一些实施方式中,包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体的分离的核酸分子包括seq id no:2259中所示的核苷酸序列。在一些实施方式中,包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体的分离的核酸分子包括seq id no:2260中所示的核苷酸序列。
[0270]
包括编码抑制atxn2的表达或活性的抑制性核酸的表达构建体的额外的分离的核酸分子可以使用seq id no:2257-2260中的任一个中所示的核苷酸序列,通过将本公开的期望的抑制性核酸序列(例如,人工mirna盒)取代到seq id no:2257-2260中的任一个的核苷酸位置344-481中来构建。
[0271]
载体和宿主细胞
[0272]
本文所描述的抑制性核酸分子(sirna、shrna、mirna)可以由载体编码。使用用于
表达本公开的抑制性核酸的载体,例如aav可以允许抑制性核酸在受试者中持续或受控地表达,而不是对受试者施用多次剂量的分离的抑制性核酸。本公开提供了包括分离的核酸的载体,所述分离的核酸包括编码本文所描述的抑制性核酸的表达构建体。载体可以是质粒、粘粒、噬菌粒、细菌人工染色体(bac)或病毒载体。病毒载体的实例包括疱疹病毒(hsv)载体、逆转录病毒载体、腺病毒载体、腺相关病毒(aav)载体、慢病毒载体、杆状病毒载体等。在一些实施方式中,逆转录病毒载体是小鼠干细胞病毒、鼠白血病病毒(例如,莫洛尼鼠白血病病毒(moloney murine leukemia virus)载体)、猫白血病病毒、猫肉瘤病毒或禽网状内皮增生症病毒载体。在一些实施方式中,慢病毒载体是hiv(人类免疫缺陷病毒,包含hiv 1型和hiv 2型、马传染性贫血病毒、猫免疫缺陷病毒(fiv)、牛免疫缺陷病毒(biv)和猿免疫缺陷病毒(siv))、马传染性贫血病毒或维斯纳-梅迪病毒载体(maedi-visna viral vector)。
[0273]
在一些实施方式中,所述载体是aav(aav)载体,如重组aav(raav)载体,所述重组aav载体通过重组方法产生。aav是单链、非包膜的dna病毒,其基因组编码用于复制(rep)的蛋白质和衣壳(cap),侧接两个itr,所述itr作为病毒基因组复制的起点。aav还含有包装序列,从而允许将病毒基因组包装到aav衣壳中。重组aav载体(raav)可以通过使用分子方法从aav的野生型基因组中移除全部或部分野生型基因组(例如,rep、cap),并且用如异源核酸序列(例如,编码抑制性核酸的核酸分子)等非天然核酸取代从aav的野生型基因组中获得。通常,对于aav,一个或两个反向末端重复(itr)序列保留在所述aav载体中。在一些实施方式中,所述raav载体包括编码本公开的抑制性核酸的表达构建体,所述抑制性核酸侧接两个顺式作用aav itr(5'itr和3'itr)。功能性itr序列对于aav病毒颗粒的挽救、复制和包装是必需的。因此,aav载体在本文中被定义为至少包含以顺式方式复制和包装病毒所需的那些序列(例如,功能性itr)。在一些实施方式中,每个aav itr是全长itr(例如,长度约为145bp,并且含有功能性rep结合位点(rbs)和末端解链位点(trs))。在一些实施方式中,所述itr中的一个或两个被修饰,例如,通过插入、缺失或取代,条件是所述itr提供功能性挽救、复制和包装。在一些实施方式中,经修饰的itr缺少功能性末端解链位点(trs)并用于产生自互补的aav载体(scaav载体)。在一些实施方式中,经修饰的itr是被称为aitr的aav2 itr的截短版本(d序列和trs缺失)。
[0274]
在一些实施方式中,所述aav载体包括5'itr,所述5'itr包括seq id no:2257-2260中的任一个的核苷酸1-106或由其组成。在一些实施方式中,所述aav载体包括3'itr,所述3'itr包括seq id no:2257-2260中的任一个的核苷酸2192-2358或由其组成。在一些实施方式中,所述aav载体包括:5'itr,所述5'itr包括seq id no:2257的核苷酸1-106或由其组成;以及3'itr,所述3'itr包括seq id no:2257的核苷酸2192-2358或由其组成;5'itr,所述5'itr包括seq id no:2258的核苷酸1-106或由其组成;以及3'itr,所述3'itr包括seq id no:2258的核苷酸2192-2358或由其组成;5'itr,所述5'itr包括seq id no:2259的核苷酸1-106或由其组成;以及3'itr,所述3'itr包括seq id no:2259的核苷酸2192-2358或由其组成;或5'itr,所述5'itr包括seq id no:2260的核苷酸1-106或由其组成;以及3'itr,所述3'itr包括seq id no:2260的核苷酸2192-2358或由其组成。
[0275]
在一些实施方式中,所述raav载体是哺乳动物血清型aav载体(例如,aav基因组和源自哺乳动物血清型aav的itr),包含灵长类动物血清型aav载体或人血清型aav载体。在一
些实施方式中,所述aav载体是嵌合aav载体。在一些实施方式中,所述itr选自以下aav血清型:aav1、aav2、aav3、aav4、aav5、aav6、aav7、aav8、aav9、aav9.47、aav9(hu14)、aav10、aav11、aav 12、aavrh8、aavrh10、aav-dj8、aav-dj、aav-php.a、aav-php.b、aavphp.b2、aavphp.b3、aavphp.n/php.b-dgt、aavphp.b-est、aavphp.b-ggt、aavphp.b-atp、aavphp.b-att-t、aavphp.b-dgt-t、aavphp.b-ggt-t、aavphp.b-sgs、aavphp.b-aqp、aavphp.b-qqp、aavphp.b-snp(3)、aavphp.b-snp、aavphp.b-qgt、aavphp.b-nqt、aavphp.b-egs、aavphp.b-sgn、aavphp.b-egt、aavphp.b-dst、aavphp.b-dst、aavphp.b-stp、aavphp.b-pqp、aavphp.b-sqp、aavphp.b-qlp、aavphp.b-tmp、aavphp.b-ttp、aavphp.s/g2a12、aavg2a 15/g2a3、aavg2b4、aavg2b5以及其变体。
[0276]
其它表达控制序列可以存在于与所述抑制性核酸可操作地连接的所述raav载体中,包括转录起始序列、终止序列、启动子序列、增强子序列、阻遏子序列、剪接位点序列、聚腺苷酸化(polya)信号序列中的一种或多种,或其任何组合。
[0277]
aav优先包装全长基因组,即与天然基因组大小大致相同且不太大或太小的基因组。然而,编码抑制性核酸序列的表达盒相当小。为了避免包装片段化的基因组,可以将填充序列与编码本公开的抑制性核酸的表达构建体连接,并且侧接5'itr和3'itr以扩展可包装基因组,从而产生在所述itr之间的长度接近正常的基因组。在一些实施方式中,包括填充序列和编码本公开的抑制性核酸序列的表达盒的raav载体在5'itr和3'itr之间具有约4.7kb的总长度。在一些实施方式中,所述raav载体是自互补的raav载体,所述自互补的raav载体包括填充序列和编码本公开的抑制性核酸序列的表达盒,并且在5'itr和3'itr之间具有约2.4kb的总长度。用于本公开的raav载体的示例性填充序列包括:包括seq id no:1522的核苷酸348-2228或由其组成的序列和包括seq id no:2257-2260中的任一个的核苷酸489-2185或由其组成的序列。
[0278]
raav载体可以具有一个或多个aav野生型基因全部或部分缺失。在一些实施方式中,所述raav载体是复制缺陷型的。在一些实施方式中,所述raav载体缺乏功能性rep蛋白和/或衣壳蛋白。在一些实施方式中,所述raav载体是自互补的aav(scaav)载体。
[0279]
在一些实施方式中,所述raav载体从5'itr到3'itr包括seq id no:2257-2260中的任一个中所示的核苷酸序列。在一些实施方式中,所述raav载体从5'itr到3'itr包括seq id no:2257中所示的核苷酸序列。在一些实施方式中,所述raav载体从5'itr到3'itr包括seq id no:2258中所示的核苷酸序列。在一些实施方式中,所述raav载体从5'itr到3'itr包括seq id no:2259中所示的核苷酸序列。在一些实施方式中,所述raav载体包括seq id no:2260中所示的核苷酸序列。
[0280]
本公开的重组aav载体可以被一种或多种aav衣壳蛋白包裹以形成raav颗粒。“raav颗粒”或“raav病毒粒子”是指感染性、复制缺陷型病毒,所述病毒包含aav蛋白壳,所述aav蛋白壳包裹包括所关注的转基因的raav载体,其两侧各自侧接5'aav itr和3'aav itr。raav颗粒在合适的宿主细胞中产生,所述宿主细胞具有指定raav载体的序列,其中引入了aav辅助功能(helper function)和附属功能(acessory function),以使宿主细胞能够编码包装raav载体(含有所关注的转基因序列)所需的aav多肽进入感染性raav颗粒用于随后的基因递送。
[0281]
使用宿主细胞培养将重组aav载体包装到aav衣壳蛋白中的方法是本领域已知的。
在一些实施方式中,包装raav载体所需的一种或多种组分(例如,rep序列、cap序列和/或附属功能)可以由已被工程化以含有一种或多种所需的组分的稳定宿主细胞提供(例如,通过载体)。aav包装所需组分的表达可能受宿主包装细胞中诱导型或组成型启动子的控制。aav辅助载体通常用于提供反式作用的aav rep和/或cap基因的瞬时表达,以补充aav复制所必需的缺失的aav功能。在一些实施方式中,aav辅助载体缺乏aav itr并且既不能复制也不能自我包装。aav辅助载体可以是质粒、噬菌体、转座子、粘粒、病毒或病毒粒子的形式。
[0282]
在一些实施方式中,raav颗粒可以使用三重转染方法产生(参见例如,美国专利第6,001,650号,通过全文引用的方式并入本文)。在此方法中,所述raav颗粒是通过用将被包装到raav颗粒的raav载体(包括转基因)、aav辅助载体和附属功能载体转染宿主细胞产生的。在一些实施方式中,所述aav辅助功能载体支持有效的aav载体产生,而不产生任何可检测的野生型aav病毒粒子(例如,含有功能性rep和cap基因的aav病毒粒子)。所述附属功能载体编码非aav源性病毒和/或细胞功能的核苷酸序列,aav依赖于所述非aav源性病毒和/或细胞功能进行复制(例如,“附属功能”)。所述附属功能包含aav复制所需的功能,包括但不限于涉及aav基因转录的激活、阶段特异性aav mrna剪接、aav dna复制、cap表达产物的合成以及aav衣壳组装的那些部分。基于病毒的附属辅助功能可以源自任何已知的辅助病毒,如腺病毒、疱疹病毒(除了单纯疱疹病毒1型)和牛痘病毒。在一些实施方式中,其中将所述aav辅助功能和附属功能克隆在单个载体上的双重转染方法用于生成raav颗粒。
[0283]
所述aav衣壳是确定所述raav颗粒的这些组织特异性的重要元件。因此,可以选择具有衣壳组织特异性的raav颗粒。在一些实施方式中,所述raav颗粒包括选自以下aav血清型的衣壳蛋白:aav1、aav2、aav3、aav4、aav5、aav6、aav7、aav8、aav9、aav9.47、aav9(hu14)、aav10、aav11、aav 12、aavrh8、aavrh10、aav-dj8、aav-dj、aav-php.a、aav-php.b、aavphp.b2、aavphp.b3、aavphp.n/php.b-dgt、aavphp.b-est、aavphp.b-ggt、aavphp.b-atp、aavphp.b-att-t、aavphp.b-dgt-t、aavphp.b-ggt-t、aavphp.b-sgs、aavphp.b-aqp、aavphp.b-qqp、aavphp.b-snp(3)、aavphp.b-snp、aavphp.b-qgt、aavphp.b-nqt、aavphp.b-egs、aavphp.b-sgn、aavphp.b-egt、aavphp.b-dst、aavphp.b-dst、aavphp.b-stp、aavphp.b-pqp、aavphp.b-sqp、aavphp.b-qlp、aavphp.b-tmp、aavphp.b-ttp、aavphp.s/g2a12、aavg2a 15/g2a3、aavg2b4、aavg2b5以及其变体。在一些实施方式中,所述aav衣壳选自能够穿过血脑屏障的血清型,例如,aav9、aavrh.10、aav-php-b或其变体。在一些实施方式中,所述aav衣壳是嵌合aav衣壳。在一些实施方式中,所述aav颗粒是假型化aav,具有来自不同aav血清型的衣壳和基因组。
[0284]
在一些实施方式中,所述raav颗粒能够转导cns的细胞。在一些实施方式中,所述raav颗粒能够转导cns的非神经元细胞或神经元细胞。在一些实施方式中,所述cns细胞是神经元、神经胶质细胞、星形胶质细胞或小胶质细胞。
[0285]
另一方面,本公开提供了用包括本文所描述的抑制性核酸或载体的raav转染的宿主细胞。在一些实施方式中,所述宿主细胞是原核细胞或真核细胞。在一些实施方式中,所述宿主细胞是哺乳动物细胞(例如,hek293t、cos细胞、hela细胞、kb细胞)、细菌细胞(大肠杆菌(e.coli))、酵母细胞、昆虫细胞(sf9、sf21、果蝇、蚊子)等。
[0286]
药物组合物
[0287]
在一些方面,本公开提供了药物组合物,所述药物组合物包含抑制性核酸、包括表
达构建体的分离的核酸或如本文所描述的载体以及药学上可接受的载体。如本文所用,术语“药学上可接受的”是指在正确医学判断的范围内适合于与细胞和/或组织接触使用而不会产生过多毒性、刺激、过敏反应或其它问题或并发症的与合理的益处/风险比相称的那些化合物、材料、组合物和/或剂型。
[0288]
如本文所使用的,术语“药学上可接受的载体”意指如液体或固体填充剂、稳定剂、分散剂、悬浮剂、稀释剂、赋形剂、增稠剂、溶剂或包封材料等药学上可接受的材料、组合物或载体,涉及在患者内或向患者,载运或输送在本发明内有用的化合物,使得所述化合物可以执行其预期功能。在与制剂的其它成分相容并且对接触的细胞或组织无害的意义上来讲,每种载体必须是“可接受的”。可以包含在本发明实践中使用的药物组合物中的额外的成分是本领域已知的并且描述于例如《雷明顿氏药物科学(remington's pharmaceutical sciences)》(genaro编辑,宾夕法尼亚州伊斯顿市麦克出版公司(mack publishing co.,easton,pa),1985)中,其通过引用并入本文。
[0289]
正如医学领域所熟知的,任何一位患者的剂量取决于许多因素,包含患者的体型、体重、体表面积、年龄、实现治疗效果所需的抑制性rna表达的表达水平、抑制性核酸的稳定性、正在治疗的具体疾病、疾病阶段、性别、施用时间和途径、一般健康状况以及同时施用的其它药物。在一些实施方式中,将本文所描述的raav颗粒以每名受试者约1
×
106vg(病毒基因组)至约1
×
10
16
vg或以下的量施用于受试者:约1
×
106、2
×
106、3
×
106、4
×
106、5
×
106、6
×
106、7
×
106、8
×
106、9
×
106、1
×
107、2
×
107、3
×
107、4
×
107、5
×
107、6
×
107、7
×
107、8
×
107、9
×
107、1
×
108、2
×
108、3
×
108、4
×
108、5
×
108、6
×
108、7
×
108、8
×
108、9
×
108、1
×
109、2
×
109、3
×
109、4
×
109、5
×
109、6
×
109、7
×
109、8
×
109、9
×
109、1
×
10
10
、2
×
10
10
、3
×
10
10
、4
×
10
10
、5
×
10
10
、6
×
10
10
、7
×
10
10
、8
×
10
10
、9
×
10
10
、1
×
10
11
、2
×
10
11
、2.1
×
10
11
、2.2
×
10
11
、2.3
×
10
11
、2.4
×
10
11
、2.5
×
10
11
、2.6
×
10
11
、2.7
×
10
11
、2.8
×
10
11
、2.9
×
10
11
、3
×
10
11
、4
×
10
11
、5
×
10
11
、6
×
10
11
、7
×
10
11
、7.1
×
10
11
、7.2
×
10
11
、7.3
×
10
11
、7.4
×
10
11
、7.5
×
10
11
、7.6
×
10
11
、7.7
×
10
11
、7.8
×
10
11
、7.9
×
10
11
、8
×
10
11
、9
×
10
11
、1
×
10
12
、1.1
×
10
12
、1.2
×
10
12
、1.3
×
10
12
、1.4
×
10
12
、1.5
×
10
12
、1.6
×
10
12
、1.7
×
10
12
、1.8
×
10
12
、1.9
×
10
12
、2
×
10
12
、3
×
10
12
、4
×
10
12
、4.1
×
10
12
、4.2
×
10
12
、4.3
×
10
12
、4.4
×
10
12
、4.5
×
10
12
、4.6
×
10
12
、4.7
×
10
12
、4.8
×
10
12
、4.9
×
10
12
、5
×
10
12
、6
×
10
12
、7
×
10
12
、8
×
10
12
、8.1
×
10
12
、8.2
×
10
12
、8.3
×
10
12
、8.4
×
10
12
、8.5
×
10
12
、8.6
×
10
12
、8.7
×
10
12
、8.8
×
10
12
、8.9
×
10
12
、9
×
10
12
、1
×
10
13
、2
×
10
13
、3
×
10
13
、4
×
10
13
、5
×
10
13
、6
×
10
13
、6.7
×
10
13
、7
×
10
13
、8
×
10
13
、9
×
10
13
、1
×
10
14
、2
×
10
14
、3
×
10
14
、4
×
10
14
、5
×
10
14
、6
×
10
14
、7
×
10
14
、8
×
10
14
、9
×
10
14
、1
×
10
15
、2
×
10
15
、3
×
10
15
、4
×
10
15
、5
×
10
15
、6
×
10
15
、7
×
10
15
、8
×
10
15
、9
×
10
15
或1
×
10
16
vg/受试者。在一些实施方式中,将本文所描述的raav颗粒以约1
×
106vg/kg至约1
×
10
16
vg/kg或以下的量施用于受试者:约1
×
106、2
×
106、3
×
106、4
×
106、5
×
106、6
×
106、7
×
106、8
×
106、9
×
106、1
×
107、2
×
107、3
×
107、4
×
107、5
×
107、6
×
107、7
×
107、8
×
107、9
×
107、1
×
108、2
×
108、3
×
108、4
×
108、5
×
108、6
×
108、7
×
108、8
×
108、9
×
108、1
×
109、2
×
109、3
×
109、4
×
109、5
×
109、6
×
109、7
×
109、8
×
109、9
×
109、1
×
10
10
、2
×
10
10
、3
×
10
10
、4
×
10
10
、5
×
10
10
、6
×
10
10
、7
×
10
10
、8
×
10
10
、9
×
10
10
、1
×
10
11
、2
×
10
11
、2.1
×
10
11
、2.2
×
10
11
、2.3
×
10
11
、2.4
×
10
11
、2.5
×
10
11
、2.6
×
10
11
、2.7
×
10
11
、2.8
×
10
11
、2.9
×
10
11
、3
×
10
11
、4
×
10
11
、5
×
10
11
、6
×
10
11
、7
×
10
11
、7.1
×
10
11
、7.2
×
10
11
、7.3
×
10
11
、7.4
×
10
11
、7.5
×
10
11
、7.6
×
10
11
、7.7
×
10
11
、7.8
×
10
11
、7.9
×
10
11
、8
×
10
11
、9
×
10
11
、1
×
10
12
、1.1
×
10
12
、1.2
×
10
12
、1.3
×
10
12
、1.4
×
10
12
、1.5
×
10
12
、1.6
×
10
12
、1.7
×
10
12
、1.8
×
10
12
、1.9
×
10
12
、2
×
10
12
、3
×
10
12
、4
×
10
12
、4.1
×
10
12
、4.2
×
10
12
、4.3
×
10
12
、4.4
×
10
12
、4.5
×
10
12
、4.6
×
10
12
、4.7
×
10
12
、4.8
×
10
12
、4.9
×
10
12
、5
×
10
12
、6
×
10
12
、7
×
10
12
、8
×
10
12
、8.1
×
10
12
、8.2
×
10
12
、8.3
×
10
12
、8.4
×
10
12
、8.5
×
10
12
、8.6
×
10
12
、8.7
×
10
12
、8.8
×
10
12
、8.9
×
10
12
、9
×
10
12
、1
×
10
13
、2
×
10
13
、3
×
10
13
、4
×
10
13
、5
×
10
13
、6
×
10
13
、6.7
×
10
13
、7
×
10
13
、8
×
10
13
、9
×
10
13
、1
×
10
14
、2
×
10
14
、3
×
10
14
、4
×
10
14
、5
×
10
14
、6
×
10
14
、7
×
10
14
、8
×
10
14
、9
×
10
14
、1
×
10
15
、2
×
10
15
、3
×
10
15
、4
×
10
15
、5
×
10
15
、6
×
10
15
、7
×
10
15
、8
×
10
15
、9
×
10
15
或1
×
10
16
vg/kg。
[0290]
如医学领域的技术人员所确定的,药物组合物可以以适于待治疗(或预防)的疾病或病状的方式施用。组合物的合适的剂量和合适的施用持续时间和频率将由如患者的健康状况、患者的体型(即,体重、质量或身体面积)、患者的疾病的类型和严重程度、活性成分的特定形式和施用方法等因素确定。通常,合适的剂量和治疗方案以足以提供治疗和/或预防益处(如本文所描述的,包含临床结果改善,如完全或部分缓解更加频繁、或无疾病和/或整体生存期更长或症状严重性减轻)的量提供一种或多种组合物。对于预防用途,剂量应足以预防、延缓与疾病或病症相关的疾病的发作或减轻其严重性。根据本文所描述的方法施用的组合物的预防益处可以通过进行临床前(包含体外和体内动物研究)和临床研究并通过适当的统计学、生物学和临床方法和技术分析从中获得的数据来确定,所有这些都可以由本领域技术人员容易地实践。
[0291]
组合物(例如,药物组合物)可以通过任何途径施用,包含肠内(例如,口服)、肠胃外、静脉内、肌内、动脉内、髓内、鞘内、软膜下、实质内、纹状体内、颅内、脑池内、脑内、脑室、眼内、脑室内、腰椎内、皮下、透皮、皮间、直肠、阴道内、腹膜内、局部(如通过粉末、软膏剂、霜剂和/或滴剂)、粘膜、鼻、颊、舌下;通过气管内滴注、支气管滴注和/或吸入施用;和/或作为口腔喷雾、鼻腔喷雾和/或气雾剂施用。最适当的施用途径通常将取决于各种因素,包括药剂的性质(例如,其在胃肠道的环境中的稳定性)和/或受试者的病状。在一些实施方式中,将组合物直接注射到受试者的cns中。在一些实施方式中,直接注射到cns中是脑内注射、脑实质内注射、鞘内注射、纹状体内注射、软膜下注射或其任何组合。在一些实施方式中,直接注射到cns中是直接注射到受试者的脑脊液(csf)中,任选地其中所述直接注射是脑池内注射、脑室内注射和/或腰椎内注射。
[0292]
在一些实施方式中,调配包括raav颗粒的药物组合物以减少raav颗粒的聚集,特别是在存在高raav颗粒浓度(例如,~10
13
vg/ml或更高)的情况下。用于减少raav颗粒的聚集的方法在本领域中是众所周知的,并且包括例如添加表面活性剂、ph调节、盐浓度调节等(参见例如,wright f r等人,《分子疗法(molecular therapy)》(2005)12:171-178,通过全文引用的方式并入本文)。
[0293]
试剂盒
[0294]
在一些实施方式中,本文提供的组合物可以组装成药物或研究试剂盒以促进其在治疗或研究用途中的使用。试剂盒可以包含一个或多个容器,所述容器包括:(a)如本文所描述的抑制性核酸、包括表达构建体的分离的核酸或载体;(b)使用说明书;以及任选地(c)用于将试剂盒组分(a)转导到宿主细胞中的试剂。在一些实施方式中,试剂盒组分(a)可以是适合特定用途和施用方式的药物制剂和剂量。例如,试剂盒组分(a)可以存在于单位剂量
或多剂量容器,如密封安瓿瓶或小瓶中。所述试剂盒的组分可能需要在使用前混合一种或多种组分,或者可以以预混合状态制备。所述试剂盒的组分可以是液体或固体形式,并且可能需要添加溶剂或进一步稀释。所述试剂盒的组分可以是无菌的。所述说明书可以是书面或电子形式,并且可以与试剂盒相关联(例如,书面插入、cd、dvd)或通过互联网或基于网络的通信提供。所述试剂盒可以在冷藏或冷冻温度下运输和储存。
[0295]
治疗方法
[0296]
另一方面,本公开提供了用于抑制细胞中atxn2的表达或活性的方法,所述方法包括向细胞施用本公开的组合物(例如,抑制性核酸、包括编码抑制性核酸的表达构建体的分离的核酸、载体、raav颗粒、药物组合物),从而抑制细胞中atxn2的表达或活性。在一些实施方式中,所述细胞是cns细胞。在一些实施方式中,所述细胞是cns的非神经元细胞或神经元细胞。在一些实施方式中,所述cns的非神经元细胞是神经胶质细胞、星形胶质细胞或小胶质细胞。在一些实施方式中,所述细胞在体外。在一些实施方式中,所述细胞来自具有一种或多种神经退行性疾病症状或疑似患有神经退行性疾病的受试者。在一些实施方式中,所述细胞表达具有至少22个、23个、24个、25个、26个、27个、28个、29个、30个、31个、32个、33个、34个或更多个cag三核苷酸(多聚谷氨酰胺)重复序列的atxn2。在一些实施方式中,细胞表达具有约22个或23个重复序列、24个到32个重复序列或33个到100个或更多个重复序列的atxn2。
[0297]
另一方面,本公开提供了用于抑制受试者的中枢神经系统中atxn2的表达或活性的方法,所述方法包括向所述受试者施用本公开的组合物(例如,抑制性核酸、包括编码抑制性核酸的表达构建体的分离的核酸、载体、raav颗粒、药物组合物),从而抑制所述受试者中atxn2的表达或活性。
[0298]
另一方面,本公开提供用于治疗患有或疑似患有神经退行性疾病的受试者的方法,所述方法包括向所述受试者施用本公开的组合物(例如,抑制性核酸、包括编码抑制性核酸的表达构建体的分离的核酸、载体、raav颗粒、药物组合物),从而治疗所述受试者。如本文所使用的,术语“治疗”是指预防或延缓神经退行性疾病(例如,als/ftd、阿尔茨海默氏病、帕金森氏病等)的发作;降低神经退行性疾病的严重程度;减少或预防神经退行性疾病症状特性的发展;防止神经退行性疾病症状特性或其任何组合的恶化。
[0299]
可以使用本公开的组合物在受试者中治疗的神经退行性疾病包含其中atxn2是病原体(例如sca2)的神经退行性疾病,以及其中atxn2不是病原体但修饰tdp-43病理聚集的神经退行性疾病。与tdp-43蛋白病相关的神经退行性疾病包括als、ftd、原发性侧索硬化症、进行性肌肉萎缩、边缘为主的年龄相关性tdp-43脑病、慢性创伤性脑病、路易体痴呆、皮质基底节变性、进行性核上性麻痹(psp)、关岛型痴呆帕金森氏症als复合征(g-pdc)、皮克氏病、海马硬化症、亨廷顿氏病、帕金森氏病和阿尔茨海默氏病。
[0300]
在一些实施方式中,所述受试者的特征在于具有具有至少22个、23个、24个、25个、26个、27个、28个、29个、30个、31个、32个、33个、34个或更多个cag三核苷酸(多聚谷氨酰胺)重复序列的atxn2等位基因。在一些实施方式中,所述受试者的特征在于具有具有约22个或23个重复序列、24个到32个重复序列或33个到100个或更多个重复序列的atxn2等位基因。
[0301]
在一些实施方式中,本公开的治疗方法减少、预防或减缓神经退行性疾病的一种或多种症状特性的发展或进展。神经退行性疾病的症状特性的实例包括运动功能障碍、认
知功能障碍、情绪/行为功能障碍或其任何组合。麻痹、颤抖、不稳、僵硬、抽搐、肌肉无力、肌肉痉挛、肌肉僵硬、肌肉萎缩、吞咽困难、呼吸困难、言语和语言困难(例如,口齿不清)、运动缓慢、行走困难、痴呆、抑郁、焦虑或其任何组合。
[0302]
在一些实施方式中,本公开的治疗方法包括作为单一疗法或与一种或多种用于治疗神经退行性疾病的另外的疗法组合施用。组合疗法可以意指在一种或多种另外的疗法同时、之前、之后向受试者施用本公开的组合物(例如,抑制性核酸、包括编码抑制性核酸的表达构建体的分离的核酸、载体、raav颗粒、药物组合物)。组合疗法的同时施用可以意指调配本公开的组合物(例如,抑制性核酸、包括编码抑制性核酸的表达构建体的分离的核酸、载体、raav颗粒、药物组合物)和另外的疗法用于以相同的剂型施用或以单独的剂型施用。
[0303]
在一些实施方式中,可以与本公开的抑制性核酸组合使用的一种或另外的疗法包括:靶向神经退行性疾病相关基因或转录物(例如,c9orf72)的抑制性核酸或反义寡核苷酸、靶向神经退行性疾病相关基因(例如,c9orf72)的基因编辑剂(例如,基于crispr、talen、zfn的系统),减少氧化应激的试剂,如自由基清除剂(例如,radicava(依达拉奉(edaravone))、溴麦角环肽(bromocriptine));抗谷氨酸剂(例如,利鲁唑(riluzole)、托吡酯(topiramate)、拉莫三嗪(lamotrigine)、右美沙芬(dextromethorphan)、加巴喷丁(gabapentin)和ampa受体拮抗剂(例如,他仑帕奈(talampanel));抗细胞凋亡剂(例如,米诺环素(minocycline)、苯丁酸钠和阿莫洛莫(arimoclomol));抗炎剂(例如,神经节苷脂、塞来昔布(celecoxib)、环孢霉素(cyclosporine)、尼美舒利(nimesulide)、硫唑嘌呤(azathioprine)、环磷酰胺(cyclophosphamide)、血浆置换(plasmapheresis)、醋酸格拉替雷(glatiramer acetate)和沙利度胺(thalidomide));β-内酰胺类抗生素(青霉素(penicillin)及其衍生物、头孢曲松(ceftriaxone)和头孢菌素(cephalosporin));多巴胺激动剂(普拉克索(pramipexole)、右旋普拉克索(dexpramipexole));以及神经营养因子(例如,igf-1、gdnf、bdnf、ctnf、vegf、colivelin、xaliproden、促甲状腺激素释放激素和adnf)。
[0304]
在一些实施方式中,本公开的抑制性核酸与靶向c9orf72的另外的疗法组合施用。在一些实施方式中,靶向c9orf72的额外的疗法包括靶向c9orf72转录物的抑制性核酸、c9orf72特异性反义寡核苷酸或c9orf72特异性基因编辑剂。c9orf72特异性疗法的实例在以下中进行了描述:美国专利第9,963,699号(反义寡核苷酸);pct公开第wo2019/032612号(反义寡核苷酸);美国专利第10,221,414号(反义寡核苷酸);美国专利第10,407,678号(反义寡核苷酸);美国专利第9,963,699号(反义寡核苷酸);美国专利公开us2019/0316126(抑制性核酸);美国专利公开第2019/0167815号(基因编辑);pct公开第wo2017/109757号(基因编辑),所述文献中的每个都通过全文引用的方式并入。
[0305]
在一些实施方式中,在本文所描述的任何方法中治疗的受试者是哺乳动物(例如,小鼠、大鼠),优选地为灵长类动物(例如,猴、黑猩猩)或人。
[0306]
在本文所描述的任何治疗方法中,可以通过鞘内、软膜下、实质内、纹状体内、颅内、脑池内、脑内、脑室、眼内、脑室内、腰椎内施用或其任何组合向受试者施用本公开的组合物(例如,抑制性核酸、包括编码抑制性核酸的表达构建体的分离的核酸、载体、raav颗粒、药物组合物)。
[0307]
在一些实施方式中,将本公开的组合物(例如,抑制性核酸、包括编码抑制性核酸
的表达构建体的分离的核酸、载体、raav颗粒、药物组合物)直接注射到受试者的cns中。在一些实施方式中,直接注射到cns中是脑内注射、脑实质内注射、鞘内注射、纹状体内注射、软膜下注射或其任何组合。在一些实施方式中,直接注射到cns中是直接注射到受试者的脑脊液(csf)中,任选地其中所述直接注射是脑池内注射、脑室内注射、腰椎内注射或其任何组合。
[0308]
在一些实施方式中,与未与抑制性核酸接触的细胞中atxn2的表达水平相比,本公开的方法将细胞中的atxn2表达或活性减少至少10%、至少15%、至少20%、至少25%、至少30%、至少35%、至少40%、至少45%、至少50%、至少60%、至少70%、至少80%、至少90%、至少95%或更多。在一些实施方式中,与未与抑制性核酸接触的细胞中atxn2的表达水平相比,本公开的方法将细胞中的atxn2表达或活性抑制10-20%、10-30%、10-40%、10-50%、10-60%、10-70%、10-80%、10-90%、10-95%、20-30%、20-40%、20-50%、20-60%、20-70%、20-80%、20-90%、20-95%、20-100%、30-40%、30-50%、30-60%、30-70%、30-80%、30-90%、30-95%、30-100%、40-50%、40-60%、40-70%、40-80%、40-90%、40-95%、40-100%、50-60%、50-70%、50-80%、50-90%、50-95%、50-100%、60-70%、60-80%、60-90%、60-95%、60-100%、70-80%、70-90%、70-95%、70-100%、80-90%、80-95%、80-100%、90-95%、90-100%。
[0309]
在一些实施方式中,与未治疗的受试者的cns中的atxn2的表达水平相比,本公开的方法将受试者的cns中的atxn2表达或活性降低至少10%、至少15%、至少20%、至少25%、至少30%、至少35%、至少40%、至少45%、至少50%、至少60%、至少70%、至少80%、至少90%、至少95%或更多。在一些实施方式中,与未治疗的受试者的cns中的atxn2的表达水平相比,本公开的方法将受试者的cns中的atxn2表达或活性降低10-20%、10-30%、10-40%、10-50%、10-60%、10-70%、10-80%、10-90%、10-95%、20-30%、20-40%、20-50%、20-60%、20-70%、20-80%、20-90%、20-95%、20-100%、30-40%、30-50%、30-60%、30-70%、30-80%、30-90%、30-95%、30-100%、40-50%、40-60%、40-70%、40-80%、40-90%、40-95%、40-100%、50-60%、50-70%、50-80%、50-90%、50-95%、50-100%、60-70%、60-80%、60-90%、60-95%、60-100%、70-80%、70-90%、70-95%、70-100%、80-90%、80-95%、80-100%、90-95%、90-100%。
[0310]
实施例
[0311]
实施例1:sirna序列的设计和测试以敲低人a
taxin-2
[0312]
许多标准用于选择和设计sirna序列以敲低atxn2。最初考虑的潜在sirna序列包含与enst00000377617.7(atxn2-201)互补的所有可能的22核苷酸rna。首先检查了编码人ataxin-2的人转录物。仅选择在所有五个atxn2转录物nm_002973.3(seq id no:2)、enst00000377617.7、enst00000550104.5()、enst00000608853.5()和enst00000616825.4()中发现的序列。
[0313]
然后通过与恒河猴和食蟹猴中的直系同源atxn2基因的交叉反应性过滤这组序列。如果需要,这允许在这些物种中测试序列,以在用于人的治疗之前确定含有这些抑制性核酸序列的基因疗法的活性和安全性。因此,所述序列也需要在恒河猴(猕猴(macaca mulatta))atxn2(ncbi参考序列:xm_015152804.1、xm_015152805.1、xm_015152806.1、xm_015152807.1、xm_015152809.1、xm_015152810.1、xm_015152811.1、xm_015152812.1、xm_
015152814.1、(集成id:)ensmmut00000062319和ensmmut00000074794)和食蟹猴(macaca fascicularis)atxn2(ncbi参考序列:xm_005572266.2、xm_005572267.2、xm_015431532.1、xm_015431533.1、xm_015431534.1、xm_015431535.1、xm_015431536.1、xm_015431537.1、xm_015431538.1、xm_015431539.1、xm_015431540.1、xm_015431541.1、xm_015431542.1、xm_015431543.1、xm_015431544.1、xm_015431546.1、xm_015431547.1、xm_015431548.1、xm_015431549.1、xm_015431550.1、ensmfat00000019903.1)中。还检查了恒河猴的atxn2转录物xm_015152813.1。观察到此转录物缺少外显子1和外显子2的组分(通过与人atxn2序列seq id no:2比较)。如上文针对恒河猴序列所描述的,以下macaca atxn2转录物被鉴定为在外显子1中缺少上游序列:xm_015431551.1和ensmfat00000019905.1。对于这些序列,将外显子1序列从人(seq id no:2)中添加回来,以免过滤掉所述序列。atxn2基因中编码多聚谷氨酰胺重复序列的核苷酸序列含有可能在基因组其它地方的其它多聚谷氨酰胺重复序列中发现的元件。依赖于rnaseq数据比对的自动转录物比对算法可能会错误比对与基因组其它地方的多聚谷氨酰胺编码延伸(cag重复序列)重叠的测序读段,从而少计此序列。因此,不排除atxn2上游部分中的这些序列,除非是由于从人到灵长类动物序列的非保守性。
[0314]
基于对脑rnaseq的分析,外显子12跳跃约为3%频率,因此尽管一些可变剪接同种型不包含此同种型,但并未过滤掉。
[0315]
在定义预期存在于人atxn2和关键毒理学物种中的序列后,基于标准进一步选择sirna,以降低脱靶效应的可能性并提高强atxn2敲低的可能性。检查了sirna的反义链和有义链的种子序列,即已知是内源性微小rna活性的关键决定因素的序列的碱基2-7在人、小鼠和大鼠中表达的内源性mirna中的保守性。排除了存在于任何人内源性mirna中的反义序列,以及在小鼠和大鼠中都保守的所有序列。如果种子区域在人、小鼠和大鼠中超过2个物种中存在的内源性mirna中保守,则排除有义序列。
[0316]
通过调整pelossof等人(《自然生物技术》(2017)35:350-353)公开的算法版本来计算预测的敲低排名。本质上,在公开中提供的平铺测序数据上训练支持向量机。为了在其中支持向量机尝试分离标记为阳性和阴性的训练实施例的空间中生成点,分别针对好的和坏的敲低选择特征作为加权度核。输入到支持向量机分类器的特征与pelossof等人的基本相同。对于svm模型,使用了shogun模块(版本6.1.3,python版本2.7)中的“libsvm”函数,而不是“svmlite”。
[0317]
训练集包含来自基因pcna、trp53、hras、rpa3、mcl1、hmyc、myc、bcl2和kras的18,421个shrna序列,所有基因来自pelossof等人中包含的“tile”数据集。tile数据集经验性地测试了覆盖所描述的9个基因序列的无偏差shrna库的性能。成本函数c是在一系列值上评估的,除了被遗漏的九个基因中的一个外,在所有基因上训练svm分类器,并且计算对来自留出基因的数据的性能预测的均方误差。示出了以kras作为留出基因的实施例(图1)。选择了c=4的值,所述值使测试的c值之间的均方误差最小化。
[0318]
为了进一步评估分类器的性能,在其它8个基因上训练分类器后,留出了来自数据集中另一个基因(trp53)的敲低数据。图2示出了在不包含trp53 shrna的数据上进行训练的分类器的精度-召回率曲线,所述分类器预测shrna敲低在trp53靶向shrna中的性能。也就是说,在通过给定的分类器评分过滤shrna之后,由分类器鉴定的真阳性的分数(召回率)被绘制为随真阳性与假阳性的数量(精度)而进行的变化(图2)。另外,绘制了预期随着分类
器评分的严格性增加而丢失的“阳性”shrna(高性能)shrna的预期累积分数,(图3),以及低性能shrna的排斥的改善百分比。在大约-1.5到-0.8的评分之间注意到曲线中的分离,大约是从trp53靶向shrna的评分的第25个百分位到第50个百分位。
[0319]
接下来,通过特异性考虑对sirna序列进行分类,然后根据上述分类器的评分进行排序。如上文所描述的,除了种子序列相对于内源性mirna的保守性外,特异性指标是:(a)种子序列(向导碱基2-7)与已公开的转染的sirna种子序列与细胞增殖数据集的比较(gaoao等人《自然通讯(nature communications)》(2018)9:4504),不包括已公开的测定中细胞增殖减少》70%的序列;(b)以2个或更少的错配与向导序列的前19个核苷酸互补的转录物的数量要求少于15个;以及(c)其它考虑因素,如特异性的内部算法也被考虑在内,但分类的样品少于(a)和(b)的标准。
[0320]
在按特异性过滤后,最常见的atxn2转录物中的序列按svm评分排序,并且选择排名靠前的候选序列。然而,在计算shrna的svm分类器评分时,发现以u开头的shrna的分类器评分显著增加(图4)。这与先前的预测算法(例如,vert等人,《bmc生物信息学(bmc bioinformatics)》(2006)《bmc生物信息学》7:520)和文献表明argonaute 2结合口袋与此碱基相互作用最好一致,尽管向导碱基1不与靶mrna碱基配对(boland等人,《embo报告(embo reports)》(2010)11:522-527)。因此,对于shrna设计,如果碱基是“g”或“c”,则基于与靶mrna序列的互补性,所述碱基被替代为“u”并计算对应的性能评分。以a或u开头的前93个序列(svm评分》-0.8)和从以g或c开头的shrna编辑的34个序列,具有更严格的过滤器(svm评分》0.4)。
[0321]
基于其它标准,包含额外的序列用于测试,所述标准包含:(a)与atxn2l的交叉反应性。atxn2l与atxn2具有相当大的氨基酸序列相似性。同源基因通常在细胞中执行相似的功能,并且atxn2l的敲低可能与敲低atxn2具有相似的治疗功能。因此,与atxn2和atxn2l匹配的序列可能具有额外的治疗益处,并且因此,选择了10个可能靶向atxn2和atxn2l两者的序列;(b)满足严格的脱靶匹配标准的序列,在sirna向导序列的前19个核苷酸(10个sirna)中的2个或更少的位置中有2个或更少的转录物匹配,但忽略基于svm的功效预测;(c)在向导序列的前19个核苷酸中与小鼠atxn2完美匹配或单个错配的序列。“单个错配”向导序列被定义为在12nt与19nt碱基之间与小鼠序列仅发生一个错配,而在1-11碱基中没有错配。对于向导序列完美匹配或单个错配的小鼠,放宽了特异性标准,接受的向导序列具有少于50个互补转录物,所述互补转录物具有2个或更少的错配。
[0322]
选择细胞系以筛选sirna候选物
[0323]
在选择用于测试的sirna之后,建立了体外细胞系统来评估sirna对atxn2的敲低。atxn2水平通过quantigene测定(赛默飞世尔科技公司(thermo fisher))在一组细胞系中进行评估(图5)。细胞系hepg2、kb、ht-29、lncap、c4-2和panc-1均表现出稳健的atxn2表达。为了查看细胞的剪接模式是否与相关靶组织中的atxn2的相似,包含在神经退行性疾病状态下,检查了死后人脑的rnaseq(梅奥诊所阿尔茨海默氏病遗传学研究(mayo clinic alzheimer's disease genetics studies)**;通过synapse.org平台访问)的atxn2的剪接模式,并且与来自细胞系的数据进行比较(国家癌症研究所gdc遗产档案(national cancer institute gdc legacy archive))。在图6a中,可变剪接的外显子通过跨越跳过可变剪接的外显子的基因组区的读段来鉴定。脑中的外显子10、21和24经常可变剪接。检查细胞系中
的可变剪接,hepg2与人脑相似(图6b)。由于相对于背景的高水平atxn2表达和一致的可变剪接模式,此线被选择用于atxn2 sirna研究。
[0324]
关于synapse.org平台,研究数据由以下来源提供:梅奥诊所阿尔茨海默氏病遗传研究,由佛罗里达州杰克逊维尔(jacksonville,fl)的梅奥诊所的nil
ü
fer ertekin-taner博士和steven g.younkin博士领导,使用来自梅奥诊所衰老研究、梅奥诊所阿尔茨海默氏病研究中心和梅奥诊所脑库的样品。数据收集由nia拨款p50 ag016574、r01 ag032990、u01 ag046139、r01 ag018023、u01 ag006576、u01 ag006786、r01 ag025711、r01 ag017216、r01 ag003949、ninds拨款r01 ns080820、curepsp基金会(curepsp foundation)和梅奥基金会(mayo foundation)支持。以下出版物适用:
[0325]
[1]carrasquillo等人,《自然遗传学(nat genet.)》(2009)41:192-8。
[0326]
[2]zou等人《公共科学图书馆遗传学(plos genet.)》(2012)8(6):e1002707。
[0327]
[3]allen等人《科学数据(sci data)》.(2016)3:160089。
[0328]
sirna的合成和测试
[0329]
sirna被合成为22个核苷酸的rna,其具有20bp的互补性(向导链和过客链的位置1-20的互补性)。在此,向导链是指与atxn2靶mrna互补或反义的序列,而过客链是指与向导链互补的链。也称为反义链和有义链rna的向导链和过客链在表1中示出。序列被合成为向导链和过客链。除6个序列外,所有序列均符合以下标准:单链在计算的质量的.05%以内(通过lc/ms)。至少85%的全长寡核苷酸纯度(通过hplc)。向导链和过客链退火后,通过非变性hplc,双链体纯度》90%。不符合这些标准的寡核苷酸被标记为“fail”,但为了完整性,数据被包含在内。
[0330]
将退火的sirna反向转染,在96孔板的每孔中具有sirna的lipofectamine 2000溶液之上添加20,000个细胞,以在如下所述的稀释的培养基中产生最终sirna浓度,体积为每孔0.5微升转染溶液。在一式四份的孔中测试sirna,并且温育24小时。使用atxn2和gapdh探针(赛默飞世尔科技公司)通过quantigene测定来测定细胞裂解物中的atxn2和gapdh水平。计算atxn2 mrna水平与管家基因gapdh水平的比率,并将值相对于针对用不含sirna的lipofectamine处理的细胞模拟获得的atxn2/gapdh比率归一化。
[0331]
在20nm或1nm(细胞培养基中sirna的最终计算浓度)的剂量下测试所有sirna在敲低后的atxn2水平(表4)。绘制预测的svm评分分类器与20nm sirna敲低数据,通过线性模型拟合评估,观察到显著相关性(图8)(p《10-8
,r2=0.15)。随后,在200pm下通过atxn2敲低从1nm sirna给药数据重新筛选排名最高的100个sirna(表5)。图7绘制了sirna的atxn2 mrna的敲低随沿着其转录的atxn2转录物的位置而进行的变化。
[0332]
表4:sirna单点测试数据20nm
–
atxn2 mrna的敲低
[0333]
[0334]
[0335]
[0336]
[0337][0338]
表5:sirna单点测试数据200pm
[0339]
[0340]
no:537)。“smartpool”(smp)是4个靶向atxn2的sirna(dharmacon公司;on-targetplus human atxn2 sirna smartpool,l-011772-00-0005)的组合,用作atxn2的特异性靶向的阳性对照。ntc和smartpool sirna均经过化学修饰以限制脱靶效应。
[0345]
基于成像的测定使用针对atxn2的抗体的间接免疫荧光信号来量化atxn2水平。对于这些实验,选择u2os细胞是因为其细胞体大且均匀,可以很好地观察细胞质中的ataxin-2水平。通过瞬时转染引入sirna,并且3天后将细胞固定在多聚甲醛中,然后用ataxin-2进行封闭和免疫染色,并且用赫斯特染料33342复染色以鉴定细胞核。
[0346]
使用cell profiler中开发的定制渠道对图像进行分段。首先,基于赫斯特33342信号鉴定和勾勒出细胞核。随后,核轮廓被扩展以生成环。在此环内,对于每个细胞,对来自与和抗ataxin-2结合的荧光二级抗体相对应的间接免疫荧光通道的信号进行量化。为了计算孔的atxn2信号,计算跨细胞atxn2信号的孔中细胞的平均值(通常为1000个到3500个成像细胞/孔)。使用细胞质区内的上四分位数atxn2信号。通过取信号的上四分位数,这避免了来自可能无意中不含有细胞的图像的分段区的信号的影响。
[0347]
细胞在跨多个板的96孔形式中用20nm或1nm sirna给药,其中每个板中都有对照。在每个成像板中,通过用二级抗体而非一级抗体染色且未转染的孔减去背景。这反映了由于二级抗体的非特异性结合而导致的背景强度。将ataxin-2强度值相对于用非靶向对照(“ntc”)转染的孔中的强度值归一化。由此,归一化的atxn2信号表示蛋白质水平敲低程度的代表。重要的是,用靶向sirna的荧光素酶处理的孔的atxn2信号与用ntc对照处理的细胞相似。应注意,“ntc”对照(dharmacon公司)化学经修饰以减少脱靶效应,而所有测试的靶向atxn2和靶向荧光素酶的sirna均未修饰。图9量化了sirna在20nm和1nm剂量水平下的atxn2信号的敲低。图10a和10b示出了来自敲低实验的代表性图像,其证明了来自所指示sirna的ataxin-2强度明显降低。图11绘制了在20nm或1nm sirna下随atxn2转录物位置而变化的sirna蛋白敲低数据。几乎所有这些靠前sirna在蛋白质水平上都产生了大量的sirna敲低。在1nm下时,所有这些靠前sirna都超过了smartpool sirna的敲低性能。表6和7显示了atxn2信号在各孔中的平均值和标准偏差。来自表6和7的sirna的序列在表1中提供。对于平均值和sd计算,排除离群点(离群点定义为值与各孔中值的偏差超过1.5个标准偏差且大于10%的归一化atxn2信号的孔)。离群点孔在图9中突出显示。
[0348]
表6:在20nm下的sirna处理后通过高含量成像测量的atxn2蛋白敲低
[0349]
[0350][0351]
表7:在1nm下的sirna处理后通过高含量成像测量的atxn2蛋白敲低
[0352]
[0353][0354][0355]
值得注意的是,56个靶向atxn2的序列中的53个通过此测定实现了大于60%的
155序列的跨度由mir-155周围的区定义,其中在相似物种中具有高度的进化保守性。也就是说,序列保守性与位置的图被可视化,并且使用来自内源性mir-155的基因组位置(此序列保守性下降的位置)来确定应该包含多少mir-155茎结构周围的侧接上下文。接下来,检查了mir-155环的特征,所述特征可能会影响此mir在不同表达系统中的使用。注意到mir-155环中的同源四聚体uuuu。据报道,uuuu序列可诱导聚合酶iii终止
14
,这将导致不进行茎配对的异常截短的mirna。为了中断此同源四聚体uuuu,添加了mir-155环内的顶端ugu基序。据报道,此基序还可增强mirna加工
1,2
。除了先前工程化的ug和cnnc基序3外,还添加了基底茎错配的ghg基序2以改进精确加工。
[0366]
为了将amirna支架的数量扩展到mir-155骨架之外,优先考虑报道具有高加工精度的内源性mirna的骨架。根据参考文献
15
,mir-1-1骨架在加工精度上排名最高,通过小rnaseq5具有较高的链偏倚,并且向导链位于mirna茎的3个主要臂上,与5个主要臂定位的向导链
16
相比,这可以提高加工精度。天然整合的有利序列基序包含基底错配的ghg基序和下游cnnc基序。其还具有短的测序上下文,并已成功工程化用于果蝇模型
17
中的人工mirna表达。
[0367]
可以考虑用于本公开的amirna的其它mirna支架包括:
[0368]
·
mir-100和mir-190a
–
高通量筛选鉴定了高中靶/脱靶比率
15
。
[0369]
·
mir-124和mir-132
–
运动神经元表达的mirna在als大鼠模型
18
中不会改变表达。整个als病程中的细胞类型特异性表达和一致性水平是有利的mirna特性。神经元特异性已在srnaseq跨组织表达数据库
19
中得到证实(https://ccb-web.cs.uni-saarland.de/tissueatlas/)。
[0370]
·
mir-9
–
神经元特异性表达
20
。
[0371]
·
mir-138-2、mir-122、mir-130a和mir-128被选择为天然不对称的(在小rnaseq数据集中仅观察到5'或3'链)、高度均匀(即高“5'均匀性评分”15
),没有报道进行转录后调节(例如,发生在聚集的mirna上),是mirbase上的共有mirna,具有灵活的环结构和简单的双链体茎。
[0372]
为了进一步模拟mirna骨架,可以将凸起和错配插入到向导:过客链双链体中,其方式重复在内源性mirna中观察到的凸起模式,但适用于靶向atxn2的人工mirna。表8中提供了可以对过客链进行以引入这些天然mirna模拟结构的修饰。
[0373]
表8:示例性mirna形式的设计规则
[0374][0375]
注意:对于上述,“过客”序列是指与向导序列的22个核苷酸互补的序列。这与用于描述sirna双链体的过客序列不同。错配是指以下取代规则:g-》c、c-》g、a-》t、t-》a。凸起错配过渡是指规则:t-》c、c-》a、a-》c、g-》a。凸起错配颠换是指规则:g-》t、c-》a、a-》c、t-》g。添加gu摆动是指规则:如果碱基是c,则转化为t。
[0376]
mir155-m和mir1-1骨架中atxn2靶向向导序列的初步测试
[0377]
作为atxn2靶向sirna在嵌入mirna上下文中时敲低atxn2能力的初步测试,xd-14792(seq id no:112)的向导序列作为sirna在以200pm给药时具有排名最高的atxn2 mrna敲低,被嵌入若干个mirna上下文中,如表9中所示。amirna dna序列在表9中作为seq id no:538-543提供。对应的amirna rna序列分别在表9中作为seq id no:1109-1114提供。
[0378]
表9:嵌入amirna的xd-14792序列
[0379]
[0380][0381][0382]
在变化列中:“e”是指“增强”,“s”指“密封”[0383]
在表9中,向导序列(包含向导序列、任何变体,以及其源自的亲本向导序列)以rna形式示出,人工mir序列以rna形式提供,并且当嵌入载体中时以dna形式示出。使用的mir骨架包括:(a)mir155,其保留了(fowler等人,《核酸研究》(2015)44:e48)中报道的凸起形式:
(b)mir155,没有序列凸起,产生完美互补的茎(“密封”);(c)mir1-1,其保留内源性mirna中的天然凸起形式;以及(d)具有“增强”变化的mir1-1,包含先前报道的其它mirna中的3'臂中的修饰以增强加工(auyeung等人,《细胞(cell)》2013)。图13示出了使用网络服务器mfold的若干种构建体的mirna茎的预测rna折叠之一。包含向导序列或与向导序列相邻的区中的茎中的凸起是明显的,所述凸起设计为模拟获得向导序列的周围上下文的微小rna的内源性形式的天然错配。作为对照(“911对照”),xd-14792向导序列的碱基9、10和11被修饰为互补碱基(即,取代a-》t、t-》a、c-》g或g-》c);或者(“sscr”),其中除了碱基1-7之外的所有碱基都为乱序。在两种情况下,应保留任何种子介导的脱靶活性(源自碱基1-7),而应阻止中靶atxn2切片活性。
[0384]
plvx-ef1a_mcherry-mir-1-1-xd_14890-wpre_cmv(seq id no:546)是可用于表达这些人工微小rna的代表性慢病毒载体。seq id no:546的核苷酸4275-4412(mir-1-1骨架中的xd-14890向导序列)可以用另一种所关注的人工mirna取代。在此适合包装成慢病毒的慢病毒载体中,ef1-α启动子驱动mcherry蛋白的表达。在终止密码子之后,amirna茎在3'utr下游表达。wpre元件(土拨鼠肝炎病毒转录后调节元件(woodchuck hepatitus virus posttranscriptional regulatory element))的下游增强了转录物的稳定性。衔接子可以包含在人工mirna构建体的上游或下游,以促进序列的克隆和下游检测,但预期这些衔接子不会影响微小rna的性能。下游的cmv启动子(如序列所示)或pgk启动子(如在图14所示数据转染的质粒中)驱动嘌呤霉素(puromycin)抗性蛋白的表达,用于在哺乳动物细胞中选择嘌呤霉素。这与(kampmann等人,《美国国家科学院院刊(pnas)》2015)中使用的载体设计相似。
[0385]
pcdna3.1 negfp stop atxn2 3'utr.gb(seq id no:547)表示用于生成gfp-atxn2报告系的质粒。cmv启动子用于驱动编码增强型绿色荧光蛋白(egfp)的转录物的表达。在egfp开放阅读框末尾的终止密码子之后是atxn2序列,但去除了初始atg,以使所述序列预期不会被翻译。下游单独的sv40启动子驱动neor/kanr蛋白产物的表达,这使得通过g418选择能够选择稳定整合质粒的u2os细胞。egfp荧光明亮且弥散,如果atxn2蛋白被翻译并融合到egfp,则不会像预期的那样局限于细胞质。在g418选择后通过单细胞克隆生成了若干条线,最终基于facs的均匀荧光信号分布以及对照转染的(sintc)和atxn2 sirna转染的细胞之间的较大差异选择了一条线。
[0386]
通过瞬时转染(lipofectamine 3000)将具有上述人工mirna的构建体转染到稳定表达gfp-atxn2报告的u2os细胞中。四天后,通过荧光自动细胞分选(facs)量化gfp
–
atxn2水平,通过mirna载体上编码的mcherry的表达对细胞进行门控,以分离表达人工mirna构建体的细胞。图14示出了gfp强度的中值荧光强度信号。相对于表达对照构建体的细胞(xd-14792 911和xd-14792sscr,嵌入mir-155茎骨架),嵌入人工mirna骨架mir-155或mir-1-1的xd-14792序列显著降低了atxn2 gfp报告强度。其中茎是完美互补的mir-155骨架中的“密封”xd-14792构建物(图14)当嵌入具有凸起残基的mir-155或mir-1-1中时不会与xd-14792那样多地降低atxn2 gfp报告信号。
[0387]
慢病毒形式的人工微小rna载体中atxn2靶向序列的扩增筛选
[0388]
鉴于atxn2 gfp报告的敲低的令人鼓舞的结果,一组atxn2靶向序列被克隆到上文描述的人工微小rna表达载体(seq id no:546)中。与在mrna敲低的剂量应答测试中测试的相同的atxn2靶向序列集被整合到质粒中,以实现慢病毒包装。载体被包装到慢病毒中(参
见下面的方法),并跨多个板以96孔形式转导到未经修饰的u2os细胞或缺少atxn2的u2os细胞中(如下文所描述的)。每个板都有对照以实现板式信号归一化。转导后3.5天,用多聚甲醛固定细胞,用抗atxn2抗体、抗mcherry抗体和hoechst染料(33342)封闭并染色以标定细胞核,并且如上文所描述的通过图像分割和信号强度测量来量化atxn2信号。通过抗mcherry信号区分转导的和未转导的细胞。图15示出了未转导的细胞以及野生型转导的细胞的预期mcherry信号的直方图。设置阈值使得来自未转导的野生型细胞的信号不超过此阈值,但双峰分布信号右峰中的大多数细胞(右图,野生型转导的细胞)被认为是阳性的。
[0389]
在u2os细胞中测量的背景中减去atxn2信号,其中atxn2基因被crispr破坏,其中atxn2蛋白已被证实通过蛋白质印迹分析消除。图20至21示出了敲除生成过程的数据。图20示出了用靶向ataxin-2或对照靶标的cas9-grna复合物核转染的细胞中ataxin-2信号的蛋白质印迹分析和facs分析。在多个向导中观察到ataxin-2蛋白的稳健减少,这与ataxin-2基因的编辑和破坏一致。图21a示出了生成克隆atxn2敲除细胞的工作流程,并且图21b示出了对源自cas9
–
grna核转染的细胞的单细胞克隆的蛋白质印迹分析,其中克隆43被证实对ataxin-2无效并被选择用于进一步使用。通过桑格测序(sanger sequencing)对所述克隆进行测序,并且使用ice工具(synthego公司(synthego))确认了与atxn2等位基因的破坏一致的破坏性突变的混合物。
[0390]
如图16所示,相对于用二级抗体而非初级抗ataxin-2抗体处理的野生型细胞,atxn2缺失的细胞中的信号略有增加,表明atxn2抗体的一些非特异性背景结合。这些细胞没有用病毒转导。在背景减除后,将信号相对于未转导的野生型细胞中的atxn2信号归一化。
[0391]
图17示出了跨人工微小rna构建体的atxn2信号强度的孔水平量化,其中图18中示出代表性的图像。通过超过上文定义的阈值的抗mcherry水平鉴定转导的细胞。每孔3355个细胞的中值为mcherry阳性,并且包含用于atxn2信号计算,范围为2469个到4582个细胞,标准偏差为每孔391个细胞。
[0392]
表10示出了针对嵌入增强的mir-155骨架或mir1-1骨架中的序列(表11中提供的序列)如上归一化的atxn2信号的平均值和标准偏差。通常,对于大多数但不是所有序列,当向导序列嵌入mir1-1骨架时,atxn2敲低性能更好。其中人工微小rna被工程化,使得向导碱基9、10和11互补(a-》t、t-》a、c-》g或g-》c)的911个对照中没有一个表现出敲低,从而表明atxn2信号的降低取决于微小rna对内源性atxn2转录物的直接rna干扰活性。另外,当在mir-1-1骨架中检查时,跨向导序列的蛋白质水平敲低与200pm sirna处理后hepg2细胞中的mrna敲低相关。(线性模型p《.001;r2=0.5;图19)。
[0393]
表10:amirna处理后的atxn2蛋白水平
[0394]
[0395][0396]
表11提供了嵌入在活性向导序列以及一小组对照序列的微小rna骨架表达载体中的亲本向导rna序列、amirna序列和amirna dna序列。预期在细胞中产生的向导序列以rna形式描述,并且编码向导序列(嵌入mirna)的序列以dna形式提供。
[0397]
表11:amirna序列
[0398]
[0399]
[0400]
[0401]
[0402]
[0403]
[0404]
[0405]
[0406]
[0407]
[0408]
[0409]
[0410][0411]
表12中示出了具有至少25%atxn2免疫荧光信号敲低的靶向atxn2的mirna向导序列(rna和dna版本)。表13中示出了具有至少50%atxn2免疫荧光信号敲低的靶向atxn2的mirna向导序列(rna和dna版本)。
[0412]
表12:具有至少25%atxn2敲低的mirna向导序列
[0413][0414]
表13:具有至少50%atxn2敲低的mirna序列
[0415][0416]
在aav质粒中嵌入mirna
[0417]
设想当从aav基因组表达时,如上述的mirna序列对患有神经退行性疾病的患者具有治疗益处。因此,mirna序列被插入aav顺式质粒中,侧接aav2反向末端重复序列(itr)。将mirna插入内含子,然后插入表达绿色荧光蛋白(gfp)的外显子。在终止密码子之后,插入sv40聚腺苷酸化序列以确保稳健的聚腺苷酸化。将编码mirna的转录物插入cag或人突触蛋白启动子的下游,作为聚合酶ii启动子。所述序列也被插入到h1启动子下游的载体中,其中cbh启动子控制h1 mirna插入物下游的gfp表达。序列scaav.syn.mir1-1.xd14792.gfp.sv40(seq id no:622)、scaav.syn.mir1-1.xd-14887.gfp.sv40(seq id no:623)、ssaav.cag.mir1-1.xd-14792.gfp.sv40pa(seq id no:624)、ssaav.cag.mir-1-1.xd-14887.gfp.sv40pa(seq id no:625)显示插入mirna xd-14792或xd-147887的代表性顺式质粒。对于临床构建体,gfp序列被惰性序列替代,所述惰性序列源自预期在表达时不会产生影响的基因组的部分。对于含有突触蛋白或h1启动子的载体,插入物侧接一个全长itr和一个具有截短的末端解链位点的itr。
[0418]
aav质粒通过常规的大规模dna制备生成,并且通过用限制性核酸内切酶smai消化验证itr的完整性,观察到预期的带模式。质粒用于将含有mirna的基因组包装成aav9衣壳衣壳化的病毒(载体生物实验室(vector biolabs))。通过qpcr用针对gfp的引物对aav进行滴定,以计算每ml的基因组计数。
[0419]
aav尾静脉注射
[0420]
xd-14792(seq id no:112)和xd-14887(seq id no:302)的向导序列与小鼠atxn2转录物互补,其中在xd-14792的碱基22处有一个碱基对错配。wu等人(《公共科学图书馆:综合(plos one)》(2011)6:e28580)和ohnishi等人(《生物化学与生物物理研究通讯(biochem biophys res commun)》(2005)329:516-21)表明这些3'错配不会损害敲低。
[0421]
为了评估这些病毒在体内敲低atxn2的能力,在pbs(vwr公司(vwr)#k812-500ml)中的0.001%pluronic f-68(吉博科公司(gibco)#24040-032)中将浓缩的aav稀释到每200微升3*10
11
个基因组计数的浓度。每只2个月大的c57bl/6雄性小鼠尾静脉内注射(iv)200微升病毒(对于具有cag启动子的病毒,总共注射3*10
11
gc;对于具有突触蛋白启动子的病毒,注射2*10
11
gc)。注射后十五天,处理小鼠组织用于分析。在二氧化碳诱导的安乐死和用pbs经心灌注后,立即将组织在液氮中速冻。随后将样品储存在-80℃下。
[0422]
atxn2水平的蛋白质印迹分析:
[0423]
通过在干冰上切割大约50mg右中叶肝组织样品进行蛋白质提取,将每个样品放入500微升ripa缓冲液(teknova公司(teknova)#50-843-016)中,所述缓冲液补充有蛋白酶和磷酸酶抑制剂片剂(皮尔斯公司(pierce)#a32959)、halt蛋白酶抑制剂混合物(赛默飞世尔科技公司#1861279)和pmsf(细胞信号传导技术公司(cell signaling technology)#8553s)。组织在precellys evolution均质器中被破坏(管=0.5ml ck14,方案=3
×
45秒5000rpm,15秒间隔,0℃)。将样品在冰上温育30分钟,在4℃下以17,000xg离心15分钟,并且将上清液转移到新试管中并储存在-80℃下。对蛋白质裂解物进行定量(皮尔斯公司,23225),得到每个样品大约8μg/μl的蛋白质。
[0424]
nupage系统(赛默飞世尔科技公司)用于凝胶电泳。将20μg的每种样品加载到4%到12%的bis-tris蛋白凝胶(赛默飞世尔科技公司,np0321box)上,并且在恒定200v下运行1小时。revert 700(licor公司,926-11010)用于测定蛋白质加载。使用恒定的30v和90ma在4℃下将蛋白质转移到pvdf膜(emd密理博公司(emd millipore),ipfl00005)上过夜。在室温下将膜封闭1小时(洛克兰(rockland),mb-070)。将一级抗体温育在4℃下摇动过夜,包含抗atxn2(1:1000,bd,611378)、抗gfp(1:2000,cst,2956)和β-肌动蛋白(1:2000,cst,4970)。用tbs 0.1%吐温-20洗涤4
×
5分钟,并且将二级抗体在室温下摇动温育1小时(800cw山羊抗小鼠和680rd驴抗兔各1:15,000,licor公司)。再次洗涤膜并在odyssey fc成像系统(licor公司)上进行成像。信号定量由licor image-studio lite进行。
[0425]
图22(左图)示出了来自用含有cag启动子的病毒给药的动物组织的蛋白质印迹分析。相对于缺少mirna的对照病毒,用表达mirna xd-14792mir1-1(seq id no:1133)或xd-14887mir1-1(seq id no:1149)的病毒给药的动物的肝组织显示atxn2信号显著降低,通过atxn2免疫印迹信号与β-肌动蛋白信号的比率来量化(图22(右图))。如预期的,由于突触蛋白启动子的表达是cns富集的,因此具有表达相同mirna的突触蛋白启动子的aav显示出更少的gfp表达,并且没有降低atxn2蛋白水平(数据未示出)。因此,aav介导的atxn2靶向mirna的递送可以在体内调节atxn2蛋白水平,这与治疗目标一致。
[0426]
为了评估从给药到脑脊液中的aav表达的靶向atxn2的amirna是否可以降低atxn2水平,新生小鼠在出生后第0天通过脑室内途径(i.c.v.)用具有cag或突触蛋白启动子的aav-amirna给药(图53a)。使用了在mir1-1骨架(seq id no:1133)中表达xd-14792或在mir1-1骨架(seq id no:1149)中表达xd-14887的aav。与静脉内给药实验一样,载体还包含gfp报告,以允许鉴定转导的细胞。在4周或8周后收获皮层组织,并且通过蛋白质印迹评估atxn2蛋白水平以及gfp水平(图53b-53c)。在4周和8周时针对xd-14792amirna利用cag载体并且在8周时利用突触蛋白启动子载体,观察到atxn2蛋白水平相对于来自用对照、非amirna载体(mcs)给药的动物的组织有所降低。
[0427]
为了验证经历敲低的细胞类型含有治疗目的的靶细胞类型,即神经元,用针对atxn2和gfp的抗体使来自i.c.v.给药的动物的固定皮层经受免疫荧光分析。与用对照载体给药的动物相比,在用atxn2 amirna给药的动物脑中观察到抗atxn2免疫荧光信号降低的明显证据。在单个组织切片中,转导的和未转导的细胞可以通过gfp报告的表达来区分。图54a示出了皮层的免疫荧光;在来自用表达atxn2 amirna(mir-1-1骨架中的xd-14792,seq id no:1133)的aav给药的动物的组织中,将表达gfp的神经元与无gfp的神经元进行比较显示出表达gfp的神经元中的axn2信号明显减少,与无gfp的神经元相比,所述表达gfp的神经元也将表达活性amirna。在用不含活性amirna的载体处理的动物切片中,表达gfp的神经元与不表达gfp的神经元之间的atxn2表达水平没有明显差异。类似地,图54b示出了来自用表达atxn2 mirna(mir-1-1骨架中的xd-14792,seq id no:1133)的aav或对照病毒处理的动物的小脑的切片。在用atxn2 mirna(mir-1-1骨架中的xd-14792,seq id no:1133)处理的动物中,表达gfp的神经元(也将表达atxn2 mirna)具有较低水平的atxn2信号。
[0428]
材料与方法
[0429]
用于免疫染色的atxn2 sirna转染
[0430]
将u2os细胞(未经修饰的;野生型)在sirna转染前1天以5,000个细胞/孔接种到96孔平底透明黑色聚苯乙烯tc处理的微孔板(康宁公司(corning),p/n 3094)中。在将sirna从储备溶液稀释到opti-mem i减血清培养基(吉博科公司,p/n 31985-062)中后,使用lipofectamine rnaimax转染试剂(英杰公司(invitrogen)p/n 56532)生成转染混合物。然后使用apricot s-pipette s2将转染混合物等分到u2os细胞上,并置于37c/5%co2/20%02的组织培养箱中。
[0431]
atxn2 sirna免疫染色和成像方案
[0432]
在转染后三天,将细胞固定(4%多聚甲醛/4%蔗糖最终),然后洗涤(pbs)、封闭和透化(if缓冲液:0.5%bsa、0.2%皂苷、5%山羊血清)。在if缓冲液中以1:200将一级抗体(bd 611378)应用于细胞,过夜温育。pbs洗涤后,将细胞在二级抗体(生命技术公司(life technologies),alexa fluor plus 488)中温育,然后进行dna染色(赫斯特33342)。在最终pbs洗涤后,细胞在4c下温育过夜,然后在第二天成像。使用带有20x物镜的thermo scientific invitrogen evos fl auto 2成像系统,通过自动聚焦核dna染色、捕获dna染色、然后自动重新定位以捕获atxn2信号来捕获图像,其中每个孔总共成像9个场。
[0433]
人工mirna构建体开发
[0434]
使用所有寡核苷酸共有的区对含有嵌入mir-1-1和mir-155e骨架内的atxn2靶向shrna的寡核苷酸(twist)进行pcr扩增(正向:taagcctgcaggaattgcctag(seq id no:626),反向:catgtctcgacctggcttactag(seq id no:627))。扩增后,通过凝胶电泳验证pcr产物的大小是否正确。然后将稀释的pcr产物插入xba1和ecori消化的plvx ef1α》mcherry cmv》puro构建体,类似于使用neb hifi dna组装主混合物(neb p/n m5520aa)的seq id no:546。然后将一部分反应混合物与neb稳定的感受态大肠杆菌细胞(neb p/n c3040h)在冰上温育,在42℃下热休克,在冰上恢复,然后添加s.o.c.培养基并在30℃下温育。然后将细菌培养物应用于具有抗生素羧苄青霉素(carbenicillin)的lb琼脂平板上,并在30℃下生长过夜。对单个细菌菌落进行了桑格序列验证(引物:catagcgtaaaaggagcaaca(seq id no:628))。在基于桑格测序验证正确插入后,使细菌培养物生长并纯化和量化质粒dna。
[0435]
病毒产生
[0436]
使用经过序列验证的构建体,使用lenti-x 293t细胞(塔卡拉公司(takara))和pc-pack2质粒混合物(cellecta p/n cpcp-k2a)产生慢病毒。使用lipofectamine 3000转染试剂盒(英杰公司,p/n l3000-008),用单个plvx ef1a》mcherry mir insert cmv》puro构建体和pc-pack2 plasmid mix转染lenti-x 293t。吸出含有转染的培养基并用病毒产物培养基(vpm;293t培养基 20mm hepes(吉博科公司,p/n 15630-08))替代。48小时后收集vpm并等分到96孔2.0ml深孔板(赛默飞世尔科技公司,p/n 4222)中并在-80℃下冷冻。
[0437]
病毒转导
[0438]
在将vpm添加到细胞之前,将u2os野生型(未经修饰)和atxn2敲除(c43)以5,000个细胞/孔在8小时前接种到96孔平底透明黑色聚苯乙烯tc处理的微孔板中(康宁公司,p/n 3094)。添加聚凝胺(最终8μg/ml,cellecta,p/n ltdr1)后,使用apricot s-pipette s2添加解冻的vpm。然后将细胞置于37℃/5%co2/20%o2的组织培养温育箱中。12小时后除去细胞上含有vpm和聚凝胺的培养基并且换上新鲜培养基(仅限u2os培养基),然后放置到37℃/5%co2/20%o2的组织培养温育箱中。
[0439]
atxn2 plvx ef1a》mcherry mir insert cmv》puro免疫染色和成像方案
[0440]
在更换培养基后三天(vpm后3.5天),将细胞固定(最终4%多聚甲醛/4%蔗糖),然后洗涤(pbs)、封闭和透化(if缓冲液:0.5%bsa、0.2%皂苷、5%山羊血清)。将一级抗体(atxn2;bd 611378,1:200稀释,mcherry;ab205402,1:1000稀释)应用于if缓冲液中的细胞并过夜温育。pbs洗涤后,将细胞在二级抗体(生命技术公司,alexa fluor plus 488和alexa fluor plus 647)中温育,然后进行dna染色(赫斯特33342)。在最终pbs洗涤后,细胞在4℃下温育过夜,然后在第二天成像。使用带有20x物镜的perkinelmer operetta cls高含量分析系统,通过自动聚焦板底部捕获非共焦图像,然后捕获dna信号、atxn2信号和mcherry信号,其中每个孔总共成像9个场。
[0441]
mir-155和mir-1-1转染和atxn2蛋白质印迹
[0442]
将mir-155支架的版本工程化为人工mirna,并且用于小鼠体内概念验证研究以敲低htt
10
。靶向atxn2的向导序列和对照被并入此支架序列,称为“mir-155m”,并且在转染u2os细胞后测定蛋白质敲低。
[0443]“mir-1-1e”,其中“e”表示“增强”,采用人mir-1-1支架并简单地引入下游cnnc基序。
[0444]
为了进行转染,将u2os细胞以90,000个细胞/孔接种在12孔培养皿中,24小时后,用lipofectamine 3000(赛默飞世尔科技公司)转染2微克/孔的8个ef1alpha》mcherry构建体(7个具有插入物,1个对照)。具体地,每次转染使用2μl增强剂试剂,1.5μllipofectamine试剂;在水中稀释样品到均匀量。
[0445]
第二天用evos成像,观察到大量mcherry细胞。在没有插入物的对照载体中观察到更高的表达水平。
[0446]
将孔吸出并用1ml含有1微克/ml嘌呤霉素的培养基替换。选择在周末进行,然后去除嘌呤霉素进行恢复。
[0447]
选择后三天,观察到许多死细胞。然而,mcherry的成像表明仍有大量明亮的存活细胞。吸出培养基并更换为含有200ng/ml嘌呤霉素(5倍稀释)的预热培养基。
[0448]
两天后(转染后7天),用pierce磷酸酶和蛋白酶抑制剂片剂在ripa缓冲液中裂解细胞。
[0449]
在odyssey fc(licor公司)上进行蛋白质印迹并成像。
[0450]
使用imagestudio软件(licor公司)对atxn2带和对照α-微管蛋白信号强度进行定量。
[0451]
在u2os细胞中生成atxn2敲除
[0452]
u2os细胞中的atxn2敲除细胞是使用cas9-grna rnp核转染方法进行的。简而言之,crrna和tracrrna(idt)以等摩尔比进行双链体化并与重组cas9(idt v3)复合,并且使用sf缓冲液和cm130程序(lonza 4d nucleofector)进行核转染。
[0453]
crispr向导rna选自两个crispr库来源。从cellecta crispr切割库中选择了三个crispr向导rna(gatxn2_1、gatxn2_2、gatxn2_3)(一个由于其上游位置在第2个atg之前而没有选择)。从bassik等人
26
报告的另一个crispr切割库中选择了两个另外的向导(gatxn2_4和gatxn2_5)。另外,从cellecta库中选择了非靶向对照向导。表14中提供了crispr向导rna序列以及dna形式。
[0454]
表14:用于靶向atxn2的crispr向导rna序列
[0455][0456]
核转染后,大量细胞被允许恢复5天并裂解用于蛋白质印迹分析。
[0457]
u2os克隆选择
[0458]
大量细胞也被单细胞分选到96孔板中用于克隆扩增。因为通过蛋白质印迹,向导gatxn2_1和gatxn2_5的atxn2蛋白信号减少最多(减少~90%),所以继续使用这些细胞进行单细胞克隆。在胰蛋白酶消化和单细胞悬浮后,使用sony sh800s单线态门控细胞并直接分选到u2os生长培养基中。允许细胞生长~2周到3周,并且裂解用于基因组dna提取以进行桑格测序和蛋白质提取以进行蛋白质印迹(在此设置中,每个泳道使用10μg蛋白质)
[0459]
克隆的ice确认
[0460]
使用qiagen血液和组织试剂盒提取基因组dna。基因组引物被设计成扩增向导rna切割位点周围的基因组区域,目的是通过桑格测序对切割位点进行测序,并且验证与单个克隆一致的框外indel模式。
[0461]
使用具有以下设置的primer blast(https://www.ncbi.nlm.nih.gov/tools/primer-blast/):对于向导1,由于atxn2的重复性,关闭了重复过滤器和低复杂度过滤器,但在其它方面保留了默认设置。使用snapgene的导入功能从ncbi导入“6311”。来自原间隔子序列的500个上游碱基和500个下游碱基被用作primer blast的输入。产物大小设置为400到1000,并且选择了2个不同的引物对(表15)。
[0462]
表15:atxn2 pcr引物
[0463][0464]
此外,扩增子内部测序引物被设计用于正向和反向桑格测序以读取切割位点(表16)。引物( )算法(http://www.biology.wustl.edu/gcg/prime.html)用于在此网络界面(https://www.eurofinsgenomics.eu/en/ecom/tools/sequencing-primer-design/)上设计测序引物。
[0465]
表16:atxn2测序引物
[0466]
引物名称引物序列ice_gatxn2_1_rseqagagccatccttaggtagcc[seq id no:640]ice_gatxn2_1_fseqgaggtttaattgactcatgctctg[seq id no:641]
[0467]
对于向导5,关闭了重复过滤器但打开了低复杂度过滤器,但在其它方面保持默认设置。来自原间隔子序列的500个上游碱基和500个下游碱基被用作primer blast的输入。产物大小设置为400到1000,并且选择了2个不同的引物对(表17)。
[0468]
表17:atxn2引物序列
[0469][0470]
内部测序引物由引物( )算法设计(表18)。
[0471]
表18:atxn2测序引物
[0472]
引物名称引物序列ice_gatxn2_5_fseqtcatgagcatccacaagaacag[seq id no:646]ice_gatxn2_5_rseqacgtgtgtgagtgtaactgattgc[seq id no:647]
[0473]
使用nebnext ultra ii q5 master mix(neb公司(neb),m0544s)进行pcr,gdna和引物对如上所述。扩增的产物通过琼脂糖凝胶可视化,正确大小的扩增子经过凝胶纯化并提交用于使用正向和反向测序引物的桑格测序。色谱图(.abi文件)结果已上传到inference of crispr editing(ice)工具
27
https://ice.synthego.com/#/用于桑格读段的反卷积以鉴定indel。
[0474]
通过蛋白质印迹和桑格测序证实为真正的敲除克隆的来自向导5核转染的克隆43用于进一步研究。
[0475]
ataxin-2蛋白质印迹
[0476]
为了制备裂解物,向1x ripa(teknova公司(teknova)、tris-hcl 50mm、nacl 150mm、1%triton x-100、脱氧胆酸钠1%、sds 0.1%、edta 2mm、ph 7.5)补充pierce蛋白酶/磷酸酶片剂(赛默飞世尔科技公司,a32959)并在冰上温育15分钟,在4℃下以17,000xg旋转沉降15分钟。
[0477]
pierce bca试剂盒(赛默飞世尔科技公司,23225)用于蛋白质定量,并且在sds-page凝胶电泳(nupage bis tris,赛默飞世尔科技公司)中每泳道加载20μg蛋白质。
[0478]
用10微克或20微克蛋白质、4x lds加载缓冲液(np0007)、10x样品还原剂(np0004)、20μl水制备样品。样品在70℃下加热10分钟。
[0479]
蛋白质大小梯是precision plus蛋白质双色标准(伯乐公司(bio-rad),1610374)或duo预染色蛋白质梯(licor公司,928-60000)。
[0480]
将样品加载到1.0mm
×
10孔4%到12%bis-tris蛋白凝胶(np0301pk2)上,并且用mops sds运行缓冲液(np0001)在恒定200v下运行凝胶电泳1小时20分钟以分辨更高分子量的带。
[0481]
使用不含甲醇的三羟甲基氨基甲烷(tris)/甘氨酸转移缓冲液(伯乐公司,1610734)。将包含海绵、滤纸、凝胶和膜在内的所有组分用转移缓冲液平衡至少15分钟。在用转移缓冲液平衡之前,将pvdf膜浸入甲醇中15秒。在mini trans-blot electrophoretic transfer cell(伯乐公司,1703930)中在4℃下在恒定30v、90ma下过夜进行湿转移。
[0482]
过夜转移后,将膜在室温下风干1小时。用1x tbs(无吐温)冲洗膜,并在odyssey封闭缓冲液(li-cor公司)中在室温下摇动30分钟到1小时进行封闭。
[0483]
将膜与一级抗体在4℃下以1:1000稀释度在含有0.1%吐温20的odyssey封闭缓冲液中温育过夜。小鼠atxn2抗体(bd,611378)和兔α-微管蛋白抗体(cst,2144s)用作加载对照。
[0484]
用tbs-0.1%吐温20洗涤膜4
×
5分钟。
[0485]
在含有0.1%吐温20和0.01%sds的odyssey封闭缓冲液中,用两种二级抗体在室温下以1:20,000的稀释度摇动1小时处理膜。
[0486]
二级抗体是irdye 800cw山羊抗小鼠igg(li-cor公司,926-32210)和irdye 680rd驴抗兔igg(li-cor公司,926-68073)。在具有700个和800个通道的li-cor odyssey扫描仪
(fc)上成像之前,用tbs-0.1%吐温20洗涤膜4
×
5分钟并用tbs(无吐温)冲洗。
[0487]
ataxin-2 facs
[0488]
将细胞胰蛋白酶消化,转移到96孔v底形式,每个处理一式三份测定,并且在洗涤缓冲液(pbs/0.5%bsa(无edta))中洗涤,并且用冰冷的甲醇逐滴固定,在冰上温育10分钟,然后添加200μl pbs并在4℃下摇动细胞过夜。
[0489]
将细胞以1000xg、5分钟冷旋转沉降并用冷facs洗涤缓冲液(pbs/0.5%bsa/2mm edta/0.2%皂苷)洗涤两次。以1:100应用一级抗体(bd 611378)并温育1小时,在4℃中摇动。缓冲液补充有5%山羊血清以减少非特异性结合。将细胞在冷facs洗涤缓冲液中洗涤两次。将细胞在1:100二级抗体(pe/cy7 biolegend clone rmg1-1)中温育,其中细胞重悬于含有5%山羊血清的冷facs洗涤缓冲液中,并且在冰上温育1小时。将细胞洗涤两次并重悬于冷facs洗涤缓冲液中并在attune(赛默飞世尔科技公司)上取样。
[0490]
脑室内注射
[0491]
对于脑室内注射,对出生后第0天的幼鼠进行冷冻麻醉,并且使用汉密尔顿注射器(hamilton synringe)在2mm的深度注射,每个脑室递送的最大体积为3ul。
[0492]
免疫荧光分析
[0493]
用raav i.c.v给药的动物在用raav给药4周后实施安乐死,在4%多聚甲醛中固定过夜。然后将组织冷冻保存在冷的30%蔗糖中,然后嵌入oct培养基并冷冻。在恒冷箱切片机上制备5微米冷冻切片。对于染色,将切片解冻并干燥,在pbs中洗涤两次,在95c抗原修复溶液(citra抗原修复,ph 6.0,载体实验室(vector labs)#h-3300-250)中加热10分钟,然后在室温下冷却30分钟。然后将切片在水、pbs和pbs-0.25%triton-x-100中各洗涤5分钟,并且在pbs中洗涤10分钟。然后在加湿室中用pbs中的5%山羊血清封闭切片30分钟。切片用pbs 1%bsa中的一级抗体溶液(包含:小鼠抗atxn2抗体(bd#611378),1:50;兔抗gfp抗体(细胞信号传导技术公司#25555))以1:2000在4c下处理过夜。在pbs中洗涤3次后,将切片与pbs 1%bsa中的二级抗体溶液(包含:山羊抗小鼠alexa fluor 555(赛默飞世尔科技公司#a21424)1:250、山羊抗兔alexa fluor 488(赛默飞世尔科技公司#a11008))以1:250在室温下一起温育30分钟。然后洗涤切片,并且用dapi(h-2000-10)固定在vectashield plus中。用revolve显微镜(discover echo公司(discover echo))收集图像。
[0494]
实施例2:通过慢病毒形式的a
txn
2靶向
mi
rna的平铺筛选鉴定高性能
ami
rna
[0495]
作为sirna筛选的替代方法,随后将相关的向导序列嵌入mirna骨架并逐一测试,进行了靶向atxn2的mirna的汇集筛选(“深度筛选1”)。
[0496]
atxn2靶向序列
[0497]
智人atxn2 mrna(nm_002973,转录变体1,seq id no:2)用于鉴定人工mirna的靶序列。鉴定了所有人类和灵长类动物的交叉反应序列,并且考虑了有效shrna和mirna序列的标准,设计了22-nt向导序列,包含在向导位置1处对a或u的偏好。因此,考虑到与ataxin-2构建体互补的22个核苷酸反义序列,如果第一个向导碱基是g或c,则将其转化为“u”,而以a或u开头的序列不会从与atxn2转录物上的对应位置互补的碱基改变。如上所述,u碱基在慢病毒表达构建体中编码为t。总共有2,381个靶向atxn2的序列被引入mir-16-2骨架的经修饰的变体中。此骨架的过客序列(来自向导序列的mirna茎的相反侧上的序列)是遵循表8中的规则生成的。
[0498]
毒性对照
[0499]
通过检查已允许长时间生长的细胞与最初转导的细胞中库元件的丰度,汇集的筛选可以鉴定改变细胞增殖或活力的元件。为了校准测定的动态范围,将另外的有毒元件添加到库中。选择了十个必需基因,每个基因都有十个shrna(去除2个具有被认为有问题的polyt序列的序列,因为其可能作为poliii的终止信号)。为了鉴定“必需”基因列表,检查了与k562癌细胞系
21
中的shrna和crispr向导rna并行进行的基因脱落筛选。在两个筛选中,将基因按shrna致死性排序,特别是在k562 shrna脱落中通过组合castle评分获得高评分的基因(阴性更致命)。由于在hela细胞中进行了毒性筛选,k562靠前基因被交叉并鉴定了在hela crispr切割脱落筛选
22
中评分也很高(贝叶斯因子(bayesian factor)》100)的前10个基因。选择的必需基因是:copb1、copb2、dhx15、eif3a、eif4a3、nup93、prpf8、psmb6、psmd1和sf3b2。
[0500]
为了选择靶向每个基因的10个shrna,考虑了先前公开的shrna库中的25个shrna/基因,并且根据其在脱落筛选
15
中的性能进行排序。具体地,将shrna按脱落指标(重复1和重复2中的读段计数除以质粒读段)进行排序,并且选择在所有重复中具有至少一个计数的性能最佳的shrna。
[0501]
gfp对照
[0502]
gfp对照(n=50)设计为靶向两种不同的gfp报告系统。第一个系统涉及用gfp的第11个β链(gfp
11
)与gfp
1-10
的过表达一起标记内源性atxn2以构成自互补的gfp系统
23
,第二个系统是gfp-stop-atxn2过表达报告。通过使用28个单独的21nt shrna完全平铺转录物,在向导位置1处添加a以形成22nt寡聚体序列来靶向gfp的第11个β链。使用博德研究所基因扰动平台(broad institute genetic perturbation platform,https://portals.broadinstitute.org/gpp/public/seq/search)的设计门户,使用gfp
1-10
序列作为输入,选择另外的shgfp(n=22)以靶向gfp
1-10
。尽管拆分gfp系统最终并未用于读出atxn2水平,50shgfp仍然靶向gfp-stop-atxn2报告。
[0503]
atxn2转录物乱序对照
[0504]
设计的中性对照在功效和毒性筛选中均不应有任何影响。这些元件可以用于基线归一化。靶向atxn2的向导序列被乱序,并且这些乱序向导序列中的974个用于像以前一样构建amirna。乱序后,对第一个碱基施加与靶向序列相同的规则。在此校正步骤之后,通过将随机选择的a或t的向导碱基2到22之一转化为随机选择的g或c来调整gc含量,以使此组乱序对照总体上保持相对于靶向atxn2的序列相似的gc含量。
[0505]
启动子选择
[0506]
h1启动子是一种rna聚合酶iii启动子,被选择用于驱动人工mirna表达,因为许多小组已经使用其来实现稳健的靶敲低。
[0507]
集合库克隆
[0508]
在芯片上合成寡核苷酸池(寡核苷酸长度172bp,安捷伦公司(agilent)),pcr扩增,并且通过bpl1限制性消化和t4连接酶连接克隆到prsicph1载体(cellecta)中。每个单独的mirna盒在h1启动子的控制下表达,随后是短的恒定区和17bp条形码序列,该序列唯一地标记每个mirna。这些元件被设计为含有mirna和条形码标签,以实现多种方式来对构建体进行扩增和测序以读出汇集筛选。例如,如果pcr扩增偏差会混淆高gc含量序列的表
示,比较含有向导序列的扩增子丰度与含有free条形码的扩增子丰度将解决任何差异。使用free条形码,因为其为indel校正的,并且对dna合成和ngs错误稳健
25
。通过桑格测序和下一代序列(依诺米那公司(illumina))检查库以验证没有合成错误,》99%的amirna和free条形码正确配对,并且顶部与底部amirna之间的折叠表示在四倍变化范围内。
[0509]
病毒产生
[0510]
lenti-x 293t(塔卡拉公司)细胞用于通过转染第4代包装质粒(lenti-x包装单发,塔卡拉公司)来产生慢病毒,然后用lenti-x浓缩器进行病毒浓缩并重悬于pbs中。通过感染和抗生素选择在u2os和hela细胞中对病毒进行滴定,然后通过cell-titer-glo(普洛麦格公司(promega))估计病毒单位和感染复数(moi)。
[0511]
细胞培养和转染
[0512]
u2os细胞和gfp-atxn2报告细胞系在补充有10%胎牛血清(fbs)和青霉素/链霉素(streptomycin)/谷氨酰胺的rpmi-1640中培养。hela细胞在补充有10%胎牛血清(fbs)和青霉素/链霉素/谷氨酰胺的dmem中培养。
[0513]
功效筛选
[0514]
对中靶功效进行了两次汇集慢病毒mirna筛选以鉴定降低atxn2蛋白信号的mirna,通过1)外源性gfp-stop-atxn2报告或2)facs测定中的内源性ataxin-2抗体读出atxn2水平。用汇集慢病毒库以0.1的感染倍数(moi)将细胞感染到具有聚凝胺(8μg/ml,emd密理博公司)的(~5
×
107个细胞)中,并且分布在四个t225烧瓶中。感染两天后,用2μg/ml的嘌呤霉素选择u2os细胞。moi在第5天(选择后3天)以96孔形式通过cell-titer-glo确认。在第7天收集未分选的分数(7
×
106个细胞)作为参考对照。剩余的细胞在洗涤缓冲液(pbs/0.5%bsa(无edta))中洗涤,并且在第7天用冰冷的甲醇逐滴固定,同时涡旋,比例为1ml甲醇/2
×
106个细胞,在冰上温育10分钟,然后添加10x体积的pbs,并且在4℃下摇动细胞过夜。
[0515]
将细胞以1000xg旋转沉降5分钟(康宁公司500ml离心管,431123)并重悬于冷facs洗涤缓冲液(pbs/0.5%bsa/2mm edta/0.2%皂苷)中。对细胞进行计数并以2
×
106/ml重悬于冷facs洗涤缓冲液中。
[0516]
以1:200应用一级抗体(bd 611378)并温育1小时,在4℃中摇动。缓冲液补充有5%山羊血清以减少非特异性结合。
[0517]
将细胞在冷facs洗涤缓冲液中洗涤两次。将细胞在1:200二级抗体(pe/cy7 biolegend clone rmg1-1)中温育,细胞重悬于含有5%山羊血清的2
×
106/ml冷facs洗涤缓冲液中,并且在冰上温育1小时。将细胞洗涤两次,并且以4
×
106/ml到5
×
106/ml重悬于冷facs洗涤缓冲液中,以在sony sh800s上达到每秒1000个到2000个事件(大约是仪器上的最大稳定细胞速度)。样品通过细胞过滤器直接过滤到facs管(falcon 352235)中。将分选的细胞收集在3ml pbs/10%fbs的15ml锥形瓶中。
[0518]
脱落筛选
[0519]
通过鉴定早期与晚期时间点之间的mirna脱落,还进行了针对脱靶毒性的汇集慢病毒mirna筛选。用聚凝胺(8μg/ml,emd密理博公司)以0.1的感染倍数感染hela细胞,以1000x表示(即,细胞数量》库元件数量的10,000x)。感染两天后,用0.5微克/ml的嘌呤霉素选择hela细胞。细胞传代总共10倍(~16天)。进行一式三份的筛选(3个单独感染)。
[0520]
dna加工
[0521]
使用machery nagel blood l试剂盒(facs收集;早期和晚期收集时间点)从每个样品中提取基因组dna。进行两步pcr。在第一个pcr反应中,生成了跨越向导序列和过客序列并在下游经过free条形码的扩增子。在第二个pcr反应中,生成了跨越向导序列和过客序列或free条形码的嵌套扩增子。第二个pcr旨在将illumina结合序列(p5和p7)与样品索引条形码结合,以便在illumina测序平台上进行解复用。每个不同的样本(即,facs收集或时间点)都被赋予了不同的索引。具体地,向导和过客扩增子是单索引的,i7序列包含在6nt样品条形码和p7序列的上游。相反,free条形码扩增子在p5端是单索引的,并且在p7端不包含i7序列。样品在illumina miseq上进行测序,以便向导序列和过客序列可以在配对读段中匹配,读段1使用自定义引物读取22nt向导序列,读段2是读取过客序列的标准illumina引物。free条形码也被单独扩增和测序,读段1是读取17nt free条形码的自定义引物,读段2是读取6nt样品条形码的自定义引物。通常,对于源自free条形码的扩增子和含有扩增子的向导/过客序列的丰度计算高度相似(使用free条形码和向导/过客序列之间关联的查找表)。下面的分析集中在向导序列的计数上。
[0522]
计算分析
[0523]
在直接对amirna向导序列进行测序的读段1序列中对每个向导序列的出现进行计数,不容忍测序或其它错误(即,不容忍与库输向导序列的错配)。为了估计atxn2敲低效率,将atxn2高facs收集中的向导序列计数丰度除以atxn2低facs收集中的向导序列计数丰度。atxn2低facs收集中丰富了有效敲低atxn2的序列。
[0524]
为了评估向导序列是否影响细胞毒性或减少增殖,测量了库转导后16天收集的细胞池中每个向导序列的计数比率与库转导后18小时收集的库的计数比率。
[0525]
数据在重复间高度一致。图23a绘制了两个独立重复彼此的高/低计数比对彼此。大多数点落在y=x上,表明相关性良好。图23b绘制了每个条件相对于所有其它条件的计数值的斯皮尔曼相关系数矩阵。重复是分层聚类的,聚类块表示相似的条件。应注意低与高条件之间的强反相关性,正如预期的那样,耗竭atxn2的向导预计在低和高条件下存在差异。还应注意,通过针对内源性ataxin-2蛋白的抗体染色可视化了atxn2信号的条件,以及通过atxn2 gfp报告的荧光可视化了信号的条件,所述条件是相关的。
[0526]
在计算计数比率之后,采用归一化程序通过atxn2靶向序列耗竭atxn2信号的能力对其进行排序。在图24中,示出了用于atxn2靶向向导、顶部网格和乱序序列、底部网格的高和低条件向导序列计数分布的直方图。atxn2乱序序列在高和低atxn2 facs条件下表现出尖锐的单峰计数比率分布。此分布的中值比率被认为是无效的,因此通过减去此(对数底数2-转换的)值来计算atxn2靶向mirna的txn2耗竭效应。
[0527]
检查了向导序列敲低atxn2的能力以及是否存在任何改变的增殖或细胞毒性。图25示出了此实验中三类向导序列的图:atxn2靶向序列、atxn2乱序序列和靶向必需基因的amirna(预计有毒)。如预期的,对数底数-2atxn2信号耗竭(从高到低atxn2 facs条件的计数中乱序基线校正的atxn2耗竭)以0为中心(无影响)。然而,这些序列中的许多序列在转导后16天的后期收集时间点与转导后的早期时间点相比,表现出显著的丰度变化。这与报告的这些序列的必要性一致,并且表明此系统可以引起细胞毒性或增殖障碍。
[0528]
与靶向必需基因或乱序序列的amirna相比,atxn2靶向向导序列沿着atxn2信号耗
竭轴的范围更广,靶向序列超过5个对数(碱基2),对应于高atxn2 facs收集与低atxn2 facs收集中表达这些amirna的细胞的大约32倍耗竭。
[0529]
atxn2转录物的近乎完全平铺使得能够检测ataxin-2靶向向导序列的“热点”,所述热点由其ataxin-2转录物互补区域的接近度定义。图26示出了敲低功效的图,如通过来自高atxn2 facs收集与低atxn2 facs收集的给定向导的计数耗竭所测量的。在转录物中,注意到相邻atxn2靶向向导序列表现出强atxn2敲低的多个区域。图27示出了atxn2的3'utr内区域的“放大”,并且突出显示具有异常高atxn2降低的向导序列(作为暗点),如通过计数减少所测量的。
[0530]
确认汇集筛选中pri-mirna加工精度的小rnaseq确认
[0531]
通过连续的drosha和dicer加工从mirna茎中切除向导序列。每种酶都会切割rna。在用于atxn2的此平铺筛选的mir骨架的情况下,来自对应的内源性mirna(mir 16-2)的向导序列从上游5主要臂中切除,因此向导序列由drosha从5'侧的亲本茎上切割。因为5'切割位点的位置决定了种子序列的组成,从5'核苷酸开始计数碱基2-7,所以切割位置对于确定所得向导序列的中靶和脱靶活性都很重要。因此,进行小rnaseq以评估此切割的位置。
[0532]
以包装的慢病毒形式的平铺库以高感染复数转导到u2os细胞中。通过嘌呤霉素选择以消除未转导的细胞(库载体含有嘌呤霉素选择盒)后,通过标准方法提取rna,纯化小rna并与接头连接,以便使用nextflex small rnaseq试剂盒v3进行小rna测序。pcr扩增后,所得的库在illumina miseq上进行下一代测序。大部分读段具有长度为21个、22个和23个核苷酸的序列,在22个核苷酸处有峰值,这与加工的mirna(向导和过客序列)的检测一致。为了检查5'加工的精度,计算了与已加工的向导序列的若干个模型匹配的22-mer序列的观察次数。在一个模型中,假设向导序列被正确加工。在其它模型中,假设向导序列在预期核苷酸的上游或下游进行加工。如果向导序列在预期核苷酸的上游被切割,则预期上游碱基从mirna骨架序列中并入。如果向导序列在预期核苷酸的下游被切割,则所得向导序列的第一个碱基位于预期的下游。因为库中的乱序序列通常不会相互重叠,例如,降低“碰撞”的可能性,其中通过在预期的第一个核苷酸下游的核苷酸处从茎中切除而加工的向导序列与比对atxn2转录物中一个bp移位的位置的向导序列相同,分析并平均所有乱序序列的加工位置以估计最可能的切割位置。图28绘制了在每个核苷酸处具有切割位置的向导序列的读段相对于预期的第一个核苷酸的百分比,并且显示了非常高比例的读段开始于预期的位置。
[0533]
来自汇集筛选的其它atxn2靶向序列
[0534]
通过检查与atxn2转录物互补的位置对atxn2的敲低功效(通过从高与低atxn2facs收集中耗竭来测量),注意到若干个所关注区域,其中观察到高性能靶向atxn2的向导序列簇。表19列出了这些向导序列、向导序列相对于atxn2转录物(seq id no:2)的靶向位置、插入到mirna16-2骨架中的向导序列(根据上述小rnaseq实验,其也是细胞中生成的概率最高的序列),以及为mir16-2骨架生成的过客序列。向导序列、mirna16-2形式的过客序列和amirna序列在表19中以rna形式和dna形式提供(例如,用于插入到aav的质粒中)。表19中还提供了rna和dna形式的示例性过客rna序列(例如,未针对特定mirna骨架进行修饰)。atxn2敲低的功效由信号耗竭列表示。总而言之,具有高效和低脱落可能性的序列可能代表很好的候选物,可以并入靶向atxn2的治疗载体。
[0535]
表19:来自平铺筛选的靶向atxn2的“热点”中的向导序列以及对应的过客和mirna
序列
[0536]
[0537]
[0538]
[0539]
[0540]
[0541]
[0542]
[0543]
[0544]
[0545]
[0546]
[0547]
[0548]
[0549]
[0550]
[0551]
[0552]
[0553]
[0554]
[0555]
[0556]
[0557]
[0558]
[0559]
[0560]
[0561]
[0562]
[0563]
[0564]
[0565]
[0566]
[0567]
[0568]
[0569]
[0570]
[0571]
[0572]
[0573]
[0574]
[0575][0576]
实施例3:在人神经元的慢病毒转导中测试来自汇集筛选的最高命中
[0577]
来自汇集深度筛选1(实施例2)的若干个最高命中被克隆到慢病毒载体中,包装并在干细胞源性运动神经元培养物中测试atxn2 mrna和蛋白质的敲低。h1-mir-16-2_1755-amely_v1_cmv_gfp_lenti(seq id no:1521)中给出了示例慢病毒载体,其中含有嵌入mir-16-2骨架中的atxn2转录物的amirna靶向位置1755,或者此处描述的其它载体。载体中的amirna序列(例如,seq id no:1521的核苷酸1889-2020)可以用对应的amir或对照非mirna序列(mcs)替换,但载体的其余部分保持不变。运动神经元的表征(图29)显示培养物(下文方法中描述的分化方案)生成富含具有精细的神经元突起的运动神经元的培养物。amirna被嵌入具有h1启动子以及gfp表达盒的慢病毒载体(图30a)中。在第一个实验中,在两种不同剂量下测试了两种amirna,即靶向编码序列中位置1784处的atxn2(向导序列seq id no:112)或具有mir-16-2骨架的位置4402处的atxn2(向导序列seq id no:1279)。通过mrna的qpcr分析和蛋白质的蛋白质印迹分析分别检测到atxn2 mrna和蛋白质的强敲低(图30b至30c)。在此测定中测量的蛋白质水平显示,蛋白质水平的下降幅度大于mrna水平,表明mrna的测量可能至少表示被给定的amirna降低的atxn2蛋白的量。令人惊讶的是,靶向atxn2编码序列(1784)的amirna比靶向3'utr(4402)的amirna产生更大的敲低,这与深度筛选1中这些amirna的相对性能不同。
[0578]
作为靶向编码区与3'utr的amirna的进一步研究,进行了第二个实验(图31)。在此情况下,所有神经元都以介于第一个人神经元慢病毒给药实验中测试的两个水平之间的剂量进行处理。如前所述,在这些神经元培养物中,靶向编码序列的amirna(1755(向导序列seq id no:1185)、1784(向导序列seq id no:112)、3302(向导序列seq id no:1216)、3330(向导序列seq id no:1811)和3805(向导序列seq id no:1221))产生比靶向3'utr(4402(向导序列seq id no:1279)、4242(向导序列seq id no:1233)和4502)的amirna更强的敲低。对于一些amirna,如1755(向导序列seq id no:1185)、1784(向导序列seq id no:112)和3330(向导序列seq id no:1811),mrna减少的量超过75%。
[0579]
方法
[0580]
运动神经元产生
[0581]
根据标准程序,诱导多能干细胞(gm25256,科里尔研究所(coriell institute))在无饲养层条件下,在基质胶涂覆的板上的mtesr1培养基中培养。为了开始分化,在6孔培养皿中生长的ipsc菌落用500ul relesr解离,在37c下温育3分钟,并且轻轻搅拌。添加1ml完整的mtesr1培养基以停止解离。收集细胞悬浮液,去除relesr并将细胞重悬于n2b27分化培养基中:50ml的50%mtesr1和50%nb27分化培养基(50%dmem-f12、50%neurobasal培养基、1x n-2补充剂、1x b-27补充剂、无异种、0.5x青霉素-链霉素、1x 2-巯基乙醇、20um l-抗坏血酸)。添加rock抑制剂y-27632(5微摩尔)、ldn(200nm)、sb 431542(40微摩尔)和chir 99021(3微摩尔)。然后将细胞悬液转移到75cm2超低附着u形烧瓶中24小时。然后细胞聚集成小球体。
[0582]
然后在第2天、第4天、第6天、第9天和第12天进行培养基更换。包含的培养基(全部基于n2b27分化培养基):第2天:视黄酸(1微摩尔)、sag(1微摩尔)、ldn-193189(0.2微摩尔)、sb 431542水合物(40微摩尔)、chir 99021(3微摩尔)。第4天:视黄酸(1微摩尔)、sag(1微摩尔)、ldn-193189(0.2微摩尔)。第6天:视黄酸(1微摩尔);凹陷(1微摩尔)。第9天:视黄酸(1微摩尔)、sag(1微摩尔)、dapt(10微摩尔)。第12天:dapt(10微摩尔)。到第14天,神经元球体出现并解离成板运动神经元。
[0583]
然后用木瓜蛋白酶:dna酶溶液解离神经元球体并研磨4-5x。然后将细胞悬液分入6孔板的孔中;并且在温育15分钟后进一步研磨。解离后,用dmem和敲除血清替代物(kosr)混合物使酶失活,离心,在90%dmem/10%kosr中再次洗涤,离心,并且重悬于完整的神经基础培养基中:神经基础培养基培养基、1x n-2补充剂、1x b-27补充剂、无异种、0.5x青霉素-链霉素、20um l-抗坏血酸、1%kosr、rock抑制剂y-27632(5微摩尔)、gdnf(10ng/ml)、bdnf(20ng/ml)、cntf(10ng/ml)、dapt(5微摩尔)。然后将细胞再次离心,重悬于完整的神经基础培养基中,通过40微米细胞过滤器,通过台盼蓝染色和血细胞计数器计数,然后稀释到20k/孔(96孔形式)或200k/孔(24孔格式)用于铺板在pdl/层粘连蛋白涂覆的板中。细胞在一定体积的神经基础培养基中培养:200ul/孔(96孔形式)或1ml/孔(24孔形式)。
[0584]
pdl/层粘连蛋白涂层是通过用100微克/ml的pbs中的聚-d-赖氨酸溶液在4c下过夜处理板来完成的;用pbs洗涤3次;然后在4c下用50微克/ml的pbs中的层粘连蛋白溶液处理板过夜。
[0585]
铺板48小时后,50%的培养基更换为神经元维持培养基(神经基础,具有1x n-2补充剂、1x xeno-free b-27补充剂、0.5x青霉素-链霉素、20微摩尔l-抗坏血酸,具有10ng/ml gdnf、10ng/ml bdnf、10ng/ml cntf),包含dapt(5微摩尔)。此后,每周更换3次50%的培养基,不包含dapt。
[0586]
与上述方案相关的参考文献包括:(通过双重抑制smad信号传导对人es和ips细胞进行高效的神经转化(chambers等人,《自然生物技术》(2009)27:275-280)以及(maury等人,《自然生物技术》(2014)33:89-96)。
[0587]
用于ipsc胚状体形成的试剂和设备
[0588][0589][0590]
用于胚状体解离和运动神经元培养的试剂:
[0591][0592]
用于胚状体解离和运动神经元培养的设备
[0593][0594]
慢病毒产生
[0595]
为了测试嵌入mir16-2的向导在干细胞源性运动神经元中的功效,amirna从嵌入在上文描述的慢病毒构建体中的h1启动子表达。使用lipofectamine ltx和plus试剂(赛默飞世尔科技公司,p/n 15338-100),用转染pspax2(cellecta,p/n cpcp-pax2)和pmd2.2(cellecta,cpcp-pm2g)的lenti-x 293t(塔卡拉公司,632180)细胞生成慢病毒。转染培养基改为包含viralboost试剂(alstem公司(alstem),p/n vb100)后的第二天以及之后2天后,使用lenti-x浓缩器(塔卡拉公司,p/n 631232)过滤和浓缩病毒产生培养基,并且重悬于n2b27培养基中。
[0596]
qpcr分析
[0597]
转导干细胞源性运动神经元,并且在转导后7天除去培养基,用pbs洗涤,用补充有β-巯基乙醇的缓冲液rlt裂解细胞。使用qiagen rneasy plus迷你试剂盒(凯杰公司(qiagen),p/n 74134)纯化rna,并且使用superscript vilo cdna合成试剂盒(赛默飞世尔科技公司,p/n 11754250)进行逆转录。使用taqman fast advanced master mix(赛默飞世尔科技公司,p/n 4444556)和quantstudio 6flex实时pcr系统(赛默飞世尔科技公司),使用针对atxn2(赛默飞世尔科技公司,hs01002847_m1)、gusb(赛默飞世尔科技公司,hs00939627_m1)和b2m(赛默飞世尔科技公司,hs00187842_m1)的引物/探针组计算ct值。计算4次重复的平均ct,并且使用δ-δct方法,计算atxn2与每个内部对照的δct,然后计算
未处理条件的平均值的δ-δct。如图所示计算和绘制未处理条件的归一化值的平均值。
[0598]
用表达atxn2 amirna的慢病毒处理的神经元的atxn2水平的蛋白质印迹分析
[0599]
通过将板置于冰上、吸出培养基并添加50微升到100微升冷ripa缓冲液(teknova公司#50-843-016),所述缓冲液补充有蛋白酶和磷酸酶抑制剂片剂(皮尔斯公司#a32959)、halt蛋白酶抑制剂混合物(赛默飞世尔科技公司#1861279)和pmsf(细胞信号传导技术公司#8553s)。使用单独的细胞铲彻底刮擦每个孔,倾斜板并收集裂解物,并且在冰上温育另外30分钟。将样品在4℃下以17,000xg离心15分钟,将上清液转移到新试管中并储存在-80℃下。对蛋白质裂解物进行定量(皮尔斯公司,23225),得到每个样品约40μg的总蛋白质。
[0600]
nupage系统(赛默飞世尔科技公司)用于凝胶电泳。将五μg的每种样品加载到4%到12%的bis-tris蛋白凝胶(赛默飞世尔科技公司,np0321box)上,并且在恒定200v下运行1小时。revert 700(licor公司,926-11010)用于测定蛋白质加载。使用恒定的30v和90ma在4℃下将蛋白质转移到pvdf膜(emd密理博公司(emd millipore),ipfl00005)上过夜。在室温(洛克兰(rockland),mb-070)下将膜封闭1小时。将一级抗体温育在4℃下摇动过夜,包含抗atxn2(1:1000,bd,611378)、抗gfp(1:2000,cst,2956)和β-肌动蛋白(1:2000,cst,4970)。用tbs 0.1%吐温-20洗涤4
×
5分钟,并且将二级抗体在室温下摇动温育1小时(800cw山羊抗小鼠和680rd驴抗兔各1:15,000,licor公司)。再次洗涤膜并在odyssey fc成像系统(licor公司)上进行成像。信号定量由licor image-studio lite进行。
[0601]
实施例4:在aav顺式质粒和aav产生中嵌入来自汇集筛选的最高命中
[0602]
为了测试从汇集筛选中鉴定的靠前性能amirna在嵌入aav时敲低atxn2的能力,将10个靠前mirna克隆到顺式质粒(转移质粒)中的h1启动子(seq id no:1522的核苷酸113-203)下游用于aav产生。质粒序列的实例(5'itr到3'itr)(scaav_amely_v1_h1_micropool_itr_to_itr)包括seq id no:1522的核苷酸序列;其中嵌入在mirna骨架中的期望的amirna插入seq id no:1522的核苷酸204-341中。通过用顺式质粒和辅助质粒三重转染hek293t细胞产生aav9并收集衣壳化的aav后,用quick-dna病毒试剂盒(zymo公司(zymo),p/n d3015)提取载体基因组dna以评估载体完整性。使用qubit dsdna hs测定试剂盒(赛默飞世尔科技公司,p/n q32854)对纯化的载体进行定量,通过琼脂糖凝胶电泳评估载体基因组大小,并且染色sybersafe以进行可视化。通过琼脂糖凝胶电泳评估载体基因组大小(图32)。令人惊讶的是,观察到两条带。上带以预期大小2284bp迁移,而下带迁移的距离比计算的载体大小更远,或者长度小于全长载体。提取表示全长载体的条带,然后用引物对嵌入的aimrna进行桑格测序,结果成功地对预期的amirna进行测序。而较小dna产物的提取和测序未能通过嵌入的aimrna进行测序,这表明下带可能表示以人工mirna为中心的载体截短,如(xi等人,《分子疗法》(2013)28:422-430)中所述。使用mfold(zuker核酸研究(zuker nucleic acids research)(2003)31:3406-15)计算mir16-2骨架中mir的预测的dna次级结构显示此序列形成强次级结构,吉布斯自由结合能(gibbs free binding energy)为-26.78。
[0603]
使用imagej,选择使用sybersafe染色的dna凝胶收集的图像的单个载体基因组泳道,绘制泳道的强度并量化峰。分别使用2284bp和2077bp的全长和以mir为中心的截短的载体基因组的计算长度,计算每个的相对染色强度衍生的摩尔浓度。使用这些值,全长载体百分比计算为全长的百分比除以全长和以mir为中心的截短的载体基因组的组合量(表20)
[0604]
表20:全长载体基因组的百分比
[0605]
h1 amely v1向导全长载体基因组百分比mir16-2-xd-1479268.3mir16-2-147956.7mir16-2-175570.3mir16-2-333062.5mir16-2-440264.1mir16-2-440567.8mir16-2-440671.0mir16-2-440967.2mir16-2-450264.9
[0606]
实施例5:第二汇集
mi
rna筛选
[0607]
鉴于在表达mir16-2嵌入的amirna的aav载体中观察到的截短,设计了第二个汇集amirna筛选以将来自第一个汇集筛选的最高atxn2 mirna命中的向导序列嵌入到一组不同的20个mirna骨架中。
[0608]
ds2的atxn2靶向序列选择
[0609]
从深度筛选1中选择被认为有效和安全的atxn2靶向序列进入“深度筛选2”。优先考虑在低atxn2信号facs箱中富集并表现出低脱落(比较早期和晚期时间点的表示变化最小)的序列。为了校准测定的动态范围,额外包含了一些具有高脱落的序列。由于成熟向导链的加工精度可能存在生物变异性,因此让包含有效向导的向导(通过沿atxn2转录物的位置)另外进入深度筛选2中。
[0610]
必需基因对照mirna选择
[0611]
基于深度筛选1中的性能,选择了一个子集的必需基因靶向amirna用于深度筛选2,所述amirna相对于其它必需基因靶向amirna具有“高”或“中等”脱落。
[0612]
911对照
[0613]
靶向atxn2的序列子集与其同源911对照配对。在911对照中,向导链的碱基9、10和11互补,同时过客链发生对应的变化,这样产生的成熟mirna不会切割原始向导的靶mrna。因为假定amirna“脱靶”活性的许多方面是通过与种子区(碱基2-8)的结合相互作用而发生的,所以这些911对照原则上应显示与原始mirna相似的脱靶特性,并且应有助于区分中靶与脱靶活性。
[0614]
atxn2乱序对照
[0615]
来自深度筛选1的mirna乱序对照的子集被转移到深度筛选2中。这些被认为是平均居中数据。
[0616]
atxn2骨架选择、加工增强基序和过客变化
[0617]
选择微小rna骨架作为人工mirna自然表现出高加工精度、高向导过客比和有效的靶敲低。考虑了功能筛选中的mirna性能和5'向导加工均匀性1–4。
[0618]
在mirbase中鉴定了初级mirna转录物序列。在entrezgene中确定了mirna周围的扩展序列上下文。周围具有高度哺乳动物保守的5'和3'序列用于定义最终的138nt mirna嵌入片段,所述片段将被插入集合库中。
[0619]
mfold和rnafold用于检查折叠模式并考虑吉布斯自由能,因为有证据表明,源自
mirna中广泛的次级结构的高吉布斯自由能可能在后来克隆并产生aav时产生以mir为中心的截短。
[0620]
基底茎、环、向导和过客序列通过对mirbase和mfold的茎环折叠预测来定义。选择如凸起和其它不对称等过客变化的规则来模拟内源性发夹茎中的非互补碱基配对,并将其并入库构建算法中。
[0621]
先前已经确定了能够有效加工pri-mirna骨架的序列基序。这些包含mirna前体5'端处的ug基序、茎中错配的ghg基序和3'cnnc基序。选择的许多初级mirna转录物天然含有这些基序。其中一些基序被人工并入五个骨架中,这些产生的mirna骨架用“_m”表示(例如,“mir-1-1_m”)。表21提供了深度筛选2中使用的mirna骨架序列(dna形式)。通过将表21的序列中的“t”核苷酸转化为“u”核苷酸来提供mirna骨架的rna序列。
[0622]
表21:深度筛选2中使用的mirna骨架序列
[0623]
[0624]
16-2骨架中的mir的丰度通常大大低于其它骨架。一种可能的解释是,在库扩增期间(当所有库元件都进行pcr扩增时)包含mir-16-2骨架的元件的扩增效率低于其它骨架。这可能是因为与mir-16-2骨架形成的强dna发夹。由于库中剩余的mir-16-2骨架元件数量较少,含有mir-16-2的向导的计数很低且因此很嘈杂,并且不包含在进一步分析中。
[0627]
在克隆、包装和筛选执行后(参见方法),测序数据的分析基本上与深度筛选1相同。库元件的丰度是通过与输入库元件完全匹配的测序读段来计算的。在此筛选中,没有针对atxn2水平或脱落进行基线减法。图33a示出了绘制atxn2敲低指标中两个筛选重复彼此之间的对应关系的散点图。在此情况下,绘制的是10%低atxn2信号分选箱与未分选的分选箱中向导元件的序列读段丰度的比率。对于未分选的/10%低atxn2信号比率较低的元件(即,导致atxn2耗竭的元素)具有良好的对应关系,但未分选的和10%未分选的箱中具有相似丰度的元件的对应性较低。
[0628]
图33b示出了嵌入所示骨架中的mir的敲低性能的箱形图;表22示出了性能的中值和第95个百分位。通过此指标,一些靠前性能的mir(按中值性能mirna衡量)是mir-1-1_m、mir1-1、mir-130a、mir-100和mir-100_m。然而,值得注意的是,在这些骨架中的每一个中都有靠前mirna,如通过此测定测量的(低atxn2分选池中的向导计数与未分选细胞中的向导计数的比率),在mir骨架中的性能相似。因此,此测定提供了具有强大性能的多个mir骨架。这可能是由于微处理器和dicer复合物对人工pri-mirna的良好加工。
[0629]
表22:跨mirna骨架的mirna性能
[0630] mir_具有_后缀中值九十_五1mir-1-1_m-0.6772802-2.27889372mir-1-1-0.5968477-2.18757223mir-130a-0.5111669-2.1575254mir-100-0.2912209-2.08870755mir-100_m-0.2094112-1.97587316mir-155e-0.1742495-1.88185287mir-132-0.1144013-2.02045878mir-190a-0.030798-2.08130079mir-190a_m0.04198442-2.125090410mir-1220.05806267-1.903947311mir-122_m0.17311752-1.85100912mir-155m0.50609504-1.1119213mir-124_m0.55091372-0.901604214mir-1240.57242321-0.9507815mir-1440.71869783-1.337283616mir-138-20.77876566-0.866666417mir-2231.047431760.46242634
[0631]
靶向必需基因的元件的耗竭也被用作mir骨架性能的正交评估。图34示出了18天时间点与转导后1天时间点的元件耗竭的箱形图。与atxn2敲低指标相比,此指标对各种mir骨架的“性能”排名相似。这可能是因为mir骨架在加工过程中产生成熟amirna的排序。
[0632]
表23列出了前100个amirna,按atxn2低信号分选细胞中的平均富集度排名。以rna和dna形式提供mir骨架、向导序列、互补atxn2转录物序列内的靶向位置、过客序列和amirna序列(包含mir骨架、环、atxn2靶向向导和过客)。“过客”序列是指与向导序列互补的序列,但包含根据表8中所述的规则设计的凸起和错配以模拟内源性mirna结构。应注意,在加工pri-mirna后,过客链可能会在表中所示核苷酸的下游1nt到3nt处开始,并包含源自mir盒的最后一个列出的核苷酸之外的1nt到3nt。表24列出了每个mir骨架的前10个amirna,不包含低性能骨架。靠前amirna通过atxn2低信号分选细胞中给定amir构建体的序列计数的平均富集度进行排名。以rna和dna形式提供mir骨架、向导序列、互补atxn2序列内的靶向位置、过客序列和amirna序列。
[0633]
表23:前100个amirna
[0634]
[0635]
[0636]
[0637]
[0638]
[0639]
[0640]
[0641]
[0642]
[0643]
[0644]
[0645]
[0646]
[0647]
[0648]
[0649]
[0650]
[0651]
[0652]
[0653]
[0654]
[0655]
[0656]
[0657]
[0658]
[0659]
[0660]
[0661]
[0662]
[0663]
[0664]
[0665]
[0666]
[0667]
[0668]
[0669][0670]
表24:每个mir骨架的前10个mirna
[0671]
[0672]
[0673]
[0674]
[0675]
[0676]
[0677]
[0678]
[0679]
[0680]
[0681]
[0682]
[0683]
[0684]
[0685]
[0686]
[0687]
[0688]
[0689]
[0690]
[0691]
[0692]
[0693]
[0694]
[0695]
[0696]
[0697]
[0698]
[0699]
[0700]
[0701]
[0702]
[0703]
[0704]
[0705]
[0706]
[0707]
[0708]
[0709]
[0710]
[0711]
[0712]
[0713]
[0714]
[0715]
[0716]
[0717]
[0718]
[0719]
[0720]
[0721][0722]
方法
[0723]
寡核苷酸池设计和合成:
[0724]
总共设计了7500个210bp长度的元件用于合成,大约均匀地分布在20个mirna骨架上。mir-1-1、mir-155和mir-16-2骨架中有更多元件,因为已在阵列实验中测试过的元件也包含在此筛选中。atxn2靶向序列约占库的60%。
[0725]
每个元件包含138nt pri-mirna,侧接双18nt衔接子对。外部衔接子对是mir特异性的,内部衔接子对是通用的。
[0726]
完整ds2库克隆策略
[0727]
合成寡核苷酸池(twist生物科学(twist bioscience))并在无核酸酶水中重构。为了将ef1a寡核苷酸池克隆到plvx_ef1a-mcs-wpre-cmv-puro,首先通过xbai和ecori限制性消化线性化载体并进行凝胶纯化。引物ds2_ef1a_fw和ds2_ef1a_rv用于通过10个pcr循环扩增寡核苷酸池并进行纯化。纯化的汇集插入物和纯化的线性化载体与neb hifi组合件组装、沉淀、浓缩并电穿孔到lucigen endura电感受态细胞中,回收并进行大量制备。在以下条件下对寡核苷酸池进行pcr扩增。
[0728]
pcr混合物由以下组成:
[0729]
组分体积(ul)nebnext 2x混合物(m0541l)50dmso(d9170-5vl)2betaine(西格玛公司(sigma),b0300-1vl)10100um fw引物(ds2_ef1a_fw)0.5100um rv引物(ds2_ef1a_rv)0.5无核酸酶水中的1ng ef1a寡核苷酸池37总计100
[0730]
pcr循环参数为:
[0731][0732]
通过琼脂糖凝胶提取(zymoclean凝胶dna回收试剂盒,d4002)纯化210bp长度的pcr产物。agilent tapestation high sensitivity d1000用于量化210bp峰的摩尔浓度并确认去除了污染带。
[0733]
通过以5比1的插入物与骨架摩尔比进行组装来进行集合库的hifi组装。15ul的2xhifi组装主混合物(neb公司,e2621l)和15ul插入物和骨架(约0.375pmol纯化的mir库插入物到0.075pmol纯化的骨架)并且在50℃下温育1小时。
[0734]
通过添加1ul的20mg/ml糖原、十分之一体积的3m醋酸钠(ph 5.5)和2.2x体积的乙醇沉淀组装的dna,混合并在-80℃下储存过夜。
[0735]
样品在4℃下以16,000xg旋转15分钟。去除并丢弃上清液。用1ml的70%冰冷乙醇洗涤沉淀两次并使其干燥,然后溶解在4ul无核酸酶的水中。
[0736]
将纯化的dna和0.1cm基因脉冲比色皿(伯乐公司,165-2089)置于冰上10分钟。50ul lucigen endura电感受态细胞在冰上短暂解冻。将4ul沉淀的hifi反应添加到25ul电感受态细胞中,混合并转移到比色皿中。使用以下参数对dna和细胞混合物进行电穿孔:1800伏、10uf、600欧、0.1cm比色皿。立即用1ml lucigen恢复培养基冲洗比色皿两次。在37℃下以230rpm回收细胞1小时。
[0737]
为了滴定转化的细菌,将2ul的每种培养物稀释到200ul的lb中,并将100ul或10ul这种培养物以1:100的稀释度铺板到lb琼脂平板上,加适当的抗生素。第二天计数菌落数量以确定转化体的总数。
[0738]
将液体培养物接种到适量的具有抗生素的lb中,用于最大制备。按照制造商的说明,使用qiagen plasmid maxi试剂盒制备集合质粒库。
[0739]
集合ef1a库的制备和滴定
[0740]
慢病毒是在lenti-x 293t细胞中使用takara包装质粒系统产生的。在感染和嘌呤霉素选择3天后通过cell titer glo确定功能滴度,以确定实现moi=0.1的条件。
[0741]
atxn2水平和脱落的完整库筛选的执行
[0742]
与ds1类似地进行并发atxn2水平和脱落筛选。u2os细胞在第0天被慢病毒汇集的ef1a库以2000x的覆盖率和moi=0.1感染。
[0743]
对于脱落筛选,在第1天收集了t0基线样品。在第2天添加嘌呤霉素,并在第5天通过铺板细胞进行cell titer glo滴度评估来确认moi。第7天后,去除嘌呤霉素并将细胞以最少2000万个细胞传代到第18天,然后收集t1最终细胞群。
[0744]
对于atxn2蛋白水平筛选,在第7天收集细胞并在室温下在6%蔗糖/8%pfa中固定10分钟到15分钟,以600xg离心3分钟,使用透化缓冲液(e生物科学公司(ebioscience),00-5523-00)洗涤三次,与洗涤缓冲液混合并在室温下温育15分钟到20分钟。将抗atxn2一级抗
体(1:200,bd,611378)在室温下温育30分钟到60分钟。将细胞洗涤三次,添加af647次级(1:200,百进生物公司(biolegend),405322)并温育45分钟。洗涤三次后,将细胞重悬于facs缓冲液中并在bd facsaria fusion上分选。在为单峰门控后,绘制了25%高和低atxn2门,通过在apc/ssc比率门上分选来调整细胞大小。一旦为25%高和低分选门收集了300万个到350万个细胞,剩余的细胞在10%的低门(收集了100万个细胞)上分选以进一步丰富高性能向导。通过对单峰进行分选来收集参考群。
[0745]
固定的分选细胞群用1%sds/1%碳酸氢钠解交联,并且在65c下温育过夜。使用machery nagel nucleospin l试剂盒提取基因组dna,并且进行嵌套式pcr以制备测序库。
[0746]
测序库制备
[0747]
进行嵌套pcr以产生适应illumina的测序扩增子。对从每个细胞沉淀中提取的所有基因组dna进行第一pcr反应。使用下列条件,每100ul pcr反应最多使用5ug基因组dna。
[0748][0749][0750]
在pcr之后,来自给定样品的所有反应都合并到单个管中。
[0751]
使用0.5x和0.9x双面spri珠比率对564bp预期大小的第一pcr产物进行珠纯化。具体地,将25ul spriselect(贝克曼公司(beckman),b23318)添加到50ul第一pcr产物中,通过移液充分混合,并且在室温下温育10分钟。将样品放在磁性支架上5分钟。将上清液转移到新管中。将45ul spriselect添加到转移的上清液中,通过移液充分混合,并且在室温下温育10分钟。将样品放在磁性支架上5分钟。然后去除上清液。用1ml新鲜的80%乙醇在2分钟的温育过程中将珠洗涤两次。将具有结合的dna的珠风干5分钟到10分钟并用来自machery nagel试剂盒的20ul洗脱缓冲液洗脱。
[0752]
执行第二pcr以添加具有以下条件的样品条形码和illumina衔接子:
[0753][0754]
使用0.7x和1.2x双面spri珠比率对具有300bp预期大小的第二pcr产物进行珠纯化。具体地,将35ul spriselect(贝克曼公司,b23318)添加到50ul第一pcr产物中,通过移液充分混合,并且在室温下温育10分钟。将样品放在磁性支架上5分钟。将上清液转移到新管中。将60ul spriselect添加到转移的上清液中,通过移液充分混合,并且在室温下温育10分钟。将样品放在磁性支架上5分钟。然后去除上清液。用1ml新鲜的80%乙醇在2分钟的温育过程中将珠洗涤两次。将具有结合的dna的珠风干5分钟到10分钟并用来自machery nagel试剂盒的20ul洗脱缓冲液洗脱。
[0755]
通过tapestation high sensitivity d1000(安捷伦公司)对最终珠纯化的第2pcr产物进行量化,并以等摩尔比进行多路复用,以便以在miseq(依诺米那公司)上进行测序。使用制造商的方案,将15pm库变性并与2%phix对照混合。ds2-ef1a-read1引物被掺入miseq v3筒(依诺米那公司)的位置12中。读段1设置为139个循环,索引读段设置为6个循环。
[0756]
使用fastq生成模块对数据进行解复用并进行分析。
[0757]
引物
128骨架嵌入的mir具有更均匀的凝胶模式。为了更普遍地评估含有不同mir骨架的aav的载体完整性,使用顺式质粒库,每个库都含有完整的atxn2靶向amirna向导序列,与深度筛选2中相同,用于如前所述包装aav。此实验中使用的寡核苷酸扩增策略不区分mir骨架的亲本和“_m”形式,其最初都存在于深度筛选2库中,因此库包含例如含有元件的mir-100和mir-100_m骨架;mir-1-1和mir-1-1_m骨架。图36示出与含有mir-100骨架和特定向导序列4402(seq id no:751(dna);seq id no:1279(rna))的aav一样,源自嵌入mir-100和mir-100_m骨架中的mir库的aav显示出比具有其它mir骨架的aav更均匀的凝胶电泳模式。尽管在包装后未评估顺式质粒库的具体组成,并确认其在具有不同mir骨架的库中是一致的,但对此数据的最简单解释是,平均而言,在一系列特定mir中,具有mir-100骨架的aav载体基因组比其它骨架表现出更均匀的全长大小。
[0761]
基于良好的敲低性能和良好aav载体基因组均匀性的组合特性,mir-100和稍加修饰的mir-100_m被优先考虑作为用于推进的骨架。通过将质粒库转染到hek293t细胞中并收集小rna,对包括插入未包装的aav顺式质粒scaav_amely_v1_h1(seq id no:1522;amirna插入物位于核苷酸204-341处)中的amirna的“微池”质粒库进行了测试。如上所述,构建质粒库的寡核苷酸扩增策略没有区分mir100与mir100_m骨架,因此库表示两者的混合;然而,鉴于来自亲本和_m形式骨架的mir的总体性能相似,库中骨架的混合不太可能降低检测精确加工的mirna的整体能力。整合这些小rnaseq数据以评估库中单个amirna的加工精度,如以下实施例所述。
[0762]
方法
[0763]
aav微池克隆
[0764]
为了将微池克隆到scaav_amely_v1_h1骨架中(以产生如seq id no:1522中所述的质粒),首先通过aari消化克隆位点区域并纯化琼脂糖凝胶将骨架线性化。
[0765]
使用以下条件扩增微池,使用mir-1-1作为实例。下表列出了所有mirna骨架特异性引物对。
[0766][0767]
在pcr产物上使用0.7x spri珠和1.2x spri珠比率的双面珠纯化,所述产物又用作hifi组装中的插入物。
[0768]
通过以5比1的插入物与骨架摩尔比进行组装来进行集合库的hifi组装。15ul的2xhifi组装主混合物(neb公司,e2621l)和15ul插入物和骨架(约0.375pmol纯化的mir库插入物到0.075pmol纯化的骨架)并且在50℃下温育1小时。
[0769]
通过添加1ul的20mg/ml糖原、十分之一体积的3m醋酸钠(ph 5.5)和2.2x体积的乙醇沉淀组装的dna,混合并在-80℃下储存过夜。
[0770]
样品在4℃下以16,000xg旋转15分钟。去除并丢弃上清液。用1ml的70%冰冷乙醇洗涤沉淀两次并使其干燥,然后溶解在4ul无核酸酶的水中。
[0771]
将纯化的dna和0.1cm基因脉冲比色皿(伯乐公司,165-2089)置于冰上10分钟。50ul lucigen endura电感受态细胞在冰上短暂解冻。将4ul沉淀的hifi反应添加到25ul电感受态细胞中,混合并转移到比色皿中。使用以下参数对dna和细胞混合物进行电穿孔:1800伏、10uf、600欧、0.1cm比色皿。立即用1ml lucigen恢复培养基冲洗比色皿2x。在37℃下以230rpm回收细胞1小时。
[0772]
为了滴定转化的细菌,将2ul的每种培养物稀释到200ul的lb中,并将100ul和10ul这种培养物以1:100稀释度铺板到lb琼脂平板上,加适当的抗生素。第二天计数菌落数量以确定转化体的总数。
[0773]
将液体培养物接种到适量的具有抗生素的lb中,用于最大制备。按照制造商的说明,使用qiagen plasmid maxi试剂盒制备集合质粒库。
[0774]
引物
[0775]
[0776][0777]
汇集aav产生
[0778]
aav微池用作顺式质粒,在载体生物实验室使用标准三质粒aav包装方法与ad辅助和aav9 repcap一起包装。
[0779]
粗裂解物加工和凝胶可视化
[0780]
为了提取载体基因组,粗裂解物经过4次冻融循环(37℃和干冰-乙醇浴)并通过0.45um过滤器(chemglass公司(chemglass),cls-2005-017)。在37℃下,用2ul dna酶1(neb公司,m0303l)和0.2ul rna酶a(赛默飞世尔科技公司,en0531)处理每100ul的通过30分钟。用quick viral dna试剂盒(zymo公司,d3015)提取载体基因组。使用具有sybrsafe或sybrgold染色dna的1.5%琼脂糖凝胶进行可视化。
[0781]
小rnaseq微池的汇集表达
[0782]
在6孔板中生长的细胞中,使用基于脂质的方法(lipofectamine ltx,赛默飞世尔科技公司)将嵌入质粒scaav_amely_v1_h1中的mir100和mir100_m骨架mir的微池转染到hek293细胞中。每孔接种600,000个细胞,并且在次日一式两份转染,每孔转染2.5微克微池库。第2天更换培养基,第3天在trizol中收集。总rna通过氯仿相分离提取,并且使用制造商的方案通过zymo direct-zol柱洗脱纯化。
[0783]
小rnaseq
[0784]
使用nextflex v3小rna测序试剂盒(bioo scientific公司(bioo scientific corp),nova-5132-05)制备small rnaseq库。简而言之,库制备以0.5ug到2ug rna输入开始。对每个样品进行14个到18个循环的pcr。在基于tapestation high sensitivity d1000定量150bp带汇集样品之前,进行了两轮双面珠清理。illumina适应库被多路复用并加载到miseq(依诺米那公司)上,在miseq v3试剂盒上以10%phix在9pm下加载库,读段1设置为75个循环,索引设置为6个循环。
[0785]
实施例7:嵌入
mi
r-100和
mi
r-100_m骨架中的靠前人工
mi
rna排名
[0786]
嵌入mir-100和mir-100_m骨架的靠前amirna在深度筛选2中按敲低性能排名;按向导与过客比率排名;并且按在晚期(t1,18天)与早期(t0)时间点(脱落)的最小耗竭排名。(应注意,如上所述,向导:过客比率来自小rnaseq库,所述库包含mir100和mir100_m骨架的混合物)。另外,对每个排名候选物评估了一组具有1bp或2bp错配的潜在脱靶转录物。在排除具有低向导:过客比率、低t1/t0比率的候选物以及具有仅1bp错配的接近互补性的cns表达转录物的候选物后,将一组9个活性mirna和2个911对照mirna克隆到h1启动子的顺式质粒下游中,并且与编码aav-dj衣壳组分的rep/cap辅助质粒一起包装。表25中列出了来自深度筛选2(图37)和这些候选物的小rnaseq分析的数据。对于这些选择的命中,重复1和重复2t1/t
0 log2比率的平均值均在mir100和靶向atxn2的mir100_m amirna的中值(-0.07)log2比率的1个标准偏差(0.22)之内。
[0787]
表25:来自深度筛选2和小rnaseq分析的数据
[0788][0789]
在干细胞源性运动神经元中,针对1755(向导序列seq id no:1185)和2945(向导序列seq id no:1213)的上述mirna以及911对照测试了atxn2的敲低。amirna包装在顺式质粒中以生成含有长h1启动子(seq id no:2257的核苷酸113-343)和填充序列“psg11_v5”(seq id no:2257的核苷酸489-2185)的自互补的aav-dj载体。编码从5'itr到3'itr的amirnamir100_1755(seq id no:1915)、mir100_2586(seq id no:1982)、mir100_2945(seq id no:1965)和mir100_3330(seq id no:2021)序列分别在seq id no:2257、seq id no:2258、seq id no:2259和seq id no:2260中提供。在对每个载体进行滴定后,并且基于基于血细胞计数器的铺板细胞数量的量化,以每个细胞3.16e3个和3.16e4个载体基因组的预期剂量添加载体。添加载体7天后,收集神经元并用mirneasy组织/细胞高级迷你试剂盒(凯杰公司,p/n 217604)分离rna,通过数字液滴rt-pcr评估atxn2敲低,测量atxn2表达与管家对照gusb和b2m的比率。
[0790]
图38示出了单独的数据点,表26示出了在3.16e3 vg/细胞和3.16e4 vg/细胞的两个剂量下,这些构建体的敲低的平均值和标准偏差,相对于来自未转导细胞的atxn2表达值
归一化,所述未转导的细胞用等效体积的aav稀释剂处理。
[0791]
表26:干细胞源性运动神经元中amirna在两种不同剂量下的atxn2敲低
[0792]
mir平均_atxn2_3160sdn平均_atxn2_31600sdnmir100_175526.81.7614.02.16mir100_258641.82.9629.31.06mir100_294536.71.7626.82.66mir100_327072.15.1647.92.26mir100_333044.16.3632.22.76mir100_333836.53.6623.65.56mir100_m_313330.61.0523.81.56mir100_m_330183.79.1649.13.76mir100_m_330238.03.0632.33.36未转导的1009.512
ꢀꢀꢀ
[0793]
剂量应答研究
[0794]
候选aav还在运动神经元中以更广泛的剂量范围进行了测试。如前所述,在培养7天后从培养物中分离rna,并且评估atxn2敲低。图39示出了添加的每种载体的不同浓度的敲低图。通过数字液滴rt-ddpcr测量用媒剂(pbs .001%pf-68)处理的神经元中atxn2 mrna的浓度,通过b2m表达对每个数据点进行归一化,并整体达到atxn2表达水平。通过检查,可以观察到amirna效力的差异;例如与其它amir相比,mir100_1755在较低的载体基因组暴露下表现出敲低;相对于其它载体,mir100_3301和mir100_3270似乎表现出降低的效力。
[0795]
以每个细胞3.16e3个载体基因组给药的神经元额外经过了小rna测序。表27示出了呈占总mirna的分数形式的amirna的丰度。表达水平的范围令人惊讶,并且若干amir(1755、2586、2945和3270)检测到的amirna比其它amirna少得多。
[0796]
对于这些小rna实验,读段并不像在深度筛选1库的小rna分析中那样“去重复”(通过消除具有相同侧接5'和3'4-mer随机衔接子的读段),因为在一些情况下,人工mirna的读段数量接近衔接子中核苷酸的独特组合数量。
[0797]
表27:呈占总mirna的分数形式的amirna的丰度
[0798]
向导重复1amirna/总mirna(%)重复2amirna/总mirna(%)mir100_17551.802.77mir100_25862.532.73mir100_29453.203.30mir100_32701.211.43mir100_333016.8317.75mir100_333823.8425.00mir100m_313322.8222.57mir100m_330122.0717.53mir100m_330238.0436.78
[0799]
为了评估aav amirna处理是否对神经元形态或细胞计数有任何明显影响,以1e4
载体基因组/细胞的剂量用aav或媒剂处理以96孔形式生长的神经元,7天后用hoechst固定和染色,抗isl1和抗β3微管蛋白抗体分别用于可视化核、运动神经元标志物和神经元突起。图40示出了来自用指定的amirnaaav和对照处理的培养物的代表性图像,证明没有aav mirna对神经元形态表现出明显影响。图41a示出了比较mir100_1755与mir100_1755_911(911对照,通过1755amirna的互补碱基9、10和11而对切割atxn2无活性)的放大图像。可以看出没有明显差异,这表明atxn2敲低不会导致神经元突起或核形态发生显著变化。右图量化了hoechst 核的总数(图41b)和isl1 的总核的百分比(图41c)。与媒剂处理的(pbs .001%pf-68)孔相比,在少数aav-amirna处理中观察到显著差异(p《.05),每个区域的核总数减少。然而,其这些处理的其中一种(mir100_1755)也显示出isl1 细胞分数显著增加,而对于其他aav-dj amirna,isl 神经元明显增加,这与运动神经元总数的任何变化相矛盾。用任何活性aav amirna和非活性911对照aav amirna转导的神经元之间没有显著差异。
[0800]
rnaseq研究
[0801]
在用上述aav的1e4载体基因组/细胞给药后7天,从运动神经元收集rna。每个条件有6个重复,除了mir100_1755_911,其有5个。为了确定来自aav表达的atxn2敲低是否影响神经元中的转录组,比较了用表达活性amirna的载体和表达同源911对照的载体转导的神经元之间的rna表达。图42示出了mir100_1755与mir100_1755_911和mir100_2945以及mir100_2945_911的差异表达的“火山图”。在为atxn2与所有其它基因计算的差异表达的标称p值中可以看到很大的分离。值得注意的是,在使用benjamini-hochberg程序调整多重比较的标称p值后,只有atxn2或一个其它基因分别超过了1755和2945的10%错误发现率阈值。
[0802]
为了进一步研究是否对任何预测的脱靶基因(与每个amirna的碱基2-18有2个或更少错配的转录物的集合)有任何影响,将每个amirna与来自所有其它活性amirna的数据进行比较(图43)。对于此组选择的amirna,很少有预测的脱靶超过10%的错误发现率阈值。这表明这些amirna产生了atxn2的特异性敲低。
[0803]
方法
[0804]
ddpcr aav滴定
[0805]
为了滴定aav,将每个载体在鲑鱼精子dna溶液(20ng/ul鲑鱼精子dna、0.001%pf-68、10mm tris-hcl ph 7.5、50mm kcl、1.5mm mgcl2)中连续稀释,然后在95℃下加热10分钟以从aav9衣壳中释放载体基因组。在与smai温育以减少次级结构(已知会抑制raav pcr反应)(neb公司,r0141l)后,使用dg32自动液滴发生器((伯乐公司)生成液滴,然后使用载体特异性引物/探针组进行pcr扩增。完成后,使用qx200液滴数字pcr系统(伯乐公司)分析液滴并定义阳性和阴性群体,并且应用稀释因子来确定未稀释载体原液的浓度。
[0806]
运动神经元免疫细胞化学
[0807]
运动神经元培养物在室温下在4%多聚甲醛中固定10分钟。固定的培养物在室温下在含有0.2%triton-x-100和10%驴血清溶液的pbs中渗透和封闭45分钟。然后将细胞在含有一级抗体的封闭溶液(0.1%吐温-pbs中的10%驴血清)中在4c下温育过夜。细胞用pbs-0.1%吐温洗涤3次,然后在室温下在含有二级抗体的封闭溶液中温育1小时到2小时,然后用pbs-0.1%吐温洗涤3次,并且用含有dapi的pbs溶液冲洗。在perkin elmer operetta高含量成像仪上使用20x水物镜对染色的培养物进行成像。每个孔成像40个到60
个区域。使用perkin elmer harmony软件进行细胞定量。使用graphpad prism软件进行统计分析。使用的一级抗体:tuj1(1:500稀释)isl1(1:200稀释)二级抗体:alexafluor 488和alexfluor647(1:500稀释)。
[0808][0809]
脱靶预测
[0810]
为了生成一组预测的脱靶,使用bowtie命令将amirna的碱基2
–
18与人类转录组比对:
[0811]
bowtie-n 2-l 17-e 81-seed[用于强制再现性的伪随机数]
–
nomaqround
–
tryhard
–
nomaqround
–
tryhard
–
chunkmbs 256
–‑
all
–‑
time(以及用于输入/输出处理的额外的命令)。为了确保仅发生2个或更少的错配,构建了含有要测试的amirna的bowtie比对的fastq文件输入,其中每个amirna被赋予iiiiiiiiiiiiiiiii的phred评分“掩模”,这样amirna与转录物的比对中不匹配发生超过2次将超过权重阈值。比对amirna以构建homo_sapiens.grch38.cdna.all、macaca_fascicularis.macaca_fascicularis_5.0.cdna.all或mus_musculus.grcm38.cdna.all。
[0812]
rnaseq
[0813]
干细胞源性运动神经元培养物以6孔板每孔200,000个细胞的密度铺板。铺板后6天,以每个细胞10,000个载体基因组(通过上文描述的滴定方法计算)的剂量用aav载体转导细胞。7天后,收集细胞用于rna。通过添加300ul buffer rlt plus进行转导样品的裂解,然后在-80下冷冻过夜。样品在冰上解冻并根据rneasy plus标准方案的其余部分进行加工。(qiagen rneasy plus微试剂盒((目录74034)),根据制造商的说明。所有纯化的rna样品均通过qubit(使用rnahs标准)进行量化。通过tapestation(高灵敏度rna)进一步表征选择的具有低、中和高rna浓度(总共16个)的样品以检查纯度(rine评分)并验证qubit量化。所有rine评分都在9.9到10范围内,接近最大值。
[0814]
然后将纯化的rna用作quantseq[lexogen目录#015(quantseq 3
‘
illumina的mrna-seq库制备试剂盒(fwd)]的输入。每个反应的靶rna输入为100ng(对于较低浓度的样品,使用5ul的最大输入体积)。遵循标准quantseq方案并进行以下修改:(1)使用“umi第二链合成模块”(lexogen目录号081)在步骤7中添加umi。(2)库扩增20个循环。通过qubit(dnahs)对所得的库进行量化,并且在tapestation(hs d5000)上进行qc抽查。基于qubit量化对库进行汇集,并在illumin novaseq(seqmatic公司(seqmatic))上进行测序。测序参数如下:novaseq s1运行,单个读段100bp,单个索引6bp。
[0815]
rnaseq分析
[0816]
为了分析rnaseq数据,使用seqtk将从每个样品中获得的每个单端读段拆分为分别含有umi和读段序列的fastq文件:
[0817]
seqtk trimfq-b 10raw.fastq》sequence.fastq
[0818]
seqtk trimfq-e[读段大小-6]raw.fastq》umi.fastq
[0819]
然后以批处理模式将所得序列与kallsito版本0.46.0(bray等人,《自然生物技术》201634:525-527)与转录组组装伪比对,所述转录组组装源自ensembl 96版(kmer长度=19)中存在的所有cdna的尾部600bp:
[0820]
kallisto pseudo
‑‑
umi
‑‑
quant
‑‑
single-t 8-i[kallisto_index]-o[output_file]-b[batch_file.txt]
[0821]
将注释到每个基因的所有转录物中的比对读段求和以生成基因水平计数矩阵。在下游分析中考虑了在至少一种实验条件的所有重复中观察到的具有五个或更多计数的基因。在通过limma::voom进行概率权重估计之前,将样品读段计数转换为碱基-2-log(cpm)并通过tmm(edger::calcnormfactors)进行归一化。(law等人,《基因组生物学(genome biology)》(2014)15:r29)。通过在归一化的表达值上拟合基因方式线性模型来量化差异表达的证据,从模型系数中提取倍数变化,并且使用沃尔德检验(wald test)估计相关的p-值。使用fdr方法对基因方式p值进行了多次测试校正。
[0822]
实施例8:野生型小鼠中候选
ami
rna的体内测试。
[0823]
对嵌入自互补的aav9载体中的amirna的体内性能进行了两项另外的研究。在第一项研究中,分别对mir1-1或mir100骨架中的amirna 1784和3330在含有不同启动子和填充的各种载体基因组中进行了测试。表28中提供了用于体内测试的特异性mir盒。
[0824]
表28:体内使用的特异性mir盒
[0825][0826]
载体设计,包含特异性启动子和填充将单独描述。在此,在若干种整体载体形式和启动子中比较了amirna的性能。向野生型小鼠静脉内给药aav(剂量:3.21e9 vg/克小鼠)或纹状体内注射(剂量:总共7.5e9 vg)。
[0827]
表29示出了相对于用媒剂给药的动物(具有0.001%pf-68的pbs),静脉内给药后3周在肝脏中评估的平均atxn2敲低。通过数字液滴rt-pcr评估atxn2表达,并且将敲低作为通过ddpcr测量的atxn2/hprt和atxn2/gusb比率的平均值。
[0828]
表29:i.v.amirna给药后肝脏中的atxn2敲低
[0829]
[0830]
对于纹状体样品,在收集穿孔活检后的载体生物分布因样品而异。通过数字液滴pcr评估载体分布,测量扩增识别aav载体基因组的引物/探针组与识别小鼠基因组中tert基因的引物/探针组的液滴的相关数量。由于每个细胞(2)中存在固定数量的tert基因拷贝,因此可以通过这种方式测量每个细胞(二倍体基因组)的载体基因组数量。通过评估与atxn2 mrna相同的活检组织中的aav载体分布,可以看到明显的剂量应答趋势(图44、45a至45b)。应当注意,核载体基因组相对于细胞质或细胞外载体的量没有被评估,如通过组织学方法;脑实质内注射引入的载体可能会在细胞外积累。尽管如此,明确的剂量应答表明,即使不是所有测量的载体基因组都在细胞核中,可用于表达amirna,任何此类总载体基因组暴露与功能活跃的载体基因组之间存在明显的相关性。
[0831]
为了确定amirna表达与敲低之间的关系,amirna以两种方式量化。首先,使用taqman advanced mirna cdna合成试剂盒(赛默飞世尔科技公司,p/n a28007)为所有纹状体穿孔活检样品生成库,使用的rna是用富含小rna的试剂盒(凯杰公司,p/n 217604)分离的。要为taqman qpcr生成cdna库,首先完成3'poly-a加尾,然后进行5'连接以添加衔接子。逆转录后,对cdna进行14个循环的pcr扩增,然后使用对外源性和内源性mirna特异的引物探针组对最终扩增产物的稀释液进行qpcr。使用了靶向外源性amirna设计的引物/探针组(赛默飞世尔科技公司),以及靶向内源性mirna mir-21a-5p(赛默飞世尔科技公司,p/n mmu482709_mir)和mir-124-3p(赛默飞世尔科技公司,p/n mmu480901_mir)的引物/探针组作为对照。mirna的丰度通过靶扩增发生的qpcr循环数来评估。比较靶向不同mirna的引物/探针之间发生扩增(ct)的qpcr循环,可以评估mirna的相对丰度。
[0832]
图45a至45b绘制了amirna与内源性对照之间的ct值差异,以及两种内源性mirna(mir-21和mir-124)之间的差异,以及相同样品中的载体生物分布。可以看出,随着aav载体基因组检测的增加,内源性mirna之间的ct阈值差异没有明显变化。相比之下,amirna与内源性mirna之间ct分离的预期增加与载体暴露之间似乎存在对数线性关系,这与aav暴露增加时amirna表达增加一致。
[0833]
对于样品的子集,另外进行了小rnaseq。如上所述,对每个样品量化通过总mirna表达归一化的amirna表达。由于这些样品amirna表达是通过小rnaseq和qpcr进行量化的,因此可以建立模型来确定qpcr如何预测amirna表达随总mirna而进行的变化。因此,拟合了线性模型(图46),很好地解释了两种amirna的方差(r2》.89)。
[0834]
使用此模型,qpcr评估的所有样品中mir100_3330和mir1.1.1784的amirna表达值可以转换为amirna/总mirna的绝对标度。将纹状体活检中的atxn2 mrna与此预测的amirna表达指标作图,与表达mir1.1.1784amirna的样品相比,在表达mir100-3330amirna的样品中表达的每个mirna的敲低相当多(图47)。因此,虽然随给药的载体而变化,表达mir1.1.1784的载体诱导了更多的敲低,但随表达的amirna而变化,mir100.3330诱导了更多的敲低。这表明mir100.3330在体内pri-mirna加工的大约22个核苷酸的最终产物的效力高于mir1.1.1784。
[0835]
在第二项研究中,将表达amirna mir100_1755(seq id no:1915)、mir100_2945(seq id no:1965)、mir100_3330(seq id no:2021)和mir100_2586(seq id no:1982)的自互补的载体包装在具有如上所述的顺式质粒的aav9中,所述质粒含有填充序列“psg11_v5”(seq id no:2257的核苷酸489-2185)、长h1启动子(seq id no:2257的核苷酸113-343)并
cell)》(2019)75:741-755),因此这些amir的这种3'修饰可能有助于atxn2的高敲低性能。
[0844]
表31:在位置 0或 1处起始的amirna的比例
[0845]
amirna在 0处切割的比例在 1处切割的比例mir100_175597.671.07mir100_258698.510.99mir100_294598.340.94mir100_333097.082.07
[0846]
表32量化了向导与过客链读段的比率。对于所有这些mir100骨架amir,从向导链与过客链检测到的读段的比率在体内超过300:1。这种高加工比率可以降低脱靶效应的可能性。
[0847]
表32:向导与过客链读段的比率
[0848][0849][0850]
方法
[0851]
对于静脉内注射,载体在具有0.001%pf-68的pbs中以3.21e9 vg/10微升稀释,并且基于体重通过尾静脉注射小鼠(平均总剂量为8.5e10 vg)。将小鼠置于限制器中,并且用无菌酒精擦拭物擦拭尾部以增加静脉可见度。定位侧尾静脉后,使用32号胰岛素注射器施用溶液。注射后3周,小鼠禁食4小时,通过腔静脉收集血液并处理血清用于ast和alt分析。在pbs灌注后,将肝脏切成切片并放入匀质管(precellys,p/n p000933-lysk0-a)中并在液氮中速冻。对于组织均质化,将补充有β-巯基乙醇的缓冲液rlt添加到样品中,并执行precellys cryolys evolution(贝尔廷医疗器械公司(bertin instruments)),程序设置为3
×
45秒在5000rpm下,暂停15秒。样品管以18,000xg离心3分钟,一部分匀浆用于使用allprep dna/rna/蛋白质迷你试剂盒(凯杰公司,p/n 80004)的dna、rna和蛋白质纯化,另一部分匀浆用于使用mirneasy组织/细胞高级迷你试剂盒(凯杰公司,p/n 217604)的小rna纯化。
[0852]
对于纹状体内注射,载体在具有0.001%pf-68的pbs中以7.5e9 vg/4微升稀释,小鼠在坐标(相对于前囟)前1.5毫米、外侧 /-1.6毫米和腹侧-4.0毫米处注射每半球4ul(汉密尔顿公司(hamilton)p/n 7635-01)超过5分钟。注射后3周后,用冷pbs经心脏灌注小鼠,将脑置于基质中(cellpoint科技公司(cellpoint scientific),alto acrylic 1mm mouse brain coronal 40-75gm),并且切除含有注射部位的2mm冠状切片。在冠状切片中,收集左右纹状体的2mm穿孔活检并放入单独的均质管(precellys,p/n p000933-lysk0-a)中,然后在液氮中速冻。对于组织均质化,将补充有β-巯基乙醇的缓冲液rlt添加到样品中,并执行precellys cryolys evolution(贝尔廷医疗器械公司)进行均质化,程序设置为3
×
45秒在5000rpm下,暂停15秒。样品管以18,000xg离心3分钟,一部分匀浆用于使用allprep dna/rna/蛋白质迷你试剂盒(凯杰公司,p/n 80004)的dna、rna和蛋白质纯化,另一部分匀浆用
于使用mirneasy组织/细胞高级迷你试剂盒(凯杰公司,p/n 217604)的小rna纯化。
[0853]
实施例9:aav载体表达的靶向atxn2的
ami
rna在非人灵长类动物中的药理学研究
[0854]
在非人类灵长类动物中进行测试以通过临床相关施用途径在与神经退行性疾病相关的组织中通过靶向atxn2的amirna raav载体确定atxn2的敲低。要评估的组织包含脊髓腹角、运动皮层和小脑,所述组织与如als或脊髓小脑性共济失调-2等神经退行性疾病相关。
[0855]
在非人灵长类动物中,通过鞘内颈椎(it-c)导管将测试制品(1
×
10
12
vg到1
×
10
14
vg的amirna,所述amirna用h1启动子表达并包装在aav9中)或媒剂施用到小脑延髓池中。将体重约为2.5kg到4kg的雄性和雌性食蟹猴(macaca fascicularis)植入鞘内颈部导管,用于剂量施用和样品收集。通过植入的鞘内导管以0.3ml/分钟的速率施用包括单剂量2.5ml媒剂或测试制品(每个测试制品4只动物),随后施用0.1ml媒剂以从导管冲洗剂量。在施用后5周到26周,处死动物,并且选择的组织用于生物分析和组织学评估。相对于媒剂组,在用包装在aav9中的具有h1启动子的atxn2 amirna处理后,评估atxn2蛋白和mrna水平的抑制。
[0856]
载体评估
[0857]
用于在非人灵长类动物中给药的测试制品通过多种测定进行评估。一项评估是空衣壳和完整衣壳的分析超速离心(auc)以及聚集体的量化。在重力场的力作用下收集吸光度扫描作为材料沉积物。在离心期间实时监测样品沉降曲线,从而对分子大小和形状进行绝对测量。随时间的分布运动用于计算沉降系数。将原始数据拟合到拉姆方程(lamm equation)获得连续分布,并且每个峰下的面积与溶液中存在的量成正比。预期空衣壳在65秒时沉降,部分衣壳在65秒与95秒之间沉降,完整衣壳在95秒时沉降,并且在》110秒时聚集。表明大多数完整衣壳的测量结果是期望的。
[0858]
aav9衣壳elisa用于评估完整的aav9衣壳。捕获抗体检测未组装的衣壳蛋白上不存在的构象表位。adk9抗体在aav9滴定elisa中用作捕获和检测抗体。通过将aav9衣壳elisa与载体基因组滴度进行比较,预期测定结果将证实auc评估。
[0859]
通过鲎属变形细胞裂解物(lal)评估内毒素。需要对低于10eu/ml的细菌内毒素进行检测和定量。
[0860]
通过直接接种评估生物负载,并且低于10cfu/100ml是期望的。
[0861]
对于批次释放和稳定性,进行基因疗法产物效力的体外效力测定。在2v6.11或lec2细胞中,通过rt-qpcr的amirna表达、rt-ddpcr的atxn2 mrna水平和atxn2蛋白facs的atxn2蛋白水平评估体外效力。在转导前,可以用1ug/ml松甾酮a(英杰公司,h10101)、50mu/ml神经氨酸酶(西格玛公司,n7885)和2mm羟基脲(西格玛公司,h8627)对细胞进行预处理。载体的系列稀释液用于处理96孔形式的细胞,在4℃下温育30分钟,然后在应用载体后温育90分钟。然后将板转移到37℃。2天到3天后,在每个剂量下评估amirna、atxn2 mrna和atxn2蛋白。
[0862]
一些实验中的体内效力在对非人灵长类动物给药之前进行测试,并且通过向野生型c57bl/6小鼠静脉内单剂量(如8.5e10vg/克)施用测试制品来评估。收集肝活检,均质化,并且通过allprep dna/rna/蛋白质迷你试剂盒(凯杰公司,80004)提取dna和rna以评估肝脏中的载体分布和atxn2 mrna敲低。
[0863]
在helarc32细胞中进行用于评估载体感染性的中值组织培养感染剂量(tcid50)。helarc32稳定表达aav2 rep和cap基因,并且所述测定涉及在96孔板中系列稀释载体并与腺病毒5辅助病毒共感染、裂解细胞、提取dna、对载体基因组进行qpcr或ddpcr以评估在稀释范围内每孔感染的细胞数量。
[0864]
生物分布和药效学活性
[0865]
来自raav载体和对照处理的动物的非人类灵长类脑和脊髓组织通过穿孔活检收集或在尸检时作为平板收集并速冻。通过添加补充有β-巯基乙醇的缓冲液rlt(凯杰公司)将样品均质化。使用在6500rpm下15秒和10秒中断的3个循环执行基于陶瓷珠的均质化(precellys,ck14 2ml)。dna、rna和蛋白质用allprep dna/rna/蛋白质迷你试剂盒(凯杰公司,80004)提取,并且小rna用mirneasy组织/细胞高级迷你试剂盒(凯杰公司,217604)提取。
[0866]
为了从raav给药的非人灵长类动物中分离运动神经元,在尸检时将脊髓组织冷冻在液氮中。使用用于lcm的arcturus histogene quick h&e stain生成冷冻切片并染色,并且使用arctururs xt lcm系统从每个切片中解剖运动神经元。使用picopure试剂盒(赛默飞世尔科技公司)提取lcm样品中的dna和rna。
[0867]
对于组织学评估,尸检时收集非人灵长类动物的脑和脊髓组织并且将其用10%中性缓冲福尔马林固定24小时,转移到70%乙醇中3天到10天并嵌入到石蜡块中。切割五微米切片,安装在载玻片上,并且针对苏木精和伊红染色用于组织学,或在单独的方案中染色用于免疫组织化学或原位杂交。
[0868]
通过ddpcr评估来自用raav给药的动物组织中的载体生物分布。具体地,使用扩增载体的启动子和/或填充区的引物探针并与对宿主基因组特异的引物探针进行比较,并且结果表示为每个二倍体基因组的载体基因组。
[0869]
为了分离富含运动神经元的组织材料中的生物分布,通过ddpcr对从激光捕获显微切割(lcm)捕获的脊髓神经元中分离的dna评估载体生物分布。具体地,使用扩增载体的启动子和/或填充区的引物探针并与宿主二倍体基因组进行比较,并且结果表示为每个二倍体基因组的载体基因组。如含有运动神经元的运动皮层以及含有浦肯野细胞的小脑等富含其它疾病相关细胞类型的组织材料中的生物分布可以通过相同的ddpcr方法在来自这些脑区域的组织穿孔中进行评估。
[0870]
为了测量脊髓运动神经元中的atxn2敲低,通过rt-ddpcr评估通过激光捕获显微切割捕获的脊髓神经元中的atxn2 mrna。使用atxn2阳性液滴与管家基因(gusb、b2m、tbp或其它)的比率,通过比较amirna处理的受试者相对于媒剂治疗组的脊髓神经元来评估atxn2 mrna的敲低。相对于媒剂给药的动物,在用atxn2靶向amirna给药的动物中,脊髓神经元中atxn2的显著敲低是期望的。
[0871]
脊髓神经元、皮质运动神经元、小脑浦肯野细胞和其它相关组织中的atxn2 mrna也通过组织切片中的原位杂交(ish)和组织穿孔中的rt-ddpcr进行评估。通过原位杂交,atxn2 mrna的敲低通过amirna处理的受试者相对于媒剂组的比较进行半定量评估。这些组织中的显著敲低是期望的,脊髓和皮质运动神经元中atxn2 mrna的减少与als尤其相关,而浦肯野细胞中的敲低与sca2尤其相关。通过rt-ddpcr,如上所述评估敲低。
[0872]
通过免疫组织化学评估脊髓神经元、皮质运动神经元、小脑和其它脑组织中的
atxn2蛋白。固定载玻片使用标准方案用单克隆atxn2抗体(bd,611378)或多克隆atxn2抗体(西格玛公司,hpa018295-100ul)染色。免疫组织化学用于半定量评估atxn2蛋白的敲低,并且atxn2水平相对于媒剂处理的动物显著降低是期望的。
[0873]
可以进行的通过施用到非人灵长类动物脑脊液中给药的atxn2 amirna载体药理学的其它测定包含使用或珠技术的atxn2测定;或者使用mirna-ish或mirna rt-qpcr从组织或体液中进行amirna检测测定。
[0874]
块状组织中的atxn2蛋白通过αlisa进行评估。捕获抗体是单克隆atxn2抗体(bd,611378),并且检测抗体是多克隆atxn2抗体(蛋白技术集团公司(proteintech),21776-1-ap)。csf中的atxn2蛋白通过自定义atxn2 simoa测定(quanterix公司(quanterix))进行评估。
[0875]
参考文献
[0876]
1.auyeung vc,ulitsky i,mcgeary se,bartel dp.超越次级结构:初级序列决定因素允许pri-mirna发夹进行加工(beyond secondary structure:primary-sequence determinants license pri-mirna hairpins for processing).《细胞》.2013.doi:10.1016/j.cell.2013.01.031
[0877]
2.fang w,bartel dp.定义初级微小rna和实现微小rna基因从头设计的特征菜单(the menu of features that define primary micrornas and enable de novo design of microrna genes).《分子细胞》.2015.doi:10.1016/j.molcel.2015.08.015
[0878]
3.fowler dk,williams c,gerritsen at,washbourne p.在增强的mir-155骨架中改进人工微小rna的敲低:有效多靶rnai的设计者指南(improved knockdown from artificial micrornas in an enhanced mir-155backbone:a designer's guide to potent multi-target rnai).《核酸研究》.2015.doi:10.1093/nar/gkv1246
[0879]
4.fellmann c,hoffmann t,sridhar v等人用于有效单拷贝rnai优化的微小rna骨架(an optimized microrna backbone for effective single-copy rnai).《细胞报告(cell rep)》.2013.doi:10.1016/j.celrep.2013.11.020
[0880]
5.kozomara a,birgaoanu m,griffiths-jones s.mirbase:从微小rna序列到功能(mirbase:from microrna sequences to function).《核酸研究》.2019.doi:10.1093/nar/gky1141
[0881]
6.watanabe c,cuellar tl,haley b.对第一代、第二代和第三代发夹系统的定量评估揭示了基于哺乳动物载体的rnai的局限性(quantitative evaluation of first,second,and third generation hairpin systems reveals the limit of mammalian vector-based rnai).《rna生物学(rna biol)》.2016.doi:10.1080/15476286.2015.1128062
[0882]
7.zuker m.用于核酸折叠和杂交预测的mfold网络服务器(mfold web server for nucleic acid folding and hybridization prediction).《核酸研究》.2003.doi:10.1093/nar/gkg595
[0883]
8.chung kh,hart cc,al-bassam s等人基于bic/mir-155的用于rna干扰的多顺反子rna聚合酶ii表达载体(polycistronic rna polymerase ii expression vectors for rna interference based on bic/mir-155).《核酸研究》.2006.doi:10.1093/nar/
gkl143
[0884]
9.lebbink rj,lowe m,chan t,khine h,wang x,mcmanus mt.聚合酶ii启动子强度决定了微小rna适应的shrna的功效(polymerase ii promoter strength determines efficacy of microrna adapted shrnas).《公共科学图书馆
·
综合》.2011.doi:10.1371/journal.pone.0026213
[0885]
10.pfister el,chase ko,sun h等人使用表达靶向人亨廷顿蛋白的人工mirna的pol ii而非pol lii启动子进行安全有效的沉默(safe and efficient silencing with a pol ii,but not a pol lii,promoter expressing an artificial mirnatargeting human huntingtin).《分子疗法-核酸(mol ther-nucleic acids)》.2017.doi:10.1016/j.omtn.2017.04.011
[0886]
11.dirren e,aebischer j,rochat c,towne c,schneider bl,aebischer p.sod1在运动神经元或胶质细胞中的沉默挽救了als小鼠的神经肌肉功能(sod1 silencing in motoneurons or glia rescues neuromuscular function in als mice).《临床和转化神经病学年鉴(ann clin transl neurol)》.2015.doi:10.1002/acn3.162
[0887]
12.pfister el,dinardo n,mondo e等人在亨廷顿氏病的转基因绵羊模型中人工mirna可减少整个纹状体中的人突变体亨廷顿蛋白(artificial mirnas reduce human mutant huntingtin throughout the striatum in a transgenic sheep model of huntington'sdisease).《人类基因疗法(hum gene ther)》.2018.doi:10.1089/hum.2017.199
[0888]
13.borel f,gernoux g,cardozo b等人治疗raavrh10介导成年sod1g93a小鼠和非人灵长类动物中的sod1沉默(therapeutic raavrh10 mediated sod1 silencing in adult sod1g93amice and nonhuman primates).《人类基因疗法》.2016.doi:10.1089/hum.2015.122
[0889]
14.gao z,herrera-carrillo e,berkhout b.3型pol iii启动子的rna聚合酶ii活性(rnapolymerase ii activity of type 3pol iii promoters).《分子疗法-核酸》.2018.doi:10.1016/j.omtn.2018.05.001
[0890]
15.kampmann m,horlbeck ma,chen y等人用于基于全基因组筛选的稳健rna干扰的下一代库(next-generation libraries for robust rna interference-based genome-wide screens).《美国国家科学院院刊(proc natl acad sci u s a)》.2015.doi:10.1073/pnas.1508821112
[0891]
16.gu s,jin l,zhang y等人shrna和前mirna的环位置对于体内dicer加工的准确性至关重要(the loop position of shrnas and pre-mirnas is critical for the accuracy of dicer processing in vivo).《细胞》.2012.doi:10.1016/j.cell.2012.09.042
[0892]
17.chang k,marran k,valentine a,hannon gj.在mir-1骨架中生成表达shrna的转基因果蝇(generation of transgenic drosophila expressing shrnas in the mir-1backbone).《冷泉港实验方案(cold spring harb protoc)》.2014.doi:10.1101/pdb.prot080762
[0893]
18.hoye ml,koval ed,wegener aj等人微小rna分析揭示了als模型中运动神经
元疾病的标志物(microrna profiling reveals marker of motor neuron disease in als models).《神经科学杂志(j neurosci)》.2017.doi:10.1523/jneurosci.3582-16.2017
[0894]
19.ludwig n,leidinger p,becker k等人mirna表达在人体组织中的分布(distribution of mirna expression across human tissues).《核酸研究》.2016.doi:10.1093/nar/gkw116
[0895]
20.otaegi g,pollock a,hong j,sun t.微小rna mir-9通过调节发育中脊髓的foxp1水平来修饰运动神经元柱(microrna mir-9modifies motor neuron columns by a tuning regulation of foxp1 levels in developing spinal cords).《神经科学杂志》.2011.doi:10.1523/jneurosci.4330-10.2011
[0896]
21.morgens dw,deans rm,li a,bassik mc.crispr/cas9和rnai筛选必需基因的系统比较(systematic comparison of crispr/cas9 and rnai screens for essential genes).《自然生物技术》2016.doi:10.1038/nbt.3567
[0897]
22.hart t,chandrashekhar m,aregger m等人高分辨率crispr筛选揭示了健康基因和基因型特异性癌症责任(high-resolution crispr screens reveal fitness genes and genotype-specific cancer liabilities).《细胞》.2015.doi:10.1016/j.cell.2015.11.015
[0898]
23.leonetti md,sekine s,kamiyama d,weissman js,huang b.用于高通量gfp标记内源性人蛋白质的可扩展策略(ascalable strategy for high-throughput gfp tagging of endogenous human proteins).《美国国家科学院院刊》.2016.doi:10.1073/pnas.1606731113
[0899]
24.sack lm,davoli t,xu q,li mz,elledge sj.哺乳动物基因筛选中的错误来源(sources of error in mammalian genetic screens).《g3基因、基因组、基因(g3 genes,genomes,genet)》.2016.doi:10.1534/g3.116.030973
[0900]
25.hawkins ja,jones sk,finkelstein ij,press wh.用于高通量测序的indel校正dna条形码(indel-correcting dnabarcodes for high-throughput sequencing).《美国国家科学院院刊》.2018.doi:10.1073/pnas.1802640115
[0901]
26.morgens dw,wainberg m,boyle ea等人在高通量筛选中使用cas9毒性对脱靶活性进行基因组规模测量(genome-scale measurement of off-target activity using cas9 toxicity in high-throughput screens).《自然通讯》.2017.doi:10.1038/ncomms15178
[0902]
27.hsiau t,maures t,waite k等人从桑格跟踪数据推断crispr编辑(inference of crispr edits from sanger trace data).《生物预印本期刊(biorxiv)》.2018.doi:10.1101/251082
[0903]
上述各个实施例可以组合以提供另外的实施例。在本说明书中引用的和/或在申请数据表中列出的所有美国专利、美国专利申请出版物、美国专利申请、外国专利、外国专利申请和非专利出版物,包括但不限于2020年2月7日提交的美国临时申请第62/971,873号以全文引用的方式并入本文中。如果需要,则可以修改实施例的方面以采用各个专利、申请和出版物的概念来提供又另外的实施例。
[0904]
鉴于以上详细描述,可以对实施例进行这些以和其它改变。一般而言,在以下权利要求书中,所使用的术语不应当被解释为将权利要求书限制于本说明书和权利要求书中所公开的具体实施例,而是应当被解释为包含所有可能的实施例连同此类权利要求有权获得的等效物的整个范围。因此,权利要求并不受本公开的限制。
再多了解一些
本文用于创业者技术爱好者查询,仅供学习研究,如用于商业用途,请联系技术所有人。
