1.本发明涉及医学领域,特别涉及一种跨膜受体基因甲基化作为年轻乳腺癌诊断的潜在标志物。
背景技术:
2.乳腺癌是全球女性发病率最高的恶性肿瘤,也是患癌女性死亡的主要原因。2018年全球新发208.8万例乳腺癌患者,有62.6万名乳腺癌患者死于乳腺癌[bray f,ferlay j, soerjomataram i,siegel rl,torre la,jemal a.global cancer statistics 2018:globocanestimates of incidence and mortality worldwide for 36cancers in 185countries.ca cancer j clin. 2018;68(6):394-424.]。中国乳腺癌的发病率每年以3%-5%的比例稳定增长,据此预计在2021 年中国将会有2.5亿乳腺癌患者,而欧美国家每年增长率仅有0.2%-0.5%[li t,mello-thoms c, brennan pc.descriptive epidemiology of breast cancer in china:incidence,mortality,survival andprevalence.breast cancer res treat.2016;159(3):395-406.]。中国女性被检出乳腺癌的年龄主要集中在30至59岁,比欧美国家平均发病年龄早十年[chen w,zheng r,baade pd,zhang s, zeng h,bray f,et al.cancer statistics in china,2015.ca cancer j clin.2016;66(2):115-32.]。
[0003]
发病年龄低于35岁的乳腺癌被称为早发性乳腺癌,在欧美国家约占3%-5%,而中国的比例是其2倍以上;在中国甚至存在部分超年轻乳腺癌患者,首诊年龄低于25岁,这在欧美国家极其罕见[han w,kang sy,korean breast cancer s.relationship between age at diagnosisand outcome of premenopausal breast cancer:age less than 35years is a reasonable cut-off fordefining young age-onset breast cancer.breast cancer res treat.2010;119(1):193-200.brinton la, sherman me,carreon jd,anderson wf.recent trends in breast cancer among younger women inthe united states.j natl cancer inst.2008;100(22):1643-8.]。早发性乳腺癌往往首诊临床分期晚、肿瘤细胞分化差、脉管瘤栓、人表皮生长因子受体-2(her-2)阳性及雌激素受体(er)阴性的比率更高[anastasiadi z,lianos gd,ignatiadou e,harissis hv,mitsis m.breast cancer inyoung women:an overview.updates surg.2017;69(3):313-7.],早发性乳腺癌患者的预后较差,相比于老年乳腺癌患者,年轻是乳腺癌患者独立的预后不良因素[fredholm h,eaker s,frisell j, holmberg l,fredriksson i,lindman h.breast cancer in young women:poor survival despiteintensive treatment.plos one.2009;4(11):e7695.cancello g,maisonneuve p,rotmensz n,vialeg,mastropasqua mg,pruneri g,et al.prognosis and adjuvant treatment effects in selected breastcancer subtypes of very young women(《35years)with operable breast cancer.ann oncol. 2010;21(10):1974-81]。因此,降低早发性乳腺癌患者死亡率的关键
在于早诊断早治疗。
[0004]
表观遗传学是一种不涉及dna序列改变但可遗传的基因表达调控方式,并能够遗传给下一代[nicoglou a,merlin f.epigenetics:a way to bridge the gap between biological fields.studhist philos biol biomed sci.2017;66:73-82]。dna甲基化是表观遗传调控的重要方式之一,是指在dna甲基化转移酶的作用下,在基因组cpg二核苷酸的胞嘧啶5'碳位共价键结合一个甲基基团[bird a.perceptions of epigenetics.nature.2007;447:396-398]。大量研究表明,dna 甲基化能引起染色质结构、dna构象、dna稳定性及dna与蛋白质相互作用方式的改变,从而控制基因表达[moore ld,le t,fan g.dna methylation and its basic function. neuropsychopharmacology.2013;38:23-38]。ptch2(patched 2)基因位于1p34.1,编码包含1203 个氨基酸的12次跨膜蛋白,为hedgehog通路的主要成员,其主要在皮肤和睾丸组织中高表达[skoda am,simovic d,karin v,kardum v,vranic s,serman l.the role of the hedgehogsignaling pathway in cancer:a comprehensive review.bosn j basic med sci.2018;18(1):8-20.]。 ptch2基因异常表达已证实与痣样基底细胞癌综合征、成神经管细胞瘤、骨髓增生性疾病相关[lacour jp.carcinogenesis of basal cell carcinomas:genetics and molecular mechanisms.br j dermatol.2002;146suppl 61:17-9.ptch2 gene alterations in keratocystic odontogenic tumorsassociated with nevoid basal cell carcinoma syndrome.journal of peking university(healthsciences).2008;40(1):15-8.klein c,zwick a,kissel s,forster cu,pfeifer d,follo m,et al.ptch2 loss drives myeloproliferation and myeloproliferative neoplasm progression.j exp med. 2016;213(2):273-90.smyth i,narang ma,evans t,heimann c,nakamura y,chenevix-trench g, et al.isolation and characterization of human patched 2(ptch2),a putative tumour suppressorgene inbasal cell carcinoma and medulloblastoma on chromosome 1p32.hum mol genet. 1999;8(2):291-7.]。目前关于ptch2基因与乳腺癌的关系在动物与细胞研究中未见报道,外周血ptch2基因与人群乳腺癌关系的研究还未见报道,ptch2甲基化与中国人群早发性乳腺癌关系的研究未见报道。
技术实现要素:
[0005]
本发明的目的是提供一种跨膜受体基因甲基化作为年轻乳腺癌诊断的潜在标志物。
[0006]
第一方面,本发明要求保护甲基化ptch2基因作为标志物在制备产品中的应用。
[0007]
其中,所述产品具有如下任一功能:
[0008]
(1)辅助诊断乳腺癌或预测乳腺癌患病风险;
[0009]
(2)辅助区分乳腺癌和健康对照;
[0010]
(3)辅助区分年龄≤35岁的乳腺癌患者和健康对照;
[0011]
(4)辅助区分年龄>35且<45岁的乳腺癌患者和健康对照;
[0012]
(5)辅助区分年龄≥45岁的乳腺癌患者和健康对照;
[0013]
(6)辅助区分临床分期为0期和/或i期的乳腺癌患者和健康对照;
[0014]
(7)辅助区分临床分期为ii期的乳腺癌患者和健康对照;
[0015]
(8)辅助区分临床分期为iii期和/或ⅳ期的乳腺癌患者和健康对照;
[0016]
(9)辅助区分肿瘤大小为tis和/或t1的乳腺癌患者和健康对照;
[0017]
(10)辅助区分肿瘤大小为t2的乳腺癌患者和健康对照;
[0018]
(11)辅助区分肿瘤大小为t3和/或t4的乳腺癌患者和健康对照;
[0019]
(12)辅助区分分子分型为luminal a型的乳腺癌患者和健康对照;
[0020]
(13)辅助区分分子分型为luminal b型的乳腺癌患者和健康对照;
[0021]
(14)辅助区分分子分型为her2扩增型的乳腺癌患者和健康对照;
[0022]
(15)辅助区分分子分型为基底样型的乳腺癌患者和健康对照;
[0023]
(16)辅助区分ki67蛋白表达量高的乳腺癌患者和健康对照;
[0024]
(17)辅助区分ki67蛋白表达量低的乳腺癌患者和健康对照;
[0025]
(18)辅助区分p53基因突变率高的乳腺癌患者和健康对照;
[0026]
(19)辅助区分p53基因突变率低的乳腺癌患者和健康对照。
[0027]
其中,所述健康对照为现在及曾经均没有患过癌症且血常规指标都在参考范围内。所述 (3)中,所述健康对照年龄≤35岁;所述(4)中,所述健康对照年龄>35且<45岁;所述(5)中,所述健康对照年龄≥45岁。所述ki67蛋白表达量高是指通过免疫组化检测组织中ki67染色细胞的比例≥30%;相应的,所述ki67蛋白表达量低是指通过免疫组化检测组织中ki67染色细胞的比例<30%。所述p53基因突变率高是指通过免疫组化检测组织中p53 突变阳性染色细胞的比例≥30%;相应的,所述p53基因突变率低是指通过免疫组化检测组织中p53突变阳性染色细胞的比例<30%。下同。
[0028]
第二方面,本发明要求保护用于检测ptch2基因甲基化水平的物质在制备产品中的应用。
[0029]
其中,所述产品具有如下前文(1)-(19)中任一所示功能。
[0030]
第三方面,本发明要求保护用于检测ptch2基因甲基化水平的物质和储存有数学模型建立方法和/或使用方法的介质在制备产品中的应用。
[0031]
其中,所述产品具有如下前文(1)-(19)中任一所示功能。
[0032]
所述数学模型可按照包括如下步骤的方法获得:
[0033]
(a1)分别检测n1个a类型样本和n2个b类型样本的ptch2基因甲基化水平;
[0034]
(a2)取步骤(a1)获得的所有样本的ptch2基因甲基化水平数据,按照a类型样本和b类型样本的分类方式,通过二分类逻辑回归法建立数学模型,确定分类判定的阈值。
[0035]
其中,(a1)中的n1和n2均为50以上的正整数。
[0036]
所述数学模型的使用方法可包括如下步骤:
[0037]
(b1)检测待测样本的ptch2基因甲基化水平;
[0038]
(b2)将步骤(b1)获得的所述待测样本的ptch2基因甲基化水平数据代入所述数学模型,得到检测指数;然后比较检测指数和阈值的大小,根据比较结果确定所述待测样本是 a类型样本还是b类型样本。
[0039]
在本发明的具体实施方式中,所述阈值设为0.5。大于0.5归为一类,小于0.5归为另外一类,等于0.5作为不确定的灰区。其中a类型和b类型为相对应的两分类,二分类的分组,哪一组是a类型,哪一组是b类型,要根据具体的数学模型来确定,无需约定。
[0040]
在实际应用中,所述阈值也可根据最大约登指数确定(具体可为最大约登指数对
应的数值)。大于阈值归为一类,小于阈值归为另外一类,等于阈值作为不确定的灰区。其中a类型和b类型为相对应的两分类,二分类的分组,哪一组是a类型,哪一组是b类型,要根据具体的数学模型来确定,无需约定。
[0041]
所述a类型样本和所述b类型样本为如下中的任一种:
[0042]
(c1)乳腺癌患者和健康对照;
[0043]
(c2)年龄≤35岁的乳腺癌患者和健康对照;
[0044]
(c3)年龄>35且<45岁的乳腺癌患者和健康对照;
[0045]
(c4)年龄≥45岁的乳腺癌患者和健康对照;
[0046]
(c5)临床分期为0期和/或i期的乳腺癌患者和健康对照;
[0047]
(c6)临床分期为ii期的乳腺癌患者和健康对照;
[0048]
(c7)临床分期为iii期和/或ⅳ期的乳腺癌患者和健康对照;
[0049]
(c8)肿瘤大小为tis和/或t1的乳腺癌患者和健康对照;
[0050]
(c9)肿瘤大小为t2的乳腺癌患者和健康对照;
[0051]
(c10)肿瘤大小为t3和/或t4的乳腺癌患者和健康对照;
[0052]
(c11)分子分型为luminal a型的乳腺癌患者和健康对照;
[0053]
(c12)分子分型为luminal b型的乳腺癌患者和健康对照;
[0054]
(c13)分子分型为her2扩增型的乳腺癌患者和健康对照;
[0055]
(c14)分子分型为基底样型的乳腺癌患者和健康对照;
[0056]
(c15)ki67蛋白表达量高的乳腺癌患者和健康对照;
[0057]
(c16)ki67蛋白表达量低的乳腺癌患者和健康对照;
[0058]
(c17)p53基因突变率高的乳腺癌患者和健康对照;
[0059]
(c18)p53基因突变率低的乳腺癌患者和健康对照。
[0060]
第四方面,本发明要求保护前文第三方面中所述的“储存有数学模型建立方法和/或使用方法的介质”在制备产品中的应用。
[0061]
其中,所述产品具有如下前文(1)-(19)中任一所示功能。
[0062]
第五方面,本发明要求保护一种试剂盒。
[0063]
本发明要求保护的试剂盒包括用于检测ptch2基因甲基化水平的物质。所述试剂盒的用途可为前文(1)-(19)中的至少一种。
[0064]
进一步地,所述试剂盒中还可含有前文第三方面或第四方面中所述的“储存有数学模型建立方法和/或使用方法的介质”。
[0065]
第六方面,本发明要求保护一种系统。
[0066]
本发明所要求保护的系统,可包括:
[0067]
(d1)用于检测ptch2基因甲基化水平的试剂和/或仪器;
[0068]
(d2)装置,所述装置包括单元m和单元n。
[0069]
所述单元m用于建立数学模型,包括数据采集模块、数据分析处理模块和模型输出模块。
[0070]
所述数据采集模块被配置为采集(d1)检测得到的n1个a类型样本和n2个b类型样本的ptch2基因甲基化水平数据。
[0071]
所述数据分析处理模块被配置为接收由所述数据采集模块发送的n1个a类型样本
和n2 个b类型样本的ptch2基因甲基化水平数据,并基于所述数据采集模块采集的n1个a类型样本和n2个b类型样本的ptch2基因甲基化水平数据,按照a类型样本和b类型样本的分类方式,通过二分类逻辑回归法建立数学模型。
[0072]
所述模型输出模块被配置为输出所述数据分析处理模块建立的数学模型。
[0073]
所述单元n用于确定待测样本是a类型样本还是b类型样本,包括数据输入模块、数据运算模块、数据比较模块和结论输出模块。
[0074]
所述数据输入模块被配置为输入(d1)检测得到的待测者的ptch2基因甲基化水平数据。
[0075]
所述数据运算模块被配置接收由所述数据输入模块发送的所述待测者的ptch2基因甲基化水平数据,并将所述待测者ptch2基因甲基化水平数据代入所述数学模型,计算得到检测指数。
[0076]
所述数据比较模块被配置为接收所述数据运算模块发送的所述检测指数,并将所述检测指数与阈值进行比较。
[0077]
所述结论输出模块被配置为根据所述数据比较模块的比较结果输出所述待测样本是a 类型样本还是b类型样本的结论。
[0078]
所述a类型样本和所述b类型样本为如下中的任一种:
[0079]
(c1)乳腺癌患者和健康对照;
[0080]
(c2)年龄≤35岁的乳腺癌患者和健康对照;
[0081]
(c3)年龄>35且<45岁的乳腺癌患者和健康对照;
[0082]
(c4)年龄≥45岁的乳腺癌患者和健康对照;
[0083]
(c5)临床分期为0期和/或i期的乳腺癌患者和健康对照;
[0084]
(c6)临床分期为ii期的乳腺癌患者和健康对照;
[0085]
(c7)临床分期为iii期和/或ⅳ期的乳腺癌患者和健康对照;
[0086]
(c8)肿瘤大小为tis和/或t1的乳腺癌患者和健康对照;
[0087]
(c9)肿瘤大小为t2的乳腺癌患者和健康对照;
[0088]
(c10)肿瘤大小为t3和/或t4的乳腺癌患者和健康对照;
[0089]
(c11)分子分型为luminal a型的乳腺癌患者和健康对照;
[0090]
(c12)分子分型为luminal b型的乳腺癌患者和健康对照;
[0091]
(c13)分子分型为her2扩增型的乳腺癌患者和健康对照;
[0092]
(c14)分子分型为基底样型的乳腺癌患者和健康对照;
[0093]
(c15)ki67蛋白表达量高的乳腺癌患者和健康对照;
[0094]
(c16)ki67蛋白表达量低的乳腺癌患者和健康对照;
[0095]
(c17)p53基因突变率高的乳腺癌患者和健康对照;
[0096]
(c18)p53基因突变率低的乳腺癌患者和健康对照。
[0097]
其中,n1和n2均可为50以上正整数。
[0098]
在本发明的具体实施方式中,所述阈值设为0.5。大于0.5归为一类,小于0.5归为另外一类,等于0.5作为不确定的灰区。其中a类型和b类型为相对应的两分类,二分类的分组,哪一组是a类型,哪一组是b类型,要根据具体的数学模型来确定,无需约定。
[0099]
在实际应用中,所述阈值也可根据最大约登指数确定(具体可为最大约登指数对
应的数值)。大于阈值归为一类,小于阈值归为另外一类,等于阈值作为不确定的灰区。其中a类型和b类型为相对应的两分类,二分类的分组,哪一组是a类型,哪一组是b类型,要根据具体的数学模型来确定,无需约定。
[0100]
在前文各方面中,所述“ptch2基因甲基化水平”可为ptch2基因中如下(e1)-(e6) 所示片段中全部或部分cpg位点的甲基化水平。所述甲基化ptch2基因为ptch2基因中如下(e1)-(e6)所示片段中全部或部分cpg位点甲基化。
[0101]
(e1)seq id no.1所示的dna片段或与其具有80%以上同一性的dna片段;
[0102]
(e2)seq id no.2所示的dna片段或与其具有80%以上同一性的dna片段;
[0103]
(e3)seq id no.3所示的dna片段或与其具有80%以上同一性的dna片段;
[0104]
(e4)seq id no.4所示的dna片段或与其具有80%以上同一性的dna片段;
[0105]
(e5)seq id no.5所示的dna片段或与其具有80%以上同一性的dna片段;
[0106]
(e6)seq id no.6所示的dna片段或与其具有80%以上同一性的dna片段。
[0107]
进一步地,所述“全部或部分cpg位点”可为如下任一:
[0108]
(f1)seq id no.1所示的dna片段的5个可区分cpg位点中的全部或任意4个或任意3个或任意2个或任意1个;
[0109]
(f2)seq id no.2所示的dna片段的7个可区分cpg位点中的全部或任意6个或任意5个或任意4个或任意3个或任意2个或任意1个;
[0110]
(f3)seq id no.3所示的dna片段的7个可区分cpg位点中的全部或任意6个或任意5个或任意4个或任意3个或任意2个或任意1个;
[0111]
(f4)seq id no.4所示的dna片段的6个可区分cpg位点中的全部或任意5个或任意4个或任意3个或任意2个或任意1个;
[0112]
(f5)seq id no.5所示的dna片段的7个可区分cpg位点中的全部或任意6个或任意5个或任意4个或任意3个或任意2个或任意1个;
[0113]
(f6)seq id no.6所示的dna片段的4个可区分cpg位点中的全部或任意3个或任意2个或任意1个;
[0114]
(f7)seq id no.1所示的dna片段的5个可区分cpg位点和seq id no.2所示的dna 片段的7个可区分cpg位点;
[0115]
(f8)seq id no.1所示的dna片段的5个可区分cpg位点和seq id no.3所示的dna 片段的7个可区分cpg位点;
[0116]
(f9)seq id no.1所示的dna片段的5个可区分cpg位点和seq id no.4所示的dna 片段的6个可区分cpg位点;
[0117]
(f10)seq id no.1所示的dna片段的5个可区分cpg位点和seq id no.5所示的 dna片段的7个可区分cpg位点;
[0118]
(f11)seq id no.1所示的dna片段的5个可区分cpg位点和seq id no.6所示的 dna片段的4个可区分cpg位点;
[0119]
(f12)seq id no.2所示的dna片段的7个可区分cpg位点和seq id no.3所示的 dna片段的7个可区分cpg位点;
[0120]
(f13)seq id no.2所示的dna片段的7个可区分cpg位点和seq id no.4所示的 dna片段的6个可区分cpg位点;
片段的7个可区分cpg位点、seq id no.4所示的dna片段的6个可区分cpg位点和seq
ꢀꢀ
id no.6所示的dna片段的4个可区分cpg位点;
[0157]
(f50)seq id no.1所示的dna片段的5个可区分cpg位点、seq id no.3所示的dna 片段的7个可区分cpg位点、seq id no.5所示的dna片段的7个可区分cpg位点和seq
ꢀꢀ
id no.6所示的dna片段的4个可区分cpg位点;
[0158]
(f51)seq id no.1所示的dna片段的5个可区分cpg位点、seq id no.4所示的dna 片段的6个可区分cpg位点、seq id no.5所示的dna片段的7个可区分cpg位点和seq
ꢀꢀ
id no.6所示的dna片段的4个可区分cpg位点;
[0159]
(f52)seq id no.2所示的dna片段的7个可区分cpg位点、seq id no.3所示的dna 片段的7个可区分cpg位点、seq id no.4所示的dna片段的6个可区分cpg位点和seq
ꢀꢀ
id no.5所示的dna片段的7个可区分cpg位点;
[0160]
(f53)seq id no.2所示的dna片段的7个可区分cpg位点、seq id no.3所示的dna 片段的7个可区分cpg位点、seq id no.4所示的dna片段的6个可区分cpg位点和seq
ꢀꢀ
id no.6所示的dna片段的4个可区分cpg位点;
[0161]
(f54)seq id no.2所示的dna片段的7个可区分cpg位点、seq id no.3所示的dna 片段的7个可区分cpg位点、seq id no.5所示的dna片段的7个可区分cpg位点和seq
ꢀꢀ
id no.6所示的dna片段的4个可区分cpg位点;
[0162]
(f55)seq id no.2所示的dna片段的7个可区分cpg位点、seq id no.4所示的dna 片段的6个可区分cpg位点、seq id no.5所示的dna片段的7个可区分cpg位点和seq
ꢀꢀ
id no.6所示的dna片段的4个可区分cpg位点;
[0163]
(f56)seq id no.3所示的dna片段的7个可区分cpg位点、seq id no.4所示的dna 片段的6个可区分cpg位点、seq id no.5所示的dna片段的7个可区分cpg位点和seq
ꢀꢀ
id no.6所示的dna片段的4个可区分cpg位点;
[0164]
(f57)seq id no.1所示的dna片段的5个可区分cpg位点、seq id no.2所示的dna 片段的7个可区分cpg位点、seq id no.3所示的dna片段的7个可区分cpg位点、seq id
ꢀꢀ
no.4所示的dna片段的6个可区分cpg位点和seq id no.5所示的dna片段的7个可区分cpg位点;
[0165]
(f58)seq id no.1所示的dna片段的5个可区分cpg位点、seq id no.2所示的dna 片段的7个可区分cpg位点、seq id no.3所示的dna片段的7个可区分cpg位点、seq id
ꢀꢀ
no.4所示的dna片段的6个可区分cpg位点和seq id no.6所示的dna片段的4个可区分cpg位点;
[0166]
(f59)seq id no.1所示的dna片段的5个可区分cpg位点、seq id no.2所示的dna 片段的7个可区分cpg位点、seq id no.3所示的dna片段的7个可区分cpg位点、seq id
ꢀꢀ
no.5所示的dna片段的7个可区分cpg位点和seq id no.6所示的dna片段的4个可区分cpg位点;
[0167]
(f60)seq id no.1所示的dna片段的5个可区分cpg位点、seq id no.2所示的dna 片段的7个可区分cpg位点、seq id no.4所示的dna片段的6个可区分cpg位点、seq id
ꢀꢀ
no.5所示的dna片段的7个可区分cpg位点和seq id no.6所示的dna片段的4个可区分cpg位点;
[0168]
(f61)seq id no.1所示的dna片段的5个可区分cpg位点、seq id no.3所示的dna 片段的7个可区分cpg位点、seq id no.4所示的dna片段的6个可区分cpg位点、seq id
ꢀꢀ
no.5所示的dna片段的7个可区分cpg位点和seq id no.6所示的dna片段的4个可区分cpg位点;
[0169]
(f62)seq id no.2所示的dna片段的7个可区分cpg位点、seq id no.3所示的dna 片段的7个可区分cpg位点、seq id no.4所示的dna片段的6个可区分cpg位点、seq id
ꢀꢀ
no.5所示的dna片段的7个可区分cpg位点和seq id no.6所示的dna片段的4个可区分cpg位点;
[0170]
(f63)seq id no.1所示的dna片段的5个可区分cpg位点、seq id no.2所示的dna 片段的7个可区分cpg位点、seq id no.3所示的dna片段的7个可区分cpg位点、seq idno.4所示的dna片段的6个可区分cpg位点、seq id no.5所示的dna片段的7个可区分 cpg位点和seq id no.6所示的dna片段的4个可区分cpg位点。
[0171]
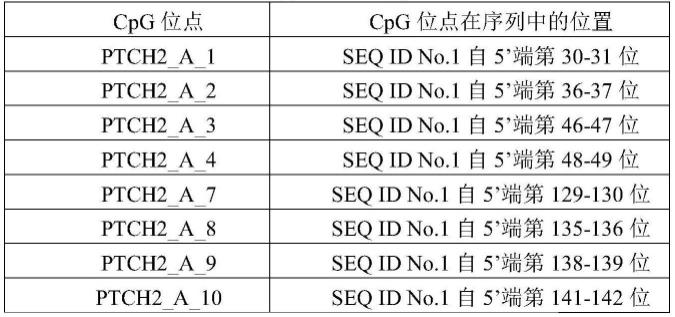
其中,所述seq id no.1所示的dna片段的5个可区分cpg位点为:seq id no.1自 5’端起的第30-31位、第36-37位、第46-47位和第48-49位所示cpg位点(ptch2_a_1.2.3.4);第129-130位、第135-136位、第138-139位、第141-142位和第147-148位所示cpg位点 (ptch2_a_7.8.9.10.11);第172-173位所示cpg位点(ptch2_a_13);第197-198位和第 200-201位所示cpg位点(ptch2_a_14.15);第253-254位所示cpg位点(ptch2_a_19)。
[0172]
所述seq id no.2所示的dna片段的7个可区分cpg位点为:seq id no.2自5’端起的第31-32位所示cpg位点(pcth2_b_1);第72-73位所示cpg位点(pcth2_b_2);第 124-125位所示cpg位点(pcth2_b_6);第173-174位所示cpg位点(pcth2_b_7);第 226-227位、第230-231位、第234-235位和第236-237位所示cpg位点(pcth2_b_9.10.11.12);第246-247位所示cpg位点(pcth2_b_13);第304-305位所示cpg位点(pcth2_b_14)。
[0173]
所述seq id no.3所示的dna片段的7个可区分cpg位点为:seq id no.3自5’端起的第29-30位所示cpg位点(pcth2_c_1);第41-42位所示cpg位点(pcth2_c_2);第 74-75位所示cpg位点(pcth2_c_3);第84-85位所示cpg位点(pcth2_c_4);第91-92 位所示cpg位点(pcth2_c_5);第108-109位所示cpg位点(pcth2_c_6);第116-117 位所示cpg位点(pcth2_c_7)。
[0174]
所述seq id no.4所示的dna片段的6个可区分cpg位点为:seq id no.4自5’端起的第117-118位所示cpg位点(pcth2_d_1);第152-153位所示cpg位点(pcth2_d_2);第163-164位所示cpg位点(pcth2_d_3);第202-203位所示cpg位点(pcth2_d_4);第260-261位所示cpg位点(pcth2_d_7);第267-268位所示cpg位点(pcth2_d_8)。
[0175]
所述seq id no.5所示的dna片段的7个可区分cpg位点为:seq id no.5自5’端起的第98-99位所示cpg位点(pcth2_e_1);第177-178位所示cpg位点(pcth2_e_3);第180-181位和第220-221位所示cpg位点(pcth2_e_4.5);第232-233位所示cpg位点(pcth2_e_6);第250-251位所示cpg位点(pcth2_e_7);第259-260位和第270-271位所示cpg位点(pcth2_e_8.9);第280-281位和第318-319位所示cpg位点 (pcth2_e_10.11)。
[0176]
所述seq id no.6所示的dna片段的4个可区分cpg位点为:seq id no.6自5’端起的第27-28位所示cpg位点(pcth2_f_1);第75-76位所示cpg位点(pcth2_f_2);第 114-115位所示cpg位点(pcth2_f_3);第278-279位所示cpg位点(pcth2_f_4)。
pcth2_a_14.15、pcth2_a_19、pcth2_b_1、pcth2_b_2、pcth2_b_6、pcth2_b_7、 pcth2_b_9.10.11.12、pcth2_c_1、pcth2_c_2、pcth2_c_3、pcth2_c_4、pcth2_c_5、 pcth2_c_6、pcth2_c_7、pcth2_d_1、pcth2_d_2、pcth2_d_3、pcth2_d_4、 pcth2_d_7、pcth2_e_3、pcth2_e_4.5、pcth2_e_6、pcth2_e_7、pcth2_e_8.9、 pcth2_e_10.11、pcth2_f_1、pcth2_f_2、pcth2_f_3和/或pcth2_f_4。
[0190]
进一步地,当辅助区分分子分型为基底样型的乳腺癌患者和健康对照时,所述“全部或部分cpg位点”可为:pcth2_a_1.2.3.4、pcth2_a_7.8.9.10.11、pcth2_a_13、 pcth2_a_14.15、pcth2_a_19、pcth2_b_1、pcth2_b_2、pcth2_b_6、pcth2_b_7、 pcth2_b_9.10.11.12、pcth2_c_1、pcth2_c_2、pcth2_c_3、pcth2_c_4、pcth2_c_5、 pcth2_c_6、pcth2_c_7、pcth2_d_1、pcth2_d_2、pcth2_d_3、pcth2_d_4、 pcth2_d_7、pcth2_d_8、pcth2_e_1、pcth2_e_3、pcth2_e_4.5、pcth2_e_6、 pcth2_e_7、pcth2_e_8.9、pcth2_e_10.11、pcth2_f_1、pcth2_f_2、pcth2_f_3和/ 或pcth2_f_4。
[0191]
进一步地,当辅助区分ki67蛋白表达量高的乳腺癌患者和健康对照时,所述“全部或部分cpg位点”可为:pcth2_a_1.2.3.4、pcth2_a_7.8.9.10.11、pcth2_a_13、pcth2_a_19、 pcth2_b_1、pcth2_b_2、pcth2_b_7、pcth2_b_9.10.11.12、pcth2_c_1、pcth2_c_2、 pcth2_c_3、pcth2_c_4、pcth2_c_5、pcth2_d_1、pcth2_d_2、pcth2_d_3、 pcth2_d_4、pcth2_d_7、pcth2_d_8、pcth2_e_1、pcth2_e_3、pcth2_e_4.5、 pcth2_e_6、pcth2_e_7、pcth2_e_8.9、pcth2_e_10.11、pcth2_f_2和/或pcth2_f_3。
[0192]
进一步地,当辅助区分ki67蛋白表达量低的乳腺癌患者和健康对照时,所述“全部或部分cpg位点”可为:pcth2_a_1.2.3.4、pcth2_a_7.8.9.10.11、pcth2_a_13、pcth2_a_19、 pcth2_b_1、pcth2_b_2、pcth2_b_7、pcth2_b_9.10.11.12、pcth2_c_1、pcth2_c_2、 pcth2_c_3、pcth2_c_4、pcth2_c_5、pcth2_d_1、pcth2_d_2、pcth2_d_3、 pcth2_d_4、pcth2_e_1、pcth2_e_3、pcth2_e_4.5、pcth2_e_6、pcth2_e_7、 pcth2_e_8.9、pcth2_e_10.11、pcth2_f_1、pcth2_f_2和/或pcth2_f_3。
[0193]
进一步地,当辅助区分p53基因突变率高的乳腺癌患者和健康对照时,所述“全部或部分cpg位点”可为:pcth2_a_1.2.3.4、pcth2_a_7.8.9.10.11、pcth2_a_13、pcth2_a_19、 pcth2_b_1、pcth2_b_2、pcth2_b_7、pcth2_b_9.10.11.12、pcth2_c_1、pcth2_c_2、 pcth2_c_3、pcth2_c_4、pcth2_c_5、pcth2_d_1、pcth2_d_2、pcth2_d_3、 pcth2_d_4、pcth2_d_7、pcth2_d_8、pcth2_e_1、pcth2_e_3、pcth2_e_4.5、 pcth2_e_6、pcth2_e_7、pcth2_e_8.9、pcth2_e_10.11、pcth2_f_2和/或pcth2_f_3。
[0194]
进一步地,当辅助区分p53基因突变率低的乳腺癌患者和健康对照时,所述“全部或部分cpg位点”可为:pcth2_a_1.2.3.4、pcth2_a_7.8.9.10.11、pcth2_a_13、pcth2_a_19、 pcth2_b_1、pcth2_b_2、pcth2_b_7、pcth2_b_9.10.11.12、pcth2_c_1、pcth2_c_2、 pcth2_c_3、pcth2_c_4、pcth2_c_5、pcth2_d_1、pcth2_d_2、pcth2_d_3、 pcth2_d_4、pcth2_e_1、pcth2_e_3、pcth2_e_4.5、pcth2_e_6、pcth2_e_7、 pcth2_e_8.9、pcth2_e_10.11、pcth2_f_1、pcth2_f_2和/或pcth2_f_3。
[0195]
在本发明的具体实施方式中,有些相邻的甲基化位点在利用飞行时间质谱进行dna甲基化分析时由于几个cpg位点位于一个甲基化片段上,峰图无法区分,因而在进行甲基化水平分析、以及构建和使用相关数学模型时将其按照一个甲基化位点进行处理。
[0196]
在上述各方面中,所述用于检测pcth2基因甲基化水平的物质可包含(或为)用于扩增pcth2基因全长或部分片段的引物组合。所述用于检测pcth2基因甲基化水平的试剂可包含(或为)用于扩增pcth2基因全长或部分片段的引物组合;所述用于检测pcth2基因甲基化水平的仪器可为飞行时间质谱检测仪。当然所述用于检测pcth2基因甲基化水平的试剂中还可包含进行飞行时间质谱所用的其他常规试剂。
[0197]
进一步地,所述部分片段可为如下中至少一个片段:
[0198]
(g1)seq id no.1所示的dna片段或其包含的dna片段;
[0199]
(g2)seq id no.2所示的dna片段或其包含的dna片段;
[0200]
(g3)seq id no.3所示的dna片段或其包含的dna片段;
[0201]
(g4)seq id no.4所示的dna片段或其包含的dna片段;
[0202]
(g5)seq id no.5所示的dna片段或其包含的dna片段;
[0203]
(g6)seq id no.6所示的dna片段或其包含的dna片段;
[0204]
(g7)与seq id no.1所示的dna片段或其包含的dna片段具有80%以上同一性的 dna片段;
[0205]
(g8)与seq id no.2所示的dna片段或其包含的dna片段具有80%以上同一性的 dna片段;
[0206]
(g9)与seq id no.3所示的dna片段或其包含的dna片段具有80%以上同一性的 dna片段;
[0207]
(g10)与seq id no.4所示的dna片段或其包含的dna片段具有80%以上同一性的 dna片段;
[0208]
(g11)与seq id no.5所示的dna片段或其包含的dna片段具有80%以上同一性的 dna片段;
[0209]
(g12)与seq id no.6所示的dna片段或其包含的dna片段具有80%以上同一性的 dna片段。
[0210]
在本发明中,所述引物组合具体可为引物对a和/或引物对b和/或引物对c和/或引物对d和/或引物对e和/或引物对f。
[0211]
所述引物对a为引物a1和引物a2组成的引物对;所述引物a1为seq id no.7或seqid no.7的第11-37位核苷酸所示的单链dna;所述引物a2为seq id no.8或seq id no.8 的第32-56位核苷酸所示的单链dna。
[0212]
所述引物对b为引物b1和引物b2组成的引物对;所述引物b1为seq id no.9或seqid no.9的第11-35位核苷酸所示的单链dna;所述引物b2为seq id no.10或seq id no.10 的第32-56位核苷酸所示的单链dna。
[0213]
所述引物对c为引物c1和引物c2组成的引物对;所述引物c1为seq id no.11或seqid no.11的第11-36位核苷酸所示的单链dna;所述引物c2为seq id no.12或seq id no.12 的第32-56位核苷酸所示的单链dna。
[0214]
所述引物对d为引物d1和引物d2组成的引物对;所述引物d1为seq id no.13或seqid no.13的第11-35位核苷酸所示的单链dna;所述引物d2为seq id no.14或seq id no.14 的第32-56位核苷酸所示的单链dna。
[0215]
所述引物对e为引物e1和引物e2组成的引物对;所述引物e1为seq id no.15或seq
id no.15的第11-35位核苷酸所示的单链dna;所述引物e2为seq id no.16或seq id no.16 的第32-56位核苷酸所示的单链dna。
[0216]
所述引物对f为引物e1和引物e2组成的引物对;所述引物e1为seq id no.17或seqid no.17的第11-35位核苷酸所示的单链dna;所述引物e2为seq id no.18或seq id no.18 的第32-56位核苷酸所示的单链dna。
[0217]
另外,本发明还要求保护一种区分待测样本为a类型样本还是b类型样本的方法。该方法可包括如下步骤:
[0218]
(a)可按照包括如下步骤的方法建立数学模型:
[0219]
(a1)分别检测n1个a类型样本和n2个b类型样本的pcth2基因甲基化水平(训练集);
[0220]
(a2)取步骤(a1)获得的所有样本的pcth2基因甲基化水平数据,按照a类型和b 类型的分类方式,通过二分类逻辑回归法建立数学模型,确定分类判定的阈值。
[0221]
其中,(a1)中的n1和n2均为50以上的正整数。
[0222]
(b)可按照包括如下步骤的方法确定所述待测样本为a类型样本还是b类型样本:
[0223]
(b1)检测所述待测样本的pcth2基因甲基化水平;
[0224]
(b2)将步骤(b1)获得的所述待测样本的pcth2基因甲基化水平数据代入所述数学模型,得到检测指数;然后比较检测指数和阈值的大小,根据比较结果确定所述待测样本的类型是a类型还是b类型。
[0225]
在本发明的具体实施方式中,所述阈值设为0.5。大于0.5归为一类,小于0.5归为另外一类,等于0.5作为不确定的灰区。其中a类型和b类型为相对应的两分类,二分类的分组,哪一组是a类型,哪一组是b类型,要根据具体的数学模型来确定,无需约定。
[0226]
在实际应用中,所述阈值也可根据最大约登指数确定(具体可为最大约登指数对应的数值)。大于阈值归为一类,小于阈值归为另外一类,等于阈值作为不确定的灰区。其中a类型和b类型为相对应的两分类,二分类的分组,哪一组是a类型,哪一组是b类型,要根据具体的数学模型来确定,无需约定。
[0227]
所述a类型样本和所述b类型样本可为如下中的任一种:
[0228]
(c1)乳腺癌患者和健康对照;
[0229]
(c2)年龄≤35岁的乳腺癌患者和健康对照;
[0230]
(c3)年龄>35且<45岁的乳腺癌患者和健康对照;
[0231]
(c4)年龄≥45岁的乳腺癌患者和健康对照;
[0232]
(c5)临床分期为0期和/或i期的乳腺癌患者和健康对照;
[0233]
(c6)临床分期为ii期的乳腺癌患者和健康对照;
[0234]
(c7)临床分期为iii期和/或ⅳ期的乳腺癌患者和健康对照;
[0235]
(c8)肿瘤大小为tis和/或t1的乳腺癌患者和健康对照;
[0236]
(c9)肿瘤大小为t2的乳腺癌患者和健康对照;
[0237]
(c10)肿瘤大小为t3和/或t4的乳腺癌患者和健康对照;
[0238]
(c11)分子分型为luminal a型的乳腺癌患者和健康对照;
[0239]
(c12)分子分型为luminal b型的乳腺癌患者和健康对照;
[0240]
(c13)分子分型为her2扩增型的乳腺癌患者和健康对照;
cpg位点进行甲基化水平和乳腺癌的相关性分析。
[0257]
ptch2_a片段(seq id no.1)位于hg19参考基因组chr1:45285516-45286616,正义链。
[0258]
ptch2_b片段(seq id no.2)位于hg19参考基因组chr1:45307298-45309298,反义链。
[0259]
ptch2_c片段(seq id no.3)位于hg19参考基因组chr1:45303415-45306414,正义链。
[0260]
ptch2_d片段(seq id no.4)位于hg19参考基因组chr1:45296445-45298445,正义链。
[0261]
ptch2_e片段(seq id no.5)位于hg19参考基因组chr1:45291204-45293204,正义链。
[0262]
ptch2_f片段(seq id no.6)位于hg19参考基因组chr1:45287930-45288460,正义链。
[0263]
ptch2_a片段中的位点信息如表1所示。
[0264]
ptch2_b片段中的位点信息如表2所示。
[0265]
ptch2_c片段中的位点信息如表3所示。
[0266]
ptch2_d片段中的位点信息如表4所示。
[0267]
ptch2_e片段中的位点信息如表5所示。
[0268]
ptch2_f片段中的位点信息如表6所示。
[0269]
表1、ptch2_a片段中cpg位点信息
[0270][0271][0272]
表2、ptch2_b片段中cpg位点信息
[0273]
cpg位点cpg位点在序列中的位置ptch2_b_1seq id no.2自5’端第31-32位ptch2_b_2seq id no.2自5’端第72-73位ptch2_b_6seq id no.2自5’端第124-125位
ptch2_b_7seq id no.2自5’端第173-174位ptch2_b_9seq id no.2自5’端第226-227位ptch2_b_10seq id no.2自5’端第230-231位ptch2_b_11seq id no.2自5’端第234-235位ptch2_a_12seq id no.2自5’端第236-237位ptch2_a_13seq id no.2自5’端第246-247位ptch2_a_14seq id no.2自5’端第304-305位
[0274]
表3、ptch2_c片段中cpg位点信息
[0275]
cpg位点cpg位点在序列中的位置ptch2_c_1seq id no.3自5’端第29-30位ptch2_c_2seq id no.3自5’端第41-42位ptch2_c_3seq id no.3自5’端第74-75位ptch2_c_4seq id no.3自5’端第84-85位ptch2_c_5seq id no.3自5’端第91-92位ptch2_c_6seq id no.3自5’端第108-109位ptch2_c_7seq id no.3自5’端第116-117位
[0276]
表4、ptch2_d片段中cpg位点信息
[0277]
cpg位点cpg位点在序列中的位置ptch2_d_1seq id no.4自5’端第117-118位ptch2_d_2seq id no.4自5’端第152-153位ptch2_d_3seq id no.4自5’端第163-164位ptch2_d_4seq id no.4自5’端第202-203位ptch2_d_7seq id no.4自5’端第260-261位ptch2_d_8seq id no.4自5’端第267-268位
[0278]
表5、ptch2_e片段中cpg位点信息
[0279][0280][0281]
表6、ptch2_f片段中cpg位点信息
[0282]
cpg位点cpg位点在序列中的位置
ptch2_f_1seq id no.6自5’端第27-28位ptch2_f_2seq id no.6自5’端第75-76位ptch2_f_3seq id no.6自5’端第114-115位ptch2_f_4seq id no.6自5’端第278-279位
[0283]
针对六个片段(ptch2_a片段、ptch2_b片段、ptch2_c片段、ptch2_d片段、ptch2_e 片段和ptch2_f片段)设计特异性pcr引物,如表7所示。其中,seq id no.7、seq id no.9、 seq id no.11、seq id no.13、seq id no.15和seq id no.17为正向引物,seq id no.8、 seq id no.10、seq id no.12、seq id no.14、seq id no.16和seq id no.18为反向引物; seq id no.7、seq id no.9、seq id no.11、seq id no.13、seq id no.15和seq id no.17 中自5’端第1至10位为非特异性标签,seq id no.9、seq id no.13、seq id no.15和seqid no.17第11至35位,seq id no.11第11至36位,seq id no.7第11至37位为特异性引物序列;seq id no.8、seq id no.10、seq id no.12、seq id no.14、seq id no.16和seqid no.18中自5’端第1至31位为非特异性标签,seq id no.8、seq id no.10、seq id no.12、 seq id no.14、seq id no.16和seq id no.18第32至56位为特异性引物序列。引物序列中不包含snp和cpg位点。
[0284]
表7、ptch2甲基化引物序列
[0285]
扩增片段引物序列号引物序列(5
’–3’
)ptch2_aseq id no.7aggaagagaggggagtgtaagtattttgagggtatttptch2_aseq id no.8cagtaatacgactcactatagggagaaggctaccatctcctccaaaaaaaataaaaptch2_bseq id no.9aggaagagagttttgtaaggtttgtgatgttttgaptch2_bseq id no.10cagtaatacgactcactatagggagaaggctaaaacaaaatttccccatattaaccptch2_cseq id no.11aggaagagaggaggttagtatggttaatatggtgatptch2_cseq id no.12cagtaatacgactcactatagggagaaggctccaacataaaataaactaaaccccaptch2_dseq id no.13aggaagagaggagttttggggaagtttatgtttttptch2_dseq id no.14cagtaatacgactcactatagggagaaggctacaaaccccacaacctacaatctacptch2_eseq id no.15aggaagagagtttatagagggtttttggtaggaggptch2_eseq id no.16cagtaatacgactcactatagggagaaggctaactcaacccttaaaatttacccaaptch2_fseq id no.17aggaagagagattagggaggtttatgatagttgggptch2_fseq id no.18cagtaatacgactcactatagggagaaggctcttaaataaaaaacatcctcctccc
[0286]
实施例2、ptch2基因甲基化检测及结果分析
[0287]
一、研究样本
[0288]
于2010年1月1日至2020年6月30日收集乳腺癌患者进行调查,总共纳入224名乳腺癌患者。于2018年1月1日至2020年6月30日对年龄匹配的无癌健康女性进行调查,总共纳入223名年龄匹配的无癌健康女性。
[0289]
乳腺癌患者调查内容包括收集调查对象的一般人口学资料信息如年龄、籍贯、民族等;询问疾病史、用药史、家族史等,主要包括癌症史,癌症家族史,初潮年龄等;收集组织病理检查结果如乳腺癌种类、肿瘤大小、淋巴转移数目、是否转移、临床分期。乳腺癌类型定义为:非浸润性导管内癌:癌细胞局限在乳腺导管内且未突破导管壁基底膜;非浸润性原位癌:小叶原位癌,癌细胞未突破末梢乳管或腺体泡基底膜;早期浸润性导管癌:癌细胞突破管壁基底膜并开始向间质浸润;早期浸润性小叶癌:癌细胞突破末梢乳管或腺体泡基底膜,
但仍局限于小叶内;浸润性癌:包括乳头状癌、髓样癌、小管癌、腺样囊性癌、粘液腺癌、大汗腺样癌、鳞状细胞癌等分化程度较高的类型,或包括浸润性导管癌、浸润性小叶癌、硬癌、髓样癌、单纯癌、腺癌等分化层度较低的类型。根据乳腺癌tnm分级规则,肿瘤大小分级定义为:导管原位癌和小叶原位癌为tis;肿瘤直径≤2cm为t1;肿瘤直径>2cm且≤5cm 为t2;肿瘤直径>5cm为t3,肿瘤侵犯胸壁或皮肤为t4。淋巴结转移分级定义:区域淋巴结无转移为n0;同侧淋巴结转移且可活动为n1;同侧转移性淋巴结相互融合或与其他组织固定或临床无证据显示腋淋巴结转移的情况下,存在临床明显的内乳淋巴结转移为n2;同侧锁骨下淋巴结转移;或有临床证据显示腋淋巴结转移的情况下,存在临床明显的内乳淋巴结转移;或同侧锁骨上淋巴结转移,伴或不伴腋淋巴结或内乳淋巴结转移为n3。是否转移定义为:无远处转移为m0,有远处转移为m1。根据乳腺癌tnm分级规则,乳腺癌患者临床分期定义为:0期为tisn0m0乳腺癌患者;ⅰ期为t1n0m0乳腺癌患者;ⅱ期包含t0n1m0、 t1n1m0、t2n0m0;ⅲ期为t0n2m0、t1n2m0、t2n2m0、t3n1m0、t3n2m0、t4n0m0、 t4n1m0、t4n2m0、tisn3m0、t1n3m0、t2n3m0、t3n3m0和t4n3m0分级的乳腺癌患者;ⅳ期为任何肿瘤大小分级和淋巴结分级,且伴有远端转移的乳腺癌患者。免疫组化结果如雌激素受体(er)水平、孕激素受体(pr)水平、人表皮生长因子受体-2(her2)水平和ki67蛋白水平等。er阳性的定义为免疫组化er阳性的细胞核占比≥1%;pr阳性的定义为免疫组化pr阳性的细胞核占比≥1%;her2阳性的定义为免疫组化在≥10%的肿瘤细胞中观察到弱至中等的完全膜染色;ki67蛋白水平高表达的定义为免疫组化在≥30%的细胞中观察到中等至强的核染色;p53基因突变率高的定义为通过免疫组化检测组织中p53突变阳性染色细胞的比例≥30%;p53基因突变率低的定义为通过免疫组化检测组织中p53突变阳性染色细胞的比例<30%。通过免疫组化检测突变型p53基因的蛋白产物,由于突变型p53蛋白产物半衰期较未突变的p53蛋白产物长,故由此判断p53的突变情况。需要指出的是临床上仅可检测出存积且稳定的突变型p53蛋白。lumina a型乳腺癌的定义为hr阳性,her2阴性;luminab型乳腺癌定义为hr阳性,her2阳性或阴性(分为两种类型:1:er阳性,her2阴性, pr阴性或低表达,ki-67小于14%;2:er阳性,her2阳性,pr和ki-67指标不限定); her2扩增型乳腺癌定义为hr阴性,her2阳性;基底样型乳腺癌的定义为er、pr和her2 均为阴性,又称为三阴性乳腺癌。hr阳性指整张组织切片中阳性染色的肿瘤细胞占所有肿瘤细胞的比例,当≥1%的肿瘤细胞核呈现不同程度的着色时,为阳性。阴性:当<1%的肿瘤细胞核呈现不同程度的着色或完全无着色时,判定为阴性。(引用自考我国《乳腺癌雌、孕激素受体免疫组织化学检测指南(2015版)》)。被调查者要求清晨空腹采血,收集外周静脉血(抗凝血1管),用于检测生化指标主要包括红细胞计数、红细胞比积、白细胞计数、淋巴细胞百分比、中心粒细胞百分比、嗜碱性粒细胞百分比、嗜酸性粒细胞百分比、单核细胞百分比、血小板计数、血小板平均体积、平均血红蛋白含量等。
[0290]
无癌健康女性定义为现在及曾经均没有患过癌症且血常规指标均在参考范围内的女性,对其调查内容包括一般人口学资料信息如年龄、籍贯和民族等;被调查者要求清晨空腹采血,收集外周静脉血(抗凝血1管),用于检测生化指标主要包括红细胞计数、红细胞比积、白细胞计数、淋巴细胞百分比、中心粒细胞百分比、嗜碱性粒细胞百分比、嗜酸性粒细胞百分比、单核细胞百分比、血小板计数、血小板平均体积、平均血红蛋白含量等。
[0291]
乳腺癌病例平均年龄38.65
±
5.78岁,对照平均年龄38.37
±
7.11岁,两组年龄差异无统计学意义(p>0.05)。
[0292]
二、甲基化检测
[0293]
1、提取血液样本的总dna。
[0294]
2、将步骤1制备的血液样本总dna进行重亚硫酸盐处理后,未发生甲基化的胞嘧啶(c) 被转化成尿嘧啶(u),而甲基化的胞嘧啶保持不变,即原来cpg位点的c碱基经重亚硫酸盐处理后转化为c或u。
[0295]
3、以步骤2经过重亚硫酸盐处理的dna为模板,采用表7中的6对特异引物对通过dna 聚合酶按照常规pcr反应要求的反应体系进行pcr扩增,6对引物都采用相同的常规pcr 体系,且6对引物都按照以下程序进行扩增。
[0296]
pcr反应程序为:95℃,4min
→
(95℃,20s
→
56℃,30s
→
72℃,2min)45个循环
→
72℃, 5min
→
4℃,1h。
[0297]
4、取步骤3的扩增产物,通过飞行时间质谱进行dna甲基化分析,具体方法如下:
[0298]
(1)向5μl pcr产物中加入2μl虾碱性磷酸盐(sap)溶液(0.3ml sap[0.5u] 1.7ml h2o)然后按照以下程序在pcr仪中孵育(37℃,20min
→
85℃,5min
→
4℃,5min);
[0299]
(2)取出2μl步骤(1)得到的sap处理后的产物,根据说明书加入5μl t-cleavage反应体系中,然后在37℃孵育3h;
[0300]
(3)取步骤(2)的产物,加入19μl去离子水,再用6μg resin在旋转摇床进行去离子化孵育 1h;
[0301]
(4)2000rpm室温离心5min,将微量上清由nanodispenser机械手臂上样384spectrochip;
[0302]
(5)飞行时间质谱分析;获得的数据用spectroacquire v3.3.1.3软件收集,通过 massarrayepityper v1.2软件实现可视化。
[0303]
上述飞行时间质谱检测使用的试剂均来试剂盒(t-cleavage masscleave reagent autokit,货号:10129a);上述飞行时间质谱检测使用的检测仪器为massarray
○
r analyzer chipprep module 384,型号:41243;上述数据分析软件为检测仪器自带软件。
[0304]
三、质量控制
[0305]
问卷调查:制定严谨的调查问卷,统一衡量标准;调查前对调查员统一培训和考核;正式调查前进行预调查,及时发现和总结问题;问卷由专门的质控人员现场质控,质量不合格的问卷退回调查员重新对研究对象调查;问卷资料双轨录入,并进行一致性检验;现场收集的血标本及时送检。严格按照实验操作要求进行,定期对操作环境紫外消毒;正式实验前进行预实验;随机抽取5%的样本重复飞行时间质谱检测,保证结果一致率达99%以上。双人判读实验结果,整理甲基化数据,保证数据的真实准确性。通过质谱实验,共获得36个可以区别的g峰图。采用spectroacquire v3.3.1.3软件根据含g峰和a峰面积比较,计算甲基化水平(spectroacquire v3.3.1.3软件可自动通过计算峰面积得到每个样本在每个cpg位点的甲基化水平)。
[0306]
四、统计分析
[0307]
呈正态分布的计量资料采用均数
±
标准差表示,计数资料用率表示,应用成组t检验进行病例组和对照组一般人口学资料分布差异的比较分析。呈非正态分布的计量资料采用中位数 (四分位数间距)表示,采用mann-whitney u检验进行病例组和对照组之间差异
比较。非条件 logistic回归模型用于ptch2甲基化和乳腺癌之间的关联分析,校正年龄,以每10%甲基化降低计算比值比(odds ratio,or)和95%可信区间(confidence interval,ci)。用spearman秩相关系数评估两变量之间的统计相关性。通过logistic回归和受试者工作特征曲线(receiveroperating characteristic curve,roc)评价多个cpg位点的组合对乳腺癌预警和早期诊断的价值。以双侧p<0.05为差异有统计学意义,所有数据均通过spss 25.0进行统计分析。
[0308]
五、结果分析
[0309]
1、ptch2甲基化与乳腺癌的关联分析
[0310]
ptch2甲基化与乳腺癌的关联分析结果显示,乳腺癌病例ptch2_a片段4个位点 [cpg_1.2.3.4(0.90vs.0.87)、cpg_7.8.9.10.11(0.92vs.0.87)、cpg_13(0.20vs.0.17)、cpg_19(0.25vs.0.21)]、ptch2_b片段4个位点[cpg_1(0.23vs.0.19)、cpg_2(0.54vs.0.51)、 cpg_7(0.54vs.0.51)、cpg_9.10.11.12(0.94vs.0.90)]、ptch2_c片段5个位点[cpg_1(0.26vs. 0.22)、cpg_2(0.38vs.0.34)、cpg_3(0.24vs.0.22)、cpg_4(0.13vs.0.11)、cpg_5(0.19vs. 0.15)]、ptch2_d片段5个位点[cpg_1(0.64vs.0.60)、cpg_2(0.26vs.0.23)、cpg_3(0.64vs. 0.60)、cpg_4(0.26vs.0.23)、cpg_7(0.29vs.0.26)]、ptch2_e片段5个位点[cpg_1(0.24vs. 0.22)、cpg_3(0.36vs.0.33)、cpg_4.5(0.29vs.0.26)、cpg_8.9(0.32vs.0.30)、cpg_10.11(0.15 vs.0.13)]和ptch2_f片段3个位点[cpg_2(0.28vs.0.25)、cpg_3(0.38vs.0.34)、cpg_4(0.53 vs.0.52)]的甲基化水平显著高于对照,具体结果见表8。而在将发病年龄按≤35岁,35岁至 45岁以及≥45岁分层以后,乳腺癌发病年龄≥45岁的乳腺癌病例与≥45岁健康无癌对照cpg 位点甲基化水平分别为ptch2_a片段5个位点[cpg_1.2.3.4(0.89vs.0.86)、 cpg_7.8.9.10.11(0.89vs.0.86)、cpg_13(0.19vs.0.16)、cpg_14.15(0.16vs.0.14)、cpg_19(0.27 vs.0.25)]、ptch2_b片段7个位点[cpg_1(0.16vs.0.18)、cpg_2(0.52vs.0.49)、cpg_6(0.74 vs.0.73)、cpg_7(0.52vs.0.50)、cpg_9.10.11.12(0.93vs.0.92)、cpg_13(1.00vs.1.00)、cpg_14 (1.00vs.1.00)]、ptch2_c片段7个位点[cpg_1(0.22vs.0.20)、cpg_2(0.35vs.0.32)、cpg_3 (0.22vs.0.20)、cpg_4(0.11vs.0.09)、cpg_5(0.16vs.0.14)、cpg_6(0.29vs.0.27)、cpg_7(0.10 vs.0.08)]、ptch2_d片段6个位点[cpg_1(0.61vs.0.59)、cpg_2(0.23vs.0.22)、cpg_3(0.61 vs.0.59)、cpg_4(0.24vs.0.21)、cpg_7(0.20vs.0.19)、cpg_8(0.34vs.0.33)]、ptch2_e片段7个位点[cpg_1(0.24vs.0.21)、cpg_3(0.33vs.0.15)、cpg_4.5(0.20vs.0.19)、cpg_6(0.10 vs.0.09)、cpg_7(0.15vs.0.13)、cpg_8.9(0.28vs.0.26)、cpg_10.11(0.14vs.0.12)]和ptch2_f 片段4个位点[cpg_1(0.29vs.0.27)、cpg_2(0.26vs.0.24)、cpg_3(0.37vs.0.35)、cpg_4(0.51 vs.0.50)];logistic回归结果显示,校正白细胞亚型比例、年龄等协变量后,ors(95%cis)每-10%甲基化分别为ptch2_a[cpg_1.2.3.4:0.80(0.66
–
0.95),p=0.021;cpg_7.8.9.10.11:0.86(0.58
ꢀ–
0.91),p=0.025;cpg_13:0.81(0.77
–
0.95),p=0.029;cpg_14.15:0.61(0.32
–
0.86),p=0.042; cpg_19:0.75(0.56
–
0.86),p=0.031]、ptch2_b[cpg_1:1.03(0.51
–
2.06),p=0.934;cpg_2: 0.48(0.22
–
0.94),p=0.043;cpg_6:0.97(0.86
–
1.78),p=0.085;cpg_7:0.48(0.22
–
0.92), p=0.036;cpg_9.10.11.12:0.74(0.42
–
0.96),p=0.033;cpg_13:1.00(1.00
–
1.00),p=0.545,cpg_14:1.00(1.00
–
1.00),p=1.000]、ptch2_c[cpg_1:0.98(0.97
–
0.99),p=
0.043;cpg_2: 0.76(0.59
–
0.88),p=0.029;cpg_3:0.97(0.86
–
1.78),p=0.175;cpg_4:0.90(0.77
–
0.99), p=0.049;cpg_5:0.82(0.35
–
0.90),p=0.029;cpg_6:0.84(0.64
–
0.92),p=0.027;cpg_7: 0.99(0.77
–
1.29),p=0.884]、ptch2_d[cpg_1:0.71(0.56
–
0.90),p=0.022;cpg_2:0.89(0.79
ꢀ–
0.98),p=0.046;cpg_3:0.71(0.56
–
0.90),p=0.022;cpg_4:0.87(0.72
–
0.97),p=0.046; cpg_7:0.99(0.77
–
1.29),p=0.964;cpg_8:0.72(0.52
–
1.01),p=0.597]、ptch2_e[cpg_1: 0.74(0.42
–
0.93),p=0.041;cpg_3:0.82(0.66
–
0.91),p=0.035;cpg_4.5:0.92(0.50
–
1.70), p=0.795;cpg_6:0.55(0.12
–
0.99),p=0.049;cpg_7:0.72(0.52
–
1.01),p=0.597;cpg_8.9: 0.78(0.36
–
0.98),p=0.045;cpg_10.11:0.77(0.50
–
0.91),p=0.045]和ptch2_f[cpg_1: 0.82(0.71
–
0.92),p=0.037;cpg_2:0.74(0.43
–
0.91),p=0.024;cpg_3:0.84(0.75
–
0.97), p=0.046;cpg_4:0.91(0.82
–
0.93),p=0.040](表9)。乳腺癌发病年龄35至45岁的乳腺癌病例与35至45岁的健康无癌对照上述cpg位点甲基化水平分别为ptch2_a片段5个位点 [cpg_1.2.3.4(0.91vs.0.87)、cpg_7.8.9.10.11(0.92vs.0.89)、cpg_13(0.19vs.0.18)、 cpg_14.15(0.16vs.0.16)、cpg_19(0.26vs.0.24)]、ptch2_b片段7个位点[cpg_1(0.22vs. 0.19)、cpg_2(0.54vs.0.52)、cpg_6(0.75vs.0.74)、cpg_7(0.54vs.0.52)、cpg_9.10.11.12(0.94 vs.0.90)、cpg_13(1.00vs.1.00)、cpg_14(1.00vs.1.00)]、ptch2_c片段7个位点[cpg_1(0.25 vs.0.22)、cpg_2(0.37vs.0.34)、cpg_3(0.24vs.0.22)、cpg_4(0.13vs.0.11)、cpg_5(0.20vs. 0.15)、cpg_6(0.31vs.0.29)、cpg_7(0.13vs.0.11)]、ptch2_d片段6个位点[cpg_1(0.64vs. 0.60)、cpg_2(0.26vs.0.23)、cpg_3(0.64vs.0.60)、cpg_4(0.26vs.0.23)、cpg_7(0.28vs. 0.25)、cpg_8(0.33vs.0.32)]、ptch2_e片段7个位点[cpg_1(0.24vs.0.22)、cpg_3(0.35vs. 0.18)、cpg_4.5(0.28vs.0.27)、cpg_6(0.12vs.0.12)、cpg_7(0.18vs.0.16)、cpg_8.9(0.32vs. 0.30)、cpg_10.11(0.15vs.0.13)]和ptch2_f片段4个位点[cpg_1(0.34vs.0.31)、cpg_2(0.23 vs.0.20)、cpg_3(0.38vs.0.36)、cpg_4(0.55vs.0.52)];logistic回归结果显示,校正白细胞亚型比例、年龄等协变量后,ors(95%cis)每-10%甲基化分别为ptch2_a[cpg_1.2.3.4: 0.82(0.66
–
0.95),p=0.001;cpg_7.8.9.10.11:0.82(0.58
–
0.87),p=0.005;cpg_13:0.81(0.77
ꢀ–
0.95),p=0.009;cpg_14.15:0.90(0.54
–
3.24),p=0.420;cpg_19:0.83(0.62
–
0.86),p=0.011]、 ptch2_b[cpg_1:0.71(0.54
–
0.98),p=0.048;cpg_2:0.76(0.48
–
0.86),p=0.011;cpg_6: 0.95(0.72
–
1.26),p=0.736;cpg_7:0.82(0.66
–
0.98),p=0.014;cpg_9.10.11.12:0.66(0.50
–ꢀ
0.86),p=0.009;cpg_13:1.00(1.00
–
1.00),p=0.738;cpg_14:1.00(1.00
–
1.00),p=1.000]、 ptch2_c[cpg_1:0.36(0.23
–
0.70),p=0.024;cpg_2:0.75(0.48
–
0.91),p=0.012;cpg_3: 0.85(0.72
–
0.95),p=0.048;cpg_4:0.55(0.45
–
0.82),p=0.007;cpg_5:0.84(0.51
–
0.92), p=0.007;cpg_6:0.74(0.68
–
0.90),p=0.033;cpg_7:0.85(0.66
–
0.95),p=0.041]、 ptch2_d[cpg_1:0.96(0.94
–
0.98),p=0.009;cpg_2:0.53(0.33
–
0.86),p=0.013;cpg_3: 0.96(0.94
–
0.98),p=0.009;cpg_4:0.53(0.33
–
0.86),p=0.013;cpg_7:0.61(0.42
–
0.92), p=0.044;cpg_8:0.99(0.79
–
1.24),p=0.937]、ptch2_e[cpg_1:0.86(0.66
–
0.92),p=0.043; cpg_3:0.89(0.85
–
0.98),p=0.010;cpg_4.5:0.89(0.85
–
0.98),p=0.049;cpg_6:1.03(0.67
ꢀ–
1.60),p=0.887;cpg_7:1.22(0.87
–
1.70),p=0.244;cpg_8.9:0.91(0.71
–
94),p=0.041; cpg_10.11:0.65(0.37
–
0.85),p=0.027]和
ptch2_f[cpg_1:0.67(0.92
–
0.49),p=0.012;cpg_2: 0.57(0.42
–
0.78),p=4.35e-4;cpg_3:0.45(0.36
–
0.68),p=2.50e-5;cpg_4:0.66(0.50
–
0.88), p=0.035](表10)。乳腺癌发病年龄≤35岁的乳腺癌病例与对照上述cpg位点甲基化水平分别为ptch2_a片段5个位点[cpg_1.2.3.4(0.94vs.0.87)、cpg_7.8.9.10.11(0.94vs.0.90)、 cpg_13(0.23vs.0.19)、cpg_14.15(0.19vs.0.16)、cpg_19(0.27vs.0.24)]、ptch2_b片段7个位点[cpg_1(0.23vs.0.18)、cpg_2(0.58vs.0.55)、cpg_6(0.78vs.0.74)、cpg_7(0.58vs.0.54)、 cpg_9.10.11.12(0.94vs.0.90)、cpg_13(0.99vs.0.99)、cpg_14(1.00vs.1.00)]、ptch2_c片段7个位点[cpg_1(0.29vs.0.26)、cpg_2(0.42vs.0.38)、cpg_3(0.26vs.0.22)、cpg_4(0.14vs. 0.11)、cpg_5(0.22vs.0.16)、cpg_6(0.35vs.0.32)、cpg_7(0.15vs.0.11)]、ptch2_d片段6 个位点[cpg_1(0.67vs.0.62)、cpg_2(0.30vs.0.25)、cpg_3(0.67vs.0.62)、cpg_4(0.30vs. 0.25)、cpg_7(0.19vs.0.18)、cpg_8(0.32vs.0.31)]、ptch2_e片段7个位点[cpg_1(0.27vs. 0.20)、cpg_3(0.33vs.0.20)、cpg_4.5(0.37vs.0.29)、cpg_6(0.15vs.0.12)、cpg_7(0.22vs. 0.19)、cpg_8.9(0.35vs.0.29)、cpg_10.11(0.15vs.0.12)]和ptch2_f片段4个位点[cpg_1 (0.35vs.0.31)、cpg_2(0.25vs.0.21)、cpg_3(0.40vs.0.36)、cpg_4(0.60vs.0.56)];logistic回归结果显示,校正白细胞亚型比例、年龄等协变量后,ors(95%cis)每-10%甲基化分别为 ptch2_a[cpg_1.2.3.4:0.32(0.23
–
0.63),p=2.62e-4;cpg_7.8.9.10.11:0.78(0.70
–
0.89), p=6.35e-4;cpg_13:0.71(0.60
–
0.85),p=7.44e-4;cpg_14.15:0.57(0.44
–
0.72),p=1.26e-4; cpg_19:0.51(0.45
–
0.58),p=2.10e-4]、ptch2_b[cpg_1:0.60(0.37
–
0.96),p=0.035;cpg_2: 0.76(0.62
–
0.91),p=0.004;cpg_6:0.81(0.66
–
0.92),p=0.021;cpg_7:0.66(0.42
–
0.84), p=8.00e-4;cpg_9.10.11.12:0.67(0.42
–
0.88),p=0.002;cpg_13:0.62(0.29
–
1.34),p=0.223; cpg_14:1.00(1.00
–
1.00),p=1.000]、ptch2_c[cpg_1:0.32(0.10
–
0.54),p=1.40e-5;cpg_2: 0.19(0.01
–
0.41),p=6.26e-4;cpg_3:0.56(0.27
–
0.74),p=0.001;cpg_4:0.63(0.50
–
0.78), p=6.93e-4;cpg_5:0.64(0.32
–
0.82),p=7.21e-4;cpg_6:0.72(0.53
–
0.90),p=0.005;cpg_7: 0.63(0.50
–
0.78),p=6.93e-4]、ptch2_d[cpg_1:0.48(0.27
–
0.67),p=7.22e-4;cpg_2:0.36(0.22
ꢀ–
0.64),p=4.23e-4;cpg_3:0.48(0.27
–
0.67),p=7.22e-4;cpg_4:0.36(0.22
–
0.64),p=4.23e-4; cpg_7:1.00(0.72
–
1.42),p=0.939;cpg_8:0.99(0.79
–
1.24),p=0.937]、ptch2_e[cpg_1: 0.63(0.43
–
0.93),p=0.019;cpg_3:0.71(0.56
–
0.90),p=0.005;cpg_4.5:0.59(0.41
–
0.86), p=0.006;cpg_6:0.60(0.34
–
0.99),p=0.026;cpg_7:0.77(0.51
–
0.98),p=0.033;cpg_8.9: 0.50(0.31
–
0.83),p=0.006;cpg_10.11:0.07(0.02
–
0.23),p=9.00e-5]和ptch2_f[cpg_1: 0.44(0.27
–
0.67),p=0.001;cpg_2:0.64(0.37
–
0.93),p=1.60e-4;cpg_3:0.52(0.33
–
0.93), p=6.31e-6;cpg_4:0.50(0.32
–
0.61),p=0.002](表11)。
[0311]
表8、224例乳腺癌病例和223例健康对照ptch2基因cpg位点的甲基化水平比较
[0312][0313][0314]
注:iqr,四分位数间距;ptch2_a:外显子1;ptch2_b:内含子1;ptch2_c:内含子2;ptch2_d: 外显子5;ptch2_e:外显子19;ptch2_f:外显子22。
[0315]
表9、35例乳腺癌病例(发病年龄≥45岁)和48例健康对照(年龄≥45岁)ptch2基因
cpg位点的甲基化水平比较
[0316][0317][0318]
注:iqr,四分位数间距;ptch2_a:外显子1;ptch2_b:内含子1;ptch2_c:内含子2;ptch2_d: 外显子5;ptch2_e:外显子19;ptch2_f:外显子22。
[0319]
表10、134例乳腺癌病例(发病年龄35岁至45岁)和117例健康对照(年龄35岁至45岁)ptch2基因 cpg位点的甲基化水平比较
[0320][0321][0322]
注:iqr,四分位数间距;ptch2_a:外显子1;ptch2_b:内含子1;ptch2_c:内含子2;
ptch2_d: 外显子5;ptch2_e:外显子19;ptch2_f:外显子22。
[0323]
表11、55例乳腺癌病例(发病年龄≤35岁)和58例健康对照(年龄≤35岁)ptch2基因cpg位点的甲基化水平比较
[0324][0325]
[0326]
注:iqr,四分位数间距;or:优势比;ci:可信区间;ptch2_a:外显子1;ptch2_b:内含子1; ptch2_c:内含子2;ptch2_d:外显子5;ptch2_e:外显子19;ptch2_f:外显子22;
[0327]
2、ptch2甲基化与乳腺癌临床分期的相关分析
[0328]
将乳腺癌病例按临床分期,将病例分成stage0&ⅰ、stageⅱ和stageⅲ&ⅳ三组,将三组病例分别与健康对照比较ptch2基因甲基化水平差异,结果表明,ptch2_a片段5个位点 (cpg_1.2.3.4、cpg_7.8.9.10.11、cpg_13、cpg_14.15、cpg_19)、ptch2_b片段7个位点 (cpg_1、cpg2、cpg_6、cpg_7、cpg_9.10.11.12、cpg_13、cpg_14)、ptch2_c片段7 个位点(cpg_1、cpg_2、cpg_3、cpg_4、cpg_5、cpg_6、cpg_7)、ptch2_d片段6个位点(cpg_1、cpg_2、cpg_3、cpg_4、cpg_7、cpg_8)、ptch2_e片段7个位点(cpg_1、 cpg_3、cpg_4.5、cpg_6、cpg_7、cpg_8.9、cpg_10.11)和ptch2_f片段4个位点(cpg_1、 cpg_2、cpg_3、cpg_4)甲基化程度在对照与三组病例存在显著差异(表12)。
[0329]
表12、乳腺癌病例不同临床分期ptch2基因cpg位点甲基化水平比较(224例乳腺癌病例vs.223例健康对照)
[0330]
[0331][0332]
注:
“‑”
表示数值缺失,因为两组数值极为接近,无法计算出对应的p值。
[0333]
3、ptch2甲基化与乳腺癌肿瘤大小的相关分析
[0334]
将乳腺癌病例按肿瘤大小,将病例分成tis&t1、t2和t3&t4三组,将三组分别与健康对照ptch2基因甲基化水平进行比较,结果表明,ptch2_a片段5个位点(cpg_1.2.3.4、 cpg_7.8.9.10.11、cpg_13、cpg_14.15、cpg_19)、ptch2_b片段7个位点(cpg_1、cpg2、 cpg_6、cpg_7、cpg_9.10.11.12、cpg_13、cpg_14)、ptch2_c片段7个位点(cpg_1、cpg_2、 cpg_3、cpg_4、cpg_5、cpg_6、cpg_7)、ptch2_d片段6个位点(cpg_1、cpg_2、cpg_3、 cpg_4、cpg_7、cpg_8)、ptch2_e片段7个位点(cpg_1、cpg_3、cpg_4.5、cpg_6、cpg_7、 cpg_8.9、cpg_10.11)和ptch2_f片段4个位点(cpg_1、cpg_2、cpg_3、cpg_4)三组与健康对照的甲基化程度均存在显著差异(表13)。
[0335]
表13、乳腺癌病例不同肿瘤大小ptch2基因cpg位点甲基化水平比较(224例乳腺癌病例vs.223例健康对照)
[0336][0337]
注:
“‑”
表示数值缺失,因为两组数值极为接近,无法计算出对应的p值。
[0338]
4、ptch2甲基化与乳腺癌分子分型的相关分析
[0339]
将乳腺癌病例按分子分型,将病例分成luminal a型、luminal b型、her2扩增型和基底样型四组,分别将四组与健康对照ptch2基因甲基化水平进行比较,结果表明,ptch2_a 片段5个位点(cpg_1.2.3.4、cpg_7.8.9.10.11、cpg_13、cpg_14.15、cpg_19)、ptch2_b片段7个位点(cpg_1、cpg2、cpg_6、cpg_7、cpg_9.10.11.12、cpg_13、cpg_14)、ptch2_c 片段7个位点(cpg_1、cpg_2、cpg_3、cpg_4、cpg_5、cpg_6、cpg_7)、ptch2_d片段 6个位点(cpg_1、cpg_2、cpg_3、cpg_4、cpg_7、cpg_8)、ptch2_e片段7个位点(cpg_1、 cpg_3、cpg_4.5、cpg_6、cpg_
7、cpg_8.9、cpg_10.11)和ptch2_f片段4个位点(cpg_1、 cpg_2、cpg_3、cpg_4)各分子分型乳腺癌病例与健康对照均存在显著性差异(表14)。
[0340]
表14、乳腺癌病例不同分子分型ptch2基因cpg位点甲基化水平比较(224例乳腺癌病例vs.223例健康对照)
[0341][0342][0343]
注:
“‑”
表示数值缺失,因为两组数值极为接近,无法计算出对应的p值。
[0344]
5、ptch2甲基化与乳腺癌ki67蛋白表达量的相关分析
[0345]
将乳腺癌病例按ki67蛋白表达量,将病例分成ki67蛋白表达量高(免疫组化检测组织中 ki67染色细胞的比例≥30%)和ki67蛋白表达量低(免疫组化检测组织中ki67染色细胞的比例<30%)两组,分别将比较两组与健康对照ptch2基因甲基化水平的差异,结果表明, ptch2_a片段5个位点(cpg_1.2.3.4、cpg_7.8.9.10.11、cpg_13、cpg_14.15、cpg_19)、 ptch2_b片段7个位点(cpg_1、cpg2、cpg_6、cpg_7、cpg_9.10.11.12、cpg_13、cpg_14)、 ptch2_c片段7个位点(cpg_1、cpg_2、cpg_3、cpg_4、cpg_5、cpg_6、cpg_7)、ptch2_d 片段6个位点(cpg_1、cpg_2、cpg_3、cpg_4、cpg_7、cpg_8)、ptch2_e片段7个位点 (cpg_1、cpg_3、cpg_4.5、cpg_6、cpg_7、cpg_8.9、cpg_10.11)和ptch2_f片段4个位点(cpg_1、cpg_2、cpg_3、cpg_4),两组与健康对照甲基化程度均存在显著性差异(表 15)。
[0346]
表15、乳腺癌病例不同ki67蛋白表达量ptch2基因cpg位点甲基化水平比较(224例乳腺癌病例vs.223 例健康对照)
[0347]
[0348][0349]
注:
“‑”
表示数值缺失,因为两组数值极为接近,无法计算出对应的p值。
[0350]
6、ptch2甲基化与乳腺癌p53基因突变状态的相关分析
[0351]
将乳腺癌病例按p53基因突变状态,将病例分成p53基因突变高(免疫组化检测组织中 p53突变阳性染色细胞的比例≥30%)和p53基因突变率低(免疫组化检测组织中p53突变阳性染色细胞的比例<30%)两组,分别比较两组与健康对照ptch2基因甲基化水平间差异,结果表明,ptch2_a片段5个位点(cpg_1.2.3.4、cpg_7.8.9.10.11、cpg_13、cpg_14.15、 cpg_19)、ptch2_b片段7个位点(cpg_1、cpg2、cpg_6、cpg_7、cpg_9.10.11.12、cpg_13、 cpg_14)、ptch2_c片段7个位点(cpg_1、cpg_2、cpg_3、cpg_4、cpg_5、cpg_6、cpg_7)、 ptch2_d片段6个位点(cpg_1、cpg_2、cpg_3、cpg_4、cpg_7、cpg_8)、ptch2_e片段7个位点(cpg_1、cpg_3、cpg_4.5、cpg_6、cpg_7、cpg_8.9、cpg_10.11)和ptch2_f 片段4个位点(cpg_1、cpg_2、cpg_3、cpg_4)两组与健康对照甲基化程度均存在显著性差异(表16)。
[0352]
表16、乳腺癌病例不同p53基因突变状态ptch2基因cpg位点甲基化水平比较(224例乳腺癌病例vs.223 例健康对照)
[0353]
[0354][0355]
注:
“‑”
表示数值缺失,因为两组数值极为接近,无法计算出对应的p值。
[0356]
7、ptch2甲基化与年龄的相关性
[0357]
ptch2甲基化与年龄的相关性结果显示,在对照人群中,ptch2_a片段5个位点 (cpg_1.2.3.4、cpg_7.8.9.10.11、cpg_13、cpg_14.15、cpg_19)、ptch2_b片段5个位点 (cpg_1、cpg_2、cpg_6、cpg_7、cpg_9.10.11.12)、ptch2_c片段2个位点(cpg_1、cpg_2、 cpg_3、cpg_4、cpg_5、cpg_6、cpg_7)、ptch2_d片段4个片段(cpg_1、cpg_2、cpg_3、 cpg_4)、ptch2_e片段7个位点(cpg_1、cpg_3、cpg_4.5、cpg_6、cpg_7、cpg_8.9、 cpg_10.11)和ptch2_f片段4个位点(cpg_1、cpg_2、cpg_3、cpg_4)甲基化程度与年龄呈负相关,且乳腺癌病例ptch2基因cpg甲基化水平与年龄的相关性较健康对照ptch2甲基化水平更强(表17)。
[0358]
表17、224例乳腺癌病例和223例健康对照ptch2基因cpg位点的甲基化水平与年龄的相关性
[0359][0360]
8、ptch2基因甲基化对乳腺癌预警和早期诊断的价值
[0361]
本发明建立的用于辅助乳腺癌诊断的数学模型可以达到如下目的:
[0362]
(1)辅助区分乳腺癌患者和健康对照;
[0363]
(2)辅助区分年龄≤35岁的乳腺癌患者和健康对照;
[0364]
(3)辅助区分年龄>35且<45岁的乳腺癌患者和健康对照;
[0365]
(4)辅助区分年龄≥45岁的乳腺癌患者和健康对照;
[0366]
(5)辅助区分临床分期为0期和/或i期的乳腺癌患者和健康对照;
[0367]
(6)辅助区分临床分期为ii期的乳腺癌患者和健康对照;
[0368]
(7)辅助区分临床分期为iii期和/或ⅳ期的乳腺癌患者和健康对照;
[0369]
(8)辅助区分肿瘤大小为tis和/或t1的乳腺癌患者和健康对照;
[0370]
(9)辅助区分肿瘤大小为t2的乳腺癌患者和健康对照;
[0371]
(10)辅助区分肿瘤大小为t3和/或t4的乳腺癌患者和健康对照;
[0372]
(11)辅助区分分子分型为luminal a型的乳腺癌患者和健康对照;
[0373]
(12)辅助区分分子分型为luminal b型的乳腺癌患者和健康对照;
[0374]
(13)辅助区分分子分型为her2扩增型的乳腺癌患者和健康对照;
[0375]
(14)辅助区分分子分型为基底样型的乳腺癌患者和健康对照;
[0376]
(15)辅助区分ki67蛋白表达量高的乳腺癌患者和健康对照;
[0377]
(16)辅助区分ki67蛋白表达量低的乳腺癌患者和健康对照;
[0378]
(17)辅助区分p53基因突变率高的乳腺癌患者和健康对照;
[0379]
(18)辅助区分p53基因突变率低的乳腺癌患者和健康对照。
[0380]
数学模型的建立方法如下:
[0381]
(a)数据来源:步骤一中列出乳腺癌患者224例和年龄匹配的无癌健康女性223例的离体血液样本的目标cpg位点(表1-表6中的一种或多种的组合)甲基化水平(检测方法同步骤二)。
[0382]
数据可根据实际需要加入年龄、白细胞计数等已知参数来提高判别效率。
[0383]
(b)模型建立
[0384]
根据需要选取任意两类不同类型患者数据即训练集(例如:发病年龄≤35岁的乳腺癌患者和健康对照、发病年龄35岁至45岁的乳腺癌患者和健康对照、发病年龄≥45岁的乳腺癌患者和健康对照、临床分期为0期和/或i期的乳腺癌患者和健康对照、临床分期为ii期的乳腺癌患者和健康对照、临床分期为iii期和/或ⅳ期的乳腺癌患者和健康对照、肿瘤大小为tis 和/或t1的乳腺癌患者和健康对照、肿瘤大小为t2的乳腺癌患者和健康对照、肿瘤大小为 t3和/或t4的乳腺癌患者和健康对照、分子分型为luminal a型的乳腺癌患者和健康对照、分子分型为luminal b型的乳腺癌患者和健康对照、分子分型为her2扩增型的乳腺癌患者和健康对照、分子分型为基底样型的乳腺癌患者和健康对照、ki67蛋白表达量高的乳腺癌患者和健康对照、ki67蛋白表达量低的乳腺癌患者和健康对照、p53基因突变率高的乳腺癌患者和健康对照、p53基因突变率低的乳腺癌患者和健康对照)作为用于建立模型的数据,使用 sas,r,spss等统计软件使用二分类逻辑回归的统计方法通过公式建立数学模型。数学模型公式计算出的最大约登指数对应的数值为阈值或直接设定0.5为阈值,待测样品经过测试和代入模型计算后得到的检测指数大于阈值归为一类(b类),小于阈值归为另外一类(a类),等于阈值作为不确定的灰区。在对新的待测样品进行预测来判断属于哪一类时,首先通过 dna甲基化的测定方法检测该待测样品ptch2基因上一个或者多个cpg位点的甲基化水平,然后将这些甲基化水平的数据代入上述数学模型,计算得到所述待测样本对应的检测指数,然后比较所述待测样本对应的检测指数和阈值的大小,根据比较结果确定所述待测样本属于哪一类样本。
[0385]
举例:将训练集中ptch2基因单个cpg位点的甲基化水平或者多个cpg位点组合的
甲基化水平的数据通过sas、r、spss等统计软件使用二分类逻辑回归的公式建立用于区分a 类和b类的数学模型。该数学模型在此为二类逻辑回归模型,具体为: log(y/(1-y))=b0 b1x1 b2x2 b3x3
…
. bnxn,其中y为因变量即将待测样品的一个或者多个甲基化位点的甲基化值代入模型以后得出的检测指数,b0为常量,x1~xn为自变量即为该测试样品的一个或者多个甲基化位点的甲基化值(每一个值为0~1之间的数值),b1~bn为模型赋予每一个位点甲基化值的权重。具体应用时,先根据训练集中已经检测的样本的一个或者多个 dna甲基化位点的甲基化程度(x1~xn)及其已知的分类情况(a类或者b类,分别对y赋值0 和1)建立数学模型,由此确定该数学模型的常量b0以及各个甲基化位点的权重b1~bn,并由该数学模型计算出的以最大约登指数对应的数值为阈值或直接设定0.5为划分的阈值。待测样品经过测试和代入模型计算后得到的检测指数即y值大于阈值归为b类,小于阈值归为a 类,等于阈值作为不确定的灰区。其中a类和b类为相对应的两分类(二分类的分组,哪一组a类,哪一组是b类,要根据具体的数学模型来确定,在此不做约定)。对受试者的样品进行预测来判断属于哪一类时,首先采集受试者的血液,然后从中提取dna。将提取的dna 通过重亚硫酸盐转化后,用dna甲基化的测定方法对受试者ptch2基因的单个cpg位点的甲基化水平或者多个cpg位点组合的甲基化水平进行检测,然后将检测得到的甲基化数据代入上述数学模型。如果该受试者的ptch2基因一个或者多个cpg位点的甲基化水平代入上述数学模型后计算出来的值即检测指数大于阈值,则该受试者判定与训练集中检测指数大于阈值的归属一类(b类);如果该受试者的ptch2基因一个或者多个cpg位点的甲基化水平数据代入上述数学模型后计算出来的值即检测指数小于阈值,则该受试者跟训练集中检测指数小于阈值的归属一类(a类);如果该受试者的ptch2基因一个或者多个cpg位点的甲基化水平数据代入上述数学模型后计算出来的值即检测指数等于阈值,则不能判断该受试者是a类还是b类。
[0386]
举例:举例说明ptch2_e的全部cpg位点(ptch2_e_cpg_1、ptch2_e_cpg_3、 ptch2_e_cpg_4.5、ptch2_e_cpg_6、ptch2_e_cpg_7、ptch2_e_cpg_8.9、 ptch2_e_cpg_10.11)的甲基化以及数学建模在用于女性中发现乳腺癌患者(早期诊断乳腺癌) 的应用:将乳腺癌患者和对照训练集(在此为:224例乳腺癌患者和223对照)中已经检测的 ptch2_e的全部cpg位点的甲基化水平的数据以及患者的年龄、白细胞计数通过spss软件或r软件使用二分类逻辑回归的公式建立用于女性中发现乳腺癌患者(早期诊断乳腺癌)的数学模型。该数学模型在此为二类逻辑回归模型,由此确定该数学模型的常量b0以及各个甲基化位点的权重b1~bn,在此例中具体为:log(y/(1-y))= log(y/(1-y))=1.302-0.512*ptch2_e_1 2.013*ptch2_e_3 1.548*ptch2_e_4.5-2.024*ptch2_ e_6-0.199*ptch2_e_7-1.900*ptch2_e_8.9-9.561*ptch2_e_10.11-0.528*白细胞计数 0.029*年龄,其中y为因变量即将待测样品的ptch2_e的全部cpg位点的甲基化值以及年龄、白细胞计数代入模型以后得出的检测指数。通过最大约登指数得到的诊断阈值或设定0.5 为阈值的情况下,待测样品的ptch2_e的全部cpg位点的甲基化水平经过测试后连同其年龄、白细胞计数的信息代入模型进行计算,得到的检测指数即y值小于阈值归为无乳腺癌的健康人,大于阈值归为乳腺癌患者,等于阈值则不确定为乳腺癌患者还是对照。此模型的曲线下面积(auc)计算结果为0.71(表18)。具体受试者判断方法举例如下所示,从两位受试者 (甲,乙)分别采集血液提取dna,将提取的dna通过重亚硫酸盐转化后,用dna甲基化的测定方法对受试者的ptch2_e的全部cpg位点的
甲基化水平进行检测。然后将检测得到的甲基化水平数据连同受试者的年龄、白细胞计数的信息代入上述数学模型。甲受试者经数学模型后计算出来的值为0.35小于0.5,则甲受试者判定为无乳腺癌的健康人(与临床诊断相符);乙受试者的ptch2_e的全部cpg位点的甲基化水平数据代入上述数学模型后计算出来的值为0.58大于0.5,则乙受试者判定乳腺癌患者(与临床诊断相符)。
[0387]
(c)模型效果评价
[0388]
根据上述方法,分别建立用于发现年龄≤35岁、35岁至45岁、≥45岁的乳腺癌患者的数学模型,并且通过受试者曲线(roc曲线)对其有效性进行评价。roc曲线得出的曲线下面积(auc)越大,说明模型的区分度越好,分子标志物越有效。采用不同cpg位点进行数学模型构建后的评价结果如表18所示。表18中,1个cpg位点代表 ptch2_a/ptch2_b/ptch2_c/ptch2_d/ptch2_e/ptch2_f扩增片段中任意一个cpg位点的位点,2个cpg位点代表ptch2_a/ptch2_b/ptch2_c/ptch2_d/ptch2_e/ptch2_f扩增片段中任意2个cpg位点的组合,3个cpg位点代表 ptch2_a/ptch2_b/ptch2_c/ptch2_d/ptch2_e/ptch2_f扩增片段中任意3个cpg位点的组合,
……
以此类推。表中的数值为不同位点组合评价结果的范围值(即任意个cpg位点组合方式的结果均在此范围内)。
[0389]
本研究结果显示,ptch2基因甲基化(所有cpg位点)用于早期诊断年龄≤35岁、年龄35 岁至45岁、年龄≥45岁和整体(不分年龄)的女性乳腺癌患者的roc曲线下面积分别为0.92、 0.84、0.77、0.86,通过最大约登指数得到的诊断阈值对应的敏感度分别为97.1%、87.3%、 83.3%和89.2%,特异度分别为80.9%、78.7%、0.677%和80.8%,提示ptch2基因甲基化对乳腺癌有很好的预警和早期诊断效果(表18)。
[0390]
表18、ptch2基因甲基化对乳腺癌预警和早期诊断的价值
[0391]
[0392]
[0393]
[0394]
[0395]
[0396][0397]
以上对本发明进行了详述。对于本领域技术人员来说,在不脱离本发明的宗旨和范围,以及无需进行不必要的实验情况下,可在等同参数、浓度和条件下,在较宽范围内实施本发明。虽然本发明给出了特殊的实施例,应该理解为,可以对本发明作进一步的改进。总之,按本发明的原理,本技术欲包括任何变更、用途或对本发明的改进,包括脱离了本技术中已公开范围,而用本领域已知的常规技术进行的改变。按以下附带的权利要求的范围,可以进行一些基本特征的应用。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。
