使用单细胞分析的改良的变体调用程序
1.交叉引用
2.本技术要求于2019年10月2日提交的美国临时专利申请第62/909,670号的权益和优先权,所述美国临时专利申请的全部公开内容出于所有目的以引用方式在此并入。
背景技术:
3.测序技术通常会生成具有介于0.5%-2%范围内的误差的序列读段,所述误差源自pcr和测序误差。旨在调用细胞群体中的变体的变体调用程序通常因这些误差而鉴别出假阳性,这对变体调用程序的准确性造成负面影响。减少假阳性的常规策略通常采用硬截止(hard cutoff);然而,实施这些硬截止会消除大量真阳性,所述问题通常称为丢失数据问题。因此,需要可更好地鉴别假阳性而不牺牲真阳性的改良的变体调用程序。
技术实现要素:
4.本文描述通过两步过程改良变体调用的实施方案,所述两步过程涉及1)通过细胞特异性过程对序列读段中的碱基进行误差校正,和2)使用误差校正的序列读段在细胞群体中进行变体调用。碱基中的误差通常源自pcr误差、测序误差、测序比对误差或校正误差中的任一个。在此处,两步过程使得能够鉴别并校正错误碱基,由此使得能够进行更准确的变体调用。在多个实施方案中,碱基的误差校正涉及实施用于校正错误碱基的第一经训练机器学习模型,下文称为误差校正模型。因此,误差校正模型使得能够校正来自单个细胞的序列读段。通过细胞特异性方式进行碱基的误差校正与校正源自批量测序的序列读段相比是有利的。例如,序列读段中的碱基误差可源自单细胞,且因此可针对单细胞一起校正这些碱基误差。在多个实施方案中,细胞群体中的变体调用涉及实施第二经训练机器学习模型,下文称为变体调用程序模型。变体调用程序模型分析校正的序列读段以调用更可能为存在于细胞群体中的真变体的变体。总之,涉及实施误差校正模型和变体调用程序模型的两步过程实现调用真变体的更高的准确性。此可用于鉴别可能与疾病(例如癌症)有关的真变体。
5.本文公开用于调用细胞群体的一种或多种变体的方法,所述方法包括:从细胞群体的细胞获得多个序列读段;对于细胞群体中的多个细胞,校正从细胞获得的序列读段,所述校正包括:鉴别序列读段的不同于参考碱基的所关注碱基;应用误差校正模型来分析所关注碱基的单细胞特征,所述误差校正模型经训练以预测所关注碱基的概率;和校正源自细胞的序列读段的所关注碱基;通过聚集细胞群体的细胞的校正的序列读段来生成细胞群体特征,校正的序列读段包含校正的碱基;和将变体调用程序模型应用于源自聚集的序列读段的细胞群体特征以鉴别细胞群体中的一种或多种变体。
6.在多个实施方案中,单细胞特征包括所关注碱基周围的上下文序列、所关注碱基的测序深度、所关注碱基的等位基因频率和所关注碱基周围的窗口中的碱基的等位基因频率。在多个实施方案中,鉴别序列读段的所关注碱基包括将包含参考碱基与错配碱基之间的转换的可能性的转换矩阵应用于在错配碱基的序列读段中观察到一定比例的核苷酸碱基的概率。在多个实施方案中,鉴别序列读段的所关注碱基还包括:确定在错配碱基的序列
读段中观察到一定比例的核苷酸碱基的概率;和比较确定的概率与来自转换矩阵的转换的可能性。在多个实施方案中,响应于确定的概率大于转换的可能性,从而将错配碱基鉴别为所关注碱基。在多个实施方案中,转换矩阵是使用训练数据生成,所述训练数据包含源自一个或多个细胞样品群体的序列读段。在多个实施方案中,转换矩阵是使用来自细胞群体的细胞的多个序列读段生成。在多个实施方案中,当校正细胞群体的一个或多个细胞的序列读段时,转换矩阵中的转换可能性被动态更新。
7.在多个实施方案中,误差校正模型是神经网络。在多个实施方案中,误差校正模型是深度学习神经网络,所述深度学习神经网络包括一个或多个层,所述一个或多个层学习所关注碱基周围的基序和局部序列上下文。在多个实施方案中,校正源自细胞结果的多个序列读段的一个或多个序列读段包括校正至少25%的不同于参考碱基的所关注碱基。
8.在多个实施方案中,细胞群体特征包括以下中的一者或多者:杂合子调用百分比、杂合子调用的中值变体等位基因频率(vaf)、杂合子调用的中值基因型质量、杂合子调用的中值读段深度、纯合子调用百分比、纯合子调用的中值vaf、纯合子调用的中值基因型质量、纯合子调用的中值读段深度、参考调用百分比、用于纯合子调用的读段深度的变异系数(cv)、用于杂合子调用的读段深度的cv、纯合子调用的基因型质量的cv、杂合子调用的基因型质量的cv、用于纯合子调用的vaf的cv、用于杂合子调用的vaf的cv、用于纯合子调用的平均vaf与中值vaf之间的差、用于杂合子调用的平均vaf与中值vaf之间的差和扩增子gc百分比。
9.在多个实施方案中,变体调用程序模型预测所关注杂合变体或所关注纯合变体中的至少一个。在多个实施方案中,变体调用程序模型还预测不确定的变体。在多个实施方案中,变体调用程序模型是使用训练数据来训练,所述训练数据包含源自一种或多种细胞系的序列读段和存在于一种或多种细胞系中的已知杂合或纯合变体的指示。在多个实施方案中,与常规gtak变体调用程序相比,误差校正模型和变体调用程序模型的应用在0.5%的检测限值(lod)下实现真变体阳性预测值的至少两倍增加。在多个实施方案中,误差校正模型和变体调用程序模型的应用在0.5%的检测限值(lod)下实现至少0.6的真变体阳性预测值。在多个实施方案中,源自细胞的多个序列读段是通过单细胞工作流分析来确定。在多个实施方案中,参考碱基是根据参考基因组序列来确定。在多个实施方案中,参考碱基是根据从对照细胞获得的一个或多个序列读段来确定。
10.另外,本文公开用于调用细胞群体的一种或多种变体的非暂时性计算机可读介质,所述非暂时性计算机可读介质包括如下指令,所述指令在由处理器执行时使得处理器:从细胞群体的细胞获得多个序列读段;对于细胞群体中的多个细胞,校正从细胞获得的序列读段,所述校正包括:鉴别序列读段的不同于参考碱基的所关注碱基;应用误差校正模型来分析所关注碱基的单细胞特征,所述误差校正模型经训练以预测所关注碱基的概率;和校正源自细胞的序列读段的所关注碱基;通过聚集细胞群体的细胞的校正的序列读段来生成细胞群体特征,校正的序列读段包含校正的碱基;和将变体调用程序模型应用于源自聚集的序列读段的细胞群体特征以鉴别细胞群体中的一种或多种变体。
11.在多个实施方案中,单细胞特征包括所关注碱基周围的上下文序列、所关注碱基的测序深度、所关注碱基的等位基因频率和所关注碱基周围的窗口中的碱基的等位基因频率。在多个实施方案中,使得处理器鉴别序列读段的所关注碱基的指令还包括在由处理器
执行时使得处理器应用转换矩阵的指令,所述转换矩阵包含参考碱基与错配碱基之间的转换的可能性。
12.在多个实施方案中,使得处理器鉴别序列读段的所关注碱基的指令还包括如下指令,所述指令在由处理器执行时使得处理器:确定在错配碱基的序列读段中观察到一定比例的核苷酸碱基的概率;和比较确定的概率与来自转换矩阵的转换的可能性。在多个实施方案中,响应于确定的概率大于转换的可能性,将错配碱基鉴别为所关注碱基。在多个实施方案中,转换矩阵是使用训练数据生成,所述训练数据包含源自一个或多个细胞样品群体的序列读段。在多个实施方案中,转换矩阵是使用来自细胞群体的细胞的多个序列读段生成。在多个实施方案中,当校正细胞群体的一个或多个细胞的序列读段时,转换矩阵中的转换可能性被动态更新。
13.在多个实施方案中,误差校正模型是神经网络。在多个实施方案中,误差校正模型是深度学习神经网络,所述深度学习神经网络包括一个或多个层,所述一个或多个层学习所关注碱基周围的基序和局部序列上下文。在多个实施方案中,校正源自细胞结果的多个序列读段的一个或多个序列读段包括校正至少25%的不同于参考碱基的所关注碱基。在多个实施方案中,细胞群体特征包括以下中的一者或多者:杂合子调用百分比、杂合子调用的中值变体等位基因频率(vaf)、杂合子调用的中值基因型质量、杂合子调用的中值读段深度、纯合子调用百分比、纯合子调用的中值vaf、纯合子调用的中值基因型质量、纯合子调用的中值读段深度、参考调用百分比、用于纯合子调用的读段深度的变异系数(cv)、用于杂合子调用的读段深度的cv、纯合子调用的基因型质量的cv、杂合子调用的基因型质量的cv、用于纯合子调用的vaf的cv、用于杂合子调用的vaf的cv、用于纯合子调用的平均vaf与中值vaf之间的差、用于杂合子调用的平均vaf与中值vaf之间的差和扩增子gc百分比。
14.在多个实施方案中,变体调用程序模型预测所关注杂合变体或所关注纯合变体中的至少一个。在多个实施方案中,变体调用程序模型还预测不确定的变体。在多个实施方案中,变体调用程序模型是使用训练数据来训练,所述训练数据包含源自一种或多种细胞系的序列读段和存在于一种或多种细胞系中的已知杂合或纯合变体的指示。在多个实施方案中,与常规gtak变体调用程序相比,误差校正模型和变体调用程序模型的应用在0.5%的检测限值(lod)下实现真变体阳性预测值的至少两倍增加。在多个实施方案中,误差校正模型和变体调用程序模型的应用在0.5%的检测限值(lod)下实现至少0.6的真变体阳性预测值。在多个实施方案中,源自细胞的多个序列读段是通过单细胞工作流分析来确定。在多个实施方案中,参考碱基是根据参考基因组序列来确定。在多个实施方案中,参考碱基是根据从对照细胞获得的一个或多个序列读段来确定。
15.另外,本文公开系统,所述系统包括:单细胞分析工作流装置,所述单细胞分析工作流装置经配置以生成细胞群体中的细胞的多个序列读段;计算装置,所述计算装置通信耦接到单细胞分析工作流装置,计算装置经配置以:从细胞群体的细胞获得多个序列读段;对于细胞群体中的多个细胞,校正从细胞获得的序列读段,所述校正包括:鉴别序列读段的不同于参考碱基的所关注碱基;应用误差校正模型来分析所关注碱基的单细胞特征,所述误差校正模型经训练以预测所关注碱基的概率;和校正源自细胞的序列读段的所关注碱基;通过聚集细胞群体的细胞的校正的序列读段来生成细胞群体特征,校正的序列读段包含校正的碱基;和将变体调用程序模型应用于源自聚集的序列读段的细胞群体特征以鉴别
细胞群体中的一种或多种变体。在多个实施方案中,单细胞特征包括所关注碱基周围的上下文序列、所关注碱基的测序深度、所关注碱基的等位基因频率和所关注碱基周围的窗口中的碱基的等位基因频率。
16.在多个实施方案中,鉴别序列读段的所关注碱基包括:将包含参考碱基与错配碱基之间的转换的可能性的转换矩阵应用于在错配碱基的序列读段中观察到一定比例的核苷酸碱基的概率。在多个实施方案中,鉴别序列读段的所关注碱基包括:确定在错配碱基的序列读段中观察到一定比例的核苷酸碱基的概率;和比较确定的概率与来自转换矩阵的转换的可能性。在多个实施方案中,响应于确定的概率大于转换的可能性,将错配碱基鉴别为所关注碱基。在多个实施方案中,转换矩阵是使用训练数据生成,所述训练数据包含源自一个或多个细胞样品群体的序列读段。在多个实施方案中,转换矩阵是使用来自细胞群体的细胞的多个序列读段生成。在多个实施方案中,当校正细胞群体的一个或多个细胞的序列读段时,转换矩阵中的转换可能性被动态更新。
17.在多个实施方案中,误差校正模型是神经网络。在多个实施方案中,误差校正模型是深度学习神经网络,所述深度学习神经网络包括一个或多个层,所述一个或多个层学习所关注碱基周围的基序和局部序列上下文。在多个实施方案中,校正源自细胞结果的多个序列读段的一个或多个序列读段包括校正至少25%的不同于参考碱基的所关注碱基。
18.在多个实施方案中,细胞群体特征包括以下中的一者或多者:杂合子调用百分比、杂合子调用的中值变体等位基因频率(vaf)、杂合子调用的中值基因型质量、杂合子调用的中值读段深度、纯合子调用百分比、纯合子调用的中值vaf、纯合子调用的中值基因型质量、纯合子调用的中值读段深度、参考调用百分比、用于纯合子调用的读段深度的变异系数(cv)、用于杂合子调用的读段深度的cv、纯合子调用的基因型质量的cv、杂合子调用的基因型质量的cv、用于纯合子调用的vaf的cv、用于杂合子调用的vaf的cv、用于纯合子调用的平均vaf与中值vaf之间的差、用于杂合子调用的平均vaf与中值vaf之间的差和扩增子gc百分比。
19.在多个实施方案中,变体调用程序模型预测所关注杂合变体或所关注纯合变体中的至少一个。在多个实施方案中,变体调用程序模型还预测不确定的变体。在多个实施方案中,变体调用程序模型是使用训练数据来训练,所述训练数据包含源自一种或多种细胞系的序列读段和存在于一种或多种细胞系中的已知杂合或纯合变体的指示。
20.在多个实施方案中,与常规gtak变体调用程序相比,误差校正模型和变体调用程序模型的应用在0.5%的检测限值(lod)下实现真变体阳性预测值的至少两倍增加。在多个实施方案中,误差校正模型和变体调用程序模型的应用在0.5%的检测限值(lod)下实现至少0.6的真变体阳性预测值。在多个实施方案中,参考碱基是根据参考基因组序列来确定。在多个实施方案中,参考碱基是根据从对照细胞获得的一个或多个序列读段来确定。
21.附图中几个视图的简述
22.参考以下描述和附图将更好地理解本发明的这些和其他特征、方面和优点,其中:
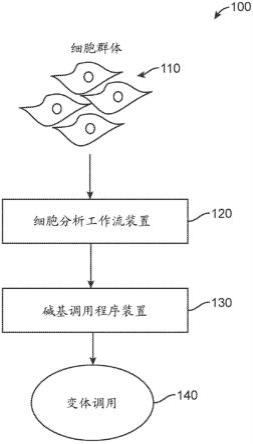
23.图(fig.)1描绘根据实施方案的整体系统环境,所述整体系统环境包括细胞分析工作流装置和用于鉴别变体调用的碱基调用程序装置。
24.图2是根据实施方案的碱基调用程序装置的单独模块的方块图。
25.图3a是根据实施方案用于校正源自单细胞的序列读段的流程图。
26.图3b描绘根据实施方案使用校正的序列读段来调用细胞群体的变体的流程图。
27.图4a描绘根据实施方案的误差校正模型的实施。
28.图4b描绘根据实施方案的变体调用程序模型的实施。
29.图5描绘用于实施参考图1-4描述的系统和方法的示例性计算装置。
30.图6描绘碱基误差的示例性分布,其中大部分碱基误差仅在一种细胞中观察到。
31.图7是转换矩阵的示例性描绘。
32.图8a和图8b是不同位置的6个序列读段的堆积的示例性描绘。
33.图9a描绘误差校正模型的示例性输入和输出。
34.图9b描绘使用由误差校正模型预测的概率校正所关注碱基的实例。
35.图10展示校正四个不同细胞群体中的20%-35%碱基作为实施误差校正模型的结果。
36.图11展示在实施误差校正模型和变体调用程序模型后真变体的改良的阳性预测值。
具体实施方式
37.定义
38.除非另有规定,否则如下文所阐述的对权利要求书和说明书中使用的术语进行定义。
39.短语“错配碱基”和“替代碱基”可互换使用且是指某一位置的碱基,所述碱基不同于同一位置的已知参考碱基。在一些情况下,错配碱基被错误地鉴别(例如在测序期间被错误地鉴别)。碱基的错误鉴别可源自多个来源,例如pcr误差、测序误差、测序比对误差和/或校正误差。为提供实例,参考位置的已知碱基可为腺嘌呤(a)。错配碱基或替代碱基是指同一位置的除腺嘌呤(a)外的碱基(例如,碱基是鸟嘌呤(g)、胞嘧啶(c)或胸腺嘧啶(t)中的任一个)。
40.短语“参考碱基”是指具有已知核苷酸碱基的已知碱基。在一个实施方案中,参考碱基是根据参考基因组序列来确定。在一个实施方案中,参考碱基是根据从对照细胞获得的一个或多个序列读段来确定。
41.短语“误差校正模型”是指预测模型或机器学习模型,它经实施以分析所关注碱基,使得所关注碱基可被校正。通常,误差校正模型经实施来以细胞特异性方式分析所关注碱基。在一个实施方案中,误差校正模型分析针对所关注碱基生成的堆积,所述堆积量化源自单细胞的序列读段的碱基。在此类实施方案中,来自包括所关注碱基的单细胞的序列读段可一起校正。
42.短语“所关注碱基”是指与参考碱基相比发生错配的源自细胞的序列读段中的碱基。在多个实施方案中,通过应用转换矩阵,所关注碱基可能是错误碱基。通常,通过误差校正模型分析针对所关注碱基生成的堆积以确定所关注碱基是否可能是错误碱基。
43.短语“单细胞特征”是指与单细胞的序列读段中的所关注碱基相关的特征。在多个实施方案中,通过误差校正模型分析单细胞特征以确定对应于四种核苷酸碱基(腺嘌呤、鸟嘌呤、胞嘧啶和胸腺嘧啶)的概率分布,所述概率分布代表所关注碱基为四种核苷酸碱基中的一者的可能性。单细胞特征的实例包括所关注碱基周围的上下文序列、所关注碱基的测
序深度、所关注碱基的等位基因频率和所关注碱基周围的窗口中的碱基的等位基因频率。
44.短语“变体调用程序模型”是指预测模型或机器学习模型,它经实施以调用细胞群体的变体。变体调用程序模型分析源自细胞群体的校正的序列读段的细胞群体特征,所述序列读段已经受误差校正(例如,使用误差校正模型校正)。在一个实施方案中,变体调用程序模型接收细胞群体特征作为输入且预测候选变体的分类。在一个实施方案中,变体调用程序模型从先前校正的序列读段提取细胞群体特征且基于所提取细胞群体特征预测候选变体的分类。
45.短语“细胞群体特征”是指与源自细胞群体的校正的序列读段的候选变体相关的特征。通过变体调用程序模型分析细胞群体特征以预测细胞群体的真变体。细胞群体特征的实例包括杂合子调用百分比、杂合子调用的中值变体等位基因频率(vaf)、杂合子调用的中值基因型质量、杂合子调用的中值读段深度、纯合子调用百分比、纯合子调用的中值vaf、纯合子调用的中值基因型质量、纯合子调用的中值读段深度、参考调用百分比、用于纯合子调用的读段深度的变异系数(cv)、用于杂合子调用的读段深度的cv、纯合子调用的基因型质量的cv、杂合子调用的基因型质量的cv、用于纯合子调用的vaf的cv、用于杂合子调用的vaf的cv、用于纯合子调用的平均vaf与中值vaf之间的差、用于杂合子调用的平均vaf与中值vaf之间的差和扩增子gc百分比。
46.短语“候选变体”是指与参考碱基相比发生错配的细胞群体的序列读段中的碱基。通常,变体调用程序模型经实施以确定候选变体是否是真变体,例如纯合变体或杂合变体。
47.短语“真变体”是指存在于细胞群体的一个或多个细胞中的遗传变体。
48.概述
49.本文所述的实施方案是指进行碱基的细胞特异性误差校正且还使用误差校正的序列读段进行变体鉴别的改良的变体调用程序。在多个实施方案中,细胞特异性误差校正涉及实施误差校正模型,且变体鉴别涉及实施变体调用程序模型。总之,与误差校正模型和/或变体调用程序模型相反的采用硬截止的常规变体调用方法(例如基因组分析工具盒(gatk))相比,本文所述的变体调用方法在调用存在于细胞中的真变体方面实现更高的准确性。关于gatk中所用的硬过滤器的进一步描述参见de summa,s.、malerba,g.、pinto,r.等人,gatk hard filtering:tunable parameters to improve variant calling for next generation sequencing targeted gene panel data.bmc bioinformatics 18,119(2017),所述参考文献的全文以引用方式在此并入。
50.参考图1,它描绘根据实施方案的整体系统环境实施方案100,其包括细胞分析工作流装置120和用于变体调用的碱基调用程序装置130。获得细胞群体110。在多个实施方案中,细胞群体110可由从受试者或患者获得的测试样品分离而来。在多个实施方案中,细胞群体110包括取自健康受试者的健康细胞。在多个实施方案中,细胞群体110包括取自受试者的患病细胞。在一个实施方案中,细胞群体110包括取自先前经诊断患有癌症的受试者的癌细胞。例如,癌细胞可为可在经诊断患有癌症的受试者的血流中获得的肿瘤细胞。作为另一实例,癌细胞可为通过肿瘤活检获得的细胞。
51.细胞分析工作流装置120是指处理细胞并生成测序用核酸的装置。在多个实施方案中,细胞分析工作流装置120是指包括处理细胞并生成测序用核酸的一种或多种装置的系统。在多个实施方案中,细胞分析工作流装置120是工作流装置,它从单细胞生成核酸,由
此使得能够随后鉴别序列读段和序列读段所源自的单个细胞。在多个实施方案中,细胞分析工作流装置120可通过将单个细胞囊封到乳液中、裂解乳液内的细胞、对乳液中的细胞裂解物进行细胞条码化并在乳液中进行核酸扩增反应来进行单细胞处理。因此,可收集扩增的核酸并进行测序。美国申请第14/420,646号描述单细胞工作流过程的示例性实施方案的进一步描述,所述美国申请的全文在此以引用方式并入本文。
52.在具体实施方案中,细胞分析工作流装置120可为tapestri
tm
平台、indrop
tm
系统、nadia
tm
仪器或chromium
tm
仪器中的任一个。在多个实施方案中,细胞分析工作流装置120包括用于对核酸测序以生成序列读段的测序仪。
53.碱基调用程序装置130经配置以从细胞分析工作流装置120接收序列读段并处理序列读段以调用一种或多种变体140。在多个实施方案中,碱基调用程序装置130被通信耦接到细胞分析工作流装置120,且因此直接从细胞分析工作流装置120接收序列读段。碱基调用程序装置130对序列读段中的所关注碱基进行误差校正且然后调用细胞群体110中的可能变体。在具体实施方案中,碱基调用程序装置130通过细胞特异性工作流过程校正序列读段中的所关注碱基,且随后使用校正的序列读段调用细胞群体中的变体。总之,此细胞特异性误差校正和细胞群体变体调用两步过程使得能够在细胞群体110中进行更准确的变体调用140。
54.碱基调用程序装置
55.图2是根据图1中所述的实施方案的碱基调用程序装置130的方块图。如图2中所显示,碱基调用程序装置130包括碱基鉴别模块210、碱基校正模块220、细胞群体模块230、碱基调用程序模块240和训练模块250。在一些实施方案中,碱基调用程序装置130的模块可不同于图2中所显示的实施方案进行排列。例如,可通过除碱基调用程序装置130外的装置实施训练模块250(如以虚线显示)且可通过其它装置进行下文针对训练模块250描述的方法。
56.通常,碱基鉴别模块210分析源自单个细胞的序列读段且鉴别与参考碱基相比发生错配的一个或多个所关注碱基。碱基鉴别模块210基于每个细胞鉴别所关注碱基。例如,碱基鉴别模块210分析来自第一细胞的序列读段以确定来自第一细胞的序列读段的所关注碱基。碱基鉴别模块210还分析来自第二细胞的序列读段以确定来自第二细胞的序列读段的所关注碱基等等。来自不同细胞的序列读段可使用条码技术彼此进行区分,pct/us2016/016444进一步描述所述条码技术的实例,所述申请的全文以引用方式在此并入。另外,对于每一细胞,碱基鉴别模块210生成对应于细胞的所关注碱基的序列读段的堆积且将堆积提供到碱基校正模块220以确定是否校正所关注碱基中的任一个。
57.在多个实施方案中,碱基鉴别模块210获得与参考基因组比对的序列读段。作为实例,碱基鉴别模块210可以可读文件格式(例如sam(序列比对图)文件格式或bam(二进制比对图)文件格式)获得序列读段。
58.给定比对的序列读段,碱基鉴别模块210在源自细胞的序列读段中鉴别一个或多个所关注碱基。在多个实施方案中,碱基鉴别模块210分析每一错配碱基以确定错配碱基是否是所关注碱基。
59.在多个实施方案中,为鉴别所关注碱基,碱基鉴别模块210应用过滤器来确定在来自细胞的位置处是否至少阈值数量的序列读段具有不同于该位置处参考碱基的特定核苷酸碱基。在多个实施方案中,如果在该位置超过阈值数量的序列读段具有不同于参考碱基
的核苷酸碱基,那么碱基鉴别模块210将该碱基鉴别为所关注碱基用于后续校正。
60.在多个实施方案中,该位置的序列读段的阈值数量是固定值。在多个实施方案中,序列读段的阈值数量大于1000个、大于2000个、大于3000个、大于4000个、大于5000个、大于6000个、大于7000个、大于8000个、大于9000个、大于10,000个、大于20,000个、大于30,000个、大于40,000个、大于50,000个、大于75,000个、大于100,000个、大于150,000个、大于200,000个、大于250,000个或大于500,000个序列读段。在多个实施方案中,序列读段的阈值数量大于来自细胞的该位置的总序列读段的5%、大于来自细胞的该位置的总序列读段的10%、大于来自细胞的该位置的总序列读段的20%、大于来自细胞的该位置的总序列读段的30%、大于来自细胞的该位置的总序列读段的40%、大于来自细胞的该位置的总序列读段的50%、大于来自细胞的该位置的总序列读段的60%、大于来自细胞的该位置的总序列读段的70%、大于来自细胞的该位置的总序列读段的75%、大于来自细胞的该位置的总序列读段的80%、大于来自细胞的该位置的总序列读段的85%、大于来自细胞的该位置的总序列读段的90%或大于来自细胞的该位置的总序列读段的95%。
61.在多个实施方案中,碱基鉴别模块210通过应用转换矩阵来鉴别所关注碱基。在此类实施方案中,应用转换矩阵包括比较转换矩阵的概率与反映观察到序列读段的一定比例的核苷酸碱基的可能性的概率。
62.首先提到的转换矩阵包括代表特定位置的参考碱基的核苷酸与观察到的碱基的核苷酸之间的转换频率的概率。通常,代表转换矩阵的转换频率的概率使得碱基鉴别模块210能够区分可能因误差(pcr误差、测序误差等)引起的错配碱基与并非因误差产生的错配碱基。
63.在多个实施方案中,对于给定参考碱基(例如a、c、g或t),转换矩阵包括参考碱基在序列读段中被观察为的不同碱基的概率。在多个实施方案中,转换矩阵包括12个概率值(例如,3个概率值反映参考碱基到错配碱基的转换)。在多个实施方案中,转换矩阵包括16个概率值。此包括每一参考碱基的在序列读段中观察到的碱基与参考碱基匹配的概率。下文图7中描述示例性转换矩阵。
64.图7是转换矩阵的示例性描绘。在此处,转换矩阵包括参考碱基(例如y轴上的“ref”)和观察到的碱基(例如x轴上的“观察到的碱基”)的名称。转换矩阵中的每个细胞包括代表参考碱基的核苷酸被观察为观察到的碱基的核苷酸的概率的可能性值。例如,转换矩阵的第一行指示对于已知腺嘌呤参考碱基“a”,观察到的碱基与参考腺嘌呤碱基匹配的概率是99%(第一行)。然而,在一些情况下,在序列读段中不同地观察到的参考腺嘌呤碱基。例如,对于已知腺嘌呤参考碱基“a”,观察到的碱基与参考腺嘌呤碱基错配的概率是0.26%(第一行第二列指示观察到的胸腺嘧啶碱基)、0.61%(第一行第三列指示观察到的鸟嘌呤碱基)和0.13%(第一行第四列指示观察到的胞嘧啶碱基)。
65.在一些实施方案中,转换矩阵先前从一种或多种先前样品生成。先前样品可包括细胞群体的细胞或可包括细胞群体混合物的细胞。在此类实施方案中,转换矩阵用作可在不同样品中应用的参考。因此,转换矩阵可用于鉴别不同样品的所关注碱基。在多个实施方案中,碱基鉴别模块210生成经受变体调用过程的每一样品的转换矩阵。因此,在此类实施方案中,在鉴别所关注碱基时,碱基鉴别模块210为每一样品应用不同的转换矩阵。此在一些情况下可能是优选的,这是因为误差可以样品依赖性方式产生。
66.在多个实施方案中,碱基鉴别模块210使用碱基鉴别模块210正在分析的至少部分相同的序列读段生成转换矩阵来鉴别所关注碱基。在此类实施方案中,随着所关注碱基被校正(例如使用如下文所述的误差校正模型校正),碱基鉴别模块210可动态更新转换矩阵中的概率,以反映校正碱基的新核苷酸碱基。作为碱基鉴别模块210如何生成转换矩阵的实例,对于具有参考碱基“a”的位置,碱基鉴别模块210确定在该位置具有四种核苷酸碱基(a、c、t或g)中的任一种的序列读段的比例。因此,碱基鉴别模块210量化具有参考碱基“a”的位置的四种核苷酸碱基的概率分布。碱基鉴别模块210可确定参考核苷酸碱基“c”、“t”和“g”的转换的概率。
67.在多个实施方案中,碱基鉴别模块210确定反映在序列读段中对该位置观察到所述比例的核苷酸碱基的可能性的概率。在一些实施方案中,概率可表示为:
68.p(腺嘌呤=w,胞嘧啶=x,鸟嘌呤=y,胸腺嘧啶=z|n读段)
69.其中w是在该位置观察到的具有腺嘌呤核苷酸碱基的序列读段的数量,其中x是在该位置观察到的具有胞嘧啶核苷酸碱基的序列读段的数量,其中y是在该位置观察到的具有胸腺嘧啶核苷酸碱基的序列读段的数量,其中z是在该位置观察到的具有胸腺嘧啶核苷酸碱基的序列读段的数量,且其中n是在该位置观察到的序列读段的总数量。
70.在一些实施方案中,概率反映在序列读段中对该位置观察到所述比例的错配核苷酸碱基的可能性。在此处,概率可表示为:
71.p(碱基1=x,碱基2=y,碱基3=z|n读段)
72.其中碱基1、碱基2和碱基3是指与参考碱基不匹配的核苷酸碱基,其中x是在该位置观察到的具有碱基1的序列读段的数量,其中y是在该位置观察到的具有碱基2的序列读段的数量,其中z是在该位置观察到的具有碱基3的序列读段的数量,且其中n是在该位置观察到的序列读段的总数量。
73.碱基鉴别模块210比较反映对该位置观察到所述比例的核苷酸碱基的可能性的概率与转换矩阵的概率。在多个实施方案中,如果比较产生反映观察到所述比例的核苷酸碱基的可能性的概率大于转换矩阵的概率,那么碱基鉴别模块210将碱基鉴别为所关注碱基。因此,所关注碱基随后可经受校正。如果比较产生反映观察到所述比例的核苷酸碱基的可能性的概率小于转换矩阵的概率,那么碱基鉴别模块210不将碱基鉴别为所关注碱基。因此,碱基不经受校正且仍为错配碱基。
74.作为鉴别所关注碱基的整体实例,碱基鉴别模块210可鉴别该位置的大多数序列读段具有腺嘌呤(参考碱基)到鸟嘌呤(观察到的碱基)错配。转换矩阵包括反映从参考腺嘌呤碱基转换成观察到的鸟嘌呤碱基的可能性的概率。假设此概率为0.01。碱基鉴别模块210可确定,观察到除参考碱基外的所述比例的核苷酸碱基(例如观察到鸟嘌呤、胞嘧啶或胸腺嘧啶核苷酸碱基)的概率为0.05。碱基鉴别模块210比较观察到所述比例的核苷酸碱基的概率(0.05)与转换矩阵的概率(0.01)。在此处,鉴于观察到所述比例的核苷酸碱基的概率(0.05)大于转换矩阵的概率(0.01),碱基鉴别模块210将碱基鉴别为所关注碱基。
75.在多个实施方案中,在已鉴别出所关注碱基的情况下,碱基鉴别模块210生成每一所关注碱基的序列读段的堆积。特定来说,碱基鉴别模块210生成包括序列读段的堆积,所述序列读段包括位于所关注碱基上游x个位置和下游y个位置的碱基。在多个实施方案中,x和y是相同的值。在其它实施方案中,x和y是不同的值。在多个实施方案中,x可为所关注碱
基上游的1个位置、2个位置、3个位置、4个位置、5个位置、6个位置、7个位置、8个位置、9个位置、10个位置、15个位置、20个位置、25个位置、30个位置、40个位置、50个位置、60个位置、70个位置、80个位置、90个位置、100个位置、110个位置、120个位置、130个位置、140个位置或150个位置。在多个实施方案中,y可为所关注碱基下游的1个位置、2个位置、3个位置、4个位置、5个位置、6个位置、7个位置、8个位置、9个位置、10个位置、15个位置、20个位置、25个位置、30个位置、40个位置、50个位置、60个位置、70个位置、80个位置、90个位置、100个位置、110个位置、120个位置、130个位置、140个位置或150个位置。
76.在多个实施方案中,碱基鉴别模块210生成堆积,使得对于位于所关注碱基位置上游和下游的位置,堆积包括指示所述比例的具有四种核苷酸碱基(例如腺嘌呤、鸟嘌呤、胞嘧啶或胸腺嘧啶)中的一者的序列读段的概率。例如,堆积可体现为矩阵,使得对于堆积中的每一位置,矩阵包括鉴别所述比例的在该位置具有相应腺嘌呤、鸟嘌呤、胞嘧啶或胸腺嘧啶的序列读段的概率。
77.碱基校正模块220应用误差校正模型来确定所关注碱基的可能核苷酸。因此,碱基校正模块220可校正源自细胞的一个或多个序列读段中的所关注碱基。校正的序列读段代表随后可用于调用真变体的改良的序列读段。通常,碱基校正模型220通过细胞特异性过程校正序列读段。在此处,碱基校正模型220可校正第一细胞的序列读段中的所关注碱基,但无法校正第二细胞的序列读段中的相同碱基。由于误差(例如pcr误差、测序误差、测序比对误差或校正误差)可出现在单个细胞中,通过碱基校正模型220进行的方法使得能够基于每个细胞校正序列读段以解决这些误差。
78.碱基校正模块220接收对所关注碱基生成的堆积。在一个实施方案中,碱基校正模块220将所关注碱基的堆积作为输入应用于误差校正模型。在此处,误差校正模型可提取并分析堆积的单细胞特征,包括所关注碱基周围的上下文序列、所关注碱基的测序深度、所关注碱基的等位基因频率和所关注碱基周围的窗口中的碱基的等位基因频率。在多个实施方案中,“窗口”是指位于所关注碱基上游的x个碱基和位于所关注碱基下游的y个碱基。在多个实施方案中,x和y可彼此独立地为2个碱基、3个碱基、4个碱基、5个碱基、6个碱基、7个碱基、8个碱基、9个碱基、10个碱基、20个碱基、30个碱基、40个碱基、50个碱基、60个碱基、70个碱基、75个碱基、80个碱基、90个碱基、100个碱基、150个碱基、200个碱基、300个碱基、400个碱基或500个碱基。作为实例,误差校正模型可为从堆积提取单细胞特征并分析单细胞特征的神经网络(例如深度学习神经网络)。在一些实施方案中,碱基校正模块220进行特征提取过程以从堆积提取单细胞特征。在此类实施方案中,单细胞特征可作为输入提供到误差校正模型。在多个实施方案中,误差校正模型输出对应于四种核苷酸碱基(腺嘌呤、鸟嘌呤、胞嘧啶和胸腺嘧啶)的概率分布,所述概率分布代表基于所分析的单细胞特征,所关注碱基是四种核苷酸碱基中的一者的可能性。
79.在多个实施方案中,碱基校正模型220基于由误差校正模型输出的概率分布将所关注碱基校正为不同的核苷酸碱基。在一个实施方案中,碱基校正模型220将所关注碱基校正为在由误差校正模型输出的分布概率中具有最高概率的核苷酸碱基。在此处,校正的核苷酸碱基代表存在于细胞中的可能碱基。为将所关注碱基校正为不同的核苷酸碱基,碱基校正模型220校正包括所关注碱基的一个或多个序列读段以反映正确的核苷酸碱基。总之,碱基校正模型220使用更准确地反映细胞的序列的校正的核苷酸碱基再生成校正的序列读
段。
80.在多个实施方案中,碱基校正模型220校正源自具有所关注碱基的单细胞的所有序列读段,使得在校正后,校正的序列读段包括正确的碱基。在多个实施方案中,碱基校正模型220校正源自具有所关注碱基的单细胞的序列读段的一部分。例如,具有所关注碱基的一些序列读段可具有正确的碱基且因此无需校正。作为另一实例,具有所关注碱基的一些序列读段可为低置信度读段且可丢弃而非校正。在多个实施方案中,碱基校正模型220以可读文件格式(例如bam文件格式或sam文件格式)生成校正的序列读段。
81.细胞群体模块230根据细胞群体的校正的序列读段来确定细胞群体特征。通常,细胞群体模块230分析基于每个细胞组织的校正的序列读段,且确定描述细胞群体的细胞群体特征。
82.细胞群体模块230鉴别细胞群体中在已对细胞的序列读段进行误差校正后保留的一种或多种候选变体。在多个实施方案中,候选变体包括在已校正序列读段后保留的所有变体。在多个实施方案中,细胞群体模块230进行过滤,使得候选变体是在已校正序列读段后保留的所有变体的子集。例如,如果碱基满足一个或多个标准,那么细胞群体模块230鉴别特定位置的候选变体。在多个实施方案中,一个或多个标准用作硬截止,它包括以下中的一者或两者:1)最小等位基因频率和2)在该位置具有错配碱基的最少数量的细胞。
83.在多个实施方案中,为确定细胞群体的细胞群体特征,细胞群体模块230基于每个细胞聚集校正的序列读段,且然后使用聚集的序列读段确定细胞群体的细胞群体特征。例如,对于每一细胞,细胞群体模块230可量化在每一位置具有特定核苷酸碱基(例如a、c、t或g)的序列读段的比例。细胞群体模块230随后通过分析量化的序列读段比例来确定细胞群体的细胞群体特征。
84.在多个实施方案中,细胞群体模块230确定一种或多种候选变体中的每一种的细胞群体特征。作为具体实例,细胞群体特征可为特定候选变体的杂合子调用百分比(例如,在特定位置,第一拷贝的候选变体与参考碱基相比是错配的且第二拷贝的候选变体与参考碱基匹配的细胞的百分比)。因此,对于细胞,细胞群体模块230聚集细胞的校正的序列读段且确定细胞的候选变体是否是杂合子调用。细胞群体模块230在细胞群体的细胞中重复此过程以导出具有对应于候选变体的杂合子调用的细胞的百分比。对于其它候选变体,细胞群体模块230确定具有其它候选变体中的每一种的杂合拷贝的细胞的百分比。
85.细胞群体特征的实例包括(但不限于)杂合子调用百分比、杂合子调用的中值变体等位基因频率(vaf)、杂合子调用的中值基因型质量、杂合子调用的中值读段深度、纯合子调用百分比、纯合子调用的中值vaf、纯合子调用的中值基因型质量、纯合子调用的中值读段深度、参考调用百分比、用于纯合子调用的读段深度的变异系数(cv)、用于杂合子调用的读段深度的cv、纯合子调用的基因型质量的cv、杂合子调用的基因型质量的cv、用于纯合子调用的vaf的cv、用于杂合子调用的vaf的cv、用于纯合子调用的平均vaf与中值vaf之间的差、用于杂合子调用的平均vaf与中值vaf之间的差和扩增子gc百分比。
86.碱基调用程序模块240应用变体调用程序模型以预测细胞群体的一种或多种真变体。在多个实施方案中,碱基调用程序模块240将候选变体的细胞群体特征提供到变体调用程序模型作为输入。变体调用程序模型分析细胞群体特征并输出对候选变体的预测。
87.在多个实施方案中,变体调用程序是从多个可能的分类中输出候选变体的分类的
分类器。在一些实施方案中,变体调用程序模型是输出候选变体的两种分类中的一者的分类器。作为实例,变体调用程序模型可输出真变体或假阳性变体的分类。作为另一实例,变体调用程序模型可输出关于真变体类型的分类,例如纯合变体或杂合变体中的一者。在一些实施方案中,变体调用程序模型是输出候选变体的两种以上的可能分类中的一者的分类器。作为实例,变体调用程序模型可输出纯合变体、杂合变体或假阳性变体的分类。在一些实施方案中,变体调用程序模型输出不确定变体的分类。不确定变体可代表低置信度调用,它可能需要额外分析来确认不确定变体是否是真变体。在一些实施方案中,变体调用程序模型输出非变体(例如假阳性变体)的分类。
88.训练模块250通常实施用于生成误差校正模型和变体调用程序模型中的一者或两者的方法。在多个实施方案中,训练模块250由除碱基调用程序装置130以外的装置或系统来实施。例如,训练模块250可由第三方实施。在此情况下,第三方生成误差校正模型和变体调用程序模型中的一者或两者。第三方随后可将经训练的误差校正模型和经训练的变体调用程序模型中的一者或两者提供到碱基调用程序装置130。
89.在多个实施方案中,训练模块250训练误差校正模型。训练模块250可采用机器学习实施的方法来训练误差校正模型,例如线性回归算法、逻辑回归算法、决策树算法、支持向量机分类、朴素贝叶斯分类(bayes classification)、k-最近邻分类、随机森林算法、深度学习算法、梯度提升算法和维数缩减技术(例如流形学习、主要组分分析、因子分析、自动编码器正则化和独立组分分析)中的任一者或其组合。在多个实施方案中,训练模块250采用监督学习算法、非监督学习算法、半监督学习算法(例如部分监督)、转移学习、多任务学习或其任一组合来训练误差校正模型。
90.训练模块250使用误差校正训练样品来训练误差校正模型。在多个实施方案中,误差校正训练样品包括源自单个细胞的训练序列读段。此类训练样品可以常用文件格式(例如sam或bam文件格式)表示。在多个实施方案中,误差校正训练样品中的训练序列读段包括具有与参考碱基相比发生错配的已知所关注碱基的序列读段。这些训练序列读段可源自已知在已知所关注碱基的位置具有遗传变体的单个细胞。
91.在多个实施方案中,误差校正训练样品可使用指示存在于细胞中的遗传变体的已知碱基的参考基础事实来标记。在多个实施方案中,用于已知碱基的标记可为整数(例如0、1、2和3),其中每一整数值指示用于已知碱基的核苷酸碱基(例如a、c、t或g中的一者)。在多个实施方案中,用于已知碱基的标记可结构化为向量(例如1
×
4矩阵,例如[0,0,0,1])。在此类实例中,矩阵中的每一细胞对应于四种核苷酸碱基中的一种。“0”值指示相应核苷酸碱基并非已知碱基,而“1”值指示相应核苷酸碱基为已知碱基。
[0092]
在多个实施方案中,误差校正训练样品包括:1)源自具有所关注碱基的细胞的一个或多个训练序列读段,和2)指示已知碱基的标记。在多个实施方案中,训练模块250使用误差校正训练样品的一个或多个训练序列读段创建不同大小的训练堆积。因此,误差校正模型可使用源自训练样品的训练序列读段的堆积来以迭代方式训练。在训练迭代期间调整误差校正模型的参数,使得误差校正模型可更好地预测所关注碱基的概率分布。
[0093]
在多个实施方案中,训练模块250训练变体调用程序模型。训练模块250可采用机器学习实施的方法来训练变体调用程序模型,例如线性回归算法、逻辑回归算法、决策树算法、支持向量机分类、朴素贝叶斯分类、k-最近邻分类、随机森林算法、深度学习算法、梯度
提升算法和维数缩减技术(例如流形学习、主要组分分析、因子分析、自动编码器正则化和独立组分分析)中的任一者或其组合。在多个实施方案中,训练模块250采用监督学习算法、非监督学习算法、半监督学习算法(例如部分监督)、转移学习、多任务学习或其任一组合来训练变体调用程序模型。
[0094]
训练模块250使用变体调用程序训练样品来训练变体调用程序模型。在多个实施方案中,变体调用程序训练样品包括训练序列读段,所述训练序列读段包括已知变体或已知参考碱基。在多个实施方案中,变体调用程序训练样品包括源自训练序列读段的细胞群体特征。
[0095]
变体调用程序训练样品可用指示变体分类的参考基础事实来标记。在一个实施方案中,参考基础事实区分真变体与假阳性变体。在一个实施方案中,参考基础事实区分不同真变体,例如纯合变体和异质变体。在一个实施方案中,参考基础事实区分纯合变体、杂合变体与参考碱基(例如非变体)。
[0096]
在多个实施方案中,变体调用程序训练样品的标记先前可通过其它测序方法(例如批量测序方法)来确定和/或确认。在多个实施方案中,变体调用程序训练样品的标记先前可至少部分地基于存在于某些细胞系中的已知遗传变体来确定。在多个实施方案中,标记可为指示变体是真变体还是假阳性变体的二进制值(例如0或1值)。在一些实施方案中,根据变体调用程序模型经设计以预测的分类的数量,标记可为不同整数值(例如0、1、2、3等)。例如,对于预测纯合变体、杂合变体和参考碱基(例如非变体)的变体调用程序模型,标记可为三个整数值(例如0、1和2),每一整数值对应于分类中的一者。
[0097]
在多个实施方案中,每一变体调用程序训练样品包括:1)具有已知参考碱基或已知变体的细胞群体的训练序列读段,和2)指示对应于训练序列读段的已知参考碱基或已知变体的存在的标记。因此,变体调用程序模型可使用每一变体调用程序训练样品以迭代方式来训练。在多个实施方案中,在训练迭代期间调整变体调用程序模型的参数,使得变体调用程序模型可更好地预测细胞群体的序列读段是否具有参考碱基或真变体。
[0098]
用于调用细胞群体的变体的方法
[0099]
现参考图3a和图3b中所显示的流程图300和350,它们描述两步过程,所述两步过程涉及1)通过细胞特异性过程对序列读段中的碱基进行误差校正,和2)使用误差校正的序列读段在细胞群体中进行变体调用。
[0100]
图3a是根据实施方案用于校正源自单细胞的序列读段的流程图300。在步骤305处,从细胞获得序列读段。在多个实施方案中,将来自一个细胞的序列读段与来自另一细胞的序列读段区分开(例如先前使用条码技术进行区分)。另外,此类序列读段可与参考基因组进行比对。
[0101]
在步骤310处,通过校正序列读段中的错误碱基来校正细胞的序列读段。步骤310是涉及步骤315、320和325的细胞特异性过程。在多个实施方案中,对于细胞群体的一个或多个细胞中的每一者,步骤315、320和325平行进行。在多个实施方案中,对于细胞群体的一个或多个细胞中的每一者,步骤315、320和325依序进行。总之,步骤315、320和325可对细胞群体的一个或多个细胞中的每一者生成校正的序列读段。
[0102]
步骤315涉及鉴别来自细胞的序列读段的所关注碱基,所关注碱基不同于参考碱基。在多个实施方案中,鉴别所关注碱基涉及应用转换矩阵来确定碱基错配是否可能归因
于误差。应用转移矩阵涉及比较转移矩阵的概率与反映观察到序列读段的一定比例的核苷酸碱基的可能性的概率。
[0103]
步骤320涉及应用误差校正模型来预测所关注碱基的概率。在多个实施方案中,误差校正模型分析源自对所关注碱基生成的堆积的单细胞特征并输出概率分布。
[0104]
步骤325涉及校正所关注碱基。在此处,所关注碱基被校正为对应于所预测概率的不同碱基。来自含有所关注碱基的细胞的一个或多个序列读段可被校正为不同碱基。
[0105]
图3b描绘根据实施方案使用校正的序列读段来调用细胞群体的变体的流程图350。在此处,在细胞群体水平上进行步骤355、360和365,由此使得能够调用细胞群体中的真变体。
[0106]
步骤355涉及从细胞群体中的校正的序列读段生成细胞群体特征。在多个实施方案中,步骤355涉及使用校正的序列读段对细胞群体中的候选变体生成细胞群体特征。步骤360涉及将变体调用程序模型应用于细胞群体特征。在多个实施方案中,候选变体的细胞群体特征作为输入应用于变体调用程序模型。变体调用程序模型可重复应用于不同候选变体以确定每一候选变体是否是可能的真变体。
[0107]
在步骤365处,基于变体调用程序模型的输出,调用细胞群体中的一种或多种变体。在多个实施方案中,调用变体包括将候选变体调用为纯合变体、杂合变体或不确定变体中的一者。
[0108]
总之,通过流程图300和350鉴别的细胞群体的被调用变体代表超出使用常规变体调用程序管路的常规被调用变体的改良。因此,被调用变体可为多种应用提供信息,所述应用的实例包括表征异常细胞和/或疾病(例如癌症)。
[0109]
误差校正模型和变体校正模型的实施方案
[0110]
在具体实施方案中,误差校正模型和变体校正模型是机器学习模型。误差校正模型和变体校正模型中的每一者可使用训练数据来训练。在训练后,可部署误差校正模型和变体校正模型(例如根据上文参考图3a和图3b所述的过程部署)。
[0111]
在多个实施方案中,误差校正模型和变体校正模型中的一者或两者是以下中的任一者:回归模型(例如,线性回归、逻辑回归或多项式回归)、决策树、随机森林、支持向量机器、朴素贝叶斯模型、k-均值聚类或神经网络(例如,前馈网络、卷积神经网络(cnn)、深度神经网络(dnn)、自动编码器神经网络、生成对抗网络或递归网络(例如,长短期记忆网络(lstm)、双向递归网络、深度双向递归网络)。
[0112]
在多个实施方案中,误差校正模型和变体校正模型中的一者或两者具有一个或多个参数,例如超参数或模型参数。超参数通常是在训练之前确立。超参数的实例包括学习速率、决策树的深度或叶子、深度神经网络中的隐藏层数、k-均值聚类中的聚类数、回归模型中的惩罚和与成本函数相关的正则化参数。通常在训练期间调整误差校正模型和变体校正模型中的一者或两者的模型参数。模型参数的实例包括与神经网络层中的节点相关的权重、支持向量机器中的支持向量和回归模型中的系数。使用训练数据训练(例如调整)机器学习模型的模型参数以改良机器学习模型的预测力。
[0113]
在一些实施方案中,误差校正模型和变体校正模型中的一者或两者是参数模型,其中模型的一个或多个参数定义自变量与因变量之间的相依性。在多个实施方案中,参数型模型的各个参数经训练以最小化损失函数,训练通过基于梯度的数值优化算法(例如批
量梯度算法、随机梯度算法等)进行。在一些实施方案中,误差校正模型和变体校正模型中的一者或两者是非参数模型,其中模型结构是根据训练数据确定且并不严格基于固定参数集。
[0114]
图4a描绘根据实施方案的误差校正模型410的实施。在此实施方案中,误差校正模型410分析包括所关注碱基的堆积,其中堆积是从源自单细胞的序列读段生成。在多个实施方案中,误差校正模型410分析源自对所关注碱基生成的堆积的单细胞特征。单细胞特征是与所关注碱基相关的特征,包括所关注碱基周围的上下文序列、所关注碱基的测序深度、所关注碱基的等位基因频率和所关注碱基周围的窗口中的碱基的等位基因频率。基于单细胞特征,误差校正模型410输出代表所关注碱基是另一碱基的可能性的碱基概率(例如腺嘌呤、胸腺嘧啶、鸟嘌呤和胞嘧啶中的一者、两者、三者或四者的概率)的分布。
[0115]
在具体实施方案中,误差校正模型410是神经网络。在一些实施方案中,误差校正模型410是深度学习神经网络。误差校正模型410可被结构化有两个、三个、四个、五个、六个、七个、八个、九个或十个层。误差校正模型410的层包含一个或多个节点。层中的节点可连接到其它层的其它节点,节点之间的连接与参数相关。一个节点处的值可表示为连接到特定节点的节点值的组合,所述组合由与该特定节点相关的激活函数映射的相关参数加权。
[0116]
图4b描绘根据实施方案的变体调用程序模型的实施。在图4b中所显示的实施方案中,变体调用程序模型420分析源自细胞群体的校正的序列读段的细胞群体特征。变体调用程序模型420输出变体的分类。在一些实施方案中,变体的分类是真变体或假阳性变体中的一者。在一些实施方案中,变体的分类是纯合变体或杂合变体中的一者。在一些实施方案中,变体的分类是纯合变体、杂合变体或不确定变体中的一者。
[0117]
在一些实施方案中,变体调用程序模型420接收序列读段或序列读段的堆积而非细胞群体特征作为输入。在此类实施方案中,在实施变体调用程序模型420之前,无需从聚集的读段提取细胞群体特征。在一些实施方案中,聚集的读段可被编译(例如在堆积中编译)且聚集的读段的堆积可作为输入提供到变体调用程序模型420来预测变体分类。例如,可为在误差校正后与参考碱基相比是错配的碱基编译聚集的读段的堆积。变体调用程序模型420分析对碱基生成的堆积并预测碱基的变体分类。
[0118]
在具体实施方案中,变体调用程序模型420是神经网络。在一些实施方案中,变体调用程序模型420是深度学习神经网络。变体调用程序模型420可被结构化有两个、三个、四个、五个、六个、七个、八个、九个或十个层。变体调用程序模型420的层包含一个或多个节点。层中的节点可连接到其它层的其它节点,节点之间的连接与参数相关。一个节点处的值可表示为连接到特定节点的节点值的组合,所述组合由与该特定节点相关的激活函数映射的相关参数加权。
[0119]
用于测序和读段比对的方法
[0120]
本文所公开本发明的实施方案涉及核酸的测序和序列读段与参考基因组的比对。在多个实施方案中,对核酸测序并比对序列读段与参考基因组的步骤是通过测序仪(例如细胞分析工作流装置120的测序仪)来进行,如上文参考图1所述。因此,经测序和比对的序列读段可通过碱基调用程序装置130来分析,且更具体来说可通过碱基鉴别模块210(参见图2)来分析以鉴别所关注碱基。
[0121]
序列读段可以通过可商购获得的下一代测序(ngs)平台来实现,所述ngs平台包括执行边合成边测序、边连接边测序、焦磷酸测序、使用可逆终止子化学测序、使用连接磷的荧光核苷酸测序或实时测序中的任一者的平台。例如,扩增的核酸可以在illumina miseq平台上进行测序。
[0122]
当进行焦磷酸测序时,将ngs片段的文库克隆,通过使用包被有与衔接子互补的寡核苷酸的颗粒捕获一个基质分子来原位扩增。每个含有相同类型基质的颗粒被放置在“油包水”类型的微气泡中,并且使用称为乳液pcr的方法克隆扩增基质。扩增后,乳液被破坏,并且颗粒被堆叠于在测序反应期间充当流动池的滴定微微板(picoplate)的单独孔中。在存在测序酶和发光报告剂诸如萤光素酶的情况下将四种dntp试剂中的每一种有序地多次施用到流动池中。在将适宜dntp添加到测序引物的3
′
端的情形下,所得atp在孔内产生闪光,使用ccd照相机记录所述闪光。有可能实现大于或等于400个碱基的读段长度,并且有可能获得序列的106个读段,从而产生高达500百万个碱基对(兆字节)的序列。voelkerding等人,clinical chem.,55:641-658,2009;maclean等人,nature rev.microbiol.,7:287-296;美国专利第6,210,891号;美国专利第6,258,568号描述关于焦磷酸测序的其它细节;所述文献中的每一者的全文以引用方式在此并入。
[0123]
在solexa/illumina平台上,以短读段的形式产生测序数据。在这种方法中,ngs片段文库的片段被捕获在包被有寡核苷酸锚定分子的流动池的表面上。将锚定分子用作pcr引物,但由于基质的长度和所述分子靠近其它邻近的锚定寡核苷酸,通过pcr延伸导致分子形成“拱顶”,其中所述分子与相邻锚定寡核苷酸杂交,并且在流动池表面上形成桥接结构。这些dna环被变性和切割。然后使用可逆染色终止子对直链进行测序。所述序列中包含的核苷酸通过检测包含之后的荧光来确定,其中在下一个dntp添加循环之前去除每个荧光剂和封闭剂。关于使用illumina平台测序的其它细节参见voelkerding等人,clinical chem.,55:641-658,2009;maclean等人,nature rev.microbiol.,7:287-296;美国专利第6,833,246号;美国专利第7,115,400号;美国专利第6,969,488号;所述文献中的每一者的全文以引用方式在此并入。
[0124]
使用solid技术对核酸分子进行测序包括使用乳液pcr克隆扩增ngs片段文库。此后,将含有基质的颗粒固定在玻璃流动池的衍生化表面上,并且用与衔接寡核苷酸互补的引物退火。然而,使用它代替使用指示的引物进行3'延伸来获得5'磷酸基团用于连接含有两个探针特异性碱基和随后6个简并碱基和四种荧光标记中的一者的测试探针。在solid系统中,测试探针具有在每个探针的3'端的两个碱基和在5'端的四种荧光染料中的一者的16种可能的组合。荧光染料的颜色和因此每个探针的属性对应于某一颜色空间编码方案。在探针比对、探针连接和荧光信号检测的多个循环后,在变性后使用与原始引物相比移位一个碱基的引物进行第二个测序循环。以这种方式,可以通过计算重构矩阵的序列;对矩阵碱基检查两次,这导致准确性增加。关于使用solid技术测序的其它细节参见voelkerding等人,clinical chem.,55:641-658,2009;maclean等人,nature rev.microbiol.,7:287-296;美国专利第5,912,148号;美国专利第6,130,073号;所述文献中的每一者的全文以引用方式并入。
[0125]
在特定的实施方案中,使用来自helicos biosciences的heliscope。通过添加聚合酶和连续添加荧光标记的dntp试剂来实现测序。接通导致出现对应于dntp的荧光信号,
并且在每个dntp添加循环之前,ccd照相机捕获到指定的信号。序列的读段长度从25-50个核苷酸变化,其中每个分析工作周期的总产量超过10亿个核苷酸对。关于使用heliscope进行测序的其它细节参见voelkerding等人,clinical chem.,55:641-658,2009;maclean等人,nature rev.microbiol.,7:287-296;美国专利第7,169,560号;美国专利第7,282,337号;美国专利第7,482,120号;美国专利第7,501,245号;美国专利第6,818,395号;美国专利第6,911,345号;美国专利第7,501,245号;所述文献中的每一者的全文以引用方式并入。
[0126]
在一些实施方案中,使用roche测序系统454。测序454涉及两个步骤。在第一步骤中,将dna切割成约300-800个碱基对的片段,并且这些片段具有钝端。然后将寡核苷酸衔接子连接到片段的末端。衔接子作为引物用于片段的扩增和测序。片段可例如使用含有5'-生物素标签的衔接子连接到dna捕获珠粒,例如链霉亲和素包被的珠粒。在油-水乳液的液滴内通过pcr扩增连接到颗粒的片段。结果是克隆扩增的dna片段在每个珠粒上有多个拷贝。在第二阶段,颗粒被捕获到孔中(体积为几皮升)。平行地对每个dna片段进行焦磷酸测序。添加一个或多个核苷酸可生成光信号,它被记录在测序仪器的ccd照相机上。信号强度与所纳入的核苷酸数量成比例。焦磷酸测序使用焦磷酸(ppi),其在添加核苷酸时被释放。在腺苷5'磷酸硫酸酯存在下,使用atp硫酸化酶将ppi转化成atp。萤光素酶使用atp将萤光素转化为氧化萤光素,并且作为这种反应的结果,产生进行检测和分析的光。关于进行测序454的其它细节参见margulies等人(2005)nature 437:376-380,所述文献的全文以引用方式在此并入。
[0127]
离子激流技术是一种基于检测dna聚合期间释放的氢离子的dna测序方法。微孔含有待测序的ngs片段文库的片段。微孔层下是超灵敏离子传感器isfet。所有层都包含在半导体cmos芯片内,类似于电子工业中使用的芯片。当将dntp纳入不断增长的互补链中时,释放出激发超灵敏离子传感器的氢离子。如果模板序列中存在均聚物重复,那么在一个循环中将包含多个dntp分子。这导致对应量的氢原子被释放,并且与更高的电信号成比例。此技术不同于其他不使用修饰的核苷酸或光学装置的测序技术。关于离子洪流技术的其它细节参见science 327(5970):1190(2010);美国专利申请公开第20090026082号、美国专利申请公开第20090127589号、美国专利申请公开第20100301398号、美国专利申请公开第20100197507号、美国专利申请公开第20100188073号和美国专利申请公开第20100137143号,所述文献中的每一者的全文以引用方式并入。
[0128]
在多个实施方案中,从ngs方法获得的测序读段可通过质量进行过滤且根据条码序列使用本领域已知的任何算法(例如python脚本barcodecleanup.py)分组。在一些实施方案中,如果给定测序读段的超过约20%的碱基具有小于q20的质量评分(q-评分),此表明碱基调用准确性小于约99%,那么可丢弃给定测序读段。在一些实施方案中,如果超过约5%、约10%、约15%、约20%、约25%、约30%具有小于q10、q20、q30、q40、q50、q60或更大的q-评分,此分别表明碱基调用准确性小于约90%、小于约99%、小于约99.9%、小于约99.99%、小于约99.999%、小于约99.9999%或更大,那么可丢弃给定测序读段。
[0129]
在一些实施方案中,可以丢弃与含有少于50个读段的条形码相关联的所有测序读段,以确保表示单细胞的所有条形码组含有足够数量的高质量读段。在一些实施方案中,可丢弃与含有少于30个、少于40个、少于50个、少于60个、少于70个、少于80个、少于90个、少于100个或更多个读段的条码相关的所有测序读段以确保代表单细胞的条码组的质量。
[0130]
具有共有条形码序列的序列读段(例如,意味着序列读段源自同一细胞)可以使用本领域已知的方法与参考基因组比对以确定比对位置信息。比对位置信息可以指示参考基因组中与给定序列读段的开始核苷酸碱基和结束核苷酸碱基对应的区域的开始位置和结束位置。参考基因组中的区域可与靶基因或基因区段相关。示例性比对器算法包括bwa、bowtie、与参考序列的拼接转录物比对(star)、tophat或hisat2。关于比对序列读段与参考序列的其它细节描述于美国申请第16/279,315号中,所述美国申请的全文以引用方式在此并入。在多个实施方案中,可生成并输出具有sam(序列比对图)格式或bam(二进制比对图)格式的输出文件,用于后续分析。
[0131]
系统和/或计算机实施方案
[0132]
本文所述的实施方案还涉及用于进行上文所述的变体调用方法的示例性系统和计算机实施方案。后续描述涉及细胞分析工作流装置120和碱基调用程序装置130,如上文参考图1所述。
[0133]
在多个实施方案中,细胞分析工作流装置120包括至少微流体装置,它经配置以用试剂囊封细胞、用反应混合物囊封细胞裂解物并进行核酸扩增反应。例如,微流体装置可以包括流体地连接的一个或多个流体通道。因此,通过第一通道的水性流体和通过第二通道的载体流体的组合可生成乳液液滴。在各种实施方案中,微流体装置的流体通道可以具有毫米或更小量级(例如,小于或等于约1毫米)的至少一个横截面尺寸。微通道设计和尺寸的其它细节描述于国际专利申请第pct/us2016/016444号和美国专利申请第14/420,646号,所述专利申请中的每一者的全文以引用方式并入。微流体装置的一个实例是tapestri
tm
平台。
[0134]
在多个实施方案中,细胞分析工作流装置120还可包括以下中的一者或多者:(a)温度控制模块,用于控制主题装置的一个或多个部分和/或其中的液滴的温度且可操作地连接到微流体装置,(b)可操作地连接到微流体装置的检测模块,即检测器,例如光学成像仪,(c)可操作地连接到微流体装置的培育器,例如细胞培育器,和(d)可操作地连接到微流体装置的测序仪。一个或多个温度和/或压力控制模块提供对装置的一个或多个流动通道中的载体流体的温度和/或压力的控制。作为实例,温度控制模块可以是调节用于执行核酸扩增的温度的一个或多个热循环仪。一个或多个检测模块(即检测器,例如光学成像仪)经配置以检测一个或多个液滴的存在或其一个或多个特性,包括它们的组成。在一些实施方案中,检测模块经配置以识别一个或多个流动通道中的一个或多个液滴的一种或多种组分。所述测序仪是被配置成执行测序(诸如下一代测序)的硬件装置。测序仪的实例包括illumina测序仪(例如,miniseq
tm
、miseq
tm
、nextseq
tm 550 series或nextseq
tm 2000)、roche测序系统454和thermo fisher科学测序仪(例如ion genestudio s5系统、ion torrent genexus系统)。
[0135]
图5描绘用于实施参考图1-4描述的系统和方法的示例性计算装置。在多个实施方案中,示例性计算装置500用作图1中所述的碱基调用程序装置130用于进行误差校正和调用变体。计算装置的实例可以包括个人计算机、台式计算机、膝上型计算机、服务器计算机、集群内的计算节点、信息处理器、手持装置、多处理器系统、基于微处理器或可编程的消费者电子装置、网络pc、小型计算机、主机计算机、移动电话、pda、平板电脑、传呼机、路由器、交换机等。
[0136]
如图5中所显示,在一些实施方案中,计算装置500包括耦接到芯片组504的至少一个处理器502。芯片组504包括存储器控制器集线器520和输入/输出(i/o)控制器集线器522。存储器506和图形适配器512耦接到存储器控制器集线器520,且显示器518耦接到图形适配器512。存储装置508、输入接口514和网络适配器516耦接到i/o控制器集线器522。计算装置500的其他实施方案具有不同的架构。
[0137]
存储装置508是非暂时性计算机可读存储介质,例如硬盘驱动器、光盘只读存储器(cd-rom)、dvd或固态存储器装置。存储器506保存处理器502使用的指令和数据。输入接口514是触摸屏接口、鼠标、轨迹球或其它类型的输入接口、键盘或其某种组合,并且用于将数据输入到计算装置500中。在一些实施方案中,计算装置500可经配置以经由用户的手势从输入接口514接收输入(例如命令)。图形适配器512在显示器518上显示图像和其它信息。网络适配器516将计算装置500耦接到一个或多个计算机网络。
[0138]
计算装置500被调适来执行用于提供本文描述的功能的计算机程序模块。如本文中所使用,术语“模块”是指用于提供指定功能的计算机程序逻辑。因此,模块可以在硬件、固件和/或软件中实施。在一个实施方案中,程序模块存储在存储装置508上,加载到存储器506中,并由处理器502执行。
[0139]
计算装置500的类型可以不同于本文所述的实施方案。例如,计算装置500可缺少上述组件中的一些,例如图形适配器512、输入接口514和显示器518。在一些实施方案中,计算装置500可包括用于执行存储在存储器506上的指令的处理器502。
[0140]
进行碱基误差校正和变体调用的方法可在硬件或软件或二者的组合中实施。在一个实施方案中,提供非暂时性机器可读存储介质,例如上文所述的非暂时性机器可读存储介质,所述介质包括用机器可读数据编码的数据存储材料,当使用被编程有使用所述数据的指令的机器时,所述数据存储材料能够执行用于进行本文所公开的碱基误差校正和变体调用方法的指令。上文所述的方法的实施方案可以在可编程计算机上执行的计算机程序中实现,所述可编程计算机包括处理器、数据存储系统(包括易失性和非易失性存储器和/或存储元件)、图形适配器、输入接口、网络适配器、至少一个输入装置和至少一个输出装置。显示器耦接至图形适配器。程序代码被应用于输入数据以执行上文所述的功能并生成输出信息。以已知方式将输出信息应用于一个或多个输出装置。计算机可为例如常规设计的个人计算机、微型计算机或工作站。
[0141]
每个程序可用高级程序或面向对象的程序化语言来实施以与计算机系统通信。然而,若需要,程序可以汇编或机器语言来实施。在任一情形下,语言都可为编译的或解释的语言。每个这样的计算机程序优选地存储在可由通用或专用目的可程序化计算机读取的存储介质或装置(例如rom或磁盘)上,用于当计算机读取存储介质或装置以执行本文所述的程序时配置并操作计算机。所述系统还可视为作为配置有计算机程序的计算机可读存储介质来实施,其中如此配置的存储介质使计算机以特定和预定义的方式操作以执行本文所述的功能。
[0142]
可在多种介质中提供签名模式和其数据库以便于它们的使用。“介质”是指含有本发明的签名模式信息的制品。本发明的数据库可记录在计算机可读介质(例如计算机可直接读取和访问的任何介质)上。此类介质包括(但不限于):磁性存储介质,例如软盘、硬盘存储介质和磁带;光存储介质,例如cd-rom;电存储介质,例如ram和rom;以及这些类别的混合
体,例如磁/光存储介质。本领域技术人员可容易地了解如何使用当前已知任一计算机可读介质来创建包含当前数据库信息记录的制品。“记录的”是指使用如本领域中已知的任何此类方法在计算机可读介质上存储信息的过程。基于用于访问所存储信息的手段,可选择任何方便的数据存储结构。可以使用多种数据处理器程序和格式进行存储,例如文字处理文本文件、数据库格式等。
[0143]
实施例
[0144]
实施例1:在应用误差校正模型之前,在序列读段中观察到的碱基误差
[0145]
图6描绘碱基误差的示例性分布,其中大部分碱基误差仅在一种细胞中观察到。图6中量化的误差是指在未应用误差校正模型的情况下存在于序列读段中的误差。
[0146]
数据是从细胞系样品内部生成,通过tapestri
tm
运行,并使用tapestri
tm
标准管路分析。获得每个细胞的误差(错配)且计算细胞中误差的频率以生成该图。具体来说,仅在1个细胞中观察到序列读段中的大多数误差,在多于1个细胞中观察到有限数量的序列读段误差。此表明对单个细胞中的序列读段进行校正可减少被错误地鉴别为与参考碱基相关的匹配碱基或错配碱基的误差(例如假阳性和/或假阴性)数量。换句话说,如果源自细胞的序列读段的碱基被确定为误差,那么源自相同细胞的其它序列读段的相同碱基更可能是误差。因此,进行单个细胞的序列读段的细胞特异性误差校正比常规方法(例如通过批量处理获得的误差校正读段)更准确和/或快速。
[0147]
实施例2:实施误差校正模型的示例性方法
[0148]
通常,下文关于图7-10描述的用于实施误差校正模型的示例性方法是指对源自单个细胞的序列读段中的碱基进行误差校正。
[0149]
对样品生成转换矩阵,例如图7中所显示的转换矩阵。通过量化样品的400万个读段的已知碱基来生成转换矩阵的概率,将这些读段与参考基因组进行比对。对于已知参考碱基(例如腺嘌呤、胸腺嘧啶、鸟嘌呤或胞嘧啶的已知参考碱基),测定4百万个探针中4种核苷酸碱基中的每一者的观察量,以生成转换矩阵的相对概率。
[0150]
鉴别细胞序列读段中的错配碱基。对于每个碱基,计算多项式概率,所述多项式概率反映在序列读段的所述位置观察到所述比例的替代碱基(例如,不同于参考碱基的3种核苷酸碱基中的任一者)的可能性。具体来说,根据以下公式计算位置的多项式概率:
[0151]
p(碱基1=x,碱基2=y,碱基3=z|n读段)
[0152]
其中碱基1、碱基2和碱基3是指与参考碱基不匹配的核苷酸碱基,其中x是在该位置观察到的具有碱基1的序列读段的数量,其中y是在该位置观察到的具有碱基2的序列读段的数量,其中z是在该位置观察到的具有碱基3的序列读段的数量,且其中n是在该位置观察到的序列读段的总数量。
[0153]
比较碱基的多项式概率与转换矩阵的转换概率。转换概率反映从参考核苷酸碱基转换成观察到的核苷酸碱基的可能性。如果多项式概率大于转换矩阵的转换概率,那么将碱基鉴别为所关注碱基。如果多项式概率小于转变矩阵的转变概率,那么不将碱基鉴别为所关注碱基。
[0154]
为每一所关注碱基创建堆积。图8a和图8b是不同位置的6个序列读段的堆积的示例性描绘。图8a和图8b各自描绘示例性位置0-14(顶行)。图8a进一步鉴别每个相应位置(第二行)的参考碱基以及六个比对序列读段中每一者的碱基。图8b描绘在六个序列读段中已
量化的每个碱基的概率。本领域技术人员可容易地理解,基因组中的额外位置(例如数千或数百万个位置)、额外参考碱基(例如数千或数百万个参考碱基)和额外序列读段碱基(例如数千或数百万个额外序列读段)可包括在示例性堆积中。
[0155]
在此处,对与参考碱基相比发生错配的所关注碱基生成实例性堆积。具体来说,对位置7生成实例性堆积。参考碱基指示位置7的胞嘧啶碱基,但6个序列读段中的5个(83%)包括错配的鸟嘌呤碱基。
[0156]
图9a描绘误差校正模型的示例性输入和输出。在此实施例中,堆积(例如图8b中所显示的堆积)作为输入提供到误差校正模型来校正所关注碱基。在此处,误差校正模型是深度学习神经网络(dnn)。使用若干不同的超参数优化误差校正模型以鉴别每一超参数的最佳值。超参数包括(但不限于)核正则化系数、学习速率、层数、激活函数和优化器。
[0157]
误差校正模型分析堆积的单细胞特征,包括所关注碱基周围的上下文序列、所关注碱基的测序深度和所关注碱基的等位基因频率。
[0158]
误差校正模型输出四种核苷酸碱基(腺嘌呤、胞嘧啶、鸟嘌呤、胸腺嘧啶)的概率分布,其中每个概率指示所关注碱基是特定碱基的可能性。在图9a所显示的实施例中,误差校正模型输出概率分布,表明所关注碱基是腺嘌呤的可能性为20%,所关注碱基是胞嘧啶的可能性为0%,所关注碱基是鸟嘌呤的可能性为70%,且所关注碱基是胸腺嘧啶的可能性为10%。
[0159]
图9b描绘使用由误差校正模型预测的概率校正所关注碱基的实例。图9b中所显示的前两列鉴别碱基的位置,包括碱基所定位的染色体以及碱基的参考位置。第三列鉴别所关注碱基已经被校正到的校正碱基,此取决于误差校正模型输出的概率。在此处,第四列显示由误差校正模型输出的概率。
[0160]
具体来说,对于第一行,输出的概率表明所关注碱基最可能是腺嘌呤核苷酸碱基,假设它具有最高的概率(例如,0.6748)。因此,所关注碱基被校正为腺嘌呤。对于第二行,输出的概率表明所关注碱基最可能是胞嘧啶核苷酸碱基,假设它具有最高的概率(例如,0.9127)。对于第三行,输出的概率表明所关注碱基最可能是胞嘧啶核苷酸碱基,假设它具有最高的概率(例如,0.83465)。对于第四行,输出的概率表明所关注碱基最可能是胸腺嘧啶核苷酸碱基,假设它具有最高的概率(例如,0.6193)。
[0161]
图10展示校正四个不同细胞群体中的20%-35%碱基作为实施误差校正模型的结果。通过单细胞工作流装置(例如)处理四种细胞系中的每一者,且单细胞dna经测序以生成序列读段。对于每一细胞,将误差校正模型应用于误差校正源自细胞的序列读段中的所关注碱基。
[0162]
总之,将误差校正模型应用于单细胞dna序列读段可鉴别并校正很大比例的错误碱基,所述错误碱基可能是由于pcr误差、测序误差、测序比对误差或校正误差中的任一者引起。这些校正的序列读段使得能够进行更准确的变体调用,如下文实施例3中所述。
[0163]
实施例3:实施变体调用程序模型的方法
[0164]
对序列读段进行误差校正后,过滤变体以去除不符合阈值(例如最小等位基因频率和突变细胞数)的变体。对于剩余变体中的每一变体,计算细胞群体特征:杂合子调用百分比、杂合子调用的中值变体等位基因频率(vaf)、杂合子调用的中值基因型质量、杂合子调用的中值读段深度、纯合子调用百分比、纯合子调用的中值vaf、纯合子调用的中值基因
型质量、纯合子调用的中值读段深度、参考调用百分比、用于纯合子调用的读段深度的变异系数(cv)、用于杂合子调用的读段深度的cv、纯合子调用的基因型质量的cv、杂合子调用的基因型质量的cv、用于纯合子调用的vaf的cv、用于杂合子调用的vaf的cv、用于纯合子调用的平均vaf与中值vaf之间的差、用于杂合子调用的平均vaf与中值vaf之间的差和扩增子gc百分比。
[0165]
使用从19个样品中获得的细胞的细胞群体特征来训练变体调用程序模型,在此情况下,该模型是多类神经网络分类器。下表1公开训练样品。对于这些样品,将已知变体基于各自样品中存在的已知真变体(根据批量测序方法确认)进行分类(异质变体、纯合变体或参考碱基)。训练样品包括来自各种稀释度高达0.1%的细胞混合物的各种样品,以及通过tapestri仪器处理并在各种测序仪上测序的临床样品。由于训练数据具有类别不平衡,其中某些类别与其它类别相比具有少得多的调用,所以对较小的类别进行上采样。将模型的超参数使用具有已知真标记的验证数据进行迭代调整。一旦模型实现足够的准确性,训练就停止,且然后对新样品以预测模式使用该模型来鉴别那些样品中的顶级变体。
[0166]
使用十三个测试样品来评估变体调用程序模型的性能。下表2公开测试样品。图11展示在13个样品中实施变体调用程序模型之后真变体的改良的阳性预测值。通过两步误差校正模型和变体预测模型,实现显著改良的中值阳性预测值(ppv)。具体来说,在13个样品的大多数中观察到在0.5%lod时ppv改良2-3倍。与采用硬截止过滤器的常规gatk模型相比,观察到2-3倍的改良,这与误差校正模型和/或变体预测模型相反。
[0167]
表1:用于训练变体调用程序模型的训练样品。
[0168][0169]
表2:用于验证变体调用程序模型的测试样品。
[0170][0171]
总之,这些结果证实,误差校正模型和变体调用程序模型的应用实现变体调用的显著改良。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。