1.本发明属于生物技术领域,具体涉及一种全基因组羟甲基化转化试剂及其应用。
背景技术:
2.dna甲基化是基因组dna的一种主要表观遗传修饰形式,研究表明dna甲基化与癌症之间有非常密切的联系,可以将其作为肿瘤早期诊断和预后评价的重要指标。过去认为,哺乳动物dna甲基化非常稳定,但越来越多的研究表明这一过程是动态可逆的。因此,近十几年来人们致力寻找dna甲基化的逆反应,即直接将dna去甲基化,但未能成功。而第六种碱基5-羟甲基胞嘧啶(5-hydroxy-methylcytosine,5hmc)的发现为dna去甲基化研究提供了新的思路。目前,相关的分析检测与测序技术快速发展,大大促进了人们对5hmc的探索研究以及对相关的生物学功能的认识。大量的研究显示5hmc是5mc在去甲基化过程中的一种重要的中间体,实际上,基因组中5hmc含量较低,这与其是一种短暂产生的中间体的推断是相一致的。虽然,5hmc的生物学功能还不是很明确,但是可以明确的是5hmc和tets(ten-eleven translocation蛋白)在全基因组的表观遗传重组和组织特异性基因表达的调控中起到了非常重要的作用。在复杂的生理过程中要深入研究5hmc的生物学功能,最关键的就是准确而高通量的对其进行定位,并且能够进一步对不同组织基因组dna中的5hmc进行定量。
3.得益于dna测序技术的蓬勃发展,dna甲基化测序技术得到了突破性进展。dna甲基化测序实质上是通过重亚硫酸盐反应,将含有修饰位点的测序转化为正常dna测序的过程。首先,对待测基因组dna进行重亚硫酸盐处理,未甲基化胞嘧啶转化为尿嘧啶,而甲基化胞嘧啶不发生变化,然后进行pcr扩增,克隆到载体后,可采用一般的dna测序技术测序,包括下一代dna测序技术和单分子dna测序技术。通过与未处理基因组dna的序列进行比对,判断基因组dna的甲基化位点及覆盖程度。但是,经典的重亚硫酸盐测序法也不能区分5mc和5hmc。经重亚硫酸盐处理后,未甲基化的胞嘧啶可通过脱氨基和脱磺酸基作用转化为尿嘧啶,再经pcr扩增转变成t,而5mc经处理后不能转化为胞嘧啶c,pcr扩增后仍为c,从而可将5mc与未修饰的c区分。但5hmc由于不能脱氨基而形成5-亚甲基磺酸胞嘧啶(5-methylenesμl-phonate,cms)。因此,在重亚硫酸盐测序中5hmc和5mc都不能脱氨基,从而pcr扩增后二者不能产生序列差异,即5mc和5hmc不能被区分。另外,在pcr过程中cms还具有抑制dna聚合酶的效果。并且,重亚硫酸盐处理后的dna损伤大,建好的文库中at占比>80%,碱基严重不平衡,上机检测需要混合其他全基因组文库进行测序,操作不便。所以,需要开发针对5hmc的检测方法,从而达到单碱基分辨率的测序效果。
技术实现要素:
4.本发明旨在至少解决上述现有技术中存在的技术问题之一。为此,本发明提出一种全基因组羟甲基化转化试剂,可特异性地识别并转换5hmc,即只将羟甲基化修饰的胞嘧啶转换成胸腺嘧啶。
5.本发明还提出一种具有上述全基因组羟甲基化转化试剂的试剂盒。
6.本发明还提出一种上述全基因组羟甲基化转化试剂和上述试剂盒的应用。
7.根据本发明的一个方面,提出了一种全基因组羟甲基化转化试剂,所述试剂包括氧化剂和还原剂,所述氧化剂包括金属氧化物,所述还原剂包括2-甲基吡啶硼烷,所述氧化剂可特异性地识别并转换5hmc,并将5hmc氧化成5-甲酰基胞嘧啶(5fc),所述还原剂可将5-甲酰基胞嘧啶(5fc)还原为二氢尿嘧啶(dhu),pcr扩增时将dhu当做“t”,从而实现转化目的。
8.在本发明的一些实施方式中,所述全基因组羟甲基化转化试剂将5-羟甲基胞嘧啶转化为胸腺嘧啶。
9.在本发明的一些实施方式中,所述金属氧化物为钌酸盐氧化剂、mno2、kmno4中的至少一种。
10.在本发明的一些实施方式中,所述钌酸盐氧化剂为k2ruo4。
11.在本发明的一些实施方式中,所述氧化剂还包括氢氧化钠、氢氧化钾、氢氧化镁和氢氧化钙的一种。采用含氢氧根的强碱性物质,在强碱性环境下,双链dna解旋,但不涉及磷酸二酯键的断裂,其一级结构(碱基顺序)保持不变。
12.在本发明的一些实施方式中,所述还原剂还包括乙酸钠缓冲溶液,作为促进2-甲基吡啶硼烷进行底物还原的反应缓冲体系。
13.在本发明的第二方面,提出了一种试剂盒,所述试剂盒包括上述全基因组羟甲基化转化试剂。
14.在本发明的一些实施方式中,所述试剂盒还包括阳性标准品。
15.在本发明的一些实施方式中,所述阳性标准品为seq id no.1所示序列。
16.在本发明的一些实施方式中,所述试剂盒还包括pcr扩增试剂。
17.在本发明的一些实施方式中,所述pcr扩增试剂包含dna聚合酶。
18.在本发明的第三方面,提出了上述全基因组羟甲基化转化试剂和上述试剂盒的应用,所述应用为在全基因组羟甲基化测序、dna羟甲基化测序或靶向区域dna羟甲基化测序中的用途。
19.在本发明的第四方面,提出了一种全基因组羟甲基化测序文库的构建方法,包括使用上述试剂或试剂盒将5-羟甲基胞嘧啶转化为胸腺嘧啶。
20.在本发明的一些实施方式中,所述方法包括以下步骤:
21.s1、片段化dna;
22.s2、片段化dna末端修复加a;
23.s3、加a产物加接头得到加接头的dna文库;
24.s4、将加接头的dna文库进行氧化,得到氧化的dna文库;
25.s5、将氧化的dna文库进行还原得到还原的dna文库;
26.s6、将还原后的dna文库进行pcr扩增得到最终文库dna。
27.在本发明的一些实施方式中,所述方法包括将待从5-羟甲基胞嘧啶到胸腺嘧啶转化的dna文库进行氧化反应的步骤,所述步骤包括:将氧化反应体系与氧化剂混合进行氧化反应;
28.以48.5μl为计,所述氧化反应体系如下:
29.dna文库,80-120ng;
30.1m naoh,1.5-3μl;
[0031]1×
ssc,补足至48.5μl。
[0032]
在本发明的一些实施方式中,在dna文库进行氧化反应的步骤中,反应条件为:将氧化反应体系于35-40℃,300-600rpm反应20-60min后加入氧化剂,于0-4℃孵育0.5h-2h。
[0033]
在本发明的一些实施方式中,还包括将氧化反应体系得到的产物加入所述还原剂进行还原反应的步骤。
[0034]
在本发明的一些实施方式中,在所述的方法中,将扩增后的文库进行二代测序,测序平台包括但不限于illumina平台、mgi或gene seq平台;优选的,所述测序平台为illumina平台。
[0035]
在本发明的第五方面,提出了利用上述方法构建的全基因组羟甲基化测序文库。
[0036]
根据本发明的实施方式,至少具有以下有益效果:本发明方案的全基因组羟甲基化转化试剂,可特异性地识别并转换5hmc,只将羟甲基化修饰的胞嘧啶转换成t,而甲基化修饰的胞嘧啶不进行转换,解决了现有技术中基于重亚硫酸氢盐转化构建的甲基化测序文库所存在的碱基不平衡,测序数据使用率低的缺陷,组分简单,成本低,效率高;同时本发明方案还提供了一种全基因组羟甲基化的测序文库的构建,方法简单高效,构建得到的测序文库无需中重亚硫酸盐的处理,可直接用于测序,碱基平衡、基因组比对率高。
附图说明
[0037]
下面结合附图和实施例对本发明做进一步的说明,其中:
[0038]
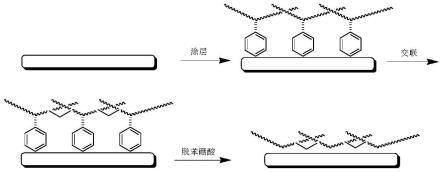
图1为本发明实施例2中的全基因组羟甲基化转化试剂原理图;
[0039]
图2为本发明实施例2中的构建文库的操作流程图;
[0040]
图3为本发明测试例中的不同氧化剂的对dna的损伤的对比结果图。
具体实施方式
[0041]
以下将结合实施例对本发明的构思及产生的技术效果进行清楚、完整地描述,以充分地理解本发明的目的、特征和效果。显然,所描述的实施例只是本发明的一部分实施例,而不是全部实施例,基于本发明的实施例,本领域的技术人员在不付出创造性劳动的前提下所获得的其他实施例,均属于本发明保护的范围。
[0042]
试剂来源:naoh购自西陇科学;高钌酸钾购自sigma公司;无菌水购自北京索莱宝生物科技有限公司;2-甲基吡啶硼烷购自sigma公司;dmso购自北京索莱宝生物科技有限公司;吡啶硼烷溶液购自sigma-aldrich。
[0043]
实施例1全基因组羟甲基化转化试剂
[0044]
本实施例制备了一种全基因组羟甲基化转化试剂,包括氧化剂和还原剂,氧化剂为k2ruo4,由浓度为15mm k2ruo4和50mm naoh组成,还原剂为浓度为5mol/l 2-甲基吡啶硼烷溶液。
[0045]
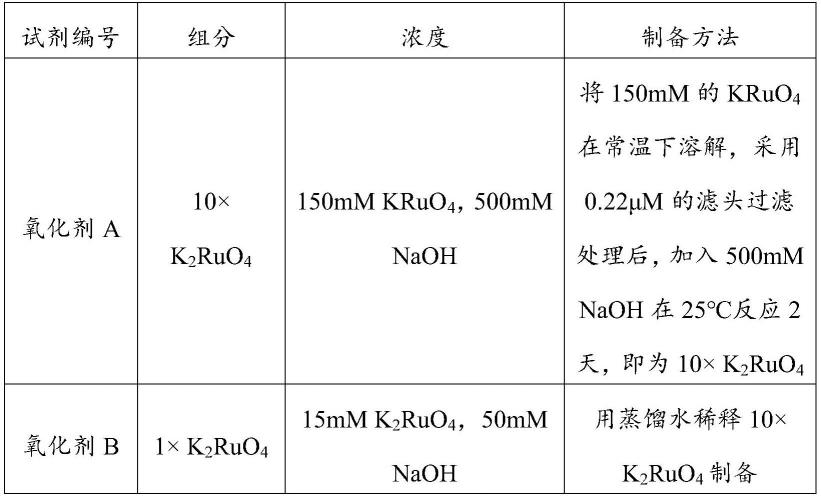
(1)氧化剂的配方如表1所示。
[0046]
表1
[0047][0048]
(2)还原剂的配方如表2所示。
[0049]
表2
[0050][0051]
实施例2全基因组羟甲基化非重亚硫酸氢盐测序文库
[0052]
本实施例提供了一种全基因组羟甲基化非重亚硫酸氢盐测序文库,全基因组羟甲基化转化试剂原理示意图和建库操作流程图如图1-2所示,具体构建方法为:
[0053]
1、甲基化和羟甲基化阳性标准品的制备
[0054]
本发明使用的阳性标准品是人工合成的一段含有甲基化和羟甲基化位点的序列,具体序列如下,/i5medc/表示甲基化修饰的胞嘧啶,/55hydmec/表示羟甲基化修饰的胞嘧啶,序列如表3所示,由上海生工合成。
[0055]
表3
[0056][0057]
将上述合成的序列按照合成产物说明书用10mm tris-hcl(ph8.0)定量稀释至100μm,然后进行退火处理。退火体系如表4所示。
[0058]
表4
[0059][0060][0061]
10
×
anealing buffer的具体配方为:1l 10
×
anealing buffer中含有100mm tris-hcl(ph8.0),500mm nacl,10mm edta。退火反应参数如表5所示。
[0062]
表5
[0063][0064]
退火结束后,退火产物即为阳性标准品储存液(40μm),可以直接用于或者适量稀释后用于连接反应,也可以在-20℃冻存备用。后续方案中所用阳性标准品即为上述退火产
物。
[0065]
2、dna片段化
[0066]
取100ng-1000ng的基因组gdna进行超声打断,基因组gdna包括人基因组gdna和外源性内参阳性dna(在本实施例中外源性内参阳性dna为上述的人工合成的阳性标准品),打断后的gdna用2
×
磁珠纯化回收,32μl elution buffer(组分:10mm tris-hcl,1mm edta,ph8.0)洗脱,即得片段化dna。本实施例所用的磁珠为ampure xp beads(购自贝克曼)。
[0067]
3、末端修复和加a
[0068]
(1)取步骤2中的片段化dna进行末端修复和加a,其反应体系如表6所示。
[0069]
表6
[0070]
mix组分体积(μl)片段化的dna30末端修复加a-缓冲液11.5末端修复加a-酶mix3.5db补足至50μl合计50
[0071]
注:末端修复加a-缓冲液、酶mix均由enzymatics公司提供。
[0072]
(2)反应条件如表7所示。
[0073]
表7
[0074]
步骤温度(℃)时间末端修复3730min加a7220min保持4∞
[0075]
注:步骤3与下一步反应可中断,期间需将制备得到的末端修复和加a的产物,暂存于4℃,暂存时间以不超过4h。
[0076]
4、连接反应及连接产物纯化
[0077]
(1)将步骤3中得到的末端修复和加a的产物使用连接酶进行连接加接头(购自idt公司),其反应体系如表8所示。
[0078]
表8
[0079]
mix组分体积(μl)末端修复和加a的产物50接头4连接缓冲液33dna连接酶3合计90
[0080]
注:先加入接头,混匀后再加入体系中的其他组分,接头为适用于illumina测序平台,且各接头带有任意符合碱基平衡的index序列,均由idt公司提供。连接缓冲液、dna连接酶均由enzymatics公司提供。
[0081]
(2)反应条件:20℃,15min;65℃,10min;4℃,待机。
[0082]
注:在65℃结束前取出至冰盒上,立即进行下一步磁珠纯化回收。
[0083]
(3)上述连接产物用1x磁珠纯化回收,30μl db洗脱。
[0084]
1)连接产物加入1x磁珠。
[0085]
2)震荡混匀,离心。
[0086]
3)室温孵育10min,使磁珠与dna结合。
[0087]
4)置于磁力架5min,待溶液澄清。
[0088]
5)弃上清。
[0089]
6)加入200μl 80%乙醇。
[0090]
7)静置30s。
[0091]
8)弃上清。
[0092]
9)重复步骤6-8。
[0093]
10)离心,去除上清液。
[0094]
11)室温晾干,磁珠没有明显的发亮或干裂。
[0095]
12)30μl db洗脱。
[0096]
13)磁珠纯化后的产物采用bio-rad micro bio-spin 6 columns(ssc缓冲)(购自bio-rad)进行纯化回收,操作步骤严格按照产品操作说明书进行,回收完成后得到纯化的连接产物。
[0097]
5、氧化反应及产物纯化
[0098]
(1)取步骤4中得到的纯化连接产物按表9所示进行混合。
[0099]
表9
[0100][0101][0102]
(2)反应条件:在37℃,400rpm条件下变性30min。
[0103]
(3)反应完,立即取出,冰上孵育5min。
[0104]
(4)加入1.5μl k2ruo4溶液(即体系中k2ruo4终浓度为0.45mm),冰上孵育1h。
[0105]
(5)反应产物采用bio-rad micro bio-spin 6 columns(ssc缓冲)(购自bio-rad)进行纯化回收,操作步骤严格按照产品操作说明书进行,得到氧化后的dna文库。
[0106]
6、还原反应及产物纯化
[0107]
(1)向步骤5得到的k2ruo4氧化的dna文库加入配制好的还原反应体系(还原体系中2-甲基吡啶硼烷溶液的终浓度为2m),反应体系如表10所示。
[0108]
表10
[0109]
反应混合物体积(μl)
氧化的dna文库205m 2-甲基吡啶硼烷溶液203m乙酸钠10
[0110]
(2)反应条件:70℃,850rpm条件下反应3h。
[0111]
(3)反应物采用zymo oligo clean&concentrator纯化柱(购自zymo research)进行纯化回收。操作步骤严格按照产品操作说明书进行。
[0112]
7、pcr扩增
[0113]
(1)将步骤6中获得的还原的dna文库进行扩增,扩增引物为primer p5(序列:5
’‑
aatgatacggcgaccaccga-3’),primer p7(序列:5
’‑
caagcagaagacggcatacga-3’)配制pcr扩增体系如表11所示。
[0114]
表11
[0115][0116][0117]
(2)pcr反应程序如表12所示。
[0118]
表12
[0119][0120]
(3)上述pcr产物用1
×
磁珠纯化回收,30μl db洗脱。洗脱产物即为待测文库。
[0121]
对比例1
[0122]
本对比例提供了一种全基因组羟甲基化非重亚硫酸氢盐测序文库,构建方法与实施例2的区别仅在于:步骤5氧化反应及产物纯化的步骤不同,本对比例步骤5采用的氧化剂
为kruo4溶液,kruo4溶液在体系中,kruo4终浓度为0.45mm,naoh的终浓度为2.5mm,具体方案为:
[0123]
(1)步骤3中的连接产物按反应体系如表13所示。
[0124]
表13
[0125]
反应混合物体积连接产物dna100ng1m naoh2.5μl1
×
ssc(ph=7.0)补足至48.5μl合计48.5μl
[0126]
(2)反应条件:在37℃,400rpm条件下变性30min。
[0127]
(3)反应完,立即取出,冰上孵育5min。
[0128]
(4)加入1.5μl kruo4溶液,冰上孵育1h。
[0129]
(5)反应产物采用bio-rad micro bio-spin 6 columns(ssc缓冲)(购自bio-rad)进行纯化回收,操作步骤严格按照产品操作说明书进行。
[0130]
对比例2
[0131]
本对比例提供了一种全基因组羟甲基化非重亚硫酸氢盐测序文库,构建方法与实施例2的区别仅在于:步骤6还原反应及产物纯化的步骤不同,本对比例步骤6采用的还原剂吡啶硼烷溶液在体系中的终浓度为2mol/l,具体方案为:
[0132]
(1)将步骤4 k2ruo4氧化的dna文库加入配制好的还原反应体系,反应体系如表14所示。
[0133]
表14
[0134]
反应混合物体积(μl)氧化的dna文库3010m吡啶硼烷溶液103m乙酸钠10
[0135]
(2)反应条件:37℃条件下,在速度为850rpm的恒温混匀仪上反应16h。
[0136]
(3)反应物采用zymo oligo clean&concentrator纯化柱(购自zymo research)进行纯化回收。操作步骤严格按照产品操作说明书进行。
[0137]
试验例
[0138]
1、氧化剂和还原剂的筛选
[0139]
本试验例测试了实施例2和对比例1-2制备的全基因组羟甲基化非重亚硫酸氢盐测序文库的转化率。
[0140]
氧化剂的筛选测试,包括以下步骤:分别将实施例2和对比例1制备的全基因组羟甲基化非重亚硫酸氢盐测序文库进行上机测序,测序过程中不添加其他全基因组文库进行碱基平衡。
[0141]
还原剂的筛选测试,包括以下步骤:分别将实施例2和对比例2制备的全基因组羟甲基化非重亚硫酸氢盐测序文库进行上机测序,测序过程中不添加其他全基因组文库进行碱基平衡。
[0142]
本发明用到的测序平台是illumina的nextseq平台,测序步骤严格按照供应商的
要求使用。对下机数据进行分析,对外源性阳性内参的测序数据进行统计分析,分别统计待测文库中阳性内参转化前的reads数和转化后的reads数,进行羟甲基化转化率的计算,计算公式为:转化率=转化reads数/(未转化和转化总reads数)。
[0143]
阳性内参转化前(即未转化)序列为:
[0144]
ctgcccaccagccatcatgtcggaccccgcggtcaacgcgcagctggatgggatcatttcggacttcgaaggtgggtgctgga(seq id no.1)。
[0145]
阳性内参转化后序列为:
[0146]
ctgcccaccagccatcatgtcggaccctgcggtcaacgcgcagctggatgggatcatttcggacttcgaaggtgggtgctgga(seq id no.3)。
[0147]
实验结果:不同氧化试剂的转化率对比结果如表15所示,从表中可以看出k2ruo4的转化率明显优于kruo4,不同还原剂的转化率结果如表16所示,从表中可以看出,2-甲基吡啶硼烷的转化率明显优于吡啶硼烷。
[0148]
表15
[0149][0150]
表16
[0151][0152][0153]
2、不同氧化剂对dna损伤
[0154]
在氧化剂实际应用中除了需要考虑氧化剂的氧化性,还需要考虑试剂对底物的损伤程度。因此,需要根据具体的应用环境来选择合适的氧化剂。本发明从大量的氧化剂中筛选得到可以用于可特异性地识别并转换5hmc,并将5hmc氧化成5-甲酰基胞嘧啶(5fc)的2种
氧化剂,kruo4和k2ruo4;本测试例对比了2种不同的氧化剂对dna损伤情况。
[0155]
实验方案:分别采用氧化剂kruo4和k2ruo4氧化dna,氧化的dna为实施例2中的阳性标准品,在氧化反应体系中,两个不同氧化剂的终浓度均为0.45mm,氧化的dna投入量均为100ng,氧化剂kruo4的氧化方案同对比例1一致,氧化剂k2ruo4的氧化方案同实施例2一致,将两个方案中氧化后的dna进行电泳分析。
[0156]
结果如图3所示,从图中可以看出,在相同的条件下,氧化剂kruo4对dna的损伤更大,氧化后的dna条带模糊,且有明显的降解趋势。由此可见,氧化剂k2ruo4在氧化dna的同时,对dna的损伤更小,更有利于低起始量dna的文库构建。
[0157]
3、全基因组羟甲基化非重亚硫酸氢盐测序文库的转化率测试
[0158]
采用本发明筛选出来的氧化试剂k2ruo4和还原试剂2-甲基吡啶硼烷以及采用实施例2中的技术路径进行羟甲基化转化实验和建库,将文库进行上机测序,具体统计结果如下表。
[0159]
表17
[0160][0161][0162]
由测序结果和表17的转化率结果可知,通过本发明的技术路径在不转化5mc的基础上可实现5hmc的单碱基检测,且5hmc的转化率较高,可达82.12%。同时,测序过程中未添加其他全基因组文库进行碱基平衡,illumina的nextseq测序仪在测序过程中未出现qv值降低,q30曲线也未出现波动,q30仍可达90%以上,下机数据采用生物信息学软件分析时,未出现数据分离困难情况,文库数据量产出正常。
[0163]
上面结合附图对本发明实施例作了详细说明,但是本发明不限于上述实施例,在所属技术领域普通技术人员所具备的知识范围内,还可以在不脱离本发明宗旨的前提下作出各种变化。此外,在不冲突的情况下,本发明的实施例及实施例中的特征可以相互组合。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。