中尺度工程化肽和选择方法
1.相关申请的交叉引用
2.本技术要求2019年5月31日提交且标题为“中尺度工程化肽和选择方法”的美国专利申请第62/855,767号的优先权和权益,所述美国专利申请以全文引用的方式并入本文中。
背景技术:
3.治疗领域的许多基础研究都旨在识别和开发具有理想特性的新分子,例如新的肽治疗剂或新的肽免疫原,从中开发新的治疗性抗体。然而,标准的分子发现范式依赖于使用随机过程的随机取样来识别有前景的功能分子。然后,这些候选分子将通过多轮评估和测试,希望它们将具有特定用途所需的活性、功能、药物动力学和/或其它所需特征。此系统从筛选随机组开始,通常会导致失败,不满足一个或多个所需的特征。因此,需要的是开发并入计算、化学和生物设计元件的工程化肽的方法。
技术实现要素:
4.在一些方面,本文提供了一种工程化肽,其中所述工程化肽具有介于1kda与10kda之间的分子量,包含至多50个氨基酸,并且包含:空间相关拓扑约束的组合,其中一个或多个约束是参考目标导出的约束;并且其中工程化肽的10%至98%的氨基酸满足一个或多个参考目标导出的约束,其中满足一个或多个参考目标导出的约束的氨基酸与参考目标具有小于的主链均方根偏差(rsmd)结构同源性。
5.在一些实施例中,满足一个或多个参考目标导出的约束的氨基酸与参考目标具有10%至90%的序列同源性。在一些实施例中,它们与参考的范德华表面积重叠为至在某些实施例中,所述组合包含至少两个或至少五个参考目标导出的约束。在一些实施例中,约束的组合包含一个或多个并非从参考目标导出的约束。在一些实施例中,一个或多个非参考目标导出的约束描述了所需的结构、动力学、化学或功能特征,或其任何组合。在更进一步的实施例中,一个或多个约束独立地与生物反应或生物功能相关。在一些实施例中,与生物反应或生物功能相关的工程化肽中的至少一部分原子在拓扑上被约束于参考目标中的二级结构元件,例如β-片层或α螺旋。
6.在其它方面,本文提供了一种选择工程化肽的方法,其包含:
7.识别参考目标的一个或多个拓扑特征;
8.为每个拓扑特征设计空间相关约束,以产生从所述参考目标导出的空间相关拓扑约束的组合;
9.将候选肽的空间相关拓扑特征与从所述参考目标导出的空间相关拓扑约束的组合进行比较;以及
10.选择具有空间相关拓扑特征的候选肽,所述特征与从所述参考目标导出的所述空间相关拓扑约束的组合重叠,以产生所述工程化肽。
11.在一些实施例中,每个特征之间的重叠独立地小于或等于由以下中的一个或多个确定的75%平均百分比误差(mpe):总拓扑约束距离(tcd)、拓扑聚类系数(tcc)、欧几里得距离(euclidean distance)、功率距离、索格尔距离(soergel distance)、堪培拉距离(canberra distance)、索伦森距离(sorensen distance)、杰卡德距离(jaccard distance)、马氏距离(mahalanobis distance)、汉明距离(hamming distance)、相似性定量估计(qel)或链拓扑参数(ctp)。在某些实施例中,一个或多个约束是从以下各者导出:每个残基能量、每个残基相互作用、每个残基波动、每个残基原子距离、每个残基化学描述符、每个残基溶剂暴露、每个残基氨基酸序列相似性、每个残基生物信息学描述符、每个残基非共价键合倾向、每个残基角、每个残基范德华半径、每个残基二级结构倾向、每个残基氨基酸邻接或每个残基氨基酸接触。在一些实施例中,一种或多种候选肽的特征通过计算机模拟确定。在更进一步的实施例中,一个或多个约束独立地与生物反应或生物功能相关。在一些实施例中,与生物反应或生物功能相关的工程化肽中的至少一部分原子在拓扑上被约束于参考目标中的二级结构元件,例如β-片层或α螺旋。
12.在更进一步的方面,本文提供包含两种或更多种选择导向多肽的组合物,其中每一种多肽独立地为包含一种或多种正导向特征的正选择分子,或包含一种或多种负导向特征的负选择分子,其中每个特征类型独立地选自由以下组成的组:氨基酸序列、多肽二级结构、分子动力学、化学特征、生物功能、免疫原性、参考目标多特异性、跨物种参考目标反应性、所需参考目标超过非所需参考目标的选择性、序列和/或结构同源家族内参考目标的选择性、具有相似蛋白质功能的参考目标的选择性、从具有高序列和/或结构同源性的较大非所需目标家族选择不同所需参考目标的选择性、对不同参考目标等位基因或突变的选择性、对不同参考目标残基水平化学修饰的选择性、对细胞类型的选择性、对组织类型的选择性、对组织环境的选择性、对参考目标结构多样性的耐受性、对参考目标序列多样性的耐受性以及对参考目标动力学多样性的耐受性;并且其中两种或更多种多肽中的至少一种是如本文所述的工程化肽。
13.在一些实施例中,两种或更多种多肽中的至少一种是正选择分子,并且两种或更多种多肽中的至少一种是负选择分子。在一些实施例中,两种或更多种多肽中的至少一种是天然蛋白质。在某些实施例中,至少一对对应的正选择和负选择分子包含至少一种共有特征类型,其中正选择分子包含正特征并且负选择分子包含负特征。
14.在另外的方面,本文提供了用包含两个或更多个如本文所述的选择导向分子的组合物筛选结合分子文库的方法,所述方法包含使候选结合分子池经受至少一轮选择,其中每轮选择包含:
15.针对负选择分子筛选所述池的至少一部分的负选择步骤;和
16.针对正选择分子筛选所述池的至少一部分的正选择步骤;
17.其中每一轮内的选择步骤的顺序和轮次的顺序导致选择与替代顺序不同的池子集。
18.在一些实施例中,结合分子文库是噬菌体文库或细胞文库,例如b细胞文库或t细胞文库。在一些实施例中,所述方法包含两轮或更多轮选择,或三轮或更多轮选择。在某些实施例中,每一轮包含不同的选择分子集。在一些实施例中,至少两轮包含相同的负选择分子,或相同的正选择分子,或两者。在一些实施例中,所述方法包含在继续进行下一轮选择
之前分析从一轮选择中获得的池子集。
附图说明
19.专利或申请文件至少包含一张彩图。在请求并支付必要的费用后,将由专利局提供带有彩图的本专利或专利申请公开案的副本。本技术可参考以下描述与附图结合理解。
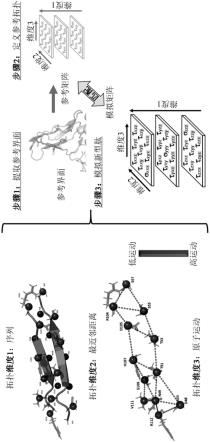
20.图1提供了展示三个空间相关拓扑约束的示例性组合的构建的示意图,用于选择如本文所述的工程化肽。
21.图2提供了确定参考导出的空间相关拓扑约束和其在选择工程化肽(中尺度分子,mem)中的使用的一些示例性方法中所涉及的步骤的示意图。
22.图3a-3c提供了展示使用本文所述的方法选择一组工程化肽的示意图。图3a显示了关于参考中感兴趣的界面的空间相关拓扑信息的提取,以及其在定义用于选择工程化肽的拓扑约束中的用途。图3b提供了详细说明计算机模拟筛选步骤的示意图,展示了如何丢弃不匹配的候选物,同时保留与拓扑匹配的候选物。图3c呈现了已鉴定的前12个选定的工程化肽候选物。
23.图4a-4b提供了第二组示意图,展示了使用本文所述的方法基于不同的参考参数集选择不同组的工程化肽。图4a显示了空间相关拓扑信息的提取和拓扑矩阵的构造。图4b提供了由计算机模拟比较候选物与拓扑约束选择的前8种工程化肽候选物的列表。
24.图5是提供示例性可编程体外选择的设计概述的示意图,所述选择使用如本文所述的工程化肽,并且还使用天然蛋白质作为正(t)或负(x)选择分子。
25.图6a-6h提供了五种工程化肽的选择的概述,以及它们在用于噬菌体淘选的可编程体外选择方案中的用途。图6a展示了选择vegf作为参考目标,以及确定从中导出空间相关拓扑信息并用于构建空间相关拓扑约束的组合的vegf部分(步骤1)。接着将此组合用于候选工程化肽的计算机模拟筛选以鉴定正选择分子和负选择分子(步骤2)。为了稳定交联选项,经由计算机进一步筛选所选的候选物。一旦获得鉴定的、稳定的工程化肽,它们就被用于构建用于噬菌体淘选的可编程体外选择方案。图6b显示了基于用于选择工程化肽的参考目标(vegf的一部分)的空间相关拓扑约束的分析和鉴定。图6c、图6d、图6e分别展示了第一、第二和第三候选工程化肽的构建,以及参数的导出以与图6b中开发的约束组合进行比较。图6f列出了每个mem与参考目标相比的平均百分比误差(mpe),以及它们基于mpe的排名。图6g显示了如何根据参考目标将额外的约束集添加到组合中。在图6h中,此额外约束集用于评估候选物mem 1。此比较的mpe为36.6%。
26.图7a是vegf的带状图,其中参考部分用于选择所示的工程化肽(r82-h90)。图7b是基于从图7a中的目标参考产生的约束选择的5种候选工程化肽的带状图。表1中列出了序列和均方根rmsip。图7d显示了描述参考目标中表位的两个最主要运动的两个特征向量,将表位中的十个c
ɑ
原子的x、y和z分量以及特征向量的特征值制成表;结构显示了表位中每个ca原子沿特征向量1(箭头)和特征向量2(箭头)的投影。根据定义,特征向量是正交的。图7e是描述参考目标(左)和mem(右)的表位中最主要运动(模式)的特征向量。叠加在表位上的mem结构与mem变体id和rmsip一起显示。图7f提供了描述参考目标(左)和mem(右)的表位中第二主要运动(模式)的特征向量。叠加在表位上的mem结构与mem变体id和rmsip一起显示。图7g提供了参考目标和mem的结构,以及沿着三个最主要运动(模式,特征向量1-3)的相关投
影,所述运动与它们在用于计算rmsip的内积矩阵中的位置有关。示出所用的rmsip方程式仅供参考。
27.图8显示了从实验数据或计算机模拟生成的参考目标(顶部)和mem(底部)的结构集合和坐标协方差矩阵。表位是参考目标右上方的较暗部分。
28.图9概述了体外可编程选择设计,使用四种工程化肽(也称为中尺度工程化分子,或mem)进行正或负选择。包括mem的原子运动和拓扑分数以供参考。序列作为seq id no:1-4提供。
29.图10a-10d是使用来自图9的针对贝伐单抗的不同工程化肽的结合生物传感器分析的图。
30.图11描述了八种不同的淘选程序,其中七种包括作为一个或多个选择分子的工程化肽,且第八种程序使用常规天然蛋白质进行选择。每个程序都分别淘选了一个初始hu scfv文库。
31.图12a和12b是vegf elisa反应图,比较了针对使用图11中描述的不同淘选程序选择的结合配偶体的vegf结合反应。如图12a中所示,mem程序化体外选择不会显著降低全长目标结合倾向,具有特定的mem程序输入,但不是所有输入。水平条表示平均值;p12与p7之间的显著差异:p值《0.0001。如图12b中所示,mem程序化体外选择以统计显著的方式指向推定的表位选择性克隆。水平条表示平均值,p12相对于p6:p值为0.024;p12相对于p9:p值为0.0004;p12相对于p10:p值为0.049。
32.图13a-13h是展示使用图11中描述的不同淘选程序选择的结合配偶体与smem工程化肽相对于vegf(参考)的结合的图。
33.图14a-14i是展示使用图11中描述的不同淘选程序选择的结合配偶体在vegf与贝伐单抗(0nm、67pm、670pm、6.7nm)的剂量反应性竞争的交叉阻断分析中的结合的图。
34.图15是从图11中概述的不同选择程序中的每一个获得的具有确认的交叉阻断特征的不同克隆的图。
35.图16是从图11中概述的选择程序产生的所有fab的结合、交叉阻断、cdr序列和种系使用的总结。
36.图17和图18是图17中列出的所有fab的elisa结合结果。
37.图19显示了随机克隆相对于选自图11中概述的选择程序的克隆的贝伐单抗阻断倾向分数(0nm、67pm、670pm、6.7nm)。elisa z分数(smem vegf-imem) 贝伐单抗阻断z分数。
38.图20总结了从图11中描述的淘选程序中随机均匀选择克隆的交叉阻断富集。
39.图21是显示如何制备所选克隆的下一代测序样本的示意图。使用2
×
250配对末端测序运行,克隆出表达载体恒定部分的单个重链和轻链序列。然后连接末端并注释读段(例如,使用pyig)。从使用每个选择程序选择的克隆获得的读段显示在条形图中。
40.图22展示了不同淘选轮次的克隆性分析(不同抗体的数量)和标准化香农分析(shannon analysis)。
41.图23显示了图11中描述的不同筛选程序的克隆性。
42.图24a-24l是种系使用热图和降维图,分析了第1轮(图24a-24d)、第2轮(图24e-24h)和第3轮(图24i-24l)的不同筛选轮次和程序如何塑造所得选定池的多样性。
43.图25a-25b总结了从每个选择程序(x轴中的s#)分离的克隆以及其与vegf和工程
化肽smem的结合。
44.图26是从每个程序的每一轮获得的独特mab命中的富集率的总结,所述命中被证实与vegf结合并交叉阻断贝伐珠单抗,并且在不使用工程化肽的常规淘选(程序12)中未被鉴定出来。
45.图27是通过常规淘选程序(12)获得的mab命中的富集率的总结,所述命中被证实与vegf结合但不是推定的表位选择性mab命中。
46.图28总结了从不同淘选程序获得的不同克隆与smem或vegf的结合。
47.图29是靶向pd-l1上提议的治疗表位参考位点的第二组示例性程序化体外选择方案的示意图。空间相关拓扑约束从这个推定位点导出,组合并用于经由计算机筛选与约束组合具有重叠特征的工程化肽。接着将这些用于在初始hu scfv文库的噬菌体淘选中进行数轮选择。
48.图30提供了根据图29中的示意图选择的三种工程化肽的模型化结构和肽序列。序列作为seq id no:5-7提供。
49.图31a-31d是从参考(图31a)以及工程化肽smem(图31b)、nmem(图31c)和imem(图31d)导出的原子距离和氨基酸描述符矩阵。与参考拓扑相比时,smem、nmem和imem拓扑的平均百分比误差分别为3.58%、0.84%和19.3%。
50.图31e-31g是生物传感器结合图,展示了图30中描述的工程化肽与阿维鲁单抗(avelumab)之间的结合。nmem与阿维鲁单抗结合的kd为43.4μm。
51.图32a-32c是生物传感器结合图,展示了图30中描述的工程化肽与度伐单抗(durvalumab)之间的结合。
52.图33是使用图30中描述的工程化肽中的一种或多种的程序化体外选择淘选程序以及使用天然蛋白质的传统淘选方法(c1)的差异的总结。图30中的工程化肽smem、nmem和imem是图33中的smem#1、smem#5和imem。
53.图34是使用图33中描述的每个淘选程序选择的克隆的pd-l1elisa结合反应的图和总结。
54.图35是使用图33中描述的每个淘选程序选择的克隆的针对smem#1的elisa结合反应的图和总结。
55.图36是使用图33中描述的每个淘选程序选择的克隆的针对nmem#5的elisa结合反应的图和总结。
56.图37是使用图33中描述的每个淘选程序选择的克隆的针对pd-l1和smem#1的elisa表位选择性反应的图和总结。
57.图38是使用图33中描述的每个淘选程序选择的克隆的针对pd-l1和nmem#5的elisa表位选择性反应的图和总结。
58.图39a-39u是比较图34-38的不同elisa结合反应的图,展示了使用不同程序选择的结合配偶体的选择性。
59.图40是总结用于分析从图33中描述的选择程序获得的克隆的抗pd-l1淘选elisa命中识别标准的表。
60.图41a-41c是对于使用图33中描述的不同淘选程序选择的结合配偶体比较对smem#1和nmem#5相比于pd-l1(分别为图41a和42b)以及smem#1相比于nmem#5(图41c)的不
同elisa结合反应的图。
61.图42a-42f是比较图33中描述的所有程序的不同elisa反应和确认的tx mab x-阻断剂的图。
62.图43总结了来自图33中描述的程序的23个不同的克隆,如从交叉阻断命中和它们的序列中确定。
63.图44是从图33中描述的每个程序获得的确认的交叉阻断不同克隆的图表。
64.图45a是从图33中描述的每个程序获得的随机选择的克隆的阻断倾向的图。阻断被评估为通过pd-l1与阿维鲁单抗或度伐单抗结合的克隆的阻断。阻断倾向被评估为elisa z分数(smem1 smem5 pd-l1-imem) max(阿维鲁单抗阻断z分数,度伐单抗阻断z分数)。
65.图45b和45c总结了从图45a中评估的不同程序获得的克隆的阻断倾向。图45c中的阴影条目是通过使用天然蛋白质的常规选择方法获得的。
66.图46是与对照(常规方法)相比,在使用图33中描述的程序获得的克隆池中观察到的交叉阻断富集的总结。
67.图47是可用于选择如本文所述的工程化肽的拓扑矩阵的实例。
68.图48是可用于选择如本文所述的工程化肽的拓扑约束化学描述符向量的实例。
69.图49是可用于选择如本文所述的工程化肽的示例性lx2矩阵。
70.图50是可用于选择如本文所述的工程化肽的二级结构相互作用描述符的示例性sxsxm矩阵。
71.图51是显示可用于选择如本文所述的工程化肽的示例性工程化肽的簇和tcc向量的示例性图。
72.图52是可用于选择如本文所述的工程化肽的示例性lxm拓扑约束矩阵。
73.图53是可用于选择如本文所述的工程化肽的示例性二级结构索引和查找表。
74.图54是从vegf淘选程序获得的数据的另一种表示。s1是指抗vegf淘选程序6,s2是指抗vegf淘选程序13,且c为常规全长vegf程序。
75.图55是图24i中提供的数据的另一种表示。s1是指抗vegf淘选程序6,s2是指抗vegf淘选程序13,且c为常规全长vegf程序。
76.图56是图26中提供的数据的另一种表示。s1是指抗vegf淘选程序6,s2是指抗vegf淘选程序13,且c为常规全长vegf程序。
77.图57a-57e是来自程序化体外选择的所选表位上克隆的vegf(灰色实线)和交叉阻断(虚线)结合数据的图。
78.图58a-58c是来自全长体外选择的表位外选择的克隆的vegf结合数据的图。
79.图59a-59b总结了针对抗vegf程序化体外选择(红色)和常规体外选择(灰色)的抗体克隆命中cdr环序列多样性。
80.图60是使用如本文所述的示例性工程化肽,使用本文所述的可编程体外选择方法选择的克隆的序列比对。顶行是在所有程序化的体外选择程序中选择的前五个表位上克隆的重链序列的比对;第二行是使用常规方法,使用vegf和bsa作为选择分子选择的前五个表位外克隆的重链序列的比对;第三行是在所有程序化体外选择程序中选择的前五个表位上克隆的轻链序列的比对;并且底行是使用常规方法与vegf和bsa选择的克隆的轻链序列的比对。
81.图61是工程化多肽设计的示例性方法的示意图。
82.图62是使用机器学习模型进行工程化多肽设计的示例性方法的示意图。
具体实施方式
83.本文提供了选择中尺度工程化肽的方法,以及包含所述工程化肽的组合物和使用所述工程化肽的方法。例如,供本文提了在体外抗体选择中使用工程化肽的方法。
84.本公开的工程化肽在1kda与10kda之间,在本文中称为“中尺度”。在一些实施例中,这种尺寸的工程化肽可能具有某些优势,例如蛋白质样功能性、从中选择候选物的大理论空间、细胞渗透性和/或结构和动力学可变性。
85.本文提供的方法包含识别多个空间相关的拓扑约束,其中一些可能从参考目标导出;构建所述约束的组合;将候选肽与所述组合进行比较;以及选择具有与所述组合重叠的约束的候选物。通过使用空间相关的拓扑约束,可以取决于预期用途、所需功能或另一所需特性将工程化肽的不同方面包括在所述组合中。此外,在一些实施例中,并非所有约束都必须从参考目标导出。通过此类方法,在一些实施例中,选择的工程化肽不是参考目标的简单变体(例如可以通过肽诱变或单个参考的渐进修饰获得),而是可以具有与参考肽不同的整体结构,同时仍保留所需的功能特征和/或关键子结构。
86.本文进一步提供了使用所述工程化肽的方法,其包括使用一种或多种工程化肽的可编程体外选择的方法。此类选择可用于例如抗体的识别。
87.这些方法和工程化肽在下文更详细地描述。
88.i.选择工程化肽的方法
89.在一些方面,本文提供选择工程化肽的方法,其包含:
90.识别参考目标的一个或多个拓扑特征;
91.为每个拓扑特征设计空间相关约束,以产生参考目标导出的约束的组合;
92.将候选肽的空间相关拓扑特征与从参考目标导出的组合进行比较;以及
93.选择具有空间相关拓扑特征的候选肽,所述特征与从参考目标导出的约束的组合重叠。
94.在一些实施例中,所述组合中包括一个或多个并非从参考目标导出的额外空间相关拓扑约束。
95.a.空间相关拓扑约束
96.本文所述的工程化肽是基于它们与空间相关拓扑约束的组合匹配的紧密程度来选择。这种组合也可以使用“张量”的数学概念来描述。在此类组合(或张量)中,每个约束在三维空间中独立描述(例如,空间相关),并且这些约束在三维空间中的组合提供例如不同所需特征以及它们相对于位置的所需水平(如果适用)的代表性“地图”。在一些实施例中,此地图不基于线性或以其它方式预先确定的氨基酸主链,因此可以允许结构中可以满足所需组合的灵活性,如所描述。例如,在一些实施例中,“地图”包括空间区域,其中规定的约束限制可以被两个相邻的氨基酸充分满足—在一些实施例中,这些氨基酸可以直接结合(例如,两个连续氨基酸)而在其它实施例中,氨基酸彼此不直接结合,而是可以通过肽的折叠在空间中聚集在一起(例如,不是连续氨基酸)。单独的约束本身也不一定基于结构,但可以包括例如化学描述符和/或功能描述符。在一些实施例中,约束包括结构描述符,例如所需
的二级结构或氨基酸残基。在某些实施例中,独立地选择每个约束。
97.例如,图1是展示空间相关拓扑约束的代表性组合的构造的示意图。图1中的三个约束是序列、最近邻距离和原子运动,其中最近邻距离和原子运动组合成一个图形。如图所示,一些约束的映射无关于主链的位置(例如,某些侧链的原子运动),因此与仅改变参考支架上的一个或多个位置相比,允许尝试多得多种类的结构配置。三种不同的约束和其空间描述被组合成一个矩阵(例如,张量),且接着可以将一系列候选肽与这种组合进行比较,以识别满足所需标准的新工程化肽。在一些实施例中,组合中还包括一个或多个额外的非参考导出的约束。可以将候选肽与定义的组合进行比较,例如,使用计算机模拟方法来评估每个候选肽对所需组合的约束,并评估候选肽的匹配程度。然后可以使用本领域技术人员已知的标准肽合成方法合成与规定组合具有期望重叠水平的所述候选物,并对其进行评估。
98.在一些实施例中,约束的组合包含至少3、至少4、至少5、至少6、至少7、至少8、至少9、至少10、至少11、至少12、3至12、3至10、3至8、3至6、或3、或4、或5、或6个独立选择的空间相关拓扑约束。一个或多个约束是从参考目标导出。在一些实施例中,每个约束是从参考目标导出。在其它实施例中,至少一个约束是从参考目标导出,而其余约束并非从参考目标导出。例如,在一些实施例中,1至9个约束、1至7个约束、1至5个约束或1至3个约束是从参考目标导出,且1至9个约束、1至7个约束、1至5个约束或1至3个约束并非从参考目标导出。
99.一旦构建了约束条件的组合,就将一系列候选肽与所述组合进行比较以识别满足所需标准的一个或多个新的工程化肽。在一些实施例中,将至少5、至少10、至少15、至少20、至少25、至少30、至少40、至少50、至少60、至少70、至少80、至少90、至少100、至少125、至少150、至少175、至少200或至少250个或更多个候选肽与所述组合进行比较以识别满足所需标准的一个或多个新的工程化肽。在一些实施例中,例如比较了超过250个候选肽、超过300个候选肽、超过400个候选肽、超过500个候选肽、超过600个候选肽或超过750个候选肽。在一些实施例中,拓扑特征模拟用于评估候选肽与约束组合相比的拓扑特征重叠(如果有的话)。在一些实施例中,还将一个或多个候选肽与参考目标进行比较,并且评估候选肽拓扑特征与参考目标拓扑特征的重叠(如果有的话)。在一些实施例中,从超过5、超过10、超过20、超过30、超过40、超过50、超过60、超过70、超过80、超过90或超过100个不同的肽和拓扑特征模拟的计算样本中识别工程化肽并选择工程化肽,其中在总取样群体中,与参考目标相比,所选择的工程化肽具有最高的拓扑特征重叠。
100.用于构建所需组合(例如,所需张量)的空间相关拓扑约束可以各自独立地从广泛的可能特征组中选择。这些可以包括例如描述结构、动力学、化学或功能特征或其任何组合的约束。
101.结构约束可以包括例如原子距离、氨基酸序列相似性、溶剂暴露、角、ψ角、二级结构或氨基酸接触,或其任何组合。
102.动态约束可以包括例如原子波动、原子能、范德华半径、氨基酸邻接或非共价键合倾向。原子能可以包括例如两个原子之间的成对吸引能、两个原子之间的成对排斥能、原子级溶剂化能、两个原子之间的成对带电吸引能、两个原子之间的成对氢键合吸引能或非共价键合能,或其任何组合。
103.化学特征可以包括例如化学描述符。此类化学描述符可包括例如疏水性、极性、原子体积、原子半径、净电荷、logp、hplc保留、范德华半径、电荷模式或h键合模式或其任何组
合。
104.功能特征可以包括例如生物信息学描述符、生物反应或生物功能。生物信息学描述符可以包括例如blosum相似性、pka、zscale、克鲁恰尼特性(cruciani properties)、基德拉因子(kidera factors)、vhse量表、protfp、ms-whim分数、t量表、st量表、跨膜倾向、蛋白质埋藏面积、螺旋倾向、片层倾向、卷曲倾向、转向倾向、免疫原性倾向、抗体表位出现率和/或蛋白质界面出现率,或其任何组合。
105.在一些实施例中,设计约束结合关于以下各者的信息:每个残基能量、每个残基相互作用、每个残基波动、每个残基原子距离、每个残基化学描述符、每个残基溶剂暴露、每个残基氨基酸序列相似性、每个残基生物信息学描述符、每个残基非共价键合倾向、每个残基角、每个残基范德华半径、每个残基二级结构倾向、每个残基氨基酸邻接或每个残基氨基酸接触。在一些实施例中,这些特征用于参考目标中总残基的子集,或约束的总组合的总残基的子集,或其组合。在一些实施例中,一个或多个不同特征用于一个或多个不同残基。即,在一些实施例中,一个或多个特征用于残基子集,并且至少一个不同特征用于不同的残基子集。在一些实施例中,用于设计一个或多个约束的一个或多个所述特征由计算机模拟确定。合适的计算机模拟方法可以包括例如分子动力学模拟、蒙特卡洛模拟(monte carlo simulation)、粗粒度模拟、高斯网络模型(gaussian network model)、机器学习或其任何组合。
106.在一些实施例中,从一个类别中选择多个约束。例如,在一些实施例中,所述组合包含两个或更多个独立地为一种类型的生物反应的约束。在一些实施例中,两个或更多个约束独立地为一种类型的二级结构。在某些实施例中,两个或更多个约束独立地为一种类型的化学描述符。在其它实施例中,所述组合不包含重叠的约束类别。
107.在一些实施例中,一个或多个约束独立地与生物反应或生物功能相关。在一些实施例中,所述约束是空间定义的原子级约束,或空间定义的形状/面积/体积级约束(例如可由若干不同原子组成满足的特征形状/面积/体积),或空间定义的动态级约束(例如可由若干不同的原子组成满足的特征动态或动态集)。
108.在一些实施例中,一种或多种约束是从与生物功能或生物反应相关的蛋白质结构或肽结构导出。例如,在一些实施例中,一种或多种约束是从细胞外域,例如g蛋白偶联受体(gpcr)细胞外域或离子通道细胞外域导出。在一些实施例中,一种或多种约束是从蛋白质-蛋白质界面连接导出。在一些实施例中,一种或多种约束是从蛋白质-肽界面连接,例如mhc-肽或gpcr-肽界面导出。在某些实施例中,被约束于此类蛋白质或肽结构的原子或氨基酸是与生物功能或生物反应相关的原子或氨基酸。在一些实施例中,被约束于此类蛋白质或肽结构的工程化肽中的原子或氨基酸是从参考目标导出的原子或氨基酸。在一些实施例中,一个或多个约束是从参考目标的多态性区域(例如,个体之间经受等位基因变异的区域)导出。
109.在一些实施例中,生物反应或生物功能选自由以下组成的组:基因表达、代谢活性、蛋白质表达、细胞增殖、细胞死亡、细胞因子分泌、激酶活性、表观遗传修饰、细胞杀伤活性、炎症信号、趋化性、组织浸润、免疫细胞谱系定型、组织微环境修饰、免疫突触形成、il-2分泌、il-10分泌、生长因子分泌、γ干扰素分泌、转化生长因子β分泌、基于免疫受体酪氨酸的激活基序活性、基于免疫受体酪氨酸的抑制基序活性、抗体定向的细胞毒性、补体定向的
细胞毒性、生物途径激动作用、生物途径拮抗作用、生物途径重定向、激酶级联修饰、蛋白水解途径修饰、蛋白稳态途径修饰、蛋白质折叠/途径、翻译后修饰途径、代谢途径、基因转录/翻译、mrna降解途径、基因甲基化/乙酰化途径、组蛋白修饰途径、表观遗传途径、免疫定向清除、调理作用、激素信号传导、整合素途径、膜蛋白信号转导、离子通道通量和g蛋白偶联受体反应。
110.在一些实施例中,与生物功能或生物反应相关的一个或多个原子选自由以下组成的组:碳、氧、氮、氢、硫、磷、钠、钾、锌、锰、镁、铜、铁、钼和镍。在某些实施例中,原子选自由以下组成的组:氧、氮、硫和氢。
111.在一些实施例中,其中约束之一是与生物功能或生物反应相关的一种或多种氨基酸,和/或工程化肽包含与生物功能或生物反应相关的一种或多种氨基酸,所述一种或多种氨基酸独立地选自由以下组成的组:20种蛋白型天然存在的氨基酸、非蛋白型天然存在的氨基酸和非天然氨基酸。在一些实施例中,非天然氨基酸是化学合成的。在某些实施例中,一种或多种氨基酸选自20种蛋白型天然存在的氨基酸。在其它实施例中,一种或多种氨基酸选自非蛋白型天然存在的氨基酸。在更进一步的实施例中,一种或多种氨基酸选自非天然氨基酸。在更进一步的实施例中,一种或多种氨基酸选自20种蛋白型天然存在的氨基酸、非蛋白型天然存在的氨基酸和非天然氨基酸的组合。
112.虽然用于选择如本文所述的工程化肽的约束的组合包含从参考目标导出的至少一个约束,但在一些实施例中,所述组合的一个或多个约束并非从参考目标导出。因此,在某些实施例中,所选的工程化肽包含一种或多种不与参考目标共有的特征。
113.在一些实施例中,从参考目标导出并在组合中使用的一个或多个约束描述了在参考目标中观察到的特征的逆特征。因此,例如,参考目标可具有一定的正电荷模式,从所述参考目标导出与电荷相关的约束,并且导出的约束描述类似的模式,但具有中性电荷或负电荷。因此,在一些实施例中,一个或多个逆约束是从参考目标导出并且包括在组合中。例如,此类逆约束可例如适用于选择工程化肽作为某些分析或淘选方法的对照分子,或作为本文所述的可编程体外选择方法中的负选择分子。
114.在一些实施例中,空间定义的拓扑约束的组合包含一个或多个非参考导出的拓扑约束。在一些实施例中,一个或多个非参考导出的拓扑约束加强或稳定一个或多个二级结构元件、加强原子波动、改变肽总疏水性、改变肽溶解度、改变肽总电荷、使得能够在经标记或无标记分析中进行检测、使得能够在体外分析中进行检测、使得能够在体内分析中进行检测、使得能够从复杂混合物中捕获、使得能够进行酶处理、使得细胞膜能够具透性、使得能够与二级目标结合或改变免疫原性。在某些实施例中,一个或多个非参考导出的拓扑约束对从参考目标导出的约束(或随后选择的肽)的组合中的一个或多个原子或氨基酸进行约束。例如,在一些实施例中,约束的组合包括从参考目标导出的二级结构,并且约束的组合还包含稳定二级结构元件的约束(例如,通过额外的氢键合或疏水相互作用,或侧链堆叠,或盐桥,或二硫键),其中稳定约束不存在于参考目标中。在另一实例中,在一些实施例中,约束(或随后选择的肽)的组合包含一个或多个从参考目标导出的原子或氨基酸,并且约束的组合还包括在从目标参考导出的原子或氨基酸的至少一部分中加强原子波动的约束,其中约束不存在于目标参考中。在一些实施例中,一个或多个非参考导出的约束是逆约束。例如,在一些实施例中,构建约束的两种组合以选择具有逆特征的工程化肽。在一些此
类实施例中,约束的第一组合将包含从参考目标导出的一个或多个约束,以及并非从参考目标导出的一个或多个约束;并且约束的第二组合将包含从参考目标导出的相同的一个或多个约束,以及第一组合的一个或多个非参考目标约束的逆约束。
115.d.参考目标
116.可以使用任何合适的参考目标来导出一个或多个空间相关的拓扑约束以用于本文提供的方法中。在一些实施例中,参考目标是全长天然蛋白质。在其它实施例中,参考目标是全长天然蛋白质的一部分。在更进一步的实施例中,参考目标是非天然蛋白质或其部分。
117.例如,在一些实施例中,参考目标是细胞表面受体、或跨膜蛋白、或信号传导蛋白、或多蛋白复合物、或蛋白质-肽复合物、或其部分。在一些实施例中,参考目标是感兴趣蛋白质的一部分,其中感兴趣蛋白质参与生物体,例如人类的疾病过程。在一些实施例中,感兴趣蛋白质参与癌症的生长或转移,或参与发炎性病症,并且参考目标是所述感兴趣蛋白质的作为推定表位的部分。因此,在一些实施例中,本文提供的方法可用于选择一种或多种可用作免疫原的工程化肽,并可用于产生感兴趣蛋白质的抗体。可能感兴趣的蛋白质的实例包括例如pd-1、pd-l1、cd25、il2、mif、cxcr4或vegf。因此,在一些实施例中,参考目标是pd-1、pd-l1、cd25、il2、mif、cxcr4或vegf,或其一部分,例如表位。在一些实施例中,本文提供的方法可用于选择一种或多种作为免疫原的工程化肽,并且可用于产生一种或多种与导出目标参考的蛋白质特异性结合的抗体。在更进一步的实施例中,本文提供的方法可用于选择一种或多种工程化肽,其进而可用于选择感兴趣蛋白质的一种或多种结合配偶体,例如抗体或fab展示噬菌体。
118.c.约束的比较
119.在一些实施例中,一个或多个约束(例如,参考导出的或非参考导出的)通过分子模拟(例如分子动力学)或实验室测量(例如nmr)或其组合来确定。一旦导出并组合了约束,在一些实施例中,使用计算蛋白质设计(例如,rosetta)生成工程化肽候选物。在一些实施例中,使用其它取样肽空间的方法。然后可以对候选工程化肽进行动力学模拟以获得已选择的约束参数。为参考目标生成原子波动的协方差矩阵,为每个候选工程化肽中的残基生成协方差矩阵,并比较这些协方差矩阵以确定重叠。执行主分量分析以计算每个协方差矩阵的特征向量和特征值-参考目标的一个协方差矩阵和每个候选工程化肽的一个协方差-并且保留那些具有最大特征值的特征向量。
120.特征向量描述了在一组模拟分子结构中观察到的最主要、第二主要、第三主要、第n主要的运动。不希望受任何理论束缚,如果候选工程化肽像参考目标一样移动,则其特征向量将类似于参考目标的特征向量。特征向量的相似性对应于它们的分量(以每个ca原子为中心的3d向量)对齐,指向相同的方向。参考目标与候选工程化肽之间的示例性特征向量比较展示于图7d-7g中。
121.在一些实施例中,候选工程化肽与参考目标特征向量之间的这种相似性是使用两个特征向量的内积计算的。如果两个特征向量相互成90度,则内积值为0,或者如果两个特征向量正好指向同一方向,则内积值为1。不希望受理论束缚,因为特征向量的排序是基于它们的特征值,并且由于分子动力学(md)模拟对两个不同分子的潜在能量景观进行取样的随机性,特征值在那些不同分子之间可能不一定相同,在一些实施例中,需要多个不同等级
的特征向量之间的内积(例如工程化肽的特征向量1乘参考目标的特征向量2、3、4等)。此外,分子运动很复杂,并且可能涉及不止一种(或不止几种)主要/主运动模式。因此,在一些实施例中,计算候选工程化肽中的所有特征向量对与参考目标之间的内积。这会产生一个内积矩阵,其维数由所分析的特征向量的数量决定。例如,对于10个特征向量,内积矩阵为10
×
10。通过计算100个(如果10
×
10)内积的均方根值,可以将此内积矩阵提炼为单个值。这是均方根内积(rmsip)。rmsip的方程式展示于图7g中。从这个比较中,选择与定义的约束组合具有相似性的一种或多种候选工程化肽。
122.e.额外步骤
123.在一些实施例中,一种或多种工程化肽的选择包含一个或多个额外步骤。例如,在一些实施例中,基于与空间相关拓扑约束的定义组合的相似性来选择工程化肽候选物,如本文所述,且接着进行一个或多个分析以确定一种或多种额外特征,以及一个或多个结构调整以赋予或加强所述所需特征。例如,在一些实施例中,例如通过分子动力学模拟分析所选候选物以确定分子的整体稳定性和/或特定折叠结构的倾向。在一些实施例中,对工程化肽进行一种或多种修饰以赋予或加强所需的稳定性水平或所需折叠结构的所需倾向。此类修饰可包括例如安装一个或多个交联(例如二硫键)、盐桥、氢键合相互作用或疏水相互作用,或其任何组合。
124.本文提供的方法可进一步包含针对一种或多种所需特征,例如所需的结合相互作用或活性来分析一种或多种所选工程化肽。可以视需要使用任何合适的分析来测量所需特征。
125.ii.所选工程化肽
126.在其它方面,本文提供工程化肽,例如通过本文所述的方法选择的工程化肽。在一些实施例中,工程化肽的分子量为1kda至10kda,并且包含至多50个氨基酸。在某些实施例中,工程化肽的分子量为2kda至10kda、2kda至10kda、3kda至10kda、4kda至10kda、5kda至10kda、6kda至10kda、7kda至10kda、8kda至10kda、9kda至10kda、1kda至9kda、1kda至8kda、1kda至7kda、1kda至6kda、1kda至5kda、1kda至4kda、1kda至3kda或1kda至2kda。在某些实施例中,工程化肽包含至多45个氨基酸、至多40个氨基酸、至多35个氨基酸、至多30个氨基酸、至多25个氨基酸、至多20个氨基酸、至少5个氨基酸、至少10个氨基酸、至少15个氨基酸、至少20个氨基酸、至少25个氨基酸、至少30个氨基酸、至少35个氨基酸或至少40个氨基酸。
127.在某些实施例中,工程化肽包含空间相关拓扑约束的组合,其中一个或多个约束是参考目标导出的约束。在一些实施例中,本文所述的任何约束都可以组合使用。在更进一步的实施例中,工程化肽的10%至98%的氨基酸满足一个或多个参考目标导出的约束(例如,如果工程化肽包含50个氨基酸,则5至49个氨基酸满足一个或多个参考目标导出的约束)。在一些实施例中,工程化肽的20%至98%、30%至98%、40%至98%、50%至98%、60%至98%、70%至98%、80%至98%、90%至98%、10%至90%、10%至80%、10%至70%、10%至60%、10%至50%、10%至40%、10%至30%或10%至20%的氨基酸满足一个或多个参考目标导出的约束。在更进一步的实施例中,满足一个或多个参考目标导出的约束的一种或多种氨基酸与参考目标的主链均方根偏差(rsmd)结构同源性为小于小于小于小于小于小于或小于在一些实施例中,工程化
肽的分子量为1kda至10kda;包含至多50个氨基酸;空间相关拓扑约束的组合,其中一个或多个所述约束是参考目标导出的约束;工程化肽的10%至98%的氨基酸满足一个或多个参考目标导出的约束;并且满足一个或多个参考目标导出的约束的氨基酸与参考目标的主链均方根偏差(rsmd)结构同源性为小于
128.在一些实施例中,满足一个或多个参考目标导出的约束的工程化肽的氨基酸与参考目标具有10%至90%序列同源性、20%至90%序列同源性、30%至90%序列同源性、40%至90%序列同源性、50%至90%序列同源性、60%至90%序列同源性、70%在90%序列同源性或80%至90%序列同源性。在一些实施例中,满足一个或多个参考目标导出的约束的氨基酸与参考的范德华表面积重叠为至或或至或至或至或至或至或至或至或至或至或至或至或至
129.工程化肽满足的约束的组合可包含两个或更多个、三个或更多个、四个或更多个、五个或更多个、六个或更多个、或七个或更多个参考目标导出的约束。如本公开的其它地方所述,所述组合可包含一个或多个并非从参考目标导出的约束。这些参考导出的约束和非参考导出的约束(如果存在)可以独立地是本文所述的任何约束,例如本文所述的任何结构、动力学、化学或功能特性,或其任何组合。
130.在一些实施例中,当与参考目标相比时,工程化肽包含至少一个结构差异。此类结构差异可包括例如序列差异、氨基酸残基数、原子总数、总亲水性、总疏水性、总正电荷、总负电荷、一个或多个二级结构、形状因子、泽尼克描述符(zernike descriptor)、范德华表面、结构图节点和边、体积表面、静电势表面、疏水势表面、局部直径、局部表面特征、骨架模型、电荷密度、亲水密度、表面积与体积比、两亲密度或表面粗糙度,或其任何组合。在一些实施例中,当与参考目标中适用于特征类型的特征相比时,一种或多种特征(例如本文所述的一种或多种特征)的差异为至少10%、至少20%、至少30%、至少40%、至少50%、至少60%、至少70%、至少80%、至少90%、至少100%或大于100%。例如,在一些实施例中,差异是原子总数,并且工程化肽具有比参考目标多至少10%、至少20%或至少30%的原子,或比参考目标少至少10%、至少20%或至少30%的原子。在一些实施例中,差异在于总正电荷,并且工程化肽的总正电荷比参考目标大至少10%、至少20%、至少30%、至少40%或至少50%(例如,更正),而在其它实施例中,工程化肽的总正电荷比参考目标小至少10%、至少20%、至少30%、至少40%或至少50%(例如,更负)。
131.在一些实施例中,空间定义的拓扑约束的组合包括参考目标中不存在的一个或多个二级结构元件。因此,在一些实施例中,工程化肽包含参考目标中不存在的一个或多个二级结构元件。在一些实施例中,组合和/或工程化肽包含在参考目标中未发现的一个二级结构元件、两个二级结构元件、三个二级结构元件、四个二级结构元件或多于四个二级结构元件。在一些实施例中,每个二级结构元件独立地选自由螺旋、片层、环、转角和卷曲组成的组。在一些实施例中,参考目标中不存在的每个二级结构元件独立地为α-螺旋、β-桥、β-股、3
10
螺旋、π-螺旋、转角、环或卷曲。
132.在一些实施例中,工程化肽包含与生物反应或生物功能相关的一个或多个原子、或一个或多个氨基酸、或其组合。在一些实施例中,生物反应或生物功能选自由以下组成的组:基因表达、代谢活性、蛋白质表达、细胞增殖、细胞死亡、细胞因子分泌、激酶活性、表观遗传修饰、细胞杀伤活性、炎症信号、趋化性、组织浸润、免疫细胞谱系定型、组织微环境修饰、免疫突触形成、il-2分泌、il-10分泌、生长因子分泌、γ干扰素分泌、转化生长因子β分泌、基于免疫受体酪氨酸的激活基序活性、基于免疫受体酪氨酸的抑制基序活性、抗体定向的细胞毒性、补体定向的细胞毒性、生物途径激动作用、生物途径拮抗作用、生物途径重定向、激酶级联修饰、蛋白水解途径修饰、蛋白稳态途径修饰、蛋白质折叠/途径、翻译后修饰途径、代谢途径、基因转录/翻译、mrna降解途径、基因甲基化/乙酰化途径、组蛋白修饰途径、表观遗传途径、免疫定向清除、调理作用、激素信号传导、整合素途径、膜蛋白信号转导、离子通道通量和g蛋白偶联受体反应。
133.在某些实施例中,参考目标包含一个或多个与生物反应或生物功能(例如本文所述的一者)相关的原子;工程化肽包含一个或多个与生物反应或生物功能(例如本文所述的一者)相关的原子;并且工程化肽中的所述原子的原子波动与参考目标中的所述原子的原子波动重叠。因此,例如,在一些实施例中,原子本身是不同的原子,但它们的原子波动重叠。在其它实施例中,原子是相同的原子,并且它们的原子波动重叠。在更进一步的实施例中,原子独立地相同或不同。在一些实施例中,所述重叠为大于0.25的均方根内积(rmsip)。在一些实施例中,重叠是rmsip大于0.3、大于0.35、大于0.4、大于0.45、大于0.5、大于0.55、大于0.6、大于0.65、大于0.7、大于0.75、大于0.8、大于0.85、大于0.9或大于0.95。在某些实施例中,rmsip如下计算:
134.其中n是工程化肽拓扑约束的特征向量,且v是参考目标拓扑约束的特征向量。
135.在一些实施例中,工程化肽包含与生物反应或生物功能相关的原子或氨基酸(或其组合),并且所述原子或氨基酸或组合的至少一部分是从参考目标导出,并且工程化肽中的原子或氨基酸集和参考目标中的原子或氨基酸集的某些约束可以通过矩阵来描述。在一些实施例中,矩阵是lxl矩阵。在其它实施例中,矩阵是sxsxm矩阵。在更进一步的实施例中,矩阵是lx2角矩阵。
136.例如,在一些实施例中,与生物反应或生物功能相关的工程化肽中的原子或氨基酸的原子波动由lxl矩阵描述;所述原子或氨基酸的一部分是从参考目标导出;并且所述部分的参考目标中的原子波动由lxl矩阵描述。在一些实施例中,每个集的邻接(与氨基酸位置相关)由相应的lxl矩阵描述。在某些实施例中,对从参考目标导出的工程化肽的部分,工程化肽lxl原子波动或邻接矩阵的所有矩阵元件(i,j)的平均百分比误差(mpe)相对于参考目标原子波动或邻接矩阵的相应(i,j)元件为小于或等于75%。在一些实施例中,对于从参考目标导出的工程化肽的部分,mpe相对于参考目标矩阵中的相应元件为小于70%、小于65%、小于60%、小于55%、小于50%、小于45%或小于40%。在一些实施例中,其中矩阵表示原子波动,l是氨基酸位置数,并且如果(i,j)原子距离小于或等于则原子波动矩阵
元件中的(i,j)值分别是第i个和第j个氨基酸的分子内原子波动的总和,或者如果(i,j)原子距离大于或者如果(i,j)在对角线上,则为零。或者,在一些实施例中,原子距离可用作原子波动矩阵元件(i,j)的加权因子而不是0或1乘数。在某些实施例中,第i个和第j个原子波动和距离可以通过分子模拟(例如分子动力学)和/或实验室测量(例如nmr)来确定。在其它实施例中,其中矩阵表示邻接,l是氨基酸位置数,并且如果原子距离小于或等于则邻接矩阵元件(i,j)中的值分别是第i个与第j个氨基酸之间的分子内原子距离,或者如果原子距离大于或者如果(i,j)在对角线上,则为零。或者,在一些实施例中,原子距离可用作邻接矩阵元件(i,j)的加权因子而不是0或1乘数。在某些实施例中,第i个和第j个原子距离可以通过分子模拟(例如分子动力学)和/或实验室测量(例如nmr)来确定。
137.在某些实施例中,对于从参考目标导出的工程化肽的部分,与工程化肽中的反应或功能相关的原子或氨基酸具有拓扑约束化学描述符向量和相对于由相同化学描述符描述的参考小于75%的平均百分比误差(mpe),其中化学描述符向量中的每个第i元件对应于氨基酸位置索引。在一些实施例中,对于从参考目标导出的工程化肽的部分,mpe相对于由相同化学描述符描述的参考为小于70%、小于65%、小于60%、小于55%、小于50%、小于45%或小于40%。示例性向量呈现在图48中。
138.在更进一步的实施例中,矩阵是lx2角矩阵,并且与工程化肽中的反应或功能相关的原子或氨基酸具有相对于从参考目标导出的工程化肽的部分中的参考角矩阵小于75%的mpe,其中l是氨基酸位置数,且ψ值分别在维数(l,1)和(l,2)中。在一些实施例中,相对于从参考目标导出的工程化肽的部分中的参考角矩阵,mpe为小于70%、小于65%、小于60%、小于55%、小于50%、小于45%或小于40%。在一些实施例中,值通过分子模拟(例如分子动力学)、基于知识的结构预测或实验室测量(例如nmr)来确定。示例性lx2矩阵显示在图49中。
139.在一些实施例中,矩阵是sxsxm二级结构元件相互作用矩阵,并且相对于从参考目标导出的工程化肽的部分中的参考二级结构元件关系矩阵,与工程化肽中的反应或功能相关的原子或氨基酸具有小于75%的平均百分比误差(mpe),其中s是二级结构元件的数量,且m是相互作用描述符的数量。在一些实施例中,相对于从参考目标导出的工程化肽的部分中的参考二级结构元件关系矩阵,mpe为小于70%、小于65%、小于60%、小于55%、小于50%、小于45%或小于40%。相互作用描述符可以包括例如氢键合、疏水堆积、范德华相互作用、离子相互作用、共价桥、手性、取向或距离,或其任何组合。在二级结构元件相互作用矩阵索引中,(i,j,m)=第i个与第j个二级结构元件之间的第m个相互作用描述符值。示例性sxsxm矩阵呈现于图50中。
140.如本文所述的不同矩阵的平均百分比误差(mpe)可如下计算:
[0141][0142]
其中n是工程化肽(engn)和相应参考(refn)的拓扑约束向量或矩阵位置索引,总计为向量或矩阵位置n。拓扑矩阵的示例性实例提供于图47中。
[0143]
在一些实施例中,相比于参考目标,工程化肽具有小于75%的mpe。在某些实施例中,相比于参考目标,工程化肽具有小于70%、小于65%、小于60%、小于55%、小于50%、小于45%或小于40%的mpe。在一些实施例中,mpe由以下各者确定:总拓扑约束距离(tcd)、拓扑聚类系数(tcc)、欧几里得距离、功率距离、索格尔距离、堪培拉距离、索伦森距离、杰卡德距离、马氏距离、汉明距离、相似性定量估计(qel)或链拓扑参数(ctp)。
[0144]
a.二级结构元件
[0145]
在一些实施例中,工程化肽的至少一部分在拓扑上被约束于一个或多个二级结构元件。在一些实施例中,与工程化肽中的生物反应或生物功能相关的原子或氨基酸在拓扑上被约束于一个或多个二级结构元件。在一些实施例中,二级结构元件独立地为片层、螺旋、转角、环或卷曲。在一些实施例中,二级结构元件独立地为α螺旋、β-桥、β-股、3
10
螺旋、π-螺旋、转角、环或卷曲。在某些实施例中,至少一部分工程化肽在拓扑上受约束的一种或多种二级结构元件存在于参考目标中。在一些实施例中,工程化肽的至少一部分在拓扑上受约束于二级结构元件的组合,其中每个元件独立地选自由片层、螺旋、转角、环和卷曲组成的组。在更进一步的实施例中,每个元件独立地选自由α-螺旋、β-桥、β-股、3
10
螺旋、π-螺旋、转角、环和卷曲组成的组。
[0146]
在一些实施例中,二级结构元件是平行或反平行的片层。在一些实施例中,片层二级结构包含大于或等于2个残基。在一些实施例中,片层二级结构包含小于或等于50个残基。在更进一步的实施例中,片层二级结构包含2至50个残基。片层可以是平行或反平行的。在一些实施例中,平行片层二级结构可被描述为具有平行的两个股i、j(i和j股的n末端相反取向),以及残基i:j的氢键合模式。在一些实施例中,反平行片层二级结构也可被描述为具有反平行的两个股i、j(i和j股的n末端相同取向),以及残基i:j-1、i:j 1的氢键合模式。在某些实施例中,可以通过基于知识或分子动力学模拟和/或实验室测量来确定股的取向和氢键合。
[0147]
在一些实施例中,二级结构元件为螺旋。螺旋可以是右旋或左旋的。在一些实施例中,螺旋具有介于2.5与6.0之间的残基每转角(残基/转角)值和介于与之间的间距。在一些实施例中,残基/转角和间距由基于知识或分子动力学模拟和/或实验室测量确定。
[0148]
在一些实施例中,二级结构元件为转角。在一些实施例中,转角包含2至7个残基和1个或更多个残基间氢键。在一些实施例中,转角包含2、3或4个残基间氢键。在某些实施例中,转弯由基于知识或分子动力学模拟和/或实验室测量确定。
[0149]
在更进一步的实施例中,二级结构元件为卷曲。在某些实施例中,卷曲包含2至20个残基和零个预测的残基间氢键。在一些实施例中,这些卷曲参数由基于知识或分子动力学模拟和/或实验室测量确定。
[0150]
在更进一步的实施例中,工程化肽包含一个或多个从参考目标导出的原子或氨基酸,其中所述原子或氨基酸具有二级结构。在一些实施例中,这些原子或氨基酸与生物反应或生物功能相关。在一些实施例中,对于从参考目标导出的工程化肽的部分,工程化肽中的原子或氨基酸的二级结构基序向量相对于参考目标二级结构基序向量具有大于0.25的余弦相似性,其中向量的长度是二级结构基序的数量,且第i个向量位置的值定义了从查找表中导出的二级结构基序(例如螺旋、片层)的身份。在一些实施例中,每个基序包含两个或更
多个氨基酸。在某些实施例中,基序包括例如α螺旋、β-桥、β-股、3
10
螺旋、π-螺旋、转角和环。在一些实施例中,对于从参考目标导出的工程化肽的部分,相对于参考目标二级结构基序向量的余弦相似性为大于0.3、大于0.35、大于0.4、大于0.45或大于0.5。图53中提供了示例性二级结构索引和查找表。余弦相似性可如下计算:
[0151][0152]
其中a为二级结构基序标识符的肽向量,b为二级结构基序标识符的参考向量,n为二级结构基序向量的长度,且i为第i个二级结构基序。
[0153]
在一些实施例中,可以使用总拓扑约束距离(tcd)将从参考目标导出的工程化肽的一个或多个原子或氨基酸与相应参考目标原子或氨基酸进行比较。在一些实施例中,所述从参考目标导出的工程化肽原子或氨基酸的总tcd相对于参考目标中的相应原子的tcd距离为 /-75%,其中如果两个分子内拓扑约束的成对距离小于或等于则它们相互作用。在一些实施例中,被比较的工程化肽中的原子或氨基酸与生物功能或生物反应相关。在一些实施例中,两个原子或氨基酸的第i、第j成对距离可以通过分子模拟(例如分子动力学)和/或实验室测量(例如nmr)来确定。用于计算总拓扑约束距离(tcd)的示例性方程式为:
[0154][0155]
其中i、j是氨基酸(i,j)的分子内位置索引,s
ij
是约束s(i)与s(j)之间的差异,如果氨基酸(i,j)在相互作用阈值内,则(i,j)=1,且l是肽或相应参考目标中的氨基酸位置数。或者,在一些实施例中,(i,j)可以用作s
ij
差的加权因子而不是0或1乘数。
[0156]
在一些实施例中,可以使用链拓扑参数(ctp)将从参考目标导出的工程化肽的一个或多个原子或氨基酸与相应参考目标原子或氨基酸进行比较。在一些实施例中,所述工程化肽原子或氨基酸的ctp相对于参考目标中的相应原子或氨基酸的ctp为 /-50%,其中链内拓扑相互作用是小于或等于的成对距离。在一些实施例中,被比较的工程化肽中的原子或氨基酸与生物功能或生物反应相关。在一些实施例中,第i、第j成对距离可以通过分子模拟(例如分子动力学)和/或实验室测量(例如nmr)来确定。用于评估ctp的示例性方程式为:
[0157][0158]
其中i、j是氨基酸(i,j)的位置索引,s
ij
是拓扑约束s(i)与s(j)之间的差异,如果氨基酸(i,j)在链拓扑相互作用阈值内,则(i,j)=1,l是肽或相应参考目标中的氨基酸位置数,且n是工程化肽或参考目标中满足拓扑相互作用阈值的链内接触总数。或
者,在一些实施例中,(i,j)可以用作s
ij
差的加权因子而不是0或1乘数。
[0159]
在一些实施例中,可以使用相似性定量估计(qel)将从参考目标导出的工程化肽的一个或多个原子或氨基酸与相应参考目标原子或氨基酸进行比较。在一些实施例中,所述工程化肽原子或氨基酸的qel相对于参考目标中的相应原子或氨基酸的qel为 /-50%。在一些实施例中,被比较的工程化肽中的原子或氨基酸与生物功能或生物反应相关。用于确定qel的示例性方程式是:
[0160][0161]
其中di是第i个氨基酸或原子位置的拓扑约束,或者是组合第i个氨基酸或原子位置的多个拓扑约束的组合函数(例如线性回归函数),且n是肽或参考目标中的氨基酸或原子位置数。
[0162]
在一些实施例中,可以使用拓扑聚类系数(tcc)向量和平均百分比误差(mpe)将从参考目标导出的工程化肽的一个或多个原子或氨基酸与相应参考目标原子或氨基酸进行比较。在一些实施例中,tcc向量和mpe相对于参考目标中的相应原子或氨基酸的tcc为小于75%,其中向量的每个元件(i)是第i个氨基酸位置的拓扑聚类系数,分子内簇由小于或等于的相互作用边缘距离和从第i个氨基酸位置开始的两个边缘i-j、j-l定义。在一些实施例中,被比较的工程化肽中的原子或氨基酸与生物功能或生物反应相关。在一些实施例中,第i、第j和第l边缘距离可以通过分子模拟(例如分子动力学)和/或实验室测量(例如nmr)确定。用于评估第i个位置的拓扑聚类系数的示例性方程式为:
[0163][0164]
其中如果分子内氨基酸位置(i,j)、(i,l)、(j,l)分别在相互作用边缘阈值内,则(i,j)=1,(i,l)=1,(j,l)=1,s
ijl
是第i、第j和第l个氨基酸的拓扑约束的组合(例如总和),l是肽向量或相应参考目标向量中的氨基酸位置数,nc为第i个氨基酸的分子内相互作用氨基酸位置数,满足边缘阈值和从第i个氨基酸开始的两个边缘i-j、j-l。或者,在一些实施例中,(i,j)、(i,l)和(j,l)可以作为聚类系数向量元件(i)的加权因子而不是0或1乘数。图51中提供了显示示例性工程化肽的簇和tcc向量的示例性图。
[0165]
在更进一步的实施例中,可以使用以下各者的lxm拓扑约束矩阵和平均百分比误差(mpe)将从参考目标导出的工程化肽的一个或多个原子或氨基酸与相应的参考目标原子或氨基酸进行比较:跨越所有m维的欧几里得距离、功率距离、索格尔距离、堪培拉距离、索伦森距离、杰卡德距离、马氏距离或汉明距离。lxm矩阵元件(l,m)含有第l个氨基酸位置的第m个约束值,其中l是氨基酸位置的数量,且m是不同拓扑约束的数量。在一些实施例中,工程化肽lxm矩阵的mpe相对于相应参考目标原子或氨基酸的矩阵为小于75%。在一些实施例中,mpe为小于70%、小于65%、小于60%、小于55%、小于50%或小于45%。在一些实施例中,被比较的工程化肽中的原子或氨基酸与生物功能或生物反应相关。示例性lxm矩阵提供于图52中。
[0166]
iii.可编程体外选择
[0167]
在其它方面,本文进一步提供了使用本文所述的工程化肽,使用一系列程序化选择步骤选择结合配偶体的方法,其中至少一个选择步骤包括评估潜在结合配偶体池与工程化肽的相互作用。
[0168]
在一些实施例中,本文提供了使用两个或更多个选择分子来引导结合分子的选择的方法。在一些实施例中,所述方法包括对候选结合分子池进行至少一轮选择,其中每一轮包含至少一个负选择步骤,其中针对负选择分子筛选所述池的至少一部分,和至少一个正选择步骤,其中针对正选择分子筛选所述池的至少一部分。在一些实施例中,所述方法包含至少两轮、至少三轮、至少四轮、至少五轮、至少六轮、至少七轮、至少八轮、至少九轮、至少十轮或更多轮,其中每一轮独立地包含至少一个负选择步骤和至少一个正选择步骤。在一些实施例中,每一轮独立地包含多于一个负选择步骤,或多于一个正选择步骤,或其组合。图5提供了详述三轮选择的示例性示意图,其中第一轮和第三轮包含多于一个负选择步骤,并且第一轮进一步包含多于一轮正选择。如方案中所示,第一轮使用了两个负选择分子(“诱饵”),并且第三轮使用了三个负选择分子。此外,第一轮使用了两个正选择分子。
[0169]
在其中所述方法包含多于一轮的一些实施例中,独立地选择每个负选择分子和正选择分子。在其它实施例中,可以在多于一轮中使用相同的负选择分子,或相同的正选择分子,或其组合。例如,在图5中,在第1轮中使用的相同负选择分子在第3轮中再次使用,另外的第三负选择分子也包括在第3轮中。在某些实施例中,可以在每轮选择中独立地选择负选择步骤和正选择步骤的顺序。因此,例如,在一些实施例中,所述方法包含一轮或多轮选择,其中每一轮首先包含负选择步骤,且接着包含正选择步骤。在其它实施例中,所述方法包含一轮或多轮选择,其中每一轮首先包含正选择步骤,且接着包含负选择步骤。在更进一步的实施例中,所述方法包含一轮或多轮选择,其中每一轮独立地包含负选择步骤和正选择步骤,其中在每一轮中,负选择步骤独立地在正选择步骤之前或在正选择步骤之后。
[0170]
此类选择方法使用正( )和负(-)步骤来引导候选结合分子文库朝向和远离某些所需特征,例如结合特异性或结合亲和力。通过对正选择分子和负选择分子使用多个步骤,可以逐步引导候选物池以选择需要的特征和反对不需要的特征。此外,在一些实施例中,每一轮内的每个步骤的顺序以及轮次相对于彼此的顺序可以引导不同方向上的选择。因此,例如,在一些实施例中,与首先是(-)选择接着是( )选择的情况相比,包含一轮( )选择接着是(-)选择的方法将产生不同的最终候选物池。将此推断至包含多轮的方法,即使总体上使用相同的正选择分子和负选择分子,选择步骤的顺序也可能导致所选候选物的最终池不同。
[0171]
在一些实施例中,使用具有另一种选择分子的逆特征的选择分子。这可能适用于例如确保使用正选择分子鉴定(或因负选择分子而被排除)的候选结合配偶体是因为所需性状(或非所需性状),而不是因为独立的、不相关的结合相互作用而被鉴定(或排除)。为了去除通过不相关相互作用结合的结合配偶体,可以使用与选择分子具有相似或相同结构和特征,不同之处在于传达所需性状(或非所需性状)的残基/结构的逆选择分子。例如,如果需要与正选择分子中的特定电荷模式相互作用,则可以使用逆负选择分子,所述分子已经用不带电残基和/或相反电荷的残基替换了提供所述电荷模式的残基。因此,对于某些选择分子,多个不同的对应逆选择分子可为可能的。
[0172]
在本文提供的选择方法中,至少一种选择分子是如本文所述的工程化肽。在一些实施例中,使用多于一种工程化肽。在一些实施例中,每个工程化肽独立地是正选择分子或负选择分子。在某些实施例中,在一轮或多轮选择中使用的每个选择分子独立地是工程化肽。在其它实施例中,至少一个不是工程化肽的分子用作选择分子。此类不是工程化肽的选择分子可包含例如天然存在的多肽或其一部分。在其它实施例中,一个或多个不是工程化肽的选择分子可包含例如非天然存在的多肽或其部分。例如,在一些实施例中,一个或多个选择分子(例如,正选择分子或负选择分子)是免疫原、抗体、细胞表面受体、或跨膜蛋白、或信号传导蛋白、或多蛋白复合物,或肽-蛋白质复合物,或其任何部分,或其任何组合。在一些实施例中,一个或多个选择分子是pd-1、pd-l1、cd25、il2、mif、cxcr4或vegf,或这些中的任一个的一部分,或这些中的任一个的抗体(例如贝伐单抗(bevacizumab)、阿维鲁单抗(avelumab)或度伐单抗(durvalumab))。
[0173]
在每个步骤中选择支持或对抗的正特征和负特征可以选自多种性状,并且可以根据最终获得的一个或多个结合分子的所需特征进行调整。此类所需特征可以取决于例如一种或多种结合分子的预期用途。例如,在一些实施例中,本文提供的方法用于支持一种或多种正特征(例如高特异性)和对抗一种或多种负特征(例如交叉反应性)来筛选候选抗体。应理解,在一个上下文中被认为是正特征的特征在另一上下文中可能是负特征,且反之亦然。因此,在一些实施例中,一个系列的选择轮次中的正选择分子在不同系列的选择轮次时,或在选择不同类型的结合分子时,或选择相同类型的结合分子但出于不同的目的时可以是负选择分子。
[0174]
在一些实施例中,每个选择特征独立地选自由以下组成的组:氨基酸序列、多肽二级结构、分子动力学、化学特征、生物功能、免疫原性、参考目标多特异性、跨物种参考目标反应性、所需参考目标超过非所需参考目标的选择性、序列和/或结构同源家族内参考目标的选择性、具有相似蛋白质功能的参考目标的选择性、从具有高序列和/或结构同源性的较大非所需目标家族选择不同所需参考目标的选择性、对不同参考目标等位基因或突变的选择性、对不同参考目标残基水平化学修饰的选择性、对细胞类型的选择性、对组织类型的选择性、对组织环境的选择性、对参考目标结构多样性的耐受性、对参考目标序列多样性的耐受性以及对参考目标动力学多样性的耐受性。在一些实施例中,每个选择特征是不同类型的选择特征。在其它实施例中,两个或更多个选择特征是不同特征但属于相同类型。例如,在一些实施例中,两个或多个选择特征是多肽二级结构,其中一个是针对所需多肽二级结构的正选择,且一个是针对非所需多肽二级结构的负选择。在一些实施例中,两个或更多个选择特征是针对细胞类型的选择性,其中正选择特征是针对特定所需细胞类型的选择性,且负选择特征是针对特定非所需细胞类型的选择性。在一些实施例中,两个或更多个、三个或更多个、四个或更多个、五个或更多个或六个或更多个选择特征是相同类型的。
[0175]
在另一方面,本文提供包含两种或更多种选择导向多肽的组合物,其中每种多肽独立地为包含一种或多种正导向特征的正选择分子,或包含一种或多种负导向特征的负选择分子。在一些实施例中,此类特征可选自由以下组成的组:氨基酸序列、多肽二级结构、分子动力学、化学特征、生物功能、免疫原性、参考目标多特异性、跨物种参考目标反应性、所需参考目标超过非所需参考目标的选择性、序列和/或结构同源家族内参考目标的选择性、具有相似蛋白质功能的参考目标的选择性、从具有高序列和/或结构同源性的较大非所需
目标家族选择不同所需参考目标的选择性、对不同参考目标等位基因或突变的选择性、对不同参考目标残基水平化学修饰的选择性、对细胞类型的选择性、对组织类型的选择性、对组织环境的选择性、对参考目标结构多样性的耐受性、对参考目标序列多样性的耐受性以及对参考目标动力学多样性的耐受性。
[0176]
因此,在另外的方面,本文提供一种用本文所述的选择导向组合物筛选结合分子文库的方法,其中每轮选择包含:针对负选择分子筛选池的至少一部分的负选择步骤;和针对正选择分子筛选所述池的至少一部分的正选择步骤;其中每一轮内的选择步骤的顺序和轮次的顺序导致选择与替代顺序不同的池子集。
[0177]
在一些实施例中,使用如本文所述的选择导向多肽的组合物或如本文所述的筛选方法评估的结合配偶体是噬菌体文库,例如含有fab的噬菌体文库;或细胞文库,例如b细胞文库或t细胞文库。
[0178]
在本文提供的筛选方法的一些实施例中,所述方法包含两轮或更多轮、三轮或更多轮、四轮或更多轮、五轮或更多轮、六轮或更多轮、或七轮或更多轮的选择。在一些实施例中,其中存在多于一轮,每一轮包含不同的选择分子集。在一些实施例中,其中存在多于一轮,至少两轮包含相同的负选择分子、相同的正选择分子或两者。
[0179]
在筛选方法的一些实施例中,所述方法包含在继续进行下一轮选择之前分析池子集。在某些实施例中,每个子集池分析独立地选自由以下组成的组:肽/蛋白质生物传感器结合、肽/蛋白质elisa、肽文库结合、细胞提取物结合、细胞表面结合、细胞活性分析、细胞增殖分析、细胞死亡分析、酶活性分析、基因表达谱、蛋白质修饰分析、蛋白质印迹和免疫组织化学。在一些实施例中,基因表达谱包含子集池的全序列库分析,例如下一代测序。在一些实施例中,统计学和/或信息学评分或机器学习训练用于在一轮或多轮选择中评估池的一个或多个子集。
[0180]
在一些实施例中,通过分析来自一轮选择的子集池来确定用于后一轮的正和/或负选择分子的身份和/或顺序。在一些实施例中,统计学和/或信息学评分或机器学习训练用于在一轮或多轮选择中评估池的一个或多个子集,以确定后一轮(例如下一轮,或程序中更进一步的一轮)的正和/或负选择分子的身份和/或顺序。
[0181]
在更进一步的实施例中,选择方法包括在进行下一轮选择之前修饰从一轮选择获得的子集池。此类修饰可包括例如子集池的遗传突变、子集池的遗传耗竭(例如,选择子集池的子集以向前推进选择)、子集池的遗传富集(例如,增加池的大小)、至少一部分子集池的化学修饰或至少一部分子集池的酶修饰,或其任何组合。在一些实施例中,统计学和/或信息学评分或机器学习训练用于评估子集池并确定在选择中向前移动修改的子集池之前要进行的一个或多个修改。在某些实施例中,此类统计学和/或信息学评分或机器学习训练还用于确定用于后一轮选择的正和/或负选择分子的身份和/或顺序。
[0182]
可以使用任何合适的分析来评估每个步骤中结合配偶体池与选择分子的结合。在一些实施例中,直接评估结合,例如通过直接检测结合配偶体上的标记。此类标记可以包括例如荧光标记,例如荧光团或荧光蛋白。在其它实施例中,间接评估结合,例如使用夹心分析。在夹心分析中,结合配偶体与选择分子结合,且接着添加二级标记试剂以标记结合的结合配偶体。接着检测此二级标记试剂。夹心分析组分的实例包括用抗his标签抗体或his标签特异性荧光探针检测到的his标签结合配偶体;用标记的链霉亲和素或标记的亲和素检
测的生物素标记的结合配偶体;或使用抗结合配偶体抗体检测到的未标记的结合配偶体。
[0183]
在一些实施例中,在每个步骤中选择的结合配偶体是基于结合信号或剂量反应,使用任何数量的可用检测方法来鉴定的。这些检测方法可以包括例如成像、荧光激活细胞分选(facs)、质谱法或生物传感器。在一些实施例中,定义命中阈值(例如中值信号),并且任何高于所述信号的信号都被标记为推定的命中基序。
[0184]
iv.使用工程化肽生产抗体
[0185]
本文提供的并通过本文提供的方法鉴定的工程化肽可用于例如产生一种或多种抗体。在一些实施例中,抗体是单克隆或多克隆抗体。因此,在一些实施例中,本文提供通过用免疫原使动物免疫而产生的抗体,其中免疫原是如本文提供的工程化肽。在一些实施例中,动物是人、兔、小鼠、仓鼠、猴等。在某些实施例中,猴是食蟹猴、猕猴或恒河猴。用工程化肽使动物免疫可包含例如向动物施用至少一剂包含肽和任选的佐剂的组合物。在一些实施例中,从动物产生抗体包含分离表达抗体的b细胞。一些实施例进一步包含将b细胞与骨髓瘤细胞融合以产生表达抗体的杂交瘤。在一些实施例中,使用工程化肽产生的抗体可以与人和猴,例如食蟹猴交叉反应。
[0186]
本文提供的描述阐述了许多示例性配置、方法、参数等。然而,应认识到,此类描述不打算作为对本公开的范围的限制,而是打算作为示例性实施例的描述提供。
[0187]
示例性实施例
[0188]
实施例i-1.一种工程化肽,其中所述工程化肽具有介于1kda与10kda之间的分子量并且包含至多50个氨基酸,并且其中所述工程化肽包含:
[0189]
空间相关拓扑约束的组合,其中一个或多个所述约束是参考目标导出的约束;并且
[0190]
其中所述工程化肽的10%至98%的所述氨基酸满足一个或多个参考目标导出的约束,
[0191]
其中满足所述一个或多个参考目标导出的约束的所述氨基酸与所述参考目标的主链均方根偏差(rsmd)结构同源性为小于
[0192]
实施例i-2.根据实施例i-1所述的工程化肽,其中满足一个或多个参考目标导出的约束的氨基酸与参考目标具有10%至90%的序列同源性。
[0193]
实施例i-3.根据实施例i-1或i-2所述的工程化肽,其中满足一个或多个参考目标导出的约束的氨基酸与参考的范德华表面积重叠为导出的约束的氨基酸与参考的范德华表面积重叠为至
[0194]
实施例i-4.根据实施例i-1至i-3中任一项所述的工程化肽,其中所述组合包含至少两个参考目标导出的约束。
[0195]
实施例i-5.根据实施例i-1至i-4中任一项所述的工程化肽,其中所述组合包含至少五个参考目标导出的约束。
[0196]
实施例i-6.根据实施例i-1至i-5中任一项所述的工程化肽,其中所述约束的组合包含一个或多个并非从参考目标导出的约束。
[0197]
实施例i-7.根据实施例i-6所述的工程化肽,其中所述一个或多个非参考目标导出的约束描述了所需的结构、动力学、化学或功能特征,或其任何组合。
[0198]
实施例i-8.根据实施例i-1至i-7中任一项所述的工程化肽,其中所述约束独立地
选自由以下组成的组:
[0199]
原子距离;
[0200]
原子波动;
[0201]
原子能;
[0202]
化学描述符;
[0203]
溶剂暴露;
[0204]
氨基酸序列相似性;
[0205]
生物信息学描述符;
[0206]
非共价键合倾向;
[0207]
角;
[0208]
ψ角;
[0209]
范德华半径;
[0210]
二级结构倾向;
[0211]
氨基酸邻接;和
[0212]
氨基酸接触。
[0213]
实施例i-9.根据实施例i-1至i-8中任一项所述的工程化肽,其中一个或多个约束独立地为原子波动。
[0214]
实施例i-10.根据实施例i-1至i-9中任一项所述的工程化肽,其中一个或多个约束独立地为化学描述符。
[0215]
实施例i-11.根据实施例i-1至i-10中任一项所述的工程化肽,其中一个或多个约束独立地为原子距离。
[0216]
实施例i-12.根据实施例i-1至i-11中任一项所述的工程化肽,其中一个或多个约束独立地为二级结构。
[0217]
实施例i-13.根据实施例i-1至i-12中任一项所述的工程化肽,其中一个或多个约束独立地为范德华表面。
[0218]
实施例i-14.根据实施例i-1至i-13中任一项所述的工程化肽,其中一个或多个约束独立地与生物反应或生物功能相关。
[0219]
实施例i-15.根据实施例i-1至i-14中任一项所述的工程化肽,其包含一个或多个与生物反应或生物功能相关的原子。
[0220]
实施例i-16.根据实施例i-1至i-15中任一项所述的工程化肽,其包含一个或多个与生物反应或生物功能相关的氨基酸。
[0221]
实施例i-17.根据实施例i-14至i-16中任一项所述的工程化肽,其中所述生物反应或生物功能选自由以下组成的组:基因表达、代谢活性、蛋白质表达、细胞增殖、细胞死亡、细胞因子分泌、激酶活性、表观遗传修饰、细胞杀伤活性、炎症信号、趋化性、组织浸润、免疫细胞谱系定型、组织微环境修饰、免疫突触形成、il-2分泌、il-10分泌、生长因子分泌、γ干扰素分泌、转化生长因子β分泌、基于免疫受体酪氨酸的激活基序活性、基于免疫受体酪氨酸的抑制基序活性、抗体定向的细胞毒性、补体定向的细胞毒性、生物途径激动作用、生物途径拮抗作用、生物途径重定向、激酶级联修饰、蛋白水解途径修饰、蛋白稳态途径修饰、蛋白质折叠/途径、翻译后修饰途径、代谢途径、基因转录/翻译、mrna降解途径、基因甲
基化/乙酰化途径、组蛋白修饰途径、表观遗传途径、免疫定向清除、调理作用、激素信号传导、整合素途径、膜蛋白信号转导、离子通道通量和g蛋白偶联受体反应。
[0222]
实施例i-18.根据实施例i-15所述的工程化肽,其中所述参考目标包含一个或多个与生物反应或生物功能相关的原子,
[0223]
且其中在与生物反应或生物功能相关的所述工程化肽中的所述一个或多个原子的原子波动与在与生物反应或生物功能相关的所述参考目标中的所述一个或多个原子的原子波动重叠。
[0224]
实施例i-19.根据实施例i-18所述的工程化肽,其中所述重叠为大于0.25的均方根内积(rmsip)。
[0225]
实施例i-20.根据实施例i-19所述的工程化肽,其中所述重叠具有大于0.75的均方根内积(rmsip)。
[0226]
实施例i-21.根据实施例i-18至i-20中任一项所述的工程化肽,其中与生物反应或生物功能相关的所述工程化肽中的至少一部分所述原子在拓扑上受约束于所述参考目标中的二级结构元件。
[0227]
实施例i-22.根据实施例i-21所述的工程化肽,其中所述二级结构元件为β-片层。
[0228]
实施例i-23.根据实施例i-21所述的工程化肽,其中所述二级结构元件为α螺旋。
[0229]
实施例i-24.根据实施例i-21所述的工程化肽,其中所述二级结构元件为转角,其中所述转角包含2至7个残基,并且包含至少一个残基间氢键。
[0230]
实施例i-25.根据实施例i-21所述的工程化肽,其中所述二级结构元件为卷曲,其中所述卷曲包含2至20个残基。
[0231]
实施例i-26.根据实施例i-25所述的工程化肽,其中所述卷曲不包含残基间氢键。
[0232]
实施例i-27.根据实施例i-21至i-26中任一项所述的工程化肽,其中与生物反应或生物功能相关的所述工程化肽中的至少一部分所述原子在拓扑上受约束于两个或更多个独立地选自以下组成的组的二级结构元件:β-片层、α螺旋、转角和卷曲。
[0233]
实施例i-28.根据实施例i-1至i-27中任一项所述的工程化肽,其中一个或多个空间相关拓扑约束为原子距离。
[0234]
实施例i-29.根据实施例i-1至i-28中任一项所述的工程化肽,其中一个或多个空间相关拓扑约束为原子能。
[0235]
实施例i-30.根据实施例i-29所述的工程化肽,其中每个原子能独立地为两个原子之间的成对吸引能、两个原子之间的成对排斥能、原子级溶剂化能、两个原子之间的成对带电吸引能、两个原子之间的成对氢键合吸引能或非共价键合能。
[0236]
实施例i-31.根据实施例i-1至i-30中任一项所述的工程化肽,其中一个或多个空间相关拓扑约束为化学描述符。
[0237]
实施例i-32.根据实施例i-31所述的工程化肽,其中每个化学描述符独立地为疏水性、极性、体积、净电荷、logp、高效液相色谱保留或范德华半径。
[0238]
实施例i-33.根据实施例i-1至i-32中任一项所述的工程化肽,其中一个或多个空间相关拓扑约束为生物信息学描述符。
[0239]
实施例i-34.根据实施例i-33所述的工程化肽,其中每个生物信息学描述符独立地为blosum相似性、pka、zscale、克鲁恰尼特性、基德拉因子、vhse量表、protfp、ms-whim分
数、t量表、st量表、跨膜倾向、蛋白质埋藏面积、螺旋倾向、片层倾向、卷曲倾向、转向倾向、免疫原性倾向、抗体表位出现率或蛋白质界面出现率。
[0240]
实施例i-35.根据实施例i-1至i-34中任一项所述的工程化肽,其中一个或多个空间相关拓扑约束为溶剂暴露。
[0241]
实施例i-36.根据实施例i-1至i-35中任一项所述的工程化肽,其中所述一个或多个参考目标导出的约束中的至少一个为gpcr胞外域。
[0242]
实施例i-37.根据实施例i-1至i-36中任一项所述的工程化肽,其中所述一个或多个参考目标导出的约束中的至少一个为离子通道胞外域。
[0243]
实施例i-38.根据实施例i-1至i-37中任一项所述的工程化肽,其中所述一个或多个参考目标导出的约束中的至少一个为蛋白质-蛋白质或肽-蛋白质界面连接。
[0244]
实施例i-39.根据实施例i-1至i-38中任一项所述的工程化肽,其中所述一个或多个参考目标导出的约束中的至少一个是从所述目标的多态性区域导出。
[0245]
实施例i-40.根据实施例i-1至i-39中任一项所述的工程化肽,其包含一个或多个与生物反应或生物功能相关的原子,其中所述一个或多个原子中的每一个独立地选自由以下组成的组:碳、氧、氮、氢、硫、磷、钠、钾、锌、锰、镁、铜、铁、钼和镍。
[0246]
实施例i-41.根据实施例i-1至i-40中任一项所述的工程化肽,其包含一个或多个与生物功能或生物反应相关的氨基酸,其中所述一个或多个氨基酸中的每一个独立地为蛋白型天然存在的氨基酸、非蛋白型天然存在的氨基酸或化学合成的非天然氨基酸。
[0247]
实施例i-42.根据实施例i-1至i-41中任一项所述的工程化肽,其中当与所述参考目标相比时,所述工程化肽具有至少一个结构差异。
[0248]
实施例i-43.根据实施例i-42所述的工程化肽,其中所述至少一个结构差异独立地选自由以下组成的组:序列、氨基酸残基数、原子总数、总亲水性、总疏水性、总正电荷、总负电荷、一个或多个二级结构、形状因子、泽尼克描述符、范德华表面、结构图节点和边、体积表面、静电势表面、疏水势表面、局部直径、局部表面特征、骨架模型、电荷密度、亲水密度、表面积与体积比、两亲密度和表面粗糙度。
[0249]
实施例i-44.根据实施例i-16所述的工程化肽,其中所述一个或多个二级结构的差异是与所述参考目标相比,所述工程化肽中存在一个或多个额外的二级结构元件,其中每个额外的二级结构元件独立地选自由α螺旋、β-片层、环、转角和卷曲组成的组。
[0250]
实施例i-45.根据实施例i-1至i-44中任一项所述的工程化肽,其中10%至90%的所述氨基酸满足一个或多个非参考目标导出的拓扑约束。
[0251]
实施例i-46.根据实施例i-45所述的工程化肽,其中所述一个或多个非参考目标导出的拓扑约束加强预先指定的功能。
[0252]
实施例i-47.根据实施例i-46所述的工程化肽,其中所述
[0253]
非参考导出的拓扑约束加强或稳定所述肽的所述参考导出部分中的二级结构元件;
[0254]
非参考导出的拓扑约束加强所述肽的所述参考导出部分中的原子波动;
[0255]
非参考导出的拓扑约束改变肽总疏水性;
[0256]
非参考导出的拓扑约束改变肽溶解度;
[0257]
非参考导出的拓扑约束改变肽总电荷;
[0258]
非参考导出的拓扑约束使得能够在经标记或无标记分析中进行检测;
[0259]
非参考导出的拓扑约束使得能够在体外分析中进行检测;
[0260]
非参考导出的拓扑约束使得能够在体内分析中进行检测;
[0261]
非参考导出的拓扑约束使得能够从复杂混合物中捕获;
[0262]
非参考导出的拓扑约束使得能够进行酶处理;
[0263]
非参考导出的拓扑约束使得细胞膜能够具透性;
[0264]
非参考导出的拓扑约束使得能够与二级目标结合,以及
[0265]
非参考导出的拓扑约束改变免疫原性。
[0266]
实施例i-48.一种选择工程化肽的方法,其包含:
[0267]
识别参考目标的一个或多个拓扑特征;
[0268]
为每个拓扑特征设计空间相关约束,以产生从所述参考目标导出的空间相关拓扑约束的组合;
[0269]
将候选肽的空间相关拓扑特征与从所述参考目标导出的空间相关拓扑约束的组合进行比较;以及
[0270]
选择具有空间相关拓扑特征的候选肽,所述特征与从所述参考目标导出的所述空间相关拓扑约束的组合重叠,以产生所述工程化肽。
[0271]
实施例i-49.根据实施例i-48所述的方法,其中每个特征之间的所述重叠独立地小于或等于由以下中的一个或多个确定的75%平均百分比误差(mpe):总拓扑约束距离(tcd)、拓扑聚类系数(tcc)、欧几里得距离、功率距离、索格尔距离、堪培拉距离、索伦森距离、杰卡德距离、马氏距离、汉明距离、相似性定量估计(qel)或链拓扑参数(ctp)。
[0272]
实施例i-50.根据实施例i-48或i-49所述的方法,其中一个或多个约束是从以下各者导出:每个残基能量、每个残基相互作用、每个残基波动、每个残基原子距离、每个残基化学描述符、每个残基溶剂暴露、每个残基氨基酸序列相似性、每个残基生物信息学描述符、每个残基非共价键合倾向、每个残基角、每个残基范德华半径、每个残基二级结构倾向、每个残基氨基酸邻接、每个残基氨基酸接触。
[0273]
实施例i-51.根据实施例i-48至i-50中任一项所述的方法,其中所述一种或多种候选肽的特征通过计算机模拟确定。
[0274]
实施例i-52.根据实施例i-51所述的方法,其中所述计算机模拟包含分子动力学模拟、蒙特卡洛模拟、粗粒度模拟、高斯网络模型、机器学习或其任何组合。
[0275]
实施例i-53.根据实施例i-48至i-52中任一项所述的方法,其中所述一种或多种候选肽的特征通过实验表征确定。
[0276]
实施例i-54.根据实施例i-48至i-53中任一项所述的方法,其中满足所述一个或多个参考目标导出的约束的所述氨基酸与所述参考目标具有10%至90%的序列同源性。
[0277]
实施例i-55.根据实施例i-48至i-54中任一项所述的方法,其中满足所述一个或多个参考目标导出的约束的所述氨基酸与所述参考的范德华表面积重叠为至
[0278]
实施例i-56.根据实施例i-48至i-55中任一项所述的方法,其中所述组合包含至少两个参考目标导出的约束。
[0279]
实施例i-57.根据实施例i-48至i-56中任一项所述的方法,其中所述组合包含至
少五个参考目标导出的约束。
[0280]
实施例i-58.根据实施例i-48至i-57中任一项所述的方法,其中所述约束的组合包含一个或多个并非从参考目标导出的约束。
[0281]
实施例i-59.根据实施例i-58所述的方法,其中所述一个或多个非参考目标导出的约束描述了所需的结构、动力学、化学或功能特征,或其任何组合。
[0282]
实施例i-60.根据实施例i-48至i-59中任一项所述的方法,其中所述约束独立地选自由以下组成的组:
[0283]
原子距离;
[0284]
原子波动;
[0285]
原子能;
[0286]
化学描述符;
[0287]
溶剂暴露;
[0288]
氨基酸序列相似性;
[0289]
生物信息学描述符;
[0290]
非共价键合倾向;
[0291]
角;
[0292]
ψ角;
[0293]
范德华半径;
[0294]
二级结构倾向;
[0295]
氨基酸邻接;和
[0296]
氨基酸接触。
[0297]
实施例i-61.根据实施例i-48至i-60中任一项所述的方法,其中一个或多个约束独立地为原子波动。
[0298]
实施例i-62.根据实施例i-48至i-61中任一项所述的方法,其中一个或多个约束独立地为化学描述符。
[0299]
实施例i-63.根据实施例i-48至i-62中任一项所述的方法,其中一个或多个约束独立地为原子距离。
[0300]
实施例i-64.根据实施例i-48至i-63中任一项所述的方法,其中一个或多个约束独立地为二级结构。
[0301]
实施例i-65.根据实施例i-48至i-64中任一项所述的方法,其中一个或多个约束独立地为范德华表面。
[0302]
实施例i-66.根据实施例i-48至i-65中任一项所述的方法,其中一个或多个约束独立地与生物反应或生物功能相关。
[0303]
实施例i-67.根据实施例i-48至i-66中任一项所述的方法,其中所述工程化肽包含一个或多个与生物反应或生物功能相关的原子。
[0304]
实施例i-68.根据实施例i-48至i-66中任一项所述的方法,其中所述工程化肽包含一个或多个与生物反应或生物功能相关的氨基酸。
[0305]
实施例i-69.根据实施例i-66至i-68中任一项所述的方法,其中所述生物反应或生物功能选自由以下组成的组:基因表达、代谢活性、蛋白质表达、细胞增殖、细胞死亡、细
胞因子分泌、激酶活性、表观遗传修饰、细胞杀伤活性、炎症信号、趋化性、组织浸润、免疫细胞谱系定型、组织微环境修饰、免疫突触形成、il-2分泌、il-10分泌、生长因子分泌、γ干扰素分泌、转化生长因子β分泌、基于免疫受体酪氨酸的激活基序活性、基于免疫受体酪氨酸的抑制基序活性、抗体定向的细胞毒性、补体定向的细胞毒性、生物途径激动作用、生物途径拮抗作用、生物途径重定向、激酶级联修饰、蛋白水解途径修饰、蛋白稳态途径修饰、蛋白质折叠/途径、翻译后修饰途径、代谢途径、基因转录/翻译、mrna降解途径、基因甲基化/乙酰化途径、组蛋白修饰途径、表观遗传途径、免疫定向清除、调理作用、激素信号传导、整合素途径、膜蛋白信号转导、离子通道通量和g蛋白偶联受体反应。
[0306]
实施例i-70.根据实施例i-66所述的方法,其中所述参考目标包含一个或多个与生物反应或生物功能相关的原子,
[0307]
且其中在与生物反应或生物功能相关的所述工程化肽中的所述一个或多个原子的原子波动与在与生物反应或生物功能相关的所述参考目标中的所述一个或多个原子的原子波动重叠。
[0308]
实施例i-71.根据实施例i-70所述的方法,其中所述重叠为大于0.25的均方根内积(rmsip)。
[0309]
实施例i-72.根据实施例i-71所述的方法,其中所述重叠具有大于0.75的均方根内积(rmsip)。
[0310]
实施例i-73.根据实施例i-67至i-69中任一项所述的方法,其中与生物反应或生物功能相关的所述工程化肽中的至少一部分所述原子在拓扑上受限于所述参考目标中的二级结构元件。
[0311]
实施例i-74.根据实施例i-73所述的方法,其中所述二级结构元件为β-片层。
[0312]
实施例i-75.根据实施例i-73所述的方法,其中所述二级结构元件为α螺旋。
[0313]
实施例i-76.根据实施例i-73所述的方法,其中所述二级结构元件为转角,其中所述转角包含2至7个残基,并且包含至少一个残基间氢键。
[0314]
实施例i-77.根据实施例i-73所述的方法,其中所述二级结构元件为卷曲,其中所述卷曲包含2至20个残基。
[0315]
实施例i-78.根据实施例i-73所述的方法,其中所述卷曲不包含残基间氢键。
[0316]
实施例i-79.根据实施例i-67至i-69中任一项所述的方法,其中与生物反应或生物功能相关的所述工程化肽中的至少一部分所述原子在拓扑上受限于两个或更多个独立地选自以下组成的组的二级结构元件:β-片层、α螺旋、转角和卷曲。
[0317]
实施例i-80.根据实施例i-48至i-79中任一项所述的方法,其中一个或多个空间相关拓扑约束为原子距离。
[0318]
实施例i-81.根据实施例i-48至i-80中任一项所述的方法,其中一个或多个空间相关拓扑约束为原子能。
[0319]
实施例i-82.根据实施例i-81所述的方法,其中每个原子能独立地为两个原子之间的成对吸引能、两个原子之间的成对排斥能、原子级溶剂化能、两个原子之间的成对带电吸引能、两个原子之间的成对氢键合吸引能或非共价键合能。
[0320]
实施例i-83.根据实施例i-48至i-82中任一项所述的方法,其中一个或多个空间相关拓扑约束为化学描述符。
[0321]
实施例i-84.根据实施例i-83所述的方法,其中每个化学描述符独立地为疏水性、极性、体积、净电荷、logp、高效液相色谱保留或范德华半径。
[0322]
实施例i-85.根据实施例i-48至i-84中任一项所述的方法,其中一个或多个空间相关拓扑约束为生物信息学描述符。
[0323]
实施例i-86.根据实施例i-85所述的方法,其中每个生物信息学描述符独立地为blosum相似性、pka、zscale、克鲁恰尼特性、基德拉因子、vhse量表、protfp、ms-whim分数、t量表、st量表、跨膜倾向、蛋白质埋藏面积、螺旋倾向、片层倾向、卷曲倾向、转向倾向、免疫原性倾向、抗体表位出现率或蛋白质界面出现率。
[0324]
实施例i-87.根据实施例i-48至i-86中任一项所述的方法,其中一个或多个空间相关拓扑约束为溶剂暴露。
[0325]
实施例i-88.根据实施例i-48至i-87中任一项所述的方法,其中所述一个或多个参考目标导出的约束中的至少一个为gpcr胞外域。
[0326]
实施例i-89.根据实施例i-48至i-88中任一项所述的方法,其中所述一个或多个参考目标导出的约束中的至少一个为离子通道胞外域。
[0327]
实施例i-90.根据实施例i-48至i-89中任一项所述的方法,其中所述一个或多个参考目标导出的约束中的至少一个为蛋白质-蛋白质或蛋白质-肽界面连接。
[0328]
实施例i-91.根据实施例i-48至i-90中任一项所述的方法,其中所述一个或多个参考目标导出的约束中的至少一个是从所述目标的多态性区域导出。
[0329]
实施例i-92.根据实施例i-48至i-91中任一项所述的方法,其中所述工程化肽包含一个或多个与生物反应或生物功能相关的原子,其中所述一个或多个原子中的每一个独立地选自由以下组成的组:碳、氧、氮、氢、硫、磷、钠、钾、锌、锰、镁、铜、铁、钼和镍。
[0330]
实施例i-93.根据实施例i-48至i-92中任一项所述的方法,其中所述工程化肽包含一个或多个与生物功能或生物反应相关的氨基酸,其中所述一个或多个氨基酸中的每一个独立地为蛋白型天然存在的氨基酸、非蛋白型天然存在的氨基酸或化学合成的非天然氨基酸。
[0331]
实施例i-94.根据实施例i-48至i-93中任一项所述的方法,其中当与所述参考目标相比时,所述工程化肽具有至少一个结构差异。
[0332]
实施例i-95.根据实施例i-94所述的方法,其中所述至少一个结构差异独立地选自由以下组成的组:序列、氨基酸残基数、原子总数、总亲水性、总疏水性、总正电荷、总负电荷、一个或多个二级结构、形状因子、泽尼克描述符、范德华表面、结构图节点和边、体积表面、静电势表面、疏水势表面、局部直径、局部表面特征、骨架模型、电荷密度、亲水密度、表面积与体积比、两亲密度和表面粗糙度。
[0333]
实施例i-96.根据实施例i-95所述的方法,其中所述一个或多个二级结构的差异是与所述参考目标相比,所述工程化肽中存在一个或多个额外的二级结构元件,其中每个额外的二级结构元件独立地选自由α螺旋、β-片层、环、转角和卷曲组成的组。
[0334]
实施例i-97.根据实施例i-48至i-96中任一项所述的方法,其中所述工程化肽的10%至90%的所述氨基酸满足一个或多个非参考目标导出的拓扑约束。
[0335]
实施例i-98.根据实施例i-97所述的方法,其中所述一个或多个非参考目标导出的拓扑约束加强预先指定的功能。
[0336]
实施例i-99.根据实施例i-98所述的方法,其中:
[0337]
非参考导出的拓扑约束加强或稳定所述肽的所述参考导出部分中的二级结构元件;
[0338]
非参考导出的拓扑约束加强所述肽的所述参考导出部分中的原子波动;
[0339]
非参考导出的拓扑约束改变肽总疏水性;
[0340]
非参考导出的拓扑约束改变肽溶解度;
[0341]
非参考导出的拓扑约束改变肽总电荷;
[0342]
非参考导出的拓扑约束使得能够在经标记或无标记分析中进行检测;
[0343]
非参考导出的拓扑约束使得能够在体外分析中进行检测;
[0344]
非参考导出的拓扑约束使得能够在体内分析中进行检测;
[0345]
非参考导出的拓扑约束使得能够从复杂混合物中捕获;
[0346]
非参考导出的拓扑约束使得能够进行酶处理;
[0347]
非参考导出的拓扑约束使得细胞膜能够具透性;
[0348]
非参考导出的拓扑约束使得能够与二级目标结合,或
[0349]
非参考导出的拓扑约束改变免疫原性,
[0350]
或其任何组合。
[0351]
实施例i-100.一种包含两种或更多种选择导向多肽的组合物,其中每一种多肽独立地为包含一种或多种正导向特征的正选择分子,或包含一种或多种负导向特征的负选择分子,其中每个特征类型独立地选自由以下组成的组:
[0352]
氨基酸序列,
[0353]
多肽二级结构,
[0354]
分子动力学,
[0355]
化学特征,
[0356]
生物学功能,
[0357]
免疫原性,
[0358]
参考目标多特异性,
[0359]
跨物种参考目标反应性,
[0360]
所需参考目标超过非所需参考目标的选择性,
[0361]
序列和/或结构同源家族内参考目标的选择性,
[0362]
具有相似蛋白质功能的参考目标的选择性,
[0363]
从具有高序列和/或结构同源性的较大非所需目标家族选择不同所需参考目标的选择性,
[0364]
对不同参考目标等位基因或突变的选择性,
[0365]
对不同参考目标残基水平化学修饰的选择性,
[0366]
对细胞类型的选择性,
[0367]
对组织类型的选择性,
[0368]
对组织环境的选择性,
[0369]
对参考目标结构多样性的耐受性,
[0370]
对参考目标序列多样性的耐受性,以及
[0371]
对参考目标动力学多样性的耐受性;
[0372]
并且其中两种或更多种多肽中的至少一种是根据实施例i-1所述的工程化肽。
[0373]
实施例i-101.根据实施例i-100所述的组合物,其中所述两种或更多种多肽中的至少一种是正选择分子,并且所述两种或更多种多肽中的至少一种是负选择分子。
[0374]
实施例i-102.根据实施例i-100或i-101所述的组合物,其中所述两种或更多种多肽中的至少一种是天然蛋白质。
[0375]
实施例i-103.根据实施例i-100至i-102中任一项所述的组合物,其包含至少一对对应的正选择和负选择分子,所述分子包含至少一种共有特征类型,其中所述正选择分子包含所述正特征并且所述负选择分子包含所述负特征。
[0376]
实施例i-104.一种筛选具有根据实施例i-100所述的组合物的结合分子文库的方法,所述方法包含使候选结合分子池经受至少一轮选择,其中每轮选择包含:
[0377]
针对负选择分子筛选所述池的至少一部分的负选择步骤;和
[0378]
针对正选择分子筛选所述池的至少一部分的正选择步骤;
[0379]
其中每一轮内的选择步骤的顺序和轮次的顺序导致选择与替代顺序不同的池子集。
[0380]
实施例i-105.根据实施例i-104所述的方法,其中所述结合分子文库为噬菌体文库。
[0381]
实施例i-106.根据实施例i-105所述的方法,其中所述结合分子文库为细胞文库。
[0382]
实施例i-107.根据实施例i-106所述的方法,其中所述结合分子文库为b细胞文库。
[0383]
实施例i-108.根据实施例i-106所述的方法,其中所述结合分子文库为t细胞文库。
[0384]
实施例i-109.根据实施例i-104至i-108中任一项所述的方法,其包含两轮或更多轮选择。
[0385]
实施例i-110.根据实施例i-104至i-109中任一项所述的方法,其包含三轮或更多轮选择。
[0386]
实施例i-111.根据实施例i-109或i-110所述的方法,其中每一轮包含不同的选择分子集。
[0387]
实施例i-112.根据实施例i-109或i-110所述的方法,其中至少两轮包含相同的负选择分子,或相同的正选择分子,或两者。
[0388]
实施例i-113.根据实施例i-109至i-112中任一项所述的方法,其包含在继续进行下一轮选择之前分析从一轮选择中获得的所述池子集。
[0389]
实施例i-114.根据实施例i-113所述的方法,其中所述子集池分析确定在一轮或多轮后续选择中使用的正和/或负选择分子集。
[0390]
实施例i-115.根据实施例i-113或i-114所述的方法,其中每个子集池分析独立地选自由以下组成的组:肽/蛋白质生物传感器结合、肽/蛋白质elisa、肽文库结合、细胞提取物结合、细胞表面结合、细胞活性分析、细胞增殖分析、细胞死亡分析、酶活性分析、基因表达谱、蛋白质修饰分析、蛋白质印迹和免疫组织化学。
[0391]
实施例i-116.根据实施例i-113至i-115中任一项所述的方法,其中一轮或多轮后
续选择中使用的所述正、负或正和负选择分子由子集池分析的统计学/信息学评分或机器学习训练确定。
[0392]
实施例i-117.根据实施例i-109至i-116中任一项所述的方法,其中在进入下一轮选择之前修饰从一轮选择获得的所述子集池。
[0393]
实施例i-118.根据实施例i-117所述的方法,其中所述子集池分析确定一轮或多轮后续选择中使用的所述正、负或正和负选择分子;且在进入下一轮选择之前修饰所述子集池。
[0394]
实施例i-119.根据实施例i-117或i-118所述的方法,其中每个修饰独立地选自选自遗传突变、遗传耗竭、遗传富集、化学修饰和酶修饰的组。
[0395]
实例
[0396]
以下实例仅为说明性的且不打算以任何方式限制本公开的任何方面。
[0397]
实例1:使用vegf表位作为参考目标来选择工程化肽
[0398]
如图6a和7a所示,vegf的推定治疗表位被鉴定为工程化肽选择的参考目标,并且确定了原子距离和氨基酸描述符拓扑结构(图6b)。使用动态模拟获得参考目标的原子距离和氨基酸描述符拓扑,并为参考目标中的表位生成原子波动的协方差矩阵。接下来,使用计算蛋白质设计(例如rosetta)、对候选物进行的动力学模拟以及确定的原子距离和氨基酸描述符拓扑生成不同的工程化肽候选物(图6c-6e)。比较这些拓扑的这些平均百分比误差(mpe)(图6g-6h)。mpe值为:参考拓扑相对于候选物1拓扑:6.03%;参考拓扑相对于候选物2拓扑:6.00%;以及参考拓扑相对于候选物3拓扑:22.8%。
[0399]
将额外的约束添加到组合中以评估一种候选工程化肽-原子波动(图6g-6h)。比较此候选物与vegf导出的参考目标之间更高维的拓扑相似性,mpe为36.6%。
[0400]
实例2:使用vegf表位作为参考目标来选择工程化肽
[0401]
使用在以上实例1中鉴定的相同参考目标,开发了第二组工程化肽。使用计算蛋白质设计(例如rosetta)或其它取样肽空间的方法生成工程化肽候选物,并对候选物进行动力学模拟。为参考目标表位和对应于参考目标表位中的残基的候选物中的残基生成原子波动的协方差矩阵。
[0402]
进行主分量分析以计算每个协方差矩阵的特征向量和特征值
‑‑‑
参考目标一个协方差矩阵且每个候选物一个协方差
‑‑‑
并且只保留那些具有最大特征值的特征向量(图8)。特征向量描述了在一组模拟分子结构中观察到的最主要、第二主要、第三主要、第n主要的运动。如果候选物像参考表位一样移动,则其特征向量将类似于参考目标(表位)的特征向量。特征向量的相似性对应于它们的分量(以每个ca原子为中心的3d向量)对齐
‑‑‑
指向相同的方向(图7d-7g)。候选物与参考目标特征向量之间的这种相似性是使用两个特征向量的内积计算的。如果两个特征向量相互成90度,则内积值为0,或者如果两个特征向量正好指向同一方向,则内积值为1。
[0403]
由于特征向量的排序是基于它们的特征值,并且由于分子动力学模拟对两个不同分子的潜在能量景观进行取样的随机性,特征值在那些不同分子之间可能不一定相同,需要多个不同等级的特征向量之间的内积(例如候选物的特征向量1乘参考目标的特征向量2、3、4等)。此外,不希望受任何理论束缚,分子运动是复杂的并且可能涉及不止一种(或不止几种)主要/主运动模式。
[0404]
为了解决这两个挑战,计算了候选物和参考目标中的所有特征向量对之间的内积。这产生了一个内积矩阵,其维数由所分析的特征向量的数量决定-对于10个特征向量,内积矩阵为10
×
10。通过计算内积的均方根值,此内积矩阵被提炼为单个值。这是均方根内积(rmsip)。
[0405]
主分量分析(pca)将3l
×
3l维数坐标协方差矩阵(l是原子数)简化为特征向量集φ(参考目标)和ψ(mem),以及特征值λ。集φ含有参考目标的n个特征向量并且集ψ含有mem的n个特征向量ψj,其中特征向量按其关联的特征值在其各自的集中排序。具有最大特征值的特征向量占总坐标协变的最大部分。计算每个和ψj特征向量的内积,以比较参考目标与mem之间的运动的相似性。和ψj特征向量的所有内积组合的均方根呈现工程化肽候选物(mem)与参考目标(rmsip)的运动的总体相似性(图8)。
[0406]
表1显示了5种候选工程化肽相对于vegf参考表位的rmsip结果。这些数据是从使用rosetta设计生成的1000个候选物的总模拟中取样的,所述设计具有候选物相对于参考静态结构rmsd截止值。在1000个候选物中,xtr-1000-t0的rosetta(静态结构)能量最低(越低越有利),但rmsip动态匹配中等。候选物xtr-1000-b1和b2具有最高的动态匹配分数(例如,由rmsip计算的它们的运动与参考目标的运动最匹配)。候选物xtr-1000-w1和w2的动态匹配分数最低,表明在这1000个候选数据集中展示了rmsip动态范围,rmsip范围为0.772-0.545。与vegf参考表位比对的候选物的结构显示在图7b中。
[0407]
表1
[0408]
参考表位qimrikphqgqhige mem变体idmem序列rmsipxtr-1000-t0qqimcikphqgqcigeaeealkitaka0.673xtr-1000-b1sqimcikphqgqhigetsedcdkaaks0.772xtr-1000-b2sqicrikphqgqhcgetsedadkaaks0.766xtr-1000-w1qqimcikphqgqcigeaeevykkrkks0.545xtr-1000-w2qqimcikphqgqcigeaeeyytkakrs0.550
[0409]
实例3:使用工程化肽进行vegf推定表位的程序化体外噬菌体选择
[0410]
在一系列噬菌体淘选程序中使用实例1中描述的三种工程化肽和遵循类似程序开发的另外的第四种工程化肽。这些肽显示在图9中。其中两个肽是正选择分子(umem和smem),且两个是负选择分子(imem2和imem1)。smem肽是高拓扑参考匹配,且umem是低拓扑参考匹配。两个imem肽是零拓扑参考匹配,并作为smem和umem的逆形式包括在内,用于选择由于除所需结合相互作用以外的原因将与smem或umem结合的结合配偶体。使用生物传感器分析对生物素结合肽的分析证实了与贝伐单抗的结合,这是通过候选拓扑结构与参考目标的相似性预测的。
[0411]
octet/生物传感器筛选:使用单循环动力学分析设计在octet red384仪器上评估不同工程化肽的亲和力。肽分别进行评估,并通过生物素连接子固定到生物传感器的链霉亲和素涂布的尖端。剩余的开放链霉亲和素位点用生物胞素封闭。在传感器尖端上洗涤分析物,且记录分析物中的分子与肽的结合。对于此分析,分析物是0.19μm到1.5μm的贝伐单抗连续稀释液。一式两份地运行每一分析。还运行了对照,所述对照仅使用缓冲液(以控制
传感器漂移)和来自人nd血清的纯化igg的单独对照(以控制非特异性igg结合)。
[0412]
设计了七个不同的淘选程序,每个程序包含三轮,每一轮包含正选择步骤和负选择步骤(图11)。每个程序都使用至少一种工程化肽作为选择分子。还包括使用常规方法的常规选择(vegf作为正目标,且bsa作为负目标,针对非特异性结合进行选择)。三轮淘选后,738个克隆被选择用于elisa反应分析。
[0413]
淘选方案从人类初始scfv文库开始,且在溶液中进行淘选,选择分子与生物素结合(但仍在溶液中)。对于每一轮,起始池首先在溶液中与负选择分子结合,且接着将链霉亲和素涂布的底物(例如磁珠)施加至混合物以结合负选择分子。因此,池中与负选择分子结合的任何噬菌体也与链霉亲和素涂布的支撑物结合。去除剩余的溶液,且接着将此流过液带入正选择步骤。流过液与正选择分子组合,使其结合,且接着将链霉亲和素涂布的固体底物施加至混合物。在此步骤中,结合的噬菌体被保留,而剩余的未结合的噬菌体被去除。然后洗脱结合的噬菌体。使用30分钟培养用洗脱的噬菌体转染大肠杆菌,将转染的细胞分裂用于下一代测序和dna分离以进行分析,且接着扩增噬菌体以用于后续的淘选轮次。对于每个淘选程序,在每一轮中首先进行负选择,且然后进行正选择。
[0414]
然后使用elisa分析从七个淘选程序加上常规淘选方法中的每一个获得的候选池对vegf和smem正选择分子(imem校正)的反应,以评估与全长vegf和推定表位smem的结合。这些elisa测试的分析显示在图12a-12b和13a-13h中。这些结果表明,使用工程化肽的体外选择程序不会降低全长vegf结合倾向,并且它们在淘选克隆中产生了推定的表位选择性结合偏倚。还在用于阻断贝伐单抗:vegf结合的交叉阻断elisa分析中测试候选池(与0nm、67pm、670pm和6.7nm的贝伐珠单抗的剂量反应竞争)。这些结果显示在图14a-14i和表2中,且从每个程序获得的确认的交叉阻断克隆的总计数总结在图15中。这些证明了使用工程化肽的可编程体外选择程序能够从交叉阻断贝伐单抗的完整克隆文库中分离克隆,贝伐单抗共享用于导出工程化肽的参考目标表位。
[0415]
表2
[0416]
[0417][0418]
通过sanger测序对表现出交叉阻断行为的克隆进行测序,且发现确认了11个不同的克隆。使用工程化肽从程序化体外选择中获得的那些克隆显示于表3a中。通过用vegf和bsa进行常规选择获得的那些克隆列于表3b中。图17总结了为进一步测试产生的所有fab的结合、交叉阻断、cdr序列和种系使用。图17和图18显示了表3a和3b中所列的fab的elisa结合结果。这些证明可编程体外选择以不同于常规淘选的方式引导抗体cdr环多样性和ig种系使用。
[0419]
表3a
[0420]
[0421][0422]
表3b
[0423][0424]
表4.批准的tx mab
[0425][0426]
使用以下方程式对选择池进行评分:
[0427]
阻断倾向=sum(x-阻断斜率,(smem vegf)-imem),其中x-阻断斜率、smem和vegf为稳健z分数。
[0428]
评分基本原理:如果通过显著的(通过稳健z分数)负斜率观察到阻断反应,则阻断倾向为vegf结合与x-阻断斜率的z分数的组合。阻断倾向总结于图19中,并总结于下表中。
[0429]
表6.从不同程序选择方案(s#)获得的克隆和阻断倾向的总结。
[0430]
[0431][0432]
还评估了与对照(常规)程序相比的不同选择程序的交叉阻断富集,与常规程序(仅使用vegf和bsa作为选择分子)相比,使用所有体外选择程序进行均匀随机取样,至少四个使用工程化肽的程序显示富集,总结在图20中。交叉阻断富集的统计检验是如下的kruskal-wallis检验:
[0433]
1.对来自所有淘选程序的96个克隆进行随机均匀取样,测量交叉阻断活动
[0434]
2.对所有96个克隆的交叉阻断进行排名
[0435]
3.执行kruskal-wallis检验以计算相对于对照的每个程序的平均交叉阻断秩
[0436]
4.x-阻断富集=100%*(程序交叉阻断平均秩-对照平均秩)/(对照平均秩)
[0437]
还对这些克隆进行了下一代测序(ngs),以在基因组水平上获得有关cdr环的信息。图21提供了ngs样本制备的示意图。简而言之,通过在表达载体的恒定部分克隆出单独的重链和轻链序列来制备样本。使用2
×
250配对末端测序运行,并使用例如pyig等工具连接和注释读段。
[0438]
分析序列以确定两个独特的序列是否实际上是不同的抗体,而不是测序错误,称为“克隆性”。还使用了标准化香农评估,如图22中所示。每个程序的每一轮的克隆性的总结在图23中示出。
[0439]
虽然仅使用全长蛋白质(vegf)的经典淘选方法确实聚焦于多样性(程序12),但工程化肽程序化淘选方法聚焦于库多样性的效率至少提高了2倍。图24a-24l是配对频率比较和维数图,分析了第1轮(图24a-24d)、第2轮(图24e-24h)和第3轮(图24i-24l)的不同筛选轮次如何塑造所得选定池的多样性。
[0440]
工程化肽(mem)程序化体外选择分离出不同的抗体克隆型,与第一轮选择中的常规方法相比,种系使用的多样性更高。与全长抗原和umem相比,使用基于smem的体外选择在第1轮产生更多样化的轻链种系使用。基于mem的体外选择程序在第2轮相对于全长抗原产生不同的重链种系使用。体外选择程序中使用的mem的顺序和身份会影响重链种系使用。基于mem的体外选择程序在第2轮相对于全长抗原产生不同的轻链种系使用。体外选择程序中使用的mem的顺序和身份会影响轻链种系使用。基于mem的体外选择程序在第3轮相对于全长抗原产生不同且更多样化的重链种系使用。体外选择程序中使用的mem的顺序和身份会影响重链种系使用和多样性。基于mem的体外选择程序在第3轮相对于全长抗原产生不同且更多样化的轻链种系使用。体外选择程序中使用的mem的顺序和身份会影响轻链种系使用和多样性。
[0441]
不同噬菌体淘选程序如何聚焦于fab命中的总结提供于图25a和25b中。
[0442]
图26中所示的总结每个程序的每轮淘选的表位上(smem)vegf命中频率的图表明工程化肽体外选择方案鉴定了独特的mab命中,证实与vegf结合并交叉阻断贝伐单抗,其中许多的这些命中在常规方法中未鉴定。图27总结了每个程序的每轮淘选的表位外vegf命中频率,证明了常规程序鉴定的mab命中被证实与vegf结合,但不是推定的表位选择性mab命中。图28总结了结合。
[0443]
实例4:使用工程化肽对pd-l1治疗表位进行程序化体外噬菌体选择
[0444]
使用pd-l1上的经鉴定治疗表位参考目标位点,通常按照与实例2中所述类似的方案设计一系列工程化肽(mem),如图29-31d中所总结。使用生物传感器评估了这三种工程化肽smem、nmem(均为正选择分子)和imem(具有逆特征的负选择分子)与两种抗pd-l1阿维鲁单抗和度伐单抗的结合能力(已知两种抗体均与参考目标表位结合),数据显示于图31a-32c中。设计了一系列使用工程化肽的五个不同的淘选程序,以及使用常规选择分子pd-l1和bsa的对照程序,如图33中所示,并用于筛选展示于噬菌体上的初始人类ig scfv格式文库。使用了与在上文实例3中所述相似的淘选方案。对于每个淘选程序,在每一轮中首先进行负选择,且然后进行正选择。
[0445]
使用每个程序选择的所得池对pd-l1和不同工程化肽的elisa反应总结于图34-38中,且比较不同程序的完整elisa反应提供于图39a-39u中。还使用具有所需结合行为的不同组合的不同选择过滤标准分析了选择的池,如图40中所总结。下表2a和2b提供了从elisa结果中选择的不同克隆的总结,所述结果进一步通过交叉阻断分析获得。
[0446]
表2a.从交叉阻断分析的elisa结果中选择的抗pd-l1淘选克隆(完整列表:包括elisa命中和对照)
[0447]
[0448]
表2a.从交叉阻断分析的elisa结果中选择的抗pd-l1淘选克隆(完整列表:包括elisa命中和对照)
[0449][0450]
使用0nm、67pm、670pm和6.7nm的阿维鲁单抗或度伐单抗的剂量反应性pd-l1竞争来分析这些elisa命中,以鉴定34个推定的交叉阻断克隆命中。阻断倾向计算如下:elisa z分数(smem1 smem5 pd-l1-imem) max(阿维鲁单抗阻断z分数,度伐单抗阻断z分数)。结果
的总结提供于下表3中。
[0451]
表3.交叉阻断elisa反应和阻断倾向的总结
[0452][0453]
elisa反应提供于图42a-42f中。对从交叉阻断命中鉴定的23个不同克隆进行测序(通过sanger测序),并在图43中列出。图44中提供了跨淘选程序的交叉阻断命中的不同克隆计数的总结。
[0454]
分析这些结果以确定是否有任何体外选择程序产生交叉阻断pd-l1:阿维鲁单抗/度伐单抗的克隆的随机选择富集。基于elisa和交叉阻断数据,至少两个使用工程化肽的程序显示出富集,所述数据使用与传统程序(仅使用pd-l1和bsa作为选择分子)相比的来自所有体外选择程序的均匀随机取样的克隆。克隆的结果和总结显示在图45a-46中(图45c中的阴影条目来自常规淘选)。分析中使用了以下基本原理:评分基本原理:如果通过显著(通过稳健z分数)负斜率观察到阻断反应,则阻断倾向为pd-l1、mem结合和x-阻断斜率的z分数的组合,其中使用的x-阻断z分数是阿维鲁单抗相对于度伐单抗的最大z分数,因为这些tx mab在表面上具有略微不同的表位。
[0455]
实例5:用于选择工程化肽的机器学习模型
[0456]
使用参考目标,参考目标(序列)的拓扑特征被识别和编码在支架蓝图中(图61,顶部)。支架蓝图可以约束工程化多肽中的氨基酸序列以匹配参考目标中的氨基酸顺序。序列
同源性可以被约束于100%(参考目标中的每个氨基酸对应于蓝图中的一个氨基酸),或者可以允许序列同源性较低,例如,10至90%同源性。可将支架蓝图转换为向量表示(图61,左侧)并用于生成候选多肽,所述多肽的空间相关拓扑特征与从参考目标导出的空间相关拓扑约束的组合重叠以产生工程化肽,每个支架蓝图都基于重叠的评分分配了一个标签(图61,右侧)。
[0457]
机器学习(ml)模型可以在训练数据上进行训练,所述训练数据包括支架蓝图的表示和相应的分数。所述表示可以是例如一维数字向量、二维字母数字数据矩阵、三维标准化数字张量。更具体地,在一些情况下,所述表示是包括插入支架残基位置数的有序列表的向量。可以使用此类表示,因为可以从目标结构推断目标残基的顺序,因此所述表示不需要鉴定目标残基位置的氨基酸一致性。可以使用计算蛋白质建模(例如rosetta remodeler)生成支架蓝图的分数,所述建模确定每个支架蓝图的能量项。然后可以基于计算蛋白质建模生成的能量项来计算分数。
[0458]
ml模型可以是例如提升决策树算法、决策树的集合、极端梯度提升(xgboost)模型、随机森林、支持向量机(svm)等。一旦经过训练,ml模型就会被执行,以从一组支架蓝图生成一组预测分数。如果预测分数高于所需分数,则可以通过计算蛋白质建模来模拟与预测分数对应的支架蓝图,以生成真实分数。可以比较真实分数和预测分数以确定ml模型的再训练。在一些植入中,训练和执行步骤可以如图62中所示进行迭代,直到预测出具有所需分数的最佳/改进的支架蓝图。然后将最佳/改进的支架蓝图转化为工程化肽。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。
