1.本发明属于植物生物技术领域。更具体地,涉及一种高效、广靶向识别几乎所有nnn-pam、更宽的编辑窗口、无靶序列偏好性、可检测的自靶向sgrna效应、以及脱靶效率低的植物腺嘌呤单碱基编辑器pyl-hyabe8e-spry及其构建与应用。
背景技术:
2.基因编辑技术,尤其是由crispr/cas9介导的基因编辑系统,通过在目标位点诱导dna双链断裂(dsb)来实现基因的敲除破坏。然而,作物的重要农艺性状通常由关键基因的点突变或单核苷酸多态性(snp)决定。碱基编辑工具,包括腺嘌呤碱基编辑器(abes)和胞嘧啶碱基编辑器(cbes)(komor et al.,2016,nature,533:420-424;rees and liu.,2018,19:770-788),它们由人工改造的单链dna脱氨酶和cas缺刻酶变体(cas9n,d10a)组成,在不需要供体模板及不造成dsb的条件下实现c-t或a-g的碱基精准替换,将基因编辑技术由破坏基因的“剪刀”转变为改变特定碱基的“校正器”,是植物功能基因组研究和作物的遗传改良的有效工具。
3.与cbe相比,基于tada7.10开发的abe7.10系统具有更高的编辑产物纯度(几乎没有非a-g的转换或插入缺失突变),但其在植物中的编辑效率相对较低(hua et al.,2019,molecular plant,12:1003-1014;molla and yang.,2019,trends biotechnol,37:1121-1142;zeng et al.,2020,plant biotechnology journal,18:1348-1350)。最近的报道称,使用新型的tada8e腺嘌呤脱氨酶,能够提高abe在哺乳动物和水稻中的编辑效率(richter et al.,2020,nature biotechnol,38:883-891;yan et al.,2021,molecular plant,14:722-731),但仍然缺乏全面的靶点序列偏好性系统分析,以及受限于特定的pam靶位点和相对狭窄的编辑活性窗口。目前使用较广的cas9变体仅能识别ngg-pam(spcas9)或ngn-pam(spcas9-ng),可识别的靶位点非常有限。另外,碱基编辑器的编辑活性窗口一般仅存在于目标序列上一个狭窄的范围(m
4-m8,m代表a或c),而对于靠近pam的碱基几乎不能编辑。因此,开发适用于植物的高效、广靶向、编辑窗口拓宽以及无序列偏好性的新型植物腺嘌呤单碱基编辑器。
技术实现要素:
4.本发明所要解决的技术问题在于克服现有技术中存在的植物腺嘌呤单碱基编辑效率低,常用abes靶点选择受限于ngg-和ngn-pam,编辑活性窗口受限在a
4-a8,往往导致目标碱基a不能被成功编辑为g,从而限制了abes在植物中的广泛应用的缺陷和不足,提供一种高效、广靶向识别几乎所有nnn-pam、更宽的编辑窗口、无靶序列偏好性、可检测的自靶向sgrna效应、以及脱靶效率低的植物腺嘌呤单碱基编辑器pyl-hyabe8e-spry及其构建与应用。
5.本发明的第一个目的在于提供一种用于植物基因编辑的spcas9变体融合蛋白tada8e-dbd-spry。
id no.1、seq id no.5所示,均按水稻密码子偏好性进行优化。
23.优选地,编码分别连接脱氨酶tada8e与dna单链结合域dbd,dna单链结合域dbd与spcas9缺刻酶变体spryn的柔性连接序列的核苷酸序列分别如seq id no.6、seq id no.7所示,均按水稻密码子偏好性进行优化。
24.进一步优选地,所述多核苷酸序列如seq id no.15所示,是按bpnls-tada8e-linker 1-dbd-linker 2-spryn-bpnls顺序融合,所有组分bpnls-tada8e、linker 1-dbd和linker 2-spryn-bpnls均按水稻密码子偏好性进行优化。
25.本发明还提供含有上述任一所述多核苷酸序列的质粒载体。
26.本发明还提供上述任一项所述spcas9变体融合蛋白tada8e-dbd-spry或任一所述多核苷酸序列或所述质粒载体在制备植物腺嘌呤碱基编辑器中的应用。
27.一种植物腺嘌呤碱基编辑器,是将编码上述任一项所述spcas9变体融合蛋白tada8e-dbd-spry的多核苷酸序列构建至植物转化载体上得到。
28.优选地,所述植物转化载体为双元载体pylcrispr/cas9pubi-h。
29.本发明还提供所述植物腺嘌呤碱基编辑器的构建方法,先制备得到编码spcas9变体融合蛋白tada8e-dbd-spry的完整融合dna序列,再组装到双元载体pylcrispr/cas9pubi-h的pst i和bamh i之间,获得编辑器pyl-hyabe8e-spry。
30.本发明还提供所述植物腺嘌呤单碱基编辑器在植物基因组单碱基编辑中的应用。
31.优选地,所述植物为水稻。
32.与现有技术相比,本发明具有以下有益效果:
33.本发明提供了一种适用于高效、广靶向、编辑窗口广、无靶点序列偏好性、无非a-g副产物、脱靶效率低、且可以多靶点编辑的spcas9变体融合蛋白tada8e-dbd-spry,并基于此融合蛋白的植物腺嘌呤单碱基编辑系统。所述植物腺嘌呤单碱基编辑系统具有高的a-g的替换效率,能对几乎所有nnn-pam的靶点进行编辑,且无靶点序列偏好性,主要编辑活性窗口在a
3-a
11
之间,存在低的自靶向t-dna的编辑效应,但并无sgrna依赖的脱靶效应。克服了使用脱氨酶tada7.10和cas9n或cas9n-ng的传统的abe7.10编辑器编辑效率低、可选择靶位点少等缺点,更有利于植物基因功能的研究和作物的遗传改良。
附图说明
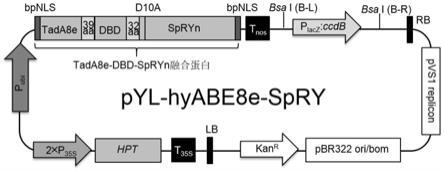
34.图1为植物腺嘌呤碱基编辑器pyl-hyabe8e-spry载体图谱。
35.图2为pyl-hyabe8e-spry、pyl-abe8e-spry和abe7.10-ng编辑器(hua et al.,2019,molecular plant,12:1003-1014)编辑效率对比。(a)具有不同靶位点的pyl-hyabe8e-spry与pyl-abe8e-spry的图谱。(b)pyl-hyabe8e-spry和pyl-abe8e-spry和abe7.10-ng在16个靶点中的编辑效率对比,括号内为突变的植株数比总植株数,靶点中的pam用下划线凸显,能被pyl-hyabe8e-spry编辑的a碱基用加粗字体凸显。
36.图3为pyl-hyabe8e-spry、pyl-abe8e-spry和abe7.10-ng的靶点偏好性和突变类型对比分析。a.从测试的16个靶点中统计ngn、nan、ntn和ncn靶点的平均编辑效率。b.突变类型分析,ho:homozygous mutation(纯合突变);he:heterozygous mutation(杂合突变);bi:bi-allelic mutation(双等位突变)。c.从可编辑的a
1-a
14
窗口区间发生所有突变中分析5’ga,5’aa,5’ca(a为被编辑的碱基)的偏好性;d.从可编辑的a
1-a
14
窗口区间发生所有突
变中分析ag3’,aa3’,at3’,ac3’(a为被编辑的碱基)的偏好性。
37.图4为pyl-hyabe8e-spry、pyl-abe8e-spry和abe7.10-ng活性窗口比较。统计a
1-a
14
编辑活性窗口中各位点的a-g平均编辑效率绘制。
38.图5为pyl-hyabe8e-spry和pyl-abe8e-spry自靶向编辑效率分析。通过高通量测序t0代t-dna中的sgrna表达盒和统计自靶向编辑效率,显示16个靶位点中,大部分靶点能检测到sgrna的自编辑效应,且pyl-hyabe8e-spry与pyl-abe8e-spry的自靶向编辑效率没有显著性差异。
39.图6为pyl-hyabe8e-spry和pyl-abe8e-spry脱靶效率分析。在t1,t2,t3,t4,t5,t8,t12,t14,t15的on-target靶点中选择≤2个碱基不匹配(针对20bp的靶序列)off-target位点用于脱靶效率分析,和on-target靶序列差异的碱基(包含20bp靶序列和3bp的pam序列)用下划线凸显,靶点的pam加粗凸显。用t0转化株基因组dna为模板pcr扩增潜在的脱靶位点做高通量测序分析,结果显示,仅在t1靶点的pyl-abe8e-spry编辑事件中检测到低水平的脱靶效应,而在pyl-hyabe8e-spry的编辑事件中没有检测到脱靶效应。
具体实施方式
40.以下结合说明书附图和具体实施例来进一步说明本发明,但实施例并不对本发明做任何形式的限定。除非特别说明,本发明采用的试剂、方法和设备为本技术领域常规试剂、方法和设备。
41.除非特别说明,以下实施例所用试剂和材料均为市购。
42.实施例1 植物腺嘌呤单碱基编辑器pyl-hyabe8e-spry和pyl-abe8e-spry的构建
43.脱氨酶tada8e、dbd、spryn和bpnls的蛋白序列根据已公布的序列(thuronyi et al.,2019,nature biotechnology,37:1070-1079;richter et al.,2020,nature biotechnology,38:883-891;walton et al.,2020,science,368:290-296;zhang et al.,2020,nature cell biology,22:740-750),按bpnls-tada8e、linker 1-dbd和linker 2-spryn-bpnls三个片段组合,由武汉genecreate公司按照水稻密码子偏好优化核酸序列直接合成。其中,优化后的n端bpnls、tada8e、dbd、spryn、c端bpnls、linker 1、linker 2的核酸序列依次如seq id no.1~7所示,编码氨基酸序列依次如seq id no.8~14所示:
44.seq id no.1(n端bpnls功能组分的dna序列,54bp):
45.aagaggacagccgacggctctgagttcgagtccccgaagaagaagcgcaaggtc
46.seq id no.2(tada8e功能组分的dna序列,498bp):
47.agcgaggtcgagttctcccacgagtactggatgaggcacgccctcacactcgctaagagagctagggacgagagagaggttccagttggcgccgtgctcgtgctcaacaatcgcgttatcggcgaaggctggaatcgcgccattggcctccatgatccaaccgcgcatgccgagattatggcccttagacaaggcggcctcgtgatgcagaactacaggctcatcgacgcgaccctctacgtgaccttcgagccatgcgttatgtgcgctggcgccatgattcactctaggatcggcagagtggtgttcggcgtgcgcaattctaaaagaggcgctgcgggctccctcatgaacgtcctcaattacccgggcatgaaccaccgcgtcgagatcacagagggcatcctcgctgatgagtgcgctgctctcctctgcgacttctacaggatgccacgccaggtgttcaacgcccagaagaaggcccagtcctccatcaat
48.seq id no.3(dna单链结合域dbd的dna序列,342bp):
49.atggccatgcagatgcagctcgaggccaatgccgatacctccgtcgaggaagagtctttcggcccgcag
ccaatttccaggcttgagcagtgcggcatcaacgccaacgacgtgaagaagctcgaagaggccggcttccataccgttgaggccgttgcctacgcgccgaagaaagagctgatcaacatcaagggcatctccgaggccaaggcggacaagattcttgccgaggccgctaagctcgtgccgatgggctttacaaccgccaccgagttccatcagcgccgctctgagatcatccagatcaccaccggctccaaagagctggacaagctccttcaa
50.seq id no.4(spcas9变体spryn功能组分的dna序列,4101bp):
51.gacaagaagtactccatcggcctcgctatcggcaccaacagcgtcggctgggcggtgatcaccgacgagtacaaggtcccgtccaagaagttcaaggtcctgggcaacaccgaccgccactccatcaagaagaacctcatcggcgccctcctcttcgactccggcgagacggcggagcgtacccgcctcaagcgcaccgcccgccgccgctacacccgccgcaagaaccgcatctgctacctccaggagatcttctccaacgagatggcgaaggtcgacgactccttcttccaccgcctcgaggagtccttcctcgtggaggaggacaagaagcacgagcgccaccccatcttcggcaacatcgtcgacgaggtcgcctaccacgagaagtaccccactatctaccaccttcgtaagaagcttgttgactctactgataaggctgatcttcgtctcatctaccttgctctcgctcacatgatcaagttccgtggtcacttccttatcgagggtgaccttaaccctgataactccgacgtggacaagctcttcatccagctcgtccagacctacaaccagctcttcgaggagaaccctatcaacgcttccggtgtcgacgctaaggcgatcctttccgctaggctctccaagtccaggcgtctcgagaacctcatcgcccagctccctggtgagaagaagaacggtcttttcggtaacctcatcgctctctccctcggtctgacccctaacttcaagtccaacttcgacctcgctgaggacgctaagcttcagctctccaaggatacctacgacgatgatctcgacaacctcctcgctcagattggagatcagtacgctgatctcttccttgctgctaagaacctctccgatgctatcctcctttcggatatccttagggttaacactgagatcactaaggctcctctttctgcttccatgatcaagcgctacgacgagcaccaccaggacctcaccctcctcaaggctcttgttcgtcagcagctccccgagaagtacaaggagatcttcttcgaccagtccaagaacggctacgccggttacattgacggtggagctagccaggaggagttctacaagttcatcaagccaatccttgagaagatggatggtactgaggagcttctcgttaagcttaaccgtgaggacctccttaggaagcagaggactttcgataacggctctatccctcaccagatccaccttggtgagcttcacgccatccttcgtaggcaggaggacttctaccctttcctcaaggacaaccgtgagaagatcgagaagatccttactttccgtattccttactacgttggtcctcttgctcgtggtaactcccgtttcgcttggatgactaggaagtccgaggagactatcaccccttggaacttcgaggaggttgttgacaagggtgcttccgcccagtccttcatcgagcgcatgaccaacttcgacaagaacctccccaacgagaaggtcctccccaagcactccctcctctacgagtacttcacggtctacaacgagctcaccaaggtcaagtacgtcaccgagggtatgcgcaagcctgccttcctctccggcgagcagaagaaggctatcgttgacctcctcttcaagaccaaccgcaaggtcaccgtcaagcagctcaaggaggactacttcaagaagatcgagtgcttcgactccgtcgagatcagcggcgttgaggaccgtttcaacgcttctctcggtacctaccacgatctcctcaagatcatcaaggacaaggacttcctcgacaacgaggagaacgaggacatcctcgaggacatcgtcctcactcttactctcttcgaggatagggagatgatcgaggagaggctcaagacttacgctcatctcttcgatgacaaggttatgaagcagctcaagcgtcgccgttacaccggttggggtaggctctcccgcaagctcatcaacggtatcagggataagcagagcggcaagactatcctcgacttcctcaagtctgatggtttcgctaacaggaacttcatgcagctcatccacgatgactctcttaccttcaaggaggatattcagaaggctcaggtgtccggtcagggcgactctctccacgagcacattgctaaccttgctggttcccctgctatcaagaagggcatccttcagactgttaaggttgtcgatgagcttgtcaaggttatgggtcgtcacaagcctgagaacatcgtcatcgagatggctcgtgagaaccagactacccagaagggtcagaagaactcgagggagcgcatgaagaggattgaggagggtatcaaggagcttggttctcagatccttaaggagcaccctgtcgagaacacccagctccagaacgagaagctctacctctactacctccagaacggtagggatatgtacgttgaccaggagctcgacatcaacaggctttctgactacgacgtcgaccacattgttcctcagtctttccttaaggatgactccatcgacaacaaggtcctcacgaggtccgacaagaacaggggtaagtcggacaacgtccct
tccgaggaggttgtcaagaagatgaagaactactggaggcagcttctcaacgctaagctcattacccagaggaagttcgacaacctcacgaaggctgagaggggtggcctttccgagcttgacaaggctggtttcatcaagaggcagcttgttgagacgaggcagattaccaagcacgttgctcagatcctcgattctaggatgaacaccaagtacgacgagaacgacaagctcatccgcgaggtcaaggtgatcaccctcaagtccaagctcgtctccgacttccgcaaggacttccagttctacaaggtccgcgagatcaacaactaccaccacgctcacgatgcttaccttaacgctgtcgttggtaccgctcttatcaagaagtaccctaagcttgagtccgagttcgtctacggtgactacaaggtctacgacgttcgtaagatgatcgccaagtccgagcaggagatcggcaaggccaccgccaagtacttcttctactccaacatcatgaacttcttcaagaccgagatcaccctcgccaacggcgagatccgcaagcgccctcttatcgagacgaacggtgagactggtgagatcgtttgggacaagggtcgcgacttcgctactgttcgcaaggtcctttctatgcctcaggttaacatcgtcaagaagaccgaggtccagaccggtggcttctccaaggagtctatccgtccaaagagaaactcggacaagctcatcgctaggaagaaggattgggaccctaagaagtacggtggtttcctctggcctactgtcgcctactccgtcctcgtggtcgccaaggtggagaagggtaagtcgaagaagctcaagtccgtcaaggagctcctcggcatcaccatcatggagcgctcctccttcgagaagaacccgatcgacttcctcgaggccaagggctacaaggaggtcaagaaggacctcatcatcaagctccccaagtactctcttttcgagctcgagaacggtcgtaagaggatgctggcttccgctaagcagctccagaagggtaacgagcttgctcttccttccaagtacgtgaacttcctctacctcgcctcccactacgagaagctcaagggttcccctgaggataacgagcagaagcagctcttcgtggagcagcacaagcactacctcgacgagatcatcgagcagatctccgagttctccaagcgcgtcatcctcgctgacgctaacctcgacaaggtcctctccgcctacaacaagcaccgcgacaagcccatccgcgagcaggccgagaacatcatccacctcttcacgctcacgaggctcggcgcccctcgtgctttcaagtacttcgacaccaccatcgacccgaagcagtacaggtccaccaaggaggttctcgacgctactctcatccaccagtccatcaccggtctttacgagactcgtatcgacctttcccagcttggtggtgat
52.seq id no.5(c端bpnls功能组分的dna序列,54bp):
53.aagaggacagctgatggctctgagttcgagtccccgaagaagaagcgcaaggtc
54.seq id no.6(linker 1的dna序列,117bp):
55.ccatcttgcaggacaagggccattgccgaggatctcgctgaggatcaagctgaggctaggctcctcgagccagccaaggctcaaccacaaaaggccgctgaagaggtggccgaagag
56.seq id no.7(linker 2的dna序列,96bp):
57.tctggcggctctagcggcggctcatctggatctgagacaccaggcacatccgagtccgctacaccagagtcatcaggcggctcctccggcggctcc
58.seq id no.8(n端bpnls的氨基酸序列,18aa):
59.krtadgsefespkkkrkv
60.seq id no.9(tada8e的氨基酸序列,166aa):
61.sevefsheywmrhaltlakrarderevpvgavlvlnnrvigegwnraiglhdptahaeimalrqgglvmqnyrlidatlyvtfepcvmcagamihsrigrvvfgvrnskrgaagslmnvlnypgmnhrveitegiladecaallcdfyrmprqvfnaqkkaqssin
62.seq id no.10(dna单链结合域dbd的氨基酸序列,114aa):
63.mamqmqleanadtsveeesfgpqpisrleqcginandvkkleeagfhtveavayapkkelinikgiseakadkilaeaaklvpmgfttatefhqrrseiiqittgskeldkllq
64.seq id no.11(spcas9变体spryn的氨基酸序列,1367aa):
65.dkkysiglaigtnsvgwavitdeykvpskkfkvlgntdrhsikknligallfdsgetaertrlkrtarr
rytrrknricylqeifsnemakvddsffhrleesflveedkkherhpifgnivdevayhekyptiyhlrkklvdstdkadlrliylalahmikfrghfliegdlnpdnsdvdklfiqlvqtynqlfeenpinasgvdakailsarlsksrrlenliaqlpgekknglfgnlialslgltpnfksnfdlaedaklqlskdtydddldnllaqigdqyadlflaaknlsdaillsdilrvnteitkaplsasmikrydehhqdltllkalvrqqlpekykeiffdqskngyagyidggasqeefykfikpilekmdgteellvklnredllrkqrtfdngsiphqihlgelhailrrqedfypflkdnrekiekiltfripyyvgplargnsrfawmtrkseetitpwnfeevvdkgasaqsfiermtnfdknlpnekvlpkhsllyeyftvyneltkvkyvtegmrkpaflsgeqkkaivdllfktnrkvtvkqlkedyfkkiecfdsveisgvedrfnaslgtyhdllkiikdkdfldneenediledivltltlfedremieerlktyahlfddkvmkqlkrrrytgwgrlsrklingirdkqsgktildflksdgfanrnfmqlihddsltfkediqkaqvsgqgdslhehianlagspaikkgilqtvkvvdelvkvmgrhkpeniviemarenqttqkgqknsrermkrieegikelgsqilkehpventqlqneklylyylqngrdmyvdqeldinrlsdydvdhivpqsflkddsidnkvltrsdknrgksdnvpseevvkkmknywrqllnaklitqrkfdnltkaergglseldkagfikrqlvetrqitkhvaqildsrmntkydendklirevkvitlksklvsdfrkdfqfykvreinnyhhahdaylnavvgtalikkypklesefvygdykvydvrkmiakseqeigkatakyffysnimnffkteitlangeirkrplietngetgeivwdkgrdfatvrkvlsmpqvnivkktevqtggfskesirpkrnsdkliarkkdwdpkkyggflwptvaysvlvvakvekgkskklksvkellgitimerssfeknpidfleakgykevkkdliiklpkyslfelengrkrmlasakqlqkgnelalpskyvnflylashyeklkgspedneqkqlfveqhkhyldeiieqisefskrviladanldkvlsaynkhrdkpireqaeniihlftltrlgaprafkyfdttidpkqyrstkevldatlihqsitglyetridlsqlggd
66.seq id no.12(c端bpnls功能组分的氨基酸序列,18aa):
67.krtadgsefespkkkrkv
68.seq id no.13(linker 1的氨基酸序列,39aa):
69.pscrtraiaedlaedqaearllepakaqpqkaaeevaee
70.seq id no.14(linker 2的氨基酸序列,32bp):
71.sggssggssgsetpgtsesatpessggssggs;
72.将优化合成的bpnls-tada8e和linker 2-spryn-bpnls通过overlaping pcr分别连接到linker 1-dbd的两侧,形成完整融合的tada8e-dbd-spry,再用gibson组装的方法将其克隆到双元载体pylcrispr/cas9pubi-h(ma et al.,2015,molecular plant,8:1274-1284)的pst i和bamh i之间,获得高效、广靶向编辑器pyl-hyabe8e-spry。此外,将bpnls-tada8e与linker 2-spryn-bpnls通过overlaping pcr形成完整融合的tada8e-spry,并克隆到双元载体pylcrispr/cas9pubi-h的pst i和bamh i之间,获得编辑器pyl-abe8e-spry,作为对照考察dbd在碱基编辑中的作用。以先前hua等公布的编辑器pyl-abe7.10-cas9n-ng(简称abe7.10-ng)(hua et al.,2019,molecular plant,12:1003-1014)作为编辑效率比较的对照。编码tada8e-dbd-spry融合蛋白的完整dna序列和融合蛋白tada8e-dbd-spry的完整氨基酸序列分别如seq id no.15、seq id no.16所示:
73.seq id no.15(编码tada8e-dbd-spry融合蛋白的完整dna序列,5268bp):
74.atgaagaggacagccgacggctctgagttcgagtccccgaagaagaagcgcaaggtcagcgaggtcgagttctcccacgagtactggatgaggcacgccctcacactcgctaagagagctagggacgagagagaggttccagttggcgccgtgctcgtgctcaacaatcgcgttatcggcgaaggctggaatcgcgccattggcctccatgatccaaccgcgcatgccgagattatggcccttagacaaggcggcctcgtgatgcagaactacaggctcatcgacgcgaccctctacgtgaccttcgagccatgcgttatgtgcgctggcgccatgattcactctaggatcggcagagtggtgttcggcgtg
cgcaattctaaaagaggcgctgcgggctccctcatgaacgtcctcaattacccgggcatgaaccaccgcgtcgagatcacagagggcatcctcgctgatgagtgcgctgctctcctctgcgacttctacaggatgccacgccaggtgttcaacgcccagaagaaggcccagtcctccatcaatccatcttgcaggacaagggccattgccgaggatctcgctgaggatcaagctgaggctaggctcctcgagccagccaaggctcaaccacaaaaggccgctgaagaggtggccgaagagatggccatgcagatgcagctcgaggccaatgccgatacctccgtcgaggaagagtctttcggcccgcagccaatttccaggcttgagcagtgcggcatcaacgccaacgacgtgaagaagctcgaagaggccggcttccataccgttgaggccgttgcctacgcgccgaagaaagagctgatcaacatcaagggcatctccgaggccaaggcggacaagattcttgccgaggccgctaagctcgtgccgatgggctttacaaccgccaccgagttccatcagcgccgctctgagatcatccagatcaccaccggctccaaagagctggacaagctccttcaatctggcggctctagcggcggctcatctggatctgagacaccaggcacatccgagtccgctacaccagagtcatcaggcggctcctccggcggctccgacaagaagtactccatcggcctcgctatcggcaccaacagcgtcggctgggcggtgatcaccgacgagtacaaggtcccgtccaagaagttcaaggtcctgggcaacaccgaccgccactccatcaagaagaacctcatcggcgccctcctcttcgactccggcgagacggcggagcgtacccgcctcaagcgcaccgcccgccgccgctacacccgccgcaagaaccgcatctgctacctccaggagatcttctccaacgagatggcgaaggtcgacgactccttcttccaccgcctcgaggagtccttcctcgtggaggaggacaagaagcacgagcgccaccccatcttcggcaacatcgtcgacgaggtcgcctaccacgagaagtaccccactatctaccaccttcgtaagaagcttgttgactctactgataaggctgatcttcgtctcatctaccttgctctcgctcacatgatcaagttccgtggtcacttccttatcgagggtgaccttaaccctgataactccgacgtggacaagctcttcatccagctcgtccagacctacaaccagctcttcgaggagaaccctatcaacgcttccggtgtcgacgctaaggcgatcctttccgctaggctctccaagtccaggcgtctcgagaacctcatcgcccagctccctggtgagaagaagaacggtcttttcggtaacctcatcgctctctccctcggtctgacccctaacttcaagtccaacttcgacctcgctgaggacgctaagcttcagctctccaaggatacctacgacgatgatctcgacaacctcctcgctcagattggagatcagtacgctgatctcttccttgctgctaagaacctctccgatgctatcctcctttcggatatccttagggttaacactgagatcactaaggctcctctttctgcttccatgatcaagcgctacgacgagcaccaccaggacctcaccctcctcaaggctcttgttcgtcagcagctccccgagaagtacaaggagatcttcttcgaccagtccaagaacggctacgccggttacattgacggtggagctagccaggaggagttctacaagttcatcaagccaatccttgagaagatggatggtactgaggagcttctcgttaagcttaaccgtgaggacctccttaggaagcagaggactttcgataacggctctatccctcaccagatccaccttggtgagcttcacgccatccttcgtaggcaggaggacttctaccctttcctcaaggacaaccgtgagaagatcgagaagatccttactttccgtattccttactacgttggtcctcttgctcgtggtaactcccgtttcgcttggatgactaggaagtccgaggagactatcaccccttggaacttcgaggaggttgttgacaagggtgcttccgcccagtccttcatcgagcgcatgaccaacttcgacaagaacctccccaacgagaaggtcctccccaagcactccctcctctacgagtacttcacggtctacaacgagctcaccaaggtcaagtacgtcaccgagggtatgcgcaagcctgccttcctctccggcgagcagaagaaggctatcgttgacctcctcttcaagaccaaccgcaaggtcaccgtcaagcagctcaaggaggactacttcaagaagatcgagtgcttcgactccgtcgagatcagcggcgttgaggaccgtttcaacgcttctctcggtacctaccacgatctcctcaagatcatcaaggacaaggacttcctcgacaacgaggagaacgaggacatcctcgaggacatcgtcctcactcttactctcttcgaggatagggagatgatcgaggagaggctcaagacttacgctcatctcttcgatgacaaggttatgaagcagctcaagcgtcgccgttacaccggttggggtaggctctcccgcaagctcatcaacggtatcagggataagcagagcggcaagactatcctcgacttcctcaagtctgatggtttcgctaacaggaacttcatgcagctcatccacgatgactctcttaccttcaaggaggatattcagaaggctcaggtgtccggtcagggcgactctctccacgagcacattgctaaccttgctggttcccctgctatcaagaagggcatccttcagactgttaaggtt
gtcgatgagcttgtcaaggttatgggtcgtcacaagcctgagaacatcgtcatcgagatggctcgtgagaaccagactacccagaagggtcagaagaactcgagggagcgcatgaagaggattgaggagggtatcaaggagcttggttctcagatccttaaggagcaccctgtcgagaacacccagctccagaacgagaagctctacctctactacctccagaacggtagggatatgtacgttgaccaggagctcgacatcaacaggctttctgactacgacgtcgaccacattgttcctcagtctttccttaaggatgactccatcgacaacaaggtcctcacgaggtccgacaagaacaggggtaagtcggacaacgtcccttccgaggaggttgtcaagaagatgaagaactactggaggcagcttctcaacgctaagctcattacccagaggaagttcgacaacctcacgaaggctgagaggggtggcctttccgagcttgacaaggctggtttcatcaagaggcagcttgttgagacgaggcagattaccaagcacgttgctcagatcctcgattctaggatgaacaccaagtacgacgagaacgacaagctcatccgcgaggtcaaggtgatcaccctcaagtccaagctcgtctccgacttccgcaaggacttccagttctacaaggtccgcgagatcaacaactaccaccacgctcacgatgcttaccttaacgctgtcgttggtaccgctcttatcaagaagtaccctaagcttgagtccgagttcgtctacggtgactacaaggtctacgacgttcgtaagatgatcgccaagtccgagcaggagatcggcaaggccaccgccaagtacttcttctactccaacatcatgaacttcttcaagaccgagatcaccctcgccaacggcgagatccgcaagcgccctcttatcgagacgaacggtgagactggtgagatcgtttgggacaagggtcgcgacttcgctactgttcgcaaggtcctttctatgcctcaggttaacatcgtcaagaagaccgaggtccagaccggtggcttctccaaggagtctatccgtccaaagagaaactcggacaagctcatcgctaggaagaaggattgggaccctaagaagtacggtggtttcctctggcctactgtcgcctactccgtcctcgtggtcgccaaggtggagaagggtaagtcgaagaagctcaagtccgtcaaggagctcctcggcatcaccatcatggagcgctcctccttcgagaagaacccgatcgacttcctcgaggccaagggctacaaggaggtcaagaaggacctcatcatcaagctccccaagtactctcttttcgagctcgagaacggtcgtaagaggatgctggcttccgctaagcagctccagaagggtaacgagcttgctcttccttccaagtacgtgaacttcctctacctcgcctcccactacgagaagctcaagggttcccctgaggataacgagcagaagcagctcttcgtggagcagcacaagcactacctcgacgagatcatcgagcagatctccgagttctccaagcgcgtcatcctcgctgacgctaacctcgacaaggtcctctccgcctacaacaagcaccgcgacaagcccatccgcgagcaggccgagaacatcatccacctcttcacgctcacgaggctcggcgcccctcgtgctttcaagtacttcgacaccaccatcgacccgaagcagtacaggtccaccaaggaggttctcgacgctactctcatccaccagtccatcaccggtctttacgagactcgtatcgacctttcccagcttggtggtgataagaggacagctgatggctctgagttcgagtccccgaagaagaagcgcaaggtctag。
75.seq id no.16(融合蛋白tada8e-dbd-spry的完整氨基酸序列,1755aa):
76.mkrtadgsefespkkkrkvsevefsheywmrhaltlakrarderevpvgavlvlnnrvigegwnraiglhdptahaeimalrqgglvmqnyrlidatlyvtfepcvmcagamihsrigrvvfgvrnskrgaagslmnvlnypgmnhrveitegiladecaallcdfyrmprqvfnaqkkaqssinpscrtraiaedlaedqaearllepakaqpqkaaeevaeemamqmqleanadtsveeesfgpqpisrleqcginandvkkleeagfhtveavayapkkelinikgiseakadkilaeaaklvpmgfttatefhqrrseiiqittgskeldkllqsggssggssgsetpgtsesatpessggssggsdkkysiglaigtnsvgwavitdeykvpskkfkvlgntdrhsikknligallfdsgetaertrlkrtarrrytrrknricylqeifsnemakvddsffhrleesflveedkkherhpifgnivdevayhekyptiyhlrkklvdstdkadlrliylalahmikfrghfliegdlnpdnsdvdklfiqlvqtynqlfeenpinasgvdakailsarlsksrrlenliaqlpgekknglfgnlialslgltpnfksnfdlaedaklqlskdtydddldnllaqigdqyadlflaaknlsdaillsdilrvnteitkaplsasmikrydehhqdltllkalvrqqlpekykeiffdqskngyagyidggasqeefykfikpilekmdgteellvklnredllrkqrtfdngsiphqihlgelhailrrqedfypflkdnrekiekiltfripyyvgplargnsrfawmtrkseetitpwnfeevvdkgasaqsfiermtnfdknlpnekvlpkhsllyeyftvyneltkvkyvtegmr
kpaflsgeqkkaivdllfktnrkvtvkqlkedyfkkiecfdsveisgvedrfnaslgtyhdllkiikdkdfldneenediledivltltlfedremieerlktyahlfddkvmkqlkrrrytgwgrlsrklingirdkqsgktildflksdgfanrnfmqlihddsltfkediqkaqvsgqgdslhehianlagspaikkgilqtvkvvdelvkvmgrhkpeniviemarenqttqkgqknsrermkrieegikelgsqilkehpventqlqneklylyylqngrdmyvdqeldinrlsdydvdhivpqsflkddsidnkvltrsdknrgksdnvpseevvkkmknywrqllnaklitqrkfdnltkaergglseldkagfikrqlvetrqitkhvaqildsrmntkydendklirevkvitlksklvsdfrkdfqfykvreinnyhhahdaylnavvgtalikkypklesefvygdykvydvrkmiakseqeigkatakyffysnimnffkteitlangeirkrplietngetgeivwdkgrdfatvrkvlsmpqvnivkktevqtggfskesirpkrnsdkliarkkdwdpkkyggflwptvaysvlvvakvekgkskklksvkellgitimerssfeknpidfleakgykevkkdliiklpkyslfelengrkrmlasakqlqkgnelalpskyvnflylashyeklkgspedneqkqlfveqhkhyldeiieqisefskrviladanldkvlsaynkhrdkpireqaeniihlftltrlgaprafkyfdttidpkqyrstkevldatlihqsitglyetridlsqlggdkrtadgsefespkkkrkv;
77.具体构建方法如下:
78.构建过程中用到的引物如表1所示:
79.表1 用于pyl-hyabe8e-spry和pyl-abe8e-spry的基础载体改造
[0080][0081][0082]
1)使用f-hytada8e-spry-1/r-hytada8e-spry-1(seq id no.17和seq id no.18)引物,以公司合成的tada8e质粒作为模板,扩增bpnls-tada8e片段。
[0083]
pcr体系(15μl):2
×
phanta max buffer 7.5μl,10mm dntps mix 0.35μl,phanta max polymerase 0.35μl,tada8e 10ng,10μm f-hytada8e-spry-1 0.35μl,10μm r-hytada8e-spry-1 0.35μl,ddh2o补足到15μl。
[0084]
pcr程序:预变性95℃ 2min,28个pcr循环(95℃ 10s,56℃ 15s,72℃ 30s),延伸
no.22)引物,以第一轮扩增的bpnls-tada8e’和linker 2-spryn-bpnls’片段作为模板,扩增bpnls-tada8e-linker 2-spryn-bpnls融合dna片段。
[0101]
pcr体系(15μl):2
×
phanta max buffer 7.5μl,10mm dntps mix 0.35μl,phanta max polymerase 0.35μl,第一轮扩增的bpnls-tada8e’和linker2-spryn-bpnls’片段各取0.1μl,10μm f-hytada8e-spry-1 0.35μl,10μm r-hytada8e-spry-3 0.35μl,ddh2o补足到15μl。
[0102]
pcr程序:预变性95℃ 2min,28个pcr循环(95℃ 10s,56℃ 15s,72℃ 5min),延伸72℃ 6min。
[0103]
(8)用genstar纯化试剂盒,纯化扩增的bpnls-tada8e-linker 1-dbd-linker 2-spryn-bpnls和bpnls-tada8e-linker 2-spryn-bpnls融合dna片段的pcr产物。用pst i和bamh i酶切pylcrispr/cas9pubi-h(ma et al.,2015,molecular plant,8:1274-1284):10x faster digest buffer,pst i 0.5μl,bamh i 0.5μl,pylcrispr/cas9pubi-h 300ng,ddh2o补足到10μl,37℃反应1h,胶回收载体骨架,用于gibson组装反应(neb#e5510s):2x mix 5μl,bpnls-tada8e-linker 1-dbd-linker 2-spryn-bpnls或bpnls-tada8e-linker 2-spryn-bpnls 60ng,胶回收载体骨架90ng,ddh2o补足到10μl,50℃反应50min。分别取1.5μl gibson的连接产物,电激转化大肠杆菌dh10b,在卡那霉素抗性(kana)lb平板上,筛选转化单克隆。并将阳性克隆送测序,从而分别获得pyl-hyabe8e-spry和pyl-abe8e-spry基础载体质粒。
[0104]
实施例2.pyl-hyabe8e-spry具有更高的腺嘌呤碱基编辑效率和广靶向能力
[0105]
参考本发明人团队前期发表的文献(ma et al.,2015,molecular plant,8:1274-1284;ma and liu,2016,current protocols in molecular biology,115:31.6.1-31.6.21;曾栋昌等,2018,中国科学:生命科学,48:783-794),分别构建小核rna基因启动子(osu6a、osu6b和osu3)驱动不同靶点的sgrna表达盒,用“金门组装,golden gate”方式插入pyl-hyabe8e-spry和pyl-abe8e-spry双元载体(ma et al.,2015,molecular plant,8:1274-1284),转化水稻,并对转化体靶点测序,分析pyl-hyabe8e-spry和pyl-abe8e-spry编辑效率,并以pyl-abe7.10-cas9n-ng(hua et al.,2019,molecular plant,12:1003-1014)作为编辑效率比较的对照。
[0106]
1.t1~t16的靶点引物设计
[0107]
为了与先前的abe7.10系统编辑效率进行比较,我们从hua等公布的结果中挑选了4个具有ngn-pam的靶位点(t1~t4)用作评价abe8e系统编辑效率的对照。此外,在基因组中寻找具有nan-,ntn-和ncn-pam的靶位点(t5~t16)。利用我们开发的网上程序crispr-ge网页(http://skl.scau.edu.cn/)(xie et al.,2018,molecular plant,11:720-735),进入引物设计(primerdesign)子程序,primerdesign-v分支程序,选择对应启动子,勾上复选框,并选择method2,点击design,自动生成16个靶点引物gr-t#与u#-t#。
[0108]
2.t1~t16的sgrna表达盒的overlapping pcr拼接
[0109]
按照我们前期发表的文献(ma et al.,2015,molecular plant,8:1274-1284;ma and liu,2016,current protocols in molecular biology,115:31.6.1-31.6.21;曾栋昌等,2018,中国科学:生命科学,48:783-794)操作,通过两轮pcr,获得两侧具有bsa i酶切位点的小rna启动子驱动的sgrna表达盒。
[0110]
第一轮pcr,利用设计的u#-t#/gr-t#引物将靶点序列引入到osu6/osu3启动子下游和sgrna序列的上游。在一个pcr体系中,利用u-f引物(seq id no.25,表2)与gr-t#引物配对,pcr扩增获得含靶点的启动子序列;利用gr-r引物(seq id no.26,表2)与u#-t#引物配对,pcr扩增获得含靶点的sgrna序列。pcr体系(20μl):2
×
phanta max buffer 10.0μl,10mm dntps mix 0.4μl,phanta max polymerase 0.3μl,pylgrna-osu6/3(含启动子和sgrna质粒)3ng,10μm u-f 0.4μl,10μm gr-t#0.2μl,10μm grna-r 0.4μl,10μm u#-t#0.2μl,ddh2o补足到20μl。pcr程序:预变性95℃ 1min,28个pcr循环(95℃ 10s,58℃ 15s,72℃ 20s),延伸72℃ 1min。
[0111]
第二轮pcr,拼接小rna启动子驱动的sgrna表达盒,并在pcr产物两侧加上bsa i酶切位点。t1~t4作为一组(target group 1,tg1),t5、t6、t9、t10、t13、t14作为一组(target group 2,tg2),t7、t8、t11、t12、t15、t16作为一组(target group 3,tg3),分别构建1个4靶点和2个6靶点的sgrna表达盒载体。pcr体系(50μl):2
×
phanta max buffer 25.0μl,10mm dntps mix1.0μl,phanta max polymerase 1.0μl,10
×
稀释上一轮pcr产物1.0μl,4靶点分别使用的引物,10μm的pps-r/pgs-2(t1),pps-2/pgs-3(t2),pps-3/pgs-4(t3),pps-4/pgs-l(t4)各1.0μl;6靶点分别使用的引物,10μm pps-r/pgs-2(t1),pps-2/pgs-3(t2),pps-3/pgs-4(t3),pps-4/pgs-5(t4),pps-5/pgs-6(t5),pps-6/pgs-l(t6)各1.0μl(表2,3),ddh2o补足到50μl,pcr程序同上述第一轮pcr。使用genstar纯化试剂盒纯化第二轮pcr产物。
[0112]
3.含不同靶点的sgrna表达盒敲除载体的构建
[0113]
使用基于bsa i酶切和连接的“金门”克隆方法,以“边切边连接”方式(ma and liu,2016,current protocols in molecular biology,115:31.6.1-31.6.21;曾栋昌等,2018,中国科学:生命科学,48:783-794),分别组装tg1,tg2和tg3;把tg1和tg2两组小核rna启动子驱动的sgrna表达盒分别克隆到双元载体pyl-hyabe8e-spry上;把tg1和tg3两组小核rna启动子驱动的sgrna表达盒分别克隆到双元载体pyl-abe8e-spry上(图2a)。15μl反应体系:10
×
cutsmart buffer 1.5μl,10mm atp 1.5μl,pyl-hyabe8e-spry或pyl-abe8e-spry质粒80~100ng,纯化后的sgrna表达盒10~15ng,bsa i-hf 10u,t4 dna ligase 35u,ddh2o补足到15μl。用pcr仪变温循环进行酶切连接反应:37℃ 10min,接着10-12循环(37℃ 5min,10℃ 3min,20℃ 5min);最后37℃ 3min。透析连接产物后,电激转化进入dh10b细胞,在卡拉抗性(kan)lb板上筛选,用引物对sp-l1/sp-r(seq id no.43和seq id no.44,表2),根据文献(ma and liu,2016,current protocols in molecular biology,115:31.6.1-31.6.21;曾栋昌等,2018,中国科学:生命科学,48:783-794),做菌落pcr,筛选阳性克隆,最终用引物sp-l1测序确定。
[0114]
表2 构建多个sgrna表达盒的通用引物
[0115][0116][0117]
注1:bsa i酶切末端设计成非回文序列,可产生高效的连接(golden gate ligation)。
[0118]
注2:如果连接多于8个sgrna表达盒,需要自行设计更多组pgs和pps引物,每组含有互补的非回文bsa i酶切末端。
[0119]
表3 组装不同数量sgrna表达盒的第二轮pcr引物组合
[0120][0121]
4.pyl-hyabe8e-spry具有较高的编辑效率。
[0122]
利用农杆菌介导的水稻愈伤转化,把上述含有tg1和tg2两组不同小rna启动子驱动的sgrna的pyl-hyabe8e-spry载体以及含有tg1和tg3两组不同小rna启动子驱动的sgrna的pyl-abe8e-spry转化水稻愈伤(图2a),提取t0代转化植株的叶片dna作为模板,扩增单碱基编辑靶位点的dna片段,回收后进行高通量测序,通过将测序结果和参考序列比对,统计、比较pyl-abe7.10-ng、pyl-abe8e-spry和pyl-hyabe8e-spry的编辑效率。结果表明pyl-hyabe8e-spry的平均编辑效率最高(75.2%),其中在t1、t5、t9、t14靶点的编辑效率高达100.0%(图2b)。而pyl-abe8e-spry的平均编辑效率为36.6%,pyl-abe7.10-ng则只有4.8%(图2b)。
[0123]
5.pyl-abe8e-spry和pyl-hyabe8e-spry显示出几乎无pam限制的编辑活性,突变类型丰富且无靶点序列偏好性。
[0124]
传统的spcas9和cas9-ng只能分别识别具有ngg-和ngn-pam的靶点,其可编辑范围有限,为了探究spry介导的单碱基编辑器pyl-abe8e-spry和pyl-hyabe8e-spry是否能在nnn-pam靶点中实现高效编辑,我们从16个靶点中统计了pyl-abe8e-spry和pyl-hyabe8e-spry在ngn-、nan-、ntn-和ncn-pam靶点的平均编辑效率(pyl-abe7.10-ng只统计ngn-pam靶点),结果显示,pyl-abe8e-spry和pyl-hyabe8e-spry不但在ngn-pam靶点中显示出高于pyl-abe7.10-ng的编辑效率,在nan-、ntn-和ncn-pam靶点中也具有较高的编辑效率,且pyl-hyabe8e-spry的效率更高(图3a)。t0代的测序结果显示,pyl-abe8e-spry和pyl-hyabe8e-spry的编辑产物主要以纯合和双等位突变为主,突变类型较丰富,而pyl-abe7.10-ng只有杂合突变(图3b)。此外,我们统计了所有测试的载体在a
1-a
14
活性窗口上5’ga、5’aa、5’ca和ag3’、aa3’、at3’、ac3’基序的平均编辑效率,并分析了它们的碱基偏好。从统计结果显示,pyl-abe8e-spry和pyl-hyabe8e-spry在上述靶点序列中的编辑效率没有显著性差异,均具有较高的编辑效率(图3c,d)。
[0125]
6.pyl-hyabe8e-spry有更宽的编辑窗口
[0126]
根据t0代的测序结果,我们统计了t1~t16中16个靶点在a
1-a
14
每个位点中a-g的平均编辑效率,绘制成编辑活性窗口图。结果显示,pyl-hyabe8e-spry(主要在a
3-a
13
,≥
10%)具有比pyl-abe8e-spry(主要在a
4-a9,≥10%)更宽的编辑窗口(图4)。更宽编辑窗口的脱氨酶搭配广靶向的cas蛋白更有助于单碱基编辑技术在动植物基因组编辑中的运用。
[0127]
7.pyl-abe8e-spry和pyl-hyabe8e-spry具有一定的自靶向sgrna的活性
[0128]
由于spry可以识别几乎所有nnn-pam,因此spry除了靶向基因组靶点,同时还可能靶向t-dna自身的sgrna的靶点(ren et al.,2021,nature plants 7:25-33)。为了探究本研究的pyl-abe8e-spry和pyl-hyabe8e-spry自靶向t-dna自身sgrna靶位点的效率,直接扩增t0代转化苗的sgrna片段并进行高通量测序,结果显示pyl-abe8e-spry和pyl-hyabe8e-spry均具有一定的自靶向sgrna活性,二者效率并没有显著性差异(平均分别为18.8%和20.5%)(图5)。
[0129]
8.pyl-hyabe8e-spry未检测到脱靶效应
[0130]
利用我们课题组开发的网上程序crispr-ge网页(http://skl.scau.edu.cn/)(xie et al.,2018,molecular plant,11:720-735),进入引物设计(off-target)子程序,输入t1~t5、t8、t12、t14、t15的靶点序列,从这9个靶点中选择靶序列(pam的5’端20bp序列)具有≤2个碱基不匹配的脱靶潜在位点用于脱靶效率分析(图6)。用t0转化株基因组dna为模板,通过pcr扩增上述潜在的脱靶位点进行高通量测序分析。结果显示,pyl-hyabe8e-spry在所有1或2个碱基不匹配的潜在脱靶位点未发现脱靶效应,而pyl-abe8e-spry在t1-off 1中检测到低(2.4%)的脱靶效应(图6)。
[0131]
综上所述,本发明开发的新型高效植物广靶向腺嘌呤碱基编辑器pyl-hyabe8e-spry比先前的abe编辑系统(如目前广泛使用的pyl-abe7.10-ng编辑器),具有更高的效率、几乎能靶向所有nnn-pam位点、编辑窗口广、无靶点序列偏好性和脱靶效率低等优点,能够广泛的用于作物基因功能筛选、大规模饱和突变、编辑调控元件、引入提前终止密码子或进行可变剪接等操作。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。