用于检测奶牛产奶性状的snps标记及应用
技术领域
1.本发明涉及分子遗传育种技术领域,具体涉及用于检测奶牛产奶性状的snps标记及应用。
背景技术:
2.奶业是我国畜牧业的重要组成部分,是国家农业发达程度的重要标志。奶业的发展对促进畜牧业产业升级,优化农业产业结构,增加农民收入,提高国民身体素质,都具有重要意义。近年来我国奶业规模化牧场发展迅速,尤其奶牛单产提升比较快,随着乳品市场的发展,乳品加工企业在鲜奶收购过程中按质论价,如果只重视产奶量,而忽视乳成分,会对奶牛场经济效益产生很大的影响,乳品质主要包括乳蛋白率、乳脂率等,提升乳品质量关系着国计民生,也是牧场的生命线。
3.当前奶制品行业急需降本提质增效,不断满足人民群众对高端营养乳制品的需求,因此延伸发展特色高端原奶产品,培育新的利润增长点显得尤为必要。随着分子生物学技术的快速发展,也促进了分子育种技术的应用,因此通过基因手段进行奶牛优质育种成为主流育种技术。
技术实现要素:
4.本发明为了加快具有优质产奶性状的奶牛育种,发现了23个与奶牛优奶性状关联的23个奶牛基因snp位点,所述产奶性状为产奶量和/或乳脂量和/或乳蛋白量;
5.该23个snp位点如下:
6.所述23个snp位点为snp位点5:g.55955109c》t、snp位点5:g.55956175c》t、snp位点19:g.20146771c》t、snp位点19:g.20148799c》t、snp位点5:g.27838883c》t、snp位点14:g.611019g》a、snp位点14:g.611020c》a、snp位点14:g.1112736g》a、snp位点20:g.31888449a》t、snp位点8:g.81002473t》c、snp位点17:g.63962768g》a、snp位点17:g.63878254t》c、snp位点17:g.63911823t》c、snp位点29:g.26500312c》t、snp位点29:g.26500510c》t、snp位点29:g.26500517g》c、snp位点23:g.10587144c》t、snp位点23:g.10588840c》g、snp位点23:g.10589485c》t、snp位点23:g.10591575t》c、snp位点23:g.10591588t》c、snp位点19:g.50784393t》c、snp位点20:g.11480078t》c。
7.其中,该23个snp位点在鉴定或辅助鉴定奶牛产奶性状中可作为检测靶标或作为基因型鉴定物质。
8.本发明还提供一种鉴定或辅助鉴定奶牛产奶性状的方法,包括如下步骤:检测供式奶牛基因上述23个snp位点中至少一个的基因型,鉴定snp位点基因型是否为优势基因型;所述优势基因型为该snp位点所有基因型中对应产奶性状最优的基因型;所述产奶性状为产奶量和/或乳脂量和/或乳蛋白量。以及该方法在奶牛筛选或奶牛育种中的应用。
9.本发明还提供引物对1-22中的任一一种或引物组合;
10.1-22个引物对包括正向引物1f-22f、反向引物1r-22r,每个引物对包括与其序号
对应的正向引物与反向引物,所述正向引物1f-22f如序列1-22所示的单链dna分子或将序1-22删除或增加或改变一个或几个核苷酸,且与序列1-22具有相同功能的核苷酸;所述反向引物1r-22r如序列表23-44所示,的单链dna分子或将序23-44删除或增加或改变一个或几个核苷酸,且与序列23-44具有相同功能的核苷酸。
11.本发明还提供用于鉴定奶牛产奶性状的基因探针,所述探针为根据权利引物对1-22中任一一种或引物组合制备。
12.本发明还提供引物对1-22中任一一种或引物组合或上述探针的应用,为如下(a)-(f)中任一种:
13.(a)鉴定或辅助鉴定奶牛产奶性状;
14.(b)奶牛筛选;
15.(c)奶牛育种;
16.(d)制备用于鉴定或辅助鉴定奶牛产奶性状的试剂盒;
17.(e)制备奶牛筛选的试剂盒;
18.(f)制备奶牛育种的试剂盒。
19.本发明的有益效果为:本发明发现并验证了奶牛基因中与产奶性状关联的23个snp位点,为奶牛育种提供了有效的鉴定方法。
附图说明
20.下面结合附图和具体实施方式对本发明作进一步详细的说明。
21.图1是本发明实施例四rs523864914、rs208784982、rs134128952、rs135863798、rs382372600、rs109234250/rs109326954的分型散点图;
22.图2是本发明实施例四rs137492431、rs385640152、rs382234923、rs209975092、rs133967247、rs110819816的分型散点图;
23.图3是本发明实施例四rs208190614、rs43142495、rs211556205、rs442346530、rs210197882、rs110284249的分型散点图;
24.图4是本发明实施例四rs133659402、rs110283961、rs41919999、rs42590258的分型散点图;
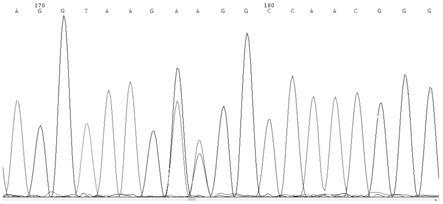
25.图5是本发明rs109234250/rs109326954测序图。
具体实施方式
26.下面结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述。在下面的描述中阐述了很多具体细节以便于充分理解本发明,但是本发明还可以采用其他不同于在此描述的其它方式来实施,本领域技术人员可以在不违背本发明内涵的情况下做类似推广,因此本发明不受下面公开的具体实施例的限制。
27.下述实施例中的产奶量为个体305天产奶量指自母牛产犊第一天开始到第305天为止的产奶总量。当实际挤奶天数不足305天时,以实际乳脂量作为305天乳脂量;当实际挤奶天数超出305天,306天以后的乳脂量不计在内。
28.下述实施例中的乳脂量指的是305天乳脂量,305天乳脂量=乳脂率
×
305天产奶量,乳脂率由每月dhi(dhi,dairy herd improvement)测定所得,通过同一泌乳期内3次以
上dhi数据绘制泌乳曲线计算可得该泌乳期平均乳脂率;
29.下述实施例中的乳蛋白量指的是305天乳蛋白量,305天乳蛋白量=乳蛋白率
×
305天产奶量,乳蛋白率由每月dhi(dhi,dairy herd improvement)测定所得,通过同一泌乳期内3次以上dhi数据绘制泌乳曲线计算可得该泌乳期平均乳蛋白率。
30.上述产奶量、乳脂率、乳脂量、乳蛋白率、乳蛋白量均为第一泌乳期统计的结果。
31.实施例1、前期组学研究
32.本发明以3头中国荷斯坦牛三个泌乳阶段(干奶期,泌乳初期,泌乳高峰期)的肝脏组织为试验材料,对其进行rna-seq和small rna-seq测序分析,整合rna-seq和small rna-seq测序结果,根据cerna调控机制,构建三个比较组lncrna-mirna-mrna互作网络图(competing endogenous rnas,cernas)。筛选出12个基因,分别是:ddit、rpl23a、nr4a、dgat、eef1d、ghr、fbp、acacb、saa、cdkn1a、fasn、pik3r。
33.实施例2、基因多态性检测
34.一、选择北京地区共26头中国荷斯坦公牛作为基因多态性检测的试验群体,将26头中国荷斯坦公牛随机均分为两组,利用核酸分析仪准确测定其冻精基因组dna的浓度,并将这些dna均稀释成浓度为50ng/μl,等量混合成两个池dna,作为模板进行pcr扩增。
35.二、根据牛ddit、rpl23a、nr4a、dgat、eef1d、ghr、fbp、acacb、saa3.2、cdkn1a、fasn、pik3r基因序列设计引物。
36.三、以步骤一得到的池dna为模板,分别以步骤二各引物进行pcr扩增,得到pcr扩增产物。
37.四、将pcr扩增产物进行测序。
38.测序结果为ddit3基因上发现了2个snp位点、rpl23a基因上发现了2个snp位点、nr4a1基因上发现了1个snp位点、dgat1基因上发现了2个snp位点、eef1d基因上发现了1个snp位点、ghr基因上发现了1个snp位点、fbp2基因上发现了1个snp位点、acacb基因上发现了3个snp位点、saa3.2基因上发现了3个snp位点、cdkn1a基因上发现了5个snp位点、fasn基因上发现了1个snp位点、pik3r1基因上发现了1个snp位点,具体如表1所示。
39.表1奶牛乳成分snp位点(23个)信息表
40.序号snp idrs.number染色体号snp位置1ddit3-5:g.55955109c》trs523864914chr5559551092ddit3-5:g.55956175c》trs208784982chr5559561753rpl23a-19:g.20146771c》trs134128952chr19201467714rpl23a-19:g.20148799c》trs135863798chr19201487995nr4a1-5:g.27838883c》trs382372600chr5278388836dgat1-14:g.611019g》ars109234250chr146110197dgat1-14:g.611020c》ars109326954chr146110208eef1d-14:g.1112736g》ars137492431chr1411127369ghr-20:g.31888449a》trs385640152chr203188844910fbp2-8:g.81002473t》crs382234923chr88100247311acacb-17:g.63962768g》ars209975092chr176396276812acacb-17:g.63878254t》crs133967247chr1763878254
13acacb-17:g.63911823t》crs110819816chr176391182314saa3.2-29:g.26500312c》trs208190614chr292650031215saa3.2-29:g.26500510c》trs43142495chr292650051016saa3.2-29:g.26500517g》crs211556205chr292650051717cdkn1a-23:g.10587144c》trs442346530chr231058714418cdkn1a-23:g.10588840c》grs210197882chr231058884019cdkn1a-23:g.10589485c》trs110284249chr231058948520cdkn1a-23:g.10591575t》crs133659402chr231059157521cdkn1a-23:g.10591588t》crs110283961chr231059158822fasn-19:g.50784393t》crs41919999chr195078439323pik3r1-20:g.11480078t》crs42590258chr2011480078
41.注:snp id为snp所在基因名称与snp名称组成。
42.五、一致性验证
43.通过对26个样本的直接sanger测序进行检测,发现rs109234250/rs109326954为联合突变,rs109234250/rs109326954对应测序图如图5所示,rs编号对应的snp位点见表1。检测出的各个snp位点对应的引物如表2所示;
44.表2不同snp位点对应引物以及对应荧光标记
45.[0046][0047]
注:f为正向引物,r为反向引物,fam、hex为荧光标记
[0048]
进一步地,使用表2中各个荧光标记引物对26个样本采用微流控imap平台进行pcr扩增,根据荧光信号的散点图,确定测定的个体基因型。检测23个位点,其基因型与26个参考样本测序的基因型匹配。
[0049]
pcr反应体系:
[0050][0051]
其中,primer mix为“f”、“r”引物各12μm以及通用引物30μm,各snp位点对应通用引物如表3所示。
[0052]
pcr反应条件:
[0053][0054]
表3各snp位点对应通用引物
[0055][0056]
[0057]
实施例3、关联分析
[0058]
一.获得供试群体
[0059]
供试群体由1095头奶牛组成
[0060]
二.构建文库
[0061]
通过dna试剂盒提取供试群体中个体的基因组dna并分别标记保存。
[0062]
三.获得群体基因分型
[0063]
使用表2中各个荧光标记引物以及通用引物对1095个样本采用微流控i map平台进行pcr扩增,根据荧光信号的散点图,确定测定个体基因型;
[0064]
pcr扩增体系以及pcr条件参考实施例二。
[0065]
四、检测产奶性状
[0066]
供试群体中的每头牛,分别进行产奶性状检测。
[0067]
产奶性状包括如下五个指标:产奶量、乳脂量、乳脂率、乳蛋白量和乳蛋白率。
[0068]
每个个体的记录依次包括牛只个体号、父号、母号、祖父号、祖母号、外祖父号、外祖母号、出生日期、泌乳期、产犊日期、产奶量、乳脂率和乳蛋白率13项信息。
[0069]
乳脂量=乳脂率
×
产奶量;
[0070]
乳蛋白量=乳蛋白率
×
产奶量。
[0071]
五、单个snp位点与性状的关联分析模型
[0072]
采用sas 9.2软件中的mixed过程对产奶性状的五个指标和基因型进行关联分析。关联分析采用动物模型,具体模型如下:
[0073]
y=μ hys b
×
m g a e
[0074]
其中,y:产奶性状(个体产奶量、乳脂量、乳脂率、乳蛋白量或乳蛋白率)观察值,μ:总体均值,hys:场年季效应,b:协变量m的回归系数,m:产犊月龄效应,g:基因型效应,a:个体随机加性遗传效应,e:随机残差效应。
[0075]
关联分析统计结果如表4所示。
[0076]
表4关联分析结果
[0077]
[0078]
[0079][0080]
注:同列数据肩标不同小写字母表示差异显著(p《0.05),不同大写字母表示差异极显著(p《0.01);*表示差异显著关联(p《0.05);**表示差异极显著关联(p《0.01);本数据中各个基因型仅代表探测到荧光信号的样本试验数据,未记录部分为未探测到荧光信号样本,“》”为优势基因,例如ddit3-5:g.55955109c》t中c相较t为优势,或例如acacb-17:g.63962768g》a,其基因型为cc、ct、tt,则a与t对应,c与g对应,则g或c相较a或t优势。
[0081]
经关联分析得出不同snp位点对应不同基因型与乳质性状之间的联系,结合表4可以根据这些snp位点的基因型对单个奶牛个体进行产奶性状分析,并判断那种基因型具有优势,例如分子标记g.20146771c》t与产奶量、乳脂量、乳脂率和乳蛋白量均达到极显著(p<0.01),该标记与乳蛋白率关联不显著(p>0.05);分子标记dgat1-14:g.611020c》a与分子标记dgat1-14:g.611019g》a对应snp位点为联合突变,其均与产奶量、乳脂率和乳蛋白量达到极显著(p<0.01),不与乳脂量、乳蛋白率关联不显著(p>0.05)。
[0082]
实施例4
[0083]
利用本发明提供的引物制备微流控芯片118个奶牛样本进行测试,检测分析,具体见表5以及图1-图4,能证明本发明提供的技术能够良好的检测出上述样本关于这23个高乳位点的分型。
[0084]
表5 118个样本乳成分位点验证统计表
[0085][0086]
显然,所描述的实施例仅仅是本发明的一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。
