病变dna识别方法、装置、电子设备及存储介质
技术领域
1.本发明涉及生物信息技术和神经网络应用技术领域,尤其涉及一种病变dna识别方法、装置、电子设备及存储介质。
背景技术:
2.血浆、尿液和脑脊液等体液中的无细胞dna(cfdna)主要是由凋亡或坏死的细胞释放到体液中的降解dna片段。在组织病变和癌症早期,当患者没有明显的临床症状时,病变细胞或癌细胞中的dna状态就已经发生了变化并会释放到体液中,因此在癌症或者各种疾病患者的体液中可以检测到微量的循环病变dna(ctdna)。随着高通量测序技术的发展,通过在大量cfdna测序数据中识别微量的病变信号ctdna正在成为疾病诊断的新型液体活检方法。
3.但现有的技术手段对病变dna序列的识别能力较差,因此,本发明有必要提供一种基于神经网络的病变dna识别方法。
技术实现要素:
4.本发明提供一种病变dna识别方法、装置、电子设备及存储介质,用以解决现有技术中对病变dna序列的识别能力较差、要求大量标注数据的问题。
5.第一方面,本发明实施例提供一种病变dna识别方法,所述方法包括:
6.获取待测dna序列并对所述待测dna序列进行预处理以得到多个dna序列片段;
7.针对每个dna序列片段,将其输入已训练的第一神经网络模型以输出与该dna序列片段对应的第一特征向量,所述第一特征向量包含所述dna序列片段中的不同部分之间的自相关信息;
8.针对每个第一特征向量,将其输入已训练的第二神经网络模型以输出与该第一特征向量对应的第二特征向量,并将该第一特征向量和与其对应的第二特征向量相加,以得到合并特征向量,其中,所述第二特征向量包含所述第一特征向量中的不同部分之间的长短期依赖关系信息;
9.将每个所述合并特征向量输入已训练的分类器以得到分类结果,所述分类结果指示输入的所述待测dna序列是否为病变dna。
10.在本发明的一个实施例中,所述针对每个dna序列片段,将其输入已训练的第一神经网络模型以输出与该dna序列片段对应的第一特征向量包括:
11.对该dna序列片段进行嵌入编码处理以得到具有预设的固定维度的第一编码向量、第二编码向量、以及第三编码向量,所述第一编码向量包含该dna序列片段的内容编码信息,所述第二编码向量包含该dna序列片段的位置编码信息,所述第三编码向量包含分段编码信息;
12.将所述第一编码向量、第二编码向量、以及第三编码向量相加以得到合并编码向量;
13.对所述合并编码向量进行自注意力处理以得到对应的自注意力向量;
14.对所述自注意力向量进行归一化以及残差处理以得到该dna序列片段对应的所述第一特征向量。
15.在本发明的一个实施例中,所述针对每个第一特征向量,将其输入已训练的第二神经网络模型以输出与该第一特征向量对应的第二特征向量包括:
16.将该第一特征向量作为初始向量输入所述已训练的第二神经网络模型;
17.所述已训练的第二神经网络模型中的每个神经单元执行如下操作:
18.对接收到的所述初始向量或者上一级神经单元输出的向量按照预设规则进行过滤处理,以丢弃部分元素;
19.对经过滤的向量进行增补处理,以按照预设规则挑选本单元中存储的信息替换所丢弃的元素;
20.基于经增补的向量确定该神经单元的输出向量;
21.其中,所述已训练的第二神经网络模型中的最后一级神经单元输出所述第二特征向量。
22.在本发明的一个实施例中,所述方法还包括:
23.在获取待测dna序列之前对所述第一神经网络模型、所述第二神经网络模型以及所述分类器按照如下方式进行训练:
24.收集用于训练的数据集,所述数据集包括已标注的病变dna序列和非病变dna序列;
25.将所述数据集按照预设比例分为训练集和测试集,并分别利用所述训练集和所述测试集对所述第一神经网络模型、所述第二神经网络模型和所述分类器进行训练和测试;
26.基于测试结果调整所述第一神经网络模型、所述第二神经网络模型和所述分类器的参数,直至达到预设的精度要求,以得到所述已训练的第一神经网络模型、第二神经网络模型以及分类器。
27.在本发明的一个实施例中,在所述收集用于训练的数据集之前,所述方法还包括:
28.获取预设dna序列,所述预设dna序列为已公开的全基因组dna序列;
29.对所述预设dna序列进行预处理后得到多个dna序列片段;
30.利用所述dna序列片段对所述第一神经网络模型进行预训练。
31.在本发明的一个实施例中,所述对所述预设dna序列进行预处理后得到多个dna序列片段包括:
32.将所述预设dna序列中含有未知碱基的片段删除;以及
33.将删除所述未知碱基后得到的dna序列按照预设长度切分成所述多个的dna序列片段。
34.在本发明的一个实施例中,所述利用所述dna序列片段对所述第一神经网络模型进行预训练包括:
35.在对该dna序列片段进行嵌入编码处理之前,对该dna序列片段中的一部分碱基进行遮蔽处理,并且针对每个被遮蔽的碱基,基于该碱基的上下游序列预测该被遮蔽的碱基位置处的碱基,并用预测出的碱基替代所述被遮蔽的碱基。
36.在本发明的一个实施例中,所述对所述第一神经网络模型、所述第二神经网络模
型以及所述分类器进行训练还包括:
37.在训练所述第一神经网络模型、所述第二神经网络模型和所述分类器的过程中,通过梯度下降法和优化交叉熵损失函数训练所述第一神经网络模型、所述第二神经网络模型以及所述分类器,直至达到所述预设的精度要求。
38.在本发明的一个实施例中,所述得到所述已训练的第一神经网络模型、第二神经网络模型以及分类器之后,所述方法还包括:
39.根据预设评价指标构建目标函数对所述第一神经网络模型、所述第二神经网络模型和所述分类器的训练结果进行评价;
40.其中,所述与预设评价指标包括下列中的任意组合:精确率、召回率以及准确率。
41.在本发明的一个实施例中,根据下列各式计算所述精确率、召回率以及准确率:
42.精确率:tp/(tp fp);
43.召回率:tp/(tp fn);
44.准确率:(tp tn)/(tp fp fn tn);
45.其中,fn表示被识别为非病变dna,但是事实上是病变dna;fp表示被识别为包含病变dna,但事实上非病变dna;tn表示被识别为非病变dna片段,事实上也是非病变dna;tp表示被识别为病变dna,事实上也是病变dna。
46.在本发明的一个实施例中,所述第一神经网络模型是基于转换器的双向编码表征模型,所述第二网络模型是长短期记忆网络模型。
47.在本发明的一个实施例中,所述基于转换器的双向编码表征模型是基于多头自注意力机制的神经网络模型,并且其具有12层网络结构,以及其隐藏层具有768维度。
48.本发明实施例还提供一种病变dna识别装置,所述装置包括:
49.预处理模块,用于获取待测dna序列并对所述待测dna序列进行预处理以得到多个dna序列片段;
50.第一特征向量生成模块,用于针对每个dna序列片段,将其输入已训练的第一神经网络模型以输出与该dna序列片段对应的第一特征向量,所述第一特征向量包含所述dna序列片段中的不同部分之间的自相关信息;
51.第二特征向量生成模块,用于针对每个第一特征向量,将其输入已训练的第二神经网络模型以输出与该第一特征向量对应的第二特征向量,并将该第一特征向量和与其对应的第二特征向量相加,以得到合并特征向量,其中,所述第二特征向量包含所述第一特征向量中的不同部分之间的长短期依赖关系信息;
52.分类模块,用于将每个所述合并特征向量输入已训练的分类器以得到分类结果,所述分类结果指示输入的所述待测dna序列是否为病变dna。
53.在本发明的一个实施例中,所述装置还包括:
54.训练模块,所述训练模块用于:
55.收集用于训练的数据集,所述数据集包括已标注的病变dna序列和非病变dna序列;
56.将所述数据集按照预设比例分为训练集和测试集,并分别利用所述训练集和所述测试集对所述第一神经网络模型、所述第二神经网络模型和所述分类器进行训练和测试;
57.基于测试结果调整所述第一神经网络模型、所述第二神经网络模型和所述分类器
的参数,直至达到预设的精度要求,以得到所述已训练的第一神经网络模型、第二神经网络模型以及分类器。
58.第三方面,本发明还提供一种电子设备,包括存储器、处理器及存储在存储器上并可在处理器上运行的计算机程序,所述处理器执行所述程序时实现上述第一方面任一种所述病变dna识别方法的步骤。
59.第四方面,本发明还提供一种非暂态计算机可读存储介质,其上存储有计算机程序,该计算机程序被处理器执行时实现上述第一方面任一种所述病变dna识别方法的步骤。
60.本发明提供的一种病变dna识别方法、装置、电子设备及存储介质,采用预训练的第一神经网络模型,可避免从零开始构建模型并节省训练时间和计算资源,并通过结合第一神经模型和第二神经网络模型的病变dna识别方法,可提高病变dna识别的准确率。
附图说明
61.为了更清楚地说明本发明或现有技术中的技术方案,下面将对实施例或现有技术描述中所需要使用的附图作一简单地介绍,显而易见地,下面描述中的附图是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的实施例。
62.图1是本发明实施例提供的病变dna识别方法的流程示意图;
63.图2是本发明实施例提供的病变dna识别的示意图;
64.图3是本发明实施例提供的训练神经网络模型的流程图;
65.图4是本发明实施例提供的注意力处理的示意图;
66.图5是本发明实施例提供的长短期记忆网络的示意图;
67.图6是本发明提供的病变dna识别装置的结构示意图;
68.图7是本发明提供的电子设备的结构示意图。
具体实施方式
69.为使本发明的目的、技术方案和优点更加清楚,下面将结合本发明中的附图,对本发明中的技术方案进行清楚、完整地描述,显然,所描述的实施例是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有作出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
70.本发明的说明书和权利要求书及上述附图中的术语“第一”、“第二”等是用于区别类似的对象,而不必用于描述特定的顺序或先后次序。应该理解这样使用的数据在适当情况下可以互换,以便这里描述的实施例能够以除了在这里图示或描述的内容以外的顺序实施。
71.以下对本发明涉及的技术背景进行描述:
72.病变dna(ctdna)在体液无细胞dna(cfdna)中的比例被证明与病变负荷一致,因此识别出cfdna中的ctdna可以成为估计病变存在和严重程度的一种有效方法。
73.虽然近年来许多研究将深度学习应用于dna序列中,包括卷积神经网络(cnn)的架构、循环神经网络(rnn)的模型,例如,长短期记忆网络(lstm)和门控循环单元(gru)网络。然而cnn通常是无法在远程上下文中捕获语义依赖性,因为它具有提取本地特征受过滤器
大小的限制。而rnn模型(lstm、gru),虽然能够长期学习依赖处理所有过去的状态并将上下文信息压缩为具有长输入的瓶颈序列,但在顺序执行时会遇到梯度消失和效率低的问题。此外,大多数现有模型都需要大量标注数据,导致在数据稀缺场景中的性能和适用性有限,其中带有标签的高质量数据获得昂贵且耗时。
74.基于此,本发明提供一种病变dna识别方法、装置、电子设备及存储介质,用以解决现有技术中对病变dna序列的识别能力较差、要求大量标注数据的问题。
75.本发明采用预训练的第一神经网络模型,可避免从零开始构建模型并节省训练时间和计算资源,通过结合第一神经模型和第二神经网络模型的病变dna识别方法,可提高病变dna识别的准确率,同时本发明在只有少量标注数据的情况下,仍然能够对病变dna识别有较高的准确率。
76.下面结合图1-图7描述本发明的一种病变dna识别方法、装置、电子设备及存储介质。
77.图1是本发明实施例提供的病变dna识别方法的流程示意图,图2是本发明实施例提供的病变dna识别的示意图,如图1、图2所示。本发明实施例提供一种病变dna识别方法,所述方法包括:
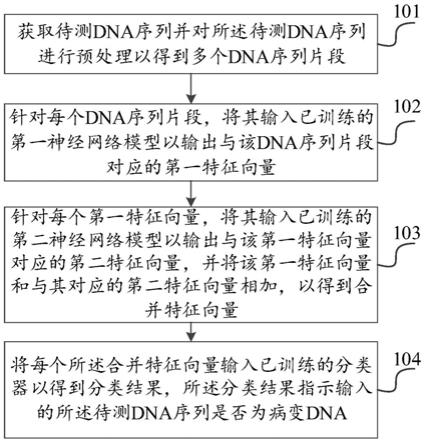
78.步骤101,获取待测dna序列并对所述待测dna序列进行预处理以得到多个dna序列片段。
79.需要说明的是,如果所述待测dna序列比较短(小于512个碱基的长度),那么不需要对所述dna序列进行预处理,即不需要切断成多个dna序列片段。如果所述待测dna序列比较长(例如大于512个碱基的),那么就需要对所述dna序列进行预处理,即需要以512个碱基为单位切断成多个dna序列片段。
80.步骤102,针对每个dna序列片段,将其输入已训练的第一神经网络模型以输出与该dna序列片段对应的第一特征向量,所述第一特征向量包含所述dna序列片段中的不同部分之间的自相关信息。
81.示例性地,所步骤102包括:
82.步骤1021,对该dna序列片段进行嵌入编码处理以得到具有预设的固定维度的第一编码向量、第二编码向量、以及第三编码向量,所述第一编码向量包含该dna序列片段的内容编码信息,所述第二编码向量包含该dna序列片段的位置编码信息,所述第三编码向量包含分段编码信息。
83.步骤1022,将所述第一编码向量、第二编码向量、以及第三编码向量相加以得到合并编码向量。
84.具体地,输入向量为对所述dna序列中的每个dna序列片段中的每个碱基生成词嵌入向量和片段嵌入向量,对词嵌入向量和片段嵌入向量求和得到输入向量,通过位置编码得到每个碱基在dna序列中的位置嵌入向量,将位置嵌入向量和输入向量求和,得到整个dna序列片段中每个碱基对应的碱基向量(即编码向量)。
85.步骤1023,对所述合并编码向量进行自注意力处理以得到对应的自注意力向量。
86.步骤1024,对所述自注意力向量进行归一化以及残差处理以得到该dna序列片段对应的所述第一特征向量。
87.具体地,对所述碱基向量进行自注意力处理后再进行相加与标准化处理,这里的
相加是将输入注意力前的碱基向量和注意力处理后得到的向量相加,然后将相加后得到的向量进行标准化,这里的标准化是指层标准化,最后通过前馈网络传输,再进行前馈网络传输的前后向量相加与标准化处理,得到所述第一特征向量。
88.步骤103,针对每个第一特征向量,将其输入已训练的第二神经网络模型以输出与该第一特征向量对应的第二特征向量,并将该第一特征向量和与其对应的第二特征向量相加,以得到合并特征向量,其中,所述第二特征向量包含所述第一特征向量中的不同部分之间的长短期依赖关系信息。
89.示例性地,所述步骤103包括:
90.步骤1031,将该第一特征向量作为初始向量输入所述已训练的第二神经网络模型。
91.步骤1032,所述已训练的第二神经网络模型中的每个神经单元执行如下操作:
92.对接收到的所述初始向量或者上一级神经单元输出的向量按照预设规则进行过滤处理,以丢弃部分元素。
93.对经过滤的向量进行增补处理,以按照预设规则挑选本单元中存储的信息替换所丢弃的元素。
94.基于经增补的向量确定该神经单元的输出向量。
95.其中,所述已训练的第二神经网络模型中的最后一级神经单元输出所述第二特征向量。
96.步骤104,将每个所述合并特征向量输入已训练的分类器以得到分类结果,所述分类结果指示输入的所述待测dna序列是否为病变dna。
97.示例性地,所述第一神经网络模型是基于转换器的双向编码表征模型(bidirectional encoder representations from transformers,简称bert),所述第二网络模型是长短期记忆网络模型(long short-term memory,简称lstm)。
98.在进行上述步骤对待测dna序列进行预测之前,需要对上述第一神经网络模型、所述第二神经网络模型以及所述分类器进行训练,以下针对上述第一神经网络模型、第二神经网络模型以及分类器进行训练和测试进行具体描述。
99.图3是本发明实施例提供的训练神经网络模型的流程图,如图3所示。所述在获取待测dna序列之前对所述第一神经网络模型、所述第二神经网络模型以及所述分类器按照如下方式进行训练:
100.步骤301,收集用于训练的数据集,所述数据集包括已标注的病变dna序列和非病变dna序列。
101.步骤302,将所述数据集按照预设比例分为训练集和测试集,并分别利用所述训练集和所述测试集对所述第一神经网络模型、所述第二神经网络模型和所述分类器进行训练和测试。
102.步骤303,基于测试结果调整所述第一神经网络模型、所述第二神经网络模型和所述分类器的参数,直至达到预设的精度要求,以得到所述已训练的第一神经网络模型、第二神经网络模型以及分类器。
103.以下对上述步骤301~303进行具体描述。
104.上述步骤301中,所述收集用于训练的数据集,所述数据集包括已标注的病变dna
序列和非病变dna序列,包括:
105.在所述步骤301之前,通过获取预设dna序列对第一神经网络模型进行预训练,得到初步训练过的第一神经网络模型,即对所述第一神经网络模型进行预训练。具体包括:
106.步骤3011,获取预设预设dna序列,所述预设dna序列为已公开的全基因组dna序列,并对所述预设dna序列进行预处理后得到多个dna序列片段。
107.示例性地,所述预设dna序列是人类的全基因组数据,约32亿个碱基组成的dna序列,所述dna序列可以包括腺嘌呤(a)、胸腺嘧啶(t)、胞嘧啶(c)、鸟嘌呤(g)、不同碱基上的修饰(如胞嘧啶的甲基化修饰,可以人为的用其他未使用过的字母表示)以及未知碱基(n)。
108.例如,某个dna序列片段为:
109.atcggcttcattggcg。
110.其中,将所述预设dna序列进行预处理的步骤包括:将序列中含有未知碱基n的片段删除,并将删除所述未知碱基n后得到的dna序列按照预设长度随机切分成多种长短不一的dna序列片段。
111.因此,将所述基因组数据进行预处理的目标是要删除dna序列中含有大片段的未知碱基n。
112.示例性,例如按照预设长度为512(表示512个碱基)的长度切分成多个dna序列片段,该预设的长度还可以是其他任意长度。
113.步骤3012,利用所述预设dna序列对所述第一神经网络模型进行预训练,得到初步训练过的第一神经网络模型。
114.示例性地,在对某dna序列片段进行嵌入编码处理之前,对该dna序列片段中的一部分碱基进行遮蔽(mask)处理,并且针对每个被遮蔽的碱基,基于该碱基的上下游序列预测该被遮蔽的碱基位置处的碱基,并用预测出的碱基替代所述被遮蔽的碱基。
115.示例性地,输入向量为对所述dna序列中的每个dna序列片段中的每个碱基生成词嵌入向量和片段嵌入向量,对词嵌入向量和片段嵌入向量求和得到输入向量,通过位置编码得到每个碱基在dna序列中的位置嵌入向量,将位置嵌入向量和输入向量求和,得到整个dna序列片段中每个碱基对应的碱基向量(如图2和图4所示)。
116.例如,dna序列片段“cls at c g g c t t c a t t g g c g sep”,cls表示一个dna序列片段的开始符号,sep表示一个dna序列片段的结束符号,一个碱基一个词,得到16个词。每个词用768维的向量表示,如a词用[0.001,0.123,0.234,
…
]向量表示,向量里面含有768个浮点数字组成。需要说明的是,多少个碱基一个词,是可以任意设置,比如设置两个碱基一个词、三个碱基一个词、5个碱基一个词。
[0117]
其中,对所述dna序列做embedding(嵌入,可以理解为“向量”)处理,采用了三个embedding相加的方式,包括token embeddings词嵌入、segment embeddings段嵌入以及position embeddings位置嵌入三个向量。token embeddings是对每个碱基做编码,position embeddings为位置编码,即在碱基向量中加入了碱基的位置信息,便于区分不同位置的碱基。segment embeddings都统一设置为0,即只有一条dna序列片段,表示每次只输入一条dna序列片段,例如每次只输入长度为512的一条dna序列片段。
[0118]
示例性地,对所述碱基向量进行自注意力处理后再进行相加与标准化处理,这里的相加是将输入注意力前的碱基向量和注意力处理后得到的向量相加,然后将相加后得到
的向量进行标准化,这里的标准化是指层标准化,最后通过前馈网络传输,再进行前馈网络传输的前后向量相加与标准化处理,得到预训练结果。
[0119]
其中,所述预训练结果包括词向量(即第一特征向量)、神经网络每一层的输出以及注意力分数,所述词向量表征词与词之间的关系以及每一个词与所述dna序列的关系,每一个词由预设个碱基构成。
[0120]
示例性地,自注意力处理流程如图4所示。放缩点积注意力机制包括对输入的query向量和key向量进行点积、放缩、屏蔽(可选操作)、归一化操作,以及将归一化操作的结果与value向量进行点积。所述注意力处理函数由下式表示:
[0121][0122]
其中,q,k,v的输入值均为碱基向量,dk表示调节因子,softmax表示归一化函数。
[0123]
基于上述注意力处理函数,对同一条dna序列片段的embedding向量进行线性变换得到query向量(即第一编码向量),key向量(即第二编码向量),value向量(即第三编码向量),之后计算注意力分数(注意力分数为上述函数的attention(q,k,v))。具体操作是每个碱基向量都要分别与三个权重矩阵wq,wk,wv线性相乘,生成每个碱基对应的query(q),key(k),value(v)三个向量。
[0124]
例如:query(q)=embedding*wq;key(k)=embedding*wq;value(v)=embedding*wq。
[0125]
所述第一神经网络模型的自注意力机制将dna序列中的某个碱基对序列中的所有碱基做一次注意力(attention)处理,即用这个碱基的query向量q与每个碱基的key向量k做点积。再通过softmax函数归一化出权重,点乘value向量v得到所有碱基的加权和,算出每个碱基对于这个碱基的相对权重,最终作为这个碱基的输出向量。
[0126]
所述第一神经网络模型的前馈网络会将经过attention之后输出的所有不同碱基词的向量都分别输入到一个全连接层中,完成前馈网络传播。
[0127]
需要说明的是,第一神经网络模型的相加与标准化处理是一残差网络,其会将该层的输入与其标准化后的输出进行相加。此外,第一神经网络模型还引入了多头注意力机制(multi-head attention),多头注意力机制相当于多个attention集成(例如用n个attention),再将n个输出进行合并(concat)得到最终的输出,从而提高算法的效果和稳定性。通过第一神经网络模型的自注意力(self-attention)机制得到dna序列中每个碱基与其他碱基的对应关系,即对应的全局dna序列语义信息的向量表示,第一神经网络模型采用self-attention机制克服了rnn无法并行、运行慢的缺点,同时通过masklm(mask language model简称mask lm,即基于mask机制的预训练语言模型)来达到训练深度双向预训练模型,使dna序列信息量更大,且dna语义理解更准确。训练完所述第一神经网络模型,得到预训练结果,并保存所述预训练结果对应的模型参数。
[0128]
示例性地,所述模型参数主要是神经网络里面的每一层的一些权重w、偏置b以及超参数(神经网络的层数和神经元的个数)。
[0129]
例如,如果需要对输入的向量(embedding,也可称为“嵌入”)做线性变换,则会用到线性的公式,y=wx b,x就是输入的embedding向量,w和b则是参数。
[0130]
示例性地,所述第一神经网络模型为bert模型,所述第二神经网络模型为长短期记忆网络模型(long short-term memory,简称lstm)。lstm是一种特殊的rnn,主要是为了解决长序列训练过程中的梯度消失和梯度爆炸问题。简单来说,就是相比普通的rnn,lstm能够在更长的序列中有更好的表现。
[0131]
其中,bert模型的全称为bidirectional encoder representations from transformers。所述bert模型为12层网络结构,其隐藏层为768维,采用12头模式。bert模型具有以下优点:
[0132]
第一、能够采用mlm(masked language model)对双向的transformers(其是一个seq2seq模型,由编码器和解码器两部分组成)进行预训练,以生成深层的双向语言表征。
[0133]
第二、经预训练后bert模型,只需要添加一个额外的输出层进行微调(fine-tuning),就可以在各种各样的下游任务中取得更好的表现,在这过程中并不需要对bert模型进行任务特定的结构修改。
[0134]
由此可知,本发明所述第一神经网络模型采用bert模型的transformer特征提取器,代替了传统常用的cnn或者rnn的固有模式,利用其能够提取dna序列的动态特征、序列结构特征以及其本身并行计算的优点,此外由于其多头自注意力机制,可以获得相关性更强的dna序列特征和dna上下文信息。然后将训练得到的词向量和lstm输出的特征向量求和形成残差结构,接着把求和得到的结果向量,通过全连接层结合softmax分类器得到不同种类的概率,实现dna序列的分类。本发明通过所述第一神经网络模型和所述第二神经网络模型的结合应用,能够提高dna分类的准确性和稳定性,具有良好的实施性和实效性。
[0135]
示例性地,所述dna序列是通过提取组织或体液中的dna,并通过一代或二代测序的方法得到的dna序列。例如针对肝癌而言,来源于肝癌组织的dna序列标签为1,来源于非肝癌组织的dna序列标签为0。肿瘤和非病变dna序列的长度可设为的范围例如100-512,表示100到512个碱基。需要说明的是,肝癌组织不纯,也存在非肝癌细胞,所以需要对肝癌组织的dna序列进行过滤,去除掉和非肝癌组织的dna一样的序列。
[0136]
此外,本发明实施例所述病变dna检测方法,可用于识别各种肿瘤的疾病,例如肝癌、肺癌等,该检测方法也可用于识别非肿瘤病变,如肝炎、肝硬化等。如果需要检测某一种疾病,则对应输入该病变的dna序列以及对应的非病变dna序列即可。另外,本发明实施例的识别结果是二分类结果,即是病变dna,或非病变dna。
[0137]
上述步骤302中,将所述数据集按照预设比例分为训练集和测试集,并分别利用所述训练集和所述测试集对所述第一神经网络模型、所述第二神经网络模型和所述分类器进行训练和测试。
[0138]
示例性地,可按照8:2的比例分为训练集和测试集。可以理解的是,也可以按照其它比例进行划分,例如6:4。本发明对于所述预设比例不做限制,具体可根据实际情况设定。
[0139]
示例性地,所述分别利用所述训练集和所述测试集对所述第一神经网络模型、所述第二神经网络模型和所述分类器进行训练和测试的步骤包括:
[0140]
步骤3021,分别将所述训练集和所述测试集输入至所述第一神经网络模型,输出所述词向量(即第一特征向量)。
[0141]
示例性地,所述词向量是通过所述第一神经网络模型学习到的向量,其表征词与词之间的关系以及每一个词与所述dna序列的关系,每一个词由预设个碱基构成,例如一个
词表示一个向量,每一个词由一个或多个碱基构成。
[0142]
步骤3022,将所述词向量输入至所述第二神经网络模型,输出特征向量(即第二特征向量),所述特征向量表征学习到病变dna与非病变dna的序列特征。
[0143]
示例性地,将第一神经网络模型(例如bert模型)与第二神经网络模型(例如lstm模型)连接的方式是:
[0144]
通过使用bert模型的get_sequence_output()方法得到序列的词向量,即bert模型输出的词向量作为lstm模型的输入。
[0145]
图5是本发明实施例提供的长短期记忆网络的示意图,如图5所示。经过lstm模型的遗忘门、输入门、输出门处理。其中,遗忘门用于决定对上一个单元输出的信息进行保留还是丢弃;记忆门用于确定什么样的新信息被存放在该单元模块(即神经单元)中。记忆门主要是针对遗忘门中丢弃的属性信息,在本单元模块找到相应的新的属性信息,添加进去,以补充丢弃的属性信息。根据本单元模块信息,确定输出值。通过遗忘门、输入门、输出门对数据进行过滤、筛选和更新,提取到更高维、有效的dna序列特征。
[0146]
示例性地,图5中c
t-1
表示上一时刻的单元状态(上一时刻可以理解为上一个词)。h
t-1
表示上一时刻lstm的输出值。x
t
表示当前时刻网络的输入值(当前时刻可以理解为当前的词)。c
t
表示当前时刻的单元状态。ht表示当前时刻lstm的输出值。σ表示sigmoid函数,公式为tanh是tanh激活函数,公式为f
t
表示遗忘门,i
t
表示记忆门,o
t
表示输出门,表示经过tanh激活函数变换后的值。
[0147]
示例性地,例如第二神经网络模型输出16*768维的特征向量(即第二特征向量),每个词用768维向量表示。
[0148]
步骤3203,将所述词向量(即第一特征向量)和所述特征向量(即第二特征向量)相加,以得到合并特征向量,将所述合并特征向量输入至所述分类器,最后输出是病变dna,或非病变dna的测试结果。
[0149]
示例性地,同时将bert模型输出的词向量和lstm模型输出的特征向量进行相加形成残差结构,该残差结构能防止梯度爆炸和消失。最后将相加得到的结果向量输入到分类器中,进行softmax分类,最终输出层可以得到是否为病变dna的二分类结果。
[0150]
示例性地,输出的二分类结构是2维向量,每个数字的取值范围维[0,1],两个数字的和为1,例如输出的二分类结果为0.23(表示非病变dna的概率)和0.77(表示病变dna的概率),这个结果说明,该dna序列更可能为病变dna。
[0151]
上述步骤303中,基于测试结果调整所述第一神经网络模型、所述第二神经网络模型和所述分类器的参数,直至达到预设的精度要求,以得到所述已训练的第一神经网络模型、第二神经网络模型以及分类器包括:
[0152]
步骤3031,对所述测试结果进行判断。
[0153]
示例性地,在训练所述第一神经网络模型、所述第二神经网络模型和所述分类器的过程中,通过梯度下降法和优化交叉熵损失函数训练所述第一神经网络模型、所述第二神经网络模型以及所述分类器,直至达到所述预设的精度要求。
[0154]
示例性地,所述交叉熵损失函数的表达式为:
[0155][0156]
其中,n表示训练样本总数,即病变dna和非病变dna的总和,y(i)表示第i个样本的真实标签(0或1),表示神经网络预测的第i个样本的预测概率,预测的概率在[0,1]的范围,表示从第1个样本到第n个样本求和。
[0157]
示例性地,通过不断降低损失函数来优化神经网络的参数。例如停止训练第一神经网络模型、第二神经网络模型以及分类器的条件包括:第一,通过设置训练总轮数,例如设置500轮、1000轮、或者10000轮。第二、观察损失函数。例如前5轮和后5轮的平均损失函数差值小于0.01的时候,可以停止训练;或者设置连续5轮,如果损失函数没有下降,那么也可以停止训练。本发明停止训练的条件不限于上述两种方式。训练完成之后保存所述第一神经网络模型、所述第二神经网络模型以及所述分类器的相关参数。
[0158]
步骤3032,如果所述测试结果未达到所述预设精度,则调整所述第一神经网络模型、所述第二神经网络模型以及分类器的模型参数,并继续对所述第一神经网络模型、第二神经网络模型以及分类器进行训练和测试。
[0159]
步骤3033,如果所述测试结果达到所述预设精度,则停止训练,得到训练完成的第一神经网络模型、第二神经网络模型以及分类器。
[0160]
示例性地,得到所述已训练的第一神经网络模型、第二神经网络模型以及分类器之后,根据预设评价指标构建目标函数对所述第一神经网络模型、所述第二神经网络模型和所述分类器的训练结果进行评价;
[0161]
其中,所述与预设评价指标包括下列中的任意组合:精确率、召回率以及准确率。
[0162]
由于本发明实施例的检测结果是分类问题,而分类问题可通过评价指标(包括精确率precision、召回率recall以及准确率accuracy)进行评价。精确率p是指分类器预测为正且预测正确的样本占所有预测为正的样本的比例,召回率r是指分类器预测为正且预测正确的样本占所有真实为正的样本的比例。accuracy是所以样本中(包括正样本和负样本),预测准确的比例。例如:
[0163]
负样本:非病变dna
[0164]
正样本:病变dna
[0165]
fn:false negative,被识别为负样本,但事实上是正样本。
[0166]
fp:false positive,被识别为正样本,但事实上是负样本。
[0167]
tn:true negative,被识别为负样本,事实上也是负样本。
[0168]
tp:true positive,被识别为正样本,事实上也是证样本。
[0169]
准确率(accuracy)的公式是accuracy=(tp tn)/(tp fp fn tn);
[0170]
定义:对于给定的测试数据集,分类器正确分类的样本数与总样本数之比。准确率越高越好。
[0171]
精确率(precision)的公式是precision=tp/(tp fp);
[0172]
定义:预测为正样本中有多少是真正的正样本。精确率越高越好。
[0173]
召回率(recall)的公式是recall=tp/(tp fn);
[0174]
定义:样本中的正样本有多少被预测正确了。召回率越高越好。
[0175]
示例性地,利用训练完成的第一神经网络模型、第二神经网络模型以及分类器对待测dna序列进行预测。在预测阶段的模型参数与训练阶段中最优的模型参数是一样,保持不变。然后将获取未标注的待测dna序列,通过训练好的第一神经网络模型将所述待测dna序列转化为第一特征向量的形式,送入训练好的第二神经网络模型输出第二特征向量,然后将所述第一特征向量和所述第二特征向量相加输入至训练好的分类器,得到预测结果,所述预测结果是病变dna的概率和非病变dna的概率。
[0176]
需要说明的是,本发明实施例提供的病变dna识别方法,可适用于基于dna序列识别的各种癌症早筛领域。例如通过识别出血浆中的病变dna对各种实体瘤和血液癌症的早期检测,通过识别大脑中脑脊液的病变dna对脑癌的早期筛查,可通过识别尿液中的病变dna对泌尿系统的早期癌症筛查等。该方法也可适用于基于dna序列识别的非癌症领域,例如肝炎、肝硬化等。通过识别血浆中的肝硬化病变dna对肝硬化进行早期检测。
[0177]
综上所述,本发明实施例提供一种结合第一神经网络模型和第二神经网络模型的病变dna识别方法,使用第一神经网络模型(例如bert预训练语言模型)对dna序列进行序列层面的第一特征向量表示,将获得的第一特征向量输入到第二神经网络模型(例如lstm模型)做进一步获取高层语义特征,输出第二特征向量。同时将第一神经网络模型输出的第一特征向量和第二神经网络模型输出的第二特征向量做跳跃连接,形成残差结构,防止梯度消失或者梯度爆炸,使得第二神经网络模型能更加准确地理解dna序列的信息,提高病变dna识别的准确率。
[0178]
并且,本发明实施例在公共数据集上进行了实验,通过多次训练、多次测试的方法,不仅提高了模型的鲁棒性,而且增强了对病变dna序列的识别能力。结果表明本发明实施例提出的病变dna识别模型的准确率和召回率都有较大提升。本发明实施例还用到了预训练模型(即第一神经网络模型)的思想,避免从零开始构建模型,通过微调(fine-tune)第一神经网络模型可大量地节省训练时间和计算资源,进一步提高检测效率。
[0179]
因此,本发明实施例提出的病变dna识别模型与传统的cnn和rnn模型通过实验对比,在公共数据集的实验结果表明本发明实施例提出的病变dna识别方法的准确率,精确率和召回率更高。
[0180]
下面对本发明提供的病变dna识别装置进行描述,下文描述的病变dna识别装置与上文描述的病变dna识别方法可相互对应参照。
[0181]
图6是本发明提供的病变dna识别装置的结构示意图,如图6所示。本发明实施例提供的一种病变dna识别装置500,所述装置包括预处理模块501、第一特征向量生成模块502、第二特征向量生成模块503以及分类模块504。其中,
[0182]
预处理模块501,用于获取待测dna序列并对所述待测dna序列进行预处理以得到多个dna序列片段。
[0183]
第一特征向量生成模块502,用于针对每个dna序列片段,将其输入已训练的第一神经网络模型以输出与该dna序列片段对应的第一特征向量,所述第一特征向量包含所述dna序列片段中的不同部分之间的自相关信息。
[0184]
第二特征向量生成模块503,用于针对每个第一特征向量,将其输入已训练的第二
神经网络模型以输出与该第一特征向量对应的第二特征向量,并将该第一特征向量和与其对应的第二特征向量相加,以得到合并特征向量,其中,所述第二特征向量包含所述第一特征向量中的不同部分之间的长短期依赖关系信息。
[0185]
分类模块504,用于将每个所述合并特征向量输入已训练的分类器以得到分类结果,所述分类结果指示输入的所述待测dna序列是否为病变dna。
[0186]
示例性地,所述病变dna识别装置,还包括:
[0187]
训练模块505(图6中未标示),所述训练模块505用于:
[0188]
收集用于训练的数据集,所述数据集包括已标注的病变dna序列和非病变dna序列;
[0189]
将所述数据集按照预设比例分为训练集和测试集,并分别利用所述训练集和所述测试集对所述第一神经网络模型、所述第二神经网络模型和所述分类器进行训练和测试;
[0190]
基于测试结果调整所述第一神经网络模型、所述第二神经网络模型和所述分类器的参数,直至达到预设的精度要求,以得到所述已训练的第一神经网络模型、第二神经网络模型以及分类器。
[0191]
示例性地,所述第一特征向量生成模块502,还用于:
[0192]
对该dna序列片段进行嵌入编码处理以得到具有预设的固定维度的第一编码向量、第二编码向量、以及第三编码向量,所述第一编码向量包含该dna序列片段的内容编码信息,所述第二编码向量包含该dna序列片段的位置编码信息,所述第三编码向量包含分段编码信息;
[0193]
将所述第一编码向量、第二编码向量、以及第三编码向量相加以得到合并编码向量;
[0194]
对所述合并编码向量进行自注意力处理以得到对应的自注意力向量;
[0195]
对所述自注意力向量进行归一化以及残差处理以得到该dna序列片段对应的所述第一特征向量。
[0196]
示例性地,第二特征向量生成模块503,还用于:
[0197]
将该第一特征向量作为初始向量输入所述已训练的第二神经网络模型;
[0198]
所述已训练的第二神经网络模型中的每个神经单元执行如下操作:
[0199]
对接收到的所述初始向量或者上一级神经单元输出的向量按照预设规则进行过滤处理,以丢弃部分元素;
[0200]
对经过滤的向量进行增补处理,以按照预设规则挑选本单元中存储的信息替换所丢弃的元素;
[0201]
基于经增补的向量确定该神经单元的输出向量;
[0202]
其中,所述已训练的第二神经网络模型中的最后一级神经单元输出所述第二特征向量。
[0203]
示例性地,所述训练模块505,还用于:
[0204]
获取预设dna序列,所述预设dna序列为已公开的全基因组dna序列;
[0205]
对所述预设dna序列进行预处理后得到多个dna序列片段;
[0206]
利用所述dna序列片段对所述第一神经网络模型进行预训练。
[0207]
示例性地,所述训练模块505,还用于:
[0208]
将所述dna序列中含有未知碱基的片段删除;以及
[0209]
将删除所述未知碱基后得到的dna序列按照预设长度切分成所述多种长短不一的dna序列片段。
[0210]
示例性地,所述训练模块505,还用于:
[0211]
在对该dna序列片段进行嵌入编码处理之前,对该dna序列片段中的一部分碱基进行遮蔽处理,并且针对每个被遮蔽的碱基,基于该碱基的上下游序列预测该被遮蔽的碱基位置处的碱基,并用预测出的碱基替代所述被遮蔽的碱基。
[0212]
示例性地,所述训练模块505,还用于:
[0213]
在训练所述第一神经网络模型、所述第二神经网络模型和所述分类器的过程中,通过梯度下降法和优化交叉熵损失函数训练所述第一神经网络模型、所述第二神经网络模型以及所述分类器,直至达到所述预设的精度要求。
[0214]
示例性地,所述训练模块505,还用于:
[0215]
根据预设评价指标构建目标函数对所述第一神经网络模型、所述第二神经网络模型和所述分类器的训练结果进行评价;
[0216]
其中,所述与预设评价指标包括下列中的任意组合:精确率、召回率以及准确率。
[0217]
示例性地,所述训练模块505,还用于:
[0218]
根据下列各式计算所述精确率、召回率以及准确率:
[0219]
精确率:tp/(tp fp);
[0220]
召回率:tp/(tp fn);
[0221]
准确率:(tp tn)/(tp fp fn tn);
[0222]
其中,fn表示被识别为非病变dna,但是事实上是病变dna;fp表示被识别为包含病变dna,但事实上非病变dna;tn表示被识别为非病变dna片段,事实上也是非病变dna;tp表示被识别为病变dna,事实上也是病变dna。
[0223]
示例性地,所述第一神经网络模型是基于转换器的双向编码表征模型,所述第二网络模型是长短期记忆网络模型。
[0224]
示例性地,所述基于转换器的双向编码表征模型是基于多头自注意力机制的神经网络模型,并且其具有12层网络结构,以及其隐藏层具有768维度。
[0225]
图7示例了一种电子设备的实体结构示意图,如图7所示,该电子设备可以包括:处理器(processor)610、通信接口(communications interface)620、存储器(memory)630和通信总线640,其中,处理器610,通信接口620,存储器630通过通信总线640完成相互间的通信。处理器610可以调用存储器630中的逻辑指令,以执行病变dna识别方法,该方法包括:
[0226]
获取待测dna序列并对所述待测dna序列进行预处理以得到多个dna序列片段;
[0227]
针对每个dna序列片段,将其输入已训练的第一神经网络模型以输出与该dna序列片段对应的第一特征向量,所述第一特征向量包含所述dna序列片段中的不同部分之间的自相关信息;
[0228]
针对每个第一特征向量,将其输入已训练的第二神经网络模型以输出与该第一特征向量对应的第二特征向量,并将该第一特征向量和与其对应的第二特征向量相加,以得到合并特征向量,其中,所述第二特征向量包含所述第一特征向量中的不同部分之间的长短期依赖关系信息;
[0229]
将每个所述合并特征向量输入已训练的分类器以得到分类结果,所述分类结果指示输入的所述待测dna序列是否为病变dna。
[0230]
此外,上述的存储器630中的逻辑指令可以通过软件功能单元的形式实现并作为独立的产品销售或使用时,可以存储在一个计算机可读取存储介质中。基于这样的理解,本发明的技术方案本质上或者说对现有技术做出贡献的部分或者该技术方案的部分可以以软件产品的形式体现出来,该计算机软件产品存储在一个存储介质中,包括若干指令用以使得一台计算机设备(可以是个人计算机,服务器,或者网络设备等)执行本发明各个实施例所述方法的全部或部分步骤。而前述的存储介质包括:u盘、移动硬盘、只读存储器(rom,read-only memory)、随机存取存储器(ram,random access memory)、磁碟或者光盘等各种可以存储程序代码的介质。
[0231]
另一方面,本发明还提供一种计算机程序产品,所述计算机程序产品包括存储在非暂态计算机可读存储介质上的计算机程序,所述计算机程序包括程序指令,当所述程序指令被计算机执行时,计算机能够执行上述各方法所提供的病变dna识别方法。
[0232]
又一方面,本发明还提供一种非暂态计算机可读存储介质,其上存储有计算机程序,该计算机程序被处理器执行时实现以执行上述各提供的病变dna识别方法。
[0233]
以上所描述的装置实施例仅仅是示意性的,其中所述作为分离部件说明的单元可以是或者也可以不是物理上分开的,作为单元显示的部件可以是或者也可以不是物理单元,即可以位于一个地方,或者也可以分布到多个网络单元上。可以根据实际的需要选择其中的部分或者全部模块来实现本实施例方案的目的。本领域普通技术人员在不付出创造性的劳动的情况下,即可以理解并实施。
[0234]
通过以上的实施方式的描述,本领域的技术人员可以清楚地了解到各实施方式可借助软件加必需的通用硬件平台的方式来实现,当然也可以通过硬件。基于这样的理解,上述技术方案本质上或者说对现有技术做出贡献的部分可以以软件产品的形式体现出来,该计算机软件产品可以存储在计算机可读存储介质中,如rom/ram、磁碟、光盘等,包括若干指令用以使得一台计算机设备(可以是个人计算机,服务器,或者网络设备等)执行各个实施例或者实施例的某些部分所述的方法。
[0235]
最后应说明的是:以上实施例仅用以说明本发明的技术方案,而非对其限制;尽管参照前述实施例对本发明进行了详细的说明,本领域的普通技术人员应当理解:其依然可以对前述各实施例所记载的技术方案进行修改,或者对其中部分技术特征进行等同替换;而这些修改或者替换,并不使相应技术方案的本质脱离本发明各实施例技术方案的精神和范围。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。