一种用于油莎豆基因分型的snp分子标记组合及其应用
技术领域
1.本发明涉及分子生物学和分子植物育种领域,尤其涉及一种用于油莎豆基因分型的snp分子标记组合及其应用。
背景技术:
2.油莎豆(cyperus esculentusl.)是一种生态高值、粮油饲兼用,极具竞争力的新型油料作物。目前国内种植的油莎豆多为上世纪六、七十年代从国外引进的品种资源。由于广泛引种繁育,油莎豆原有遗传基础已无法准确溯源,种质资源匮乏且混乱,严重制约了我国油莎豆产业的健康快速发展。当前生产上对油莎豆种质资源的鉴别主要是通过块茎形状、大小和颜色等表型方面的差异进行区分,准确度和可信度较差,难以进行准确鉴定和纯度分析。同时作物外观性状易受到环境因素的影响,在实际操作过程中也常常会导致鉴别错误。油莎豆形态特征的相似性和溯源不清,给准确鉴别和合理利用油莎豆资源,特别是油莎豆品种选育和品种权保护等造成极大的困难。进一步厘清国内油莎豆资源现状,加快油莎豆种质资源利用及优良品种选育已成为我国油莎豆产业发展中急需解决的关键问题。
3.与传统形态或生化标记鉴定方法不同,分子标记技术(如rapd、rflp、ssr、alfp、srap、snp等)能够从本质上反应生物个体的差异,具有受环境影响小、检测特异性、稳定性和准确性强等诸多优势,已广泛应用于生物多样性分析、种质资源或品种分类、遗传图谱构建、分子标记辅助选择育种、比较基因组学等领域的研究。由于油莎豆全基因组序列目前尚未公布,已有的研究主要是利用随机设计的rapd或srap引物进行检测,扩增片段特异性和稳定性较差,且不同分子标记间数据整合存在困难,因而并没有获得广泛的应用。与其他分子标记相比,snp标记(single nucleotide polymorphisms,单核苷酸多态性)具有覆盖全基因组、高通量、位点特异、共显性遗传、误检率低、数据易整合等优点,且部分标记与功能基因或植物表型相关,被国际新品种保护联盟作为首选的dna指纹标记技术之一。特别是随着测序技术的飞速发展,高通量、低成本测序技术为全基因组水平snp标记的检测提供了强有力的技术支持。现有技术并未发现采用snp标记手段对油莎豆种质资源鉴定的方案。
技术实现要素:
4.本发明的目的在于提供一种用于油莎豆基因分型的snp分子标记组合及其应用。
5.为了实现上述发明目的,本发明提供以下技术方案:
6.本发明提供了一种用于油莎豆基因分型的snp分子标记组合,所述分子标记组合至少包括1292个snp标记中的一个,所述1292个snp标记如表1所示:
7.表1 1292个snp标记
8.9.10.11.12.13.14.15.16.17.18.19.20.21.22.23.24.25.26.27.28.29.30.31.32.33.34.35.36.37.38.39.40.41.42.43.44.45.46.47.48.49.50.51.52.53.54.55.56.57.58.59.60.61.62.63.64.65.66.67.68.69.70.71.72.73.74.75.[0076][0077]
作为优选,所述snp分子标记组合至少包括1292个snp标记中的2个。
[0078]
作为优选,所述snp分子标记组合包括1292个snp标记。
[0079]
本发明还提供了所述的snp分子标记组合在油莎豆种质资源和品种基因型鉴定中的应用。
[0080]
本发明还提供了所述的snp分子标记组合在构建油莎豆dna指纹图谱中的应用。
[0081]
本发明的snp分子标记组合与现有技术相比具有如下优点:
[0082]
(1)与现有技术相比,本发明提供的1292个油莎豆snp标记具有覆盖率高、多态性丰富的优点。
[0083]
(2)与现有技术相比,本发明具有检测通量高、检测成本低等优势。
[0084]
(3)国内油莎豆种质资源和品种匮乏,现有资源溯源不清。本发明提供的snp标记或组合可以实现对油莎豆种质资源和品种的高通量检测,可广泛应用于油莎豆种质资源和品种鉴定、育种材料遗传背景分析、分子标记选择或全基因组选择育种等,具有较好的应用前景。
附图说明
[0085]
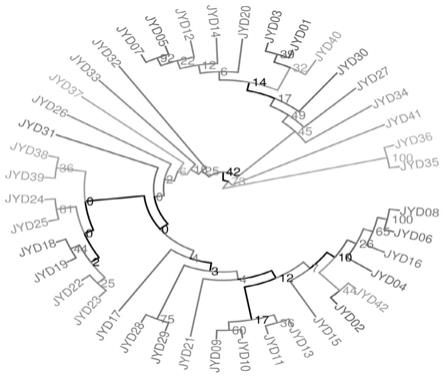
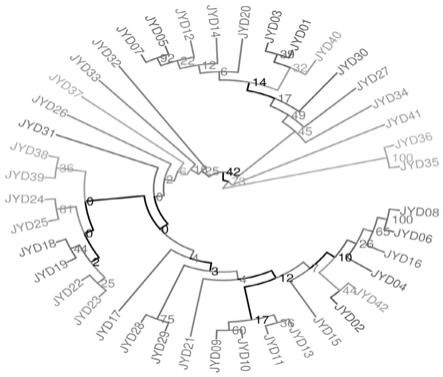
图1为本发明实施例2中利用super
‑
gbs技术对42份油莎豆测序获得的snp指纹图谱进行聚类分析的结果。
[0086]
图2为本发明实施例3中利用一代测序对42份油莎豆进行snp检测的结果。
具体实施方式
[0087]
下面结合实施例对本发明提供的技术方案进行详细说明,但是不能把它们理解为
对本发明保护范围的限定。若未特别指明,实施例均按照常规实验条件或按照制造厂商说明书建议的条件进行。
[0088]
本发明实施例中所述的42份油莎豆种质资源信息如表2所示。
[0089]
表2 42份油莎豆种质资源信息
[0090][0091][0092]
实施例1 油莎豆snp标记开发
[0093]
利用pacbio sequel ii单分子实时测序系统,对油莎豆jyd
‑
02进行de novo测序,
得到hifi reads。将hifi reads利用hifiasm软件(版本0.15.2)进行基因组组装,获得306,075,184bp的组装本。以该组装本作为参考基因组序列,对表2所述的42份油莎豆资源进行简化基因组测序(super
‑
gbs,illumina hiseq xten,pe150),得到测序样本。平均测序深度为39.3
×
,覆盖度范围为3.79%~4.99%。基于测序样本与参考基因组的比对结果,得到初步的snp和indel结果。利用vcftools软件对获得snp和indel分型结果进行过滤。过滤条件如下:(1)reads支持数(dp)不低于4;(2)剔除maf小于0.01的位点;(3)剔除snp或者indel分型缺失率高于20%的位点。共获得87398个snp位点和5009个indel位点。
[0094]
基于获得的snp位点,利用ugbs
‑
flex分析流程,对所有snp位点进行基因型转换。最低测序深度设为8,h/c/d鉴定阈值为4,缺失数据阈值为30%。从检测出的共分离snp标记中选取缺失信息最少的snp作为该组标记的代表,共筛选获得1292个snp。所述1292个油莎豆snp标记信息如表1所示。
[0095]
实施例2 1292个snp在油莎豆基因组dna指纹图谱分析中的应用
[0096]
根据实施例1中获得的1292个油莎豆snp分子标记组合,结合super
‑
gbs技术对表2的42份油莎豆资源的基因型进行检测。具体方法如下:
[0097]
(1)利用实施例1得到的参考基因组序列作为本实施例2的参考基因组序列;
[0098]
(2)利用super
‑
gbs技术构建油莎豆基因组dna测序文库,测序得到clean reads;
[0099]
(3)利用bowtie2软件将clean reads与参考基因组序列进行比对,比对结果利用gatk软件进行snp或indel检测,得到油莎豆snp位点;
[0100]
(4)基于所述的snp分子标记组合,利用ugbs
‑
flex对油莎豆snp位点进行基因型转换,得到油莎豆dna指纹图谱。
[0101]
所述基因组dna的提取方法采用ctab法;
[0102]
利用此方法构建表2中的42份油莎豆指纹图谱,结果如表3所示。
[0103]
表3中的snp编号中的1~1292分别代表cesnp0001~cesnp1292。
[0104]
对42份油莎豆种质资源进行聚类分析,结果如图1所示。图1显示国内油莎豆资源大致可以分为3类,其中,从非洲喀麦隆引进的2份种质资源(jyd
‑
35和jyd
‑
36)较为相近;来源于西班牙(jyd
‑
41)、喀麦隆(jyd
‑
34)及国内部分资源(如jyd
‑
14等)遗传上较为相近;而来源于俄罗斯(jyd
‑
42)、马里(jyd
‑
33)和多数国内资源(如jyd
‑
23等)则更为相近。这一结果与表型鉴定的结果较为吻合。本发明筛选的1292个油莎豆snp标记适用于油莎豆指纹图谱的建立,便于比较不同资源或品种之间的亲缘关系,适于不同油莎豆种质资源和品种的准确鉴定。
[0105]
表3 42份油莎豆指纹图谱
[0106]
[0107]
[0108]
[0109]
[0110]
[0111]
[0112]
[0113]
[0114]
[0115]
[0116]
[0117]
[0118]
[0119]
[0120]
[0121]
[0122]
[0123]
[0124]
[0125]
[0126]
[0127]
[0128]
[0129]
[0130]
[0131]
[0132]
[0133]
[0134]
[0135]
[0136]
[0137]
[0138]
[0139]
[0140]
[0141]
[0142]
[0143]
[0144]
[0145]
[0146]
[0147]
[0148]
[0149]
[0150]
[0151]
[0152]
[0153]
[0154]
[0155]
[0156]
[0157]
[0158]
[0159]
[0160]
[0161]
[0162]
[0163]
[0164]
[0165]
[0166]
[0167]
[0168]
[0169]
[0170]
[0171]
[0172]
[0173]
[0174]
[0175]
[0176]
[0177]
[0178]
[0179]
[0180]
[0181]
[0182]
[0183]
[0184]
[0185]
[0186]
[0187]
[0188]
[0189]
[0190]
[0191]
[0192]
[0193]
[0194]
[0195]
[0196]
[0197]
[0198]
[0199]
[0200]
[0201][0202]
实施例3 用于鉴定油莎豆种质资源的核心snp及应用
[0203]
基于实施例1中获得的油莎豆snp标记,计算其中纯合子的占比以及分离比,将纯合子占比高且分离比均一的位点挑选出作为核心snp位点。本发明从1292个snp中筛选73个核心snp标记,具体分子标记信息如表4所示。利用上述核心snp序列信息,设计pcr检测引物或kasp引物,对油莎豆基因组dna进行pcr扩增。采用一代测序(sanger法)或荧光检测,分析不同油莎豆资源snp位点变异情况。
[0204]
表4中所述的snp编号对应表5引物中的snp编号。
[0205]
利用一代测序对snp位点检测包括如下步骤。
[0206]
基于73个油莎豆核心snp标记信息,设计检测引物,具体引物序列如表5所示。
[0207]
表5.用于油莎豆核心snp检测的引物序列
[0208]
[0209][0210]
表4 73个核心snp
[0211]
[0212]
[0213]
[0214]
[0215]
[0216][0217]
利用ctab法提取油莎豆基因组dna。将提取得到的油莎豆基因组dna的浓度稀释至50ng/μl作为pcr扩增模板。
[0218]
pcr扩增及电泳检测:pcr扩增体系(25ul)为:10
×
pcr缓冲液2.5ul,10mmol/l dntps 0.5ul,5u/ul taq酶0.5ul,样品dna 1.0ul,上下游引物混合物(各10umol/l)1.0ul,ddh2o 19.5ul。pcr扩增程序为:95℃5min;94℃30s,60℃30s,72℃45s,共35个循环;72℃10min。pcr产物经1.5%琼脂糖凝胶电泳检测。
[0219]
snp检测:将电泳检测结果显示条带的pcr产物进行测序,并根据测序结果分析snp
位点变异情况。结果如图2所示。
[0220]
以上所述仅是本发明的优选实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。